Method Article
Kulturmethoden zur Bestimmung der Grenze der Detektion und des Überlebens in Transportmedien von Campylobacter Jejuni in humanen Fäkalproben
In diesem Artikel
Zusammenfassung
Obwohl die Hockerkultur für Campylobacter ungenau ist, gilt sie immer noch als Goldstandard für die Identifizierung. Methoden zur Bestimmung der Nachweisgrenze und des Überlebens in Transportmedien von C. jejuni im menschlichen Stuhl werden beschrieben und mit einem neuen Immunoassay mit besserer Genauigkeit verglichen.
Zusammenfassung
Eine Kultur aus dem menschlichen Stuhl für die Diagnose von Campylobacter-basierteDarmerkrankung dauert mehrere Tage, eine Wartezeit, die die Stärke des Arztes und des Patienten besteuert. Eine Kultur ist auch anfällig für falsche negative Ergebnisse durch zufällige Verlust der Lebensfähigkeit während der Handhabung von Proben, Überwucherung anderer Fäkalienflora und schlechtes Wachstum mehrerer pathogener Campylobacter-Arten in traditionellen Medien. Diese Probleme können klinische Entscheidungen über die Behandlung von Patienten verwirren und haben das Feld daran gehindert, grundlegende Fragen zum Campylobacter-Wachstum und zu Infektionen zu beantworten. Wir beschreiben ein Verfahren, das die untere Grenze der Bakterienzahlen schätzt, die durch eine Kultur und eine Methode zur Quantifizierung des Überlebens von C. jejuni in Medien, die für den Transport dieses fragilen Organismus verwendet werden, nachgewiesen werden kann. In Kenntnis dieser Informationen wird es möglich, klinisch relevante Nachweisschwellen für diagnostische Tests festzulegen und unerforschte Fragen zu behandeln, ob eine nichtsymptomatische Kolonisation weit verbreitet ist, ob eine Koinfektion mit anderen enterischen Krankheitserregern häufig ist oder ob die bakterielle Belastung mit Symptomen oder schwerwiegenden Folgen korreliert. Die Studie umfasste auch Tests von 1.552 prospektiv gesammelten Patientendurchfall-Fäkalproben, die zunächst durch konventionelle Kultur klassifiziert und durch einen neuen Enzym-Immunoassay weiter getestet wurden. Positive und diskrete Proben wurden dann mit vier molekularen Methoden abgeschirmt, um einen wahr-positiven oder wahr-negativen Status zuzuweisen. Die 5 nicht-kulturfreien Methoden zeigten eine vollständige Übereinstimmung bei allen 48 positiven und diskreten Exemplaren, während die Kultur 14 (28%) falsch identifizierte. Die Proben, die fälschlicherweise durch Kultur identifiziert wurden, enthielten 13 falsch negative und 1 falsch positive Probe. Dieses Grundlegende Protokoll kann mit mehreren Campylobacter spp. verwendet werden und wird es ermöglichen, die Anzahl der Campylobacter-Bakterien, die Symptome der Gastroenteritis beim Menschen produzieren, zu bestimmen und die Prävalenzraten zu aktualisieren.
Einleitung
Die United States Centers for Disease Control (CDC) veröffentlicht enthonte vor kurzem, dass die Foodborne Diseases Active Surveillance Network (FoodNet) Überwachungsprogramm berichtet 9.723 Fälle von Labor-diagnostizierten Campylobacter-Infektionen im Jahr 20181. Dies entspricht einem Anstieg der Fallberichte von Campylobacter um 12 % gegenüber 2015-20171. Weltweit gehören Campylobacter spp. zu den häufigsten bakteriellen Darminfektionen2. Dennoch wird die Zahl der Campylobacter-basiertenDarmerkrankungen, die jedes Jahr auftreten, als untergemeldet3. Diese Unterschätzung ist vorhersehbar, da sich die meisten Patienten mit nur mäßigen Beschwerden und ohne medizinische Behandlung erholen können. Jedoch, für Patienten mit schwereren Symptomen oder die ein höheres Risiko für schwere Krankheiten sind, und die dann medizinische Versorgung suchen, ist Stuhlkultur die häufigste Methode, um zu beurteilen, ob Campylobacter der Erreger ist, der ihre Not verursacht4.
Für Campylobacter spp. ist die Stuhlkultur besonders lästig. Die häufigsten pathogenen Organismen, C. jejuni, C. coli, C. upsaliensis und C. lari, sind mikroaerophil5. Dies bedeutet, dass die Bakterien nach dem Zufallsprinzip sterben, unbekannte Raten einmal der Luft ausgesetzt. Die Zeit zwischen Probensammlung und Kulturaufbau wird so zu einer unkontrollierten Variable in der Fähigkeit, lebensfähige Campylobacter spp. nach Kultur zu erkennen.
Für die direkte Kultur der Fäkalien ist auch das langsame Wachstum von Campylobacter ein Problem. Campylobacter-Kolonien sind auch nach 48 h Inkubation sehr klein und können leicht von konkurrierenden Organismen in der Fäkalienmatrix abgedeckt werden. Platten, die Antibiotika enthalten, gegen die die meisten Stämme von C. jejuni und C. coli resistent sind, sind weit verbreitet, da die Antibiotika das Wachstum vieler (aber nicht aller) konkurrierender Fäkalienbakterien hemmen und eine bessere Visualisierung der Campylobacter-Kolonien 6ermöglichen. Andere Campylobacter-Arten wie C. lari und C. upsaliensis reagieren jedoch empfindlich auf einige dieser Antibiotika und wachsen entweder schlecht oder gar nicht. Dies trägt zur Untermeldung von Campylobacter-Infektionen dieser antibiotikaempfindlichen Artenbei 7.
Es gibt einen dritten Grund, warum eine Kultur für Campylobacter ungenau sein kann. Die Bakterien, wenn sie gestresst sind, können lebensfähig bleiben, aber "nicht kultivierbar" werden8. Dies bedeutet per definitionem, dass die Kultur die in der Probe vorhandenen Bakterien nicht erkennt. Wie oft dies geschieht, ist nicht bekannt8.
Angesichts dieser potenziellen Probleme mit der Kultur haben wir mehrere Vergleichsreferenzmethoden verwendet, so dass fehlerhafte Kulturergebnisse keinen einzelnen Vergleichstest ungenau erscheinen ließen9. Die verwendeten Kulturmethoden (z.B. Campylobacter-selektivePlatten, Transportmedium, gaserzeugende Beutel) wurden gewählt, weil sie in klinischen Laboratorien für Stuhlprobenkultur10weit verbreitet sind.
Die hier beschriebenen Kulturprotokolle wurden entwickelt, weil die geringste Anzahl von Campylobacter jejuni, die von der Kultur im menschlichen Stuhl nachgewiesen werden konnten, nicht bekannt war. Obwohl Schätzungen für die Anzahl der Koloniebildenden Einheiten (KBE) in Geflügelkot11veröffentlicht wurden, können diese Ergebnisse nicht mit menschlichem Stuhl gleichgesetzt werden, da Campylobacter spp. Kommensale bei Hühnern sind und keinen Durchfall verursachen. Diese grundlegenden Informationen werden benötigt, um die Anzahl der Campylobacter-Bakterien zu ermitteln, die Symptome einer Gastroenteritis beim Menschen produzieren, und um Virulenz zwischen Stämmen oder Arten zu vergleichen.
Protokoll
1. Aufzählung von Campylobacter in zerknirschten menschlichen Fäkalproben
HINWEIS: Alle Schritte werden mit steriler Technik und Materialien auf einer Einwegschutzfolie in einer desinfizierten laminaren Strömungssicherung durchgeführt.
VORSICHT: Live Campylobacter sind ansteckend und können Krankheiten verursachen, einschließlich Durchfall. Tragen Sie Handschuhe, einen Labormantel und eine Schutzbrille, wenn Sie mit Bakterien umgehen. Nicht Mund Pipet. Entsorgen Sie alle Materialien, die Bakterien in geeigneten Biogefahrenbehältern kontaktiert haben.
- Wachstum der Bestandskultur von Bakterien
- Besorgen Sie sich Stämme von C. jejuni (ATCC-33560) oder C. coli (ATCC 33559) (Materialtabelle) als getrocknete oder gefrorene Kulturen und rehydrieren oder tauen Bakterien gemäß den Anweisungen des Herstellers. Streichen Sie die rehydrierten Bakterien auf eine Campylobacter-spezifischeAgarplatte, um die Kultur zu starten. Inkubieren Sie die Platte 48 h bei 37 °C in einem anaeroben Glas, das einen mikroaeroben, gaserzeugenden Beutel enthält.
- Am folgenden Tag 100 ml Gehirn-Herz-Infusion (BHI) Wachstumsbrühe mit 0,5% Trypticase, 0,5% Proteasepepton, 0,0125% Natriumpyruvat und 0,0125% Natriumbisulfit zubereiten.
- Reduzieren Sie die BHI-Brühe, indem Sie den Kolben locker bedecken und in ein anaerobes Glas mit einem Beutel legen, der eine mikroaerophile Umgebung erzeugt. Brühe bei 37 °C über Nacht vorreduzieren lassen. In ähnlicher Weise reduzieren Campylobacter-spezifischePlatten, die für Koloniezählungen in den Schritten 1.1.10 und 1.2.2 verwendet werden sollen.
- Da Campylobacter luftempfindlich sind, sammeln Sie alle Materialien vor der Inokulation Brühe und nicht beim Umgang mit Kulturen. Wenn Sie bereit sind zu impfen, fügen Sie fetales Rinderserum (FBS) zu vorreduzierter Brühe auf 4% des Gesamtvolumens hinzu. Bewahren Sie 1 ml vorreduzierte Brühe auf, um als Rohling bei Messungen der optischen Dichte bei 600 nm (OD600)zu dienen.
- Entfernen Sie 3 ml vorreduzierte Brühe, die FBS enthält, und verwenden Sie Brühe, um die Starterplatte mit der Campylobacter-Kultur zu kratzen. Kratzen Sie die Platte vorsichtig mit einer Impfschlaufe und übertragen Sie dann die bakterielle Gülle in ein steriles Rohr.
- Die 100 ml vorreduzierter Brühe mit ca. 3 ml bakteriellegülle brüten und mit mäßigem Schütteln bei 115 U/min bei 37 °C in einem anaeroben Glas mit einem gaserzeugenden Beutel brüten.
- Überwachen Sie das Wachstum der Bakterien spektrophotometrisch durch Trübung bei OD600. Verwenden Sie die reservierte Brühe als Leerzeichen. Wenn das anaerobe Glas geöffnet wird, ersetzen Sie den gaserzeugenden Beutel.
- Stoppen Sie die Brüheinkubation nach 48 x 72 h oder bevor der OD600-Wert 0,4 usd erreicht.
HINWEIS: Diese OD600 entspricht in der Regel 107 bis 108 KBE/ml. Typische Ergebnisse finden Sie in Tabelle 1. - Um die Anzahl der Bakterien in der reinen Bestandskultur zu ermitteln, führen Sie acht 10-fache Verdünnungsreihen von 100 l Brühe in 900 l Verdünnungspuffer(Tabelle der Materialien) durch. Nachdem die 100 l Brühe für die erste Verdünnung entfernt wurde, kehren Sie den Kolben in ein anaerobes Glas mit frischem gaserzeugenden Beutel zurück, um den Einsatz im Spiking-Fäkalienbecken abzuwarten.
- Verwenden Sie sterile Beschichtungsperlen, um 100 l der 10-5 bis 10-7 Verdünnungen auf doppelten vorreduzierten Campylobacter-spezifischenPlatten ab Schritt 1.1.3 zu verteilen. Etikettenplatten mit verwendeter Verdünnung, in ein zweites anaerobes Glas mit gaserzeugendem Beutel legen und bei 37 °C für 48 x 72 h brüten.
HINWEIS: Siehe Abbildung 1 und Abbildung 2 für Verdünnungsschema und Fotos von Kolonien. - Nach dem Wachstum, wählen Sie die Platte mit zwischen 30 bis 300 Kolonien zu zählen. Verwenden Sie die Zählungen, um die KBE/ml der Brühekultur mit Gleichung 1 zu bestimmen:
CFU/ml auf Lager = Durchschnitt der Kolonien auf ausgewählten (doppelten) analytischen Platten (mL plattiert x Verdünnung der Platte) [Gleichung 1]
- Vorbereitung und Aufzählung von erfundenen klinischen Fäkalienproben
- Unmittelbar nachdem die Platten für analytische Zählungen in Schritt 1.1.10 vorbereitet sind, machen Sie einen zweiten Satz von Brüheverdünnungen, indem Sie 10 serielle 2-fache Verdünnungen aus der Brühe und einem Campylobacter-negativenFäkalienpool (NFP) zubereiten. Bereiten Sie beispielsweise die erste Verdünnung vor, indem Sie gleiche Volumina von Brühe und NFP (z. B. 0,1 ml) mischen und nachfolgende Verdünnungen vornehmen, indem Sie ein bestimmtes Volumen von Brühe und NFP-Gemisch in ein Rohr mit einem gleich bezeichneten Volumen NFP übertragen. Fügen Sie eine Kontrollplatte mit Brühe ohne Campylobacter hinzu, die dem Fäkalpool hinzugefügt wurde, um nichtCampylobacter-Kolonien zu identifizieren.
- Machen Sie das NFP aus de-identifizierten, durchfallkranken Patientenüberwachungsproben oder gesunden Spenderhockern, die zuvor getestet wurden und sich als Campylobacter-negativerwiesen haben, indem Sie Methoden wie einen Campylobacter-Enzymimmunoassay und 16S rRNA qPCR verwenden.
- T-Streifen 10 l jeder Campylobacter/Hocker-Verdünnung auf doppelten vorreduzierten Campylobacter-spezifischenAgarplatten. Platten in das anaerobe Glas mit einem gaserzeugenden Beutel legen und bei 37 °C für 48 x 2 h inkubieren.
- Untersuchen Sie die gestreiften Platten visuell auf Kolonien, die denen aus reinen Campylobacter-Kulturen ähneln.
HINWEIS: Der dritte Quadrant ist in der Regel dort, wo diese gefunden werden. Siehe Abbildung 1 und Abbildung 2 für Verdünnungsschema und Bilder von Koloniegröße, Farbe und Morphologie. - Wählen Sie mehrere Campylobacter-ähnlicheKolonien und Gram Fleck. Untersuchen Sie mit Hilfe der Mikroskopie mit einer Öl-Tauchlinse einen dünn gestreiften Bereich auf gramnegative gekrümmte, spiralförmige oder zigarrenförmige kleine Bakterien.
HINWEIS: Campylobacter sind gramnegativ und benötigen grundlegendes Fuchsin als Gegenfleck (anstelle des typischen Safranins), um genau visualisiert zu werden. Klassische Möwen-Flügelbakterien können gesehen werden, sind aber keine Voraussetzung. Siehe Abbildung 2 für repräsentative Mikrographik. - Wenn eine der doppelten Platten bei einer bestimmten Verdünnung 1 oder mehr Campylobacter-Kolonien vorhanden hat, betrachten Sie diese Verdünnungs-Fäkalkultur positiv.
- Betrachten Sie die letzte Verdünnung, die eine sichtbare Campylobacter-wie gramnegative Kolonie die Grenze der Kulturerkennung enthält. Verwenden Sie Gleichung 2, um die KBE/ml der positiven Verdünnung in erfundenen klinischen Fäkalienzuhaben zu berechnen:
CFU/mL in der Fäkalienprobe = Analytische KBE/ml - Verdünnung mit letzter positiver Kolonie [Gleichung 2]
HINWEIS: Typische Ergebnisse finden Sie in Tabelle 2.
- Unmittelbar nachdem die Platten für analytische Zählungen in Schritt 1.1.10 vorbereitet sind, machen Sie einen zweiten Satz von Brüheverdünnungen, indem Sie 10 serielle 2-fache Verdünnungen aus der Brühe und einem Campylobacter-negativenFäkalienpool (NFP) zubereiten. Bereiten Sie beispielsweise die erste Verdünnung vor, indem Sie gleiche Volumina von Brühe und NFP (z. B. 0,1 ml) mischen und nachfolgende Verdünnungen vornehmen, indem Sie ein bestimmtes Volumen von Brühe und NFP-Gemisch in ein Rohr mit einem gleich bezeichneten Volumen NFP übertragen. Fügen Sie eine Kontrollplatte mit Brühe ohne Campylobacter hinzu, die dem Fäkalpool hinzugefügt wurde, um nichtCampylobacter-Kolonien zu identifizieren.
2. Lebensfähigkeitsbestimmung von Campylobacter in Transportmedien gespeichert
- Mischen Sie 1 ml Campylobacter Brühe Kultur (Schritt 1.1.8) mit 1 ml NFP und bereiten 10 doppelte serielle Verdünnungen in NFP. Verdünnen Sie jede Verdünnung weiter um weitere 1:4 in Cary-Blair-Medien, so wie eine klinische Probe, die in Transportmedien zubereitet wird, behandelt wird.
- Bewahren Sie die 20 Verdünnungsrohre und eine Negativkontrolle in Cary-Blair-Medium in gekappten Rohren bei 2 °C für 96 h auf und zählen Sie Kolonien aus jeder Verdünnung, die zum Zeitpunkt Null und alle 24 h auftritt. Für die Koloniezählung, Probe der Brühe: Fäkalienrohre und Einrichtung Fäkalienkultur für Koloniezählen jeder Verdünnung, in Duplikat.
- Pro Tagesplatte 10 L Portionen der Fäkalverdünnungen auf Campylobacter-selektiver Agar und inkubieren bei 37 °C für 48 h, wie oben in den Schritten 1.1.9-1.2.7 beschrieben.
- Führen Sie eine gleichzeitige analytische Plattenanzahl des ursprünglichen Bakterienbestands (ab Schritt 1.1.8 oder eines frisch angebauten Brühenbestands) wie oben beschrieben (Schritte 1.1.9-1.1.11) durch. Berechnen Sie die KBE/ml des ursprünglichen Bakterienbestands (Gleichung 1), um die Konzentration von Bakterien in der Transportmedien-Fäkalienprobe und ihre Verdünnungen zu berechnen (Gleichung 2).
3. Nicht-kulturbezogene Assays zur Überprüfung von Kulturergebnissen
- Verwenden Sie einen Enzym-Immunoassay (EIA), der minimale falsch positive Ergebnisse12 liefert und gemäß den Anweisungen zum Einfügen von Verpackungen führt, um die Kulturergebnisse zu überprüfen.
- Verwenden Sie einen molekularen Assay, der das 16S rRNA-Gen oder ein anderes Gen einer breiten Palette von Campylobacter-Arten 13detektieren kann. Bestätigen Sie, dass der molekulare Assay mit Arten wie C. upsaliensis oder C. lari reagiert, die auf Standard-Antibiotika-haltigen Agar14schlecht wachsen. Befolgen Sie die Anweisungen des Herstellers für die Extraktion von DNA aus Fäkalienproben und die Durchführung des Tests.
HINWEIS: Bidirektionale DNA-Sequenzierung des 16S-Amplicons kann verwendet werden, um die Art von Campylobacter in einer positiven Probe zu bestätigen. Artspezifische PCR (siehe Tabelle 3 für Zielgene) können auch verwendet werden, um Arten zu identifizieren, die in diskreten oder positiven Proben vorhanden sind15.
Ergebnisse
Die Identifizierung von Campylobacter spp. Kolonien unter konkurrierenden Fäkalienflora erfordert ein scharfes Sehvermögen und ein beträchtliches Urteilsvermögen. Die niedrigste Anzahl von Kolonien, die durch Kultur nachgewiesen werden können, wurde nicht untersucht, obwohl Proben von Patienten auf 106x 109 KBE/ml16,17geschätzt wurden. Patientenproben können jedoch nicht quantitativ verwendet werden, da es keine unabhängige Methode zur Ermittlung genauer Bakterienzahlen gibt. Um diese Einschränkung zu überwinden, werden zwei gleichzeitige Messungen mit einem Bakterienbestand durchgeführt. Ein Test wird für den visuellen Nachweis von Campylobacter-Kolonien aus seriellen Verdünnungen der Stockbakterien in einer Fäkalienmatrix verwendet, wodurch klinische Proben simuliert werden; die andere wird analytisch verwendet, um die KBE/ml zu quantifizieren, die in der bakteriellen Bestandskultur zum Spiking enthalten ist (Abbildung 2A).
Die Erkennungsschwellenwerte für Campylobacter sind keine definierten Werte. Dies ist zu erwarten, da jede Fäkalienmatrix komplex und einzigartig ist und das Wachstum von Bakterien variabel ist. Ein wichtiger Parameter für den Erfolg ist die Identifizierung der punktgenauen Größenkolonien unter der konkurrierenden Fäkalienflora. Eine repräsentative Platte der Spitzenhockerkultur ist in Abbildung 2C und Abbildung 2Ddargestellt. Die negative Kontrollplatte ohne zusätzlichen Campylobacter ist wichtig, um andere Fäkalien zu identifizieren. Gram Färbung viele Kandidaten trainiert auch das Auge, um die richtigen glänzenden Kolonien und die zwischenrosa Farbe der fuchsin-gefärbten gramnegativen Bakterien zu unterscheiden und bestätigt die Morphologie der Bakterien in den ausgewählten Kolonien (Abbildung 2B). Sieben unabhängige Experimente wurden mit 5 C. jejuni und 2 C. coli Brühen durchgeführt und ergaben Schwellenwerte, die sich überlappen und von 0,3 x 5 x 106 KBE/ml überspannten. Typische Daten finden Sie in Tabelle 2. Die Nachweisgrenzen betrugen durchschnittlich 2 x 106 für C. jejuni und 1,2 x 106 KBE/ml für C. coli. Dies deutet darauf hin, dass dieKultur wahrscheinlichjejuni oder C. coli pro Gramm Fäkalienproztion auf Standard-Antibiotika-haltigem Campylobacter-spezifischemAgar, der von vielen klinischen Laboratorien verwendet wird, erkennen kann. Es gibt mehrere spezialisierte Agars mit verschiedenen Antibiotika, die verschiedene Schwellenwerte für die Erkennung von Kolonien geben können. Die hier beschriebenen Methoden sollten quantitativere und vergleichende Studien fördern, um die Genauigkeit der Kultur zu verbessern und die Vielseitigkeit neuer Medien zu erweitern. Zum Beispiel wurden 152 Kolonien auf der ersten 10-5 Platte und 144 Kolonien auf der zweiten 10-5 Platte gezählt. Der Durchschnitt zwischen den beiden Platten beträgt 148 Kolonien. Die Platten wurden mit 0,1 ml (100 l) von 10-5 Verdünnung geimpft, was nach Gleichung 1 148 x 106 (14,8 x 107) CFU/ml im Reinenkulturbestand entspricht. Als die Fäkalverdünnungen gemacht wurden, wurde die Kultur in negative Fäkalienbecken mit einem Verhältnis von 1:1 gespickt. Daher entspricht der erste Punkt (Platte "a") auf der Fäkalienkurve durch Gleichung 214,8 x 107 geteilt durch 2 und entspricht 7,4 x 107 CFU/ml. Dieses "a" Rohr wird verwendet, um 9 zusätzliche Verdünnungen zu machen. In Abbildung 1befindet sich die letzte Verdünnung mit einer sichtbaren gramnegativen Kolonie mit Campylobacter-ähnlicherMorphologie auf platte "g". Dies entspricht 1,1 x 106 KBE/ml für die Fäkalkulturschwelle der Erkennung in diesem Beispiel.
Auch wenn eine nachhaltige Lebensfähigkeit der Schlüssel zur Genauigkeit der Kultur ist, ist die Beibehaltung der Lebensfähigkeit von Campylobacter spp. bei der Handhabung und dem Versand von Proben von Patienten in Kliniken an Referenzlabore problematisch. Typische Lagerung ist die Kühlung von Proben in gewöhnlichen Kappenrohren mit Lufteinwirkung und ohne besondere Atmosphäre. Es wird angenommen, dass Proben in Transportmedien (auch als konservierte Proben bekannt) ein besseres Überleben haben, aber es gibt nur wenige Berichte, die quantitative Daten liefern18.
Die Kombination von analytischen und ausgeklügelten Probenmethoden wurde erneut verwendet, um Lebensfähigkeits- und Überlebenszeitschätzungen von C. jejuni in Transportmedien zu erhalten. Eine bakterielle Stockbrühe wurde verwendet, um zehn doppelte 2-fach bis 1024-fache Probenverdünnungen in der Fäkalienmatrix vorzubereiten. Die anfängliche Brühe wurde von den analytischen Zählungen mit einer Konzentration von 4,8 x 107 KBE/ml festgestellt. Auf Platten, die am Tag 0 hergestellt wurden, wurde C. jejuni (2 Tage später) auf der Platte mit der 32-fachen Verdünnung, die 1,5 x 106 KBE/ml entspricht, nachgewiesen. Auf den Platten, die nach dem Kühlen der Cary Blair-Fäkalienprobe für 24 Stunden hergestellt wurden, wuchs jedoch nur die 2-fache Verdünnung (entspricht 2,4 x 107 KBE/ml) sichtbare Kolonien. Kein weiterer Verlust an Lebensfähigkeit wurde bis 96 Stunden gesehen, als die Studie gestoppt wurde. Dieser Verlust an Lebensfähigkeit entspricht einem 16-fachen (94%) Verlust von kultivierbaren Organismen in weniger als 24 Stunden und weist darauf hin, dass, auch bei der Kühlung, Hocker in Cary Blair Medium mit weniger als 107 KBE/ml C. jejuni von der Kultur vermisst werdenkann.
Im Gegensatz zu den Ergebnissen der Kultur stellte die UVP das Vorhandensein von C. jejuni bei der 256-fachen Verdünnung zum anfangs Zeitpunkt und während des 4-tägigen Testzeitraums fest. Der C. jejuni-Nachweisschwelle für diese UVP mit spiked Fäkalienproben beträgt 8,4 x 104 KBE/ml. Dieser Schwellenwert liegt unter dem der Fäkalienkultur und ermöglicht eine empfindlichere und stabilere Erkennung von C. jejuni.
Um die Fähigkeit der Kultur zu testen, Campylobacter spp. in einem tatsächlichen klinischen Umfeld zu erkennen, wurden 1.552 klinische Stuhlproben durch 6 Verfahren charakterisiert: Fäkalienkultur, ein neuer Immunoassay für Campylobacter spp. und 4 molekulare Methoden. Alle Proben wurden prospektiv gesammelt und zunächst nach konventioneller Kultur in 3 Laboratorien in den Vereinigten Staaten klassifiziert und dann von der UVP überprüft. Alle kulturpositiven oder UVP/Kultur-Discrepant-Proben wurden dann mit den molekularen Methoden12gescreent. Den Exemplaren wurde ein wahr-positiver oder wahr-negativer Status zugewiesen, der auf den Ergebnissen der 5 Nichtkulturmethoden basiert. Die 5 nicht-kulturfreien Methoden zeigten eine vollständige Übereinstimmung bei allen 48 positiven und diskreten Exemplaren, während die Kultur 14 (28%) falsch identifizierte. Die Proben, die fälschlicherweise durch Kultur identifiziert wurden, enthielten 13 falsch negative und 1 falsch positive Probe.

Abbildung 1: Schema für die gleichzeitige Vorbereitung von analytischen und gespickten Fäkalienproben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
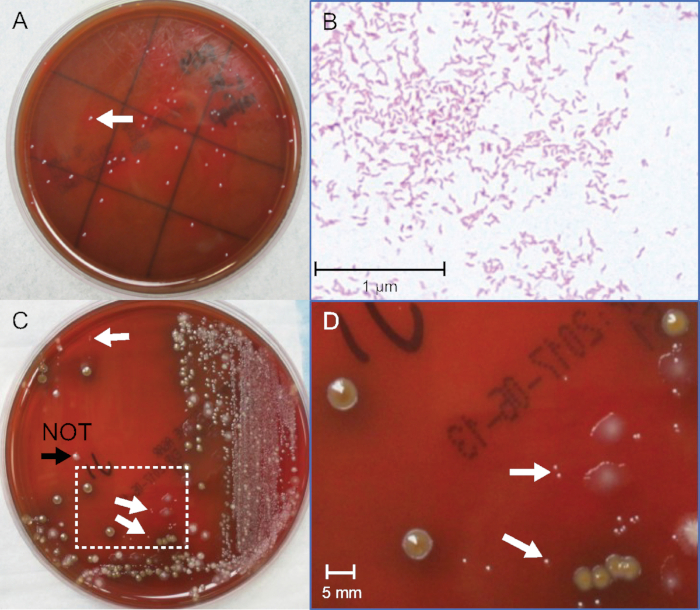
Abbildung 2: Identifizierung von C. jejuni-Kolonien aus reinen und fäkalen Kulturen. (A) Fotografie von C. jejuni Kolonien aus reiner Bakterienkultur nach 72 Stunden Inkubation. (B) Gramfleck von C. jejuni aus reiner Bakterienkultur, Öl-Immersion 400x Vergrößerung. (C) Fotografie von C. jejuni-positive spiked Fäkalienkultur nach 48 h Inkubation. (D) Vergrößerter Bereich in Kasten in (C), 10-fache Vergrößerung. Weiße Pfeile zeigen Punktgröße gram-negative C. jejuni Kolonien. Die schwarze Pfeilspitze zeigt eine Kolonie an, die etwas größer, grampositiv und nicht C. jejuniist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Kulturen | OD600 bei T0 | OD600 bei T Finale1 | Endgültige CFU/ml |
| C. jejuni | 0.146 | 0.321 | 1,28 x 107 |
| C. coli | 0.245 | 0.508 | 4,50 x 108 |
Tabelle 1: Typisches Wachstum und KBE/ml der Bestände C. jejuni und C. coli. 1C. jejuni Kultur wurde nach 48 h Inkubation gestoppt. Die C. coli-Kultur wurde nach 54 h Inkubation gestoppt.
| Verdünnungsrohr für spiked Fäkalienprobe | Anzahl Campylobacter-ähnlicheKolonien | Anzahl gramnegativer Kolonien1 | Kultur positiv? | Berechnete CFU/ml der Spike-Probe | |
| C. jejuni (1,28 x 108 KBE/ml)) | (2-fach) a | Dichten | nd2 | Ja | 6,40 x 107 |
| (4-fach) b | 20+ | Nd | Ja | 3,20 x 107 | |
| (8-fach) c | 4-10 | Nd | Ja | 1,60 x 107 | |
| (16-fach) d | Nd | Ja | 8,00 x 106 | ||
| (32-fach) e | Nd | Ja | 4,00 x 106 | ||
| (64-fach) f | 1-3 | 1 von 2 | Ja | 2,00 x 106 | |
| (128-fach) g | 2 von 3 | Ja | 1,00 x 106 | ||
| 3(256-fach) h | 1 von 2 | Ja | 5,00 x 105 | ||
| (512-fach) i | 0 von 1 | Nein | 2,50 x 105 | ||
| (1024-fach) j | 0 | Nd | Nein | Nfp | |
| C. coli (4,50 x 108 KBE/ml)) | (2-fach) a | Dichten | Nd | Ja | 2,25 x 108 |
| (4-fach) b | Nd | Ja | 1,13 x 108 | ||
| (8-fach) c | 50+ | Nd | Ja | 5,63 x 107 | |
| (16-fach) d | 30+ | Nd | Ja | 2,81 x 107 | |
| (32-fach) e | 10+ | Nd | Ja | 1,41 x 107 | |
| (64-fach) f | 3-8 | Nd | Ja | 7,03 x 106 | |
| (128-fach) g | Nd | Ja | 3,52 x 106 | ||
| 3(256-fach) h | 1-3 | 1 von 3 | Ja | 1,76 x 106 | |
| (512-fach) i | 0 von 1 | Nein | 8,79 x 105 | ||
| (1024-fach) j | 0 | Nd | Nein | Nfp |
Tabelle 2: Typische Anzahl von Kolonien auf Platten von gespickten Fäkalienproben. 1 Gramm negative Kolonien unter Campylobacter-ähnlichenKolonien, 2nd = nicht bestimmt, 3Daten in fetter Art zeigen die letzte positive Verdünnung an.
| Spezies | Genziel |
| C. jejuni | Hipo |
| C. coli | cadF |
| C. upsaliensis | cpn60 |
| C. lari | cpn60 |
| C. helveticus | cpn60 |
| C. Fötus | cpn60 |
| C. hyointestinalis | cpn60 |
| C. Concisus | cpn60 |
Tabelle 3: Gene nützlich für den Nachweis einzelner Campylobacter-Arten qPCR.
Diskussion
Die hier beschriebenen Kulturmethoden basieren auf einfachen, weit verbreiteten Techniken und Materialien, die in den meisten Laboratorien verfügbar sind10. Es ist die Kombination von analytischen und ausgeklügelten Proben, die neue Informationen über einen klinisch relevanten Nachweisschwellenwert für Fäkalienkulturen liefern. Darüber hinaus verstärkt die Beurteilung von Kulturergebnissen mit 5 separaten Assays die Schlussfolgerung, dass Campylobacter-Fäkalkultur einen signifikanten Teil der Patientenproben falsch identifiziert. Die UVP und molekulare Assays sind als Kontrollen nützlich, da sie jeweils auf einem anderen Prinzip basieren (Antigen-Interaktion mit Antikörper vs. DNA-Verstärkung) und, was wichtig ist, nicht auf die Lebensfähigkeit von Bakterien angewiesen sind. Beachten Sie, dass der für diese Studien verwendete UVP-Test gut validiert ist und nachweislich mit 4 molekularen Tests vollständig einverstanden ist12.
Kultur von Campylobacter spp. ist besonders lästig, mit Empfindlichkeit berichtet, um von 60 bis 76%19,20, und wie aus seiner 30% Rate der Nicht-Erkennung von wirklich-positiven Proben hier zu erkennen. Das Personal kann erwarten, dass die Kontrolle der UVP und molekulare Tests häufig zu positiven Ergebnissen führen, wenn Die Kulturdaten negativ sind.
Der wichtigste Schritt im Protokoll ist die Identifizierung von punktgenauen Kolonien unter konkurrierenden Fäkalienflora. Es ist nicht ungewöhnlich, dass es als Verdünnungsschwelle abwechselnd Schätzungen der Anzahl der Null- und -Null-Kolonien gibt (z. B. 2, 0, 1, 0, 0). Es ist wichtig zu erkennen, dass Kulturschwellen eine Reihe von Konzentrationen sein werden, nicht eine bestimmte KBE/ml. Nichtsdestotrotz ist die Schätzung von 1 x 106 KBE/ml Kot als untere Grenze für die Kulturerkennung gut im Vergleich zu Berichten, dass infizierte Menschen 106 bis 109Campylobacter pro Gramm Kot vergossen haben21. Veränderungen der Antibiotika oder Agarplatten und Schwankungen, die in einzelnen Fäkalienproben unvermeidlich sind, werden zweifellos die Schwellenwerte verändern. Dieses Protokoll sollte Verbesserungen in den Wachstumsmedien ermöglichen.
Diese ersten Informationen über eine Grenze für die Kulturdetektion ermöglichen es, klinisch relevante Schwellenwerte für diagnostische Tests festzulegen, und legen die mikrobiologische Grundlage, die benötigt wird, um nicht untersuchte Probleme des nicht-symptomatischen Wagens22,23 von Campylobacterzu lösen, oder wenn die bakterielle Belastung mit Symptomen oder schwerwiegenden Folgen korreliert.
Offenlegungen
Die Autoren sind Mitarbeiter von TECHLAB, Inc., die das QUIK CHEK™ Kit produziert, das in diesem Artikel als Komparator verwendet wird.
Danksagungen
Diese Studien wurden von TECHLAB, Inc. finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Anaerobic 3.5L Jar | Thermo Fisher | HP0031A | |
| AnaeroGRO Campylobacter Selective Agar | Hardy Diagnostics | AG701 | |
| Bacto Brain Heart Infusion | BD Biosciences | 237500 | |
| Bacto Protease Peptone | Life Technologies Corp | 211684 | |
| Basic Fuchsin | Fisher Scientific | B12544 | |
| BBL Trypticase Peptone | Life Technologies Corp | 211921 | |
| C. coli Type strain | ATCC | 33559 | |
| C. jejuni Type strain | ATCC | 33560 | |
| CampyGen gas generating system sachet | Thermo Fisher | CN0025A | |
| Campylobacter QUIK CHEK | TechLab, Inc. | T5047 / T31025 | |
| Cary-Blair transport medium | Fisher Scientific | 23-005-47 | |
| Coli Roller Sterile plating beads | Millipore Sigma | 71013 | |
| Dilution Buffer | Anaerobe Systems | AS-908 | |
| Fetal bovine serum | Equitech-Bio, Inc | SFBM30 | |
| Sodium bisulfite | Sigma-Aldrich | 243973 | |
| Sodium pyruvate | Sigma-Aldrich | P2256 | |
| Spectrophotometer cuvettes | USA Scientific | 9090-0460 |
Referenzen
- CDC. Annual Summaries of Foodborne Outbreaks. , Available from: https://www.cdc.gov/fdoss/annual-reports/index.html (2018).
- Kaakoush, N. O., Castaño-Rodríguez, N., Mitchell, H. M., Man, S. M. Global Epidemiology of Campylobacter Infection. Clinical Microbiology Reviews. 28 (3), 687-720 (2015).
- Pitkanen, T. a, H, M. L. Global Water Pathogen Project. Rose, J. B., Jimenez-Cisneros, B. , UNESCO (2017).
- Fitzgerald, C., et al. Multicenter Evaluation of Clinical Diagnostic Methods for Detection and Isolation of Campylobacter spp. from Stool. Journal of Clinical Microbiology. 54 (5), 1209-1215 (2016).
- Kirkpatrick, B. D., Tribble, D. R. Update on human Campylobacter jejuni infections. Current Opinion in Gastroenterology. 27 (1), 1-7 (2011).
- CDC. Incidence and Trends of Infection with Pathogens Transmitted Commonly Through Food - Foodborne Diseases Active Surveillance Network, 10 U.S. Sites, 2006-2013. Morbidity and Mortality Weekly Report. 63, 328-332 (2014).
- Jaime, A. L., et al. Campylobacter upsaliensis: Another Pathogen for Consideration in the United States. Clinical Infectious Diseases. 34 (11), 59-60 (2002).
- Bullman, S., O'Leary, J., Corcoran, D., Sleator, R., Lucey, B. Molecular-based detection of non-culturable and emerging campylobacteria in patients preseting with gastroenteritis. Epidemiology and Infection. 140, 684-688 (2012).
- Giltner, C. L., Saeki, S., Bobenchik, A. M., Humphries, R. M. Rapid Detection of Campylobacter Antigen by Enzyme Immunoassay Leads to Increased Positivity Rates. Journal of Clinical Microbiology. 51 (2), 618-620 (2013).
- M'ikanatha, N. M., et al. Culturing stool specimens for Campylobacter spp., Pennsylvania, USA. Emerging Infectious Disease. 18, 484-487 (2012).
- Al Amri, A., Senok, A. C., Ismaeel, A. Y., Al-Mahmeed, A. E., Botta, G. A. Multiplex PCR for direct identification of Campylobacter spp. in human and chicken stools. Journal of Medical Microbiology. 56 (10), 1350-1355 (2007).
- Buss, J. E., et al. Campylobacter culture fails to correctly detect Campylobacter in 30% of positive patient stool specimens compared to non-cultural methods. European Journal of Clinical Microbiology & Infectious Diseases. 38, 1087-1093 (2019).
- Wohlwend, N., Tiermann, S., Risch, L., Risch, M., Bodmer, T. Evaluation of a Multiplex Real-Time PCR Assay for Detecting Major Bacterial Enteric Pathogens in Fecal Specimens: Intestinal Inflammation and Bacterial Load Are Correlated in Campylobacter Infections. Journal of Clinical Microbiology. 54 (9), 2262-2266 (2016).
- Couturier, B. A., Hale, D. C., Couturier, M. R. Association of Campylobacter upsaliensis with Persistent Bloody Diarrhea. Journal of Clinical Microbiology. 50 (11), 3792-3794 (2012).
- Chaban, B., Musil, K. M., Himsworth, C. G., Hill, A. K. Development of cpn60-Based Real-Time Quantitative PCR Assays for the Detection of 14 Campylobacter Species and Application to Screening of Canine Fecal Samples. Applied and Environmental Microbiology. 75, 3055-3061 (2009).
- Allos, B., Blaser, M. J. Mandell, Douglas, and Bennett's Principles and Practice of Infectious Diseases, 7th ed. Mandell, G. L., Bennett, J. E., Dolin, R. , Churchill Livingstone. 2793-2802 (2009).
- Anderson, N. W., Buchan, B. W., Ledeboer, N. A. Comparison of the BD MAX Enteric Bacterial Panel to Routine Culture Methods for Detection of Campylobacter, Enterohemorrhagic Escherichia coli (O157), Salmonella, and Shigella Isolates in Preserved Stool Specimens. Journal of Clinical Microbiology. 52 (4), 1222-1224 (2014).
- Wasfy, M., Oyofo, B., Elgindy, A., Churilla, A. Comparison of preservation media for storage of stool samples. Journal of Clinical Microbiology. 33 (8), 2176-2178 (1995).
- Bessède, E., Delcamp, A., Sifre, E., Buissonniere, A., Mégraud, F. New Methods for Detection of Campylobacters in Stool Samples in Comparison to Culture. Journal of Clinical Microbiology. 49 (3), 941-944 (2011).
- Bessède, E., et al. Evaluation of the Diagnostic Accuracy of Two Immunochromatographic Tests Detecting Campylobacter in Stools and Their Role in Campylobacter Infection Diagnosis. Journal of Clinical Microbiology. 56 (4), (2018).
- Shane, A. L., et al. Infectious Diseases Society of America Clinical Practice Guidelines for the Diagnosis and Management of Infectious Diarrhea. Clinical Infectious Diseases. 65 (12), 1963-1973 (2017).
- Toledo, Z., Simaluiza, R. J., Astudillo, X., Fernández, H. Occurrence and antimicrobial susceptibility of thermophilic Campylobacter species isolated from healthy children attending municipal care centers in Southern Ecuador. Revista do Instituto de Medicina Tropical de Sao Paulo. 59, 77-77 (2017).
- Lee, G., et al. Symptomatic and asymptomatic Campylobacter infections associated with reduced growth in Peruvian children. PLoS Neglected Tropical Diseases. 7 (1), 2036(2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten