Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine Wasserstoff-Deuterium-Austausch-Massenspektrometrie -Plattform (HDX-MS) zur Untersuchung biosynthetischer Peptidenzyme
In diesem Artikel
Zusammenfassung
Lanthipeptid-Synthetasen katalysieren mehrstufige Reaktionen während der Biosynthese von Peptid-Naturprodukten. Hier beschreiben wir einen kontinuierlichen, Bottom-up, Wasserstoff-Deuterium-Austausch-Massenspektrometrie (HDX-MS) Workflow, der verwendet werden kann, um die Konformationsdynamik von Lanthipeptid-Synthetasen zu studieren, sowie andere ähnliche Enzyme, die an der Peptid-Naturproduktbiosynthese beteiligt sind.
Zusammenfassung
Die Wasserstoff-Deuterium-Austauschmassenspektrometrie (HDX-MS) ist eine leistungsfähige Methode zur biophysikalischen Charakterisierung von Enzymkonformationsveränderungen und Enzym-Substrat-Wechselwirkungen. Unter seinen vielen Vorteilen verbraucht HDX-MS nur geringe Mengen an Material, kann unter nahezu nativen Bedingungen ohne Enzym-/Substratkennzeichnung durchgeführt werden und kann räumlich aufgelöste Informationen über Enzymkonformationsdynamik liefern – auch für große Enzyme und Multiproteinkomplexe. Die Methode wird durch die Verdünnung des enzyms von Interesse in Puffer inD2O vorbereitet initiiert. Dies löst den Austausch von Protium in Peptidbindungsamiden (N-H) mit Deuterium (N-D) aus. An den gewünschten Austauschzeitpunkten werden Reaktions-Aliquots abgeschreckt, das Enzym in Peptide proteolysiert, die Peptide durch ultra-performance liquid chromatography (UPLC) getrennt und die Veränderung der Masse jedes Peptids (aufgrund des Austauschs von Wasserstoff gegen Deuterium) wird von MS aufgezeichnet. Die Menge der Deuteriumaufnahme durch jedes Peptid hängt stark von der lokalen Wasserstoffbindungsumgebung dieses Peptids ab. Peptide, die in sehr dynamischen Regionen des Enzymaustauschdeuteriums sehr schnell vorhanden sind, während Peptide aus gut geordneten Regionen viel langsamer austauschen. Auf diese Weise berichtet die HDX-Rate über die lokale Enzymkonformationsdynamik. Störungen der Deuteriumaufnahme in Gegenwart verschiedener Liganden können dann verwendet werden, um Ligandenbindungsstellen zu kartieren, allosterische Netzwerke zu identifizieren und die Rolle der Konformationsdynamik in der Enzymfunktion zu verstehen. Hier veranschaulichen wir, wie wir HDX-MS verwendet haben, um die Biosynthese einer Art von Peptid-Naturprodukten namens Lanthipeptide besser zu verstehen. Lanthipeptide sind genetisch kodierte Peptide, die posttranslational durch große, multifunktionale, konformational dynamische Enzyme modifiziert werden, die mit traditionellen strukturbiologischen Ansätzen schwer zu studieren sind. HDX-MS bietet eine ideale und anpassungsfähige Plattform zur Untersuchung der mechanistischen Eigenschaften dieser Enzymtypen.
Einleitung
Proteine sind strukturell dynamische Moleküle, die verschiedene Konformationen auf Zeitskalen abtasten, die von Bindungsschwingungen im Femtosekundenmaßstab bis hin zu Umlagerungen ganzer Proteindomänen reichen, die über viele Sekunden auftreten können1. Diese Konformationsschwankungen sind oft kritische Aspekte der Enzym-/Proteinfunktion. So sind beispielsweise konforme Veränderungen, die durch Ligandenbindung induziert werden, oft von entscheidender Bedeutung für die Modulation der Enzymfunktion, entweder durch die Organisation aktiver Standortrückstände, die für die Katalyse benötigt werden, die Definition von Substratbindungsstellen in sequenziellen kinetischen Mechanismen, die Abschirmung reaktiver Zwischenprodukte von der Umgebung oder durch Modulation der Enzymfunktion über allosterische Netzwerke. Jüngste Studien haben auch gezeigt, dass die konforme Dynamik während der gesamten Evolution konserviert werden kann und dass Störungen zu konservierten molekularen Bewegungen mit Veränderungen der Substratspezifität und der Entstehung neuer Enzymfunktionenkorreliertwerden können 2,3.
In den letzten Jahren hat sich die Wasserstoff-Deuterium-Austausch-Massenspektrometrie (HDX-MS) schnell als eine leistungsstarke Technik entwickelt, um zu untersuchen, wie Proteinkonformationslandschaften auf Störungen wie Ligandenbindung oder Mutagenese4,5,6,7reagieren. In einem typischen HDX-MS-Experiment (Abbildung 1) wird ein inD2O hergestelltes Protein in einen Puffer gegeben, der den Austausch von lösungsmittelaustauschbaren Protonen durch Deuteria auslöst. Die Wechselkursrate der Amid-Feuchtigkeit der Peptidbindungen hängt stark vom pH-Wert, der lokalen Aminosäuresequenz und der lokalen strukturellen Umgebung des Amids8ab. Amide, die mit Wasserstoffbindungswechselwirkungen (wie sie in α-Helices und β-Blättern vorhanden sind) tätig sind, tauschen sich langsamer aus als Amide in unstrukturierten Regionen des Proteins, die Massenlösungsmitteln ausgesetzt sind. Somit spiegelt das Ausmaß der Deuteriumaufnahme die Struktur des Enzyms wider. Enzyme, die konform dynamisch sind oder bei Ligandenbindung strukturelle Übergänge durchlaufen, würden eine messbare HDX-Antwort liefern.
Die mechanistische Grundlage für den langsamen Wechselkurs eines strukturierten Amids ist in Abbildung 25,8,9dargestellt. Um HDX zu durchlaufen, muss der strukturierte Bereich zunächst eine entfaltete Konformation vorübergehend abtasten, so dass die Lösungsmittelmoleküle, die den HDX-Austausch über einen bestimmten säure-basenchemischen Mechanismus katalysieren, Zugang zum austauschbaren Amid haben. Letztlich bestimmen die relativen Größen des chemischen Wechselkurses (kchem) und die Falt- und Umfaltraten (koffen und kschließen) die im ExperimentgemesseneHDX-Rate 5,8. Aus diesem einfachen kinetischen Modell geht klar hervor, dass das Ausmaß der Deuteriumaufnahme die zugrunde liegende Konformationsdynamik widerspiegelt (wie definiert durch koffen und kclose). Die meisten HDX-MS-Experimente werden in einem Bottom-up-Workflow durchgeführt, bei dem nach der Austauschreaktion das von Interesse kommende Protein in Peptide verdaut wird und die Deuteriumaufnahme durch jedes Peptid als Erhöhung der Masse7gemessen wird. Auf diese Weise ermöglicht HDX-MS die Zuordnung von Störungen der Enzym-Konformationsdynamik auf der lokalen räumlichen Skala von Peptiden, sodass der Forscher beurteilen kann, wie die Störung die Dynamik in verschiedenen Regionen des Enzyms von Interesse verändert.
Die Vorteile des HDX-MS-Ansatzes zur Aufklärung der Proteinstrukturdynamik sind zahlreich. Zunächst kann die Methode mit kleinen Mengen an nativem Protein oder auf Proteinkomplexen in Systemen mit quartärer Struktur10durchgeführt werden. Es ist nicht einmal notwendig, dass die im Test verwendete Enzympräparation hochgereinigt wird11,12, solange der Bottom-up-HDX-MS-Workflow eine ausreichende Anzahl von sicher identifizierten Peptiden bietet, die die Proteinsequenz von Interesse abdecken. Darüber hinaus kann HDX-MS Informationen über konforme Dynamiken unter nahezu nativen Bedingungen bereitstellen, ohne dass eine standortspezifische Proteinkennzeichnung erforderlich ist, wie sie in Einzelmolekülfluoreszenzstudien13verwendet würde, und es gibt keine Größenbeschränkung für den Protein- oder Proteinkomplex, die untersucht werden kann (was Ansätze wie Kernspinresonanz [NMR] Spektroskopie schwierig macht)7,14. Schließlich können zeitaufgelöste HDX-MS-Methoden eingesetzt werden, um intrinsisch ungeordnete Proteine zu untersuchen, die mit Röntgenkristallographie15,16,17,18schwer zu untersuchen sind. Die Hauptbeschränkung von HDX-MS ist, dass die Daten von geringer struktureller Auflösung sind. HDX-MS-Daten sind nützlich, um darauf hinzuweisen, wo sich die Konformationsdynamik ändert, und um gekoppelte Konformationsänderungen aufzudecken, aber sie bieten oft nicht viel Einblick in den genauen molekularen Mechanismus, der die beobachtete Veränderung antreibt. Jüngste Fortschritte in der Kombination von Elektronenfang-Dissoziationsmethoden mit Protein-HDX-MS-Daten haben gezeigt, dass Austauschstellen zu einzelnen Aminosäurerückständen19zugeordnet werden können, aber nachfolgende biochemische und strukturelle Studien sind immer noch häufig erforderlich, um Klarheit für Strukturmodelle zu schaffen, die von HDX-MS-Daten übermittelt werden.
Nachfolgend wird ein detailliertes Protokoll für die Entwicklung eines HDX-MS-Assays vorgestellt20. Die nachstehend vorgestellten Probenvorbereitungsprotokolle sollten allgemein für jedes Protein gelten, das eine gute Löslichkeit in wässrigen Puffern aufweist. Speziellere Probenvorbereitungsmethoden und HDX-MS-Workflows stehen für Proteine zur Verfügung, als in Gegenwart von Waschmitteln oder Phospholipiden21,22,23,24untersucht werden müssen. Instrumentale Einstellungen für die HDX-MS-Datenerfassung werden für ein hochauflösendes Quadrupol-Massenspektrometer beschrieben, das an das Flüssigchromatographiesystem gekoppelt ist. Daten von ähnlicher Komplexität und Auflösung könnten auf einem der handelsüblichen Flüssigchromatographie-Massenspektrometrie-Systeme (LC-MS) gesammelt werden. Wichtige Aspekte der Datenverarbeitung mit einem kommerziell erhältlichen Softwarepaket werden ebenfalls zur Verfügung gestellt. Wir stellen auch Richtlinien für die Datenerhebung und -analyse vor, die mit den Empfehlungen der breiteren HDX-MS-Community12übereinstimmen. Das beschriebene Protokoll wird verwendet, um die dynamischen strukturellen Eigenschaften von HalM2 zu untersuchen, einer Lanthipeptid-Synthetase, die die mehrstufige Reifung eines antimikrobiellen Peptids20katalysiert. Wir veranschaulichen, wie HDX-MS verwendet werden kann, um Substratbindungsstellen und allosterische Eigenschaften aufzudecken, die einer vorherigen Charakterisierung entgangen sind. Mehrere andere Protokolle zum Protein HDX-MS wurden in den letzten Jahren veröffentlicht25,26. Zusammen mit der vorliegenden Arbeit sollten diese früheren Beiträge dem Leser eine gewisse Flexibilität im experimentellen Design bieten.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Herstellung von deuterated Reagenzien und Enzymstofflösungen
- Bereiten Sie Reagenzien für die HDX-Reaktionen (einschließlich pufferr, Salze, Substrate, Liganden usw.) als 100-200-fache konzentrierte Stofflösungen in D2O (99,9% Atomfraktion D) vor. Bereiten Sie mindestens 50 ml Pufferlagerlösung vor.
HINWEIS: Für die Charakterisierung von HalM2 wurden folgende Lösungen vorbereitet: 500 mM MgCl2, 100 mM Tris (2-Carboxyethyl)phosphin (TCEP), 750 mM ATP (in HEPES-Puffer), 800 mM HEPES pD 7,1, 500 m HalA2 und 500 mM AMPPNP. - Einfrieren und lyophilisieren Sie die Stammlösungen auf Trockenheit.
- Lösen Sie sich inD2O auf und wiederholen Sie den Lyophilisierungszyklus mindestens eine zusätzliche Zeit, um so viele der austauschbaren Protonen wie möglich durch Deuteronen zu ersetzen.
- Stellen Sie die pD des deuterated HEPES Pufferbestands auf den gewünschten Wert mit konzentriertem NaOD/DCl ein, wobei die folgende Beziehung zu berücksichtigenist 27:
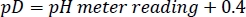
HINWEIS: Die Amid-HDX-Rate ist stark vom pL der Lösung abhängig (pL = pH oder pD)5. Verschiedene Chargen von Pufferlagerlösungen müssen auf identische Weise vorbereitet, gelagert und verwendet werden, um eine leichte pL-Drift zwischen den Experimenten zu vermeiden. - Berechnen Sie die Menge jedes Reagenzes, das für einen 300-L-HDX-Assay benötigt wird, und speichern Sie sie als Einweg-Aliquots bei -80 °C.
- Bereiten Sie eine konzentrierte Enzymstofflösung (ca. 100 bis 200 M) im protiated Enzymspeicherpuffer mit einem Zentrifugalfilter(Materialtabelle) oder einer gleichwertigen Vorrichtung vor.
HINWEIS: Der genaue Puffer- und Zentrifugalfilter Molekulargewichts-Cutoff hängt vom Protein/Enzym ab. HalM2 wird in 50 mM HEPES, pH 7,5, 100 mM KCl und 10% Glycerin gelagert. Zur Herstellung des konzentrierten Enzyms wurden 10 kDa-Filter verwendet. - Aliquot das Enzym in Einzelgebrauchsportionen und speichern Sie bei -80 °C.
HINWEIS: Dies kann ein Haltepunkt sein. Alle in Abschnitt 1 beschriebenen Lagerlösungen können im Vorfeld der HDX-Reaktionen vorbereitet werden. Bei -80 °C gelagert, sind die meisten Enzyme/deuterated Stock-Lösungen für viele Monate stabil.
2. Kalibrierung des HDX-Abschreckvolumens
- Bereiten Sie eine 300-L-HDX-Reaktion inD2O mit den in Abschnitt 1 hergestellten deuterated Reagenzien und konzentrierten Enzymvorräten vor.
- Verwenden Sie eine endgültige Enzymkonzentration von 1 bis 5 m.
- Verwenden Sie eine abschließend euterated HEPES Pufferkonzentration von mindestens 50 x 100 mM.
- Stellen Sie sicher, dass die Konzentrationen anderer Komponenten ausreichen, um die gewünschte Enzymaktivität/-funktion aufrechtzuerhalten.
- Bereiten Sie 1 L HDX-Quench-Lösung (100 mM Phosphat, 0,8 Mio. Guanidin-HCl, pH 1,9) vor. Einfrieren und Lagern in sowohl 50 ml Portionen (für langfristige Lagerbestände) als auch in 1 ml Portionen (für Einweg-Aliquots).
HINWEIS: Die genaue Zusammensetzung des Quench-Puffers hängt von dem Enzym ab, das im Proteolyseschritt des Bottom-up-HDX-MS-Workflows verwendet wird (Schritt 3.3.3). Der hier angegebene Quench-Puffer ist kompatibel mit Pepsin, der am häufigsten verwendeten Protease für HDX-MS. Wenn eine andere Protease verwendet wird, wenden Sie sich an den Protease-Lieferanten, um die Pufferkompatibilität sicherzustellen. - Kalibrieren Sie das Volumen des Löschpuffers, der benötigt wird, um den endgültigen pL des abgeschreckten HDX-Reaktionsgemisches auf einen pH-Meter-Messwert von 2,3 einzustellen.
HINWEIS: Der Lösungsmittel-H/D-Wechselkurs der Peptidamid-N-H-Bindung ist ein pH-abhängiges Verfahren, das sowohl einer Säure- als auch einer Basenkatalyse unterliegt. Der Mindestwechselkurs tritt bei einem Wert von pH 2,5 auf (pH-Meterstand = 2,3 für ein 50:50 H2O:D2O-Gemisch). So minimiert ein endgültiger pL-Wert nahe 2,5 den Wasserstoff-Rückaustausch, der während der Bottom-up-LC-MS-Analyse auftritt, wodurch das Deuterium-Etikett in den Peptiden erhalten bleibt.- Mischen Sie 50 l des HDX-Reaktionsgemisches aus Schritt 2,1 mit 50 l Löschpuffer und messen Sie den pL der abgeschreckten Mischung mit einer Mikrospitzenelektrode.
- Erhöhen Sie das Volumen der Quench-Lösung nach Bedarf, um den endgültigen pH-Meterstand auf einen Wert von 2,3 anzupassen.
- Nachdem das entsprechende Abschreckvolumen bestimmt wurde, wiederholen Sie den Abschreckvorgang mehrmals mit frischen 50 L-Aliquots aus der HDX-Reaktion (Schritt 2.1), um sicherzustellen, dass ein konsistentes endliches pL erreicht wird, wenn eine feste Menge an Quenchpuffer hinzukommt.
3. Erstellung von Referenzproben und Optimierung des Bottom-up LC-MS-Workflows
- Bereiten Sie undeuterated Referenzproben für das Protein von Interesse in Triplicate in 0,5 ml-Röhren vor. Stellen Sie sicher, dass die Bedingungen des Endreaktionsgemisches mit denen identisch sind, die in den authentischen HDX-Reaktionen (Schritt 2.1) verwendet werden, mit der Ausnahme, dass die Reaktionen in H2O mit Reagenzien-Stocklösungen hergestellt werden, die ebenfalls in H2O hergestellt sind.
- Quench die Proben wie in Schritt 2.3 durch Hinzufügen des entsprechenden Volumens des Quench-Puffers, um den endgültigen pH-Wert auf 2,5 einzustellen. Blitz einfrieren Sie die Proben in flüssigem Stickstoff und lagern Sie bei -80 °C, bis sie zur Analyse bereit sind.
- Analysieren Sie die protiated Enzymreferenzproben mit einem Bottom-up-LC-MS-Workflow.
HINWEIS: Vor der Ausführung dieser Schritte sollte das für die Datenerfassung zu verwendende LC-MS-System ordnungsgemäß kalibriert und einsatzbereit sein. Das Timing und die Temperatur aller Schritte im Bottom-up-LC-MS-Workflow müssen streng kontrolliert werden, um Unterschiede im Rückaustausch zwischen den Proben zu minimieren. Mit der in diesem Protokoll verwendeten MS-Instrumentierung (Materialtabelle Und Unterstützende Informationen), können die meisten Schritte über die Instrumentensoftware gesteuert werden. Um die Sammlung präziser Replikationen sicherzustellen, wird empfohlen, so viele Schritte im Workflow wie möglich zu automatisieren.- Entfernen Sie eine einzelne Enzymreferenzprobe (zubereitet wie in Schritt 3.2) aus dem Gefrierschrank und tauen Sie bei 37 °C für 1 min in einem Wasserbad auf.
- In genau 2 min nach dem Entfernen der Probe aus dem Gefrierschrank und dem Auftauen einen 40-L-Teil der abgeschreckten Referenzprobe in eine ultra-performance liquid chromeatography (UPLC) Kolonne (2,1 x 30 mm, 300 , 5 m) mit einer stationären Phase, die mit Pepsin (einer säurestabilen Protease) funktionalisiert ist.
- Verdauen Sie die Probe mit einer Durchflussrate von 100 l/min für 3 min bei 15 °C mit 0,1 % Ameisensäure inH2O (pH = 2,5) als Lösungsmittel.
- Sammeln Sie die Peptide, während sie von der Pepsinsäule auf eine C18-Falle-Säule bei 0,4 °C gehalten werden, um den Rückaustausch zu minimieren.
- Übergeben Sie die entsalzten Pepttide von der Trapsäule auf eine C18-Analysesäule (1 mm x 100 mm, 1,7 m, 130 °C), die bei 0,4 °C zur Trennung der Peptide gehalten und betrieben wird.
HINWEIS: Die Schritte 3.3.3-3.3.5 können von bestimmten LC-MS-Systemen automatisiert werden, die für die HDX-MS-Datenerfassung verwendet werden. Alternativ können diese Schritte unabhängig durchgeführt werden, wobei zu berücksichtigen ist, dass das Timing und die Temperatur jedes Schritts sorgfältig kontrolliert werden müssen, um einen konstant niedrigen Rückenaustausch zu erreichen. - Elute die C18-Säule mit einem Acetonitril/Wasser/0,1% Ameisensäure-Lösungsmittelsystem. Optimieren Sie den LC-Gradienten für das Protein von Interesse, um die Trennung von zu maximieren und das Deuterium-Label in den Peptiden zu erhalten.
HINWEIS: Die Daten zur Gradientenelution finden Sie in den Unterstützenden Informationen. - Unterziehen Sie den Magenverdauungsindex der Elektrospray-Ionisation (ESI) Massenspektrometrie.
HINWEIS: Die in den Unterstützenden Informationen angegebenen Quellbedingungen bieten eine ausreichende Ionisierung für die meisten Peptide.- Nach der Ionisiertim MS-Instrument führen Sie eine Gasphasen-Ionen-Mobilitätstrennung mit Stickstoff als Puffergas durch, um die Spitzenkapazität der Methode zu erhöhen.
- Nach der Ionenmobilitätstrennung unterziehen Sie die Peptidvorläuferionen einem MSE-Workflow mit abwechselnden Zyklen mit geringer Kollisionsenergie (4 V) und hoher Kollisionsenergie (21-40 V).
HINWEIS: Die abwechselnden Systeme der niedrigen und hohen Kollisionsenergie ermöglichen die gleichzeitige Erfassung von MS-Daten (niedrige Kollisionsenergie) mit MSMS-Daten (hohe Kollisionsenergie). Dies wiederum ermöglicht die zeitliche Korrelation von Vorläuferionen mit ihren jeweiligen Fragmentionen. Diese Korrelation ist für die in Abschnitt 4 beschriebene sichere Peptididentifikation von entscheidender Bedeutung. - Erkennen Sie den Peptidvorläufer und die Fragmentionen mit einem Massenanalysator mit einer Auflösungskraft von mindestens 20.000.
- Erwerben Sie gleichzeitig mit der Datenerfassung MS-Daten für einen [Glu-1]-Fibrinopeptid B (GluFib) externen Standard.
HINWEIS: Der in Schritt 3.3.7 beschriebene MS-Workflow wird als MSE-Protokoll bezeichnet. Vollständige Instrumentasteneinstellungen für ein MSE-Protokoll, das für UNTEN-up-HDX-MS geeignet ist, finden Sie in der Support Information.
- Bewerten Sie die Qualität der LC-MS-Daten.
ANMERKUNG: Unter Verwendung des oben beschriebenen Protokolls und der in den Unterstützenden Informationenangegebenen Instrumentaleinstellungen sollten die Referenzproben ein Gesamtionenchromatogramm mit einer maximalen Signalintensität von ca. 1 x 108erzeugen. Es sollten viele Peptide zwischen 3 und 9 min elutieren (Abbildung 3A-C). - Injizieren Sie 40 l Leerproben (0,1 % Ameisensäure in Wasser), um die Pepsin- und analytischen C18-Säulen zu reinigen.
ANMERKUNG: Im Allgemeinen sollten 2 x 3 Rohlinge ausreichen. - Wiederholen Sie die Schritte 3.3.1-3.3.9 für jede der dreifachen Referenzproteinproben.
4. Verarbeitung der Referenzdaten und Definition einer Peptidliste
- Analysieren Sie die rohen MSE-Daten (Schritt 3.3.7) mit der Proteomik-Software (Tabelle der Materialien). Navigieren Sie mit der Proteomics-Software zu Bibliotheken | Protein Sequenz Datenbanken, um die Protein-Datenbank zu definieren, indem Sie die Aminosäuresequenz des Proteins von Interesse importieren.
HINWEIS: Das Ziel dieses Schritts besteht darin, die Referenz-MS-Daten nach Peptiden zu durchsuchen, die aus dem Protein von Interesse abgeleitet wurden, und die MSMS-Daten (gleichzeitig mit den MS-Daten erfasst) zu verwenden, um alle vermeintlichen Peptid-Identifikationen zu validieren. - Geben Sie der Proteinsequenz einen Namen. Importieren Sie die Proteinsequenz (im FASTA-Format). Die Software wird eine in silico Verdauung des Datenbankproteins durchführen, um eine Liste von Peptiden zu generieren, die verwendet werden, um die LC-MS-Daten zu durchsuchen.
- Definieren Sie die Verarbeitungsparameter (unter dem Menü Bibliothek). Wählen Sie Electrospray MSE als Datenerfassungstyp aus. Geben Sie im Feld Sperrmasse für Ladung 2 785.8426 für das m/z für das 2+ Ion von [Glu-1]-Fibrinopeptid B (GluFib) ein und klicken Sie auf Fertig.
- Definieren Sie die Workflow-Parameter (unter dem Menü Bibliothek).
- Wählen Sie Electrospray MSE für den Suchtyp. Unter dem Workflow-| Datenbanksuchabfrageüberschrift, wählen Sie das Datenbankprotein aus, das in Schritt 4.2 im Feld Datenbank erstellt wurde.
- Ändern Sie das primäre Digest-Reagenz in unspezifisch, und löschen Sie das Feld "Fixed Modifier Reagent", indem Sie die Strg-Taste gedrückt halten, während Sie auf Carbamidomethyl Cklicken.
- Geben Sie das Ausgabeverzeichnis an, indem Sie zu Optionen navigieren, die | Automatisierungseinrichtung | Identität E. Aktivieren Sie die Kontrollkästchen für Apex 3D und Peptide 3D Output und Ion Accounting Output und geben Sie das gewünschte Verzeichnis an.
- Verarbeiten Sie die Referenzstichprobendaten.
- Erstellen Sie auf der linken Werkzeugleiste des Proteomics-Plattform-Arbeitsbereichs eine neue Platte, indem Sie mit der rechten Maustaste auf Microtiter Plateklicken. Markieren Sie drei Brunnen in der Mikrotiterplatte (eine für jede in Abschnitt 3 entnommene Referenzprobe). Klicken Sie mit der linken Maustaste in einen Brunnen, halten und ziehen Sie ihn auf drei Bohrungen.
- Rechtsklick und wählen Sie Rohdaten hinzufügenaus. Navigieren Sie in dem angezeigten Fenster zu dem Verzeichnis, das die drei Referenzdateien aus Abschnitt 3 enthält, und wählen Sie sie gleichzeitig aus.
- Klicken Sie auf weiter und wählen Sie die in Schritt 4.3 definierten Verarbeitungsparameter aus. Klicken Sie auf weiter und wählen Sie die in Schritt 4.4 definierten Workflowparameter aus. Klicken Sie dann auf Fertigstellen.
- Sobald die Rohdaten, Verarbeitungsparameter und Workflowparameter jedem Brunnen auf der Platte zugewiesen wurden, erscheinen die Bohrungen blau. Wählen Sie die Brunnen, Rechtsklick und wählen Sie Prozess neuesten Rohdaten. Klicken Sie auf die rechte untere Ecke des Fensters, um die Verarbeitung der Daten nachzuverfolgen. Sobald die Meldung Kein auszuführender Auftrag angezeigt wird, ist die Verarbeitung vollständig abgeschlossen.
- Nach Abschluss der Datenverarbeitung werden die Brunnen in der Platte grün. Klicken Sie mit der rechten Maustaste auf die Brunnen und wählen Sie Workflow-Ergebnisse anzeigen. Für jede Referenzdatendatei wird ein separates Fenster geöffnet.
- Überprüfen Sie die Daten, um sicherzustellen, dass die Mehrheit der MS-Signale in den Referenzstichprobendaten erfolgreich Peptiden zugeordnet wurden, die aus der in-silico-Verdauung des von Interesse sindden Proteins vorhergesagt wurden. Abgestimmte Peptide werden im Ausgabespektrum blau gefärbt (Abbildung 4). Doppelklicken Sie auf den OK-Filter, und überprüfen Sie, ob die Prozentuale Abdeckung größer als 99 % ist.
HINWEIS: Bei der Verarbeitung wird die Datenausgabe automatisch mit der Dateierweiterung(raw_data_file_name_IA_final_peptide) in dem in Schritt 4.5 angegebenen Verzeichnis gespeichert. - Importieren Sie die Proteomics-Softwareausgabe in die HDX-Verarbeitungssoftware (Tabelle der Materialien) für zusätzliche Schwellenwerte.
- Klicken Sie in der linken Ecke des HDX-Verarbeitungssoftwarefensters auf Daten. Klicken Sie auf PLGS-Ergebnisse importieren und auf das Add-Symbol. Wählen Sie die verarbeiteten Datendateien aus Schritt 4.9 aus, indem Sie zum entsprechenden Verzeichnis navigieren.
- Klicken Sie auf Weiter und geben Sie die folgenden Parameter an: minimale aufeinander folgende Ionen ≥ 2, Massenfehler = 5 ppm und Dateischwellenwert = 3. Klicken Sie auf Fertigstellen.
- Sobald Sie mit den Schwellenwertparametern zufrieden sind, speichern Sie das HDX-Projekt. Alle HDX-Daten werden zur Analyse und Anzeige in dieses Projekt importiert.
HINWEIS: Die im nächsten Abschnitt beschriebenen Deuterium-Austauschbeispiele müssen mit einem identischen LC-MS-Workflow verarbeitet werden. Bevor Sie mit HDX-Assays (Abschnitt 5) fortfahren, stellen Sie daher sicher, dass die Probenvorbereitung (Abschnitt 2), der Bottom-up-LC-MS-Workflow (Abschnitt 3) und die Datenverarbeitungsworkflows (Abschnitt 4) die gewünschte Reproduzierbarkeit und Sequenzabdeckung des Zielproteins bieten. Wenn einer dieser Prozesse geändert werden muss, um die Abdeckung zu verbessern, wird empfohlen, zu Schritt 2.1 zurückzukehren, neue Referenzproben in Dreifacharbeit vorzubereiten und Abschnitte 2-4 (bei gleichzeitiger Anpassung des Protokolls) zu wiederholen, um sicherzustellen, dass jedes Peptid reproduzierbar erzeugt und erkannt werden kann.
5. Durchführung von HDX-Reaktionen
- Bereiten Sie den Arbeitsbereich auf HDX-Reaktionen vor.
- Vor-aliquot-Quench-Puffer in ordnungsgemäß beschrifteten 0,5 ml-Röhren. Bereiten Sie eine andere Röhre für jeden Zeitpunkt, jede Replikation und jeden zu analysierenden biochemischen Zustand vor. Verwenden Sie das entsprechende Volumen des Löschpuffers aus Schritt 2.2, der erforderlich ist, um den endgültigen pH-Meterstandeines eines 50-L-Anteils der HDX-Reaktion auf einen Wert von 2,3 einzustellen.
- Zentrifugieren Sie kurz die 0,5 ml-Wannen, um den gesamten Löschpuffer auf den Boden des Rohres zu übertragen. Legen Sie die Rohre auf Eis.
- Füllen Sie einen kleinen Dewar mit flüssigem Stickstoff und halten Sie neben arbeitsplatz.
- Bereiten Sie die HDX-Reaktionen vor. Stellen Sie sicher, dass ein ausreichendes Reaktionsvolumen vorhanden ist, um die gewünschte Anzahl von Austauschzeitpunkten zu sammeln (ein Aliquot von 50 L für jeden gewünschten Zeitpunkt). Sammeln Sie mindestens 4 bis 5 Zeitpunkte über 3 x 4 Größenordnungen in der Zeitskala (z. B. bieten Löschzeiten von 15 s, 60 s, 300 s [5 min], 1.800 s [30 min] und 14.400 s [4 h] eine angemessene Abdeckung der Austauschdynamik für die meisten Enzyme).
- Alle deuterated Komponenten (minus Enzym) aus Schritt 1.1 in D2O vormischen.
- Für jeden zu untersuchenden biochemischen Zustand (freies Enzym, Enzym + Liganden, Enzym + Inhibitor usw.) bereiten Sie HDX-Reaktionen zumindest in Dreifache vor.
- Inkubieren Sie die Reaktionsgemische in einem temperaturgeregelten Wasserbad bei 25 °C für 10 min vor Zugabe des Enzyms.
HINWEIS: Das Enzym sollte als konzentrierte Stofflösung (ca. 100 bis 200 M, Schritt 1,6) hergestellt werden, um die Zugabe von Protium in den HDX-Assay zu minimieren. - Wenn Sie das Enzym zu einer Endkonzentration von 1 bis 5 M hinzufügen, starten Sie den Timer. Mischen Sie die Lösung sorgfältig und schnell mit einer 200-L-Pipette, um sicherzustellen, dass das Enzym gleichmäßig in der Probe verteilt ist.
- An den gewünschten Austauschzeitpunkten 50 L-Aliquots aus der HDX-Reaktion entfernen und schnell und gleichmäßig mit dem voralizitierten, eiskalten Löschpuffer in einem 0,5 ml-Rohr mischen.
HINWEIS: Die Mischvolumina und das Mischverfahren müssen so präzise und reproduzierbar wie möglich sein, um sicherzustellen, dass die gewünschte Endabschreckung pL von 2,3 in allen Proben schnell erreicht wird. Die Eiseskälte des Abschreckpuffers wird helfen, den Rückaustausch nach Denaturierung des Enzyms zu minimieren. - Unmittelbar nach dem Löschen der HDX-Probe, kappen Sie die Röhre und Blitz einfrieren in flüssigem Stickstoff.
- Fahren Sie mit dem Sammeln von Zeitpunkten fort, bis alle Tests abgeschlossen sind, und übertragen Sie die Proben zur Lagerung in den -80 °C-Gefrierschrank.
HINWEIS: Dies kann ein Haltepunkt sein. Nach dem Sammeln aller HDX-Zeitpunkte können die Proben bei -80 °C gelagert werden, bis sie für die LC-MS-Analyse bereit sind. Idealerweise sollten für alle biochemischen Zustände am selben Tag dreifache HDX-Reaktionen durchgeführt werden. Mindestens sollten alle replizierten HDX-Reaktionen für einen bestimmten biochemischen Zustand parallel am selben Tag ausgeführt werden.
- Nachdem Sie alle abgeschreckten HDX-Zeitpunkte gesammelt haben, unterwerfen Sie die Proben dem optimierten Bottom-up-LC-MS-Workflow, der wie in Schritt 3.3 beschrieben entwickelt wurde. Injizieren Sie HDX-Proben in einer randomisierten Reihenfolge mit einer angemessenen Anzahl von Rohlingen zwischen den Proben, um sicherzustellen, dass die Übertragung von Peptid minimal ist.
HINWEIS: HDX-Daten müssen nicht im MSE-Modus erfasst werden. Daher sollte das Segment der hohen Kollisionsenergie (Schritt 3.3.7.2) aus dem MS-Betriebszyklus entfernt werden. Dies sollte die einzige Änderung am Workflow sein, die in Schritt 3.3 beschrieben wird. - Bewerten Sie die Qualität der HDX-Daten, während sie gesammelt werden.
- Stellen Sie sicher, dass chromatographische Spitzen im gesamten Ionenchromatogramm der nicht euteierten Referenzproben zur gleichen Retentionszeit in den deuterated Samples erscheinen (wie in Abbildung 3D-F).
- Stellen Sie sicher, dass Massenspektren, die über bestimmte Zeitintervalle aus Referenz- und deuterated-Proben summiert werden, Hinweise auf eine Deuteration aufweisen (d. h. eine Verschiebung der Isotopenhülle einzelner Peptide auf höhere m/z-Werte in den deuterated Samples [Abbildung 6]).
6. Verarbeitung von HDX-Daten
- Importieren Sie die HDX-Daten in das in Schritt 4.11 erstellte HDX-Projekt, indem Sie auf Data | MS-Dateien in der oberen Werkzeugleiste.
- Klicken Sie bei Bedarf auf Neue Zustands- und Neue Exposition, um die biochemischen Zustände (z. B. freies Enzym, Enzym + Liganden usw.) bzw. Deuterium-Expositionszeiten zu definieren, die für die Analyse relevant sind.
- Klicken Sie auf Neu Roh, um die zu analysierenden HDX-Datendateien auszuwählen. Weisen Sie jeder importierten Rohdatendatei die entsprechenden Austauschzeiten und den biochemischen Zustand zu.
HINWEIS: Die Daten können importiert und in Batches oder auf einmal verarbeitet werden. Durch das Hinzufügen von Daten zum Projekt werden keine Analysen rückgängig gemacht, die zuvor in diesem Projekt durchgeführt wurden.
- Nachdem die Datendateien hinzugefügt wurden, klicken Sie auf Fertig stellen, um mit der Datenverarbeitung zu beginnen. Nach einer kurzen Verzögerung fragt die Software, ob der Benutzer die Daten speichern möchte, bevor er fortfährt. Klicken Sie auf ja.
HINWEIS: Die anfängliche Verarbeitung kann bis zu mehreren Stunden dauern, je nachdem, wie viele Proben analysiert werden, wie viele Peptide in der endgültigen Peptidliste (Schritt 4.10), die Größe des chromatographischen Fensters und die Häufigkeit der spektralen Erfassung sind. - Ändern Sie bei Bedarf die Verarbeitungsparameter im Menü Konfiguration, um die Ion-Suchparameter zu ändern. Stellen Sie sicher, dass Sie dieselben Ionensuchparameter für alle Daten in einem bestimmten Projekt verwenden.
7. Analyse und Visualisierung der HDX-Daten
HINWEIS: Sobald die erstmalige Verarbeitung der Rohdaten abgeschlossen ist (Schritt 6.2), wird die HDX-Verarbeitungssoftware Peptide aus der Peptidliste (generiert in Schritt 4.10) in jeder der analysierten Rohdatendateien gefunden haben. Sobald sich die Isotopenverteilung für ein Peptid in der Liste in einer Rohdatendatei befindet, stellt die HDX-Verarbeitungssoftware jedes Isotop mit einem "Stick" dar (wie in Abbildung 6C-E). Die relativen Intensitäten der Stäbe für ein bestimmtes Peptid werden dann verwendet, um die Deuteriumaufnahme relativ zu den Referenzspektren zu berechnen. Während die HDX-Verarbeitungssoftware eine bewundernswerte Aufgabe der korrekten Zuweisung von "Sticks" zu den meisten Peptiden leistet, ist weiterhin eine signifikante manuelle Kuration der Deuterium-Aufnahmewerte erforderlich.
- Analyse der Aufnahmewerte von Peptiddeuterium
- Wählen Sie das erste Peptid in der Peptidliste aus, und öffnen Sie das gestapelte Spektraldiagramm im Menü Ansichten. Scrollen Sie im gestapelten Spektrendiagrammfenster nach oben und unten, um die Massenspektren für das ausgewählte Peptid als Funktion der Deuterium-Austauschzeit zu sehen (Abbildung 7D,E).
- Weisen Sie Sticks nach Bedarf mit Mausklicks zu und dezuweisen, um sicherzustellen, dass die richtige Isotopenverteilung in den Daten gefunden wurde und dass jeder Isotopenspitzen zugewiesen wurde (zugewiesene Sticks werden blau angezeigt). Wenn Sie auf eines der Spektren im gestapelten Spektraldiagramm klicken (Abbildung 7D,E), kann der Benutzer Sticks im aktiven Datenbetrachterfenster zuweisen/entordnen ( Abbildung7C).
- Überprüfen Sie die Stickzuweisungen für jeden Ladezustand, indem Sie den Ladezustand oben im gestapelten Spektralplotfenster umschalten.
- Wiederholen Sie die Schritte 7.1.1-7.1.3 für jeden biochemischen Zustand von Interesse. Der biochemische Zustand kann auch oben im gestapelten Spektraldiagrammfenster umgeschaltet werden. Stellen Sie für die genauesten Deuteriumaufnahmedifferenzmessungen sicher, dass Stöcke für jeden biochemischen Zustand für den gleichen Ladungssatz zugewiesen werden.
- Wiederholen Sie die Schritte 7.1.1-7.1.4 für jedes Peptid in der Peptidliste.
- Überprüfen Sie die Standardabweichung der Peptid-Deuterium-Aufnahmewerte mithilfe der Abdeckungskarte.
- Greifen Sie über das Menü Ansichten auf die Abdeckungskarte zu, die jedes Peptid in der Peptidliste anzeigt, die entlang der Aminosäuresequenz des von Interesse sindden Proteins abgebildet ist (Abbildung 8C). Färben Sie die Peptide nach relativer Standardabweichung (Einheiten von Da).
- Suchen Sie die Karte visuell nach Ausreißerpeptiden mit hoher relativer Standardabweichung. Klicken Sie auf die Ausreißerpeptide in der Coverage-Karte, um die gestapelten Spektraldiagramme (Abbildung 8B) und das Datenbetrachterfenster (Abbildung 8A) mit dem Zielpeptid aufzufüllen.
- Überprüfen Sie mit dem gestapelten Spektraldiagramm sorgfältig, ob alle Ladezustände und alle Zeitpunkte des Ausreißerpeptids entsprechend zugeordnete Sticks haben.
HINWEIS: Am häufigsten weisen Peptide mit großen Standardabweichungen (>0,3 Da) Isotopspitzen auf, die von der Software nicht angemessen zugeordnet wurden (siehe Abbildung 8B). Durch das Zuweisen fehlender Sticks kann in der Regel die relative Standardabweichung eines Peptids auf <0,3 Da reduziert werden. - Ausblenden des Peptids aus der Liste, wenn die relative Standardabweichung nicht auf weniger als 0,3 Da reduziert werden kann.
- Exportieren Sie HDX-Differenzdaten zwischen zwei biochemischen Zuständen für die Kartierung auf ein strukturelles Modell des Proteins von Interesse.
- Zeigen Sie die Interessendifferenz in der Abdeckungskarte an. Klicken Sie mit der rechten Maustaste auf die Abdeckungskarte, um die Differenzdaten in eine .csv Datei zu exportieren. Exportieren der Zustandsdaten (im Format .csv), indem Sie zu Data | Exportieren von Statusdaten in der Hauptsymbolleiste.
HINWEIS: Eine angemessene Formatierung der Differenzdaten und Zustandsdatendateien finden Sie in der Unterstützungsinformation. - Importieren Sie die Differenzdaten, die Zustandsdaten und die pdb-Datei des von Interesse sindden Proteins in Deuteros28. Wählen Sie das 99%-Konfidenzintervall aus, wählen Sie Summe aktivierenaus, und verarbeiten Sie die Daten.
HINWEIS: MATLAB muss auf dem PC installiert sein, um Deuteros ausführen zu können. Deuteros verwendet die Replikationsmessungen im Datensatz, um die Standardabweichung der Aufnahmedaten für jedes Peptid zu berechnen. Diese Standardabweichung wird verwendet, um das Konfidenzintervall für einen signifikanten Austausch zu definieren, der auf den Diagrammen angezeigt wird. - Wählen Sie unter PyMOL-Optionen exportaufnahme- | Exportieren, um ein Pymol-Skript zu generieren, um Regionen mit signifikanten Austauschunterschieden auf die pdb-Struktur des Proteins von Interesse mit PyMOL-Software abzubilden.
ANMERKUNG: Bei Verwendung des in diesem Protokoll beschriebenen Workflows beträgt das 99 %-Konfidenzintervall für signifikante Deuteriumaufnahmedifferenz für ein bestimmtes Peptid zu einem bestimmten Zeitpunkt in der Regel 0,3 x 0,5 Da. Das 99%-Konfidenzintervall für die Differenz, die über alle Wechselkurszeitpunkte summiert wird, beträgt in der Regel 0,7 bis 1,0 Da.
- Zeigen Sie die Interessendifferenz in der Abdeckungskarte an. Klicken Sie mit der rechten Maustaste auf die Abdeckungskarte, um die Differenzdaten in eine .csv Datei zu exportieren. Exportieren der Zustandsdaten (im Format .csv), indem Sie zu Data | Exportieren von Statusdaten in der Hauptsymbolleiste.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Es ist notwendig, die Qualität der proteolytischen Verdauung und die Reproduzierbarkeit des Workflows für jeden Satz von Probeninjektionen zu bewerten. Daher ist es vor der Durchführung von HDX-MS-Assays wichtig, wirksame Bedingungen für die Proteolyse des Proteins von Interesse, für die Trennung von Peptiden mittels Reverse-Phase-Flüssigkeitschromatographie und Gasphasenionenmobilität sowie für den Nachweis von Peptiden mit MS zu schaffen. Zu diesem Zweck sollten zunächst die Referenzproben für das Protein von...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Der in diesem Protokoll vorgestellte HDX-MS-Workflow bietet eine bemerkenswert robuste Plattform zur Kartierung der räumlichen Verteilung strukturdynamischer Elemente in Proteinen und zur Untersuchung, wie sich diese Dynamik als Reaktion auf Störungen (Ligandenbindung, Enzym-Mutagenese usw.) ändert. HDX-MS bietet mehrere deutliche Vorteile gegenüber anderen strukturbiologischen Ansätzen, die häufig zur Untersuchung der Konformationsdynamik verwendet werden. Vor allem werden nur geringe Mengen Aniis benötigt. Mithi...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Wir haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde vom Natural Sciences and Engineering Research Council of Canada, dem Fonds de Recherche du Quebec Nature et Technologie, der Canadian Foundation for Innovation und den Start-up-Fonds der McGill University unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| [glu-1]-fibrinopeptide B (Glu-Fib) | BioBasic | NA | |
| 0.5 mL Amicon Ultracel 10k centrifugal filtration device (Millipore) | Milipore Sigma | UFC501096 | |
| acetonitrile | Fisher | A955-1 | |
| AMP-PNP | SIGMA | A2647-25MG | |
| ATP | SIGMA | a2383-5G | |
| D2O | ALDRICH | 435767-100G | |
| formic acid | Thermo Fisher | 28905 | |
| guanidine-HCl | VWR | 97063-764 | |
| HEPES | Fisher | BP310-1 | |
| Magnesium chloride | SiGMA-Aldrich | 63068-250G | |
| Potassium chloride | BioBasic | PB0440 | |
| potassium phosphate | BioBasic | PB0445 | |
| TCEP Hydrochloride | TRC Canada | T012500 | peptide was synthesized upon request |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| software | |||
| Deuteros | Andy M C Lau, et al | version 1.08 | |
| DynamX | Waters | version 3.0 | |
| MassLynx | Waters | version 4.1 | |
| Protein Lynx Global Server (PLGS) | Waters | version 3.0.3 | |
| PyMOL | Schrödinger | version 2.2.2 | |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| Instrument and equipment | |||
| ACQUITY UPLC BEH C18 analytical Column | Waters | 186002346 | |
| ACQUITY UPLC BEH C8 VanGuard Pre-column | Waters | 186003978 | |
| ACQUITY UPLC M-Class HDX System | Waters | ||
| HDX Manager | Waters | ||
| microtip pH electrode | Thermo Fisher | 13-620-291 | |
| Waters Enzymate BEH column or Pepsin solumn | Waters | 186007233 | |
| Waters Synapt G2-Si | Waters |
Referenzen
- Karplus, M., McCammon, J. A. Molecular dynamics simulations of biomolecules. Nature Structural & Molecular Biology. 9 (9), 646-652 (2002).
- Campbell, E., et al. The role of protein dynamics in the evolution of new enzyme function. Nature Chemical Biology. 12 (11), 944-950 (2016).
- Tokuriki, N., Tawfik, D. S. Protein dynamism and evolvability. Science. 324 (5924), 203-207 (2009).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chemical Society Reviews. 40 (3), 1224(2011).
- Katta, V., Chait, B. T. Conformational changes in proteins probed by hydrogen-exchange electrospray-ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 5 (4), 214(1991).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Science. 2 (4), 522(1993).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Xiao, K., et al. Revealing the architecture of protein complexes by an orthogonal approach combining HDXMS, CXMS, and disulfide trapping. Nature Protocols. 13 (6), 1403-1428 (2018).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Masson, G. R., et al. Recommendations for performing, interpreting and reporting hydrogen deuterium exchange mass spectrometry (HDX-MS) experiments. Nature Methods. 16 (7), 595-602 (2019).
- Liu, J., et al. An Efficient Site-Specific Method for Irreversible Covalent Labeling of Proteins with a Fluorophore. Scientific Reports. 5, 16883(2015).
- Marion, D., et al. Overcoming the overlap problem in the assignment of proton NMR spectra of larger proteins by use of three-dimensional heteronuclear proton-nitrogen-15 Hartmann-Hahn-multiple quantum coherence and nuclear Overhauser-multiple quantum coherence spectroscopy: application to interleukin 1.beta. Biochemistry. 28 (15), 6150-6156 (1989).
- Balasubramaniam, D., Komives, E. A. Hydrogen-exchange mass spectrometry for the study of intrinsic disorder in proteins. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1834 (6), 1202-1209 (2013).
- Goswami, D., et al. Time window expansion for HDX analysis of an intrinsically disordered protein. Journal of the American Society for Mass Spectrometry. 24 (10), 1584-1592 (2013).
- Keppel, T. R., Howard, B. A., Weis, D. D. Mapping Unstructured Regions and Synergistic Folding in Intrinsically Disordered Proteins with Amide H/D Exchange Mass Spectrometry. Biochemistry. 50 (40), 8722-8732 (2011).
- Mitchell, J. L., et al. Functional Characterization and Conformational Analysis of the Herpesvirus saimiri Tip-C484 Protein. Journal of Molecular Biology. 366 (4), 1282-1293 (2007).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Hydrogen/deuterium exchange mass spectrometry with top-down electron capture dissociation for characterizing structural transitions of a 17 kDa protein. Journal of the American Chemical Society. 131 (35), 12801-12808 (2009).
- Habibi, Y., Uggowitzer, K. A., Issak, H., Thibodeaux, C. J. Insights into the Dynamic Structural Properties of a Lanthipeptide Synthetase using Hydrogen-Deuterium Exchange Mass Spectrometry. Journal of the American Chemical Society. 141 (37), 14661-14672 (2019).
- Hebling, C. M., et al. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Analytical Chemistry. 82 (13), 5415-5419 (2010).
- Duc, N. M., et al. Effective application of bicelles for conformational analysis of G protein-coupled receptors by hydrogen/deuterium exchange mass spectrometry. Journal of the American Society for Mass Spectrometry. 26 (5), 808-817 (2015).
- Canul-Tec, J. C., et al. Structure and allosteric inhibition of excitatory amino acid transporter 1. Nature. 544 (7651), 446-451 (2017).
- Reading, E., et al. Interrogating Membrane Protein Conformational Dynamics within Native Lipid Compositions. Angewandte Chemie International Edition. 56 (49), 15654-15657 (2017).
- Hentze, N., Mayer, M. P. Analyzing protein dynamics using hydrogen exchange mass spectrometry. Journal of Visualized Experiments. (81), e50839(2013).
- Lento, C., et al. Time-resolved ElectroSpray Ionization Hydrogen-deuterium Exchange Mass Spectrometry for Studying Protein Structure and Dynamics. Journal of Visualized Experiments. (122), e55464(2017).
- Schowen, K. B., Schowen, R. L. Solvent isotope effects on enzyme systems. Methods in Enzymology. 87, 551-606 (1982).
- Lau, A. M. C., Ahdash, Z., Martens, C., Politis, A. Deuteros: software for rapid analysis and visualization of data from differential hydrogen deuterium exchange-mass spectrometry. Bioinformatics. 35 (7), 3171-3173 (2019).
- Houde, D., Berkowitz, S. A., Engen, J. R. The utility of hydrogen/deuterium exchange mass spectrometry in biopharmaceutical comparability studies. Journal of Pharmaceutical Sciences. 100 (6), 2071(2011).
- Burkitt, W., O'Connor, G. Assessment of the repeatability and reproducibility of hydrogen/deuterium exchange mass spectrometry measurements. Rapid Communications in Mass Spectrometry. 22 (23), 3893-3901 (2008).
- Rob, T., Gill, P. K., Golemi-Kotra, D., Wilson, D. J. An electrospray ms-coupled microfluidic device for sub-second hydrogen/deuterium exchange pulse-labelling reveals allosteric effects in enzyme inhibition. Lab on a Chip. 13, 2528-2532 (2013).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten