Method Article
Ein Drosophila-Modell zur Untersuchung der Wund-induzierten Polyploidisierung
In diesem Artikel
Zusammenfassung
Wund-induzierte Polyploidisierung ist eine konservierte Gewebereparaturstrategie, bei der Zellen in der Größe wachsen, anstatt sich zu teilen, um den Zellverlust zu kompensieren. Hier ist ein detailliertes Protokoll, wie man die Fruchtfliege als Modell zur Messung der Ploidie und ihrer genetischen Regulation bei der epitheliale Wundreparatur verwendet.
Zusammenfassung
Polyploidie ist ein häufiges Phänomen, dessen Auswirkungen auf die Gesundheit und Krankheit des Organismus noch immer schlecht verstanden werden. Eine Zelle ist als polyploid definiert, wenn sie mehr enthält als die diploide Kopie ihrer Chromosomen, die das Ergebnis der Endoreplikation oder Zellfusion ist. Bei der Gewebereparatur wurde eine wundinduzierte Polyploidisierung (WIP) als konservierte Heilungsstrategie von Fruchtfliegen bis wirbeltierchen festgestellt. WIP hat mehrere Vorteile gegenüber der Zellproliferation, einschließlich der Resistenz gegen onkogenes Wachstum und genotoxischen Stress. Die Herausforderung bestand darin, herauszufinden, warum polyploide Zellen entstehen und wie diese einzigartigen Zellen funktionieren. Bereitgestellt wird ein detailliertes Protokoll zur Untersuchung von WIP im erwachsenen Fruchtfliegenepithel, bei dem polyploide Zellen innerhalb von 2 Tagen nach einer Stichwunde erzeugt werden. Unter Ausnutzung des umfangreichen genetischen Werkzeugbaus von D. melanogaster wurden die Gene identifiziert, die zur Initiierung und Regulierung von WIP erforderlich sind, einschließlich Myc. Fortgesetzte Studien mit dieser Methode können zeigen, wie andere genetische und physiologische Variablen wie Geschlecht, Ernährung und Alter die Funktion von WIP regulieren und beeinflussen.
Einleitung
Drosophila melanogaster ist ein attraktives Modellsystem, um die zellulären und molekularen Mechanismen der epitheliaalen Wundreparatur zu untersuchen. Wie bei Säugetieren hängen die verwendeten Gewebereparaturmechanismen sowohl vom Gewebe als auch von seinem Entwicklungsstadium ab. Die narbenlose Wundheilung erfolgt im Fruchtfliegen-Embryo, wo sich eine Actomyosin-"Geldbörsenschnur" an der epitheliaalen Vorderkante bildet, die es der Wunde ermöglicht, sich nahtlos zu schließen1,2. Die postembryonale Wundheilung bei Larven, Pupas und erwachsenen Fruchtfliegen führt zu extrazellulären Matrix-Remodeling, Melaninnarbenbildung und Epithelzellwachstum3,4,5,6. Die Epithelzellen nehmen durch Zellfusion und Endzyklus an Größe zu, ein unvollständiger Zellzyklus, der die Mitose3,4,7,8umgeht. Dadurch wird der Zellverlust durch polyploidzelles Wachstum anstelle der Zellteilung kompensiert. Die erwachsenen Fliegenhintergut, Midgut, und follikuläre Epithel verlassen sich auch auf polyploide Zellwachstum, um Zellverlust nach Gewebeschäden9,10,11zu kompensieren.
Polyploidie ist ein bekannter Aspekt der Entwicklung des Organismus bei Pflanzen und Insekten, aber in den letzten Jahren ist es offensichtlicher geworden, dass Polyploidie eine konservierte Gewebereparaturstrategie bei Wirbeltieren ist12. Der Zebrafisch, der die Fähigkeit hat, sein Herz zu regenerieren, beruht auf polyploidem Zellwachstum, um beschädigtes Epikardinium zu heilen13. Polyploidie trägt auch zur Regeneration der Säugetierleber und Zur Reviertubie epithelische Reparatur nach akuter Verletzung14,15bei. In diesen Beispielen werden polyploide Zellen durch Endoreplikation entweder über Endocycle oder Endomitose erzeugt, was aufgrund eines Blocks in der Zytokinese12zu einer binuklierten Zelle führt. Das Rätsel ist, warum polyploide Zellen während der Wundreparatur entstehen und wie Polyploidie die Gewebefunktion beeinflusst. Jüngste Studien haben neue Erkenntnisse über die Frage geliefert, ob Polyploidie einen heilenden Vorteil oder Nachteil bietet. Im Zebrafisch Epicardium, Polyploidie verbesserte die Geschwindigkeit der Wundheilung13. Bei D. melanogaster hindgut und Säugetierleber, Polyploidie wurde festgestellt, dass Schutz gegen onkogenes Wachstum11,14. Bei erwachsenen Fliegenepithel, Es wurde vor kurzem festgestellt, dass Polyploidie Wundreparatur in Gegenwart von genotoxischem Stressermöglicht 16. Endoreplication ist resistent gegen DNA-Schäden, so dass Wundheilung ermöglicht, wenn Zellproliferation sonst gefährdet wäre17. Bei Kardiomyozyten in Maus- und Zebrafischherzen verlangsamt die Polyploidie jedoch die Heilung, was zu einer verbesserten Narbenbildung18,19führt. Daher kann Polyploidie je nach Organ und/oder Zelltyp eine vorteilhafte oder schädliche Gewebereparaturstrategie sein. Die Zugänglichkeit der D. melanogaster Genetik in Verbindung mit der Analyse der Wund-induzierten Polyploidisierung (WIP) macht sie zu einem idealen Modellsystem zur Aufklärung der molekularen und zellulären Mechanismen, die diese Wundheilungsstrategie leiten.
Hier stellen wir ein Protokoll zur Analyse von WIP im erwachsenen D. melanogaster Epithel vor. Enthalten sind Anweisungen für Fruchtfliegenverletzungen, Sezieren, Immunflecken, Montage, Bildgebung und Analyse der Reepithelisierung, Zellfusion und Endoreplikation (Ploidy). Die Bildgebungs- und Ploidienanalyse kann auch an andere Modelle angepasst werden, um zu testen, ob WIP auftritt. Es sei darauf hingewiesen, dass bei einer Zunahme des Kern-DNA-Gehalts oft eine entsprechende Zunahme der nuklearen Größe zu verzeichnen ist. Es gibt jedoch viele Beispiele in der Biologie, wo die nukleare Größe keine entsprechende Veränderung der Ploidie20widerspiegelt. Noch mehr Vorsicht ist geboten, wenn die Kerngröße im Kontext einer Wundumgebung interpretiert wird, in der sich Zellen oft ausbreiten oder dehnen, um die Wundstelle zu bedecken. Daher besteht der einzige endgültige Beweis für eine Veränderung der Ploidie darin, den DNA-Gehalt nach dieser Methode (oder anderen, wie der Sequenzierung des gesamten Genoms) zu messen21. Diese Methode erhöht die Eignung des erwachsenen D. melanogaster Bauchepithels als Modell, um die Rolle und Regulierung der Polyploidie bei der Wundreparatur zu untersuchen.
Protokoll
1. Inszenierung und Verwundung von erwachsenen Fruchtfliegen
- Wählen Sie D. melanogaster Stamm der Wahl (d.h. Epi-Gal4/ UAS-Stamm, siehe Tabelle der Materialien).
HINWEIS: Hier wird das Gal4/UAS-System verwendet, um epitheliale spezifische Genexpression (epi-Gal4) eines Gens oder RNAi nachgeschaltet von UAS zu ermöglichen. Diese Studie verwendet fluoreszierendes Membranprotein (UAS-Cd8.mRFP), mitotische Induktoren (UAS-fzrRNAi, UAS-stg) und WIP-Hemmer (UAS-E2F1RNAi; UAS-RacDN). - Sammeln Sie zwei Fläschchen mit 10-15 neu geschlossenen weiblichen Fruchtfliegen und altern Sie auf frischen Fläschchen bei 25 °C bis 3-5 Tage alt. Eine Durchstechflasche dient als unverletzte Kontrolle und die andere Durchstechflasche wird wie unten beschrieben verwundet. Die weiblichen Fliegen sollten bei Männchen gehalten werden (5 /durchsschläfst).
- Um die Fliegen zu wickeln, montieren Sie mehrere Stifthalter mit jeweils einem einzigen 0,10 mm Edelstahlstift. Stellen Sie sicher, dass das scharfe Ende des Stifts nach außen zeigt. Pins können leicht biegen oder Chip nach Punktion der Fliege und haken oder beschädigte Stifte sollten verworfen werden.
- Anästhetisieren Sie gealterte weibliche Früchte fliegen auf einemCO2-Fliegenpadunter einem Stereomikroskop und richten Sie sie mit einem Pinsel in eine Reihe aus. Tragen Sie eine Schutzbrille, halten Sie den Stifthalter in der einen Hand und zangen in der anderen, verwenden Sie Zangen, um eine Fliege mit ihrem ventralen Bauch nach oben zu positionieren.
- Punktion der erwachsenen weiblichen Fliegen innerhalb der epithelialen Pleuritregion von Tergith A4 auf beiden Seiten der ventralen Mittelliniensternite (Abbildung 1A). Das Punktieren dieser ventralen Region bietet optimalen Raum weg von den Sezierstellen, an denen Gewebekanten durch mechanische Verarbeitung gerissen werden.
- Zurück verwundete Fliegen in die Nahrungsflasche und Alter zu gewünschten Tag nach Verletzung (dpi). Die epitheliale Wundheilung beginnt bei 1 dpi und endet mit 3 dpi. Endoreplication erreicht Spitzenwerte bei 2 dpi, was ideal für den EdU-Assay ist (Abschnitt 4, Abbildung 2).
2. Fliegen Bauchsektion
HINWEIS: In diesem Schritt ist es wichtig, das ventrale Bauchgewebe nicht mit den Sezierwerkzeugen zu berühren, da es die Integrität des Epithels beeinträchtigt.
- Besorgen Sie sich alle benötigten Materialien für die Zerlegung: Grace-Lösung, Zange, Vanna-Federschere, 0,10-mm-Stifte, Sezierplatten, 9 Brunnenglas-Sezierschale, fixative Lösung (4% Paraformaldehyd in 1x PBS), 1x PBS, Tücher, Pipetten und Spitzen für 30 m und Handschuhe (siehe Tabelle der Materialien).
- Bestätigen Sie, dass Fliegen erfolgreich durch anästhesierende verwundete Fliegen auf einemCO2-Fliegenpadunter einem Stereomikroskop verletzt wurden, und überprüfen Sie das Vorhandensein der Wundnarbe (d. h. eine Melanin-Stelle am Bauch, siehe Abbildung 1B). Entsorgen Sie alle Fliegen aus der experimentellen Gruppe, die nicht erfolgreich verwundet wurden.
- Um mit der Zerlegung zu beginnen, füllen Sie einen Brunnen einer 9 Brunnenglas-Sezierschale mit Graces Lösung. Verwenden Sie ein Paar Zangen, um eine verwundete weibliche Fliege an der rückenden Seite des Thorax zu erfassen und die Fliege in den Brunnen zu tauchen, der Graces Lösung enthält.
- Mit Zangen in der gegenüberliegenden Hand, ohne den Thorax loszulassen, punktieren Sie die dorsale Nagelhaut unter tergite A6 und ziehen Sie die Nagelhaut vom hinteren Ende der Fruchtfliege. Die inneren Organe (Eierstock und Darm) kommen in der Regel bei diesem Schritt heraus. Wenn nicht, drücken Sie vorsichtig auf die Rückenseite des Bauches mit der Zange, um die verbleibenden Organe herauszupressen und in einen leeren Brunnen zu werfen.
- Schnappen Sie den vollen Bauch an der Thorax-Kreuzung oberhalb von Tergite A2 mit der Zange ab und übertragen Sie den Bauch in einen leeren Brunnen, der die Lösung von Grace mit 100 L enthält.
- Wiederholen Sie die Schritte 2.3-2.5, bis alle Fliegenabdomen seziert sind.
- Reduzieren Sie das Volumen der Grace-Lösung auf 30 l im gut gepoolten, sezierten Bauch.
- Filet die Bauchmuskeln öffnen, indem Sie den Bauch auf der Rückenseite mit der Zange in der einen Hand positionieren und dann mit der anderen Hand die untere Klinge von Vannas Federschere in die Bauchhöhle einsetzen. Schneiden Sie entlang der dorsalen Mittellinie, bis der Bauch vollständig geöffnet ist, was bis zu drei Schnitte erfordern kann(Abbildung 1C, 1D).
- Richten Sie eine trockene Sesektierungsplatte mit vier 0,10 mm Stiften pro Bauchmontagebereich ein. Jede 35 mm Sezierenvon Platte kann bis zu sieben Montagebereiche passen. Pipette 30 L von Grace Lösung auf jedem Montagebereich und übertragen Sie einen filetierten Bauch auf jedes Tröpfchen.
- Pin die Filet bauche auf die Schale auf den vier dorsalen Ecken (Abbildung 1E). Stellen Sie sicher, dass das Gewebe flach liegt, ohne das Bauchgewebe zu zerreißen oder zu überdehnen.
- Um das Gewebe zu fixieren, Pipette aus der Grace-Lösung und fügen Sie 30 l der Fix-Lösung, um den angehefteten Bauch.
VORSICHT: Tragen Sie Handschuhe beim Umgang mit der Fixlösung, da Paraformaldehyd giftig ist. - Wiederholen Sie die Schritte 2.10-2.11, bis alle filetierten Abdomen auf die Sezierenderplatte gepinnt sind.
- Platzieren Sie ein Bandetikett auf der Unterseite jeder Schale, um jede Kontroll- und Versuchsgruppe zu markieren. Feste Proben für 30-60 min bei Raumtemperatur (RT).
- Fixlösung abwaschen, indem man auf 1,5 ml 1x PBS auf jede Platte rohriert. Entsorgen Sie Fixlösung und Kunststoffe in geeigneten flüssigen oder trockenen chemischen Abfallbehältern nach institutionellen Richtlinien.
- Spülplatten 2x mit 1,5 ml 1x PBS waschen und festes Gewebe mit 1,5 ml 1x PBS in einem Kunststoffbehälter mit Deckel aufbewahren. Fügen Sie eine Schicht feuchten Papiertuchs an den Boden des Behälters und lagern Sie Proben bei 4 °C, bis sie innerhalb von 1 Woche nach der Zerlegung immunstainen können.
3. Immunfluoreszenz
- Reagenzien frisch vorbereiten (siehe Materialtabelle):Waschpufferlösung (0,3% Triton X 100, 0,3% BSA in 1x PBS). Restwaschpuffer kann bei 4 °C gespeichert und für die Dauer des 2-tägigen Färbeprotokolls verwendet werden. Bereiten Sie genügend primäre Antikörperlösung pro Test (Abbildung 2) mit Anti-FasIII (1:50 Maus Anti-Fasciclin-III) in Waschpuffer entweder Anti-Grh (1:300 Affinität gereinigt Kaninchen Anti-Grainyhead8) oder Anti-RFP (1:1,000 Kaninchen Anti-RFP). Primäre Antikörperlösungen können bei 4 °C gespeichert und mehrfach wiederverwendet werden, bis das Signal deutlich reduziert wird.
- Permeabilisieren Sie Gewebe, indem Sie 1x PBS abpfeifen, 1,5 ml Waschpuffer hinzufügen und mindestens 30 min auf einem Orbital-Shaker (80 Rpm) bei RT inkubieren.
- Waschpuffer und Fleckengewebe über Nacht mit 1,5 ml Primärantikörperlösung entfernen, auf einem Orbitalshaker (80 Rpm) bei 4 °C inkubieren. Sammeln Sie die primäre Antikörperlösung und sparen Sie in einem Rohr bei 4 °C für zukünftige Experimente.
- Zuerst die Probe schnell mit 1x PBS abspülen und dann 3x mit 1,5 ml Waschpuffer waschen. Für jede Wäsche Proben bei RT auf einem Orbitalshaker für mindestens 30 min inkubieren.
- Während der letzten Wäsche die sekundäre Antikörperlösung vorbereiten: 1:1.000 Esel Anti-Kaninchen Alexa 488 oder 568 und 1:1.000 Ziegen-Anti-Maus Alexa 488 oder 568 (oder Fluorophore nach Wahl) im Waschpuffer.
- Entfernen Sie Waschpuffer und Fleckengewebe mit 1,5 ml sekundärer Antikörperlösung. Abdecken Sie Proben mit Aluminiumfolie und inkubieren Sie auf einem Orbital-Shaker bei RT für 3 h. Alternativ können Proben über Nacht bei 4 °C auf einem Orbital-Shaker inkubiert werden.
- Waschen Sie Proben, indem Sie zuerst sekundäre Antikörperlösung entsorgen und dann die Probe schnell mit 1x PBS spülen, gefolgt von drei Waschungen mit 1,5 ml Waschpuffer. Für jede Wäsche Proben bei RT auf einem Orbitalshaker für mindestens 30 min inkubieren.
- Bereiten Sie die DAPI-Lösung vor, indem Sie Die DAPI im Waschpuffer auf 10 g/ml verdünnen. Nach der Endwäsche, Fleckenproben mit 1,5 ml DAPI-Lösung inkubieren bei RT für 30 min.
- Verwerfen Sie die DAPI-Lösung und spülen Sie Proben 2x in 1,5 ml 1x PBS. Gebeiztes Gewebe in 1,5 ml 1x PBS im Dunkeln, mit Aluminiumfolie bedeckt, bei 4 °C lagern, bis es auf einem Glasschlitten mit Deckelbeschlag montiert werden kann. Der Montageschritt sollte innerhalb von 1 Woche durchgeführt werden.
4. Zellzyklusaktivität (EdU-Assay)
- Erstellen Sie eine 10 mM EdU-Lagerlösung aus Dem Click-iT-Kit (siehe Tabelle der Materialien), indem Sie EdU-Pulver in dH20 auflösen und bis zur vollständigen Auflösung 15 min mischen. Die Stammlösung kann aliquoted (250 l pro Tube) und bei -80 °C gelagert werden.
- Feed fliegt EdU, indem zuerst edU-Bestand auf 5 mM in dH2O verdünnt wird. Trockene Hefe hinzufügen, bis die Lösung trüb ist und kurz Wirbel zu mischen. Schneiden Sie eine 0,5 ml Rohrkappe ab und legen Sie sie an den Boden der Fliegenfutter-Durchstechflasche. Schieben Sie die Kappe in das Essen, so dass es stabil ist.
- Anästhesisieren Sie die Fliegen und übertragen Sie 3-5 Tage alte Fliegen in die Durchstechflasche. Tippen Sie auf die Fliegen auf eine Kante, so dass keine in der Kappe stecken.
- Pipette 75 l Hefe-EdU-Lösung in die Kappe. Fliegen sollten jeden Tag mit frischer Hefe-EdU-Lösung gefüttert und jeden zweiten Tag in eine frische Durchstechflasche mit einer Kappe gebracht werden, um sicherzustellen, dass die Fliegen nicht im Boden der Durchstechflasche stecken bleiben.
- Um Fliegen zu übertragen, kippen Sie auf eine neue Durchstechflasche mit einer Kappe, legen Sie Fliegen in den Schlaf, tippen Sie auf Fliegen zur Seite und fügen Sie frische Hefe-EdU-Lösung hinzu.
- Am dritten Tag, verletzen Sie die Fliegen und weiterhin Hefe-EdU bis zur Zerlegung bei 2 dpi zu füttern (Abbildung 4A). Siehe Protokollabschnitt 2 für Sezier- und Fixierungsmethoden.
- Herstellung von EdU-Färbereagenzien: Waschpuffer (0,3% Triton X 100, 0,3% BSA in 1x PBS), Permeabilisationspuffer (0,5% Triton X 100 in 1x PBS), Sperrpuffer (3% BSA in 1x PBS) und Herstellung von Reagenzien aus dem EdU-Assay-Kit (siehe Tabelle der Materialien),einschließlich 1x Reaktionspuffer und 1x Reaktionspuffer-Additiv nach Anweisung des Herstellers.
- Proben 1 h waschen, bei RT in 1,5 ml Waschpuffer mischen.
- Fügen Sie 1,5 ml Permeabilisationspuffer hinzu und inkubieren Sie Proben für 20 min.
HINWEIS: Tauen und bereiten Reaktionscocktail-Lösung mit einem Volumen von 500 l / Platte. - Waschen Sie Proben 1x schnell mit 1x PBS und dann 3x schnell mit 1 ml Sperrpuffer.
- Pipette den verbleibenden Sperrpuffer abschalten und 500 L Reaktionscocktaillösung pro Platte hinzufügen. Wirbelplatten, um sicherzustellen, dass Gewebe vollständig abgedeckt sind. Inkubieren Sie in einer Schublade im Dunkeln für 1 h bei RT.
- Proben 1x schnell mit 1,5 ml Sperrpuffer waschen.
- Stain mit 1,5 ml DAPI-Lösung bei 1:5.000 im Waschpuffer für 30 min.
- 2x mit 1x PBS schnell waschen, in Folie einwickeln und im Dunkeln bei 4 °C lagern, bis probenfertig innerhalb von 3 Tagen gelagert werden.
5. Beflecktes Gewebe montieren
- Erhalten Sie alle erforderlichen Materialien für die Montage: Glasschlitten, Glasabdeckungen, klarer Nagellack, Montagemedien, ein Paar Zangen und Tücher.
- Um gefärbtes Fliegengewebe zu montieren, entpinen Bauch von der Sezieren Platte mit Zangen unter dem Stereomikroskop. Übertragen Sie Gewebe auf einen Glasdeckel, indem Sie das Gewebe mit der Zange sanft an seinen Rückenflanken greifen und darauf achten, dass der ventrale Bereich nicht mit Zangen berührt wird.
- Unter dem Stereomikroskop das Bauchgewebe so ausrichten, dass die Innenseite nach unten zum Deckelschlupf (d.h. die äußere Nagelhaut/Borsten sind nach oben). Ziehen Sie die orientierten Bauchanten an den Rand des Medientröpfchens mit den Zangen. Die Oberflächenspannung wird dazu beitragen, das Gewebe flach zu halten (Abbildung 1F).
HINWEIS: Es ist hilfreich für die Bildgebung, die Bauchmuskeln in einer Spalte oder Zeile in diesem Stadium zu organisieren. - Beschriften Sie eine Glasrutsche (d. h. steuerung oder experimentell) und nehmen Sie den Deckelaufschlag auf, indem Sie die Folie langsam näher an den Deckelschlupf bringen. Drehen Sie den Schlitten um und blots sanft mit einem Wisch, um überschüssige Montagemedien zu entfernen.
- Versiegeln Sie die Ränder des Coverslipmitmitals mit klarem Nagellack und wiederholen Sie ihn für alle verbleibenden Versuchsgruppen. Bewahren Sie Dias in einer Diabox bei 4 °C auf, bis sie bildbereit sind.
6. Bildgebung und Verarbeitung
- Stellen Sie sich den fliegenden Bauchwundenbereich vor, indem Sie zuerst die Melaninnarbe mit einem konfokalen Mikroskop (Abbildung 1B), entweder einem Punktscanner oder einer strukturierten Beleuchtung (ApoTome) mit einem 40-fachen Öl- oder Trockenobjektiv lokalisieren.
- Überprüfen Sie die Belichtung auf jedem Kanal, um sicherzustellen, dass das Signal unter der Sättigung liegt. Die Bildeinstellungen sollten auf der hellsten Stichprobengruppe basieren. Dies ist besonders wichtig für die Ploidie-Analyse, da der DAPI-Kanal im linearen Bereich bleiben muss, um den DNA-Gehalt genau zu messen.
- Nehmen Sie ein vollständiges Z-Stack-Bild in allen drei Kanälen mit einem optimalen Abstand von mindestens 0,50 m zwischen den Slices auf. Speichern Sie aufgenommene Bilder und öffnen Sie die Datei im Bildanalyseprogramm Fidschi (auch bekannt als ImageJ).
- Erstellen Sie für jedes Bild eine Z-Stack-Projektion mit der Option Summe der Slices für alle Kanäle.
- Drehen Sie die Bilder nach Bedarf, um sicherzustellen, dass die Kerne horizontal über Bilder angeordnet sind (Abbildung 3A und Abbildung 4E).
- Schneiden Sie alle Bilder auf eine rechteckige Auswahl von 300 x 300 m um die Wundstelle oder das Zentrum der unverletzten Kontrolle zentriert. Identifizieren Sie den Bereich, indem Sie ein Rechteck zeichnen und Bearbeiten | Auswahl | Angeben . Stellen Sie sicher, dass skalierte Einheiten in Mikrometern vorhanden sind, um sicherzustellen, dass für alle zu analysierenden Bilder die gleiche Größe verwendet wird.
7. Endoreplikationsanalyse (Ploidy)
- Wählen Sie mit Fidschi das Grh-Kanalfenster aus, und duplizieren Sie das Bild. Verwenden Sie dann das Schwellenwertwerkzeug, um eine Maske zu erstellen. Passen Sie den Schwellenwert manuell an, indem Sie die obere Leiste verschieben, um den Hintergrund zu minimieren, ohne dass die Kerne drastisch schrumpfen(Abbildung 4D).
- Wenn sich Kerne im Grh-Kanal im Schwellenbild berühren, verwenden Sie das Pinselwerkzeug (2 Pixel Breite) in der gleichen Farbe wie der Hintergrund, um eine Linie zwischen den Kernen zu zeichnen. Klicken Sie hier, um 1x anzuwenden, wenn Sie fertig sind, um die endgültige Maske zu generieren.
- Generieren Sie eine Region von Interesse (ROI) Karte mit der Funktion Partikel analysieren: Stellen Sie die Größe auf 5 'm-60 'm, um die meisten der Kerne zu erfassen, ohne den Hintergrund einzubeziehen.
- Passen Sie die ROI-Karte nach Bedarf im ROI-Managermanuell an. Löschen Sie alle Auswahlen, die keine Kerne sind, und fügen Sie der Liste alle Kerne hinzu, die nicht identifiziert wurden, indem Sie den Kern mit dem Freihandauswahltool umreißen und dem ROI-Manager hinzufügen (Abbildung 4D).
- Wählen Sie den DAPI-Kanal aus und klicken Sie dann im ROI-Manager auf Alle anzeigen, um die generierte ROI-Karte im Grh-Kanal auf den DAPI-Kanal anzuwenden.
- Löschen Sie alle Auswahlen, bei denen sich der Epithelkernmitriss mit nichtepitheliaalen Kernen (z. B. Kerne aus Muskel oder Fett) überlappt, aus der ROI-Karte. Grainyhead färbt nur epitheliale Kerne, während DAPI alle Kerne färbt. Überprüfen Sie, ob jede umrissene Auswahl nur einen Kern enthält, und löschen oder bearbeiten Sie Auswahlen mit mehr als einem Kern. Speichern Sie die bearbeitete ROI-Liste.
- Messen Sie die Fläche und die integrierte Dichte der einzelnen Epithelkerne in der ROI-Karte mit den Analysewerkzeugen in Fidschi. Exportieren Sie die Werte in ein Tabellenkalkulationsprogramm.
- Messen Sie den durchschnittlichen Bildhintergrund mit dem kreisförmigen Auswahlwerkzeug. Zeichnen Sie drei Kreise, die sich nicht mit Kernen in verschiedenen Bereichen des DAPI-Bildes überlappen. Fügen Sie den Bereich und die integrierte Dichte der einzelnen Kreise zu einem Tabellenkalkulationsprogramm hinzu, um die Hintergrundbildhelligkeit festzulegen.
- Beginnen Sie mit der Berechnung des durchschnittlichen Hintergrunds pro Flächeneinheit für jedes Bild, indem Sie jeden integrierten Hintergrunddichtewert durch den entsprechenden Bereich dividieren. Dann durchschnittlich die drei integrierten Dichte pro Fläche Messungen für das Bild, um den durchschnittlichen Hintergrund pro Flächeneinheit zu erhalten.
- Als Nächstes berechnen Sie den Gesamthintergrund jedes DAPI-Kerns, indem Sie die Fläche des Kerns mit dem durchschnittlichen Hintergrund pro Flächeneinheit multiplizieren. Die normalisierte DAPI-Intensität für jeden gemessenen Kern kann dann berechnet werden, indem der Gesamthintergrund jedes Kerns von seiner gemessenen integrierten Dichte subtrahiert wird.
- Durchschnitt aller normalisierten DAPI-Intensitätswerte aus der nicht verletzten Epithelsteuerung. Die unverletzten Epithelkerne hatten zuvor einen Ploidy-Wert von 2C und können als Referenz für die Berechnung der Ploidie in den Epithelkernen aus den experimentellen Bedingungen8dienen.
- Berechnen Sie die Ploidie jedes Kerns, indem Sie die normalisierte DAPI-Intensität jedes Kerns durch den normalisierten Wert aus der referenzierten nicht verletzten Epithelkontrolle (2C) dividieren und dann den Wert mit 2 multiplizieren, um der normalisierten Ploidie (C-Wert) gleichzumachen:
(Nuclear Integrated Density - Background Nuclear Integrated Density)/Average Nuclear Integrated Density (uninjured epitheliale Kerne = 2C) x 2 = epitheliale Kernploidie (C) - Graphenkerne mit Ploidy-Werten als Punktdiagramm, Histogramm oder entsprechend in ein Balkendiagramm gruppiert (d. h. 2C [0.6-2.9C], 4C [3.0-5.9C], 8C [6.0-12.9C], 16C [13.0-24.9C] und >32C [>25.0C] (Abbildung 3F).
Ergebnisse
Ein detailliertes Protokoll zur Verwendung von D. melanogaster als Modell zur Untersuchung der wundinduzierten Polyploidisierung (WIP) ist vorgesehen. Dieses Wundheilungsmodell bietet viele Vorteile gegenüber Säugetier- und anderen Fliegenmodellen von WIP. Polyploidie wurde leicht durch eine mechanische Punktion mit einem Insektenstift induziert und polyploide Zellen wurden innerhalb kurzer Zeit erzeugt (2-3 dpi) (Abbildung 1A, 1B)4. Die größte Herausforderung ist die Zerlegung des intakten Bauchgewebes ohne Störungen des Epithels. Das D. melanogaster Epithel wird leicht versehentlich mit den scharfen Sezierwerkzeugen gestoßen oder zerkratzt. Daher sollten die Schritte dieses Protokolls vor der Verwendung und Analyse geübt werden.
Erstens beschränkte sich die Verletzung auf den ventralen weiblichen Bauch, der einen großen, flachen undurchsichtigen Gewebebereich bietet, der ideal für die Bildgebung ist. Die Stichwunden wurden im Pleuritepithel zugefügt, das auf beiden Seiten der ventralen Mittelliniensterniten liegt und zwischen tergiten (T) Segmenten T4-T5 (Abbildung 1A-C) ausgerichtet ist. Diese Wundplatzierung bietet einen großen sichtbaren Bereich, der nicht durch die Sezierung gestört wird. Anspruchsvolle Schritte sind der Bauchfederschereschnitt und die Pinning-Schritte (Abbildung 1D, 1E). Der Federschnittschritt funktionierte am besten, wenn die Bauchmuskeln in einem reduzierten Volumen von Graces Lösung geschnitten wurden (30 ,L), um die Gewebebewegung zu verringern. Ein gut zentrierter Schnitt entlang der dorsalen Mittellinie war notwendig, um genügend Fläche auf den Bauchdorsalklappen zur Verfügung zu stellen, um sich auf der Sezierplatte zu öffnen (Abbildung 1C). Der Bauch muss ohne übermäßige Kraft vorsichtig an den vier Ecken fixiert werden (Abbildung 1E). Ein zu harter Stiftschub verzerrt das Bauchgewebe und könnte das Gewebe sogar in die Sezierplatte schieben. In diesem Fall muss das Gewebe entsorgt werden. Sobald das Bauchgewebe fixiert war, blieb es auf der Sezierplatte, bis die Immunfluoreszenzfärbung abgeschlossen war und die Bauchmuskeln auf einem Glasdeckel für die Bildgebung montiert waren (Abbildung 1F).
Die Wundheilung erfordert eine kontinuierliche Epithelplatte, die von Endoreplikation und Zellfusion abhängig ist4,16. Das Septat-Knotenprotein FasIII, das Zell-Zell-Verbindungen kennzeichnet, lieferte einen Indikator dafür, ob während der Vorbereitung Verarbeitungsstörungen aufgetreten sind (Abbildung 1G, 1H). Abdomen mit großen Kratzern (unbefleckter Bereich), die den Wundbereich stören, müssen entsorgt werden und wurden nicht für weitere Analysen verwendet (Abbildung 1H).
Der nächste Schritt bestand darin, intakte Proben auf Fehler in WIP zu analysieren. Dieses Protokoll enthält verschiedene Assays, um verschiedene Aspekte der WIP-Antwort zu erkennen (Abbildung 2). Die Wundreparatur war abgeschlossen, als eine zentrale, große, mehrkernige Zelle den Wundschorf bedeckte (Abbildung 3A). Hier wurde die Zellfusion durch Färbung für FasIII/Grh und Quantifizierung der Anzahl der Grh+ Epithelkerne, die im FasIII umrissenen Bereich4umfassten, nachgewiesen. Defekte im Wundverschluss oder bei der Reepithelisierung wurden festgestellt, wenn Lücken von >10 m im Epithelblatt beobachtet wurden(Abbildung 3B,roter Pfeil). Dies war z.B. der Fall, als WIP durch die Aktivierung des mitotischen Zyklus durch Expression von stg, fzrRNAigehemmt wurde, wie kürzlich berichtet16. In diesem genetischen Zustand waren 52% der Wunden nicht in der Lage, ein kontinuierliches Epithelblatt über dem Wundschorf zu bilden (Abbildung 3B, 3C).
Eine weitere Methode zur Messung der Wundreparatur in diesem Modell bestand darin, die Epithelmembran mit Epi-Gal4-Expression von UAS-mCD8-ChRFP4 zu visualisieren (Abbildung 2, Abbildung 3D). In der Kontrolle schlossen sich 91% der Epithelwunden vollständig durch 3 dpi, hemmten aber WIP durch Blockierung der Endoreplikation (E2f1RNAi) und zellfusion (RacDN) gleichzeitig, wie bereits berichtet, verursachten 92% der Epithelwunden völlig offen (Abbildung 3D, 3E)8,16. Die Aktivierung des mitotischen Zellzyklus durch Expression von stg, fzrRNAi führte auch zu einem epithelialer Wundverschlussdefekt. Durch die Visualisierung der Epithelzellmembran konnte jedoch das Ausmaß des Reepithelisierungsfehlers bestimmt werden. Die WIP-Mutation (E2f1RNAi, RacDN) Fliegenwunden waren offener als die stg, fzrRNAi Wunden (Abbildung 3D, gestrichelter roter Umriss)16. Dieser Membran-Wundheilungstest lieferte weitere Informationen über das Ausmaß des Wundreparaturfehlers. Infolgedessen konnten Reepithelisierungsfehler entweder als vollständig offen, teilweise geschlossen (d. h. >10 m Lücken) oder vollständig geschlossen gruppiert werden (Abbildung 3D, 3E).
Neben der Zellfusion wachsen Epithelzellen durch Endoreplikation in der Größe, einem unvollständigen Zellzyklus, der den Kern-DNA-Gehalt verdoppelt. Die Endoreplikation wurde sowohl durch die Zellzyklusaktivität als auch durch direkte Kern-DNA-Ploidie-Messungen(Abbildung 2 und Abbildung 4)durchgeführt. Hierbei wurde die Zellzyklusaktivität durch Einbeziehung des Thymidin-Analogas EdU (Abbildung 4A, 4B) nachgewiesen. D. melanogaster Epithelzellen traten in den Endzyklus ein, ein unvollständiger Zellzyklus, der zwischen den S- und G-Phasen ohne eine dazwischenliegende M-Phase4,12oszilliert. Die erwachsene D. melanogaster Diät wurde vor der Verletzung mit EdU+ Nahrung ergänzt und die Fliegen wurden auf einer EdU+ Diät bis zur Zerlegung bei 2 dpi aufrechterhalten (Abbildung 4A). Die EdU wurde dann mit dem Click-iT-Protokoll des Herstellers erkannt. Dieser EdU-Assay wurde verwendet, um zu bestimmen, wo, wann und wie viele Kerne ausgelöst wurden, um in die S-Phase als Reaktion auf eine Wunde einzutreten. Mit Dem Gal4/ UAS-System wurde kürzlich festgestellt, dass epitheliale spezifische Expression von myc entweder die Kompetenz von Epithelzellen blockieren ( myc RNAi ) oder die Kompetenz von Epithelzellen, in die S-Phase einzutreten, verstärken kann (mycRNAi). Als Ergebnis hat sich gezeigt, dass Myc ausreichend ist, um Endoreplikation in postmitotischen Zellen zu induzieren, auch ohne Verletzung16,22.
Als nächstes wurde die epitheliale Ploidie durch direkte Messung des Kern-DNA-Gehalts bestimmt. Epithelkerne wurden durch Immunfluoreszenzfärbung für den epitheliaalspezifischen Marker Grh (Abbildung 4D) identifiziert. In der Fidschi-Bildgebungssoftware wurden Epithelkerne systemisch identifiziert und dann mit Grh-Atomflecken angeschneit. Die Kerne wurden dann getrennt und die ROIs auf die SUMME der Stapel DAPI-Bild überlagert(Abbildung 4D,grüner Pfeil). Alle überlappenden Kerne wurden manuell gelöscht, bevor die integrierte Dichte der ausgewählten Kerne gemessen wurde(Abbildung 4D, rote Pfeile). Diese halbautomatische Methode ermöglicht es, die Verteilung und Ploidie der meisten Kerne im gesamten unverletzten und reparierten Fliegenabdominalepithel8zu quantifizieren. Wie kürzlich berichtet, setzten sich die epitheliale Kerne, die die Wunde umgaben, aus 44% polyploiden Kernen mit einem DNA-Gehalt von mehr als 3C bei 3 dpi zusammen (Abbildung 4E, 4F)16. Wie von den EdU-Ergebnissen erwartet, führte der Knockdown von myc zu einem signifikanten Block in der Endoreplikation, da nur 9% der Epithelkerne polyploid waren, während eine Überexpression von Myc zu 100% polyploiden epitheliale Kerne um die Wundstelle führte (Abbildung 4F)16. Epithelkerngröße wurde auch sichtbar durch myk-Expression mit entweder reduzierten oder vergrößerten Kernen beeinflusst. Der Kernbereich ist jedoch kein genaues Maß für die Ploidie und physiologische Wirkungen, da Faktoren wie die Zelldehnung auch die nukleare Größe beeinflussen können, ohne den Kern-DNA-Gehalt zu beeinflussen20.
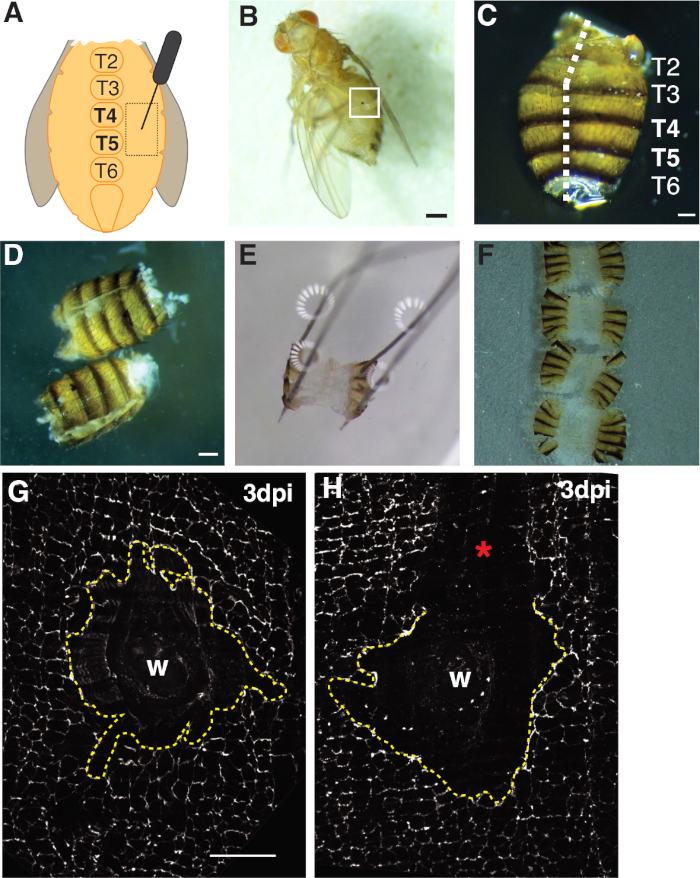
Abbildung 1: Erwachsene Früchte fliegen Bauchwunden, Sezieren und Gewebemontage. (A) Diagramm des erwachsenen Bauchwundenassays. Fliegen sollten auf beiden Seiten des Bauches bei Tergite 4 (T4) verletzt werden. (B) Erwachsene weibliche Fruchtfliege 3 dpi mit Melanin-Schorf aus Wundheilung gebildet (weiße Box). Skala bar = 50 m. (C) Sezierter Abdomen für Erwachsene, dorsale Ansicht, mit Tergiten beschriftet. Abdomen wurden auf der Mittellinie der dorsalen Seite (weiße gestrichelte Linie) abgefüllt. Skala bar = 50 m . (D) Sezierte und filetierte erwachsene Bauchvorstufen vor dem Anheften. Skala bar = 50 m. (E) Gepinnter erwachsener Bauch auf einer Sezierender Platte. In jeder der vier Ecken des Bauches wurde auf der Rückenseite eine Stecknadel platziert. Das Gewebe wurde sanft geöffnet, aber nicht gedehnt, um Einreißen zu vermeiden. (F) Erwachsene Bauchmuskeln wurden montiert und auf einem Glasdeckel mit der Innenseite des Bauches nach unten zu dem Deckelschlupf und Nagelhaut in Richtung der Glasrutsche ausgerichtet platziert. (G) FasIII Färbung der intakten Wundfläche ohne Verarbeitungsstörung und einer zentralen Syncytium (gestrichelte gelbe Linie). Skala bar = 50 m . (H) Bild einer zerkratzten Wundfläche (*) mit einem ungefärbten FasIII-Bereich, der das Syncytium stört. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
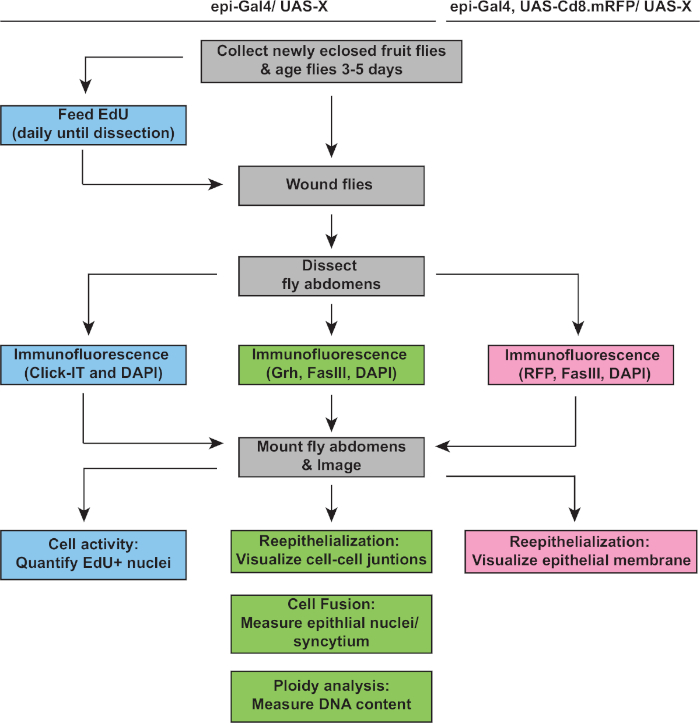
Abbildung 2: WIP-Analyseworkflow. Das Flussdiagramm zeigt die drei in dieser Studie beschriebenen Assays sowie überlappende und unterschiedliche Schritte zum Erkennen und Messen der WIP-Antwort. Der EdU-Test misst die Zellzyklusaktivität (blaue Kästen), Ploidie und Reepithelisierung werden durch Grh/FasIII-Immunostainierung (grüne Boxen) nachgewiesen, und die Expression von Membran-RFP ermöglicht die Messung des Ausmaßes des epitheliaalen Wundverschlusses (rosa Boxen). Häufige Schritte sind in grauen Boxen und D. melanogaster Stamm Genotypen sind oben aufgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
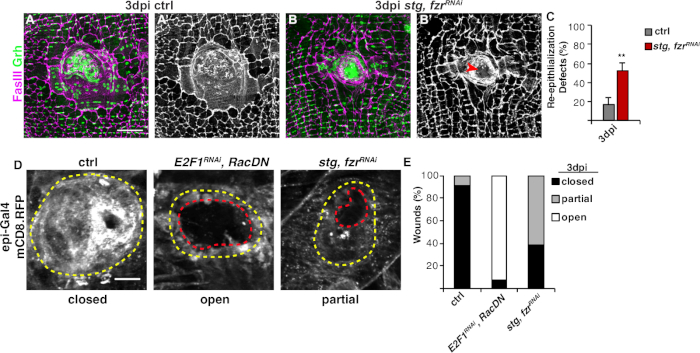
Abbildung 3: Methoden zum Nachweis der Reepithelisierung während des WIP. Die Reepithelisierung war gestört, als WIP genetisch gehemmt wurde. Immunfluoreszierende Bilder der Kontrolle (A) und stg, fzrRNAi (B) bei 3 dpi. Epithelkerne und Septat-Kreuzungen wurden mit Grh (grün) bzw. FasIII (Magenta) befleckt. (A' und B') FasIII Färbung allein zeigte, dass die Reepithelisierung beeinträchtigt war (roter Pfeil) in stg, fzrRNAi epithelium. Skala bar = 50 m . (C) Quantifizierung von Reepithelisierungsdefekten (%) bei 3 dpi (grau): Steuerung (n = 8), stg, fzrRNAi (n = 6). Fehlerbalken zeigen Standardfehler an; die statistische Signifikanz wurde mit dem T-Test des Schülers gemessen, **P < 0,01. Eine Reepithelisierung während der Wundreparatur konnte auch durch Expression eines membrangebundenen RFP mit Epi-Gal4, UAS-mCD8-RFP nachgewiesen werden. (D) Immunfluoreszenzbilder der Kontrolle, E2F1RNAi, RacDNund stg, fzrRNAi bei 3 dpi. Skalenbalken = 20 m. Wundschorf (gelber Umriss) und offener epithelialen Wundbereich (roter Umriss). (E) Quantifizierung des Wundverschlusses in Strg (n = 11), E2F1RNAi, RacDN (n = 13) und stg, fzrRNAi (n = 13). Nach Grendler et al.16. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
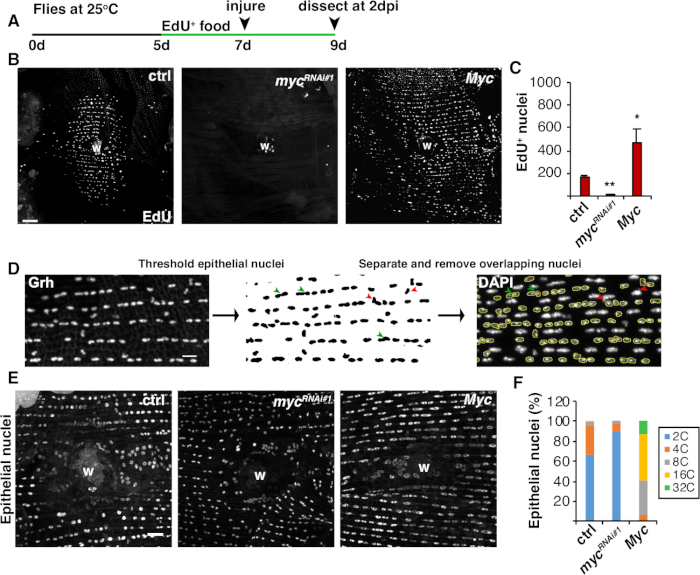
Abbildung 4: Methoden zum Erkennen der Endoreplikation während des WIP. (A) Zeitleiste des EdU-Assays: Erwachsene Drosophila wurden 2 Tage vor der Verletzung täglich 2 Tage vor der Verletzung mit 75 l von 5 mM Hefe-EdU gefüttert und bis 2 dpi fortgesetzt. (B) Immunfluoreszenzbilder des EdU-Etiketts in Fliegenstämmen, ausgedrückt mit Epi-Gal4/ UAS-System bei 2 dpi. Wundschorf (W). Skala bar = 50 m. (C) Durchschnittliche Anzahl der EdU+ Epithelkerne pro Fliege bei 2 dpi: Strg (n = 37), mycRNAi 1 (n = 10) und Myc (n = 8). Fehlerbalken zeigen Standardfehler an; Die statistische Signifikanz wurde mit dem T-Test des Schülers gemessen, *P < 0,05, **P < 0,01. (D) Schematic der Detektion und Messung der epitheliaalen Kernploidie. Epithelkerne wurden durch den Anti-Grh-Fleck in Fidschi identifiziert und angeschneit. Überlappende Epithelkerne wurden getrennt (grüne Pfeilspitzen) oder entfernt (rote Pfeilspitzen), wenn sie von nichtepitheliaalen Kernen überlagert wurden. Die integrierte Dichte und der Kernbereich des entsprechenden DAPI-Buntkernbildes wurden gemessen. (E) Epithelatomgröße (Grh) wurde durch myc-Expression bei 3 dpi verändert. (F) Epitheliale Kernploidie (%) bei 3 dpi: Strg (n = 4), mycRNAi 1 (n = 6) und Myc (n = 3). Nach Grendler et al.16. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Präsentiert wird ein detailliertes Protokoll, wie man das erwachsene D. melanogaster Bauchepithel seziert und verwendet, um zu untersuchen, wie Gene WIP regulieren, indem sie die Reepithelisierung und Endoreplikation während der Wundreparatur verändern16. Mit dieser Methode wurde das Proto-Onkogen Myc vor kurzem als wichtiger Regulator von WIP identifiziert. Myc ist für Epithelzellen erforderlich, um post-Verletzungen zu endoreplizieren und ist ausreichend für stille Epithelzellen, um sowohl in erwachsenen Fliegenepithel und Zubehör Drüsen16,22endocyclen. Es wurde auch festgestellt, dass die Umstellung von Epithelzellen auf einen mitotischen Zellzyklus durch Expression von stg, fzrRNAi ist schädlich für die Wundreparatur. Weitere Studien mit dieser Methode werden andere Gene identifizieren, die erforderlich sind, um die Reepithelisierung und Endoreplikation während des WIP zu regulieren, und zeigen sowohl Ähnlichkeiten als auch Unterschiede zur Regulierung der Polyploidie und Funktionen in einer Vielzahl von Geweben.
Dieses Modell und Verfahren bieten einzigartige Vorteile, einschließlich der einfachen Induktion der Polyploidie mit einer mechanischen Punktion und der Tatsache, dass polyploide Zellen innerhalb vonTagen4 erzeugt werden. Die Gewebesektions- und Vorbereitungsprotokolle basieren auf Larvensektionstechniken23, aber der erwachsene Fliegenbauch ist steifer und daher leicht gestört. Daher erfordert dieses Protokoll Praxis und Präzision, um ein intaktes Gewebe zu isolieren, um WIP zu untersuchen. Einmal seziert, ist das Epithel jedoch deutlich sichtbar und leicht abgebildet, was eine Momentaufnahme des Wundheilungsprozesses ergibt. Diese Methode bietet eine Fülle von Informationen über die epitheliale Organisation der erwachsenen Fliege, die Zell- und Synzytiumgröße sowie die Ploidie von Zellen und einzelnen Kernen. Während Live-Bildgebung innerhalb der intakten Fruchtfliege aufgrund ihrer undurchsichtigen Nagelhaut noch nicht möglich ist, könnte dieses Protokoll angepasst werden, um derzeit verfügbare ex vivo-Kulturbedingungen, die in D. melanogaster verwendet werden, um kurzfristige Live-Bildgebungsstudien durchzuführen24.
In Zukunft wird dieses Modell ideal sein, um Zell-zu-Zell-Überlauf und den Beitrag anderer Zelltypen zu WIP zu untersuchen, indem die Genexpression mit dem Gal4/UAS-System bei anderen Zellarten reguliert wird. Ähnliche Fragen können auch mit einer Vielzahl von genetischen und mutierten Hintergründen beantwortet werden. Der sezierte erwachsene Fliegenbauch enthält eine Vielzahl von Zelltypen, die mit dieser Methode leicht visualisiert werden können, einschließlich Fettkörper und Oenozyten, seitliche Muskelfasern, sensorische Neuronen, Luftröhre und Makrophagen-ähnliche Hämozyten. Darüber hinaus wird dieses Modell es Forschern ermöglichen, zu untersuchen, wie physiologische Variablen WIP beeinflussen, einschließlich Geschlecht, Ernährung, Infektion, Alter und Umweltstressoren. Während das Protokoll die erwachsene weibliche Fliege aufgrund ihrer größeren Größe verwendet, tritt WIP auch in der männlichen Fruchtfliege auf (Gjelsvik und Losick, unveröffentlicht). Polyploide Zellen sind gefunden worden, um während des Alterns und altersassoziierte Erkrankungen in der Säugetierleber, Gehirn, Auge, und Herz12entstehen. Das Fruchtfliegenmodell wird es Forschern ermöglichen, die Polyploidisierung in physiologischen und krankheitsbedingten Kontexten zu untersuchen, da menschliche krankheitsbedingte Gene stark konserviert sind.
Offenlegungen
nichts.
Danksagungen
Am Boston College möchten wir Dr. Eric Folker für die Verwendung der Kamera- und Stereoskopmikroskop-Setup seines Labors für die Bildgebung und Bret Judson am Boston College Imaging Core für Infrastruktur und Support danken. Wir danken auch den Fly Community Ressourcen: Bloomington Drosophila Stock Center (NIH P40OD018537), Vienna Drosophila Resource Center und TRiP Center an der Harvard Medical School (NIH/NIGMS R01-GM084947) für die Bereitstellung transgener Bestände, die in dieser Studie verwendet werden. Der Maus-FasIII-Antikörper wurde von der Developmental Studies Hybridoma Bank gewonnen, die von NICHD des NIH unterstützt wurde, und wurde an der University of Iowa, Department of Biology, Iowa City, IA, gepflegt. Die in dieser Publikation berichtete Forschung wurde vom National Institute of General Medical Sciences der National Institutes of Health unter der Award-Nummer R35GM124691 unterstützt. Der Inhalt liegt allein in der Verantwortung der Autoren und stellt nicht unbedingt die offizielle Meinung der National Institutes of Health dar.
Materialien
| Name | Company | Catalog Number | Comments |
| 35 mm Petri dishes | Fisher Scientific | FB0875713 | For creating plates to dissect in |
| 50 mL Conical Centrifuge Tubes | Fisher Scientific | 14-432-22 | For preparing staining reagents in |
| AxioImager M2 with Apotome | Zeiss | NA | For imaging samples |
| Blowgun mini | Genesee Scientific | 54-104M | For anethesizing D. melanogaster strains |
| Bovine Serum Albumin, 30% | Sigma | A7284-500ML | For immuostaining |
| Carbon dioxide tank | various distributors | N/A | For anethesizing D. melanogaster strains |
| Click-iT EdU 594 Kit | Thermofisher | C10339 | For EdU assay |
| Coverslips | Thermofisher | 3406 | For mounting |
| DAPI | Sigma | D9542-10MG | For immuostaining |
| Dissecting Plates (use Sylgard 184 Sil Elastic Kit) | Ellsworth Adhesives | 184SIL | For creating plates to dissect in. Mix epoxy as directed, let dry overnight |
| Donkey anti-Rabbit IgG (H+L) Secondary Antibody, Alexa Fluor 488 conjugate | Thermofisher | A21206 | For secondary immuostaining |
| Donkey anti-Rabbit IgG (H+L) Secondary Antibody, Alexa Fluor 568 conjugate | Thermofisher | A10042 | For secondary immuostaining |
| Drosophila tubing and fittings | Genesee Scientific | 59-124C, 59-123, 59-140 | For anethesizing D. melanogaster strains |
| Dumont #5 Forceps | Fine Science Tools | 11252-20 | For dissecting |
| epi-Gal4 | Bloomington Drosophila Stock Center (b) | b38793 | Losick et al. Current Biology, 2013 |
| epi-Gal4, UAS-mCD8.RFP | Bloomington Drosophila Stock Center (b) | b38793, b27392 | Losick et al. Current Biology, 2013 |
| Excel | Microsoft | For performing ploidy calculations | |
| Fiji/ImageJ (image analysis software) | NIH | https://imagej.nih.gov/ij | For image analysis |
| Fly food | Archon Scientific | N/A | Corn Syrup/Soy food |
| Flystuff Flypad | Genesee Scientific | 59-114 | For anethesizing D. melanogaster strains |
| Glass dissecting dish | Fisher Scientific | 13-748B | For performing dissections in |
| Glass slides | Fisher Scientific | 12-518-104C | For mounting |
| Goat anti-Mouse IgG (H+L) Secondary Antibody, Alexa Fluor 488 conjugate | Thermofisher | A11001 | For secondary immuostaining |
| Goat anti-Mouse IgG (H+L) Secondary Antibody, Alexa Fluor 568 conjugate | Thermofisher | A11031 | For secondary immuostaining |
| Grace's Insect Medium, unsupplemented | Thermofisher | 11595030 | For dissecting in |
| Insect pins | Fine Science Tools | 26002-10 | For wounding and pinning fly abdomens flat |
| Mouse anti-Fasciclin III (Drosophila) Primary Antibody | Developmental Studies Hybridoma Bank | 7G10 | For immunostaining epithelial cell-cell junctions |
| Mouting media | Vector Laboratories | H-1000 | Anti-fade mounting media to prevent photo bleaching during imaging |
| Nail polish | Electron Microscopy Sciences | 72180 | For sealing slides |
| Ortibal shaker | Fisher Scientific | 02-217-988 | For immuostaining |
| Phosphate Buffered Saline, PH 7.4 | Sigma | P3813-10PAK | For staining |
| Pin holders | Fine Science Tools | 91606-07 | For wounding |
| Rabbit anti-Grainyhead Primary Antibody | N/A | N/A | For immunostaining epithelial nuclei. Protocol to make antibody can be found (Ref. #4 and 8) |
| Rabbit anti-RFP Primary Antibody | MBL | PM005 | For immunostaining mCD8-RFP fly epithelium |
| Stereomicroscope | Olympus | SZ51 | For dissecting and mounting fly tissue |
| Triton X-100 | Sigma | 10789704001 | For immuostaining |
| UAS-E2F RNAi, UAS-RacDN | VDRC (v) and Bloomington Drosophila Stock Center (b) | v108837, b6292 | Losick et al. Current Biology, 2013 |
| UAS-fzr RNAi, UAS-Stg | VDRC (v) and Bloomington Drosophila Stock Center (b) | v25550, b56562 | Grendler et al. Development, 2019 |
| UAS-Myc | Bloomington Drosophila Stock Center (b) | b9674 | Grendler et al. Development, 2019 |
| UAS-myc RNAi | Bloomington Drosophila Stock Center (b) | b36123 | Grendler et al. Development, 2019 |
| Vannas Spring Scissors | Fine Science Tools | 15000-00 | For dissecting |
Referenzen
- Abreu-Blanco, M. T., Verboon, J. M., Liu, R., Watts, J. J., Parkhurst, S. M. Drosophila embryos close epithelial wounds using a combination of cellular protrusions and an actomyosin purse string. Journal of Cell Science. 125 (Pt 24), 5984-5997 (2012).
- Wood, W., et al. Wound healing recapitulates morphogenesis in Drosophila embryos. Nature Cell Biology. 4 (11), 907-912 (2002).
- Galko, M. J., Krasnow, M. A. Cellular and genetic analysis of wound healing in Drosophila larvae. PLoS Biology. 2 (8), E239 (2004).
- Losick, V. P., Fox, D. T., Spradling, A. C. Polyploidization and cell fusion contribute to wound healing in the adult Drosophila epithelium. Current Biology. 23 (22), 2224-2232 (2013).
- Wang, Y., et al. Integrin Adhesions Suppress Syncytium Formation in the Drosophila Larval Epidermis. Current Biology. 25 (17), 2215-2227 (2015).
- Ramos-Lewis, W., LaFever, K. S., Page-McCaw, A. A scar-like lesion is apparent in basement membrane after wound repair in vivo. Matrix Biology. 74, 101-120 (2018).
- Lee, C. W., Kwon, Y. C., Lee, Y., Park, M. Y., Choe, K. M. cdc37 is essential for JNK pathway activation and wound closure in Drosophila. Molecular Biology of the Cell. 30 (21), 2651-2658 (2019).
- Losick, V. P., Jun, A. S., Spradling, A. C. Wound-Induced Polyploidization: Regulation by Hippo and JNK Signaling and Conservation in Mammals. PLoS One. 11 (3), e0151251 (2016).
- Tamori, Y., Deng, W. M. Tissue repair through cell competition and compensatory cellular hypertrophy in postmitotic epithelia. Developmental Cell. 25 (4), 350-363 (2013).
- Xiang, J., et al. EGFR-dependent TOR-independent endocycles support Drosophila gut epithelial regeneration. Nature Communications. 8, 15125 (2017).
- Cohen, E., Allen, S. R., Sawyer, J. K., Fox, D. T. Fizzy-Related dictates A cell cycle switch during organ repair and tissue growth responses in the Drosophila hindgut. Elife. 7, e38327 (2018).
- Gjelsvik, K. J., Besen-McNally, R., Losick, V. P. Solving the Polyploid Mystery in Health and Disease. Trends in genetics: TIG. 35 (1), 6-14 (2019).
- Cao, J., et al. Tension Creates an Endoreplication Wavefront that Leads Regeneration of Epicardial Tissue. Developmental Cell. 42 (6), 600-615 (2017).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Lazzeri, E., et al. Endocycle-related tubular cell hypertrophy and progenitor proliferation recover renal function after acute kidney injury. Nature Communications. 9 (1), 1344 (2018).
- Grendler, J., Lowgren, S., Mills, M., Losick, V. P. Wound-induced polyploidization is driven by Myc and supports tissue repair in the presence of DNA damage. Development. 146 (15), 173005 (2019).
- Mehrotra, S., Maqbool, S. B., Kolpakas, A., Murnen, K., Calvi, B. R. Endocycling cells do not apoptose in response to DNA rereplication genotoxic stress. Genes & Development. 22 (22), 3158-3171 (2008).
- Patterson, M., et al. Frequency of mononuclear diploid cardiomyocytes underlies natural variation in heart regeneration. Nature Genetics. 49 (9), 1346-1353 (2017).
- Gonzalez-Rosa, J. M., et al. Myocardial Polyploidization Creates a Barrier to Heart Regeneration in Zebrafish. Developmental Cell. 44 (4), 433-446 (2018).
- Webster, M., Witkin, K. L., Cohen-Fix, O. Sizing up the nucleus: nuclear shape, size and nuclear-envelope assembly. Journal of Cell Science. 122 (Pt 10), 1477-1486 (2009).
- Knouse, K. A., Lopez, K. E., Bachofner, M., Amon, A. Chromosome Segregation Fidelity in Epithelia Requires Tissue Architecture. Cell. 175 (1), 200-211 (2018).
- Box, A. M., et al. Endocycles support tissue growth and regeneration of the adult Drosophila accessory gland. bioRxiv. , 719013 (2019).
- Karim, M. R., Moore, A. W. Morphological analysis of Drosophila larval peripheral sensory neuron dendrites and axons using genetic mosaics. Journal of Visualized Experiments. (57), e3111 (2011).
- Dai, W., Montell, D. J. Live Imaging of Border Cell Migration in Drosophila. Methods in Molecular Biology. 1407, 153-168 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten