Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Kultur der Hirnkapillarpericyten für zytosolische Kalziummessungen und Calcium-Imaging-Studien
In diesem Artikel
Zusammenfassung
Gehirn Kapillarpericyten sind wesentliche Akteure bei der Regulierung der Blut - Hirn-Schranke Eigenschaften und Blutfluss. Dieses Protokoll beschreibt, wie Kapillarperizyten des Gehirns isoliert, kultiviert, in Bezug auf den Zelltyp charakterisiert und für Untersuchungen der intrazellulären Calciumsignalisierung mit fluoreszierenden Sonden angewendet werden können.
Zusammenfassung
Pericyte werden mit Endothelzellen und astrozytischen Endfüßen in einer Struktur assoziiert, die als neurovaskuläre Einheit (NVU) bekannt ist. Gehirn Kapillarperizytenfunktion ist nicht vollständig bekannt. Pericyten wurden vorgeschlagen, an der Kapillarentwicklung, der Regulierung der Endothelbarrieredichtheit und Tranzytoseaktivität, der Regulierung des Kapillartons beteiligt zu sein und bei bestimmten Hirnpathologien eine entscheidende Rolle zu spielen.
Perizyten sind eine Herausforderung, im intakten Gehirn aufgrund der Schwierigkeiten bei der Visualisierung von Prozessen im Gehirn Parenchym zu untersuchen, sowie die Nähe zu den anderen Zellen der NVU. Das vorliegende Protokoll beschreibt eine Methode zur Isolierung und Kultur von primären Rinderhirnkapillarperizyten und deren anschließende Verwendung in Kalzium-Bildgebungsstudien, bei denen Die Wirkung von Agonisten, die an der Signalisierung des Gehirns und Pathologien beteiligt sind, untersucht werden kann. Kortikale Kapillarfragmente dürfen sich an den Boden von Kulturkolben anheften und nach 6 Tagen sind Endothelzellen und Pericyten aus den Kapillarfragmenten herausgewachsen. Die Endothelzellen werden durch sanfte Trypsinisierung entfernt und Pericyten werden für 5 weitere Tage vor dem Passieren kultiviert.
Isolierte Pericyte werden in 96-Well-Kulturplatten gesät und mit dem Calciumindikatorfarbstoff (Fura-2 Acetoxymethyl (AM)) beladen, um Messungen des intrazellulären Kalziumspiegels in einem Plattenleser-Setup zu ermöglichen. Alternativ werden Pericyte auf Coverlips gesät und in Zellkammern montiert. Nach der Belastung mit dem Kalziumindikator (Cal-520 AM) kann die Calcium-Live-Imaging mittels konfokaler Mikroskopie bei einer Anregungswellenlänge von 488 nm und einer Emissionswellenlänge von 510-520 nm durchgeführt werden.
Die hier beschriebene Methode wurde verwendet, um die ersten intrazellulären Kalziummessungen aus primären Hirnkapillarperizyten zu erhalten, die zeigen, dass Pericyte über ATP stimuliert werden und sich in vitro zusammenziehen können.
Einleitung
Hirnkapillarpericyten bilden zusammen mit Endothelzellen und Astrozyten die NVU1,2,3. Die Endothelzellen, die die strukturelle Basis der Kapillaren bilden, bilden lange zylindrische Rohre mit einem Durchmesser von 5-8 m. Die Endothelzellen sind sporadisch mit Pericyten bedeckt und von Vorsprüngen von Astrozyten umgeben; die Astrozyten-Endfüße.
Die Blut-Hirn-Schranke (BBB), die sich an den Hirnkapillaren befindet, ist der Hauptort für den Austausch von Nährstoffen, Gasen und Abfallprodukten zwischen Gehirn und Blut. Die BBB schützt auch das Gehirn vor endogene und exogene Neurotoxine und dient als Barriere für die Lieferung einer großen Anzahl von Wirkstoffverbindungen. Die Barrierefunktion ist ein Schwerpunktbereich und ein Hindernis für Pharmaunternehmen, die Medikamente des Zentralnervensystems (ZNS) entwickeln. Dies hat ein großes Interesse an der Untersuchung der Zellen der NVU in Kultur4geweckt. Hirnastrozyten und Endothelzellen wurden in einer Reihe von Studien kultiviert und charakterisiert, während die Studien und Protokolle für die Pericytkultur spärlich sind.
Zuvor veröffentlichte Protokolle haben die Erzeugung von hirnkapillaren Pericytenkulturen bis zu einem gewissen Grad beschrieben, mit einer Reihe von verschiedenen Ansätzen wie Immunpanning5, High- und Low-Glucose-Medien6, fluoreszierend-aktivierte Zellsortierung7, Gradient zentrifugation8, etc. Obwohl diese Methoden ausreichen, um Kulturen von Pericyten zu erhalten, sind einige zeitaufwändig, kostenteuer und die erhaltenen Pericyten sind aufgrund der Anzahl der Kulturpassagen, die die Pericyten dedifferenzieren können, möglicherweise nichtideal. Darüber hinaus ist das Potenzial kultivierter Pericyten in In-vitro-Signalisierungsstudien bisher ziemlich unerforscht.
Die vorliegende Arbeit konzentriert sich auf die Erzeugung von Pericytkulturen aus isolierten Rinderhirnkapillaren und die anschließende Einrichtung von Messungen und bildgebenden Studien von Veränderungen des intrazellulären Kalziums, einem wichtigen intrazellulären zweiten Botenstoff. Wir beschreiben kurz die Isolierung von Kapillaren von kortikaler Graumaterie (Details siehe Helms et al.10) und die Isolation und Kultur von Pericyten in reiner Monokultur ohne Kontamination mit endothelialen oder glialen Zellen. Wir liefern dann ein Protokoll für die Aussaat von Pericyten in 96-Well-Platten und Ladeprotokolle für die Kalziumsonde Fura-2 AM. Schließlich zeigen wir, wie Pericyte in Echtzeit konfokale Bildgebung in Mikroskopkulturkammern verwendet werden können und beschreiben die Protokolle dafür.
Protokoll
1. Vorbereitung von Puffern und Lösungen für die Zellkultivierung
- Bereiten Sie Kollagen-Stammlösung durch Auflösen von 5 mg Kollagen IV aus menschlicher Plazenta in 50 ml PBS über Nacht bei 4 °C. Aliquot die Lagerlösung in 5 ml Portionen und lagern bei -20 °C.
- Bereiten Sie Fibronectin-Stammlösung durch Auflösen von 5 mg Fibronectin in 5 ml sterilem Wasser über Nacht. Bewahren Sie die Fibronectin-Bestände in Aliquots von 500 l bei -20 °C auf. Wenn Sie auftauen, fügen Sie PBS zu einem Endvolumen von 50 ml hinzu, um die Arbeitslösung vorzubereiten und bei 4 °C zu lagern.
- Bereiten Sie Dulbeccos Modified Eagle Medium (DMEM) komplettes Medium vor, indem Sie 50 ml fetales Rinderserum (FBS), 5 ml MEM nicht essentielle Aminosäuren und 5 ml Penicillin/Streptomycin (0,1 g/L Streptomycinsulfat und 100.000 U/L Penicillin G-Natrium) zu 500 ml DMEM hinzufügen.
- Bereiten Sie 5 mg/ml Heparin-Stammlösung vor, indem Sie Heparin-Natriumsalz in PBS auflösen und durch einen 0,2 m-Filter zur Sterilisation passieren. Lagern Sie die Lagerlösung bei 4 °C.
- Bereiten Sie Wachstumsmedium (GM) unmittelbar vor der Verwendung; 10 ml DMEM-comp und 250 l Heparin-Lagerlösung pro T75-Kolben mischen.
2. Isolierung von Kapillaren aus frischem Rinderhirn
HINWEIS: Rinderhirnkapillaren werden isoliert und kultiviert, wie zuvor beschrieben (Helms et al.10).
- Sammeln Sie Gehirne von Kälbern, die nicht älter als 12 Monate sind, aus einem Schlachthof und bringen Sie sie direkt auf Eis ins Labor.
- Entfernen Sie die Meninges und sammeln Sie alle grauen Materie aus dem Gehirn mit einem Skalpell. Identifizieren Sie die Meninges als den Film, der das Gehirn und die graue Materie durch seine graue Farbe bedeckt.
- Verwenden Sie einen 40 ml Dounce Gewebeschleifer, um die graue Materie in Dulbeccos Modified Eagle Medium (DMEM) zu homogenisieren. Füllen Sie den schlanken Teil des Gewebeschleifers 1/5 mit grauer Stoffsuspension und fügen Sie DMEM hinzu, bis das schlanke Teil gefüllt ist.
- Trennen Sie die Kapillaren von freien Zellen und kleineren Gewebestücken durch Filtration des Homogenats durch einen 160-m-Nylonnetzfilter. Spülen Sie die Filter mit DMEM-comp. Holen Sie die Kapillaren ab und bündeln Sie die Suspensionen in 50 ml Zentrifugationsrohre.
- Setzen Sie die Kapillaren in DMEM-comp wieder auf und fügen Sie einen Enzymmix aus DNase I (170 U/ml), Kollagenase Typ III (200 U/ml) und Trypsin (90 U/ml) hinzu. Lassen Sie die Suspension für 1 h in einem 37 °C Wasserbad für die Verdauung der Kapillaren.
- Führen Sie die Suspension durch einen 200-m-Netzfilter und setzen Sie sie in FBS mit 10 % Dimethylsulfoxid (DMSO) wieder auf. Die Kapillaren über Nacht bei -80 °C einfrieren und am Tag danach zur Langzeitlagerung in flüssigen Stickstoff bringen.
HINWEIS: Das Protokoll kann hier angehalten werden.
3. Aussaat und Kultivierung von Rinderkapillaren
- Tag 0: Mischen Sie 0,7 ml Kollagen IV-Lager mit 6,3 ml PBS. Fügen Sie die Lösung in einen T75-Kolben und lassen Sie den Kolben für 2 h bei Raumtemperatur (RT) oder lassen Sie es über Nacht bei 4 °C.
- Entfernen Sie die Kollagenlösung aus dem Kolben und waschen Sie sie dreimal mit PBS.
- Fügen Sie 7 ml Fibronectin Arbeitslösung und lassen Sie den Kolben für 30 min bei RT. Entfernen Sie dann die Fibronectin-Lösung und säen Sie die Kapillaren unmittelbar danach.
- Während der 30 min Wartezeit eine Durchstechflasche Kapillaren in einem 37 °C Wasserbad auftauen.
- Wenn die Kapillaren aufgetaut sind, sofort in ein Zentrifugationsrohr mit 30 ml DMEM-Comp und Zentrifuge für 5 min bei 500 x g und RT übertragen. DMEM-comp aus dem Rohr nehmen und das Kapillarpellet in 10 ml frischem DMEM-comp wieder aufhängen.
- Die 10 ml-Aufhängung auf den beschichteten T75-Kolben übertragen und die Kapillaren an der Unterseite des Kolbens für 4-6 h in einem 37 °C-Inkubator bei 10%CO2kleben lassen.
HINWEIS: Die Zellwachstumsrate ist mit 10%CO2 höher als die herkömmliche 5%CO2. - Nach 4-6 h Inkubation inspizieren Sie den Kolben unter einem Lichtmikroskop. An der Unterseite des Kolbens sollten nun Kapillarfraktionen befestigt werden(Abbildung 1, Tag 0).
- Bereiten Sie GM vor und aspirieren Sie das DMEN-Comp Medium sehr vorsichtig von den Kapillaren und ersetzen Sie es durch 10 ml frisch hergestellte GM.
- Tag 2: Entfernen Sie GM aus den Kapillaren und ersetzen Sie es durch 10 ml frisch hergestellte GV. Zelluläres Auswuchs aus den Kapillaren sollte an dieser Stelle unter einem Lichtmikroskop sichtbar sein(Abbildung 1, Tag 2-3).
4. Isolierung von primären Pericyten aus Rinderhirnkapillaren
-
Tag 4: Prüfen Sie die Kapillaren unter einem Lichtmikroskop.
HINWEIS: Der Kolben sollte jetzt etwa 60-70% konfluent sein, um eine angemessene Menge an Pericyten bereitzustellen(Abbildung 1,Tag 4). Wenn dies nicht der Fall ist; den GM durch 10 ml frisches Medium ersetzen und den Kolben für einen weiteren Tag im Inkubator lassen. - Das Medium ansaugen und die Zellen in PBS sanft waschen.
- Fügen Sie 2 ml aufgetauttry Trypsin-EDTA für Endothelzellen hinzu und lassen Sie den Kolben im Inkubator für 1-3 min. Nehmen Sie den Kolben häufig heraus und beobachten Sie während dieser Zeit mit dem Mikroskop.
HINWEIS: Die Endothelzellen sollten sich aufrunden und sich vom Kolben lösen; Pericyten sollten als Zellen mit einer "Geister"-Morphologie sichtbar sein und trotzdem an der Oberfläche des Kolbens befestigt werden. Dies ist ein schwieriger und wichtiger Schritt. Es ist wichtig, die meisten Endothelzellen zu entfernen, um eine Kontamination der Pericyte-Monokultur zu vermeiden, aber eine längere Trypsinisierung kann auch die Pericyten lösen. Die Trypsinisierungszeit kann von Zeit zu Zeit leicht variieren, und daher ist es von größter Bedeutung, den Kolben während der Behandlung häufig mit dem Mikroskop zu beobachten. - Tippen Sie vorsichtig auf den Kolben, wenn die Endothelzellen begonnen haben, sich aufzurunden, um die gelockerten Endothelzellen zu lösen.
- Um die Trypsinisierung zu stoppen, fügen Sie dem Kolben 10 ml DMEM-comp hinzu. Spülen Sie den Kolben vorsichtig ein paar Mal mit dem Medium, um die Endothelzellen zu entfernen. Aspirieren Sie die Endothelzellsuspension aus dem Kolben. Die Endothelzellen können nun für andere Zwecke verwendet werden.
- Fügen Sie dem Kolben 10 ml DMEM-comp hinzu. Schauen Sie unter das Lichtmikroskop, um sicherzustellen, dass die Pericyten noch vorhanden und am Boden befestigt sind. Legen Sie den Kolben wieder in den Brutkasten, damit die pericyte-angereicherte Kultur wachsen kann.
HINWEIS: Es ist wichtig, die Kultur in den folgenden Tagen zu beobachten. Wenn es immer noch eine angemessene Menge an Endothelzellen wächst, kann eine weitere Trypsin-Behandlung durchgeführt werden. - Lassen Sie die Pericyte-Monokultur mit der Veränderung von DMEM-comp wachsen. medium jeden zweiten Tag. Überprüfen Sie das Wachstum der Zellen unter dem Lichtmikroskop(Abbildung 1, Tag 5-8).
5. Erzeugung und Lagerung einer Monokultur von primären Rinderpericyten
-
Tag 8-9: Prüfen Sie die Kapillaren unter einem Lichtmikroskop
HINWEIS: Die Pericyten sollten nun 70-80% Konfluenz erreicht haben und in Inseln im Kolben wachsen(Abbildung 1,Tag 9). Wenn die Konfluenz der Pericyten weniger als 70% beträgt, lassen Sie die Zellen für einen anderen Tag wachsen. Die Pericyten bilden keine vollständige Monoschicht, wie es die Endothelzellen tun würden. - DMEM-comp aspirieren und die Pericyten mit 7 ml PBS waschen.
- Fügen Sie 2 ml Trypsin-EDTA in den Kolben und lassen Sie es im Inkubator für 2-3 min. Stellen Sie den Kolben häufig unter das Lichtmikroskop, um zu beobachten, wann sich die Pericyten aufbahnen und sich vom Kolben lösen. Wenn sich die Pericyten aufgerundet haben, kann der Kolben sanft angezapft werden, um die Zellen zu lösen.
- Tippen Sie vorsichtig auf den Kolben, wenn die Pericyten begonnen haben, sich zu runden, um die Zellen zu lösen.
- Fügen Sie dem Kolben 10 ml DMEM-comp hinzu, um den Trypsinisierungsprozess zu stoppen. Spülen Sie den Kolben ein paar Mal mit dem Medium, um die letzten Pericyten zu lösen.
- Die 12 ml Zellsuspension in ein 50 ml Zentrifugationsrohr übertragen und mit DMEM-comp bis zu 30 ml füllen.
- Zentrifugieren Sie die Zellsuspension für 5 min bei 500 x g und RT. Aspirieren Sie die DMEM-comp. ohne das Zellpellet zu berühren. Setzen Sie das Zellpellet in 3 ml FBS mit 10% DMSO wieder auf.
- Übertragen Sie die Zellsuspension in Kryovials; Fügen Sie jeweils 1 ml hinzu, so dass es insgesamt 3 Durchstechflaschen pro T75-Flasche Pericytes gibt. Die Pericyten über Nacht bei -80 °C einfrieren und am Tag danach zur Langzeitlagerung in flüssigen Stickstoff bringen.
HINWEIS: Zellen können vor dem Einfrieren für eine spätere Schätzung des Überlebensprozentsatzes gezählt werden. Das Protokoll kann hier angehalten werden.
6. Aufbau einer Pericyte-Monokultur für Experimente
- Mantel Sie einen T75-Kolben mit Kollagen IV und Fibronectin nach dem gleichen Verfahren wie in Abschnitt 3.1-3.4 erwähnt.
- Während der Kolben mit Fibronectin beschichtet wird, tauen Sie eine Durchstechflasche mit Pericyten in einem 37 °C-Wasserbad auf.
- Übertragen Sie die nun aufgetauten Pericyten vom Kryovial auf ein Zentrifugationsrohr mit 30 ml DMEM-Comp. Zentrifugieren Sie die Zellsuspension für 5 min bei 500 x g,RT.
- Saugen Sie das Medium vorsichtig an und lassen Sie das Zellpellet an der Unterseite des Rohres zurück. Das Pellet in 10 ml DMEM-comp wieder aufhängen.
- Sammeln und übertragen Sie die Zellsuspension in den beschichteten Kolben. Lassen Sie den Kolben mit Pericyten in einem 37 °C Inkubator bei 10%CO2wachsen.
- Jeden zweiten Tag das Medium mit 10 ml frischem DMEM-Comp auffrischen.
HINWEIS: Nach 5 Tagen Wachstum sollten die Pericyten etwa 80% Konfluenz erreicht haben. Wenn die Konfluenz geringer ist, lassen Sie die Zellen für einen oder zwei Tage wachsen. Die Zellen sollten nun für weitere Experimente aussaat bereit sein.
7. Aussaat von Pericyten in einer beschichteten 96-Well-Platte
- Kollagen IV verdünnen, wie in Schritt 3.1 beschrieben. Fügen Sie jedem Brunnen 100 l in eine 96-Well-Platte und brüten Sie 2 h bei RT oder über Nacht bei 4 °C.
- Aspirieren Sie die Lösung und waschen Sie die Brunnen dreimal mit PBS.
- Fügen Sie jedem Brunnen 100 l verdünntes Fibronectin hinzu und brüten bei RT 30 min. Entfernen Sie die Fibronectin-Lösung und verwenden Sie die Platte sofort.
HINWEIS: Je nachdem, wie gut die Pericyt-Charge wächst, sollte es genügend Zellen geben, um zwei Platten zu säen. - Nehmen Sie die Pericyten aus dem Inkubator und aspirieren Sie das Medium. Waschen Sie die Zellen mit PBS.
- Fügen Sie 2 ml Trypsin-EDTA zu den Pericyten hinzu und folgen Sie dem gleichen Verfahren wie in Schritt 5.3-5.6.
- Das Medium ansaugen, ohne das Zellpellet zu beschädigen, und das Pellet in 1 ml frischem DMEM-Comp wieder aufhängen.
- Nehmen Sie 12 L Zellsuspension heraus und fügen Sie sie zu einer Zählkammer hinzu. Zählen Sie unter dem Lichtmikroskop mindestens 3 von 3x3 Gittern und verwenden Sie die durchschnittliche Zellzahl pro Raster.
- Verwenden Sie die folgende Gleichung, um das Volumen der Zellsuspension zu berechnen, das jedem Brunnen hinzugefügt werden sollte, um 10.000 Zellen pro Bohrkörper in der 96-Well-Platte auszusäen.
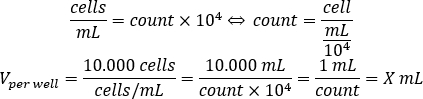
- Fügen Sie DMEM-comp und das berechnete Volumen der Zellsuspension in jedem Bohrkörper hinzu, um ein Endvolumen von 200 l zu erreichen.
- Legen Sie die 96-Well-Platte in einen 37 °C-Inkubator bei 10%CO2. Lassen Sie die Zellen für 4 Tage mit einem Wechsel des Mediums nach 2 Tagen wachsen.
8. Vorbereitung von Puffern und Lösungen für Ca2+-Imaging
- Autoklav-Abdeckungs-Zellkammern und Abdeckungen.
- Testpuffer: Fügen Sie 1,19 g HEPES zu 500 ml HBSS-Puffer für eine Endkonzentration von 10 mM HEPES hinzu. Stellen Sie den pH-Wert auf 7,4 ein.
- Bereiten Sie 20% (w/v) Pluronic F127 + 1% (v/v) polyethoxylierte Rizinusöl-Lagerlösung vor, indem Sie 0,5 g Pluronic F127-Lösung in 2,5 ml wasserfreiem DMSO in einer Glasdurchstechflasche auflösen. Auf ca. 30 min oder bis auf 40 °C erhitzen und wirbeln. Fügen Sie 25 l polyethoxyliertes Rizinusöl hinzu und lagern Sie bei RT. Nicht einfrieren.
- Bereiten Sie 2 mM Fura-2 AM Aktie vor, indem Sie 1 mg in 500 l wasserfreies DMSO auflösen. In Aliquots von 20 l bei -20 °C lichtgeschützt aufbewahren.
- Bereiten Sie die Ladelösung 5 'M Fura-2 AM vor, indem Sie 20 'L von 20% Pluronic F-127 + 1% polyethoxylierte Rizinusöl-Lagerlösung mit der 20'L von 2 mM Fura-2 AM aliquot mischen. Fügen Sie 500 L Assay-Puffer und Wirbel hinzu. Fügen Sie einem Endvolumen von 8 ml einen Assaypuffer hinzu. Die Lösung sollte unmittelbar vor gebrauchen vorbereitet und vor Licht geschützt werden.
- Bereiten Sie 4 mM Cal-520 AM vor, indem Sie 1 mg in 226,7 l wasserfreies DMSO auflösen. In Aliquots von 20 l bei -20 °C lichtgeschützt aufbewahren.
- Bereiten Sie 20 M Cal-520 AM Ladelösung vor, indem Sie 20 l von 20% Pluronischem F-127 + 1% polyethoxyliertes Rizinusöllager mit der 20 l 4 mM Cal-520 aliquot mischen. Fügen Sie 500 L Assay-Puffer und Wirbel hinzu. Fügen Sie dem Endvolumen von 4 ml einen Assaypuffer hinzu. Die Lösung sollte unmittelbar vor gebrauchen vorbereitet und vor Licht geschützt werden.
9. Beladen von Pericyten mit Fura-2 AM Calciumindikatorfarbstoff in einem Plattenleser-Setup
HINWEIS: Alle Lösungen sollten sich vor Beginn des Experiments bei RT aufstellen.
- Nehmen Sie die 96-Well-Platte mit Zellen aus dem Inkubator und aspirieren Sie das Medium aus den Brunnen. Waschen Sie die Zellen zweimal mit Assay-Puffer.
- Fügen Sie jedem Bohrgut 100 l Ladelösung hinzu und wickeln Sie die Platte mit Tinfoil, um Fotobleichungen zu vermeiden. 45 min mit 30 Umdrehungen bei RT bebrüten.
HINWEIS: Tragen Sie Fura-2 AM nicht bei 37 °C, da es interne Fächer laden kann. Denken Sie daran, Brunnen mit Zellen im Assay-Puffer anstelle des Ladepuffers zu lassen; dies sind die "Blanks", die zur Messung der Hintergrundfluoreszenz verwendet werden. - Den Ladepuffer ansaugen und die Zellen zweimal mit Assaypuffer waschen. Fügen Sie 100 l frischen Assay-Puffer hinzu und lassen Sie die Zellen 30 min bei RT brüten; Dies ermöglicht eine kontinuierliche Spaltung des AM-Esters und damit das Einfangen von Fura-2 AM in den Zellen.
- Waschen und ersetzen Sie vor der Ca2+-Imaging den Puffer mit 100 l frischem Assaypuffer.
10. Well-Platte Fluoreszenz-Messung von Pericyten in einem Plattenleser-Setup
- Stellen Sie die Temperatur des Plattenlesers auf 37 °C ein und übertragen Sie die 96-Well-Platte mit Zellen in die Probenplattenposition. Legen Sie die Reagenzplatte mit Agonist an der Reagenzplattenposition.
- Beginnen Sie mit der Messung der Belastung der Zellen, um eine gleichmäßige Beladung von Fura-2 AM in allen Brunnen zu gewährleisten.
- Führen Sie die Messungen mit anregungsfluoreszenzwellenwellungbei 340 nm/380 nm und die Emissionswellenlänge bei 510 nm durch. Fügen Sie 50 L Agonist bei Geschwindigkeit 150 l/s von der Reagenzplatte zu jedem Brunnen mit Zellen in der Probenplattenposition hinzu.
- Speichern Sie die Daten und exportieren Sie sie als xlsx-Dateien zur weiteren Analyse. Abbildung 2 zeigt die zytosolische Ca2+-Antwort,gemessen als Verhältnis zwischen den beiden Anregungswellenlängen im Zeitverlauf, bei denen die Hintergrundfluoreszenz subtrahiert wird.
HINWEIS: Der Plattenleser muss ein Dual-Mikroplattenleser sein, der Platz für ein "Zellfach" und ein "Probenfach" und ein integriertes Pipettorsystem bietet.
11. Aussaat von Pericyten in einer beschichteten Zellkammer zur Live-Bildgebung
HINWEIS: Coverlips können auch in den Boden von Kulturbrunnen gelegt, beschichtet und mit Pericyten wie oben beschrieben besät und dann in der Kammer vor Experimenten montiert werden.
- Montieren Sie einen Deckelrutsch in die Zellkammer und machen Sie ihn fest, um Leckagen zu vermeiden.
- Kollagen IV verdünnen, wie in Schritt 3.1 beschrieben. Fügen Sie jeder Zellkammer 500 l hinzu und brüten Sie 2 h bei RT oder über Nacht bei 4 °C.
- Aspirieren Sie die Kollagenlösung und waschen Sie die Kammern dreimal mit 500 l PBS.
- Fügen Sie jedem Brunnen 500 l verdünntes Fibronectin hinzu und brüten bei RT 30 min. Entfernen Sie die Fibronectin-Lösung und verwenden Sie die Zellkammer direkt danach.
- In der Zwischenzeit den Kolben mit konfluenten Pericyten herausnehmen und mit 7 ml PBS waschen.
- Fügen Sie 2 ml Trypsin-EDTA zu den Pericyten hinzu und folgen Sie dem gleichen Verfahren wie in Schritt 5.3-5.6.
- Gehen Sie vor, indem Sie die gleichen Schritte wie in Schritt 8.6-8.7 ausführen.
- Verwenden Sie die folgende Gleichung, um das Volumen der Zellsuspension zu berechnen, die jeder Kammer hinzugefügt werden sollte, um 90.000 Zellen pro Kammer auszusäen.
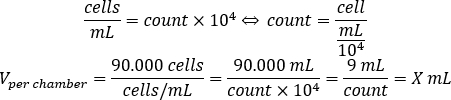
- Fügen Sie DMEM-comp und das berechnete Volumen der Zellsuspension in jeder Kammer hinzu, um ein Endvolumen von 500 l zu erreichen.
- Legen Sie die Zellkammern im Inkubator bei 37 °C, 10%CO2. Lassen Sie die Zellen für 6 Tage wachsen (oder bis konfluent).
HINWEIS: Die Pericyten wachsen auf Glasrutschen langsamer als bei Kunststoff; mehr Tage Wachstum sind notwendig.
12. Beladen von Pericyten mit Cal-520 AM Calciumindikatorfarbstoff für Live-Bildgebung
HINWEIS: Alle Lösungen sollten sich vor Beginn des Experiments bei RT aufstellen.
- Bereiten Sie den 20-M-Ladepuffer Cal-520 AM vor: Mischen Sie 20 l von 20% Pluronischem F-127 + 1% polyethoxyliertes Rizinusöl-Ölvorrat mit der 20 l 4 mM Cal-520 aliquot. Fügen Sie 500 von 'L-Assay-Puffer und Wirbel hinzu. Fügen Sie dem Endvolumen von 4 ml einen Assaypuffer hinzu. Die Lösung sollte unmittelbar vor gebrauchen vorbereitet und vor Licht geschützt werden.
HINWEIS: Schützen Sie Lösungen, die Cal-520 AM enthalten, vor Lichteinwirkung. - Nehmen Sie die Zellkammern aus dem Inkubator und aspirieren Sie das Medium. Waschen Sie die Zellen zweimal mit Assay-Puffer.
- Fügen Sie jeder Kammer 500 l Ladepuffer hinzu und inkubieren Sie bei RT 45 min.
- Den Ladepuffer ansaugen und die Zellen zweimal mit Assaypuffer waschen.
- Fügen Sie jeder Kammer 500 l frischen Assaypuffer hinzu und brüten Sie 30 min bei RT, um eine Spaltung des AM-Esters zu ermöglichen.
- Ersetzen Sie den Puffer durch 500 l frischen Assaypuffer, bevor Sie die Live-Bildgebung an einem konfokalen Mikroskop durchführen.
13. Live-Bildgebung von intrazellulären Ca2+-Ebenen
HINWEIS: Für die Bildgebung kann eine Vielzahl von Mikroskoptypen verwendet werden. Es können aufrechte oder invertierte konventionelle Fluoreszenzmikroskope sowie aufrechte oder invertierte konfokale Laserscanmikroskope mit entsprechender Anregungsquelle (488 nm) und Emissionsfiltern (510-520 nm) eingesetzt werden. Die Ziele sollten für die Fluoreszenz geeignet sein und von hoher Qualität und mit hoher numerischer Blende (NA) sein.
- Montieren Sie die Zellkammer auf der Bühne des konfokalen Mikroskops so schonend wie möglich, um Störungen der Zellen zu vermeiden.
- Wählen Sie eine Anregungswellenlänge von 488 nm, Emission bei 515 nm, sequentielle Bildaufnahme mit 5 Sekunden Intervallen, eine XY-Bildgröße von 512 x 512 Pixeln und messen Sie für 2 min, um Basis-Calciumsignale zu messen.
- Fügen Sie der Zellkammer mit einer Pipette 3 l von 100 mM ATP hinzu, und setzen Sie die sequenzielle Bildaufnahme fort. Führen Sie die Zugabe langsam und sanft aus, um das Präparat nicht zu stören und die Zellen aus dem Fokus zu bewegen.
- Beobachten Sie den Grad der Veränderungen und erhöhen Sie das Zeitintervall im Laufe der Zeit, wie erforderlich für ca. 18 min, bis keine weitere morphologische Veränderung festgestellt wird (Abbildung 3).
- Speichern Sie Zeitrafferbilder und exportieren Sie sie als TIFF- und/oder AVI-Dateien zur weiteren Analyse.
HINWEIS: Eine Durchstechflasche mit Pericyten sollte genügend Zellen für die Aussaat in 1-2 96-Well-Platten und mehrere Abdeckungen geben, was bedeutet, dass Sie Zellen für beide Arten von Kalziummessungen vorbereiten können.
Ergebnisse
Rinderhirnkapillaren wurden aus frischem Hirngewebe isoliert und Abbildung 1 stellt die Kapillarensaat und das zelluläre Auswachsen über Tage und die anschließende Reinigung von Pericyten dar. Die Kapillaren sind an Tag 1 vollständig am Kolben befestigt und am Tag 2 ist die Endothelsprossen sichtbar geworden (Abbildung 1, Tag 2). Nach 4 Tagen ist das zelluläre Auswachsen sehr markant(Abbildung 1...
Diskussion
In dieser Studie haben wir eine Methode vorgestellt, um primäre Pericyten von Rinderhirnen zu isolieren. Das beschriebene Protokoll ermöglicht die Kultur dieses ansonsten nicht zugänglichen Zelltyps. Die später erhaltene Zellkultur war eine nahezu homogene Population von Pericyten, mit wenig oder gar keiner Kontamination mit Endothelzellen und Gliazellen auf Der Grundlage von Zellmorphologie und Proteinexpression12. Darüber hinaus haben wir eine einfache und einfache Methode zum Beladen der P...
Offenlegungen
Die Autoren erklären keine konkurrierenden finanziellen Interessen.
Danksagungen
Die Autoren möchten die Förderung durch die Lundbeck Foundation Research Initiative on Brain Barriers and Drug Delivery (RIBBDD) und die Simon Hougners Family Foundation würdigen.
Materialien
| Name | Company | Catalog Number | Comments |
| ATP | Tocris | 3245 | |
| Cal-520 AM | AAT Bioquest | 21130 | |
| Cell incubator | Thermo Fisher | ||
| Centrifuge | Thermo Fisher | Heraeus Multifuge 3SR+ | Standard large volume centrifuge for spinning down cells |
| Collagen IV | Sigma Aldrich | C5533 | |
| Confocal laser scanning microscope | Carl Zeiss | Zeiss LSM 510 | Inverted microscope |
| Counting chamber | FastRead | 102 | |
| Coverslip cell chamber | Airekacells | SC15022 | |
| Cremophor EL | Sigma Aldrich | C5135 | Formerly known as Kolliphor EL |
| DMSO | Sigma Aldrich | 471267 | |
| Dulbecco's Modified Eagles Medium | Sigma Aldrich | D0819 | |
| Fetal bovine serum (FBS) | PAA/GE Healthcare | A15-101 | |
| Fibronectin | Sigma Aldrich | F1141 | |
| Fura-2 AM | Thermo Fisher | F1201 | |
| Glass coverslips 22x22 mm | VWR International | 631-0123 | |
| HBSS | Gibco | 14065-049 | |
| Heparin | Sigma Aldrich | H3149 | |
| HEPES | AppliChem Panreac | A1069 | |
| Light microscope | Olympus | Olympus CK2 | Upright light microscope with phase contrast |
| MEM nonessential amino acids | Sigma Aldrich | M7145 | |
| Microplate Reader | BMG LabTech | NOVOstar | |
| PBS | Sigma Aldrich | D8537 | Phosphate-buffered saline |
| penicillin G sodium/streptomycin sulfate | Sigma Aldrich | P0781 | |
| Pluronic F127 | Sigma Aldrich | P2443 | |
| Trypsin-EDTA | Sigma Aldrich | T4299 | |
| T-75 flask | Sigma Aldrich | CLS3972 | |
| 96-well plate | Corning incorporated | 3603 |
Referenzen
- Armulik, A., Genove, G., Betsholtz, C. Pericytes: Developmental, Physiological, and Pathological Perspectives, Problems, and Promises. Developmental Cell. 21 (2), 193-215 (2011).
- Abbott, N. J., Ronnback, L., Hansson, E. Astrocyte-endothelial interactions at the blood-brain barrier. Nature Reviews Neuroscience. 7 (1), 41-53 (2006).
- Mathiisen, T. M., Lehre, K. P., Danbolt, N. C., Ottersen, O. P. The Perivascular Astroglial Sheath Provides a Complete Covering of the Brain Microvessels: An Electron Microscopic 3D Reconstruction. Glia. 58 (9), 1094-1103 (2010).
- Helms, H. C., et al. In vitro models of the blood-brain barrier: An overview of commonly used brain endothelial cell culture models and guidelines for their use. Journal of Cerebral Blood Flow and Metabolism. 36 (5), 862-890 (2016).
- Zhou, L., Sohet, F., Daneman, R. Purification of Pericytes from Rodent Optic Nerve by Immunopanning. Cold Spring Harbor Protocols. , 608-617 (2014).
- Liu, G. H., et al. Isolation, Purification, and Cultivation of Primary Retinal Microvascular Pericytes: A Novel Model Using Rats. Microcirculation. 21 (6), 478-489 (2014).
- Nayak, R. C., Herman, I. M. Bovine retinal microvascular pericytes: isolation, propagation, and identification. Methods In Molecular Medicine. 46, 247-263 (2001).
- Nees, S., et al. Isolation, bulk cultivation, and characterization of coronary microvascular pericytes: the second most frequent myocardial cell type in vitro. American Journal of Physiology-Heart and Circulatory Physiology. 302 (1), H69-H84 (2012).
- Armulik, A., Abramsson, A., Betsholtz, C. Endothelial/pericyte interactions. Circulation Research. 97 (6), 512-523 (2005).
- Helms, H. C., Brodin, B., Milner, R. Methods in Molecular Biology. Cerebral Angiogenesis: Methods and Protocols. 1135, 365-382 (2014).
- Abbracchio, M. P., Burnstock, G., Verkhratsky, A., Zimmermann, H. Purinergic signalling in the nervous system: an overview. Trends in Neurosciences. 32 (1), 19-29 (2009).
- Cai, C., et al. Stimulation-induced rises in cerebral blood flow and local capillary vasoconstriction depend on conducted vascular responses in brain capillaries. PNAS. , (2018).
- Tigges, U., Welser-Alves, J. V., Boroujerdi, A., Milner, R. A novel and simple method for culturing pericytes from mouse brain. Microvascular Research. 84 (1), 74-80 (2012).
- Maier, C. L., Shepherd, B. R., Yi, T., Pober, J. S. Explant Outgrowth, Propagation and Characterization of Human Pericytes. Microcirculation. 17 (5), 367-380 (2010).
- Orlidge, A., Damore, P. A. Cell specific effects of glycosaminoglycans on the attachment and proliferation of vascular wall components. Microvascular Research. 31 (1), 41-53 (1986).
- Rhinehart, K., Zhang, Z., Pallone, T. L. Ca2+ signaling and membrane potential in descending vasa recta pericytes and endothelia. American Journal of Physiology-Renal Physiology. 283, F852-F860 (2002).
- Bintig, W., et al. Purine receptors and Ca2+ signalling in the human blood-brain barrier endothelial cell line hCMEC/D3. Purinergic Signalling. 8 (1), 71-80 (2012).
- Neuhaus, A. A., Couch, Y., Sutherland, B. A., Buchan, A. M. Novel method to study pericyte contractility and responses to ischaemia in vitro using electrical impedance. Journal of Cerebral Blood Flow and Metabolism. 37 (6), 2013-2024 (2017).
- Kamouchi, M., et al. Calcium influx pathways in rat CNS pericytes. Molecular Brain Research. 126 (2), 114-120 (2004).
- Das, A., et al. ATP Causes Retinal Pericytes to Contract In vitro. Experimental Eye Research. 46, 349-362 (1988).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten