Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
High-Throughput Live Imaging von Mikrokolonien zur Messung der Heterogenität in Wachstum und Genexpression
In diesem Artikel
Zusammenfassung
Hefewachstumsphänotypen werden präzise durch hochparallele Zeitraffer-Bildgebung von immobilisierten Zellen gemessen, die zu Mikrokolonien heranwachsen. Gleichzeitig können Stresstoleranz, Proteinexpression und Proteinlokalisierung überwacht werden, wodurch integrierte Datensätze generiert werden, um zu untersuchen, wie ökologische und genetische Unterschiede sowie die Genexpressionsheterogenität unter isogenen Zellen das Wachstum modulieren.
Zusammenfassung
Präzise Messungen der Heterogenität zwischen und innerhalb der Dehnung in mikrobiellen Wachstumsraten sind entscheidend für das Verständnis genetischer und ökologischer Inputs in Stresstoleranz, Pathogenität und anderen Schlüsselkomponenten der Fitness. Dieses Manuskript beschreibt einen mikroskopischen Assay, der pro Experiment etwa 105 Saccharomyces cerevisiae mikrocolonies nachverfolgt. Nach der automatisierten Zeitraffer-Bildgebung von Hefe, die in einer Multiwell-Platte immobilisiert ist, lassen sich die Wachstumsraten von Mikrokolonien mit einer benutzerdefinierten Bildanalysesoftware einfach analysieren. Für jede Mikrokolonie können auch die Expression und Lokalisation fluoreszierender Proteine und das Überleben akuter Belastungen überwacht werden. Dieser Test ermöglicht eine genaue Schätzung der durchschnittlichen Wachstumsraten der Stämme sowie eine umfassende Messung der Heterogenität in Wachstum, Genexpression und Stresstoleranz innerhalb klonaler Populationen.
Einleitung
Wachstumsphänotypen tragen entscheidend zur Hefefitness bei. Die natürliche Selektion kann effizient zwischen Linien unterscheiden, wobei die Wachstumsraten durch die Umkehrung der effektiven Bevölkerungsgröße abweichen, die 108 Individuen1 überschreiten kann. Darüber hinaus ist die Variabilität der Wachstumsraten bei Individuen innerhalb einer Bevölkerung ein evolutionär relevanter Parameter, da er als Grundlage für Überlebensstrategien wie Wetten hedging2,3,4,5,6dienen kann. Daher sind Assays, die hochgenaue Messungen von Wachstumsphänotypen und deren Verteilungen ermöglichen, für die Untersuchung von Mikroorganismen von entscheidender Bedeutung. Der hier beschriebene Mikrokolonie-Wachstumstest kann individuelle Wachstumsratenmessungen für 105 Mikrokolonien pro Experiment generieren. Dieser Test bietet daher ein leistungsfähiges Protokoll, um Hefe evolutionäre Genetik und Genomik zu studieren. Es eignet sich besonders gut, um zu testen, wie Variabilität innerhalb von Populationen von genetisch identischen Einzelzellen erzeugt, gepflegt wird und zur Eignung der Bevölkerung beiträgt7,8,9,10.
Die hier beschriebene Methode (Abbildung 1) verwendet periodisch erfasste Hellfeldbilder von Zellen mit geringer Vergrößerung, die in flüssigen Medien auf einer 96- oder 384-Well-Glasbodenplatte wachsen, um das Wachstum in Mikrokolonien zu verfolgen. Die Zellen haften an dem Lektin Concanavalin A, das den Boden der Mikroskopplatte überzieht, und bilden zweidimensionale Kolonien. Da die Mikrokolonien in einer Monoschicht wachsen, ist das Mikrokoloniegebiet stark mit der Zellnummer7korreliert. Daher können genaue Schätzungen der Wachstumsrate und Verzögerungszeit von Mikrokolonien mit einer benutzerdefinierten Bildanalysesoftware generiert werden, die die Änderungsrate der Fläche jeder Mikrokolonie nachverfolgt. Darüber hinaus kann der Versuchsaufbau die Häufigkeiten und sogar die subzellulären Lokalisationen fluoreszierend markierter Proteine überwachen, die in diesen Mikrokolonien exprimiert werden. Die nachgeschaltete Verarbeitung von Daten aus diesem Mikrokolonie-Wachstumstest kann durch benutzerdefinierte Analyse oder durch vorhandene Bildanalysesoftware erreicht werden, wie z. B. Processing Images Easily (PIE)11, ein Algorithmus für robuste Kolonieflächenerkennung und Wachstumsanalyse mit hohem Durchsatz aus Hellfeldbildern mit geringer Vergrößerung, die über GitHub12verfügbar sind.
Da wachstumsfördernde Schätzungen, die aus dem Mikrokolonie-Wachstumstest abgeleitet werden, aus einer großen Anzahl von Einzelkolonienmessungen generiert werden, sind sie extrem genau, mit Standardfehlern, die um mehrere Größenordnungen kleiner sind als die Schätzungen selbst für ein angemessen großes Experiment. Daher ist die Kraft des Assays, Wachstumsratenunterschiede zwischen verschiedenen Genotypen, Behandlungen oder Umgebungsbedingungen zu erkennen, hoch. Das Multiwell-Plattenformat ermöglicht den Vergleich zahlreicher unterschiedlicher Umgebungs- und Genotypkombinationen in einem einzigen Experiment. Wenn Stämme konstitutiv unterschiedliche fluoreszierende Marker exprimieren, können sie in der gleichen Bohrung gemischt und durch nachfolgende Bildanalyse unterschieden werden, was die Leistung weiter erhöhen könnte, indem sie eine gut-durch-gut-datennormalisierung ermöglichen.
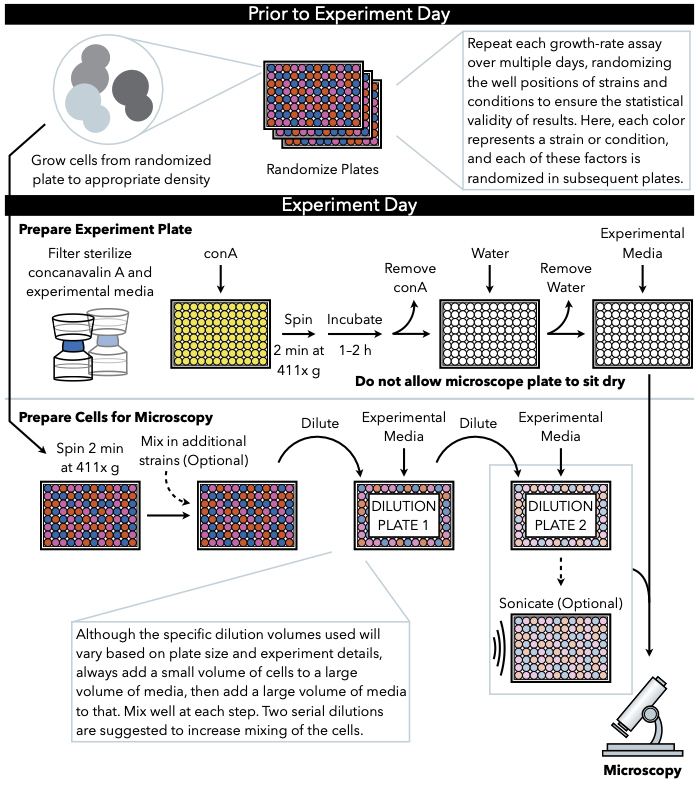
Abbildung 1:Schematische Darstellung des Protokolls. Dieses Protokoll folgt zwei Hauptschritten, die die Vorbereitung der Versuchsplatte und die Vorbereitung der Zellen auf das Bild sind. Die Randomisierung der Platten und das Wachstum der Zellen sollten vor und vor dem Experimenttag durchgeführt werden. Das wiederholte Mischen von Zellen bei jedem Schritt während der Verdünnung ist in den Schritten bis zur Beschichtung zwingend erforderlich, und daher wird die Vorbereitung der Versuchsplatte zuerst empfohlen, so dass sie sofort nach Abschluss der Zellverdünnung zur Beschichtung bereit ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Vorbereitung randomisierter Platten (vor dem Experimenttag)
- Planen Sie die Belastungen und Bedingungen, die mit dem Wachstumstest getestet werden sollen. An diesem Punkt, zufällig zuweisen Stämme und Bedingungen zu jedem Brunnen.
HINWEIS: Bei der Prüfung der Platteneinrichtung ist es ratsam, mehr als eine Replikation pro Dehnung und Wachstumszustand auf einer einzigen Platte einzuschließen, um gut verwandte Geräusche in Messungen zu berücksichtigen. Weitere Informationen finden Sie unter Diskussion. - Berechnen sie die Position jeder Dehnung und Umgebungsbedingung für Plattenreplikationen, die an verschiedenen Tagen ausgeführt werden.
- Wachsen Sie alle Zellen, die im Experiment zur Sättigung in Hefeextrakt-Pepton-Dextrose (YEPD; 2% Glukose) Medium in einem Shaker bei 30 °C (oder einer anderen geeigneten Temperatur) verwendet werden.
- Erstellen Sie die randomisierten Lagerplatten entweder manuell oder mit einem Flüssigkeitshandling-Roboter. Fügen Sie jedem Brunnen einer sterilen U-Boden-Gewebekulturplatte 10 l der bezeichneten gesättigten Zellen hinzu. Wenn mehrere Stämme in einem einzigen Brunnen getestet werden, kombinieren Sie sie an dieser Stelle nicht; Diese Kombination wird kurz vor den Zellverdünnungen am Tag des Experiments durchgeführt, um sicherzustellen, dass alle Stämme in den richtigen Konzentrationen sind, wenn sie als Mikrokolonie-Gründerzellen plattiert werden.
- Fügen Sie jedem Bohrgut jeder Platte 10 l 30 % Glycerin hinzu. Pipetten Sie nach oben und unten, so dass die Zellen und das Glycerin gut gemischt werden.
- Versiegeln Sie jede Platte mit einer Folienabdeckung und frieren Sie sofort bei -70 °C ein, bis sie einsatzbereit ist.
HINWEIS: Es ist wichtig, alle randomisierten Platten am selben Tag zu erstellen und einzufrieren, so dass die Vorwachstumsbedingungen der Zellen in jeder Platte identisch waren und keine technischen Unterschiede im Wachstumsratentest erzeugen.
2. Vorwachstum der Hefe
HINWEIS: In der Regel beginnt dies vor dem Experimenttag und ist in hohem Maße von der experimentellen Frage abhängig. Weitere Informationen finden Sie unter Diskussion.
- Entfernen Sie eine Stammplatte (10 l Hefe, 10 l Glycerin pro Bohrgut) aus dem Gefrierschrank -70 °C und fügen Sie 180 L des für das Experiment zu verwendenden Mediums hinzu. Wenn das Experiment mit nährstoffbegrenzenden Medien durchgeführt wird, nicht vorwachsen Hefe zu Sättigung in den nährstoffbegrenzenden Medien als Sporulation von Hefe auftreten kann. Stattdessen wachsen Sie in nicht-begrenzenden Medien.
- Hefe wachsen, während sie bei 30 °C schüttelt. Überlegen Sie, ob der Test mit Zellen in der Protokollphase oder in der stationären Phase ausgeführt werden soll, um zu bestimmen, ob eine verdünnung der Zellen vor dem Experiment erforderlich ist. Wenn erwartet wird, dass die Hefestämme oder Bedingungen im Test deutlich unterschiedliche Wachstumsraten aufweisen, dann wird eine zweitägige Vorwachstumsphase notwendig sein, damit alle unterschiedlichen Bedingungen die stationäre Phase erreichen.
3. Mikroskop-Setup
- Mikroskopplattenvorbereitung
- Stellen Sie sicher, dass der Mikroskop-Inkubator eingeschaltet ist und erwärmen Sie die Mikroskopkammer auf die gewünschte Wachstumstemperatur für die experimentellen Bedingungen. Bei Standardexperimenten mit Saccharomyces cerevisiae-Zellen sollte der Inkubator die Mikroskopkammer auf 30 °C erwärmen, um sicherzustellen, dass die Wachstumsbedingungen für die Zellen während des Wachstumsraten-Assays korrekt sind.
- Sanitisieren Sie die Werkbank, Pipetten und andere Werkzeuge mit 70% Ethanol. Holen Sie sich eine Mikroskopplatte und legen Sie sie auf die Bank auf einem fussel- und statisch-freien Wisch.
HINWEIS: Berühren Sie niemals den Boden der Mikroskopplatte, auch nicht mit Handschuhen, und stellen Sie die Mikroskopplatte immer auf ein fussel- und statisch-freies Wischen, wenn es eine Oberfläche berührt. Dadurch wird verhindert, dass Flecken oder Kratzer Wachstumsmessungen behindern, sobald das Experiment abgebildet wird. - 5 ml 5x Concanavalin Eine Lösung auftauen, mit Wasser auf 1x verdünnen und durch eine Spritze mit einem 0,2-mm-Filter sterilisieren.
- Filter sterilisieren alle anderen Flüssigkeiten, die im Test mit einem 0,2 m-Filter verwendet werden, einschließlich experimenteller Medien, um Kristalle oder Schmutz zu entfernen, die in den Lösungen materialisiert wurden. Das Vorhandensein von Kristallen würde die Qualität der Mikroskopiebilder verringern.
- Pipetten Sie 200 l Concanavalin Eine Lösung in jeden Brunnen der Mikroskopplatte.
- Zentrifugieren Sie die Platte für 2 min bei 411 x Schwerkraft (g) mit einem fussel- und statisch-freien Wisch unter der Platte, um sicherzustellen, dass die Concanavalin-A-Lösung den Boden jedes Brunnens gleichmäßig abdeckt und keine Luftblasen vorhanden sind.
- Bedecken Sie die Platte mit ihrem Deckel und lassen Sie sie für 1-2 h sitzen. Die genaue Zeit, in der die Platte sitzt, ist flexibel, aber es ist wichtig, zwischen verschiedenen Durchläufen des Experiments konsistent zu sein.
- Entfernen Sie die gesamte Concanavalin-A-Lösung entweder durch Absaugen oder durch gewaltsames Abladen in die Spüle oder eine Aufnahme. Achten Sie darauf, den Glasteil der Platte nicht zu berühren. Es ist akzeptabel, wenn einige Tropfen Concanavalin Eine Lösung in den Brunnen bleiben.
- Waschen Sie die Mikroskopplattenbrunnen, indem Sie 400 l steriles Wasser hinzufügen. Entfernen Sie das Wasser wie mit dem Concanavalin A im vorherigen Schritt getan. Lassen Sie die Platte nicht trocken sitzen.
- Fügen Sie sofort 185 l experimentelle Wachstumsmedien in die Platte ein. 15 l richtig verdünnte Zellen werden dieser Platte zugesetzt.
- Hefezellverdünnung
ANMERKUNG: Die folgenden Schritte beschreiben eine Verdünnung der Hefe aus einer gesättigten Kultur (ca. 108 Zellen/ml) 400-fach, um eine Konzentration von 250.000 Zellen/ml zu erreichen, von denen 15 l in der Glasbodenplatte in 400 l verdünnt werden, was eine endgültige Anzahl von etwa 4000 Zellen pro Brunnen in einer 96-Well-Platte ergibt. Bei Verwendung einer 384-Well-Platte sollte die endgültige Anzahl der Zellen pro Bohrgut etwa 700 betragen und die Verdünnungen sollten entsprechend angepasst werden. Dieses Verhältnis sollte für Zellen angepasst werden, die in der Protokollphase gesammelt werden und in reicheren oder ärmeren Medien vor dem Wachstum oder aus verschiedenen Stämmen wachsen. Die enddichte Dichte der Zellen pro Bohrkörper sollte reduziert werden, wenn der Wachstumsratentest für Zeiträume von mehr als 10 h ausgeführt wird.- Richten Sie zwei 96-Well-Kulturplatten für serielle Verdünnungen ein: Etikettieren Sie als Platte 1 und 2, und fügen Sie jeder seriellen Verdünnungsplatte 90 L experimentelle Wachstumsmedien (d. h. die Medien, in denen die Hefe auf dem Mikroskop wächst) hinzu.
HINWEIS: Unabhängig davon, welche endgültige Verdünnung verwendet wird, werden mindestens zwei serielle Verdünnungen von Zellen empfohlen, in denen jeweils ein kleines Hefevolumen in ein größeres Volumen experimenteller Medien pipettiert wird und dann eine große Menge experimenteller Medien kräftig mit einer Pipette vermischt wird (wie in den Schritten 3.2.5 und 3.2.6 unten). - Holen Sie die Platte der Zellen aus dem Vorwachstum und zentrifugieren Sie die Platte für 2 min bei 411 x g.
HINWEIS: Es ist sehr wichtig, verschiedene Brunnen in der Platte nicht zu verunreinigen. Der Zweck dieses Zentrifugationsschritts vor dem Entfernen der Folienabdeckung von Platten besteht darin, sicherzustellen, dass hefegefüllte Tröpfchen aus einem Brunnen nicht von der Folie abfliegen und in anderen Brunnen landen. Achten Sie darauf, die Platten niemals zu kippen oder zu erregen, um zu vermeiden, dass Hefe nach der Zentrifugation mit der Folienabdeckung in Berührung kommt. - Die Folie vorsichtig zurückziehen und Zellen durch kräftiges Pipetieren von Zellen mit einer Pipetten, die auf etwa die Hälfte des Gesamtvolumens in der Platte eingestellt ist, zurückblättern, während die Pipette um den Brunnen bewegt wird, um sie zu mischen. Überprüfen Sie, ob alle Zellen von der Unterseite der Brunnen resuspendiert wurden.
- Wenn mehrere Stämme in einzelnen Brunnen verwendet werden, sollten Stämme zu diesem Zeitpunkt in dem für das Experiment erforderlichen Verhältnis gemischt werden. Wenn ein Referenzstamm verwendet wird, um Wachstumsratenmessungen zu generieren, sollte das Verhältnis der Referenz zu Teststamm 1:1 betragen.
- Pipetten Sie 10 l Hefe aus Wachstumsmedien in Verdünnungsplatte 1. Fügen Sie jedem Bohrwert 100 L experimentelle Wachstumsmedien zu einem Endvolumen von 200 l pro Bohrgut hinzu. Pipetten Sie kräftig auf und ab.
- Pipetten Sie 10 l Hefe von Platte 1 in Platte 2. Fügen Sie jedem Brunnen 100 l experimentelle Wachstumsmedien hinzu und pfeifen Sie kräftig nach oben und unten, um sie zu mischen.
HINWEIS: Diese Verdünnungsschritte sind entscheidend, um Hefecluster zu trennen, die am Ende der Vorwachstumsphase zusammengeklebt sind, und sicherzustellen, dass ungefähr die gleiche Anzahl von Hefezellen in jedem Brunnen landet. Eine konsistente Anzahl von Hefen in jedem Brunnen hilft, experimentelles Rauschen und Verzerrungen bei Wachstumsratenmessungen zu entfernen (siehe repräsentative Ergebnisse).
- Richten Sie zwei 96-Well-Kulturplatten für serielle Verdünnungen ein: Etikettieren Sie als Platte 1 und 2, und fügen Sie jeder seriellen Verdünnungsplatte 90 L experimentelle Wachstumsmedien (d. h. die Medien, in denen die Hefe auf dem Mikroskop wächst) hinzu.
- Sonorisierung
HINWEIS: Beschallung ist optionalund muss nur für Hefestämme durchgeführt werden, die eine hohe Neigung haben, sich aneinander zu halten (z. B. einige wilde Stämme). Bei Laborstämmen ist eine Beschallung in der Regel nicht erforderlich und kann durch fortfahren sie mit Schritt 3.4 übersprungen werden.- Sanitisieren Sie einen 96-poligen Beschallungskopf mit 70% Ethanol, indem Sie ihn in eine 96-Well-Platte legen, die mit 70% Ethanol gefüllt ist und mit einem fussel- und statisch-freien Wisch trocknen.
- Legen Sie ein Beschallungsprogramm fest, das stark genug ist, um flockige Hefezellen zu trennen, aber keine Zellen abtötet oder erhöhte Stressreaktionen verursacht. Einige Tests sind möglicherweise erforderlich, um das beste Beschallungsprogramm für ein bestimmtes Experiment zu identifizieren. Das in diesem Experiment verwendete Beschallungsprogramm ist: Amplitude = 10, Prozesszeit = 10 s, Puls-on = 1 s, Puls-off = 1 s. Dieses genaue Programm ist wahrscheinlich nicht für alle Beschallungsgeräte anwendbar, so dass Tests vor dem Experimenttag vorgeschlagen werden.
- Mischen Sie die Hefe in der seriellen Verdünnungsplatte 2 noch einmal, indem Sie fünfmal kräftig nach oben und unten pfeifen.
- Legen Sie verdünnungsplatte 2 auf die Plattform und befestige Sie sie mit den Beschallungsstiften in der Zellsuspension, berühren Sie jedoch nicht den Boden der Platte. Führen Sie das Beschallungsprogramm mit entsprechendem Gehörschutz aus.
- Nach dem Programmläuft, reinigen Sie den Beschallungskopf mit 70% Ethanol und dann mit Wasser, und gehen Sie dann sofort zur Mikroskopplattenvorbereitung, damit die Zellen nicht wieder flumeln.
- Platte für Mikroskop vorbereiten:
- Pipetten Sie 15 l Hefe von der seriellen Verdünnungsplatte 2 in die Mikroskopplatte bis zu einem Volumen von 200 l. Fügen Sie jedem Bohrgut 200 l experimentelle Wachstumsmedien zu einem Endvolumen von 400 l pro Bohrgut hinzu und geben Sie die Spipet nach oben und unten kräftig an, um sie zu mischen.
- Bedecken Sie die Platte mit einer atmungsaktiven Membran. Es ist wichtig, die Platte mit dieser Membran gut zu versiegeln, zum Beispiel mit einer Gummiwalze.
- Um die Hefezellen an das Concanavalin A auf der Glasoberfläche zu kleben, zentrieren Sie die Platte mit einem fussel- und statisch-freien Abwischen darunter für 2 min bei 411 x g.
- Wischen Sie am Mikroskop die Ober- und Unterseite der Platte mit einem fussel- und statisch-freien Wisch ab und blasen Sie Druckluft auf die Platte, um Schmutz zu beseitigen.
- Legen Sie die Platte auf das Mikroskop, um sicherzustellen, dass sie eben ist und dass sich der A1-Brunnen in der linken oberen Ecke befindet.
4. Zeitraffermikroskopische Wachstumsrate Messungen
HINWEIS: Während der Zeitraffermikroskopie werden die folgenden Funktionen computergesteuert: x-, y- und z-Position, Rollläden und Fluoreszenzfilter. Ein hardwarebasiertes Autofokussystem ist optimal, um eine Drift der Brennebene während der Zeitraffer-Bildgebung zu verhindern. Alternativ kann eine softwarebasierte Autofokus-Schleife verwendet werden. Um die Luftfeuchtigkeit in der Mikroskopkammer aufrechtzuerhalten, wird empfohlen, während der gesamten Dauer des Experiments einen Becher mit gereinigtem Wasser in der Kammer zu halten.
- Erstellen Sie eine Liste der Positionen (x,y) zum Abbild, sodass jeder Mikroskop-Plattenbrunnen vollständig abgebildet ist. Vermeiden Sie überlappende Bilder, sodass keine Zelle mehrmals analysiert wird.
- Bild im hellen Feld mit diaskopischer Beleuchtung (DIA) bei einer Vergrößerung von 15x. Stellen Sie die Belichtungsposition auf 5 ms fest.
- Zoomen Sie das Bild digital ein, sodass Zellen deutlich sichtbar sind. Verwenden Sie die Fokussierknöpfe, um den idealen Fokus für das Experiment in den vier Brunnen an der Ecke der Platte und in einem Brunnen in der Mitte der Platte zu identifizieren. Konzentrieren Sie sich so, dass ein maximaler Kontrast der Zellen zu erhalten ist.
- Legen Sie die z-Position (oder Autofokusposition) für das Experiment als Durchschnitt der für jeden dieser Brunnen identifizierten Z/Autofokus-Positionen fest. Wenn die Mikroskopplatte gut gemacht ist und der Glasboden keine Defekte aufweist, sollten die idealen Fokuspositionen für jeden Brunnen ähnlich sein.
HINWEIS: Bei der Analyse von Bildern mit der Bildanalyse-Pipeline11,12ist es hilfreich, dass die Zellen etwas außerhalb des Fokus auf das Mikroskop liegen, so dass sich ein dunkler Rand außerhalb der Zelle und ein helles Inneres befinden, was zu einer genauen Kolonieerkennung und Größenschätzungen beihilft. - Wenn Sie fluoreszierende Stämme verwenden, identifizieren Sie die Kanäle und belichtungswerten Belichtungen, mit denen sie abbilden, und stellen Sie sicher, dass keine Pixel überbelichtet sind. Wenn Sie die Belichtungszeit für fluoreszierende Kanäle einstellen, deaktivieren Sie den Modus "Live Capture" am Mikroskop, um zu vermeiden, dass Zellen über einen längeren Zeitraum der Fluoreszenzerregung ausgesetzt werden, da dies sowohl die Zellen photobleichen als auch Stress verursachen kann.
- Richten Sie die Zeitsequenzerfassung ein, um Bilder im gewünschten Zeitintervall für die gewünschte Zeitzunahme zu erfassen.
- Führen Sie das Experiment aus.
Ergebnisse
Die Neuheit dieses Protokolls ist, dass die Wachstumsrate für einzelne Zellen innerhalb einer Population berechnet werden kann, indem ihr Wachstum in Mikrokolonien durch Zeitraffer-Bildgebung verfolgt wird (Abbildung 2A). Da Mikrokolonien aufgrund des Vorhandenseins von Concanavalin A viele Stunden planarer wachsen, können ihre Gebiete während des gesamten Experiments verfolgt werden, und eine lineare Anpassung an die Veränderung des natürlichen Protokolls des Gebiets im Laufe der Zeit ...
Diskussion
Das hier beschriebene Protokoll ist ein vielseitiger Test, der es ermöglicht, Zellwachstum und Genexpression gleichzeitig auf der Ebene einzelner Mikrokolonien zu überwachen. Die Kombination dieser beiden Modalitäten ergibt einzigartige biologische Erkenntnisse. Zum Beispiel haben frühere Arbeiten diesen Test verwendet, um eine negative Korrelation zwischen der Expression des TSL1-Gens und der Mikrokolonie-Wachstumsrate in isogenen Wildtypzellen zu zeigen, indem beide gleichzeitig7
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Naomi Ziv, Sasha Levy und Shuang Li für ihre Beiträge zur Entwicklung dieses Protokolls, David Gresham für gemeinsame Ausrüstung und Marissa Knoll für die Hilfe bei der Videoproduktion. Diese Arbeit wurde durch den Zuschuss der National Institutes of Health R35GM118170 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are recommended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
Referenzen
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization--a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten