Method Article
NMR-basiertes Fragment-Screening mit minimaler Probe, aber maximalem Automatisierungsmodus
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Das fragmentbasierte Screening mittels NMR ist eine robuste Methode zur schnellen Identifizierung von niedermolekularen Bindemitteln an Biomakromoleküle (DNA, RNA oder Proteine). Es werden Protokolle vorgestellt, die die automatisierungsbasierte Probenvorbereitung, NMR-Experimente und Aufnahmebedingungen sowie Analyse-Workflows beschreiben. Die Technik ermöglicht die optimale Nutzung von 1H und 19F NMR-aktiven Kernen für den Nachweis.
Zusammenfassung
Das fragmentbasierte Screening (FBS) ist ein gut validiertes und akzeptiertes Konzept innerhalb des Wirkstoffforschungsprozesses sowohl in der Wissenschaft als auch in der Industrie. Der größte Vorteil des NMR-basierten Fragmentscreenings ist seine Fähigkeit, nicht nur Bindemittel über 7-8 Größenordnungen der Affinität zu detektieren, sondern auch die Reinheit und chemische Qualität der Fragmente zu überwachen und so qualitativ hochwertige Treffer und minimale falsch positive oder falsch negative Ergebnisse zu erzeugen. Eine Voraussetzung innerhalb des FBS ist die Durchführung einer anfänglichen und periodischen Qualitätskontrolle der Fragmentbibliothek, die Bestimmung der Löslichkeit und chemischen Integrität der Fragmente in relevanten Puffern und die Einrichtung mehrerer Bibliotheken, um verschiedene Gerüste abzudecken, um verschiedene Makromolekül-Zielklassen (Proteine/RNA/DNA) aufzunehmen. Darüber hinaus ist eine umfassende NMR-basierte Optimierung des Screening-Protokolls in Bezug auf Probenmengen, Geschwindigkeit der Erfassung und Analyse auf der Ebene des biologischen Konstrukts/Fragmentraums, im Zustandsraum (Puffer, Additive, Ionen, pH und Temperatur) und im Ligandenraum (Ligandenanaloga, Ligandenkonzentration) erforderlich. Zumindest in der akademischen Welt wurden diese Screening-Bemühungen bisher nur in sehr begrenztem Umfang manuell durchgeführt, was zu einer begrenzten Verfügbarkeit der Screening-Infrastruktur nicht nur im Arzneimittelentwicklungsprozess, sondern auch im Zusammenhang mit der Entwicklung chemischer Sonden führte. Um den Anforderungen wirtschaftlich gerecht zu werden, werden fortschrittliche Workflows vorgestellt. Sie nutzen die Vorteile der neuesten hochmodernen Hardware, mit der die flüssige Probenentnahme auf temperaturkontrollierte Weise automatisiert in die NMR-Röhrchen gefüllt werden kann. 1H/19F NMR-Liganden-basierte Spektren werden dann bei einer bestimmten Temperatur aufgenommen. Der Probenwechsler mit hohem Durchsatz (HT-Probenwechsler) kann mehr als 500 Proben in temperaturgesteuerten Blöcken verarbeiten. Zusammen mit fortschrittlichen Softwaretools beschleunigt dies die Datenerfassung und -analyse. Des Weiteren wird die Anwendung von Screening-Routinen auf Protein- und RNA-Proben beschrieben, um die etablierten Protokolle für eine breite Anwenderbasis in der biomakromolekularen Forschung bekannt zu machen.
Einleitung
Das fragmentbasierte Screening ist heute eine häufig verwendete Methode zur Identifizierung von relativ einfachen und niedermolekularen Molekülen (MW <250 Da), die eine schwache Bindung an makromolekulare Ziele wie Proteine, DNA und RNA aufweisen. Initiale Treffer von Primär-Screens dienen als Grundlage, um ein Sekundär-Screening von kommerziell erhältlichen größeren Analoga der Hits durchzuführen und dann chemiebasierte Fragmentwachstums- oder Verknüpfungsstrategien zu nutzen. Für eine erfolgreiche fragmentbasierte Wirkstoffentdeckungsplattform (FBDD) ist im Allgemeinen eine robuste biophysikalische Methode zum Nachweis und zur Charakterisierung schwacher Treffer, eine Fragmentbibliothek, ein biomolekulares Ziel und eine Strategie für die Follow-up-Chemie erforderlich. Vier häufig angewandte biophysikalische Methoden innerhalb der Wirkstoffforschungskampagnen sind thermische Verschiebungsassays, Oberflächenplasmonenresonanz (SPR), Kristallographie und Kernspinresonanzspektroskopie (NMR).
Die NMR-Spektroskopie hat in den verschiedenen Stadien der FBDD unterschiedliche Rollen gespielt. Neben der Sicherstellung der chemischen Reinheit und Löslichkeit der Fragmente in einer Fragmentbibliothek, die in einem optimierten Puffersystem gelöst sind, können Liganden-beobachtete NMR-Experimente die Bindung von Fragmenten an ein Target mit geringer Affinität nachweisen, und die Target-beobachteten NMR-Experimente können das Bindungsepitop des Fragments abgrenzen, wodurch detaillierte Struktur-Aktivitäts-Beziehungsstudien möglich sind. Innerhalb der Epitopkartierung können NMR-basierte chemische Verschiebungsänderungen nicht nur die orthosterischen Bindungsstellen identifizieren, sondern auch allosterische Stellen, die kryptisch und nur in sogenannten angeregten Konformationszuständen des biomolekularen Ziels zugänglich sein könnten. Wenn das biomolekulare Target bereits an einen endogenen Liganden bindet, können die identifizierten Fragmenttreffer durch NMR-basierte Konkurrenzexperimente leicht als allosterisch oder orthosterisch klassifiziert werden. Die Bestimmung der Dissoziationskonstante (KD) der Ligand-Target-Wechselwirkung ist ein wichtiger Aspekt im FBDD-Prozess. NMR-basierte chemische Shift-Titrationen, entweder Liganden- oder Target-Titrationen, können problemlos durchgeführt werden, um den KD zu bestimmen. Ein großer Vorteil der NMR besteht darin, dass die Interaktionsstudien in Lösung und in der Nähe physiologischer Bedingungen durchgeführt werden. Somit können alle Konformationszustände für die Analyse der Ligand/Fragment-Wechselwirkung mit seinem Target untersucht werden. Darüber hinaus sind NMR-basierte Ansätze nicht nur auf das Screening gut gefalteter löslicher Proteine beschränkt, sondern werden auch auf größere Zielräume wie DNA, RNA, membrangebundene und intrinsisch ungeordneteProteine angewendet 1.
Fragmentbibliotheken sind ein unverzichtbarer Bestandteil des FBDD-Prozesses. Im Allgemeinen fungieren Fragmente als erste Vorläufer, die schließlich Teil (Substruktur) des neuen Inhibitors werden, der für ein biologisches Ziel entwickelt wurde. Es wurde berichtet, dass mehrere Medikamente (Venetoclax2, Vemurafenib3, Erdafitinib4, Pexidartnib5) als Fragmente begonnen haben und nun erfolgreich in den Kliniken eingesetzt werden. Typischerweise handelt es sich bei Fragmenten um organische Moleküle mit niedrigem Molekulargewicht (<250 Da) und einer hohen wässrigen Löslichkeit und Stabilität. Eine sorgfältig ausgearbeitete Fragmentbibliothek, die in der Regel einige hundert Fragmente enthält, kann bereits eine effiziente Erkundung des chemischen Raums versprechen. Die allgemeine Zusammensetzung von Fragmentbibliotheken hat sich im Laufe der Zeit weiterentwickelt und wurde meist durch das Zerlegen bekannter Medikamente in kleinere Fragmente abgeleitet oder rechnerisch entworfen. Diese verschiedenen Fragmentbibliotheken enthalten hauptsächlich flache aromatische oder Heteroatome und halten sich an die Lipinski-Regel von 5 6 oder an die aktuelle kommerzielle Trendregel von 3 7, vermeiden jedoch reaktive Gruppen. Einige Fragmentbibliotheken wurden auch von hochlöslichen Metaboliten, Naturstoffen und/oder deren Derivaten abgeleitet oder zusammengesetzt8. Eine allgemeine Herausforderung, die von den meisten Fragmentbibliotheken gestellt wird, ist die Einfachheit der nachgelagerten Chemie.
Das Zentrum für Biomolekulare Magnetresonanz (BMRZ) der Goethe-Universität Frankfurt ist Partner von iNEXT-Discovery (Infrastructure for NMR, EM and X-rays for Translational research-Discovery), einem Konsortium für strukturelle Forschungsinfrastrukturen für alle europäischen Forschenden aus allen Bereichen der biochemischen und biomedizinischen Forschung. Im Rahmen der vorherigen Initiative von iNEXT, die 2019 endete, wurde eine Fragmentbibliothek mit 768 Fragmenten erstellt, mit dem Ziel, "minimale Fragmente und maximale Vielfalt" zu erreichen, die einen großen chemischen Raum abdeckt. Darüber hinaus wurde die iNEXT-Fragmentbibliothek im Gegensatz zu allen anderen Fragmentbibliotheken auch auf der Grundlage des Konzepts der "ausgeglichenen Fragmente" entwickelt, mit dem Ziel, die nachgelagerte Synthese komplexer Liganden mit hoher Affinität zu erleichtern, und von nun an als Inhouse-Bibliothek (Diamond, Structural Genomic Consortium und iNEXT) bezeichnet.
Die Etablierung von FBDD mittels NMR erfordert Arbeitskräfte, Wissen und Instrumente. Am BMRZ wurden optimierte Arbeitsabläufe entwickelt, um die technische Unterstützung beim Fragment-Screening mittels NMR zu unterstützen. Dazu gehören Qualitätskontrolle und Löslichkeitsbewertung der Fragmentbibliothek 9, Pufferoptimierung für die ausgewählten Targets, 1H oder 19F- beobachtetes 1D-Liganden-basiertes Screening, Konkurrenzexperimente zur Unterscheidung zwischen orthosterischer und allosterischer Bindung, 2D-basierte Target-beobachtete NMR-Experimente zur Epitopkartierung und zur Charakterisierung der Wechselwirkung mit sekundären Derivaten der ursprünglichen Fragmenttreffer. Das BMRZ hat automatisierte Routinen für die Analyse von Kleinmolekül-Protein-Wechselwirkungen etabliert, wie auch bereits in der Literatur 10,11 diskutiert, und verfügt über die notwendige automatisierte Infrastruktur für das NMR-basierte Fragmentscreening. Es hat Sättigungstransferdifferenz-NMR (STD-NMR), Wasser-Liganden-Spektroskopie (waterLOGSY) und Carr-Purcell-Meiboom-Gill-basierte (CPMG-basierte) Relaxationsexperimente implementiert, um Fragmente innerhalb eines breiten Spektrums von Affinitätsregimen zu identifizieren, sowie modernste automatisierte NMR-Instrumente und Software für die Wirkstoffforschung. Während das NMR-basierte Fragment-Screening für Proteine gut etabliert ist, wird dieser Ansatz weniger häufig verwendet, um neue Liganden zu finden, die mit RNA und DNA interagieren. Das BMRZ hat den Proof-of-Concept für neue Protokolle etabliert, die die Identifizierung von niedermolekularen RNA/DNA-Wechselwirkungen ermöglichen. In den folgenden Abschnitten dieses Beitrags wird über die Anwendung von Screening-Routinen auf Protein- und RNA-Proben berichtet, um die etablierten Protokolle für eine breite Anwenderbasis in der biomakromolekularen Forschung bekannt zu machen.
Protokoll
1. Fragment-Bibliothek
- Eigene Fragmentbibliothek
HINWEIS: Im Rahmen einer der gemeinsamen Forschungsaktivitäten des iNEXT wurde eine robuste und nachgelagerte chemiefreundliche Fragmentbibliothek der ersten Generation entwickelt12 und anschließend eine zweite Generation der Bibliothek in Zusammenarbeit mit Enamine zusammengestellt und ist als DSI (Diamond-SGC-iNEXT)-poised fragment library bekannt (im Folgenden als "Inhouse-Bibliothek" bezeichnet). Diese Bibliothek kann dem BMRZ zu Sichtzwecken zur Verfügung gestellt werden.- Bewerten Sie die Fragmentbibliothek auf ihre Integrität und Löslichkeit unter Verwendung eines zuvor berichteten NMR-basierten Protokolls9.
HINWEIS: Die hauseigene Bibliothek besteht aus 768 Fragmenten mit einer sehr hohen chemischen Diversität (>200 Singletons). Die Durchführung des Screenings in Fragmentgemischen kann die Screeningkampagne erheblich beschleunigen. Die Anzahl der Fragmente in einem Mix ist jedoch aufgrund der Signalüberlappung im 1-H-NMR-Spektrum begrenzt. Die höhere chemische Vielfalt, die die hauseigene Bibliothek bietet, ermöglicht die Herstellung von Mischungen mit 12 verschiedenen Fragmenten ohne signifikante chemische Verschiebungsüberlappung in den 1H beobachteten NMR-Spektren. - 103 Fragmente der 768 Fragmente besitzen ein Fluoratom. Für das 19-F-Screening werden alle 103 Fragmente, die eine Fluorgruppe besitzen, in 5 Mischungen aufgeteilt, basierend auf einer chemischen Überlappung von mindestens 19F. Um die Signalüberlappung bei der 19-F-Abschirmung zu minimieren, verwenden Sie die chemischen Verschiebungsinformationen aus Einzelverbindungsmessungen, um Mischungen mit maximaler Anzahl von Fragmenten und minimaler Signalüberlappung zu entwerfen. Jede Mischung besteht aus 20-21 Fragmenten mit deutlichen chemischen Verschiebungen von 19F, die eine eindeutige Zuordnung der Fragmente ermöglichen.
- Bewerten Sie die Fragmentbibliothek auf ihre Integrität und Löslichkeit unter Verwendung eines zuvor berichteten NMR-basierten Protokolls9.
- Benutzerdefinierte/bereitgestellte Fragmentbibliothek
- Führen Sie Screening-Kampagnen mit der benutzerdefinierten oder bereitgestellten Fragmentbibliothek durch. Die folgenden Schritte müssen jedoch der Screening-Kampagne vorausgehen.
- Wenn nicht vorher vom Anwender vorgegeben, führen Sie eine NMR-basierte Qualitätskontrolle der Fragmente durch (am BMRZ werden dafür fortschrittliche Software-Tools eingesetzt; 9, Kapitel 6.1.1).
- Überprüfen Sie vor der Verwendung die Löslichkeit der Fragmente im Puffer der Wahl für das biomolekulare Ziel, die Strukturintegrität und die Konzentration der Fragmente.
- Entwerfen Sie die Mischung, um sowohl die Signalüberlappung in NMR-Spektren als auch die Messzeit zu verringern.
- Entwerfen Sie Mischungen gemäß Schritt 4.2.
- Durchsuchen Sie einzelne Fragmente oder eine Teilmenge von Mischungen anstelle der gesamten Bibliothek.
2. Probenvorbereitung
HINWEIS: Beim Hochdurchsatz-Screening durch NMR wird ein Pipettierroboter für die Probenvorbereitung verwendet. NMR-Spektren, aber auch Stabilitäten über mehrere Tage der Signalerfassung von Proteinen, RNAs und DNA sind extrem empfindlich gegenüber Temperaturschwankungen, so dass temperaturgesteuerte automatisierte Systeme die Stabilität der zu pipettierenden Proben erheblich erleichtern. Dazu wird ein zusätzliches Zusatzgerät, das zwischen 4 und 40 °C arbeitet, an den Pipettierroboter gekoppelt, um die NMR-Proben in einer temperierten Umgebung flüssig zu handhaben.
- Herstellung von Ligandenmischungen
- Bereiten Sie Screening-Proben für NMR-Messungen mit einem Probenvorbereitungsroboter vor. Die flexible Konfiguration des Roboters ermöglicht eine Vielzahl von Anwendungen (z. B. Rückgewinnung der Proben aus NMR-Röhrchen zurück in Lagerbehälter oder allgemeine Liquid-Handling-Aufgaben). Es können NMR-Röhrchen mit unterschiedlichen Durchmessern (1,7, 2,0, 2,5, 3,0 und 5,0 mm) verwendet werden. Das Probenrobotersystem liest zusammen mit der fortschrittlichen Steuerungssoftware den Barcode, der jedem Behältertyp zugewiesen ist, und führt das Flüssigkeitsabfüllprotokoll optimal aus.
- Verwenden Sie für die Herstellung der hauseigenen Bibliotheksligandenmischungen Barcode-Fläschchen. Die mit Barcode versehenen Fläschchen garantieren höchste Zuverlässigkeit und optimale Rückverfolgbarkeit der Proben.
- Verteilen Sie 768 Verbindungen auf 8 Platten im 96-Well-Format. Die Bestandskonzentration jedes einzelnen Fragments beträgt 50 mM in d6-DMSO/D2O (9:1). Bereiten Sie insgesamt 64 Mischungen mit jeweils 12 Fragmenten vor. Die Endkonzentration jedes Fragments in einer Mischung beträgt 4,2 mM.
HINWEIS: Der Pipettierroboter kann eine Vielzahl von Behältertypen mit unterschiedlichen Geometrien aufnehmen (Kryo- oder Autosampler-Fläschchen, 96-Well-Platten rund oder quadratisch tief, Standardfläschchen mit Barcode, Mikrozentrifugenröhrchen) und unterstützt die effiziente Durchführung des Flüssigkeitstransfers in eine Vielzahl von NMR-Röhrchen und -Racks.

Abbildung 1: (A) Hochdurchsatz-NMR-Probenvorbereitungs- und NMR-Röhrchenfüllroboter am BMRZ installiert. (B) Hochdurchsatz-Probenwechsler mit einzelnen temperaturgeregelten Racks, installiert auf einem 600-MHz-Spektrometer in der BMRZ-Anlage. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Screening-Probenvorbereitung leer (Referenzligandenspektrum) und mit Target (Ligand in Gegenwart von Target)
- Verwenden Sie für die Vorbereitung der NMR-Screening-Proben in Gegenwart des Zielbiomoleküls (Protein/RNA/DNA) und des Ligandengemisches 3-mm-NMR-HT-Probenwechslöhrchen, die aus dem Bruker-NMR-Portfolio der Standard-NMR-Röhrchen ausgewählt wurden.
- Das biomolekulare Target (z.B. 1H Screening: 10 μM RNA oder Protein) wird in einem definierten Screening-Puffer in das 3 mm NMR-Röhrchen (Endvolumen 200 μL) manuell oder mit dem Pipettierroboter überführt.
- Übertragen Sie 10 μl (z. B. 1H Screening) des Ligandengemisches im nächsten Schritt mit dem Robotersystem in die mit Barcode versehenen 3-mm-NMR-Röhrchen, die das Ziel-Biomolekül enthalten, und mischen Sie es unter Verwendung des eingebauten Protokolls der Steuerungssoftware.
HINWEIS: Die Barcode-Nummer des NMR-Röhrchens wird bequem und automatisch in den erfassten NMR-Datensatz übernommen, so dass ein ID-orientierter Workflow ohne Verwechslung gewährleistet ist. Das Zubehör zur Temperaturregelung des Pipettierroboters ermöglicht es, die vorbereiteten Proben in den NMR-Röhrchen bei konstanter Temperatur zu halten.
- Eigens definierte Bedingungen und Parameter
- Schaffen Sie optimale Pufferbedingungen für das Screening von RNA und Protein anhand der hauseigenen Fragmentbibliothek. Für die RNA am BMRZ werden folgende Proben konditioniert: 25 mM KPi, 50 mM KCl, pH 6,2. Mg2+ ist optional.
- Proteine sind extrem empfindlich gegenüber Lösungsbedingungen; Verwenden Sie Puffer, die für das Ziel Ihrer Wahl optimal sind. Erfassen Sie für jeden dieser Puffer zusätzliche Referenzspektren der Liganden, die als Leerzeichen für die Analyse dienen.
- Benutzerdefinierte Bedingungen
HINWEIS: In Fällen, in denen die intern festgelegten Bedingungen nicht geeignet sind, um die zu überprüfenden Ziele vor einem potenziellen Benutzer zu überprüfen, sollten die folgenden Schritte durchgeführt werden.- Führen Sie 1H-NMR des Puffers allein durch, um eine minimale Interferenz der Komponenten des Puffers bei der Durchführung und Analyse der beobachteten Screening-Experimente mit Liganden zu gewährleisten. Störkomponenten könnten in geeigneter Weise durch deuterierte Äquivalente ersetzt werden.
- Einschränkungen in der Musterproduktion (Zielmengen)/-konditionen und -verfügbarkeit
HINWEIS: Die Isolierung oder rekombinante Herstellung bestimmter Biomakromoleküle kann sich in bestimmten Fällen als schwierig erweisen und zu einer begrenzten Verfügbarkeit des Zielmoleküls führen, um eine erfolgreiche Wirkstoff-Screening-Kampagne durchzuführen. In Fällen begrenzter oder unbegrenzter Verfügbarkeit der Targets können die folgenden Alternativen für die Durchführung eines erfolgreichen NMR-basierten Fragmentscreenings verwendet werden.- Wenn es eingeschränkt ist, verwenden Sie ein 19F-NMR-basiertes Screening. Typische fluorierte Liganden haben ein einzelnes 19F-Signal; Verwenden Sie daher Cocktails mit 25-30 Fragmenten ohne Signalüberlappung. Es müssen weniger Signale analysiert werden, es gibt keine Signalstörungen durch Pufferkomponenten und weniger Signale, auf die man sich bei der Treffererkennung verlassen kann.
- Wenn es unbegrenzt ist, verwenden Sie größere Bildschirme wie 1H-NMR. Die größere Fragmentbibliothek kann durchsucht werden. In der Regel bestehen Fragmente aus mehr als einem Proton, was bedeutet, dass man sich bei der Analyse auf mehr Signale verlassen kann.
3. Bedingungen für die NMR-Erfassung
- Interne, allgemein definierte Bedingungen
- Spektrometer mit HT-Probenwechsler (Automation)
- Verwenden Sie für das Hochdurchsatz-Screening 96-Well-Platten, die nur mit einem HT-Probenwechsler gemessen werden können. Der HT-Probenwechsler bietet zudem die Möglichkeit, jedes Rack einzeln zu temperieren.
- Verwenden Sie für ein optimales Signal-Rausch-Verhältnis ein Spektrometer mit einer kryogenen Sonde, die entweder mit Helium oder Stickstoff gekühlt ist. Für die Automatisierung ist ein automatisiertes Tuning- und Matching-Modul (ATM) erforderlich.
- Parametersätze & Impulsfolgen
HINWEIS: Viele NMR-Experimente können Bindungsereignisse charakterisieren. Die Trefferidentifikation variiert je nach Versuchsaufbau. Die folgenden Experimente werden routinemäßig in BMRZ-Screening-Kampagnen eingesetzt. Dennoch können Änderungen für benutzerdefinierte Screening-Kampagnen und nach Benutzervorgaben vorgenommen werden.- Wenn Sie die TopSpin-Software verwenden, schließen Sie den Parametersatz für ligandenbasierte Experimente ein: SCREEN_STD, SCREEN_T1R, SCREEN_T2, SCREEN_WLOGSY. Der Parametersatz enthält alle notwendigen Parameter und die Pulsfolgen: STD: stddiffesgp.3; T1ρ: t1rho_esgp2d; T2: cpmg_esgp2d; und waterLOGSY: ephogsygpno.2.
- Verwenden Sie für alle aufgeführten Experimente die Anregungsformung13 als Wasserunterdrückung. Verwenden Sie als Referenz 1D-Anregungsmodellierung (zgesgp). Die Anzahl der Scans hängt von der Empfindlichkeit des Systems (Magnetfeldstärke und Sondenkopf), der Probenkonzentration und der Wahl des Experiments ab. Eine Empfehlung ist: 1D mit NS=64, T1ρ & T2 mit NS=128, STD mit NS=256 und waterLOGSY mit NS= 384 oder 512.
- Verwenden Sie für das 19-F-Screening sowohl 1D- als auchT-2-Experimente: 1D: F19CPD (pp=zgig) für 19 F{1 H}-Sondenkopf und F19(pp=zg) für 19F/1H-Sondenkopf; SCREEN_19F_T2 (pp = cpmgigsp).
- Verwenden Sie eine spektrale Breite von 220 ppm und eine Anregungsfrequenz von -140 ppm. Die Experimentierzeit beträgt zwischen 1 und 5 Stunden (Gewährleistung der Langzeitstabilität des Biomakromoleküls) je nach Hardware und Probenkonzentration. Für T2 sollte die CPMG-Zeit zwischen 0 ms und 200 ms liegen.
- Verarbeitung
- Zeichnen Sie die STD-, T1ρ - und T2-Experimente als Pseudo-2D auf. Um die beiden einzelnen 1D-Spektren zu bearbeiten, verwendet IconNMR das au-Programm proc_std wahlweise mit oder ohne die Option relax. Die erste Option liefert die Referenz 1D und die Differenz zweier Spektren. Die zweite Option liefert zwei getrennte Spektren mit kurzer und langer Relaxationszeit. Der waterLOGSY ist ein einzelner 1D, der mit einem negativen Signal für das Lösungsmittelsignal gephaset werden sollte.
- Spektrometer mit HT-Probenwechsler (Automation)
- Benutzerspezifische Bedingungen
- Passen Sie einen der zuvor genannten Parameter an benutzerdefinierte Bedingungen an. Wenn z. B. ein vom Benutzer bereitgestelltes Protein bei der allgemein verwendeten Temperatur nicht stabil ist, können Optimierungsexperimente durchgeführt werden, bei denen Temperatur, Konzentration, Pufferbedingungen usw. variiert werden.
4. Datenanalyse
- Fragmentbibliotheks-QC (d6-DMSO/spezifischer Puffer) und Quantifizierung
- CMC-q
HINWEIS: Die Qualitätskontrolle von Fragmentbibliotheken ist vor der Initiierung von Screening-Kampagnen unerlässlich. Darüber hinaus muss die Langzeitstabilität der Fragmentbibliothek für die Anwendung mehrerer Screening-Kampagnen sichergestellt werden, weshalb eine regelmäßige Evaluierung der Qualität der Bibliothek durchgeführt werden muss. Hierfür wird die integrierte Software CMC-q und CMC-a von TopSpin zur Qualitäts- und Quantitätsbewertung eingesetzt. CMC-q und CMC-a sind Softwaremodule innerhalb von Topspin, die eine reibungslose Erfassung, Analyse einschließlich Strukturverifikation unter Verwendung des 1-H-NMR-Spektrums aus kleinen organischen Molekülen 9 ermöglichen.- Für die Integrität sind Beurteilungsproben mit einer Fragmentkonzentration von 1 mM in d6-DMSO vorzubereiten. Bereiten Sie Proben automatisiert mit einem Pipettierroboter vor, indem Sie flüssige Probenentnahme in ein 3-mm-NMR-Röhrchen füllen.
- Zur Beurteilung der Löslichkeit wird eine Probe verwendet, die aus 1 mM-Verbindung in 50 mM Natriumphosphatpuffer bei pH 7,4, 150 mM Natriumchlorid, 90 % H2 O/ 10 %D2O und 1 mM 3-(Trimethylsilyl)propionsäure-2,2,3,3-d4-Natriumsäure (TMSP-Na) besteht.
- Erfassen Sie NMR-Spektren bei 298 K oder 293 K mit einem 600-MHz-NMR-Spektrometer, das mit einer kryogenen 5-mm-TCI-Dreifachresonanzsonde und einem HT-Probenwechsler ausgestattet ist, der 579 Proben gleichzeitig verarbeiten kann.
- Befolgen Sie zum Einrichten der CMC-q-Software die Anweisungen im Benutzerhandbuch, das die Erstellung eines IconNMR-Benutzers, die Aktivierung von FastLaneNMR und den Wechsel des HT-Probenwechslers implementiert.
- Kalibrieren Sie den 90°-Impuls und speichern Sie ihn in der TopSpin prosol-Tabelle.
- Platzieren Sie die 96-Proben-Well-Platte in einer der 5 Rack-Positionen im HT-Probenwechsler.
- Um eine SDF-Datei (Strukturdatendatei) zu laden, die die vorgeschlagene chemische Struktur, eine eindeutige Kennung und die Position jeder Probe in einer Charge im HT-Probenwechsler enthalten soll, gehen Sie im CMC-q-Setup-Fenster zu Durchsuchen und klicken Sie auf Öffnen , nachdem Sie eine Datei ausgewählt haben, die auf .sdf endet.
- Legen Sie in den Einstellungen für die CMC.q-Stapelautomatisierung den Verifizierungstyp fest, der das zu messende Experiment definiert, den IconNMR-Benutzer und das Lösungsmittel.
- Definieren Sie SDF-Dateien für den Pfad für die SDF-Datei, die Molekül-ID und die Probenposition.
- Starten Sie die Erfassung, indem Sie auf Start klicken. Klicken Sie erneut auf Akquisition starten . Das CMC-q Setup kann auch mit einem Klick auf Speichern gespeichert werden.
- Eine detaillierte Beschreibung der CMC-q-Einrichtungsschritte finden Sie in der Bedienungsanleitung von Bruker.
- CMC-a
- Verwenden Sie für CMC-a das Softwaremodul in Topspin, das die Analyse einschließlich der Strukturüberprüfung unter Verwendung des 1H-NMR-Spektrums ermöglicht, das aus kleinen organischen Molekülen9 gewonnen wird.
- CMC-q
- Mischungsdesign
HINWEIS: Ein geeignetes Mischungsdesign spielt eine wichtige Rolle für das Screening mit NMR als Plattform. Eine hohe Anzahl von Fragmenten pro Mischungen ermöglicht ein schnelleres Screening, erhöht jedoch das Risiko falsch positiver und negativer Ergebnisse. Eine niedrigere Zahl verringert dieses Risiko, erhöht aber die Zeit, die für die Durchführung des Screenings benötigt wird. Generell muss bei der Erstellung von Mischungen eine Signalüberlappung vermieden werden. Bei Verwendung der hauseigenen Bibliothek kann dies für das 1-Stunden-Screening vernachlässigt werden, da die Bibliothek speziell so konzipiert wurde, dass sie vielfältig ist und eine geringe Signalüberlappung aufweist, während eine hohe chemische Vielfalt erhalten bleibt. Das wiederum bedeutet, dass für die Erstellung der 64 Mischungen kein spezielles Designverfahren durchlaufen werden muss.- Da die 19-F-Abschirmung auf den Fragmenten der hauseigenen Bibliothek beruht, die Fluor enthalten, und die Bibliothek nicht erstellt wurde, um die Signalüberlappung für diese spezifischen Fragmente zu reduzieren, sollten Sie eine geeignete Mischung entwerfen.
- Messen Sie Einzelverbindungsspektren für alle Fragmente, die 19F enthalten.
- Beachten Sie die chemischen Verschiebungsinformationen jedes Signals.
- Wählen Sie nach diesen Informationen 20-21 Fragmente pro Mischung. Dies wiederum ergibt 5 Mischungen mit jeweils 20-21 Fragmenten ohne Signalüberlappung und ermöglicht eine halbautomatische Analyse der Daten.
- Durchführung der Hit-Identifizierung innerhalb eines Liganden beobachtete Biomakromolekül-Ligand-Wechselwirkung
HINWEIS: Es gibt unterschiedliche Definitionen eines Treffers zwischen dem 19-F- und dem 1-H-Screening-Verfahren. Die folgenden Trefferidentifikationen wurden von uns eingerichtet und folgen bestimmten Regeln. Das Thema Trefferermittlung ist sehr subjektiv und kann von Nutzer zu Nutzer unterschiedlich sein. Nichtsdestotrotz ist es von größter Bedeutung, dass sich die Regeln für die Identifizierung von Treffern nicht ändern, sobald sie vereinbart wurden, um die Validierung und Glaubwürdigkeit zu erhalten.- 1H-Bildschirm
- Um Treffer sicher zu bestimmen, werden 1D-1H-Spektren, waterLOGSY- undT2-Relaxationsexperimente sowohl in Anwesenheit als auch in Abwesenheit von Targets zur Identifizierung von Bindemitteln durchgeführt. Alle drei Experimente haben das Potenzial, ein Bindungsereignis zu zeigen. Ist in den Probenspektren im Vergleich zu den Leerspektren ein CSP von mehr als 6 Hz sichtbar, wird dies als Hinweis auf einen Treffer gewertet. Gleiches gilt, wenn ein starkes positives Signal im waterLOGSY sowie eineT2-Reduktion von mehr als 30% in den Probenspektren sichtbar ist. Bindungsereignisse können in allen drei Experimenten gezeigt werden, wenn die Probe, die Spektren enthält, mit ihren jeweiligen Leerspektren verglichen wird. Es kann jedoch sein, dass Bindungsereignisse nicht in allen drei Experimenten sichtbar sind. Aus diesem Grund wurde vereinbart, dass mindestens zwei der zuvor beschriebenen Ereignisse eintreten müssen, um ein Fragment als verbindlichen Treffer zu klassifizieren.
- Verwenden Sie das FBS-Werkzeug in TopSpin, um den Status von Fragmenten in Bindung, Mehrdeutigkeit, Unbekannt, Aggregate und Nicht-Bindung zu definieren.
- Wenn Sie mit einem Mix fertig sind, genehmigen Sie ihn im FBS-Tool.
- Klicken Sie auf der Registerkarte "Zusammenfassung" innerhalb des FBS-Projekts auf Screening-Bericht erstellen. Dadurch wird ein Fenster geöffnet, in dem eine .xlsx Datei erstellt wird. Der Benutzer kann dann zwischen allen Liganden, nur bindenden Liganden, nur bindenden Liganden und mehrdeutigen Liganden wählen, die in der Tabelle angegeben werden sollen.
- 19F-Bildschirm
- Zur Unterscheidung zwischen Non-Binder, Week-Binder und Strong-Binder dividiert man den Integrationsquotienten zwischen der 200-ms-Target-Messung und der 200-ms-Blindmessung durch den Quotienten aus 0-ms-Target-Messung und 0-ms-Blindmessung:
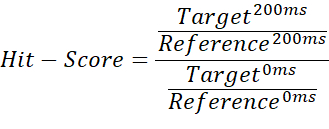
HINWEIS: Dies ergibt Werte im Bereich von 0 bis ~1 (die Trefferbewertung), sodass Schwellenwerte für jeden Bindungsstatus zugewiesen werden können. - Verwenden Sie den Durchschnitt der Referenzmessung von 200 ms als Basisschwellenwert, um Fälle zu markieren, in denen der Trefferwert 1 überschreitet. Dies kann der Fall sein, wenn die importierten Integrale negative Werte enthalten oder die Referenzmessung höher als die Sollmessung ist. Ein Trefferwert von ≤ 0,67 gilt als schwacher Treffer, < 0,33 als starker Treffer und alles > 0,67 als kein Treffer. Ein Beispiel ist in Abbildung 2 dargestellt.
- Zur Unterscheidung zwischen Non-Binder, Week-Binder und Strong-Binder dividiert man den Integrationsquotienten zwischen der 200-ms-Target-Messung und der 200-ms-Blindmessung durch den Quotienten aus 0-ms-Target-Messung und 0-ms-Blindmessung:
- 1H-Bildschirm
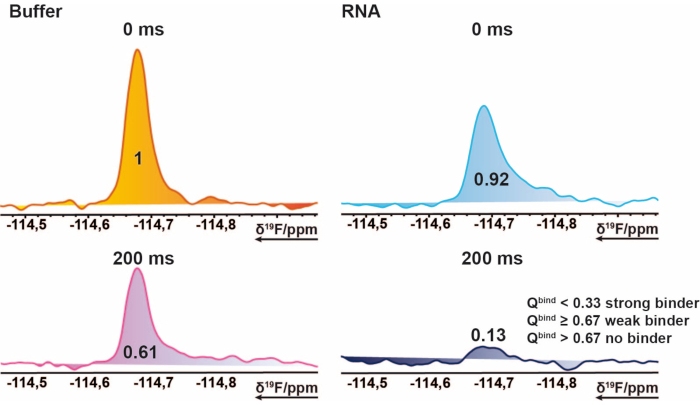
Abbildung 2: Trefferidentifikation für das 19F-Screening. Ausschnitt aus 19F CPMG NMR-Spektren einer beispielhaften Verbindung. Diese bildliche Darstellung erklärt die Eigenschaften eines Bindemittels. 19F-CPMG-Spektren einer Verbindung, die aus Mischungsproben in An- und Abwesenheit von RNA aufgenommen wurden. Die Werte stellen die normierten ganzzahligen Werte des entsprechenden Peaks dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Datenanalyse
- Vorbereiten von Daten für die Analyse
HINWEIS: Es ist wichtig, dass die erfassten Daten keine sichtbaren Mängel aufweisen. Dies bedeutet, dass Daten, bei denen das Shimming problematisch war oder die Wasserunterdrückung unzureichend war, für die Analyse nicht berücksichtigt werden sollten. Vielmehr empfiehlt es sich, die Daten erneut aufzuzeichnen und sicherzustellen, dass mit der Probe alles in Ordnung ist (z. B. keine Luftblasen), mit der Temperatur, dem Shimming und der Wasserunterdrückung. Die Korrektheit der Daten kann beim Vergleich von DMSO-Signalen immer beurteilt werden. - 1H-Siebung
- Um 1-H-Screening-Daten zu analysieren, verwenden Sie das FBS-Tool (erfordert zusätzliche Lizenz) in TopSpin 4.0.9.
- Befolgen Sie die Anweisungen im FBS-Toolhandbuch, um mit der Datenanalyse zu beginnen. Die folgenden Schritte fassen die im Handbuch beschriebene Vorgehensweise zusammen.
- Speichern Sie die BMRZ-NMR-Daten von Screening-Kampagnen so, dass jede verschiedene Screening-Mischung ein eigenes Verzeichnis hat, in dem ein Unterverzeichnis die verschiedenen an der Probe gemessenen Experimente enthält.
- Um das FBS-Tool zu verwenden, speichern Sie die Referenzspektren, die alle gespeicherten Daten von Proben ohne das biomolekulare Ziel, aber mit den Mischungen sowie der gemessenen Einzelverbindung enthalten, in verschiedenen /nmr-Verzeichnissen. Dies ist wichtig, da das FBS-Tool nach dem Verzeichnispfad jedes einzelnen fragt.
HINWEIS: Das FBS-Tool erkennt ein Verzeichnis als Screening-Projekt, wenn die folgenden Datensätze in demselben Verzeichnis gespeichert wurden, in dem die Mischungen einer Screening-Probe gespeichert sind (csv, FragmentScreen-XML-Dokumente und BAK-Datei). - Wenn Sie TopSpin 4.0.9 verwenden, erstellen Sie einen direkten Pfad zu dem Verzeichnis, das die erfassten Daten enthält, ein sogenanntes DIR. Wählen Sie das Verzeichnis /nmr, in dem alle Mischungen ein eigenes Verzeichnis haben sollen.
- Um das FBS-Werkzeug eines gerasterten Samples zu starten, ziehen Sie das Symbol-FBS-Projekt in die Mitte des TopSpin-Fensters. Im gewählten Verzeichnis sollte das FBS-Projektsymbol erscheinen, wenn diese Datensätze zuvor in das Verzeichnis kopiert wurden.
- Das Fenster Fragment Based Screening Options sollte sich automatisch öffnen, wenn ein neues FBS-Projekt zum ersten Mal geladen wird. Wählen Sie in diesem Fenster eine Cocktaildatei aus. Die Cocktail-Datei ist eine csv-Datei, die die Zuweisung des Namens der Mixe, den Namen jedes Fragments und deren Aufteilung in die Mixe enthält. Definieren Sie außerdem einen Referenzligandenspektrenordner, der alle gemessenen Spektren der einzelnen Fragmente enthält. Definieren Sie schließlich einen leeren Referenz-Experimentordner, bei dem es sich in der Regel um den Ordner handelt, der die Datensätze der Mischungen ohne das untersuchte Ziel enthält.
- Die Fragment Based Screening Options haben eine Registerkarte namens Spectra types , auf der man die untersuchten Spektren sowie die Farbe für die Anzeige der Spektren definieren kann. Legen Sie den Spectype entsprechend den zuvor verarbeiteten Daten fest. Definieren Sie auf der Registerkarte Anzeigelayout die Spektren, die entsprechend ihren Spezifikationstypen miteinander verglichen werden sollen.
- Drücken Sie OK , um das FBS-Projekt zu starten.
- Während der Betrachtung der Daten öffnet sich ein separates Fenster, in dem alle Cocktailmischungen und alle Liganden jeder Mischung in einer Tabelle zusammengefasst sind. Mit einem Doppelklick auf eine Zelle öffnen sich die entsprechenden Datensätze, die z.B. 1H 1D Blank Spektren mit dem Datensatz vergleichen, der das Ziel enthält.
- Stellen Sie vor der Zuweisung von Bindemitteln sicher, dass die Referenzpeaks (DMSO aller Messungen sowie der einzelnen Verbindungen) zueinander passen und die gleiche chemische Verschiebung aufweisen. Wenn Unterschiede festgestellt werden, korrigieren Sie diese mit der seriellen Verarbeitungsoption von TopSpin.
- Die Option für die serielle Verarbeitung befindet sich auf der Registerkarte " Prozess " unter "Erweitert". Es wendet Änderungen auf alle ausgewählten Spektren aus einem Datensatz an. Auf diese Weise können Spectypes einfach Experimentnummern zugewiesen werden und alle Spektren können auf einmal verschoben werden, um sie an der Referenz auszurichten.
- 19F-Screening
- Für die erste Analyse der 19F-Mischungen erstellen Sie für jede Mischung eine Integrationsdatei. Um den Integrationsbereich zu definieren, klicken Sie auf der Registerkarte Analyse auf die Funktion Integrieren. Stellen Sie sicher, dass für jedes Fragment in der Mischung ein eindeutiger Integrationsbereich für die entsprechenden 19F-Singale definiert ist.
- Verwenden Sie die Schaltfläche Integrationsbereiche speichern/ exportieren, um die Integrationsdatei für die zukünftige Verwendung zu exportieren. Speichern Sie alle verwendeten Integrationsdateien in C:\Bruker\TopSpin4.0.9\exp\stan\nmr\lists\intrng oder im entsprechenden Pfad des TopSpin-Installationsverzeichnisses.
- Öffnen Sie für 19F-Daten einen Datensatz mit oder ohne das untersuchte Ziel.
- Um die Integrationsdatei in das aktuelle Spektrum zu laden, öffnen Sie erneut den Reiter Analyse , gehen Sie auf Integrieren und laden Sie über die Schaltfläche Integrationsregionen lesen/importieren die entsprechende Integrationsdatei. Dadurch werden alle definierten Regionen dieser Datei in das aktuelle Spektrum geladen.
- Speichern Sie und kehren Sie zurück, um eine Liste aller integrierten Regionen auf der Registerkarte "Integrale " zu finden. Kopieren Sie dies in eine Tabellenkalkulation oder ein anderes Tool, das für die weitere Analyse der Daten verwendet wird.
- Wiederholen Sie diesen Vorgang für jede Mischung, mit und ohne Ziel.
- Datenmanagement
- Um die Benutzerfreundlichkeit und Produktivität zu erhöhen, sollten Sie einen einheitlichen Arbeitsablauf für die weitere Analyse und Speicherung der erfassten Daten einrichten. Verwenden Sie sowohl für das 1-H- als auch für das 19-F-Screeningjeweils eine speziell entwickelte Tabelle.
ANMERKUNG: Für das 1-H-Screening wurde dies ausschließlich für das Datenmanagement und zur Zusammenfassung jedes Ziels verwendet, während für das 19-F-Screeningder in Kapitel 4.3 erläuterte Quotient verwendet wurde, um jedes Fragment automatisch als Treffer/kein Treffer zu kennzeichnen, nachdem die integralen Daten hineinkopiert wurden. Dies verringert das Risiko menschlicher Fehler während der Analyse, vorausgesetzt, die Datei wurde ordnungsgemäß eingerichtet, und erleichtert den Austausch von Informationen, da alle wichtigen Informationen an einem Ort in einer Datei gesammelt werden, die von praktisch jedem geöffnet werden kann, ohne dass weitere Programme für einen ersten Blick auf die Daten erforderlich sind.
- Um die Benutzerfreundlichkeit und Produktivität zu erhöhen, sollten Sie einen einheitlichen Arbeitsablauf für die weitere Analyse und Speicherung der erfassten Daten einrichten. Verwenden Sie sowohl für das 1-H- als auch für das 19-F-Screeningjeweils eine speziell entwickelte Tabelle.
- Vorbereiten von Daten für die Analyse
Ergebnisse
Qualitätskontrolle der Fragmentbibliothek
Die Fragmente aus der hauseigenen Bibliothek wurden als 50 mM Stammlösungen in 90 % d6-DMSO und 10 % D2 O geliefert (10 %D2O sorgen für eineMinimierung des Verbindungsabbaus durch wiederholte Gefrier-Tau-Zyklen14). Die Einzelproben bestanden aus 1 mM Ligand in 50 mM Phosphatpuffer (25 mM KPi pH 6,2 + 50 mM KCl + 5 mM MgCl2), pH 6,0 in 90%H2O/9% D2O/1% d6-DMSO. 1H-NMR-Experimente von Fragmenten aus der iNEXT-Bibliothek wurden mit einem 500/600 MHz NMR-Spektrometer gemessen. Diese Daten wurden weiter verwendet, um die einzelnen Verbindungen in 1-H-Screening-Kampagnen mit der CMC-q-Software zu identifizieren, die es dem Benutzer ermöglicht, Spektren vollständig und automatisiert zu erfassen, und mit dem Analyse-Addon CMC-a wurde die Qualität (Löslichkeit und Integrität) von Fragmenten bewertet. Die Ergebnisse der automatisierten Analyse von CMC-a werden ähnlich wie in Abbildung 3 dargestellt als grafische Ausgabe dargestellt. Die grafische Ausgabe zeigt eine Darstellung einer 96-Well-Platte. Ein rot gefärbter Kreis bedeutet, dass dieses Fragment eine Inkonsistenz in der Struktur oder Konzentration aufweist. Grün eingefärbte Vertiefungen zeigen an, dass das Fragment konsistent ist.
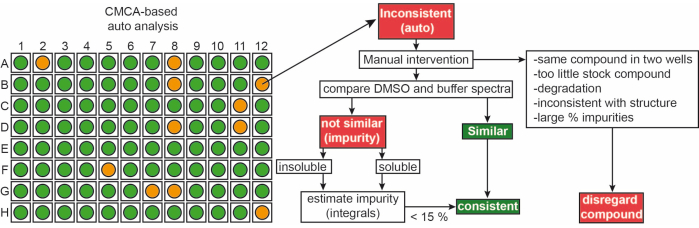
Abbildung 3: Qualitätskontrolle der Fragmentbibliothek. Schematische Darstellung der CMC-a-basierten automatisierten Ausgabe. Fragmenteigenschaften wie Konzentration und strukturelle Integrität werden bewertet. Grün steht für konsistent, Orange steht in diesem Fall für inkonsistent. Inkonsistente Fragmente werden gemäß dem gezeigten Workflow manuell überarbeitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ungefähr 65 % bzw. 35 % der Fragmente wurden sowohl im DMSO als auch im Puffer als konsistent bzw. inkonsistent klassifiziert. Darüber hinaus wurden 30% der inkonsistent klassifizierten Liganden nach einer sorgfältigen manuellen Inspektion der Spektren konsistent9.
19F Gemischausführung
103 Fragmente, die eine oder mehrere Fluorgruppen aus der hauseigenen Bibliothek enthielten, wurden in 5 Mischungen (A, B, C, D, E) aufgeteilt. Jede Mischung besteht aus 20 bis 21 Fragmenten. In diesem Fall mussten die Mischungen sorgfältig ausgelegt werden, um Signalüberlappungen zu vermeiden. 19F-transversale Relaxationsexperimente wurden für jedes Gemisch gemessen, das CPMG-Pulsfolgen anwendete. Diese Experimente können durch Variation der Relaxationsverzögerungen modifiziert werden. Die chemische Verschiebung der Mischungen A-E um 19F ist in Abbildung 4 zu sehen.
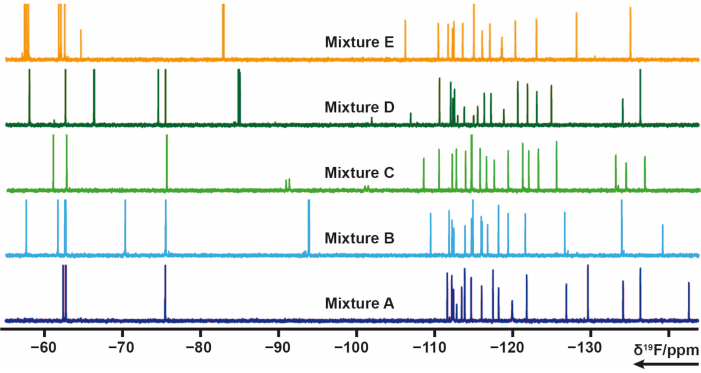
Abbildung 4: 19F 1D-NMR-Spektren von Mischungsproben aus der hauseigenen Bibliothek. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Probenvorbereitung
Die Probenvorbereitung im 19F-Screening-Verfahren erfolgte entweder manuell oder mit automatisiertem Pipettieren mittels Pipettierroboter. Die Fragmente in jeder Mischung wiesen eine Konzentration von 2,5 mM in 90 % d6-DMSO und 10 %D2O auf. Das Endvolumen einer Screening-Probe betrug 170 μL mit 5 %D2Oals Lockmittel. Jedes Gemisch wurde zweimal pipettiert, einmal in eine pufferhaltige Lösung (ohne Target) und einmal in eine Target-haltige Pufferlösung. Das Verhältnis von Target und Fragment wurde auf 1:1 eingestellt, was zu einer endgültigen Target/Ligand-Konzentration von 50 μM führte. Zusätzlich sind Kontrollproben das Zielbiomolekül im Screening-Puffer ohne Mischung, um die Target-Integrität zu gewährleisten, sowie eine Kontrollprobe nur mit Puffer undD2O,um die Pufferqualität zu gewährleisten.
Bei den NMR-Screening-Daten von 19 F-1D und 19F-CPMG-T2 handelte es sich um Messungen gemäß Abschnitt 3.1. Im Falle von RNA wurde beispielsweise eine Jump-Return-Echosequenz (pp = zggpjrse,15) für die einzelne Zielprobe im Puffer erfasst.
Datenanalyse
Das 19F-Screening-Verfahren wurde unter anderem auf den TPP-Riboswitch thiM aus E. coli und die Protein-Tyrosinkinase (PtkA) aus M. tuberculosis angewendet16. Die 19F Screening-Bibliothek enthält 103 Fragmente, die in 5 Mischungen unterteilt sind, die von Mix A bis E beschriftet sind. Die Vorbereitung von Screening-Proben kann manuell ohne den Einsatz eines Probenpipettierroboters durchgeführt werden. 40 μM thiM RNA enthaltende Lösung (Pufferbedingungen) wurde mit 3,2 μL aus den Mischungen gemischt. Weitere Kontrollproben wurden aus reinem Puffer, Puffer mit 5% DMSO (zuvor die Stabilität des Biomakromoleküls in Gegenwart der gewünschten DMSO-Konzentration sichergestellt) und Puffer mit RNA hergestellt. Diese 13 Screening-Proben wurden präpariert und in 3 mm NMR-Röhrchen überführt. Barcodes von NMR-Röhrchen werden gescannt und jede Mischung in An- und Abwesenheit von RNA sowie Kontrollproben wurden gemäß den oben genannten 19F-NMR-Experimenten gemessen, die bei 298 K durchgeführt wurden. Das Screening der thiM-RNA gegen die hauseigene Bibliothek wurde durch Durchführung vonT2-Messungen mit CPMGs von 0 ms und 200 ms für jede unterschiedliche Probe durchgeführt. Das korrekte Shimming und die Wasserunterdrückung wurden nach Abschluss der Messungen durch den Vergleich aller DMSO-Peaks in Bezug auf Linienverbreiterung und Intensitätsverlust der zusätzlich gemessenen 1H 1D-Experimente für alle Proben überwacht. Die Verarbeitung der erhaltenen CPMG T219F Relaxationsspektren erfolgte mit Hilfe eines zuvor vorbereiteten bzw. automatisierten Makros in TopSpin. Die Datenanalyse wurde gemäß den Anweisungen im Protokollabschnitt durchgeführt. Die von TopSpin erhaltenen integralen Daten (gemäß den Anweisungen im Protokoll) können schnell und einfach mit einer vorgefertigten Tabelle oder einem ähnlichen Programm ausgewertet werden, indem die richtigen Bedingungen und Schwellenwerte festgelegt werden. Wie bereits beschrieben, sind Schwellenwerte nützlich, um Bindemittel, schwache Bindemittel oder Nicht-Bindemittel zu definieren. Abbildung 5 zeigt typische Ergebnisse von CPMG-Spektren von thiM-RNA bzw. PtkA. In einigen Fällen war eine weitere fachliche Überarbeitung erforderlich.
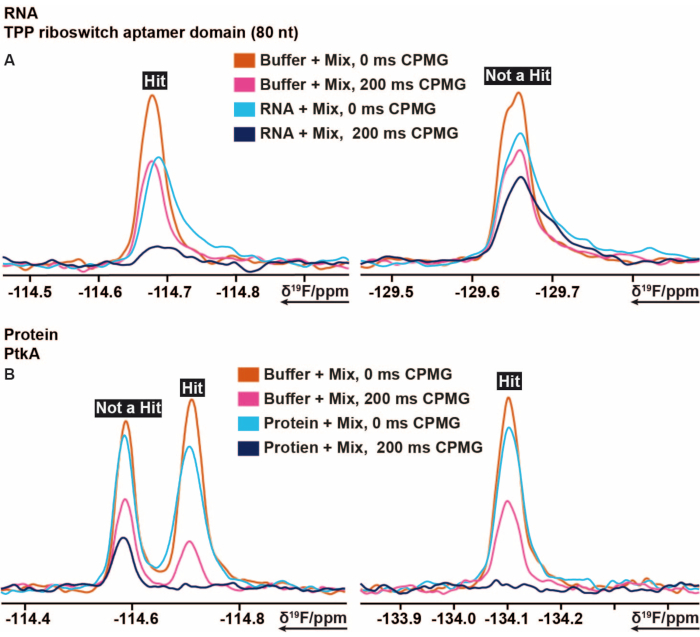
Abbildung 5: Ausschnitt aus 19 F CPMG NMR-Spektren, die die Intensitätsänderungen zeigen, die durch verschiedene Verzögerungszeiten von CPMG-basierten Experimenten erzielt wurden . (A) Darstellung eines Binders (Hit) und eines Nicht-Binders in einem 19F-Fragment-basierten Screening, das auf TPP-Riboswitch-thiM-RNA aus E. coli durchgeführt wurde. (B) Darstellung eines Bindemittels und eines Nicht-Bindemittels im 19F Fragment-basierten Screening, das auf PtkA von M. tuberculosis durchgeführt wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
1h Screening
Mischungsdesign
Die verwendete hauseigene Bibliothek ist so vielfältig, dass für die1-Stunden-Siebung kein Mischungsdesign durchgeführt wurde. Das bedeutet, dass 64 Mischungen hergestellt wurden, indem 12 nach dem Zufallsprinzip ausgewählt wurden, um sie in einer Mischung zu mischen.
Probenvorbereitung
Für das 1-Stunden-Screening einer exemplarischen SARS-CoV-2-RNA wurde ein automatisiertes Pipettieren mit einem Pipettierroboter durchgeführt, um die Proben vorzubereiten. Die Fragmente in jeder Mischung wiesen eine Konzentration von 4,2 mM in 90 % d6-DMSO und 10 %D2O auf. Das Endvolumen einer Screening-Probe betrug 200 μL mit 5 %D2Oals Lockmittel. 64 Proben, die jeweils eine andere Mischung in 25 mM KPi, 50 mM KCl bei pH 6,2 enthielten, wurden ohne Ziel-RNA pipettiert. Jeweils wurden 64 Proben mit Ziel-RNA pipettiert, die jeweils eine andere Mischung enthielten. Das RNA:Ligand-Verhältnis wurde auf 1:20 eingestellt, woraus sich eine RNA-Konzentration von 10 μM und eine Ligandenkonzentration von 200 μM ergibt.
Datenanalyse
Für die 1-Stunden-Analyse wurde das FBS-Tool in TopSpin verwendet. Um festzustellen, ob es sich bei einem Fragment um einen Treffer handelt, wurden 1D-Chemikalienverschiebungs-, waterLOGSY- und T2-Relaxationsexperimente durchgeführt. Für dieT2-Relaxation wurde eine Intensitätsabnahme von mehr als 30 % als Treffer gewertet, während für die chemische Verschiebung eine Verschiebung von mehr als 6 Hz als Cut-off gewertet wurde. Der waterLOGSY musste eine signifikante Signaländerung aufweisen (in diesem Fall von negativ zu positiv). Wenn zwei dieser drei Kriterien positiv waren, wurde ein Fragment als Treffer gewertet. Zwei Beispiele hierfür sind in Abbildung 6 zu sehen.
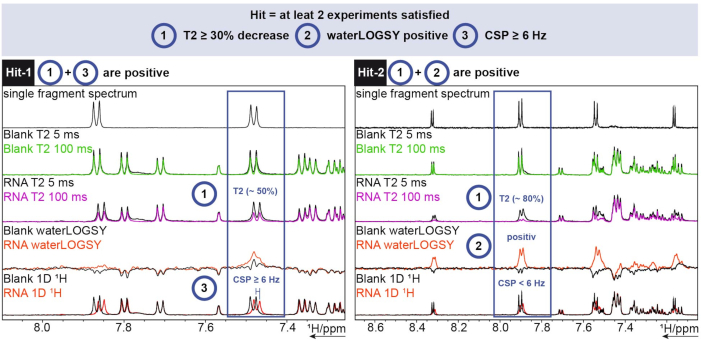
Abbildung 6: 1-H-Screening an einer beispielhaften SARS-CoV-2-RNA mit Trefferbestimmungskriterien. Erfassung von drei verschiedenen Experimenten (1 H T2 CPMG (5/100 ms), waterLOGSY und 1D 1H). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Hit-1 zeigt eineT2-Abnahme von ~50% und einen CSP ≥ 6 Hz. Der waterLOGSY zeigt keine ausreichend signifikante Signaländerung, um ebenfalls als positiv gewertet zu werden. Da zwei von drei Experimenten positiv verlaufen, wird dieses Fragment als Treffer gewertet. Für Hit-2 zeigt der T2 eine Abnahme der Signalintensität von ~80% und für den waterLOGSY ist eine deutliche Signaländerung zu erkennen. Der CSP reicht in diesem Fall nicht aus, aber da die beiden vorherigen Kriterien positiv sind, wird er dennoch als Treffer gezählt.
Diskussion
Vielseitigkeit des NMR-basierten Fragment-/Wirkstoff-Screenings. Das BMRZ hat erfolgreich modernste automatisierte NMR-Instrumente sowie STD-NMR-, waterLOGSY- und Relaxationsexperimente implementiert, um Fragmente innerhalb eines breiten Affinitätsbereichs für die Wirkstoffforschung zu identifizieren. Die installierte Hardware umfasst einen Hochdurchsatz-Probenvorbereitungsroboter und eine Hochdurchsatz-Probenspeicher-, Wechsler- und Datenerfassungseinheit, die einem 600-MHz-Spektrometer zugeordnet sind. Eine kürzlich erworbene Kryosonde für 1 H, 19 F, 13C und 15 N gewährleistet die erforderliche Empfindlichkeit für die vorgeschlagenen Messungen und ermöglicht eine Entkopplung von 1 H (1) während der 19F-Detektion. Diese Sonde ist mit der neuesten Generation von NMR-Konsolen verbunden, die die Möglichkeit bieten, die fortschrittlichen Software-Tools von Bruker zu verwenden, einschließlich CMC-q, CMC-assist, CMC-se und FBS (in TopSpin enthalten). Das fragmentbasierte Screening-Tool (FBS) ist in der neuesten Version von TopSpin enthalten und hilft bei der Analyse der Hochdurchsatzdaten, die aus STD-, waterLOGSY- und T2/T 1-r-Relaxationsexperimenten bestehen. Die flüssige 1D-1H-Probenentnahme kann mit Hilfe des Probenfüllroboters automatisiert in die NMR-Röhrchen gefüllt werden. In der Regel wird ein Block von 96 Röhrchen (3 mm) in etwa zwei Stunden befüllt. Die 96-Well-Platten-Racks werden direkt im HT-Probenwechsler positioniert, der den Barcode des Blocks liest und die NMR-Röhrchen den von der Automatisierungssoftware (IconNMR) gesteuerten Experimenten zuordnet. Fünf 96-Well-Platten-Racks können gleichzeitig im HT-Probenwechsler gelagert und programmiert werden. Die Temperatur der einzelnen Racks kann separat gesteuert und geregelt werden. Zusätzlich kann jede einzelne Probe vor der Messung auf die gewünschte Temperatur vorkonditioniert werden (Vorwärmen und Röhrchentrocknung zur Entfernung von kondensierter Feuchtigkeit).
Eignung für ein breites Anwendungsspektrum. Eine der breiten Anwendungen dieses automatisierten NMR-basierten Screenings ist die Identifizierung und Entwicklung neuartiger Liganden, die an ein biomakromolekulares Ziel (DNA/RNA/Proteine) binden. Zu diesen Liganden können orthosterische und allosterische Inhibitoren gehören, die typischerweise nicht-kovalent binden. Darüber hinaus wird die FBDD mittels NMR in der Regel als erster Schritt zur Auswahl vielversprechender Verbindungen eingesetzt, wobei die zu erfüllenden Anforderungen die Verfügbarkeit des biomolekularen Targets in ausreichenden Mengen sind. Dieses Ziel gliedert sich in zwei Hauptaufgaben.
Aufgabe eins ist die Entwicklung und Charakterisierung einer internen Fragmentbibliothek aus folgenden Gründen: anfängliche und periodische Qualitätskontrolle, Charakterisierung und Quantifizierung von mehr als 1000 Fragmenten; Bestimmung der Löslichkeit der Fragmente in Puffern, die für jedes Target, insbesondere für Proteintargets, optimiert sind; und die Einrichtung mehrerer Bibliotheken, um verschiedene Gerüste unterzubringen und sich auf andere Makromolekülklassen auszudehnen. Aufgabe zwei ist die Integration von Arbeitsabläufen für das fragmentbasierte Wirkstoffdesign (FBDD) mittels NMR mit: automatisiertem 1D-Liganden-Beobachtungs-Screening (1H und 19F beobachtet); automatisierte Ersatzassays (Konkurrenzexperimente mit (natürlichen) Liganden) zur Unterscheidung von orthosterischer und allosterischer Bindung; automatisierte Sekundärscreenings mit mehreren Fragmenten; automatisiertes 2D-Protein-Screening und Sekundär-Screening einer Reihe von Derivaten um einen ersten Treffer unter Verwendung der EU-OPENSCREEN-Bibliothek oder einer anderen Bibliothek; und Re-Profiling-Screening der FDA-Bibliothek gegen die ausgewählten Ziele.
Darüber hinaus kann die Metabotypisierung verschiedener Zelllinien (krankheitsrelevant) durchgeführt werden, um die regulatorischen Mechanismen zu entschlüsseln, die die Kontrolle des Zellzyklus und den Stoffwechsel miteinander verbinden. Darüber hinaus gibt es eine funktionelle Charakterisierung von RNA/DNA/Protein-Regulationselementen in vivo und in vitro zur Optimierung der Konstrukt-/Domänenoptimierung (Stabilitätsoptimierung für strukturelle Untersuchungen (Puffer-, pH-, Temperatur- und Salzscreening) und eine Erweiterung des NMR-basierten Fragmentscreenings auf Membranproteine und intrinsisch ungeordnete Proteine, die für andere Techniken im Allgemeinen unzugänglich sind.
Begrenzungen. Die Verwendung von 19-F- und 1-H-Fragmentbibliotheken hat ihre Vor- und Nachteile, von denen im Folgenden einige erwähnt werden. Der größte Vorteil von 19F gegenüber 1H Messungen ist die Geschwindigkeit sowohl der eigentlichen Messzeit als auch der anschließenden Analyse, da die Mischungen fast doppelt so viele Fragmente enthalten und weniger Experimente durchgeführt werden müssen. Auch die Nachanalyse ist für das 19F-Screening einfacher, da es keine Interferenzen durch Puffer gibt und zusätzlich einen breiteren chemischen Verschiebungsbereich mit nahezu keiner Signalüberlappung für ein optimal ausgelegtes Fragmentgemisch bietet. Die Spektren selbst sind stark vereinfacht und haben in der Regel nur ein oder zwei Signale pro Fragment, abhängig von der Anzahl der Fluoratome. Die Analyse dieser Spektren kann so automatisiert werden, was wiederum Zeit spart. Dies geht auf Kosten der chemischen Vielfalt, zumindest für die in dieser Studie verwendete Bibliothek. Da nur ~13% der Bibliothek 19 F enthalten, aber natürlich alle im 1-H-Screening verwendbar sind, ist die Diversität der 19F-Screening-Fragmente geringer. Dies könnte durch die Verwendung speziell entwickelter 19-F-Bibliotheken mit mehr Fragmenten und größerer chemischer Vielfalt umgangen werden. Ein weiterer Nachteil des 19-F-Screenings ist die geringe Anzahl von Signalen pro Fragment. Fragmente bestehen in der Regel aus mehr als einem Wasserstoffatom. Daher können sich 1H beobachtete Screening-Experimente auf unterschiedliche Signale für dasselbe Fragment verlassen, um die Bindung zu erkennen. Dies führt zu einem höheren Maß an Sicherheit bei der Identifizierung von Treffern für das 1-H-Screening, während sich das 19-F-Screeningauf ein oder zwei Signale pro Fragment verlassen muss.
Es wurde ein detaillierter Bericht über die modernen, automatisierten NMR-basierten Fragment-Screening-Instrumente, die Software und die Analysemethoden und deren Protokolle vorgestellt. Die installierte Hardware umfasst einen Hochdurchsatz-Probenvorbereitungsroboter und eine Hochdurchsatz-Probenspeicher-, Wechsler- und Datenerfassungseinheit, die einem 600-MHz-Spektrometer zugeordnet ist. Ein kürzlich installierter kryogener Sondenkopf für 1 H, 19 F, 13C und 15N gewährleistet die erforderliche Empfindlichkeit für die vorgeschlagenen Messungen und ermöglicht eine1-H-Entkopplung bei 19F-Detektion. Darüber hinaus bietet die neueste Generation der NMR-Konsole die Möglichkeit, fortschrittliche Analysesoftware zur Unterstützung der Erfassung und On-the-Fly-Analyse zu verwenden. Die oben besprochene Technologie, die Arbeitsabläufe und die beschriebenen Protokolle sollten Anwendern, die FBS mittels NMR verfolgen, einen bemerkenswerten Erfolg bescheren.
Offenlegungen
Nichts.
Danksagungen
Diese Arbeit wurde von iNEXT-Discovery, Projekt Nummer 871037, gefördert durch das Horizon 2020 Programm der Europäischen Kommission.
Materialien
| Name | Company | Catalog Number | Comments |
| Bruker Avance III HD | Bruker | 600 MHz NMR Spectrometer | |
| Matrix Clear Polypropylene 2D Barcoded Open-Top Storage Tubes | 3731-11 0.75ML V-BOTTOM TUBE/LATCH RACK | ThermoFisher Scientific | Barcoded Tubes |
| Matrix SepraSeal und DuraSeal& | 4463 Cap Mat, SeptraSeal 10/CS | ThermoFisher Scientific | |
| SampleJet | Bruker | HT Sample Changer | |
| SamplePro Tube | Bruker | Pipetting Robot |
Referenzen
- Yanamala, N., et al. NMR-Based Screening of Membrane Protein Ligands. Chemical Biology & Drug Design. 75, 237-256 (2010).
- Souers, A. J., et al. ABT-199, a potent and selective BCL-2 inhibitor, achieves antitumor activity while sparing platelets. Nature Medicine. 19, 202-208 (2013).
- Su, M. C., Te Chang, C., Chu, C. H., Tsai, C. H., Chang, K. Y. An atypical RNA pseudoknot stimulator and an upstream attenuation signal for -1 ribosomal frameshifting of SARS coronavirus. Nucleic Acids Research. 33, 4265-4275 (2005).
- Perera, T. P. S., et al. Discovery & pharmacological characterization of JNJ-42756493 (Erdafitinib), a functionally selective small-molecule FGFR family inhibitor. Molecular Cancer Therapeutics. 16, 1010-1020 (2017).
- Zhang, C., et al. Design and pharmacology of a highly specific dual FMS and KIT kinase inhibitor. Proceedings of the National Academy of Sciences of the United States of America. 110, 5689-5694 (2013).
- Lipinski, C. A., Lombardo, F., Dominy, B. W., Feeney, P. J. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings. Advanced Drug Delivery Reviews. 23, 3-25 (1997).
- Congreve, M., Carr, R., Murray, C., Jhoti, H. A 'Rule of Three' for fragment-based lead discovery. Drug Discovery Today. 8, 876-877 (2003).
- Chávez-Hernández, A. L., Sánchez-Cruz, N., Medina-Franco, J. L. A Fragment Library of Natural Products and its Comparative Chemoinformatic Characterization. Molecular Informatics. 39, 2000050 (2020).
- Sreeramulu, S., et al. NMR quality control of fragment libraries for screening. Journal of Biomolecular NMR. , 00327-00329 (2020).
- Gao, J., et al. Automated NMR Fragment Based Screening Identified a Novel Interface Blocker to the LARG/RhoA Complex. PLoS One. 9, 88098 (2014).
- Peng, C., et al. Fast and Efficient Fragment-Based Lead Generation by Fully Automated Processing and Analysis of Ligand-Observed NMR Binding Data. Journal of Medicinal Chemistry. 59, 3303-3310 (2016).
- Cox, O. B., et al. A poised fragment library enables rapid synthetic expansion yielding the first reported inhibitors of PHIP(2), an atypical bromodomain. Chemical Science. 7, 2322-2330 (2016).
- Hwang, T. L., Shaka, A. J. Water Suppression That Works. Excitation Sculpting Using Arbitrary Wave-Forms and Pulsed-Field Gradients. Journal of Magnetic Resonance, Series A. 112, 275-279 (1995).
- Gossert, A. D., Jahnke, W. NMR in drug discovery: A practical guide to identification and validation of ligands interacting with biological macromolecules. Progress in Nuclear Magnetic Resonance Spectroscopy. 97, 82-125 (2016).
- Sklenar, V., Bax, A. A new water suppression technique for generating pure-phase spectra with equal excitation over a wide bandwidth. Journal of Magnetic Resonance. 75, 378-383 (1987).
- Binas, O., et al. 19F NMR-Based Fragment Screening for 14 Different Biologically Active RNAs and 10 DNA and Protein Counter-Screens. ChemBioChem. , (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten