Method Article
Ein auf Bead-Bewegung basierendes Berechnungsframework für die 3-dimensionale Analyse der Heterogenität von Biofilmmaterialien
In diesem Artikel
Zusammenfassung
Wir beschreiben eine Methode zur Analyse und Quantifizierung des Bewegungsmusters von 1 μm Carboxylenkügelchen durch heterogene bakterielle Biofilme. Der Vergleich der Bewegungsmuster kann verwendet werden, um Unterschiede in den Materialeigenschaften von Biofilmen zu quantifizieren.
Zusammenfassung
Unterschiede in den Materialeigenschaften von bakteriellen Biofilmen wurden bei Biofilmen verschiedener Bakterienarten innerhalb derselben Spezies unter unterschiedlichen Wachstumsbedingungen und nach Behandlung mit matrixmodifizierenden Molekülen beobachtet. Um die Materialeigenschaften von 3D-Biofilmen besser quantifizieren zu können, wurde ein experimenteller und rechnerischer Workflow entwickelt und angewendet, um Unterschiede zwischen Enterococcus faecalis, Salmonella enterica Serotyp Typhimurium und Escherichia coli Biofilmen sowie die Rolle der Amyloid-Curli bei der Bestätigung der Steifigkeit gegenüber Enterobacteriaceae-Biofilmen zu untersuchen. Die räumlich-zeitliche Dynamik von 1 μm Carboxylatkügelchen in Biofilmen wurde in 20 μm 3D-Biofilmen über einen Zeitraum von 20 Minuten verfolgt. Die 4D-Bildstapel wurden mit dem Mosaic-Plugin in ImageJ verarbeitet, um 3D-Trajektoriendaten der Perlenbewegung zu erzeugen. Diese Trajektoriendaten wurden mit einer neu entwickelten Bead Evaluator-Toolbox analysiert, in der Bewegungsdaten, einschließlich Trajektorienlebensdauer, Bead-Geschwindigkeiten, Zelldichten entlang der Trajektorien und Bounding-Box-Informationen, berechnet und in csv-Dateien gespeichert wurden. In diesem Beitrag wird der Arbeitsablauf vom Versuchsaufbau und der Bildaufzeichnung bis hin zur Berechnung und Analyse der Raupenbahn vorgestellt. Die Struktur der Curli-haltigen Biofilme führte zu stabileren Bead-Wechselwirkungen und weniger Bead-Bewegung als bei Curli-Mutanten und Enterokokken-Biofilmen. Die Bewegung der Beads schien bei der Messung der Bead-Geschwindigkeit und des Trajektorien-Bounding-Box-Volumens nicht stark von der Zelldichte abhängig zu sein, was die Hypothese unterstützt, dass andere Materialeigenschaften der Biofilme die Bead-Dynamik steuern. Diese Technik ist vielseitig einsetzbar für die Quantifizierung von Unterschieden in Biofilmen unterschiedlicher Matrixzusammensetzungen sowie von Biofilmen vor und nach matrixmodifizierenden Behandlungen.
Einleitung
Bakterielle Biofilme sind als Teil der menschlichen Mikrobiota allgegenwärtig und interagieren kontinuierlich mit Molekülen. Diese Moleküle reichen in der Größe von 1 nm Antibiotika und 1-3 mm großen Bakterien bis hin zu größeren Ballaststoffpartikeln im Magen-Darm-Trakt. Die Zusammensetzung von Einzel- oder Mehrspezies-Biofilmen beeinflusst die Materialeigenschaften und damit das Bewegungsmuster der Partikel durch die Biofilme 1,2,3,4,5. Ein Beispiel sind bakterielle Amyloide, die eine konservierte, fibrilläre Cross-Beta-Faltblattstrukturaufweisen 6. Amyloid curli wird von enterischen Bakterien wie Escherichia coli und Salmonella enterica Serotyp Typhimurium exprimiert, und Gene wurden in mehreren anderen Bakterienstämmen nachgewiesen7. Verschiedene Materialeigenschaften von Biofilmen werden durch curli 8,9 beeinflusst. Curli interagiert direkt mit anderen Bestandteilen der Matrix wie extrazellulärer DNA (eDNA) und Cellulose10,11. Curli umgibt die Zellen und beeinflusst die Steifigkeit der Zellmembran12 und die gesamten viskoelastischen Eigenschaften des Biofilms13. Curli vermittelt eine erhöhte Zugfestigkeit durch Bindung an Fibronektin, was zu einer Zunahme der starken Glasoberflächenbindung führt14. Eindringende Bakteriophagen binden an Curli und begrenzen die Invasion von Phagen in Biofilme15.
Bei Verwendung von Multitest-beschichteten Well-Objektträgern zur Analyse von etwa 20 μm dicken Enterococcus faecalis, E. coli und S. Typhimurium-Biofilme mittels konfokaler Mikroskopie, deutliche Unterschiede zwischen E. coli, S. Es konnten Typhimurium10,16 und E. faecalis Biofilme (aktuelle Studie) beobachtet werden. Während Enterobacteriaceae-Spezies-Biofilme ein hohes Maß an Steifigkeit aufwiesen und Bereiche mit geringer Zelldichte leicht abzubilden waren, erforderte die Erstellung klarer, hochauflösender Bilder von E. faecalis-Biofilmen mittels Linien- und Rahmenmittelung die Ausübung von Druck auf den Objektträger, um während des Bildgebungsprozesses eine ausreichende Oberflächenspannung für die Zellstabilität zu induzieren. Bakterielle Amyloide wie Curli bilden hochgeordnete Strukturen, was darauf hindeutet, dass sie relativ starr sein könnten17. Dies motivierte die Hypothese, dass Amyloid-Curli bei E. coli und S. Rigidität induzieren könnten. Typhimurium-Biofilme. Es gab keine eindeutigen Beweise dafür, dass E. faecalis unter den untersuchten Bedingungen Amyloide exprimierte. Das Protein Esp, ein Pilin-Gen, das mit pathogeneren Stämmen von E. faecalis assoziiert ist, hat kürzlich gezeigt, dass es Amyloid-Strukturen produziert18; Bei der Blastn- und Blastp-Suche wurde dieses Gen jedoch in dem in diesen Studien verwendeten E. faecalis kommensalen Stamm OG1RF nicht nachgewiesen. Das von OG1RF produzierte Pheromon cOB1 kann Amyloid-ähnliche Strukturen bilden19. Mit den gegebenen Biofilmwachstumsbedingungen und Amyloid-Nachweismethoden, die bisher für S verwendet wurden.Typhimurium-Amyloid-Färbung10 in E. faecalis, OG1RF-Amyloide konnten nicht nachgewiesen werden (Daten nicht gezeigt). Es wurde eine neue vierdimensionale (4D) Bildtechnik entwickelt, um die Gesamtmaterialeigenschaften der viskosen E. faecalis, E. coli und S. Typhimurium zu vergleichen sowie den Beitrag von Amyloid zu Enterobacteriaceae-Biofilmen unter Verwendung von Amyloid-Mutanten von S zu bestimmen. Typhimurium und E. coli.
In der Vergangenheit wurden fluoreszierende Kügelchen erfolgreich eingesetzt, um die Materialeigenschaften von Biofilmen in zwei Dimensionen (2D) mit Hilfe der Mikrorheologie 20,21,22,23,24,25 zu analysieren. Dies kann auf einen dreidimensionalen Biofilm angewendet werden, indem 2D-Optikscheiben in verschiedenen Tiefen im Biofilm26 untersucht werden. Die aktuelle Technik wurde entwickelt, um 1 μm mikroskalige Kügelchen in 3D über die Zeit zu verfolgen und sie in der 4D-Modellierung zu verwenden. Ein Teil der Begründung war das übergeordnete Konzept, die 4D-Modellierung zu nutzen, um die Bewegung von Plasmiden durch gastrointestinale Mikrobiota-Gemeinschaften zu verstehen. Es wurden fluoreszierend geladene Carboxylatkügelchen mit einem Durchmesser von 1 μm verwendet, da diese in Bezug auf Größe und Ladung gut mit E. faecalis, dem gewählten Modellorganismus für die Bewegung und Aufrechterhaltung von Plasmiden, übereinstimmen27, 28. Es wurde ein 4D-Assay zur Quantifizierung der physikalischen Eigenschaften von Biofilmen entwickelt (Abbildung 1A). In der entwickelten Methode wurden Beads zu Biofilmen hinzugefügt und ihre räumlich-zeitlichen Trajektorien durch 10-20 μm dicke Biofilme über einen Zeitraum von 10-20 Minuten aufgezeichnet. Die Trajektorien der Raute in 3D wurden dann unter Verwendung einer neu entwickelten Toolbox in Bezug auf die Länge der Trajektorie, die Raupengeschwindigkeit, das Volumen des Bounding-Box-Rings (die minimale Box, die die Trajektorie enthält) und die zelluläre Dichte des Bounding-Box-Bereichs quantifiziert. Das folgende Protokoll kann verwendet werden, um 4D-Bilddaten von Bakterien und kügelchenhaltigen Biofilmen zu generieren, die Daten mit ImageJ29 und dem Plugin Mosaic vorzuverarbeiten und die Trajektorien der Kügelchen mit einer Bead Evaluator-Toolbox zu analysieren.
Diese Technik hat mehrere Anwendungen zur Untersuchung von Materialeigenschaften sowie zur Verfolgung von Partikel- und Bakterienbewegungen in drei Dimensionen. Zum Beispiel wurde eine frühe Version dieser Technik verwendet, um die Wirkung von monoklonalen Antikörpern, die gegen Curli gerichtet sind, auf die strukturelle Integrität von Biofilmen zu charakterisieren16. Die Vollversion verfügt über mehrere Werkzeuge, um eine detailliertere Analyse der Materialeigenschaften von Biofilmen zu ermöglichen, und wird weiterhin verwendet, um die Auswirkungen der Behandlung mit monoklonalen Antikörpern auf Biofilme zu untersuchen. Partikel unterschiedlicher Ladung können verwendet werden, um die Materialladungseigenschaften der Biofilme und die Bewegung von Partikeln durch Biofilme mit unterschiedlichen Matrixzusammensetzungen zu untersuchen. Dies könnte verwendet werden, um die Ergebnisse der 2D-Mikrorheologie zu vergleichen, die die Materialeigenschaften aufzeigen, die für die Bewegung von Kügelchen verantwortlich sind, die wir in Biofilmen beobachtet haben, die nicht unter Fluss waren. Diese Technik könnte auch bei Biofilmen mit gemischten Spezies mit Regionen unterschiedlicher Biofilmzusammensetzung angewendet werden. Biofilme können in mikrofluidischen Geräten und Durchflusszellen live unter Strömung abgebildet werden, um Änderungen der Materialeigenschaften zwischen statischen und Strömungsbiofilmen sowie die Auswirkungen der Strömung auf die Bewegung von Partikeln zu untersuchen. Die Techniken können auch auf fluoreszenzmarkierte Bakterien angewendet werden, um die Bewegung exogener Bakterien durch eine Biofilmgemeinschaft zu charakterisieren. Unter Verwendung von drei Farben könnten fluoreszenzmarkierte Spenderbakterien, fluoreszenzmarkierte Empfängerbakterien und fluoreszenzmarkierte Plasmide verwendet werden, um die Bewegung, das Andocken und die Übertragung von Plasmiden zu verfolgen.
Protokoll
1. Biofilm-Vorbereitung
HINWEIS: Biofilme für die Analyse können mit jeder Methode gezüchtet werden, die es ermöglicht, dass sich der Biofilm auf einer optischen Glasoberfläche bildet. Die Biofilmstruktur sollte so gut an der optischen Oberfläche haften, dass die Struktur während der Wasch- und/oder Montageschritte des Protokolls nicht gestört wird. Im Folgenden wird die Technik für optische 96-Well-Bodenplatten und 12-mm-Glasdeckgläser in 24-Well-Platten beschrieben. Weitere Optionen sind unterschiedlich große optische Bodenplatten und optische Durchflusskammern mit und ohne Strömung.
- Aufbau von Biofilmen
- Geben Sie bakterielles Wachstumsmedium in die Vertiefungen der Platte. Für diese Studie, für E. fügen Sie 2 mL Todd-Hewitt (TH) zu einer 24-Well-Platte hinzu und fügen Sie 0,4 mL TH zu den unteren 96-Well-Platten der Optik hinzu. Für S. Typhimurium, E. coli und die isogenen csgAB-Curli-Mutanten geben 0,7 ml salzfreie Luria-Bouillon (LB) in die Vertiefungen. Wenn Sie optische Bodenkammern verwenden, fahren Sie mit Schritt 1.2 fort.
- Legen Sie 12 mm #1,5 Glasoptik-Deckgläser in eine Petrischale und bedecken Sie sie mit Ethanol.
- Entfernen Sie mit einer Pinzette das Deckglas und zünden Sie den restlichen Alkohol mit einer Flamme an. Lass den Alkohol abbrennen. Benutze einfach die Flamme, um sie in Brand zu setzen; Halten Sie das Deckglas nicht in die Flamme, da es sonst reißt. Lassen Sie das Deckglas 10-20 s abkühlen, bevor Sie es in die Vertiefung legen, um Risse zu vermeiden.
- Legen Sie das Deckglas schräg in die Vertiefung, in der sich das Medium befindet, um zu verhindern, dass es auf dem Medium liegt. Geben Sie das Deckglas nicht in eine trockene Mulde und fügen Sie dann Medium hinzu, da dies dazu führt, dass das Deckglas am Boden der Mulde kleben bleibt.
- Schieben Sie das Deckglas mit einer sterilen Pipettenspitze vorsichtig auf den Boden der Vertiefung mit dem Medium.
HINWEIS: Denken Sie daran, die Dicke (#1 oder #1,5) des Deckglases oder der optischen Bodenplatte an die Dicke der konfokalen Mikroskopoptik anzupassen.
- Inkubieren Sie den Biofilm unter geeigneten Bedingungen für das Biofilmwachstum. In diesen Studien züchten Sie E . faecalis-Biofilme als statische Kulturen bei 37 °C und E . coli-Biofilme aerob bei 30 °C.
- In diesen Studien züchteten Sie 2 Tage lang E . faecalis-Biofilme mit Mediumwechseln am Morgen und frühen Abend. Um eine Beschädigung oder Verdrängung des Biofilms zu vermeiden, neigen Sie die Platte vorsichtig ab. Platzieren Sie die Pipettenspitze in der Nähe des unteren Randes der Vertiefung und ziehen Sie das Medium langsam heraus. Fügen Sie den ersten ml frisches Medium auf die gleiche Weise hinzu. Geben Sie den zweiten ml langsam in der Nähe der Schnittstelle zwischen Medium und Vertiefung hinzu.
- Verwenden Sie in diesen Studien die Wachstumsbedingungen für eine optimale Lockenproduktion. Grow S. Typhimurium-Biofilme wurden 6-8 Tage lang bei 28 °C in einer Schräge inkubiert, die es dem Biofilm ermöglichte, sich etwa 2/3 des Weges nach oben des Objektträgers anzuheften und dann als Pellikel an der Luft-Flüssigkeits-Grenzfläche zu wachsen. Dies geschah ohne einen Mediumwechsel. Um ein Austrocknen des Mediums zu verhindern, stellen Sie die 24-Well-Platte in eine Kammer mit einer Wasserschale.
2. 4D-Bildgebung
- Vorbereitung der Biofilm-Halterung
- Verdünnen Sie Crimson 1 μm Carboxylat FluoSpheres Kügelchen 1:50 in PBS (2 x 107 Kügelchen in 1 mL PBS). Bei Verwendung von optischen Bodenkammern ist Syto9 in einer Verdünnung von 1 μl auf 300 μl für die Bead-Zubereitung hinzuzufügen.
- (fakultativ) Waschen Sie den Biofilm, um Spuren von Wachstumsmedium zu entfernen, wenn das Wachstumsmedium eine Autofluoreszenz aufweist. Waschen Sie in diesen Experimenten zweimal mit 1 ml PBS, neigen Sie die Platte vorsichtig ab, platzieren Sie die Pipettenspitze in der Nähe des unteren Randes der Vertiefung und ziehen Sie das Medium langsam heraus. Fügen Sie PBS hinzu, indem Sie die Spitze in der Nähe der unteren Kante platzieren und die Vertiefung langsam füllen. Verwenden Sie diese Technik für optische Vertiefungen am Boden zusätzlich zu Deckgläsern in einer 24-Well-Platte.
- Entfernen Sie Medium oder PBS. Verdünnte Crimson-Kügelchen, die in Nummer 2.1.1 hergestellt wurden, werden dem Biofilm zugeführt. In diesen Studien fügen Sie 1 ml Kügelchen (2x107) zu den Deckgläsern und 0,2 ml Kügelchen (4x106) und Syto9 zu den optischen Bodenplatten mit 96 Vertiefungen hinzu.
- 1 Minute bei Raumtemperatur inkubieren, um die Assoziation der Kügelchen zu ermöglichen.
- Entfernen Sie die Kügelchen und waschen Sie den Biofilm vorsichtig einmal mit PBS, um nicht assoziierte Kügelchen zu entfernen. In diesen Studien wird das Deckglas vorsichtig mit 1 ml PBS gewaschen, die Vertiefung mit 1 ml PBS aufgefüllt und mit 2.1.6 fortgefahren. Waschen Sie den Biofilm für die optischen 96-Well-Bodenplatten vorsichtig mit 0,2 mL PBS und füllen Sie die Vertiefung mit 0,2 mL PBS auf. Die untere Kammer der Optik ist nun bereit für die Abbildung, fahren Sie also mit Schritt 2.2 fort.
- (Bei Verwendung eines Deckglases) Geben Sie 1 μl Syto9 (grün fluoreszierender DNA-Farbstoff; verdünnt gemäß den Anweisungen des Herstellers) in die Mitte einer Vertiefung auf einem beschichteten 10-Well-Multitest-Objektträger. Diese beschichteten Deckgläser haben Vertiefungstiefen von 23-25 μm.
- (Bei Verwendung eines Deckglases) Entfernen Sie mit einer mit Alkoholflamme sterilisierten Pinzette vorsichtig das Deckglas aus der Vertiefung und drehen Sie es auf die Vertiefung mit dem Syto9-Tropfen um. Wenn Sie das Deckglas in 1 ml PBS belassen, lässt es sich leichter aus der Vertiefung entfernen und hilft, nicht assoziierte Kügelchen aus dem Biofilm zu waschen.
- (Bei Verwendung eines Deckglases) Versiegeln Sie das Deckglas vorsichtig mit Fingernagellack, ohne auf das Deckglas zu rutschen oder zu drücken, da dies zu Oberflächenspannungen führen kann, die die Bewegung in viskoseren, weniger starren Biofilmen stoppen.
- (Bei Verwendung eines Deckglases) Lass den Nagellack trocknen. Wischen Sie die Außenfläche des Deckglases vorsichtig mit 70 % Ethanol ab. Wischen Sie aus den oben genannten Gründen ohne Druck auf das Deckglas auf.
- Konfokale Bildgebung
HINWEIS: Verwenden Sie in diesen Studien ein konfokales Laser-Scanning-Mikroskop mit inverser spektraler Bildgebung, das mit einem konfokalen TCS-System mit dem 63x-Objektiv ausgestattet ist. Das Oszilloskop wird verwendet, um ein 4D-Video zu generieren (Abbildung 1). Der 3D-Biofilm besteht aus Z-Scheiben, die in Schritten von 0,5 μm durch 18-20 μm dicke Biofilme eingefangen werden, wodurch 36-40 Z-Schichten erzeugt werden. Die Aufnahme eines 3D-Biofilms dauert 50-60 Sekunden. Die Scheiben bilden zusammen einen Rahmen, der als 3D-Biofilm visualisiert werden kann. Dieser Vorgang wird 20 Mal wiederholt, um das 4D-Zeitraffervideo für eine Gesamtaufnahmezeit von 18-20 Minuten zu erzeugen.- Stellen Sie das Zielfernrohr so ein, dass Fluorophore erfasst werden. In diesen Studien wird Syto9 (bakterielle DNA-Färbung) mit einem 488-nm-Laser angeregt und die Emission von 495 bis 540 nm gemessen (das Leica Sp5 ist ein spektrales Bildgebungsmikroskop). Stimulieren Sie purpurrote (rote) Kügelchen mit einem 633-nm-Laser und messen Sie die Emission von 650 nm bis 700 nm. Diese Fluorophor-Einstellungen können angepasst werden, um alle gewünschten Fluorophore einzufangen.
- Wählen Sie den xyzt-Imaging-Modus.
- Identifizieren Sie einen Bereich des Biofilms mit einer Mischung aus Bereichen mit höherer und niedrigerer Dichte, um Unterschiede in den viskoelastischen Eigenschaften in dicken und dünnen Bereichen des Biofilms zu erfassen.
- Stellen Sie einen Z-Stapel mit einer Dicke von 18-20 μm ein. Vermeiden Sie bei einem auf Deckglas montierten Biofilm, dass die Ober- und Unterseite des Biofilms mit dem Glas in Berührung kommen, um Artefakte durch Perleneinschlüsse zu vermeiden (siehe Diskussion).
- Passen Sie die Verstärkung und den Offset an, um den gesamten Dynamikbereich der Intensität an der hellsten Stelle des Biofilms zu nutzen. Dadurch wird die Signalüberlappung aus den unteren Schichten des Biofilms minimiert.
- Stellen Sie die Schichtdicke auf 0,5 μm ein. Dies ermöglicht eine schnelle Bildgebung ohne Verlust von Bead-Informationen.
- Stellen Sie die Auflösung auf 512 x 512 (0,48 μm) ein. Dies ermöglicht eine schnelle Bildgebung und erzeugt dennoch Bilder mit ausreichender Auflösung, um die Biofilmstruktur und die Details der Bead-Bewegung zu erkennen.
- Minimieren Sie die Bildgebungszeit.
- Legen Sie fest, dass 20 Stapel erfasst werden.
- Speichern Sie als .lif (oder eine ähnliche konfokale Datei). Der 4D-Film kann in ImageJ erzeugt werden.
3. Generieren des 4D-Biofilmvideos mit ImageJ
- Öffnen Sie die .lif-Datei in ImageJ mit folgenden Einstellungen: Stapel mit Hyperstack anzeigen, Stapelreihenfolge XYCZT, Farbmodus: farbig, Autoskalierung aktiviert. Drücken Sie dann OK.
- Wählen Sie Bild > Farbe > Kanäle teilen.
- Wählen Sie Bild > Farbe > Kanäle zusammenführen > Komposition erstellen > OK.
- Wählen Sie Plugins > 3D-Viewer aus > Kanäle, deaktivieren Sie das blaue Häkchen > OK.
- Wählen Sie Bearbeiten > Inhalt anzeigen aus > Kontrollkästchen Begrenzung anzeigen aktiviert.
- Verwenden Sie den Mausklick und halten Sie gedrückt, um das Bild auszuwählen. Drehen Sie dann das Bild, während Sie die Klicktaste gedrückt halten. Drehen Sie das Bild so, dass sich die Unterseite des Biofilms auf der Unterseite befindet und der Winkel die Visualisierung der Kügelchen unterstützt. Lassen Sie dann das Bild los.
- Drücken Sie die rote Aufnahmetaste am unteren Rand des Fensters, um ein Video aufzunehmen.
- Speichern Sie als .avi-Datei mit jpeg als Komprimierung.
4. Generieren von Trajektoriendaten
- Installieren Sie das Open-Source-Tool ImageJ (https://imagej.net/Fiji) und das Partikelverfolgungs-Plugin Mosaic (https://imagej.net/MOSAICsuite).
- Importieren Sie die .lif-Datei, die die beiden Kanäle für Kügelchen und Bakterien enthält. Teilen Sie die Kanäle auf und speichern Sie die Dateien separat.
- Speichern Sie in ImageJ die Bildvoxelgröße (x-, y- und z-Dimensionen) und die Größe des Zeitschritts in einer Textdatei.
- Gehen Sie in ImageJ zu Plugins | Mosaikieren Sie und starten Sie den Partikel-Tracker 2D/3D.
- Geben Sie die folgenden Parameter ein: Radius: 3, Cutoff: 0.003, Per/Abs: 0.12, Link Range: 2, Displacement: 10.00 mit der auf Brownian eingestellten Dynamik. Bitte sehen Sie sich die Ergebnisse für die Begründung für die Wahl der Brownschen Partei in diesen Experimenten an.
- Generieren Sie Trajektorien und exportieren Sie die Trajektorienliste als .csv Dateien.
5. Analysieren von Trajektorien
- VRL Studio installieren (https://vrl-studio.mihosoft.eu)
- Biofilm-Projekt (https:// neurobox3d.github.io/Biofilm/) herunterladen und in VRL Studio starten
- Laden Sie die Trajektoriendatei in ImportData
- Geben Sie die X-, Y- und Z-Pixelgröße an (verwenden Sie ImageJ, um diese Werte zu finden) in ProcessTrajectories.
- Geben Sie das Frame-Intervall in ComputeVelocity an (verwenden Sie ImageJ, um diesen Wert zu finden).
- Laden Sie die bakterielle tiff-Datei (siehe Schritt 3.2) in Comdensity.
- Legen Sie Ausgabepfade für Geschwindigkeitsdaten in SaveVelocityDataToFile und Trajektoriendaten in SaveTrajectoryDataToFile fest.
- Rufen Sie SaveVelocityDataToFile und SaveTrajectoryDataToFile auf.
- Importieren Sie Daten zur Analyse in Excel. Zu diesen Daten gehören Trajektorienlängen, Trajektorienlebensspannen, Abmessungen und Volumina des Trajektorienbegrenzungsrahmens sowie durchschnittliche Sippengeschwindigkeiten und Varianzen. Die Analyse berechnet gewichtete Variablen unter Verwendung des Kanals für Syto9-markierte Bakterien, um die lokalen (innerhalb der gegebenen Trajektorien-Begrenzungsboxen) zellulären Dichten zu berechnen. Die Analyse führt zur Berechnung der gewichteten durchschnittlichen Geschwindigkeiten und Varianzen sowie der gewichteten Durchschnitte und Varianzen der Bounding-Box-Varianz.
Ergebnisse
Diese Methode wurde verwendet, um die Hypothese zu testen, dass Curli E . coli und S Steifigkeit verleihen können. Typhimurium-Biofilme, die die Bewegung der Kügelchen bei konfokalen Mikroskopie-Experimenten reduzieren. Die aktuelle Toolbox wurde verwendet, um die Materialeigenschaften des Enterococcus faecalis kommensalen Typs OG1RF mit dem Salmonella enterica Serotyp Typhimurium , E. coli und ihren jeweiligen isogenen Curli-Mutanten zu vergleichen (Abbildung 1B und ergänzendes Video 1, ergänzendes Video 2, ergänzendes Video 3, ergänzendes Video 4, ergänzendes Video 5, ergänzendes Video 6). Die Materialeigenschaften von Biofilmen könnten sich möglicherweise in Bezug auf die Steifigkeit (z. B. an eDNA gebundene Curli) oder elektrostatische und hydrophobe Wechselwirkungen zwischen den negativ geladenen Kügelchen und den Biofilmzellen und Matrixmaterialien sowie in Bezug auf die Zelldichte unterscheiden.
Reproduzierbarkeit
Die Biofilm-Toolbox wurde in Groovy30 und Java31 innerhalb des VRL-Studio32 programmiert und ermöglicht ein modulares Workflow-Design mit automatischer Generierung des User Interface (UI) aller Rechenkomponenten. Dies ermöglichte einen automatisierten Arbeitsablauf, der unbeabsichtigte Verzerrungen durch den Experimentator bei der Analyse der Ergebnisse beseitigte.
Verwendung von MSD zur Bestätigung der Art der Bewegung in den Biofilmen
Für die Analyse von Trajektorien mit Particle Tracker 2D/3D stehen verschiedene Dynamikeinstellungen für die Analyse verschiedener Raupenbewegungstypen zur Verfügung. Für diese Studien wurde die Einstellung "Brownsche Bewegung" (d.h. diffusionsgetriebene Bewegung) gewählt, da E. faecalis ein unbewegliches Bakterium ist, E. coli und Salmonella keine Flagellen in Biofilmen exprimieren und die Experimente in einem geschlossenen System ohne Strömung durchgeführt wurden. Diese Einstellung könnte durch die berechneten mittleren quadratischen Verschiebungen (MSD) der Perlen weiter validiert werden. Unter Verwendung der Definition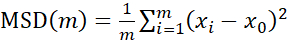 , bei der m die Anzahl der Trajektoriensegmente ist, kann die Änderung der MSD im Verlauf jeder Trajektorie berechnet werden. Lineare Trajektorien zeigen die Bewegung der diffusiven Perle an (Abbildung 2A). Unter Verwendung der quadratischen Anpassung der kleinsten Quadrate wurde das durchschnittliche Bewegungsmuster aller Kügelchen im Biofilm berechnet, wobei die dominante lineare Ordnung gezeigt und die passive Diffusion als treibende Kraft bestätigt wurde (Abbildung 2A-2F).
, bei der m die Anzahl der Trajektoriensegmente ist, kann die Änderung der MSD im Verlauf jeder Trajektorie berechnet werden. Lineare Trajektorien zeigen die Bewegung der diffusiven Perle an (Abbildung 2A). Unter Verwendung der quadratischen Anpassung der kleinsten Quadrate wurde das durchschnittliche Bewegungsmuster aller Kügelchen im Biofilm berechnet, wobei die dominante lineare Ordnung gezeigt und die passive Diffusion als treibende Kraft bestätigt wurde (Abbildung 2A-2F).
Analyse des Begrenzungsrahmens.
Die Toolbox verwendet ImageJ Mosaic und Particle Tracker 2D/3D, um Trajektorien zu generieren (Schritt 4) und generiert dann mithilfe der automatisierten Biofilmanalyse-Pipeline wichtige Daten über die Bead-Trajektorien, die zum Vergleich der Materialeigenschaften von Biofilmen verwendet werden können. Das Volumen des Begrenzungsrahmens inμm 3 wurde gemessen, indem der minimale Quader, der eine Trajektorie enthält, konstruiert und sein Volumen gemessen wurde (Abbildung 3).
E. faecalis-Biofilme weisen mit Bounding-Box-Werten von 1-6000 μm3 eine stärkere Bead-Bewegung auf (Abbildung 3B, 3C und 3D). Die Ergebnisse bestätigen, dass die Bewegung, die in einem Glasdeckglas zu sehen ist, das auf einem beschichteten Objektträger mit einer ca. 25 μm beschichteten Vertiefung montiert ist (Abbildung 3C), im Vergleich zu Biofilmen, die am Boden von optischen Glasvertiefungen gezüchtet und direkt abgebildet wurden (Abbildung 3D), gleichwertige Ergebnisse mit geringen Unterschieden liefert. Der einzige Unterschied bestand darin, dass im oberen Bereich des montierten Deckglases von E. faecalis-Biofilmen stabile Trajektorien mit einer Lebensdauer von mehr als 10 Minuten, aber gleichzeitig kleinen Begrenzungsrahmen registriert werden konnten, während in der optischen Bodenplatte eine ausgewählte Anzahl von Kügelchen mit höherer Beweglichkeit registriert werden konnte. Zusammengenommen deutet dies darauf hin, dass die Montage des Objektträgers die Oberflächenspannung des Systems an der Oberseite des Biofilms gegenüber dem Objektträger im montierten Deckglas-Biofilm verändert haben könnte, was letztendlich die Beweglichkeit einiger Kügelchen in weniger viskosen Biofilmbereichen verringerte (Abbildung 3B, 3C, 3D und 3I). Die Trajektorien, die in diese Kategorie fallen, waren zufällig ein sehr kleiner Prozentsatz, und selbst bei dieser geringen Anzahl von gefangenen Kügelchen war die durchschnittliche MSD von E. faecalis auf einem montierten Deckglas etwas höher als die MSD, die aus einem Biofilm der optischen Bodenplatte berechnet wurde (Abbildung 3).
S. Typhimurium- und E. coli-Bead-Trajektorien wiesen kleinere Bounding-Box-Volumina von 0-10 μm3 auf (Abbildung 3A, 3B, 3E und 3F), verglichen mit isogenen Curli-Mutanten mit Bounding-Boxen von 1-6000 μm3 für E. coli und 1-5000 μm3 für S. Typhimurium (Abbildung 3A, 3B, 3F und 3H), das eine größere Beweglichkeit der Kügelchen zeigt. Diese Ergebnisse deuteten darauf hin, dass das Vorhandensein des Amyloids mit einer erhöhten Steifigkeit in den Biofilmen korrelierte und mit dem Fehlen einer nennenswerten Biofilmbewegung in den Videos übereinstimmte. Die Bounding-Box-Volumina waren selbst in Bereichen mit geringer Dichte des Biofilms konstant klein (0-10μm 3). Diese Beobachtung stimmt mit früheren Beobachtungen überein, dass Curli in Regionen mit niedriger Zelldichte des Biofilms vorhanden sein können10.
Es war nicht möglich, das Verhalten von Enterobacteriaceae-Biofilmen auf optischen Bodenplatten zu vergleichen, da sie als Pellikel an der Luft-Flüssigkeits-Grenzfläche wachsen (Schritt 1.2.2). Bei der Verwendung eines Deckglases wird die Schale an der Schnittstelle mit dem Deckglas befestigt, und wenn das Deckglas entfernt wurde, legte sich die Schale auf das Deckglas, wodurch eine einzige Bildfläche entsteht. In einer optischen Bodenplatte, die schräg gezüchtet wurde, wurde die Bildgebung mit Flüssigkeit durchgeführt, die sich noch in der Vertiefung befand. Das bedeutet, dass das Furlikel immer noch über dem optischen Boden schwebt und das Furlikel aus der Arbeitstiefe eines invertierten Zielfernrohrs wie dem Leica Sp5 herausragt. Das Entfernen von genügend Medium, um den Biofilm in die Arbeitstiefe des Mikroskops zu bringen, führte dazu, dass die Probe während des 20-minütigen Bildgebungsprozesses austrocknete.
Insgesamt bestätigen die Diagramme die visuellen Beobachtungen in den ergänzenden Filmen und stimmen mit den beobachteten MSD-Unterschieden überein (Abbildung 3I und 3J).
Lebensdauer der Trajektorie
Die Lebensdauer der Trajektorie wurde als die Anzahl der aufeinanderfolgenden Frames gemessen, in denen eine Perle registriert wurde (Abbildung 3).
In den viskoseren, flüssigkeitsartigen E . faecalis-Biofilmen hatten alle Kügelchen eine Lebensdauer von weniger als 10 Minuten, und die meisten Trajektorien lagen zwischen 2 und 5 Minuten für E. faecalis-Biofilme . Kügelchen mit einer registrierten kurzen Lebensdauer konnten jedoch durch visuelle Inspektion in E . faecalis-Biofilmen über das gesamte Bildgebungszeitfenster lokalisiert werden (Ergänzendes Video 1 und 2). Somit ist es möglich, dass sich Kügelchen entlang einer registrierten Bahn bewegen, sich intermittierend von dem Biofilm lösen und eine Trajektorie beenden, und wieder mit dem Biofilm assoziieren, woraufhin eine neue Trajektorie initiiert wird. Dies würde letztendlich zu einer kurzen Lebensdauer bei kontinuierlicher Anwesenheit von Kügelchen im Biofilm führen. Es ist wichtig zu beachten, dass bei dieser Technik die Lebensdauer der Flugbahn, insbesondere in einem viskosen Biofilm, tendenziell die Gesamtzeit unterschätzt wird, die eine Perle mit dem Biofilm verbunden ist.
In S. Typhimurium-Biofilme, die kleinere Bounding-Box-Volumina aufwiesen, hatten die meisten Kügelchen (etwa 80%) eine lange Lebensdauer von 16-20 Frames, was etwa 15-20 Minuten in Echtzeit entspricht (Abbildung 3A, 3G und 3H). Im Gegensatz zu diesen trugen isogene Curli-mutierte Biofilme mobilere Kügelchen mit Bounding-Box-Volumina zwischen 1-6000 μm3 (E. coli) und 1-5000 μm3 (S. Typhimurium) (Abbildung 3A, 3B, 3F und 3H). Im Gegensatz zu E . faecalis-Biofilmen mit >70 % Trajektorien mit Bounding-Box-Volumina von mehr als 10μm 3 wiesen die Biofilme der Enterobacteriaceae-Spezies jedoch nur 30 % Bead-Trajektorien mit Bounding-Box-Volumina über 10 μm3 auf. Obwohl die Gesamtlebensdauer der Beads in Curli-mutierten Biofilmen geringer war, zeigten einige Trajektorien eine erhebliche Bead-Bewegung und eine lange Lebensdauer der Trajektorien (Abbildung 3H). Diese Beobachtung könnte darauf hindeuten, dass diese Variabilität mit unterschiedlichen Eigenschaften des Biofilmmaterials, wie z. B. der Viskoelastizität, und/oder Änderungen der Partikeloberflächenchemie, wie z. B. der Ladung, korrespondieren kann.
Analyse von Trajektorienlängen und -geschwindigkeiten der Raupe
Die Trajektorienlänge ist ein Maß für die von den Kügelchen zurückgelegte Strecke in μm. Diese Messung stimmt mit der Geschwindigkeit der Perlenbewegung in μm/s überein. In Übereinstimmung mit den größeren Bounding-Box-Volumina wiesen die Kügelchen in E . faecalis-Biofilmen 10-mal längere Trajektorien auf, 5-20 μm, im Vergleich zu <4 μm in Biofilmen, die Curli enthielten. In Übereinstimmung mit den kürzeren Trajektorien (Abbildung 4A). E. faecalis-Beads maßen bis zu 15-mal höhere Geschwindigkeiten, wobei die meisten Beads Geschwindigkeiten im Bereich von 0,01 bis 0,15 μm/s gegenüber Geschwindigkeiten <0,006 μm/s aufwiesen (Abbildung 4B). Nichtsdestotrotz maßen Curli-mutierte Biofilme insgesamt niedrigere Geschwindigkeiten und kürzere Trajektorien im Vergleich zu E. faecalis-Biofilmen , aber längere Trajektorien und höhere Geschwindigkeiten als die Curli-haltigen Elternstämme (Abbildung 4A und 4B).
Bemerkenswert ist die Tatsache, dass die fibrilläre gitterartige Struktur von curli30 die Beweglichkeit auf anisotrope Weise beeinflussen kann, wodurch die Bewegung in der xy-Ebene reduziert und eine erhöhte Beweglichkeit in z-Richtung ermöglicht wird (Abbildung 2G). Der große Trajektorienpool (ca. 800), der zu etwa 50 einzigartigen Kügelchen in lockenhaltigen Biofilmen gehört, würde mit den Einschränkungen des Mosaic Particle Tracking übereinstimmen, indem jede dieser sich schnell bewegenden Kügelchen als eine einzelne Perle in x, y und z gezählt wird. Zusätzliche Forschung und Softwareentwicklung werden notwendig sein, um diese Beobachtung zu bestätigen.
Analyse der Abhängigkeit der Kügelchenbewegung von der Zelldichte
Die Abhängigkeit der Bead-Bewegung von der zellulären Dichte wurde unter Verwendung von gewichteten durchschnittlichen Geschwindigkeiten und Varianzen sowie von Durchschnitten/gewichteten Durchschnitten und Varianzen der Bounding-Box-Volumina bestimmt. Der zweite Bildgebungskanal für Syto9-markierte Bakterien wurde verwendet, um die lokalen zellulären Dichten bei der Berechnung der gewichteten Geschwindigkeiten zu berechnen. Die zelluläre Dichte wurde berechnet, indem die Syto9-Voxeldaten über den Begrenzungsrahmen jeder Trajektorienkante gemittelt wurden (Abbildung 5, rechts). Somit kann die Raupengeschwindigkeit durch die kantenweisen (lokalen) Zelldichten gewichtet werden. Es gibt mehrere Arten von Färbungen, die zur Visualisierung von Bakterien verwendet werden können, einschließlich Färbungen für die Zellwand, die Membranen und den DNA-Gehalt. Zur Bestimmung der zellulären Dichte wurde Syto9 gewählt, da es das konsistenteste Signal liefert, unabhängig davon, welche optische Z-Schicht visualisiert wird. Hüllflecken (Zellwand und Membran) geben je nach Position der Z-Schicht ein anderes Signal. Wenn die Z-Schicht den oberen oder unteren Rand der Zelle umfasst, ist das Signal stärker, als wenn die Z-Schicht durch die Mitte der Zelle verläuft, wo nur der Umriss der Zelle gefärbt ist.
Die Raupentrajektorien aus dem roten Kanal wurden über 20 Frames verfolgt, wobei die einzelnen Trajektorien eine minimale Lebensdauer von 2 Frames und maximal 20 Frames mit 19 Trajektoriensegmenten, die die Frames verbinden, hatten (Abbildung 5). Um die Zelldichteabhängigkeit der Bead-Mobilität zu untersuchen, wurde die GFP-Intensität pro Voxel (jede der Einzelmessungen im 512x512 Voxel-Bild) bestimmt. Die Zelldichte um jedes Segment der Bead-Trajektorie wurde als lokal gemittelte Dichte im Begrenzungsrahmen des Segments berechnet.
Für einige Biofilme konnte eine statistisch signifikante Dichteabhängigkeit dokumentiert werden (Abbildung 6), vor allem für E . faecalis-Biofilme , die auf einem Glasdeckglas gezüchtet und auf einen Multiwell-Objektträger invertiert wurden (Abbildung 6A). Im Gegensatz dazu zeigten E . faecalis-Biofilme , die auf dem Boden einer 96-Well-Platte gezüchtet wurden (Abbildung 6B), keine Dichteabhängigkeit. Zusammenfassend deutet dies darauf hin, dass hochflüssige E. faecalis-Biofilme aufgrund der Montage auf einem Multi-Well-Objektträger möglicherweise leicht komprimiert werden könnten, was mit einer Verringerung der Anzahl der sich schneller bewegenden Kügelchen und derjenigen mit kleinen Bounding-Box-Volumina, die an der Oberseite des Biofilms gegen den Glasobjektträger eingeschlossen sind, konsistent ist (Abbildung 3C vs. Abbildung 3D). Sowohl die Salmonellen - als auch die E . coli-Biofilme (Abbildung 6C und 6D) und ihre isogenen Mutanten (Abbildung 6E und 6F) zeigten eine geringe bis keine Abhängigkeit von der zellulären Dichte.
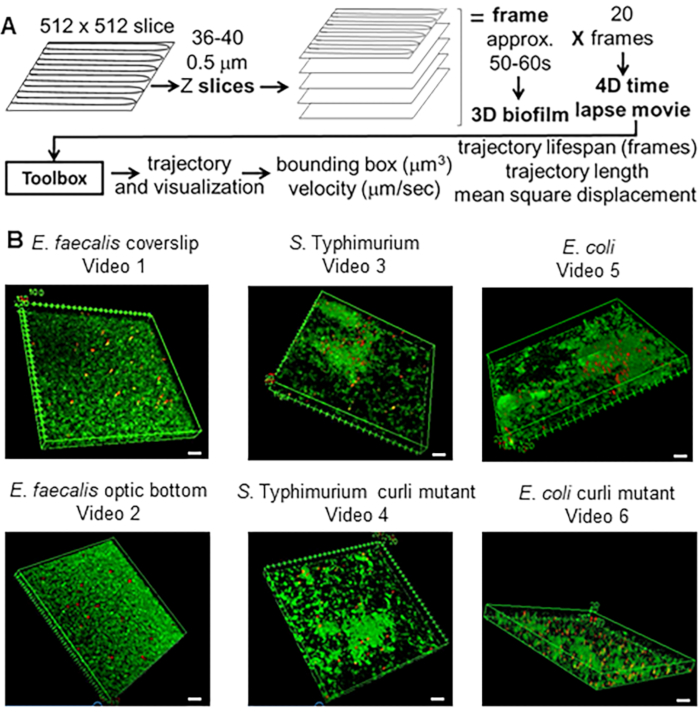
Abbildung 1. Bildgebungs- und Analysepipeline (Schritte 2-4) (A) Biofilme werden wie in 2.2 beschrieben abgebildet. Mit Hilfe der abgebildeten Biofilme (siehe 3) wurden Bead-Trajektorien erzeugt, wie in 4 beschrieben. Anhand der Trajektorien wurden relevante Daten mit der Analyse-Toolbox berechnet (siehe 5) (B) Biofilme wurden wie in Schritt 1 beschrieben auf Deckgläsern gezüchtet (E. faecalis, Video 1), S. Typhimurium (Video 3), E. coli (Video 5) und isogene Curli-Mutanten (Video 4, Video 6) oder in einer 96-Well-Platte mit optischem Boden (E. faecalis Boden, Video 2). Die weiße Maßstabsleiste ist 20 mm breit. Diese Abbildung wird mit Genehmigung von (31) übernommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
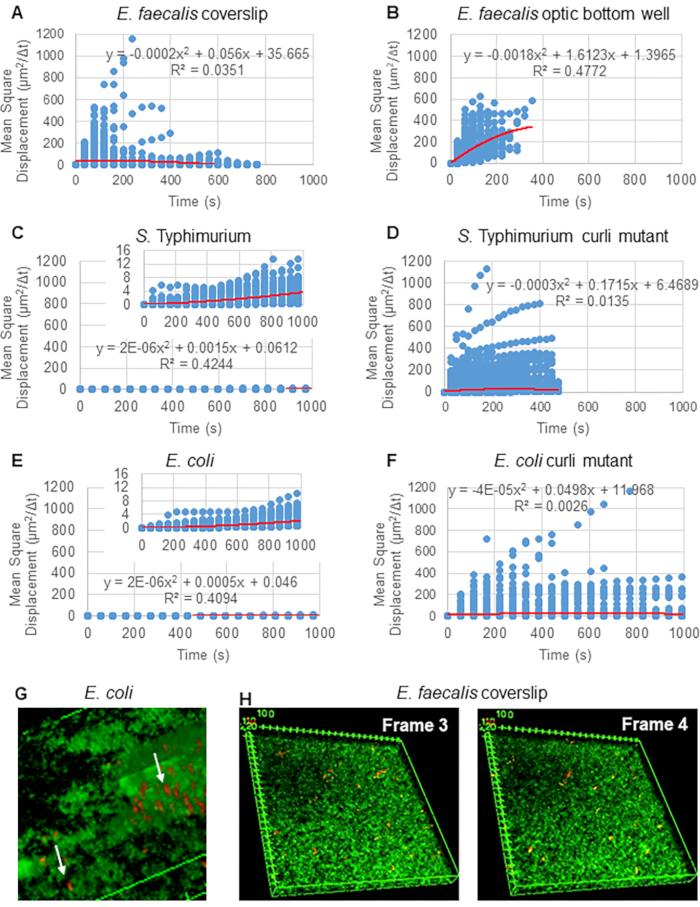
Abbildung 2. Es wurde festgestellt, dass die Bead-Bewegung in den Biofilmen diffusiv ist (Brownsche Bewegung, siehe Schritt 5) (A-F) MSD-Daten zeigen ein lineares Verhalten (rote Linie), um die Brownsche Bewegung zu validieren. Biofilme wurden gemäß Schritt 1 gezüchtet (G) Beispiel für eine elliptische Kügelchenbewegung, die in E . coli - und S. Typhimurium-Biofilmen beobachtet wurde, die aus einem Frame des E. coli 4D-Biofilm-Assays entnommen wurden (H) Beispiel für große Veränderungen der Bead-Muster zwischen Frame 3 und 4, die aus der optischen Bodenvertiefung von E. faecalis entnommen wurden. Beachten Sie, dass der Biofilm selbst einen gewissen Fluss hervorruft (Video 1 und 2), was den Anschein erweckt, dass Bild 3 und 4 unterschiedlich ausgerichtet sind. Diese Abbildung wird mit Genehmigung von (31) übernommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
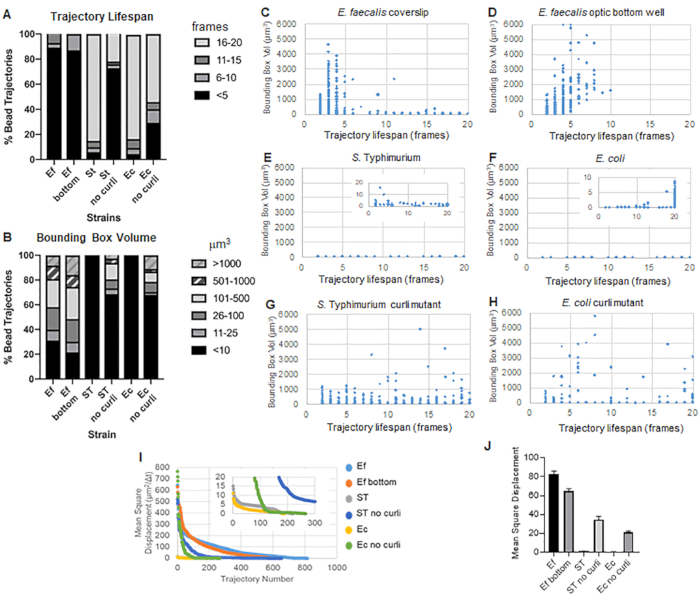
Abbildung 3. Analyse von Unterschieden in der Steifigkeit unter Verwendung von Bounding Boxes und Trajektorienlebensdauer. Biofilme wurden wie in Schritt 1 beschrieben auf Deckgläsern gezüchtet (E. faecalis, Video 1), S. Typhimurium (Video 3), E. coli (Video 5) und isogene Curli-Mutanten (Video 4, Video 6) oder in einer 96-Well-Platte mit optischem Boden (E. faecalis Boden, Video 2). Die Lebensdauer der Trajektorien wird in % der gesamten Bead-Trajektorien (A) und der Scatter-Graphen (C-H) zusammen mit den Bounding-Box-Volumina (berechnet in Schritt 5) dargestellt. (I) Vergleich der Bead-MSDs in den verschiedenen Biofilmen (H) Durchschnittliche MSDs jeder Art von Biofilm. Die Balken geben das 95 %-Konfidenzintervall der Daten an. Diese Abbildung wird mit Genehmigung von (31) übernommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
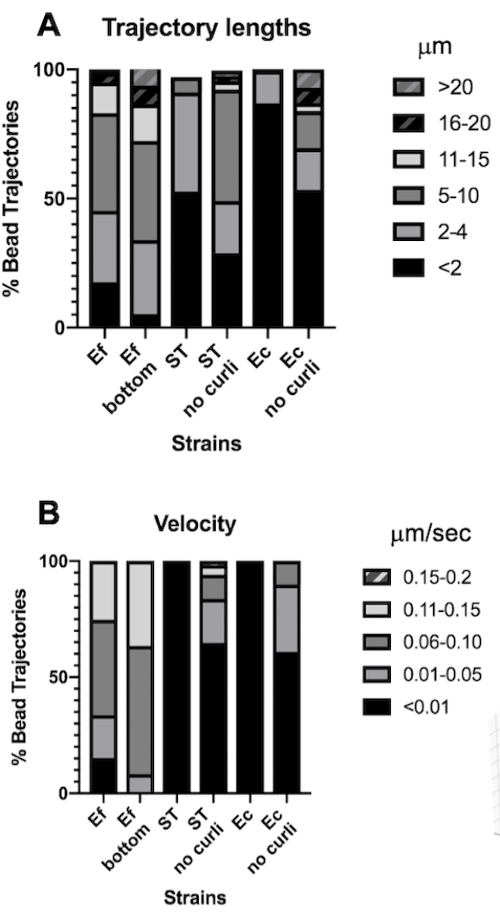
Abbildung 4. Analyse von Unterschieden in der Trajektorienlänge und der Raupengeschwindigkeit. Es wurden Biofilme gezüchtet, wie in Schritt 1 beschrieben. Die Trajektorienlängen werden in μm angegeben und in % der gesamten Bead-Trajektorien (A) dargestellt. Die Geschwindigkeit wird in μm/s angegeben und in % der gesamten Bead-Trajektorien (B) dargestellt. Diese Abbildung wurde mit Genehmigung von (31) angepasst. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
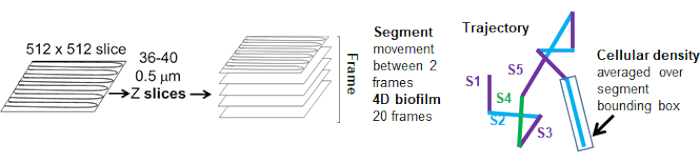
Abbildung 5. Überblick über die Analyse von Trajektoriensegmenten. Diese Abbildung wurde mit Genehmigung von (31) angepasst. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
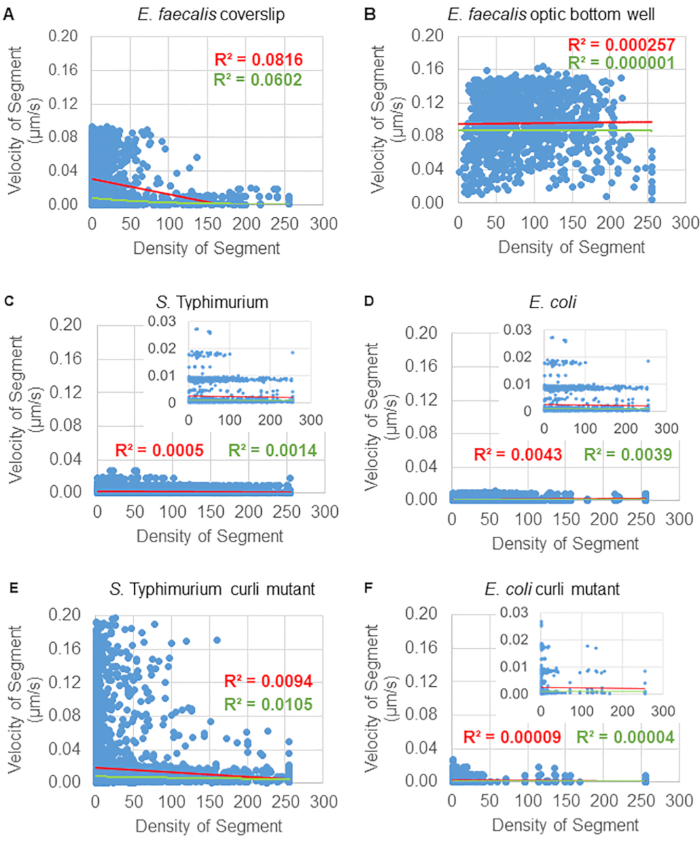
Abbildung 6. Untersuchung des Einflusses der Biofilmdichte auf die Raupengeschwindigkeit mit Hilfe der Biofilmanalyse-Pipeline (Schritt 5). Die Ergebnisse zeigten, dass die Raupengeschwindigkeit nicht ausschließlich von der Zelldichte abhängt. Es wurden Biofilme gezüchtet, wie in Schritt 1 beschrieben. Die Trajektorien wurden auf der Skala einzelner Trajektoriensegmente analysiert (Abbildung 5). Für jedes Segment wurde die Bead-Geschwindigkeit in μm/s gegen die zelluläre Dichte des Bounding-Box-Moduls aufgetragen (durchschnittliches GFP pro Voxel innerhalb des Bounding-Box). Die rote Linie zeigt die lineare Regression und die grüne Linie die Ablaufverfolgung der exponentiellen Regression. Diese Abbildung wird mit Genehmigung von (31) übernommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzendes Video 1. 4D-Video eines 24-Stunden-Biofilms von E. faecalis OG1RF, der auf einem 1,5 dicken Deckglas aus optischem Glas gewachsen ist. Das 4D-Zeitraffervideo wurde mit einem Mikroskop mit einer Auflösung von 512x512 erstellt. Eine Z-Serie von 40 Bildern wurde erzeugt, indem ein etwa 20 μm dicker Bereich eines Biofilms in 0,5 μm-Schritten abgebildet wurde. Jede Z-Serie bestand aus einem Bild und benötigte 50-60 s für die Aufnahme. Eine Reihe von 20 zusammenhängenden Bildern wurde aufgenommen, um ein 4D-Video zu erstellen. Die Videowiedergabe erfolgt mit ca. 120x. Das Video ist repräsentativ für mindestens 6 unabhängige Experimente. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Ergänzendes Video 2. 4D-Video eines 24-Stunden-Biofilms von E. faecalis OG1RF, der auf einer optischen 96-Well-Bodenplatte gewachsen ist. Das 4D-Zeitraffervideo wurde mit einem Mikroskop mit einer Auflösung von 512x512 erstellt. Eine Z-Serie von 40 Bildern wurde erzeugt, indem ein etwa 20 μm dicker Bereich eines Biofilms in 0,5 μm-Schritten abgebildet wurde. Jede Z-Serie bestand aus einem Bild und benötigte 50-60 s für die Aufnahme. Eine Reihe von 20 zusammenhängenden Bildern wurde aufgenommen, um ein 4D-Video zu erstellen. Die Videowiedergabe erfolgt mit ca. 120x. Das Video steht stellvertretend für 3 unabhängige Experimente.Bitte klicken Sie hier, um es herunterzuladen Video.
Ergänzendes Video 3. 4D-Video eines Salmonella enterica Serotyps Typhimurium Biofilm ATCC 14028, der 6-7 Tage lang auf 1,5 dicken Deckgläsern aus optischem Glas gezüchtet wurde. Das 4D-Zeitraffervideo wurde mit einem Mikroskop mit einer Auflösung von 512x512 erstellt. Eine Z-Serie von 40 Bildern wurde erzeugt, indem ein etwa 20 μm dicker Bereich eines Biofilms in 0,5 μm-Schritten abgebildet wurde. Jede Z-Serie bestand aus einem Bild und benötigte 50-60 s für die Aufnahme. Eine Reihe von 20 zusammenhängenden Bildern wurde aufgenommen, um ein 4D-Video zu erstellen. Die Videowiedergabe erfolgt mit ca. 120x. Das Video steht stellvertretend für 3 unabhängige Experimente. Bitte klicken Sie hier, um es herunterzuladen Video.
Ergänzendes Video 4. 4D-Video der Salmonella enterica Serotyp-Typhimurium-Biofilme ATCC 14028 curli (csgBA) Mutanten wurden 6-7 Tage lang auf 1,5 dicken Deckgläsern aus optischem Glas gezüchtet. Das 4D-Zeitraffervideo wurde mit einem Mikroskop mit einer Auflösung von 512x512 erstellt. Eine Z-Serie von 40 Bildern wurde erzeugt, indem ein etwa 20 μm dicker Bereich eines Biofilms in 0,5 μm-Schritten abgebildet wurde. Jede Z-Serie bestand aus einem Bild und benötigte 50-60 s für die Aufnahme. Eine Reihe von 20 zusammenhängenden Bildern wurde aufgenommen, um ein 4D-Video zu erstellen. Die Videowiedergabe erfolgt mit ca. 120x. Das Video steht stellvertretend für 3 unabhängige Experimente.Bitte klicken Sie hier, um es herunterzuladen Video.
Ergänzendes Video 5. 4D-Video von E. coli UTI89, gezüchtet auf 1,5 dicken Deckgläsern aus optischem Glas für 6-7 Tage. Das 4D-Zeitraffervideo wurde mit einem Mikroskop mit einer Auflösung von 512x512 erstellt. Eine Z-Serie von 40 Bildern wurde erzeugt, indem ein etwa 20 μm dicker Bereich eines Biofilms in 0,5 μm-Schritten abgebildet wurde. Jede Z-Serie bestand aus einem Bild und benötigte 50-60 s für die Aufnahme. Eine Reihe von 20 zusammenhängenden Bildern wurde aufgenommen, um ein 4D-Video zu erstellen. Die Videowiedergabe erfolgt mit ca. 120x. Das Video steht stellvertretend für 3 unabhängige Experimente.Bitte klicken Sie hier, um es herunterzuladen Video.
Ergänzendes Video 6. 4D-Video E. coli UTI89 curli (csgBA) Mutante, die 6-7 Tage lang auf 1,5 dicken Deckgläsern aus optischem Glas gezüchtet wurde. Das 4D-Zeitraffervideo wurde mit einem Mikroskop mit einer Auflösung von 512x512 erstellt. Eine Z-Serie von 40 Bildern wurde erzeugt, indem ein etwa 20 μm dicker Bereich eines Biofilms in 0,5 μm-Schritten abgebildet wurde. Jede Z-Serie bestand aus einem Bild und benötigte 50-60 s für die Aufnahme. Eine Reihe von 20 zusammenhängenden Bildern wurde aufgenommen, um ein 4D-Video zu erstellen. Die Videowiedergabe erfolgt mit ca. 120x. Das Video steht stellvertretend für 3 unabhängige Experimente.Bitte klicken Sie hier, um es herunterzuladen Video.
Diskussion
Kritische Schritte und Fehlerbehebung
Die größte Herausforderung bei dieser Technik besteht darin, ein aufgezogenes Deckglas mit einem sehr viskosen Biofilm wie E. faecalis zu verwenden.Das Deckglas muss sorgfältig und präzise auf dem Multiwell-Objektträger platziert werden, ohne es neu zu positionieren. Während des Versiegelungsschritts muss darauf geachtet werden, dass das Deckglas nicht nach unten gedrückt oder versehentlich über die Gleitfläche geschoben/geschoben wird. Jede Bewegung oder jeder Druck kann eine Oberflächenspannung erzeugen und die Bewegung eines viskosen Biofilms blockieren. Wenn möglich, ermöglicht der Vergleich der Materialeigenschaften von Biofilmen durch Abbildung eines Biofilms auf einer optischen Vertiefung mit einer Deckglashalterung eine Beurteilung der Technik. Bei korrekter Ausführung ähnelte eine Deckglashalterung sehr stark einem Biofilm in einer optischen Bodenplatte für E. faecalis.
Darüber hinaus sollte bei der Verwendung eines gerahmten Deckglases eine Abbildung der Grenzflächen des Biofilms mit dem Deckglas unten oder dem Objektträger ganz oben vermieden werden. Bei Verwendung eines umgekehrten Zielfernrohrs mit dem Deckglas an der Unterseite können sich an der Basis des Biofilms Kügelchen gegen das Deckglas befinden. Diese Kügelchen passieren den Biofilm und bleiben auch nach schonendem Waschen am Deckglas hängen. Sie haben x-, y- und z-Koordinaten von 0 und Bounding-Box-Koordinaten von 0. Für bestimmte Anwendungen, wie z. B. die Untersuchung der Integrität von Biofilmen nach der Behandlung, können diese Datenpunkte jedoch als Werkzeug verwendet werden. Die Fähigkeit der Kügelchen, durch einen dicken Bereich des Biofilms bis zum Boden des Deckglases einzudringen, kann zur Beurteilung der Integrität des Biofilms nach der Behandlung verwendet werden (Manuskript in Vorbereitung in Zusammenarbeit mit dem Tükel-Labor). Am oberen Rand des Biofilms, in einem viskosen Biofilm wie E. faecalis , hatten wir einige Hinweise auf eine Verdichtung, die durch das Deckglas verursacht wurde. Dies schränkte die Bewegung einiger Kügelchen an der Grenzfläche des Glasschiebers ein und führte möglicherweise zu einer gewissen Dichteabhängigkeit bei der Analyse der Perlenbewegung.
Die Waschschritte waren für die Biofilme notwendig, da das Wachstumsmedium eine starke Autofluoreszenz im grünen Kanal aufweist. Wir haben uns dafür entschieden, überschüssige Kügelchen zu verwenden und nicht assoziierte Kügelchen durch Waschen zu entfernen, um die zugehörigen Kügelchen zu maximieren und die genaueste Charakterisierung der beobachteten Regionen zu erhalten.
Die Anzahl der Kügelchen und Waschungen, die benötigt werden, um die gewünschten Datensätze zu erhalten, muss empirisch bestimmt werden. Das Vorhandensein von zu vielen Kügelchen in einem Biofilm erzeugt unmöglich große Datensätze, die schwer zu analysieren sind. Das Vorhandensein von zu wenigen Kügelchen führt nicht zu einer gründlichen Beprobung der Biofilmumgebungen. Die Kontrolle der Anzahl der hinzugefügten Kügelchen (2x107 Kügelchen in 1 ml PBS) und die Verwendung von Waschschritten führten jedoch zu einer relativ konsistenten Anzahl von Kügelchen (40-140), die je nach Struktur, räumlicher Anordnung und Zusammensetzung mit dem Biofilm assoziiert waren.
Bei der Untersuchung von Biofilmen mit gemischten viskosen und starren Bereichen können sich die Kügelchen im Laufe der Zeit in den starren Bereichen festsetzen. In diesem Fall muss die Bildgebung sofort nach dem Hinzufügen der Kügelchen gestartet werden. Dies kann oft nicht mit Deckgläsern erreicht werden, sondern erfordert optische Bodenplatten oder Durchflusszellen, bei denen die Bildgebung unmittelbar nach der Zugabe der Kügelchen und dem/den Waschschritt(en) erfolgen kann.
Modifikationen und zukünftige Anwendungen
Verwendung von mikrofluidischen Geräten. In unseren Studien erforderten die optimalen Bedingungen für die Untersuchung der Enterobacteriaceae-Biofilme das Wachstum des Biofilms als Pellikel an der Luft-Flüssigkeits-Grenzfläche. Dies schränkte den Einsatz von optischen Bodenplatten und mikrofluidischen Geräten in den Studien ein. Wenn die Bedingungen für die Biofilmbildung es jedoch zulassen, können die Biofilme in mikrofluidischen Kammern oder Durchflusszellen gezüchtet werden. Die Biofilme konnten dann gewaschen und die Kügelchen mit minimaler Störung des Biofilms durch das mikrofluidische Gerät eingeführt werden.
Zugabe von Kügelchen während des Biofilmwachstums. Wir haben uns dafür entschieden, den Biofilmen überschüssige Kügelchen hinzuzufügen und dann nicht assoziierte Kügelchen durch schonendes Waschen zu entfernen, um die Anzahl der während der Analyse vorhandenen Kügelchen zu optimieren. In den viskosen E . faecalis-Biofilmen ist es möglich, dass sich die Kügelchen während der 20-minütigen Bildgebungszeit dissoziiert und wieder assoziiert haben. Wenn eine geringe Anzahl von Kügelchen zu unterschiedlichen Zeitpunkten während des Biofilmwachstums hinzugefügt wird, könnte es möglich sein, die Kügelchen im Biofilm einzufangen, was eine genauere Charakterisierung der Biofilmbewegung in viskoseren Biofilmen ermöglicht.
Auswahl der Region, die abgebildet werden soll. Für Untersuchungen zu Materialeigenschaften ist es am besten, dicke und dünne Bereiche des Biofilms zu wählen. Bei der Untersuchung von Änderungen der Materialeigenschaften eines behandelten Biofilms können jedoch dicke konfluente Bereiche abgebildet werden, um Änderungen der viskoelastischen Eigenschaften und der Raupenpenetration in diesen Bereichen zu bestimmen. In diesem Fall ist die Suche nach Kügelchen, die den Biofilm durchdrungen haben und am Deckglas eingeklemmt wurden, ein nützliches Maß für die Auflockerung des Biofilms.
Bildgebung unter Strömung. Mit optischen Glasdurchflusszellen oder mikrofluidischen Geräten kann die Bewegung von Kügelchen oder Bakterien in einem Biofilm unter Fluss abgebildet werden. Dies kann auf unterschiedliche Weise geschehen. Dies kann durch Injektion von Kügelchen in die gesamte Kammer erfolgen, gefolgt von einer kurzen Inkubation, um die Assoziation der Kügelchen mit dem Biofilm zu ermöglichen. Die nicht assoziierten Kügelchen können durch Waschen entfernt und der Biofilm mit oder ohne Durchfluss abgebildet werden. Umgekehrt kann eine kleine Anzahl von Kügelchen in eine Seite der Kammer eingebracht und ihre Bewegung durch und im Biofilm unter Strömung verfolgt werden. Bei der Verwendung von Flow ist bei der Auswahl der Einstellungen für die Verfolgung der Mosaikperlen Vorsicht geboten (Schritt 4.5). In den aktuellen Studien war die Dynamikeinstellung Brownsch. MSD-Berechnungen bestätigten, dass die Bewegung wahrscheinlich diffusiv sein würde, was die Brownsche Einstellung zur geeigneten Einstellung machte.
Matrix-Färbung. In den aktuellen Studien wird bei der Färbung mit Syto9 die zelluläre Dichte und nicht die Dichte der Biofilmstruktur untersucht. Zum Beispiel erhöht das Vorhandensein von Amyloiden wahrscheinlich die Dichte des Matrixmaterials des Biofilms. Die Abhängigkeit der Bewegung von der Amyloiddichte konnte durch die Verwendung von Fluoreszenzmatrixfärbungen anstelle von Syto9 bestimmt werden.
Fluoreszenzmarkierte Bakterien. Fluoreszenzmarkierte Bakterien können verwendet werden, um die Bewegung exogener Bakterien durch Biofilme (z. B. plasmidhaltige Bakterien) zu verfolgen. Die Herausforderung bei fluoreszenzmarkierten Bakterien, wie z. B. Enterokokken, besteht darin, dass sie Einzelzellen, Diplokokken und kurze Ketten bilden, was die genaue Verfolgung der Bakterien erschwert. Dieser Prozess wäre einfacher, wenn die Bakterien eine einzellige Morphologie aufweisen würden.
Begrenzungen
Einschränkungen bei der Visualisierung von Trajektorien und beim Stitching.
Eine Einschränkung der Methode ist die Visualisierung von Trajektorien und das Stitching. Rekonstruierte und analysierte Trajektorien bestehen aus x-, y- und z-Punktkoordinaten, wobei nachfolgende Punkte den linearen Weg zwischen diesen Punkten definieren. Die Visualisierung solcher stückweisen linearen Trajektorien kann mit verschiedenen Werkzeugen erreicht werden. Ein Ansatz bestand darin, Python- und Jupyter-Notebooks zusammen mit den Python-Plugins Pandas und Matplotlib zu verwenden. Obwohl es möglich war, einzelne bestehende Trajektorien in dem Artikel im Journal of Bacteriology zu visualisieren, in dem diese Technik ursprünglich veröffentlicht wurde34, gab es immer noch erhebliche Einschränkungen, die in zukünftigen Forschungen angegangen werden.
Derzeit ist die Anzahl der rekonstruierten Trajektorien größer als die Anzahl der Kügelchen im Biofilm, was bedeutet, dass mehrere Trajektorien einer Perle entsprechen können. Dies kann durch ein schwaches konfokales Signal in einem Frame verursacht werden, bei dem Mosaic eine Trajektorie beendet und eine zweite initiiert. Dies kann sich in mehreren kürzeren Trajektorien für eine Perle bemerkbar machen, insbesondere bei weniger viskosen Biofilmen. Eine weitere Ursache für die große Anzahl von Trajektorien ist das Fehlen von Trajektoriennähten. Insbesondere in den optischen Bottom-Well-Biofilmen von E. faecalis bleiben die Kügelchen während der Bildgebung visuell mit dem Biofilm assoziiert (Supplemental Video 2). Es gab jedoch keine Trajektorien, die länger als 10 waren, und über 90 % der Trajektorien hatten eine Lebensdauer von 5 Frames oder weniger (Abbildung 3D). Wenn die Software verwendet wird, um nur Trajektorien oberhalb einer definierten Länge zu analysieren (z. B. bei der Verfolgung von Zellen, die in der Lage sind, Plasmide zu übertragen), können kürzere Trajektorien automatisch aus dem Datensatz entfernt werden. Es gibt jedoch auch andere Zwecke, für die das Zusammenfügen der Trajektorien sehr wichtig sein kann. Schließlich könnte die Unfähigkeit, die schnelle Bewegung der Kügelchen als eine einzige Trajektorie zu verfolgen, aufgrund der schnellen Bewegung in Z-Richtung zu mehr Trajektorien in Enterobacteriaceae-Biofilmen führen, was zu elliptisch geformten Kügelchen führt (Abbildung 2G). Die Möglichkeit, Trajektorien zu nähen, die durch schnelle anisotrope Bewegung gestört werden, wird wichtig sein, um die Wirkung der Curli-Amyloid-Matrix bei Enterobacteriaceae zu untersuchen.
Bedeutung
Es wurde ein rechnerischer Workflow entwickelt, um die Trajektorien von Beads zu untersuchen und die Materialeigenschaften von 3D-Biofilmen zu vergleichen. Der Workflow ermöglicht es den Forschern, kritische Parameter zu identifizieren, die bei der computergestützten Modellierung der Fluiddynamik in heterogenen Biofilmen verwendet werden können. Mit Hilfe dieses Open-Source-Bead-Evaluators konnte die Wirkung von bakteriellen Amyloid-Curli auf die Materialeigenschaften untersucht werden, wobei eine erhöhte Steifigkeit der Biofilmmatrix durch Curli gezeigt wurde. In einem allgemeineren Kontext kann der Evaluator verwendet werden, um Veränderungen in der Biofilmstruktur zu untersuchen, die durch Biofilmbehandlung oder verschiedene Umweltbedingungen, wie z. B. Strömung, induziert werden. So wird das Tool beispielsweise verwendet, um in Zusammenarbeit mit dem Tükel-Labor (LKSOM Temple University) die Wirkung einer Behandlung mit monoklonalen Antikörpern auf die Störung von Biofilmstrukturen zu analysieren. Der Werkzeugkasten des Raupenauswerters ist vollständig anpassbar und modular erweiterbar, wobei VRL-Studio verwendet wird, um seine Funktionen weiter zu verbessern und zu erweitern.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Arbeit in den Laboren von GQ und BAB erhielt keine spezifische Förderung von Fördergebern im öffentlichen, kommerziellen oder gemeinnützigen Sektor. Die Autoren danken Isaac Klapper, Ph.D. (Department of Mathematics, Temple University) für hilfreiche Diskussionen und Çagla Tükel (Department of Microbiology and Immunology, Temple University) für ihre Expertise in der ersten Veröffentlichung dieser Technik.
Materialien
| Name | Company | Catalog Number | Comments |
| 96-well plates, No. 1.5 Uncoated Coverslip, 5 mm Glass Diameter | MatTek | P96G1.55F | |
| Fisherbrand Cover Glasses: Circles | Fisher Scientific | 12-293-232P | 1.5 optic glass coverslip |
| Invitrogen Syto 9 Green Fluorescent Nucleic Acid Stain | Invitrogen | S34854 | |
| Molecular Probes FluoSpheres Carboxylate-modified Microspheres, 1 um, crimson fluorescent (625/645) | Molecular Probes | F8816 |
Referenzen
- Huang, R., Li, M., Gregory, R. L. Bacterial interactions in dental biofilm. Virulence. 2, 435-444 (2011).
- Nadell, C. D., Drescher, K., Wingreen, N. S., Bassler, B. L. Extracellular matrix structure governs invasion resistance in bacterial biofilms. ISME Journal. 9, 1700-1709 (2015).
- Billings, N., Birjiniuk, A., Samad, T. S., Doyle, P. S., Ribbeck, K. Material properties of biofilms - key methods for understanding permeability and mechanics. Reports on Progress in Physics. 78, 036601 (2015).
- Araújo, G. R. d. e. S., Viana, N. B., Gómez, F., Pontes, B., Frases, S. The mechanical properties of microbial surfaces and biofilms. The Cell Surface. 5, 100028 (2019).
- Tallawi, M., Opitz, M., Lieleg, O. Modulation of the mechanical properties of bacterial biofilms in response to environmental challenges. Biomaterials Science. 5, 887-900 (2017).
- Tursi, S. A., Tükel, &. #. 1. 9. 9. ;. Curli-Containing Enteric Biofilms Inside and Out, Matrix Composition, Immune Recognition, and Disease Implications. Microbiology and Molecular Biology Reviews. 82, (2018).
- Dueholm, M. S., Albertsen, M., Otzen, D., Nielsen, P. H. Curli functional amyloid systems are phylogenetically widespread and display large diversity in operon and protein structure. PLoS One. 7 (12), 51274 (2012).
- Hung, C., et al. Escherichia coli biofilms have an organized and complex extracellular matrix structure. mBio. 4, 00645 (2013).
- Kikuchi, T., Mizunoe, Y., Takade, A., Naito, S., Yoshida, S. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells. Microbiology and Immunology. 49, 875-884 (2005).
- Gallo, P. M., et al. Amyloid-DNA Composites of Bacterial Biofilms Stimulate Autoimmunity. Immunity. 42, 1171-1184 (2015).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an architectural element in spatially structured Escherichia coli biofilms. Journal of Bacteriology. 195, 5540-5554 (2013).
- Oh, Y. J. Characterization of curli A production on living bacterial surfaces by scanning probe microscopy. Biophysical Journal. 103, 1666-1671 (2012).
- Lembré, P., Di Martino, P., Vendrely, C. Amyloid peptides derived from CsgA and FapC modify the viscoelastic properties of biofilm model matrices. Biofouling. 30, 415-426 (2014).
- Oh, Y. J., et al. Curli mediate bacterial adhesion to fibronectin via tensile multiple bonds. Scientific Reports. 6, 33909 (2016).
- Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D., Drescher, K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection. Nature Microbiology. 3, 26-31 (2018).
- Tursi, S. A., et al. Salmonella Typhimurium biofilm disruption by a human antibody that binds a pan-amyloid epitope on curli. Nature Communications. 11, 1007 (2020).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15 (8), 1007978 (2019).
- Taglialegna, A., et al. The biofilm-associated surface protein Esp of Enterococcus faecalis forms amyloid-like fibers. Npj Biofilms and Microbiomes. 6, 15 (2020).
- Gour, S., Kumar, V., Rana, M., Yadav, J. K. Pheromone peptide cOB1 from native Enterococcus faecalis forms amyloid-like structures, A new paradigm for peptide pheromones. Journal of Peptide Science. 25, 3178 (2019).
- Stoodley, P., Debeer, D., Lewandowski, Z. Liquid flow in biofilm systems. Applied and Environmental Microbiology. 60, 2711-2716 (1994).
- Birjiniuk, A., et al. Single particle tracking reveals spatial and dynamic organization of the E. coli biofilm matrix. New Journal of Physics. 16, 085014 (2014).
- Chew, S. C., et al. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio. 5, 01536 (2014).
- Cao, H., et al. Revealing region-specific biofilm viscoelastic properties by means of a micro-rheological approach. Npj Biofilms and Microbiomes. 2, 5 (2016).
- Galy, O., et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical Journal. 103, 1400-1408 (2012).
- Rogers, S. S., vander Walle, C., Waigh, T. A. Microrheology of bacterial biofilms in vitro, Staphylococcus aureus and Pseudomonas aeruginosa. Langmuir. 24, 13549-13555 (2008).
- Hart, J. W., Waigh, T. A., Lu, J. R., Roberts, I. S. Microrheology and Spatial Heterogeneity of Staphylococcus aureus Biofilms Modulated by Hydrodynamic Shear and Biofilm-Degrading Enzymes. Langmuir. 35 (9), 3553-3561 (2019).
- van Merode, A. E. J., van der Mei, H. C., Busscher, H. J., Krom, B. P. Influence of culture heterogeneity in cell surface charge on adhesion and biofilm formation by Enterococcus faecalis. Journal of Bacteriology. 188, 2421-2426 (2006).
- Tariq, M., Bruijs, C., Kok, J., Krom, B. P. Link between Culture Zeta Potential Homogeneity and Ebp in Enterococcus faecalis. Applied and Environmental Microbiology. 78, 2282-2288 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ, 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- . Oracle.com Available from: https://docs.oracle.com/en/java (2021)
- Hoffer, M., Poliwoda, C., Wittum, G. Visual reflection library, a framework for declarative GUI programming on the Java platform. Computing and Visualization in Science. 16, 181-192 (2013).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15, 1007978 (2019).
- Malhotra, K., et al. Development of a New Bead Movement-Based Computational Framework Shows that Bacterial Amyloid Curli Reduces Bead Mobility in Biofilms. Journal of Bacteriology. 202, 00253 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten