Method Article
Schnelle In-vivo-Fixierung und Isolierung translationaler Komplexe aus eukaryotischen Zellen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir stellen eine Technik zur schnellen Stabilisierung translationaler (Proteinbiosynthese) Komplexe mit Formaldehydvernetzung in lebenden Hefe- und Säugetierzellen vor. Der Ansatz ermöglicht die Sezierung transienter Zwischenprodukte und dynamischer RNA:Protein-Interaktionen. Die vernetzten Komplexe können in mehreren nachgelagerten Anwendungen eingesetzt werden, z. B. in Deep-Sequencing-basierten Profilierungsmethoden, Mikroskopie und Massenspektrometrie.
Zusammenfassung
Schnelle Reaktionen mit schneller Umverteilung von Messenger(m)RNA und Veränderungen der mRNA-Translation sind relevant für die laufende homöostatische Anpassung der Zellen. Diese Anpassungen sind entscheidend für die Überlebensfähigkeit eukaryotischer Zellen und die "Schadenskontrolle" bei schwankenden Nährstoff- und Salzgehaltswerten, Temperaturen und verschiedenen chemischen und Strahlungsbelastungen. Aufgrund der hochdynamischen Natur der RNA-Level-Antworten und der Instabilität vieler RNA:RNA- und RNA:Protein-Zwischenprodukte ist es nur mit einer begrenzten Anzahl von Methoden möglich, eine aussagekräftige Momentaufnahme des zytoplasmatischen RNA-Zustands zu erhalten. Transkriptom-weite, RNA-seq-basierte Ribosom-Profiling-Experimente gehören zu den informativsten Datenquellen für die Kontrolle der Translation. Das Fehlen einer einheitlichen RNA und einer RNA:Protein-Intermediärstabilisierung kann jedoch zu unterschiedlichen Verzerrungen führen, insbesondere in den schnelllebigen zellulären Antwortwegen. In diesem Artikel stellen wir ein detailliertes Protokoll der schnellen Fixierung zur Verfügung, das auf eukaryotische Zellen unterschiedlicher Permeabilität anwendbar ist, um die Stabilisierung von RNA und RNA:Protein-Intermediär zu unterstützen. Darüber hinaus liefern wir Beispiele für die Isolierung der stabilisierten RNA:Protein-Komplexe basierend auf ihrer Co-Sedimentation mit ribosomalen und poly(ribo)somalen Fraktionen. Das abgetrennte stabilisierte Material kann anschließend als Teil von Ribosomenprofilierungsexperimenten verwendet werden, z. B. im TCP-seq-Ansatz (Translation Complex Profile Sequencing) und seinen Derivaten. Die Vielseitigkeit der Methoden im TCP-seq-Stil wurde nun durch die Anwendungen in einer Vielzahl von Organismen und Zelltypen demonstriert. Die stabilisierten Komplexe können aufgrund der Leichtigkeit der Vernetzungsumkehr auch zusätzlich affinitätsgereinigt und elektronenmikroskopisch abgebildet, in verschiedene poly(ribo)somale Fraktionen getrennt und einer RNA-Sequenzierung unterzogen werden. Daher können Methoden, die auf Snap-Chilling und Formaldehyd-Fixierung basieren, gefolgt von der sedimentationsbasierten oder anderen Art der RNA:Protein-Komplex-Anreicherung, von besonderem Interesse sein, um feinere Details der schnellen RNA:Protein-Komplexdynamik in lebenden Zellen zu untersuchen.
Einleitung
Lebende Organismen unterliegen über ihre Lebensspanne dynamischen intra- und extrazellulären Veränderungen, die schnelle Reaktionen erfordern, um die Homöostase aufrechtzuerhalten und das Überleben zu sichern. Um die Anpassung an die Umwelt zu ermöglichen, passen eukaryotische Zellen ihren Stoffwechsel über die Genexpressionskontrolle an. Die Kontrolle der Genexpression kann während der Transkription und/oder Translation ausgeübt werden; wobei translationale Antworten im Allgemeinen schneller auftreten1,2,3,4. Zum Beispiel treten translationale Veränderungen typischerweise innerhalb von 1-30 Minuten nach Beginn des Stresses auf, während Veränderungen auf Transkriptionsebene Stunden nach der Stressexpositionfolgen 3,4,5. Veränderungen der Translationsleistung werden aufgrund der anhaltenden Verfügbarkeit von Boten(m)RNA-Molekülen im Zytoplasma schneller erreicht. Umgekehrt müssen auf der Transkriptionsebene neue mRNA-Moleküle synthetisiert und in Eukaryoten verarbeitet und aus dem Kern exportiert werden, was zu erheblichen Verzögerungen in der Reaktionszeitführt 2,4,6,7,8.
Akute translationale Reaktion auf Stress ist im Allgemeinen durch eine allgemeine Abnahme der Translationsleistung gekennzeichnet, wobei die selektive Hochregulierung von Proteinen für das Zellüberleben notwendig ist1,3,4,9. Es wird angenommen, dass die Verringerung der Proteinproduktion aufgrund des hohen Energieaufwands des Prozesses entscheidend ist3,7. Um die selektive Hemmung und Hochregulierung zu erleichtern, werden translationale Reaktionen durch eine Reihe komplexer Regulationsmechanismen bedient. Regulation kann über alle Phasen der Translation ausgeübt werden: Initiierung, Dehnung, Beendigung der Polypeptid-Biosynthese und ribosomales Recycling10,11,12,13, zeigt sich aber am stärksten in der Initiationsphase5,7,9,10,13. Während der Initiierung bindet die kleine ribosomale Untereinheit (SSU), unterstützt durch eukaryotische Initiationsfaktoren (eIFs), an die 5' untranslatierte Region (UTR) der mRNA und scannt sie, bis ein Startcodon erkannt wird2,5,6,8,11,12,13. Regulierungsmechanismen zielen häufig auf eIFs ab, die das Anhängen, Scannen und starten die Codonerkennung beeinflussen. Zum Beispiel der Initiationsfaktor eIF2, ein wesentlicher Translationsfaktor, der bei der Rekrutierung einer Initiator-Met-tRNA hilftiMet zur SSU, wird oft bei Eukaryoten unter Stressbedingungen ins Visier genommen4,6,11. In Hefe kann die Phosphorylierung dieses Faktors unter Nährstoffentzug und osmotischem Stress induziert werden1,4,11,14,15, und in Säugetierzellen können Aminosäuremangel, endoplasmatischer Retikulum (ER) -Stress, UV-Stress, Virusinfektion und veränderte Sauerstoffwerte diese Reaktion auslösen8,9,11. Die schnelle Hochregulierung der spezifischen mRNA-Translation zeigt sich in der Reaktion der Säugetierzellen auf Hypoxie, die eine globale schnelle Translationshemmung und selektive Hochregulierung der Hypoxie-induzierbaren Faktoren (HIFs) Biosynthese aufweist. HIFs sind Transkriptionsfaktoren, die dann eine längerfristige zelluläre Reprogrammierung auf der DNA-Transkriptionsebene auslösen8,9,16. Ähnliche Reaktionen wurden in Hefe unter Hitzestress beobachtet, mit schneller translationaler Expression von Hitzeschockproteinen (HSPs), gefolgt von verzögerten Reaktionen auf Transkriptionsebene.17,18. Zusätzlich zu Nährstoffentzug und Hitzeschock wurden translationale Reaktionen in Hefe unter unterschiedlichem Sauerstoff untersucht.8,19Salzgehalt5, Phosphat, Schwefel20,21 und Stickstoff22,23 Stufen. Diese Forschung hat weitreichende Auswirkungen auf die industrielle Verwendung von Hefe, wie Backen und Fermentieren24,25. Translationale Reaktionen können auch dazu beitragen, das Verständnis von Krankheiten wie neurodegenerativen Erkrankungen und Herzerkrankungen zu fördern, die durch intrazelluläre Belastungen wie oxidativen Stress gekennzeichnet sind. Insgesamt sind translationale Reaktionen integraler Bestandteil der Genexpressionskontrolle und ermöglichen eine schnelle Anpassung an ein breites Spektrum von Stressbedingungen in eukaryotischen Organismen.
Um translationale Antworten zu untersuchen, sind Methoden erforderlich, die minimal verzerrte Momentaufnahmen der Übersetzungslandschaft liefern. Polysomenprofilierung ist ein klassischer Ansatz, der bei der Untersuchung der Translation über mRNA verwendet wird, bei dem poly(ribo)somale Anteile von mRNA durch Ultrazentrifugation durch Saccharosegradienten getrennt werden26,27. Der Ansatz kann verwendet werden, um Translationsniveaus für einzelne mRNAs (mit den Nachweismethoden wie Reverse Transkription und Polymerase-Kettenreaktion, RT-PCR26) oder global in Verbindung mit Hochdurchsatztechniken (Microarray oder RNA-seq28,29) zu untersuchen. Ein weiterentwickelter Ansatz ist das Ribosomen-Profiling, das die Untersuchung von Positionen länglicher Ribosomen entlang eines mRNA-Moleküls auf genomweiter Skala sowie die Rückschlüsse auf die Effizienz der Translation über Transkriptome und die Nutzung der Haupt- und alternativen Startstellenermöglicht 30,31. Das Ribosomen-Profiling beinhaltet die Isolierung und Sequenzierung von mRNA-Fragmenten, die durch ribosomale Präsenz geschützt sind. Die Ribosomenprofilierung hat einen beträchtlichen Einblick in die Translationsdynamik unter einer Reihe von Bedingungen gegeben, darunter hypoxischer Stress, Hitzeschock und oxidativer Stress31,32. Die Technik wurde an mehrere Quellmaterialtypen angepasst, darunter Hefe- und Säugetierzellen.
Während polysomen- und ribosomenprofilierung grundlegend für die Erweiterung der Fähigkeiten der Translationsforschung waren, umfasst der Prozess der Translation verschiedene translationale Zwischenprodukte und Komplexe, die mit diesen Methoden schwer zu erfassen sind11,13. Eine zusätzliche Einschränkung ergibt sich aus der mangelnden Fähigkeit, Schnellreaktionstypen zu untersuchen, da translationale Komplexe entweder in vivo durch zugesetzte spezifische Translationsinhibitoren (Antibiotika) stabilisiert werden, was zu bestimmten Ribosomenverteilungsartefakten führt, oder ex vivo bei Zelllyse spezifisch (Antibiotika) oder unspezifisch (hohe Salz- oder Magnesiumionen), was zum Entzug der kurzlebigen oder weniger stabilen Zwischenprodukte führt33. 34,35.
Formaldehyd wird häufig zur Vernetzung von Nukleinsäuren und Proteinen verwendet, z. B. in Chromatin-Immunpräzipitationsstudien (ChIP) und Crosslinking Immunoprecipitation (CLIP). Seine geringe Größe und ausgezeichnete Zellpermeabilität ermöglichen eine schnelle In-vivo-Aktion 36. Basierend auf der schnellen Formaldehydvernetzung wurde der Ribosomenprofilierungsansatz um das Translation Complex Profile Sequencing (TCP-seq)10,36,37,38,39,40erweitert. TCP-seq, zuerst in Hefe entwickelt, ermöglicht die Erfassung aller Übersetzungszwischenprodukte, einschließlich Scannen oder Post-Termination SSU-Komplexe und mehrere ribosomale Konfigurationen37,38,41,42. Die Methode wurde in mehreren Studienverwendet 10,38,39,41 ,42,von denen einige einen kombinatorischen Ansatz sowohl von Translationsinhibitoren als auch von Formaldehydvernetzung verwenden, um den Stillstand der Translation zu erleichtern. Eine weitere modifizierte Version der Technik, selektives TCP-seq39,wurde kürzlich eingesetzt, um die Immunreinigung der vernetzten Komplexe einzubeziehen und den Umfang der TCP-seq-Anwendungen zu erweitern. Die schnelle, effiziente und reversible Natur der Formaldehydvernetzung macht diese Ansätze geeignet, um transiente mRNA:Translation-Komplex-Interaktionen zu untersuchen, insbesondere im Kontext hochdynamischer Reaktionswege auf Translationsebene.
Hier beschreiben wir die Prozesse der in vivo Formaldehydvernetzung zum Zwecke einer umfassenden Stabilisierung und Isolierung von Translationskomplexen. Wir bieten separate Protokolle, die für Hefe- und Säugetierzellen nuanciert sind (Abbildung 1). Wir skizzieren weiterhin Beispiele für die anschließende Verwendung des vernetzungsstabilisierten Materials (Abbildung 1), wie z.B. für den co-gereinigten Proteinfaktornachweis mittels Immunoblotting (Western-Blotting), immununterstützter Aufreinigung (oder "Immunpräzipitation"; IP) und Anreicherung von translationalen Komplexen mit spezifischen Interessantfaktoren, Elektronenmikroskopie und RNA-Sequenzierung.
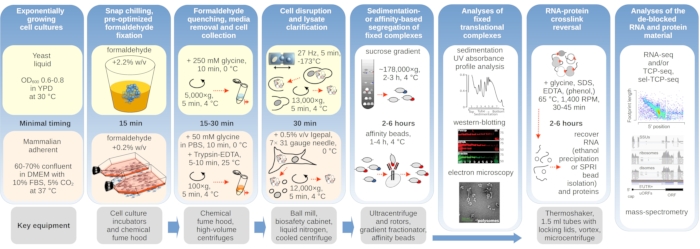
Abbildung 1: Schematische Darstellung eines Überblicks über den typischen Versuchsaufbau. Hauptschritte der in vivo Formaldehydstabilisierung translationaler Komplexe werden als Flussdiagramm dargestellt, ergänzt durch Informationen über die wichtigsten notwendigen Instrumente. Mögliche nachgelagerte Anwendungen des vernetzten Materials werden skizziert, einschließlich Beispiele, die erfolgreich eingesetzt wurden, aber nicht direkt in diesem Protokoll abgedeckt sind, wie z.B. SPRI-Perlenreinigung von RNA, RNA-Sequenzierung und Massenspektrometrie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Hefezellprotokoll
- Hefezellkultur und Fixierung
HINWEIS: Zellfixierung und -ernte werden von10,38mit Modifikationen angepasst.- 1 L Hefezellkultur (Wildtyp (WT) BY4741 werden beispielhaft angegeben) in einem Orbitalschüttler mit der optischen Ausgangsdichte von nicht mehr als 0,05 AE bei 600 nm (OD600)in geeigneten Medien (1% w/v Hefeextrakt, 2% w/v Pepton, 2% w/v Dextrose (Glucose), 40 mg/L Adeninsulfat (YPD) als Beispiel) unter den gewünschten Bedingungen (30 °C in diesem Verwendeten) aufgestellt Experiment).
- Richten Sie eine präparative Zentrifuge mit kompatiblem Rotor und Zentrifugenflaschen für die Pelletierung der flüssigen Suspensionskultur von Hefezellen ein. Für Glukosehungerexperimente pelletieren die Zellen, sobald die optische Dichte von 0,6-0,8 AE bei 600 nm (OD600)erreicht ist, mittels einer kurzen Zentrifugation bei 30 °C, 5.000 x g für 1 min.
HINWEIS: Zeichnen Sie den OD der wachsenden Zellen auf und lassen Sie die Zellen wachsen, bis der OD600 0,6-0,8 AE erreicht, wenn die exponentielle Wachstumsphase von Interesse ist. - Das Pellet sofort in warme (30 °C) YP-Medien, die keine oder wenig (0,25% w/v) Glucose enthalten, resuspendieren und die Kultur für weitere 10 min bei 30 °C in einem Orbital-Shaker-Inkubator inkubieren.
HINWEIS: Die Medienzusammensetzung kann die spätere Vernetzungseffizienz beeinträchtigen. Dieses Protokoll wurde nur mit YPD getestet. Bei der Durchführung von Hungerexperimenten ist die Einhaltung des Timings und die Minimierung der Verzögerungen zwischen den Verfahren von entscheidender Bedeutung. - Sobald die Zellen fertig sind, stellen Sie eine Eisbox im Abzug mit einem Becher auf, der 250 g sauberes Crushed Water Ice enthält. Stellen Sie sicher, dass 25 ml Stripettes und frisch gekaufte methanolstabilisierte 37% ige Formaldehydlösung in der Haube zugänglich sind. Gießen Sie die 1 L-Kultur in das Becherglas, das 25% w/v zerstoßenes Wassereis enthält.
HINWEIS: Bewahren Sie die Zellen während aller nachfolgenden Operationen auf Eis auf, bis die Zellen eingefroren sind, sofern nicht anders angegeben. - 75 ml 37 % w/v Formaldehydlösung in eine Endkonzentration von 2,2 % w/v geben und die Mischung intensiv umrühren, bis das Eis schmilzt.
- Sobald das Eis geschmolzen ist, stellen Sie einen Timer für 10 Minuten ein.
HINWEIS: Halten Sie sich an die empfohlenen Zeiten und das Temperaturschema, um reproduzierbare Fixierungsergebnisse zu erzielen. - Nach der Inkubation für 10 min wird die Kultur in die vorgekühlten Zentrifugenflaschen überführt und die Zellen durch Zentrifugation bei 4 °C, 5.000 x g für 5 min pelletiert. Während dieser Drehung ein 50-ml-Röhrchen vorkühlen und frisch zubereiteten Puffer A (mit Glycin zur Neutralisierung des verbleibenden Formaldehyds) auf Eis aufbewahren.
HINWEIS: Die genauen Pufferzusammensetzungen finden Sie in der mitgelieferten Tabelle. - Nach dem Zentrifugieren die Zentrifugenröhrchen mit der Pelletseite in Kontakt mit dem Eis auf Eis legen. Bringen Sie die Rohre in den Abzug und entsorgen Sie den Überstand in einem Formaldehyd-Abfallbehälter.
- Resuspendieren Sie das Zellpellet aus allen Röhrchen in 20 mL Puffer A mit einer 25 mL Stripette und übertragen Sie es in ein 50 mL Rohr.
HINWEIS: Diese Wäsche ist entscheidend, um eine nicht reproduzierbare Vernetzung zu vermeiden, und die Pufferzugabe darf 20 Minuten nach der Ernte der Zellen nicht überschreiten. - Machen Sie das Volumen mit Puffer A auf 40 mL und sammeln Sie die gewaschenen Zellen durch Zentrifugation bei 4 °C, 5.000 x g für 5 min.
- Verwerfen Sie den Überstand und resuspenieren Sie das Zellpellet in 40 ml Puffer A1, der Puffer A ist, der kein Glycin enthält, um eine Glycinkontamination zu entfernen.
- Pelletzellen wieder durch Zentrifugation bei 4 °C, 5.000 x g für 5 min.
- Wiederholen Sie die Wäschen mit Puffer A1 noch einmal. Entsorgen Sie den Überstand und legen Sie das Zellpellet auf Eis. Wiegen Sie das Röhrchen mit dem Pellet (die nasse Zellmasse sollte ~ 1 g pro 1 L der Zellkultur betragen).
- Hefezellaufschluss und Zytosolsammlung
- Füllen Sie eine mit Aluminiumfolie ausgekleidete Polystyrolschaumbox mit flüssigem Stickstoff bis zu einer Tiefe von ca. 3 cm. Legen Sie ein 50 ml Rohr aufrecht in die Box.
- Resuspendieren Sie das Pellet (~1 g Nasszellenmasse) in 550 μL Puffer A2 durch Pipettieren und Vortexen für 10 s. Fügen Sie 10 μL 40 U / μL RNase-Inhibitor hinzu und wirbeln Sie erneut für 10 s.
VORSICHT: Tragen Sie beim Umgang mit flüssigem Stickstoff eine geeignete Schutzausrüstung wie wärmeisolierte Handschuhe. Stellen Sie sicher, dass jeder Behälter, der zur Aufnahme von flüssigem Stickstoff verwendet wird, nicht ausläuft und dass das Rohrgestell im Inneren nicht nach oben schwimmt oder auf die Seite fällt. Arbeiten Sie in einem gut belüfteten Bereich, um Sauerstoffmangel zu vermeiden. - Tropfen Sie die Zellsuspension mit einer 1-ml-Pipette in das 50-ml-Röhrchen, das den flüssigen Stickstoff enthält.
HINWEIS: Das Tropfen muss langsam und vorsichtig durchgeführt werden, um eine Aggregation der Tröpfchen zu vermeiden. Stellen Sie sicher, dass die Tröpfchen einfrieren, bevor Sie neue Tröpfchen einführen. - Das 50-ml-Röhrchen mit den gefrorenen Zellsuspensionströpfchen auf Raumtemperatur bringen und warten, bis der flüssige Stickstoff vollständig verdampft ist. Verschließen Sie das Röhrchen mit seiner Kappe und lagern Sie die Zellpellets bei -80 °C oder fahren Sie sofort weiter.
VORSICHT: Stellen Sie sicher, dass der flüssige Stickstoff vollständig verdampft ist, bevor Sie das Rohr versiegeln. Übrig gebliebener flüssiger Stickstoff in einem abgedichteten Rohr kann einen gefährlichen Druckaufbau verursachen. - Um sich auf den nächsten Schritt vorzubereiten, 1,5 ml nukleasefreie Röhrchen und 10 mL Edelstahl-Mahlgläser auf Trockeneis vorkühlen.
- Die gefrorenen Zellsuspensionströpfchen mit einem sauberen, sterilen Spatel in die Gläser geben.
ACHTUNG: Stellen Sie sicher, dass die Mahlgläser dicht verschlossen sind. - Tauchen Sie die Mahlgläser für 1 Minute in den flüssigen Stickstoff ein, um sicherzustellen, dass die flüssige Phase unter der Verbindung bleibt. Richten Sie eine Kryomischermühle bei 27 Hz für das Rühren für 1 Minute ein.
HINWEIS: Balancieren Sie den Schleifkanister immer mit einem anderen des gleichen Modells aus, auch wenn die Probe nur einen Kanister für die Verarbeitung benötigt. - Rühren Sie die versiegelten Mahlgläser bei 27 Hz für 1 min in der Mischermühle.
- Kühlen Sie die Mahlgläser wie bisher in flüssigem Stickstoff ab und schütteln Sie bei 27 Hz für 1 min weiter in der Mischermühle.
- Die Gläser zusammen mit den 1,5 ml nukleasefreien Röhrchen in die Eisbox mit Trockeneis geben. Mit einem kleinen Stahlspatel die resultierende Pulverprobe in ~ 100 mg Aliquots in die Röhrchen überführen und die Röhrchen bei -80 °C lagern.
HINWEIS: Es wird empfohlen, ~ 600 mg der Probe pro Experiment zu verwenden, einschließlich der Polysomensedimentationsprofilanalyse, der Trennung des Zytosols in übersetzte und nicht übersetzte Fraktionen und einer weiteren Trennung der übersetzten Fraktion in SSU-, Ribosomen- und Disomefraktionen beim RNase-Aufschluss.
- Trennung der fixierten (poly)ribosomalen Komplexe von den nicht-translatierten Fraktionen des Zytosols
ANMERKUNG: Das zuvor festgelegte Verfahren10,38wird im Allgemeinen befolgt, um übersetzte RNA basierend auf ihrer Co-Sedimentation mit (Poly)Ribosomen anzureichern. Hier wird ein verfeinerter Ansatz zur Trennung der übersetzten und nicht translatierten Zytosolfraktionen vorgestellt, wodurch die Notwendigkeit entfällt, das Material zu fälschen und anschließend wieder zu lösen.- Herstellung von 2,5 ml linearen 10%-20% w/v Saccharosegradienten mit Puffer B unter Verwendung der Gefrier-Tau-Methode43 in dünnwandigen Ultrazentrifugenröhrchen (5 mL, 13 x 51 mm).
HINWEIS: Die Freeze-Thaw-Methode wird durch sequentielle Zugabe und Einfrieren von gepufferten Saccharoseschichten mit linear regressiven Konzentrationen übereinander durchgeführt. Siehe ergänzende Tabelle 1 für Einzelheiten. - Um ein diskontinuierliches 50% w/v Saccharosekissen zu erzeugen, geben Sie bei den linearen Gradienten, die auftauen und stabilisieren, langsam 0,5 ml 50% Saccharose in Puffer B direkt auf den Boden der Röhrchen mit einer 1 mL Spritze, die an einer 19 G x 1,5"-Nadel oder einer Glaskapillar mit ähnlichen /geeigneten Abmessungen befestigt ist. Vor der Dosierung vorsichtig und langsam die Spitze der Nadel oder Kapillare von oben nach unten der vorgeformten Saccharosegradienten treiben und jede Störung vermeiden, bis sie den Rohrboden erreicht.
ANMERKUNG: Anweisungen zur Herstellung von Puffer B finden Sie in der ergänzenden Tabelle 1. - Gleichen Sie die Gradienten sorgfältig aus, indem Sie die oberen Teile entfernen oder mehr als 10 w/v Saccharose in Puffer B schichten und eiskalt oder bei 4 ° C halten.
HINWEIS: Der diskontinuierliche Gradient mit der unteren 50% Saccharoseschicht wird benötigt, um Material mit höherer Sedimentationsrate zu sammeln, ohne es an der Rohrwand auszufällen. - ~100 mg der gefrorenen Zellpulverprobe bei Raumtemperatur auftauen und sofort auf Eis legen. Mischen Sie 150 μL Puffer A2 durch Pipettieren, fügen Sie RNase-Inhibitor zu 1 U / μL hinzu und mischen Sie durch Vortexing (vermeiden Sie übermäßiges Schäumen und Mischen mit der gasförmigen Phase) für 10 s.
HINWEIS: Setzen Sie alle Vorgänge fort, während Sie das Material auf Eis halten, sofern nicht anders angegeben. - Pelletieren Sie die Zelltrümmer durch Zentrifugieren der Röhrchen bei 4 °C, 13.000 x g für 5 min und gewinnen Sie den geklärten Überstand (~ 150 μL) in einem neuen 1,5 mL niedrigen Proteinbindungsröhrchen zurück.
- Laden Sie das resultierende geklärte Gemisch auf die diskontinuierlichen Saccharosegradientenröhrchen aus Schritt 1.3.3 und balancieren Sie sie sorgfältig aus.
- Ultrazentrifugieren Sie die Rohre in einem mittelgroßen Schwenklöffelrotor bei 4 °C mit einer durchschnittlichen g-Kraft von 287.980 x g (k-Faktor 49) für 1 h 30 min.
HINWEIS: Diese Bedingungen wurden voroptimiert (unter Verwendung der UV-Absorptionsspurenanalyse nach dem Ultrazentrifugationsgradienten), um die freien (nicht(poly)ribosomalen) SSUs und LSUs (große ribosomale Untereinheit) im oberen (10%-20% Saccharose) Teil des Gradienten zu erhalten, während die (poly)ribosomale Fraktion im unteren (50%) Saccharosekissen konzentriert wird, ohne das Material zu pelletieren. - Verwenden Sie eine neue sterile 1-ml-Spritze, die mit einer 19 G x 1,5-Zoll-Nadel ausgestattet ist, um die übersetzte Zytosolfraktion zu sammeln. Platzieren Sie das 5-ml-Gefälle auf einem stabilen Rack, um sicherzustellen, dass der Boden des Rohrs sichtbar ist.
- Von der Oberseite des Röhrchens die Nadel gerade in den Boden des Gradienten stecken (ohne das Röhrchen zu durchstechen) und vorsichtig, ohne Blasen zu erzeugen, genau 0,5 ml der unteren Lösung ziehen, die den übersetzten RNA-Pool enthält.
HINWEIS: Stellen Sie sicher, dass dieser Schritt in einem kalten Raum ausgeführt wird und der Schlauch fest gehalten wird. Es wird empfohlen, die gesamten 0,5 ml in einer einzigen Aufwärtsbewegung zu zeichnen, um eine Störung des Gradienten zu vermeiden. - Bestätigen Sie die (poly)ribosomale Präsenz und die Erschöpfung der SSU-, LSU- und leichteren Fraktionen in der resultierenden Mischung durch Absorptionsanzeige des Saccharosegradienten bei Ultrazentrifugationslauf.
- Konzentrieren Sie den gesammelten übersetzten RNA-Pool aus dem vorherigen Schritt mittels Ultrafiltration auf 100 μL in einem Mikrokonzentrationsgerät mit 10 kDa Cut-off regenerierter Cellulosemembran.
HINWEIS: Waschen Sie die Membran des Mikrokonzentrationsgeräts mit 0,5 ml Puffer 1 vor (siehe Abbildung 2a) und verwenden Sie die vom Hersteller empfohlenen Spinbedingungen (g). - Verdünnen Sie das Material aus dem vorherigen Schritt fünfmal (400 μL hinzufügen) mit Puffer 1 und konzentrieren Sie es wieder auf 200 μL, um ein kleineres Volumen sowie eine teilweise Entfernung der Saccharose zu ermöglichen.
HINWEIS: Es wird empfohlen, die resultierenden Gemische bis zu 6 Monate bei -80 °C aufzubewahren und als Eingangsmaterial für die RNA-seq-Bibliothekskonstruktion "total translated RNA" oder den RNase-Aufschlussschritt des TCP-seq-Bibliotheksaufbaus zu verwenden. Die "nicht übersetzte" Zytosolfraktion kann mit einem ähnlichen Verfahren von der Oberseite des Gradienten gewonnen und bei -80 °C gelagert werden.
- Herstellung von 2,5 ml linearen 10%-20% w/v Saccharosegradienten mit Puffer B unter Verwendung der Gefrier-Tau-Methode43 in dünnwandigen Ultrazentrifugenröhrchen (5 mL, 13 x 51 mm).
- RNase-Verdauung der fixierten (poly)ribosomalen Komplexe und Trennung von verdautem Material in kleine ribosomale Untereinheiten (SSU), monoribosomale (Ribosomen, RS) und diribosomale (Disome, DS) Fraktionen
HINWEIS: Das Verfahren folgt im Allgemeinen einem zuvor beschriebenen Ansatz.10,38Es werden jedoch ein modifizierter Gradiententyp, Eine Trennzeit, eine Beschleunigung und RNase-Aufschlussbedingungen verwendet, um die beste Auflösung für alle drei isolierten Fraktionen zu erreichen.- Bereiten Sie sorgfältig ausbalancierte 12,5 ml lineare 10%-40% w/v Saccharosegradienten vor, die mit Puffer 1 in 13 mL dünnwandigen Polypropylenrohren, 14 x 89 mm, unter Verwendung der Gefrier-Tau-Methode43 wie in Schritt 1.3.1 beschrieben hergestellt wurden, und beachten Sie dies.
- Bei Raumtemperatur auftauen und die Proben sofort auf Eis übertragen oder die konzentrierte und mit Saccharose erschöpfte übersetzte Zytosolfraktion aus Schritt 1.3.12 entnehmen.
HINWEIS: Setzen Sie alle Prozeduren auf Eis fort, sofern nicht anders angegeben. - Verdauung der übersetzten Zytosolfraktion durch Mischen von 4,5 E. coli RNase I pro 1 OD260 Einheit der Fraktion für 30 min bei 23 °C. Sofort den RNase-Inhibitor, der RNase I in der Lage ist, RNase I zu inaktivieren, auf 0,25 U/μL zu inaktivieren, in die Mischung geben und mischen, um RNase I zu inaktivieren.
HINWEIS: Verwenden Sie einen RNase-Inhibitor, der in der Lage ist, RNase I zu hemmen. Leiten Sie AU260 ab, indem Sie AU260 = (Absorption bei 260 nm standardisiert auf optische Dichteeinheiten, die 1 cm optischem Pfad x Volumen des Lysat in μL entsprechen) / 1.000 verwenden. - Übertragen Sie die Proben sofort auf Eis.
VORSICHT: Es ist wichtig, die empfohlenen Verdauungsbedingungen einzuhalten und die Menge der zugesetzten RNase I sorgfältig zu messen. Die hier erwähnte RNase I-Einheit ist definiert als die Menge des Enzyms, die benötigt wird, um 1 μg säurelösliches Material aus Mausleber-RNA in 30 min bei 37 °C herzustellen. Wenn der Enzymstock zu konzentriert ist, wird empfohlen, ihn mit Puffer 1 zu verdünnen, um zu vermeiden, dass sehr kleine Mengen der Lösung pipettiert werden. - Laden Sie die Reaktionsgemische auf die 10%-40% w/v Saccharosegradienten aus Schritt 1.4.1.
HINWEIS: Verwenden Sie Endvolumina im Bereich von 150-300 μL pro Gradient. Jede Reinigung erfordert mindestens zwei Gradienten. Verwenden Sie unterschiedliche Eingangsvolumina des Materials (untere AU260, 10-11 AU260, für DS und vergleichsweise höhere AU260, 13-14 AU260, für SSU oder RS), um eine optimale Trennung zu erreichen. - Ultrazentrifugieren Sie die Rohre in einem mittelgroßen Schwenklöffelrotor bei 4 °C mit einer mittleren g-Kraft von 178.305 x g (k-Faktor 143,9) für 3 h 30 min.
ACHTUNG: Wenn Ersatzwuchtröhrchen benötigt werden, gleichen Sie deren Masse und Massenverteilung mit den probenhaltigen Röhrchen aus. Verwenden Sie Ersatz-Saccharose-Gradienten, die mit einer Puffermenge überlagert sind, die der des Probenauflages entspricht, und nicht Röhrchen mit gleichmäßiger Saccharosekonzentration. - Richten Sie mindestens 30 minuten vor Abschluss des Ultrazentrifugationsspins eine Gradientenfraktioniervorrichtung ein, einschließlich des Einfüllens der 0,2 μm gefilterten Heavy Chase-Lösung (z. B. 60% Saccharose in deionisiertem Wasser, wie hier verwendet) in die Verdrängungspumpe.
HINWEIS: Es wird empfohlen, die Leitungen und Schläuche des Fraktionierers mit deionisiertem Wasser zu entkontaminieren, gefolgt von 1% -2% SDS-Lösung in entionisiertem Wasser, deionisiertem Wasser und schließlich 80% Ethanol in deionisierter Wasserlösung vor und nach den Läufen. - Passen Sie die Absorptionsmessbasislinie an, indem Sie zuerst das System mit deionisiertem Wasser füllen und die Optik gemäß den Empfehlungen des Herstellers auf Null stellen und dann die Basislinienverschiebung mit einem ersatzen unbelasteten 14 x 89 mm Saccharosegradienten kompensieren, der mit einem Puffer hergestellt wurde, der mit den Probenröhrchen identisch ist (z. B. Puffer 1).
HINWEIS: Verwenden Sie die gleiche Verschiebungsgeschwindigkeit, um die Einstellungen wie für die Probenauslesung vorzunehmen, z. B. 1,5 ml/min. - Messen Sie das Totvolumen des Verschiebungssystems, indem Sie die Zeit zwischen dem ersten Eintritt der Lösung in den optischen Pfad des Detektors und dem ersten Auftreten am Ausgang des Fraktionskollektors genau zählen.
HINWEIS: Mit der empfohlenen Geschwindigkeit von 1,5 ml/min kann die Fraktionierung bei Raumtemperatur durchgeführt werden. Es wird empfohlen, die gesammelten Fraktionen sofort auf Eis zu übertragen. - Führen Sie die Fraktionierung mit Live-Absorptionsanzeige bei 254 nm, 1,5 ml/min Verdrängungsgeschwindigkeit und Inline-Fraktionsdetektion basierend auf der erwarteten Sedimentationsposition und dem Absorptionsprofil der Proben durch. Verwenden Sie die Kollektorröhrenschaltung mit einer Zeitverzögerung, die dem zuvor gemessenen Totvolumen entspricht.
- Fraktionen, die den Positionen und der Mobilität der SSU-, RS- und DS-Komplexe entsprechen, isolieren und in neuen proteinarm bindenden 1,5-ml-Mikrozentrifugenröhrchen sammeln; die isolierten Fraktionen sofort auf Eis übertragen und einfrieren, wenn sie nicht sofort weiterverarbeitet werden.
HINWEIS: Es wird empfohlen, die gesammelten Fraktionen sofort in Trockeneis oder flüssigem Stickstoff zu schockgefrieren und bei -80 °C oder weniger für bis zu 6 Monate zu lagern.
- Entvernetzung der ribosomalen Komplexe und Isolierung der RNA zum Aufbau von RNA-seq-Bibliotheken
- Um die Vernetzung zu entblocken/umzukehren und die RNA von den assoziierten Proteinen weg zu isolieren, übertragen Sie etwa die Hälfte der gesamten Saccharosegradientenfraktionen in neue 1,5 ml Mikrozentrifugenröhrchen aus Polypropylen mit niedrigem Nukleinsäurebindungs-1,5 ml Mikrozentrifugenröhrchen (350 μL pro Röhrchen) mit Deckelsicherheits-/Verriegelungsvorrichtungen.
- Ergänzen Sie die Mischungen mit 40 μL 100% Stop-Lösung (10% SDS w/v und 100 mM EDTA), 4 μL 1 M Tris-HCl pH 2 bei 25 °C (bis 10 mM), 1,6 μL 2,5 M Glycin (bis 10 mM) und deionasefreiem Wasser, um das Endvolumen von 400 μL zu erhalten.
- Mischen Sie den Inhalt der Rohre durch Pipettieren und übertragen Sie die Rohre bei Raumtemperatur.
- Zu jedem Röhrchen wird das gleiche Volumen des sauren Phenol:Chloroform:Isoamylalkohol 125:24:1 (pH 4,0-5,0) Gemischs gegeben. Schütteln Sie die Mischungen 2 min lang kräftig mit einem auf maximale Geschwindigkeit eingestellten Wirbelmischer.
VORSICHT: Phenol und Chloroform sind ätzend und giftig. Vermeiden Sie den physischen Kontakt mit den Flüssigkeiten und arbeiten Sie in einem gut belüfteten Bereich oder unter einem Abzug. Verwenden Sie bei der Arbeit mit Phenol oder Chloroform immer Handschuhe, Laborkittel und Schutzbrille oder einen Gesichtsschutz. - Legen Sie die Rohre in einen Thermoshaker und schütteln Sie kontinuierlich bei 65 °C, 1.400 U / min für 30 min.
- Erleichtern Sie die Phasenaggregation, indem Sie die Mischung bei 12.000 x g für 10 min bei Raumtemperatur zentrifugieren.
- Sammeln Sie die oberen wässrigen Phasen und übertragen Sie sie in frische 1,5 ml Röhrchen mit niedriger Nukleinsäurebindung.
HINWEIS: Um Kreuzkontaminationen zu vermeiden, versuchen Sie nicht, die wässrigen Phasen vollständig wiederherzustellen. Ein vernünftiges Rückgewinnungsvolumen beträgt 300-350 μL. - Ergänzen Sie die gesammelten wässrigen Phasen mit 0,1 Volumen 3 M Natriumacetat (pH 5 bei 25 °C), 20 μg Glykogen (unter Verwendung von 5 μg/μL Stamm) und 2,5 Volumen absolutem Ethanol. Mischen Sie die Lösungen vorsichtig, indem Sie die Rohre für 1 min vorwirbeln.
- Die RNA wird ausgefällt, indem die Proben bei -20 °C für mindestens 2 h inkubiert werden (empfohlen über Nacht).
- Erwärmen Sie die Rohre auf Raumtemperatur und mischen Sie sie durch Wirbeln.
HINWEIS: Die Vorwärmung der Röhrchen und die anschließende Zentrifugation bei Raumtemperatur (ohne erzwungene Abkühlung) tragen dazu bei, die Co-Ausfällung und den Übertrag von Salz und Phenol zu reduzieren. Diese Bedingungen sollten nicht zu Materialverlust oder Ineffizienz der RNA-Sammlung führen, wenn sie wie beschrieben durchgeführt und ausreichend reines Ethanol verwendet wird. - Pelletieren Sie die RNA, indem Sie die Röhrchen bei 12.000 x g für 30 min bei Raumtemperatur zentrifugieren.
- Entsorgen Sie den Überstand und waschen Sie das Pellet zweimal mit 80% v / v Ethanol und sammeln Sie es jedes Mal durch Zentrifugation bei 12.000 x g für 10 min bei Raumtemperatur.
- Trocknen Sie die RNA-Pellets, indem Sie die Röhrchendeckel öffnen und die geöffneten Röhrchen für 10 min in eine auf 45 °C eingestellte Trockenblockheizung legen. Lösen Sie das resultierende getrocknete Pellet in 20 μL 1x HE-Puffer auf.
- Schätzen Sie die resultierende RNA-Konzentration mithilfe der UV-Absorptionsspektrummessung.
HINWEIS: Die Länge und Gesamtmenge der RNA-Fragmente kann mit Hilfe der Denaturierungsgelelektrophorese weiter beurteilt werden, z. B. in einem automatisierten fluoreszenzbasierten Kapillargelelektrophoreseapparat.
- Selektive Co-Immunreinigung der SSUs durch die markierten eIFs und Western-Blot-Analyse der selektiven SSU-Anreicherung
HINWEIS: Verwenden Sie ~15 AE (260 nm) der verdauten und sedimentationsgetrennten SSU-Fraktion aus Schritt 1.4.11, um eine Affinitätsreinigung mit magnetischen IgG-Kügelchen durchzuführen. Sparen Sie ~5% des SSU-Anteils als Eingangssteuerung (Eingangsfraktion, I). eIF4A-getaggt (TIF1-TAP; Tandem Affinity Purification Tag) wurde ein Hefestamm verwendet, der es auch ermöglicht, eIF4A nachzuweisen, indem er mit Anti-TAP-Antikörpern nach dem TAP-Tag sucht.- Transfer von 100 μL magnetischer IgG-Perlensuspension (1 mg der Kügelchen wurde für jeweils 15 AE (260 nm) des Lysats oder der Fraktion verwendet) in ein neues 1,5-ml-Röhrchen mit niedriger Proteinbindung; Sammeln Sie die Perlen mit einem Magnetgestell und saugen Sie sie ab.
- Waschen Sie die Magnetperlen zweimal mit 1 ml Puffer 1, indem Sie die sequentielle Resuspension durch Pipettieren und Sammeln mit dem Magnetgestell verwenden.
- Sammeln und dekantieren Sie die Perlen nach dem Waschen, während Sie sie auf dem Magnetgestell aufbewahren.
- Die SSU-Fraktion zu den gewaschenen Kügelchen geben und die Mischung für 4 h mit Einer Drehung bei 4 °C in einem Cyclomixer mit ~20 U/min inkubieren.
- Sammeln Sie die Kügelchen mit dem Magnetgestell bei 4 °C und sparen Sie den Überstand (Durchflussfraktion, FT).
- Waschen Sie die Perlen zweimal bei 4 °C mit Puffer 1, ergänzt mit 4 mM DTT, drehen Sie sich jedes Mal für 10 min im Cyclomixer und sammeln und dekantieren Sie die Perlen auf dem Magnetgestell. Bewahren Sie die Wäschen (W1- und W2-Fraktionen) auf.
- Für eine analytische Anwendung wie Western Blotting eluieren Sie das gebundene Material unter denaturierenden und reduzierenden Bedingungen durch Zugabe von LDS (Lithium-Dodecylsulfat) Polyacrylamid-Gelelektrophorese (PAGE) Probenpuffer mit pH 8,5 bis 1x und DTT bis 2 mM.
- Erhitzen Sie die Mischung bei 95 °C für 5 min in einem thermischen Block, um die Elution abzuschließen.
- Sammeln Sie die Perlen mit dem magnetischen Rack und gewinnen Sie das denaturierte Eluat (E-Fraktion) in einem frischen, proteinarm bindenden 1,5 ml Mikrozentrifugenröhrchen zurück.
- Verwenden Sie die E-Fraktion aus dem vorherigen Schritt, um sofort eine denaturierende Natriumdodecylsulfat (SDS)-SEITE auszuführen, oder lagern Sie die E-Fraktion bei -20 °C.
HINWEIS: Für eine präparative Sammlung der mit TAP-Tags angereicherten translationalen Komplexe für jede nachfolgende Anwendung verwenden Sie einen alternativen Elutionsansatz unter Verwendung der Tobacco Etch Virus (TEV) -Protease. Weitere Einzelheiten sind der ergänzenden Tabelle 1 zu entnehmen. - Um die verdünnten FT-, W1- und W2-Fraktionen zu konzentrieren, fallen Sie ihr Material aus, indem Sie 3x Volumen eiskaltes Aceton hinzufügen. Die Proben-Aceton-Mischung bei -20 °C für 3 h inkubieren.
- Pelletieren Sie den Niederschlag durch Zentrifugieren der Röhrchen bei 13.000 x g für 10 min bei 4 °C.
- Entsorgen Sie den Überstand und trocknen Sie das Pellet in den offenen Rohren bei Raumtemperatur für 30 min an der Luft.
- Lösen Sie das Pellet in 7 μL 1x LDS-Ladepuffer auf, ergänzt mit 2 mM DTT. Erhitzen Sie die Proben in einem auf 95 °C eingestellten Wärmeblock für 5 min.
- Laden Sie alle I-, FT-, W1-, W2- und E-Proben auf ein 4%-12% w/v Acrylamid-Gradienten-, Bis-Tris-Polyacrylamid-Denaturierungsgel. Führen Sie das Gel mit 1x MES SDS (2- [N-Mopholino]ethansulfonsäure, Natriumdodecylsulfat) mit einem Puffer bei 80 V laufen, bis sich der Proteinmarker (10-250 kDa) gut auflöst und der Bleifarbstoff den Boden des Gels erreicht.
HINWEIS: Es wird empfohlen, serielle Verdünnungen des WCL (Ganzzelllysat) (2-10 μg) als Kontrolle auf das Gel zu laden. Es kann mehrere Versuche erfordern, um eine vergleichbare Belastung des Gels über das Fraktionsmaterial zu erreichen. - Übertragen Sie den Proteingehalt des Gels auf eine Polyvinylidendifluorid (PVDF) -Membran durch Nasstransfermethode bei 100 V für 1 h in einem Kühlraum, wie vom Hersteller von Western-Blotting-Geräten empfohlen.
- Blockieren Sie die Membran mit einem geeigneten Blockierpuffer (auf Basis von Phosphate Buffered Saline) bei Raumtemperatur für 1 h unter ständigem Schütteln.
- Gemäß den Anweisungen des Herstellers zur Antikörperverdünnung untersuchen Sie die Membran mit einem Anti-TAP-Antikörper zum Nachweis des markierten eIF4A-Proteins, des Anti-Pab1p-Antikörpers oder des Anti-β-Aktin-Antikörpers (oder eines anderen wünschenswerten Ziels) durch über Nacht inkubation der Membran mit Blocking Buffer (PBS)-verdünntem Antikörper (1:1.000 Verdünnung) in einem Cyclomixer in einem Kühlraum.
HINWEIS: Eine 1:1.000-Antikörperverdünnung ist ein guter Ausgangspunkt. - Waschen Sie die Membran dreimal mit 1x phosphatgepufferter Kochsalzlösung, 0,2% v/v Tween 20 (PBST) für jeweils 10 min.
- Untersuchen Sie die Membran mit fluoreszierend markierten sekundären Antikörpern gemäß den Anweisungen des Herstellers, indem Sie 1 h lang in einem Cyclomixer bei Raumtemperatur inkubieren.
HINWEIS: 1:20.000 Antikörperverdünnung ist ein guter Ausgangspunkt. - Waschen Sie die Membran dreimal mit 1x PBST für jeweils 10 min. Spülen Sie die Membran kurz mit entionisiertem Wasser und dann mit absolutem Methanol ab. Trocknen und visualisieren Sie die Membran in einem fluoreszierenden Bildgebungssystem gemäß den Anweisungen des Herstellers.
HINWEIS: Die Färbung für andere Proteine kann durch die Verwendung sekundärer Antikörper mit Farbstoffen erreicht werden, die verschiedenen Fluoreszenzkanälen entsprechen (wie im hier verwendeten eIF4A-TAP vs. β-Aktin-Paar), durch sequentielles Färben oder und Färben derselben Membran oder durch Schneiden der Membran aus einem Gel, das mit sich wiederholenden Fraktionsmustern beladen ist, und durch separates Sondieren jedes Stücks mit entsprechenden Antikörpern (wie in dem hier verwendeten Pab1p-Beispiel).
2. Säugetierzellprotokoll
- Zellkultur und Fixierung von Säugetieren
- In 2 T-175-Kolben HEK293-Zellen auf 60% -70% Konfluenz in Dulbeccos Modified Eagle Medium und 10% v/v Fetal Bovine Serum bei 37 °C und 5% v/v Kohlendioxid wachsen.
HINWEIS: Das gesamte Medium wird durch Zugabe von 55 ml kommerziellem FBS in ein 500 ml kommerziell erworbenes DMEM mit hohem Glukosegehalt hergestellt, das L-Glutamin, Phenolrot und Natriumbicarbonat, jedoch kein HEPES oder Natriumpyruvat enthält. Die Zellzahlen pro T-175-Kolben bei 70% Konfluenz sollten im Bereich von 1,7-2,0 x 107 liegen. - Ersetzen Sie mindestens 3 h vor der gewünschten Fixierungszeit das Medium der T-175-Kolben durch genau 30 ml vorgewärmte Gesamtmedien und ersetzen Sie die Kolben in einem Zellinkubator.
ANMERKUNG: Stellen Sie sicher, dass das frische Medium auf die gegenüberliegende Seite des Kolbens zur Zellmonoschicht pipettiert wird, um eine Zellablösung zu vermeiden. Versuchen Sie, den Medienaustausch so schnell wie möglich durchzuführen, indem Sie minimale Gas- und Temperaturgleichgewichtsstörungen verursachen. - Sobald das Zellmedium ausgetauscht wurde, bereiten Sie Puffer und Chemikalien vor, die für die Fixierung erforderlich sind. Bereiten Sie Dulbecco's Phosphate Buffered-Saline (DPBS) mit 50 mM Glycin vor, indem Sie 10,2 ml 2,5 M Glycin-Kern in eine 500-ml-Flasche DPBS geben und mischen.
- Bereiten Sie eine Flasche DMEM mit 10% FBS gemäß Schritt 2.1.1 vor, die unter nicht sterilen Bedingungen und einem 100-ml-Aliquot von 0,25% Trypsin-EDTA verwendet wird. Beziehen Sie eine zusätzliche Flasche kommerzielles DPBS, das mit Calciumchlorid(CaCl2)und Magnesiumchlorid (MgCl2) vorformuliert ist.
HINWEIS: Die Lösungen können bei 4 °C bis zu 2 Wochen gelagert werden. - Bereiten Sie eine Eisbox bis zum Rand mit zerstoßenem Wassereis so vor, dass ein T-175-Kolben gleichmäßig darauf passt und zusammen mit den vorbereiteten Puffern, auch auf Eis, im Abzug aufbewahrt wird.
ANMERKUNG: Aufgrund der schnellen Translationsreaktion auf jede Umweltveränderung müssen alle Zeitpunkte zwischen der Entfernung der Zellkolben aus dem Inkubator und der Zugabe der Formaldehydlösung minimiert werden. - Um die Zellen zu kühlen, nehmen Sie den T-175-Kolben aus dem Inkubator und drücken Sie ihn fest gegen das Eis, um maximalen Oberflächenkontakt zu gewährleisten. Kippen Sie den Kolben im Inneren des chemischen Abzugs auf die Seite, so dass sich das Medium an der den Zellen gegenüberliegenden Seite sammelt. 168 μL 37% w/v Formaldehyd direkt in das gepoolte Medium pipettieren (bis zu einer Endkonzentration von 0,2% w/v). Mischen Sie sofort, indem Sie den Kolben vorsichtig hin und her schaukeln, schließen und positionieren Sie den Kolben auf Eis, um sicherzustellen, dass er horizontal ist und die Zellen gleichmäßig bedeckt sind.
VORSICHT: Formaldehyd ist eine schädliche Substanz mit potenziell langfristigen Nebenwirkungen und auch eine Reizung der Atemwege und der Haut. Es sollte nur in einem geeigneten chemischen Abzug gehandhabt werden. Behälter mit Formaldehyd müssen immer verschlossen sein, wenn sie sich außerhalb des Abzugs befinden.
HINWEIS: Stellen Sie sicher, dass das Formaldehyd direkt in das Zellmedium und nicht in die Kolbenwand gegeben wird. Schritt 2.1.6 sollte weniger als 1 Min. dauern. - Die Kolben für weitere 10 min auf Eis inkubieren. Gießen Sie das Medium in einen geeigneten Abfallbehälter durch die Kolbenseite gegenüber den Zellen.
- Mit einer Stripette in 30 ml Dulbecco's Phosphate Buffered Saline ohne Calcium- und Magnesiumionen pipettieren und zusätzlich 50 mM Glycin enthalten, sanft auf der den Zellen gegenüberliegenden Seite. Mischen Sie, indem Sie den Kolben schaukeln; Bringen Sie den Kolben in die horizontale Position zurück und inkubieren Sie für weitere 10 Minuten auf Eis.
- Gießen Sie die Lösung durch die den Zellen gegenüberliegende Kolbenseite ab und geben Sie vorsichtig 7 ml der Standardmäßigen 0,25% mit Trypsin-EDTA-Lösung hinzu, um die Zellen zu lösen und wieder zu resuspendieren. Den Kolben bei Raumtemperatur für 5-10 min inkubieren.
HINWEIS: Stellen Sie sicher, dass die Trypsin-EDTA-Lösung alle Zellen gleichmäßig abdeckt. Verwenden Sie periodisches sanftes Kippen und Schaukeln, um die Zellablösung zu fördern. - Stellen Sie den Kolben senkrecht auf und sammeln Sie die abgelösten Zellen mit einer Stripette, indem Sie alle verbleibenden Zellen vorsichtig von den Kolbenwänden waschen. Übertragen Sie die Suspension in ein 50 ml Rohr, das auf Eis steht.
HINWEIS: Feste Zellen können zerbrechlicher werden; Nicht intensiv oder mehr pipettieren, als zum Lösen der Zellen von der Kolbenwand erforderlich ist. - Die gesammelte Zellsuspension wird sofort mit 20 mL kompletten Medien (den nicht sterilen eiskalten Medien mit 10% FBS) ergänzt und durch vorsichtiges Umdrehen des Röhrchens vermischt.
HINWEIS: Das gesamte Zellkulturmedium (einschließlich 10% FBS) wird hinzugefügt, um das Trypsin zu neutralisieren und eine weitere Schädigung der Zellmembranen und den Zellzerfall zu verhindern. - Pelletieren Sie die Zellen, indem Sie das Röhrchen bei 100 x g für 5 min und 4 °C zentrifugieren. Zellpellets müssen gut sichtbar sein.
- Gießen Sie das Medium ab und resuspenieren Sie das Zellpellet vorsichtig in 10 ml eiskaltem DPBS mit Ca2+,Mg2+und ohne Glycin.
- Wiederholen Sie Schritt 2.1.12.
- Den Waschpuffer abgießen und das Zellpellet in 800 μL eiskaltem DPBS mit Ca2+,Mg2+,ohne Glycin, vorsichtig auf Eis resuspenieren. Übertragen Sie die resuspendierten Zellen in ein neues proteinarmes 1,5 ml Mikrozentrifugenröhrchen.
- Zentrifugieren Sie das Röhrchen bei 100 x g für 3 min und 4 °C. Verwerfen Sie den Überstand vorsichtig mit einem 1 ml Pipettierer. In diesem Stadium kann das Zellpellet bei -80 °C eingefroren werden oder zum Zelllyseschritt übergehen.
HINWEIS: Gefrorene Zellpellets können bei -80 °C bis zu 1 Jahr gelagert werden. Wir fanden heraus, dass das Einfrieren von Zellpellets die anschließende Lyse erleichtert und empfehlen das Einfrieren, auch wenn eine längerfristige Lagerung nicht geplant ist.
- In 2 T-175-Kolben HEK293-Zellen auf 60% -70% Konfluenz in Dulbeccos Modified Eagle Medium und 10% v/v Fetal Bovine Serum bei 37 °C und 5% v/v Kohlendioxid wachsen.
- Aufschluss von Säugetierzellen und Zytosolsammlung
- In einer Biosicherheitswerkbank werden 300 μL des Lysepuffers auf Basis eines nichtionischen, nicht kondensierenden Detergens und 7 μL 40 U/μL RNase-Inhibitor zugegeben. Mischen Sie gut durch Pipettieren mit einer 1 ml Spitze.
- Befestigen Sie vorsichtig eine 25 G Nadel an einer 1-3 ml Spritze und pipettieren Sie die Mischung kräftig, wobei mindestens sieben langsame Aufwärtseinlass- und schnelle Abwärtsauspuffhübe verwendet werden.
- Entsorgen Sie die Spritze und die Nadel in einen Behälter für scharfe Gegenstände und wiederholen Sie den Vorgang mit einer 0,3 ml Spritze, die mit einer 31 G Nadel ausgestattet ist.
- Entsorgen Sie die Spritze und die Nadel in einen Behälter für scharfe Gegenstände. Zentrifugieren Sie die Röhrchen bei 4 °C, 12.000 x g für 5 min, um die Zelltrümmer zu pelletieren.
- Übertragen Sie den Überstand in ein neues proteinarmes 1,5 ml Mikrozentrifugenröhrchen. Lagern Sie sowohl den Zelltrümmer (zu Kontrollzwecken) als auch das resultierende geklärte Zelllysat bei -80 °C.
ANMERKUNG: Die optische Dichte des Lysat liegt zwischen 25 und 30 AU260, wenn zwei T-175-Kolben kombiniert und die empfohlenen Volumina eingehalten werden. Die Lysate und Zelltrümmer können bei -80 °C bis zu 1 Jahr gelagert werden.
- Trennung der fixierten (poly)ribosomalen Komplexe von den nicht-translatierten Fraktionen des Zytosols
- Herstellung linearer 15%-45% w/v Saccharosegradienten in 13 ml dünnwandigen Polypropylenrohren, 14 x 89 mm, unter Verwendung der Gefrier-Tau-Methode im Allgemeinen wie in Schritt 1.3.1 des Hefeprotokolls beschrieben, jedoch unter Verwendung von Puffer 2 (Abbildung 2a).
HINWEIS: Tauen Sie die Gradienten über Nacht in einem kalten Raum bei 4 °C in der Nacht vor der Fraktionierung auf. - Laden Sie 150-250 (maximal 300) μL des Zelllysat aus dem vorherigen Schritt 2.2.5 auf die ausgeglichenen Gradienten. Das restliche Lysat wird bei -80 °C gelagert und zu Kontrollzwecken verwendet.
HINWEIS: Hier wird ein Beispiel für eine sedimentationsbasierte Segregation in polysomale, ribosomale und "freie" SSU-Fraktionen bereitgestellt. Einen alternativen Ansatz finden Sie in der bereitgestellten ergänzenden Tabelle 1. - Ultrazentrifugieren Sie die Rohre in einem mittelgroßen Schwenklöffelrotor bei 4 °C, mittlere g-Kraft 178.305 x g (k-Faktor 143,9) für 1 h 45 min.
- 30 min vor dem Schleuderabschluss wird der Gradientenfraktionierer, wie in den Hefeprotokollschritten 1.4.7-1.4.9 beschrieben, eingerichtet und zu Basislinie aufgestellt.
- Fraktionieren Sie die Gradienten im Allgemeinen, wie in den Hefeprotokollschritten 1.4.10-1.4.11 beschrieben.
HINWEIS: Dieser Schritt trennt polysomale, ribosomale und "freie" SSU-Fraktionen. Polysomale Fraktionen können in Polysomenprofilierungsexperimenten verwendet werden. - Die gesammelten Fraktionen sofort auf Eis übertragen und wenn nicht weiterverarbeitet, bei -80°C bis zu 6 Monate lagern.
HINWEIS: Wenn der Fraktionsabscheiderrohrwechsel mit der Online-Fraktionsidentifikation und -seigerung synchronisiert ist, empfehlen wir die Verwendung von bis zu 800 μL-Fraktionen (Sammelzeit von 32 s pro Fraktion bei 1,5 ml/min). Wenn die Fraktionierung ohne Verwendung der Inline-Absorptionsanzeige durchgeführt wird, wird die Verwendung von 250-500 μL-Fraktionen (10-20 s pro Fraktion bei 1,5 ml / min) empfohlen. Nach der Trennung können die Fraktionen sofort für die Immunreinigung, Elektronenmikroskopie, Denaturierung von PAGE und Western Blotting verwendet oder für nachfolgende RNA- und/oder Proteomik-Analysen einer Vernetzungsumkehr unterzogen werden.
- Herstellung linearer 15%-45% w/v Saccharosegradienten in 13 ml dünnwandigen Polypropylenrohren, 14 x 89 mm, unter Verwendung der Gefrier-Tau-Methode im Allgemeinen wie in Schritt 1.3.1 des Hefeprotokolls beschrieben, jedoch unter Verwendung von Puffer 2 (Abbildung 2a).
Ergebnisse
Translationale Komplexe reagieren empfindlich auf die ionische Zusammensetzung der Puffer, was besonders wichtig ist bei der Ultrazentrifugation, bei der die Sedimentationseigenschaften bewertet werden. Wir testeten daher mehrere Sedimentationspuffer mit geklärtem Lysat, das aus gemahlenem nicht festem Hefematerial extrahiert wurde, um Bedingungen auszuwählen, die am besten geeignet sind, translationale Komplexe aufzulösen und ribosomale Untereinheiten (SSU, LSU), Monosomen (RS) und Polysomen über den Gradienten zu trennen. Alle Puffer basierten auf der Kernzusammensetzung, die 25 mM HEPES-KOH pH 7,6 und 2 mM DTT enthielt. Die Konzentrationen von KCl,MgCl2,CaCl2und EDTA wurden über die Puffer weiter modifiziert (Abbildung 2a), und diese Komponenten wurden den Lysaten vor der Gradientenbelastung und den Saccharosegradientenpuffern vor dem Gradientenguss entsprechend zugesetzt.
In den Puffern 1 und 2 wurden wohlaufgelöste translationale Komplexe erhalten. Puffer 1 führte zu einer etwas besseren Trennung der kleinen ribosomalen Untereinheiten (SSUs) (Abbildung 2a). Das Weglassen vonMgCl2 und die Zugabe von EDTA (Puffer 3,4) führten zu einem Verlust der hohen Sedimentationseigenschaften für die meisten Polysomen und wahrscheinlich zu deren teilweiser Demontage (Abbildung 2a). Während die Zugabe von 2,5 mMCaCl2 zu etwas homogeneren polysomalen Peaks führte, war die Verbesserung marginal und die Gesamtmenge des polysomalen Materials nahm in diesem Fall (Abbildung 2a) im Vergleich zu den Puffern 1 und 2 ab. Wir haben daher Puffer 1 als Arbeitspuffer der Wahl ausgewählt.
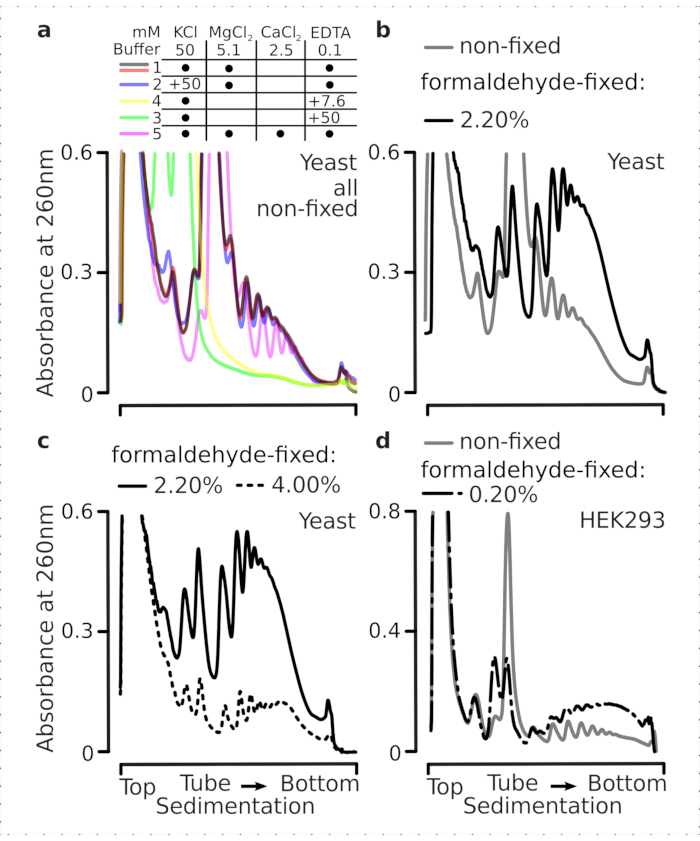
Abbildung 2: Pufferbedingungen für die extraktion translationaler Komplexe und Bewertung der stabilisierenden Wirkung der Fixierung. Gezeigt sind UV-Absorptionsprofile, die bei 260 nm für das gesamte Hefezelllysat in 10% -40% mit Saccharosegradienten getrennt wurden. a) Auswirkungen ein- und zweiwertiger Salze und der Magnesiumionenbindung auf die Sedimentation von Material, das aus nicht fixierten Hefezellen gewonnen wird. Rote und graue Linien stellen ein typisches Replikat dar. (b,c) Vergleich von Lysaten, die aus nicht fixierten (graue Linie), 2,2% (schwarze Linie) und 4,4% (schwarze gepunktete Linie) mit Formaldehyd-fixierten Hefezellen gewonnen wurden. (d) Stabilisierung von Polysomen durch die optimierten 0,2% w/v Formaldehydfixierung (schwarze gestrichelte linie) von HEK 293T-Zellen im Vergleich zu dem Material aus denselben nicht fixierten Zellen (graue Linie). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Als nächstes überprüften wir die Wirkung der polysomalen Stabilisierung durch Fixierung auf verschiedene Formaldehydkonzentrationen. Mit dem ansonsten gleichen Zellmaterial, Puffern, Zellhandhabung und Timing-Ansätzen verglichen wir Material, das aus nicht fixierten Zellen extrahiert wurde, und Zellen, die mit 2,2% und 4% w/v Formaldehyd fixiert waren (Abbildung 2b,c). Wir fanden heraus, dass 2,2% w/v Formaldehyd besser für die Fixierung geeignet waren, da es zwar die Polysomen hervorragend konservierte, wie es anhand des Polysomen-zu-Monosomen-Verhältnisses beurteilt werden kann (Abbildung 2b), aber die Gesamtausbeute des ribosomalen Materials im Vergleich zu 4% w/v Formaldehyd nicht reduzierte, was deutliche Anzeichen einer Überfixierung aufwies (Abbildung 2c).
Für das aus Säugetierzellen gewonnene Material wurde aufgrund des größeren Lysepuffer-zu-Zell-Volumenverhältnisses, das für die extraktion auf Detergenzienbasis erforderlich ist, Puffer 2 (Abbildung 2a) verwendet. Dies erzeugte gut aufgelöste translationale Komplexe bei der Sedimentation in Saccharosegradienten (Abbildung 2d). Insbesondere wurde eine viel niedrigere Konzentration von Formaldehyd von 0,2% w/v verwendet, da höhere Konzentrationen zu einem erheblichen polysomalen und ribosomalen Materialverlust führten (Daten nicht gezeigt). In Ähnlichkeit mit den mit Hefezellen erzielten Ergebnissen zeigte das vernetzungsstabilisierte Material eine bessere Konservierung der Polysomen und ein höheres Polysomen-zu-Monosomen-Verhältnis (Abbildung 2d).
Als nächstes testeten wir, ob die ausgewählten Formaldehyd-Fixierungsbedingungen effizient genug sind, um die aktiv übersetzte mRNA innerhalb der polysomalen Fraktionen als Folge der Vernetzung zu stabilisieren, und die verbesserte polysomale Ausbeute ist nicht nur eine Folge der Hemmung der Enzymfunktion und der Translationsdehnungsprogression. Wir verwendeten EDTA und hocheinwertiges Salz (KCl), um Polysomen und Ribosomen zu destabilisieren. Diese Reagenzien wurden den geklärten Hefezelllysaten zugesetzt und in alle nachfolgenden Puffer und Saccharosegradienten zusätzlich zur Puffer-1-Zusammensetzung aufgenommen.
Tatsächlich zeigte 15 mM EDTA einen geringeren Destabilisierungseffekt auf die polysomalen Fraktionen, die von den fixierten Zellen abgeleitet wurden (Abbildung 3a), was bestätigt, dass die vernetzten Komplexe robuster sind. Die destabilisierende Wirkung von EDTA kann durch erhöhung der Formaldehydkonzentration etwas überwunden werden, da Material aus den 4% w/v von Formaldehyd-fixierten Zellen einer besseren Entfaltung standhält (Abbildung 3a). Die Erhöhung der EDTA-Konzentration auf 50 mM führte jedoch zu einer Destabilisierung der meisten translationalen Komplexe sowohl unter festen als auch unter nicht festen Bedingungen, wie aus der langsameren Sedimentation des Materials und dem Fehlen gut geformter Peaks abgeleitet werden kann (Abbildung 3b). Dies kann durch die teilweise Entfaltung von Strukturen und den allgemeinen Verlust der Kompaktheit erklärt werden, anstatt durch die vollständige Dissoziation polysomaler Komponenten von der mRNA. Selbst in diesem Fall hat das vernetzte Material eine schnellere Sedimentation gezeigt (Abbildung 3b).
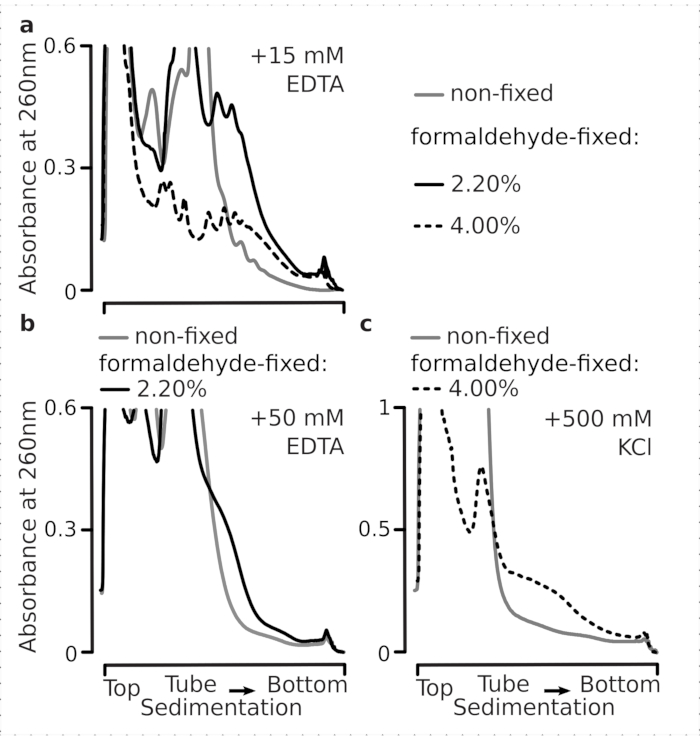
Abbildung 3: Auswirkungen der in vivo Hefe-Formaldehyd-Fixierung auf die Stabilität von Polysomen. Puffer 1 (siehe Text und Abbildung 2a)wurde in allen Experimenten verwendet. Datentyp und -darstellung wie in der Legende in Abbildung 2 beschrieben. (a) Vergleich der Addition von 15 mM EDTA zu den Zelllysaten und nachfolgenden Puffern auf die Stabilität der Polysomen, die von nicht fixierten (graue Linie), 2,2% (schwarze Linie) und 4% (schwarze gepunktete Linie) w/v von Formaldehyd-fixierten Zellen abgeleitet sind. b) identisch mit Buchstabea), jedoch für den Zusatz von 50 mM EDTA und ausgenommen 4 % w/v an formaldehydfixierten Zellen. c) identisch mit Buchstabea), jedoch für den Zusatz von 500 mM KCl und ohne 2,2 Gew.-% an Formaldehyd-fixierten Zellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ähnlich wie bei den EDTA-Effekten fanden wir bei 500 mM KCl eine deutliche Verbesserung der Stabilität mit 4% w/v Formaldehydfixierung (Abbildung 3c). Der scheinbare Verlust der Kompaktheit kann in diesem Fall auch durch eine teilweise Ablösung der Bestandteile der ribosomalen Komplexe und nicht durch ihre vollständige Dissoziation von der RNA erklärt werden. Insgesamt zeigten Polysomen, die aus Formaldehyd-fixierten Zellen gewonnen wurden, eine höhere Beständigkeit gegen Entfaltung und strukturelle Destabilisierung, was mit der Bildung zusätzlicher kovalenter Bindungen innerhalb dieser Komplexe übereinstimmt.
Während stimulierender Wachstumsbedingungen können mRNAs schnell initiiert werden, was zu einer Anhäufung mehrerer Ribosomen auf denselben mRNA-Molekülen führt, die Strukturen bilden, die als Polyribosomen oder Polysomen bekannt sind. Polysomen können durch Ultrazentrifugation in Saccharosegradienten getrennt werden, wo sie basierend auf ihrer Ordnung (Anzahl der gleichzeitig anhaftenden Ribosomen auf mRNA) sedimentieren. Wenn die Translation unterdrückt wird, gelingt es den Ribosomen nicht, früh genug eine weitere Übersetzungsrunde durchzuführen, was zu einer (teilweisen) "Demontage" von Polysomen führt, was sich als Modalverschiebung zu den Polysomen einer niedrigeren Ordnung und Akkumulation von Monosomen4,26zeigt.
Ein Modell der translationalen Reaktion, das auf der Polysomenordnungsverteilungsebene visualisiert werden kann, kann durch Glukosemangel bereitgestellt werden. Glukosemangel löst eine der dramatischsten und schnellsten translationalen hemmenden Wirkungen auf Hefeaus 1,3,40. Frühere Studien zeigten, dass innerhalb von 1 min nach Glukoseabbau, Verlust von Polysomen, Akkumulation von Monosomen und Hemmung der Translationsinitiation auftreten können4. Innerhalb von 5 Minuten nach der erneuten Glukoseergänzung wird die Translation schnell wiederhergestellt, wobei der Anstieg der Polysomen3,4 offensichtlich ist. Es wurde auch beobachtet, dass die Translation gehemmt wurde, wenn Zellen Medien ausgesetzt waren, die Glukose von 0,5% (w/v) oder weniger enthielten, und es gab keine Wirkung bei Glukosespiegeln von 0,6% (w/v) oder höher.
Wir wollten daher feststellen, ob unsere Fixierungsbedingungen für die Erhaltung der translationalen Unterschiede innerhalb der Dynamik der Glukose-Stressantwort geeignet sind, wie sie anhand des Polysomen-zu-Monosomen-Verhältnisses beurteilt werden können. Wir verglichen das Material aus den Zellen, die in der mittleren exponentiellen Phase auf hoher Glukose (2,00% w / v hinzugefügt) gezüchtet wurden, mit denen, die für 10 Minuten in Medien mit keiner oder niedriger Zugabe (0,00% bzw. 0,25% w / v) Glukose übertragen wurden. Die Fixierung wurde unter Verwendung von 2,2% w/v Formaldehyd parallel in der Kontrolle durchgeführt (nicht verhungert; schneller Medienaustausch mit gleichen Standardmedien mit 2% w/v zugesetzter Glucose, gefolgt von Inkubation für 10 min und Fixierung) und 10 min verhungert (schneller Medienaustausch mit gleichen Medien, aber niedrig 0,25 w/v oder ohne zugesetzte Glukose, gefolgt von Inkubation für 10 min und Fixierung) Zellen.
In Übereinstimmung mit den früheren Ergebnissen beobachteten wir, dass Hefezellen die Translation bei Glukosemangel stark unterdrücken (Abbildung 4a). Sowohl keine zugesetzten als auch niedrige Glukosebedingungen induzierten die Polysom-Demontage, wobei bei niedrig zugesetzter Glukose etwas, aber offensichtlich mehr Polysomen zurückgehalten wurden. Daher kann die Reaktion auf die Entfernung von Hefeglukose nicht von einem All-On- oder All-Off-Typ sein und wird allmählich abgestimmt. Polysomales Material aus den fixierten Zellen bestätigt die Erwartungen an die stabilisierende Wirkung der Formaldehydvernetzung und hat eine höhere Unterscheidung zwischen den ausgehungerten und nicht ausgehungerten Zellen gezeigt, wodurch wohl ein höherer Dynamikbereich der Reaktion erhalten bleibt (Abbildung 4b). Interessanterweise führte im Falle von Material aus den fixierten Zellen eine niedrige zugesetzte Glukosekonzentration zu der spezifischen polysomalen Häufigkeit, die im Vergleich zu den nicht fixierten Zellen viel besser von der Bedingung ohne zusätzliche Glukose unterschieden wird (Abbildung 4a). Dies ist ein starker Hinweis auf die Eignung des Formaldehydfixierungsansatzes zur Erhaltung und Erfassung relativ winziger und vorübergehender Unterschiede im Gleichgewicht hochdynamischer Prozesse, z. B. während translationaler Reaktionen.

Abbildung 4:Erfassung schneller Veränderungen der Hefetranslation bei Glukosemangel. Puffer 1 (siehe Text und Abbildung 2a)wurde in allen Experimenten verwendet. Datentyp und -darstellung wie in der Legende in Abbildung 2 beschrieben. (a)Zelllysate, die aus nicht verhungerten (graue Linie), eingeschränkt glukosehungrigen (0,25% mit zugesetzter Glukose für 10 min; braune Linie) und glukosearmen (keine zugesetzte Glukose für 10 min; rote Linie) nicht fixierten Hefezellen gewonnen werden. b) wie (a), jedoch für 2,2 % w/v Formaldehyd-fixierte Zellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die Überwachung des translationalen Status durch die Ribosomen, die mit der aktiven Translation von mRNA mittels Saccharosegradientensedimentation ("Polysomenprofilierung") assoziiertsind,ist eine weit verbreitete Technik26,27,28. In Kombination mit der quantitativen Microarray-Analyse und in jüngster Zeit mit der Hochdurchsatzsequenzierung28,44liefert das Polysomen-Profiling Informationen über Ribosom-assoziierte mRNAs-Transkriptom-weit. Mit mehreren Annahmen wurde im Bereich der Proteinbiosyntheseforschung traditionell argumentiert, dass die polysomale Präsenz ein Hinweis auf eine aktive Beteiligung an der Translation der jeweiligen mRNAs ist. Eine weitere Schlussfolgerung ist oft (aber nicht immer) gerechtfertigt, dass je mehr Ribosomen auf einer mRNA einer bestimmten Länge vorhanden sind (je höher die Ordnung der Polysomen), desto aktiver ist diese mRNA an der Translation beteiligt. Daher kann die Trennung der polysomalen Fraktion vom Rest des Materials unter dem Gesichtspunkt der Isolierung der aktiv übersetzten RNA nützlich sein. Innerhalb der Footprint-Profiling-Ansätze, insbesondere TCP-seq10,38,39, das eine separate Population der befreiten SSUs erzeugt, die aus den Scanning-, Start- und Stopp-Codon-Komplexen abgeleitet ist, kann es zusätzlich aufschlussreich sein, ribosomale Untereinheiten zu entfernen, die nicht mit den vollständigen Monosomen oder Polysomen co-sedimentieren.
Wir haben daher eine Trennung der "nicht übersetzten" mRNPs wie freie SSUs (mRNA, die an einzelne SSU oder SSUs ohne angehängte mRNA gebunden ist) vom "aktiv übersetzenden" Pool von mRNAs eingesetzt. Um dies zu erreichen, gingen wir davon aus, dass mRNAs, die an Wechselwirkungen mit entweder einer (Mono-) oder mehreren Ribosomen (Polysomen) beteiligt sind, aktiv übersetzt werden können. Solche Komplexe können durch ihren höheren Sedimentationskoeffizienten von den anderen getrennt werden. Wir schlugen auch vor, den "aktiv übersetzten" Pool von mRNAs in ein Saccharosekissen (50% w/v Saccharose) zu trennen, anstatt das Material direkt an der Rohrwand zu pelletieren. Die Zentrifugation der schnell sedimentierenden Komplexe in das Kissen ermöglichte es uns, die Trennung mittels Absorptionsprofilanzeige zu überwachen und eine höhere Leistung des solubilisierten, nicht aggregierten und nicht denaturierten Materials im Vergleich zur Pelletierung und Wiederlösung zu erreichen10,38.
Insgesamt wurden zur Reinigung der einzelnen SSUs, Ribosomen, Disome und potentiell kompakt gepackten Polysomen höherer Ordnung fest geklärte Lysate einem zweistufigen Ultrazentrifugationsprozess unterzogen (Abbildung 5). Im ersten Saccharosegradienten führte die Ultrazentrifugation zu getrennten freien SSUs und LSUs im oberen Teil (10%-20% w/v Saccharose) des Gradienten, während der vernetzte translatierte Pool einschließlich Polysomen und mRNAs, die mit einem vollständigen Ribosom assoziiert waren, am unteren Rand (50% w/v Saccharose) des Gradienten konzentriert waren (Abbildung 5a ). Die unteren 50% w/v der Saccharoseschicht, die den übersetzten mRNA-Pool enthielt, wurden dann konzentriert und ihre RNA mit RNase I verdaut, gefolgt von einer zweiten Saccharosegradienten-Ultrazentrifugation, um separate SSU-, LSU-, RS-, RNase-resistente Disome (DS) und eine geringfügige Fraktion nukleaseresistenter Polysomen höherer Ordnung zu erhalten ( Abbildung5b). Die negative Färbung mit Uranylacetat und die Bildgebung mit einem Transmissionselektronenmikroskop bestätigten die Identität der in jeder Sedimentationsstufe isolierten Komplexe (Abbildung 5).

Abbildung 5: Isolierung der gesamten übersetzten RNA-Fraktionen weg von der untranslatierten RNA. (a,c) Schematische (links) und die jeweiligen repräsentativen Ergebnisse (rechts; Datentyp und Diagramm wie in der Legende von Abbildung 2 beschrieben) von (a) erster diskontinuierlicher Saccharosegradiententrennung der nicht translatierten Zytosolfraktionen einschließlich freier SSUs und des übersetzten mRNA-Pools, identifiziert durch Co-Sedimentation mit Ribosomen und Polysomen, und(c)Trennung der einzelnen ribosomalen Komplexe, die aus dem translatierten mRNA-Pool durch kontrollierte RNase-I-Verdauung und Ultrazentrifugation durch einen zweiten linearen Saccharosegradienten in SSU-, LSU-, ribosomale (RS) und nukleaseresistente disomale (DS) Fraktionen freigesetzt wurden. Hohe (15 AU260)und niedrige (8 AU260)Mengen des nicht verhungerten Verdauguts wurden einbezogen, um die Möglichkeit einer Erhöhung der Ultrazentrifugationsbelastung zu demonstrieren, wenn kleinere Fraktionen von Interesse sind. Es können auch nukleaseresistente Polysomen höherer Ordnung identifiziert werden (z. B. Trisomen in den bereitgestellten Beispielen). (b,d) Repräsentative TEM-Abbildungen von Uranylacetat-kontrastierten Fraktionen von (a,c), bzw. wie markiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Um die Eignung des Fixierungsschemas für die Retention transienter Ribosom-assoziierter Proteine (insbesondere eIFs) zu überprüfen, testeten wir die Co-Sedimentation von eIF4A, einem labilen eIF, das dynamisch an das Ribosom gebunden ist, über die ribosomalen Fraktionen hinweg. Wir nutzten den eIF4A Tandem Affinity Purification (TAP) markierten Hefestamm (TIF1-TAP) und untersuchten das Vorhandensein von eIF4A in Material, das aus den fixierten vs. nicht fixierten Zellen gewonnen wurde, indem wir Anti-TAP-Antikörper verwendeten, verglichen mit der Häufigkeit von Pab1p als zusätzliche RNA-Bindungskontrolle, unter Verwendung von SDS-PAGE gefolgt von Western Blotting (Abbildung 6).
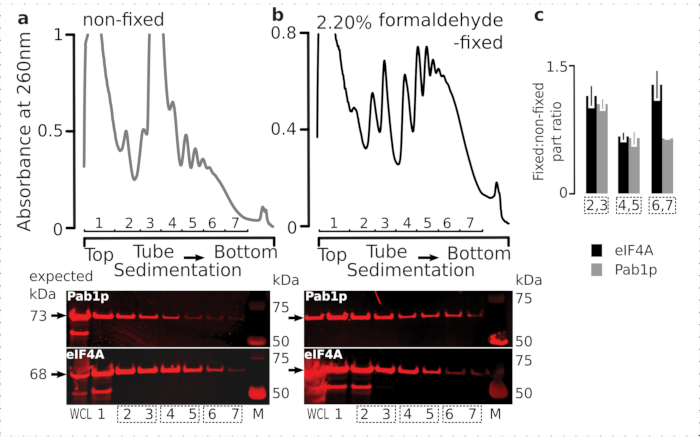
Abbildung 6: Stabilisierung transienter Proteine in den translationalen Komplexen bei in vivo Formaldehydfixierung. (a,b) ( obere Diagramme ) Ganzzelllysat (WCL) von (a) nicht fixierten und (b) 2,2% Formaldehyd-fixierten eIF4A-TAP-Hefezellen, die durch Ultrazentrifugation getrennt und visualisiert werden, wie in der Legende von Abbildung 2 beschrieben. (untere Plots) Western-Blot-Abbildung der jeweiligen Saccharose-Gradientenanteile bei Trennung des analysierten Materials in den entsprechenden Gradienten (Top-Plots) und WCL als Kontrolle. c) Durchschnittliches Verhältnis zwischen der eIF4A- oder Pab1p-Häufigkeit in den Fraktionen von festem und nicht festem Material. Die relativen Anteile (normalisiert auf das Signal aller 2-7 Fraktionen) von eIF4A (schwarze Balken) und Pab1p (graue Balken) wurden über 2,3 (SSU, LSU), 4,5 (RS, leichte Polysomen) und 6,7 (schwere Polysomen) aus den Daten von (a,b) (untere Diagramme) berechnet und ihr festes zu nicht festes Verhältnis genommen. Fehlerindikatoren zeigen die Standardabweichung des Verhältnisses vom Mittelwert an, wobei die gepoolten Brüche (gepunktete Kästchen) als Replikate behandelt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Im Einklang mit ihrer hohen Häufigkeit in den Zellen beobachteten wir eine hohe Intensität des Signals von beiden Proteinen im gesamten Zelllysat (WCL) und langsamer sedimentierenden Fraktionen, die von nicht fixierten Zellen abgeleitet wurden(Abbildung 6a,unteres Bild). Wir haben auch erhebliche Mengen dieser Proteine in der WCL nachgewiesen, die aus den festen Zellen stammen und die Effizienz der vernetzten Materialextraktion und das Fehlen unerwarteter Verluste gewährleisten(Abbildung 6b,untere Platte). Im Gegensatz zu den nicht fixierten Zellen zeigte das Material der fixierten Zellen jedoch im Vergleich zu Pab1p eine erhöhte relative Präsenz von eIF4A in den schneller sedimentierenden ribosomalen Fraktionen (Abbildung 6c). Dieses Ergebnis deutet darauf hin, dass eIF4A fester mit den Polysomen in Formaldehyd-vernetztem Material assoziiert bleibt.
Nachdem wir die positive und spezifische stabilisierende Wirkung der Vernetzung auf das Vorhandensein von eIF4A in den ribosomalen Fraktionen bestätigt hatten, verwendeten wir das feste Material aus dem eIF4A-markierten (TIF1-TAP) Hefestamm, um eIF4A-haltige Komplexe durch Affinitätsreinigung mit magnetischen IgG-Kügelchen einzufangen und anzureichern. Wir haben affinitätsangereicherte WCL-, freie SSU- und polysomale (übersetzter mRNA-Pool) Fraktionen nach der ersten Sedimentation durch Saccharosegradienten (z. B. Abschnitt 1.3 des Hefeprotokolls) sowie SSU-, LSU- und RS-Fraktionen aus der zweiten Sedimentation bei der Demontage des übersetzten Pools in einzelne Komplexe mit RNase I (z. B. Abschnitt 1.4 des Hefeprotokolls)(Abbildung 7 ). In allen Fällen, mit Ausnahme der LSU-Fraktion, konnten wir eine selektive Anreicherung des eIF4A in den gereinigten Fraktionen (Eluat, E) im Vergleich zum Vorhandensein von β-Aktin im Ausgangsmaterial (Input, I) beobachten (Abbildung 7).
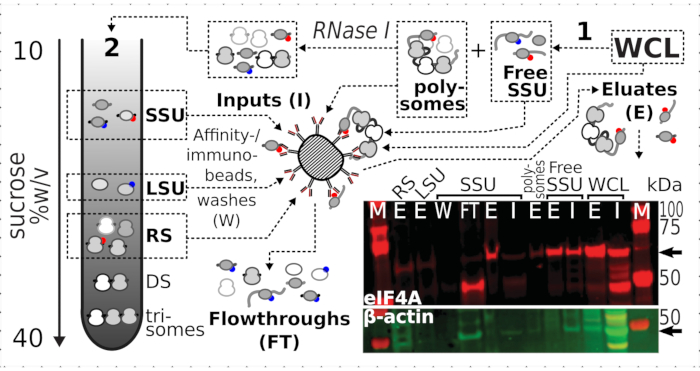
Abbildung 7: Selektive Immunreinigung von in vivo Formaldehyd-stabilisierten translationalen Komplexen durch transient assoziiertes eIF4A. Das Schema veranschaulicht die Quelle verschiedener translationaler Komplexe und eIF4A-Epitope, einschließlich der fraktionierten geklärten WCL der eIF4A-TAP-Hefezellen; freie SSUs und übersetzter RNA-Pool (Polysomen), die in der ersten Ultrazentrifugation getrennt wurden; SSU-, LSU- und RS-Fraktionen, die durch RNase I-Verdauung aus der übersetzten RNA freigesetzt und mittels zweiter Ultrazentrifugation getrennt wurden (siehe Text). Das Western-Blot-Bild bietet eine Visualisierung der eIF4A-Häufigkeit in den Fraktionen im Vergleich zur Häufigkeit der gleichzeitig gefärbten β-Aktin-Kontrolle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Tabelle 1. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Die Formaldehydfixierung ist eine bequeme und beliebte Methode, um eine schnelle In-vivo-Vernetzung der Biomoleküle10,36,45,46,47,48zuerreichen. Im Vergleich zu den anderen potenziellen Biomolekül-Targets erfordert die erfolgreiche Erfassung translationaler Komplexe eine sofortige Fixierung während des Snap-Chillings der Zellen oder anderer Materialien. Ohne die unverschichtete Stabilisierung besteht die Möglichkeit, dass verschiedene translationsbezogene Prozesse fortgesetzt werden, wodurch die komplexe Verteilung vom unbeirrten In-vivo-Zustand weg verlagert wird49. Im Vergleich zu den anderen Methoden des translationalen Stillstands und der ribosomalen Komplexstabilisierung versprechen die Schnelligkeit der Formaldehydwirkung über die Zellmembranen und die wahllose Natur der Vernetzung die Erhaltung der maximalen Vielfalt der Intermediäre des Translationskomplexes näher an ihren nativ verteiltenZuständen 50.
Der hier vorgestellte Ansatz wurde sowohl in Hefe- als auch in Säugetierzellen etabliert und optimiert, und es wurden nun Methoden von anderen Gruppen für den Einsatz in vielfältigerem biologischem Material abgeleitet, z. B. in ganzen Wirbeltieren (z. B. Zebrafischembryonen)10,38,39,49,51,52 . Obwohl diese Arbeiten zusammen die Vielseitigkeit und breite Anwendbarkeit des Ansatzes gewährleisten, kann die schnelle Formaldehydvernetzung translationaler Komplexe aufgrund der Notwendigkeit von Optimierungen und Anpassungen als etwas schwierig zu transponieren auf neue Arten von biologischem Material angesehen werden.
Eine wesentliche Voraussetzung für den Erfolg der Methode ist die Re-Optimierung der Konzentration des Formaldehyds und der Zellsammel- und -aufschlusstechnik. Weniger durchlässige, kleine und runde Hefezellen erfordern eine viel höhere (mindestens 10-fache) Formaldehydkonzentration und physikalische Störung der fixierten Zellen. Im Gegensatz dazu können große und abgeflachte anhaftende Säugetierzellen in Kultur leicht überfixiert werden und erfordern bei der Fixierung eine schonende Handhabung, während die Extraktion der fixierten Komplexe chemisch mit Membranaufschluss unter Verwendung von Reinigungsmitteln erfolgen kann. Eine Untervernetzung kann es weniger stabilen oder kurzlebigen Zwischenprodukten ermöglichen, sich zu dissoziieren oder in einen späteren Zustand zu gelangen. Eine Übervernetzung kann die Fähigkeit, ribosomale Fraktionen zu isolieren und zu untersuchen, negativ beeinflussen und selektive Verzerrungen wie eine tiefere Erschöpfung schwerer Komplexe erzeugen. In unserer Beobachtung können selbst geringfügige Veränderungen, wie die Art der verwendeten adhärenten menschlichen Zellen, die Ausbeute der wiederhergestellten vernetzten Komplexe beeinflussen und eine erneute Optimierung des Vernetzungsschemas erfordern. Wir können auch davon ausgehen, dass Zellen mit wesentlich unterschiedlichen Permeabilitätseigenschaften, wie z.B. Pflanzenzellen, eine zusätzliche umfangreiche Optimierung der Fixierungsbedingungen erfordern52. Es ist jedoch schwierig, sich eine Art von biologischem Material vorzustellen, das mit dem Ansatz völlig unvereinbar wäre.
Eine Überlegung, die für das Säugetier-Fixierungsprotokoll relevant ist, ist die Dichte und Menge des Zellmaterials, das als Input verwendet wird. Es wird empfohlen, die Zellen mindestens 2 Tage lang kontinuierlich ohne erneutes Aussäen oder andere Störungen wachsen zu lassen, um äußere Einflüsse auf die zelluläre Translationsdynamik zu vermeiden. Anwendbar für die meisten Zelltypen, aber für die Mehrheit der adhärenten Zellen, die konsistent Konfluenzniveaus von nicht mehr als 70% erreicht werden, wird sichergestellt, dass keine größeren Kontakthemmungseffekte auftreten, die sich negativ und unvorhersehbar auf die Translationsraten auswirken können.
Ein weiteres interessantes und potenziell einzigartig praktisches Merkmal der Formaldehydfixierung, das sich aus ihrer wahllosen Reaktivität ergibt, ist der stabilisierende Effekt auf translationale Komplexe in Systemen gemischter Taxonomie. Bakterielle und noch mehr translationale Komplexe von Mitochondrien, Chloroplasten und verschiedenen intrazellulären Parasiten waren bekanntermaßen schwer mit spezifischen Translationsinhibitoren zu bekämpfen. Im Gegensatz dazu sind in den TCP-seq-Daten Footprints, die dem Mitotranskriptom zugeordnet sind, in den Daten38,39,50leicht beobachtbar. Eine interessante spätere Entwicklung könnte die Verwendung des Ansatzes zur Untersuchung der Translation in ganzen Mikrogemeinschaften sein, wie in Boden-, Wasser- oder Darmproben, wo ein zuverlässiger schneller translationaler Stillstand und eine komplexe Stabilisierung mit anderen Mitteln problematisch wären.
Es sollte auch erwähnt werden, dass für das komplizierteste Material (wie hartes und / oder sperriges Gewebe) nichts die Verwendung einer Formaldehydstabilisierung unmittelbar nach Zellaufschluss und Materialhomogenisierung verhindert. Dieser Ansatz wird bereits häufig eingesetzt, um die Zelleintrittsverzögerung bei der Stabilisierung translationaler Komplexe mit spezifischen niedermolekularen Inhibitoren33,53,54,55zubeseitigen. Angesichts der Tatsache, dass die Formaldehydfixierung traditionell mit hervorragenden Ergebnissen für die Ex-vivo/In-vitro-Probenstabilisierung in Anwendungen wie der Elektronenmikroskopie45,56,57,58verwendet wurde, können wir in diesem Fall noch weniger negative Effekte erwarten, insbesondere solche, die mit der schlechten Extraktion der translationalen Komplexe aus den gründlich fixierten Zellen verbunden sind.
Unsere Ergebnisse bestätigen die Verwendbarkeit der schnellen Formaldehydfixierung zur Stabilisierung hochtransienter Komplexe, wie sie eIF4A enthalten. Bemerkenswert ist, dass Hefe eIF4A im Gegensatz zu Säugetieren viel schwächer mit dem Kappenbindungskomplex eIF4F und damit mit translationalen Komplexen im Allgemeinen assoziiert ist. eIF4A geht in der Regel bei einer umfangreichen Reinigung des ribosomalen Materials in Hefe29,59,60,61,62,63 verloren. Im in vivo-Fixhefematerial ist es jedoch möglich, eine zuverlässige Anreicherung von eIF4A in allen Fraktionen translationaler Komplexe zu erreichen, bei denen sein Vorhandensein erwartet würde. Die zuvor veröffentlichten Sel-TCP-seq-Daten haben die Anreicherung von eIF2 und eIF3 gezeigt, die stärker mit den Ribosomen assoziiert sind (aber auch eine vorübergehend auftretende co-translationale Proteinkomplex-Assemblierung zeigten)39. Somit eignet sich das Verfahren zum Nachweis sowohl stärkerer als auch schwächerer anhaftender Bestandteile der translationalen Komplexe.
Zusammenfassend haben wir einen Ansatz vorgestellt, der nützlich ist, um in erster Linie Einblicke in die Veränderungen zu gewinnen, die in der Initiationsphase der Translation auftreten und wann eine minimal gestörte ribosomale Verteilung über die mRNA erforderlich ist. Wichtig ist, dass der Ansatz für die Stabilisierung von relativ labilen und dynamischen Komponenten translationaler Komplexe wie eIF4A geeignet ist und breit eingesetzt werden kann, um den notwendigen Optimierungen unterzogen zu werden. Wir haben auch Beweise für die Nützlichkeit der Formaldehydfixierung in den Szenarien einer schnellen dynamischen Veränderung der Translation erbracht, die Untersuchungsbereiche wie schnelllebige zelluläre Reaktionen auf Umweltveränderungen oder Stressbedingungen eröffnen.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Diese Arbeit wurde durch den Australian Research Council Discovery Project Grant (DP180100111 an T.P. und N.E.S.), den National Health and Medical Research Council Investigator Grant (GNT1175388 an N.E.S.) und das Research Fellowship (APP1135928 an T.P.) unterstützt. Die Autoren würdigen die Einrichtungen von Microscopy Australia am Centre for Advanced Microscopy der Australian National University, einer Einrichtung, die von der Universität und der Bundesregierung finanziert wird.
Materialien
| Name | Company | Catalog Number | Comments |
| Yeast extract | Merck, Sigma-Aldrich | 70161 | |
| Peptone | Merck, Sigma-Aldrich | 70178 | |
| D-Glucose (Dextrose) | Merck, Sigma-Aldrich | 49139 | |
| Adenine sulphate | Amresco | 0607-50G | |
| Formaldehyde solution | Merck Sigma-Aldrich | F11635-500ML | ACS reagent, 37 wt. % in H2O, contains 10-15% Methanol as stabiliser (to prevent polymerisation) |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | Invitrogen™ byThermo Fischer Scientific | 10777019 | |
| cOmplete™, EDTA-free Protease Inhibitor Cocktail | COEDTAF-RO Roche by Merck | 11873580001 | |
| Magnesium chloride solution | (Merck/Sigma-Aldrich) | M1028 | |
| Ethylenediaminetetraacetic acid solution | (Merck/Sigma-Aldrich) | E7889 | |
| Ambion™ RNase I, cloned, 100 U/µL | Ambion | AM2294 | |
| SUPERase•In™ RNase Inhibitor (20 U/μL) | Invitrogen™ by Thermo Fisher Scientific | AM2694 | |
| Acidic phenol:chlorophorm:isoamyl alcohol 125:24:1 (pH 4.0-5.0) | (Merck/Sigma-Aldrich) | P1944-100ML | |
| Dynabeads™ Goat Anti-Mouse IgG | Invitrogen™ by Thermo Fisher Scientific) | 11033 | |
| Sodium Acetate (3 M), pH 5.5 | Invitrogen™ by Thermo Fisher Scientific) | AM9740 | |
| Glycogen (5 mg/ml) | Invitrogen™ by Thermo Fisher Scientific) | AM9510 | |
| Ethyl alcohol, Pure | Merck; Sigma Aldrich | E7023 | |
| Amersham™ Hybond® P Western blotting membranes, PVDF | Merck | GE10600023 | PVDF membrane for western blotting |
| Bolt™ 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel | Invitrogen™ by ThermoFischer Sientific | NW04120BOX | Protein gel |
| 4X Bolt™ LDS Sample Buffer | Invitrogen™ by ThermoFischer Sientific | B0007 | LDS sample loading buffer |
| Precision Plus Protein™ Kaleidoscope™ Prestained Protein Standards | BioRad | 1610375 | Protein ladder |
| 20X Bolt™ MES SDS Running Buffer | ThermoFischer Scientific | B0002 | PAGE runninjg buffer |
| Intercept® (PBS) Blocking Buffer | LI-COR | 927-70001 | Odyssey Blcoking buffer (PBS) |
| IRDye® 800CW Goat anti-Mouse IgG Secondary Antibody | LI-COR | 92632210 | |
| IRDye® 800CW Goat anti-Rabbit IgG Secondary Antibody | LI-COR | 92632211 | |
| TAP Tag Polyclonal Antibody | Invitrogen™ by ThermoFischer Sientific | CAB1001 | |
| Anti-beta Actin antibody | Abcam | ab8227 | |
| Sucrose | (Merck/Sigma-Aldrich) | 84097 | BioUltra, for molecular biology, ≥99.5% (HPLC) |
| DL-Dithiothreitol solution | (Merck/Sigma-Aldrich) | 43816 | BioUltra, for molecular biology, ~1 M in H2O |
| Terumo Syringe 1CC/mL | Terumo Syringe | 878499 | |
| Potassium chloride | (Merck/Sigma-Aldrich) | 60128 | |
| HEPES | (Merck/Sigma-Aldrich) | H3375 | |
| Dulbecco's Modified Eagle's Medium - high glucose | Sigma Aldrich | D5796 | |
| Fetal Bovine Serum | Sigma Aldrich | 12003C | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300062 | |
| Dulbecco's Phosphate Buffered Saline with Calcium and magnesium | Sigma-Aldrich | D8662 | |
| Glycine | Sigma-Aldrich | G7126 | |
| Tris hydrochloride | Merck/Sigma-Aldrich | 10812846001 | |
| Sodium dodecyl sulfate | Merck/Sigma-Aldrich | 436143 | |
| IGEPAL CA-630 | Merck/Sigma-Aldrich | I3021 | |
| Rnasin Ribonuclease Inhibitor | Promega | N2111 | |
| Stainless steel grinding jar | Retsch | 02.462.0059 | |
| MM400 mixer mill | Retsch | 20.745.0001 | |
| Gradient Fractionator | Brandel | BRN-BR-188 | |
| Thermomixer R | Eppendorf | Z605271 | |
| Nanodrop spectrophotometer | Thermo Fisher Scientific | ND-2000 | |
| 0.5-ml microcentrifuge tubes with locking devices | Eppendorf Safe-Lock | 30121023 | |
| Mini Gel Tank | (Thermo Fisher Scientific) | A25977 | PAGE running tank |
| 5 mL, Open-Top Thinwall Ultra-Clear Tube, 13 x 51mm | Beckman-Coulter | 344057 | |
| 13.2 mL, Certified Free Open-Top Thinwall Polypropylene, 14 x 89mm - 50Pk | Beckman-Coulter | 331372 | |
| Amicon Ultra-0.5 ultrafiltration devices | Merck | UFC5030 | Ultracel-30 regenerated cellulose membrane, 0.5 mL sample volume |
| Thermo Sorvall Evolution RC Floor Super Speed Centrifuge | Cambridge Scientific | 15566 | |
| Beckman Coulter Optima L-90K | GMI | 8043-30-1191 | |
| Nunc EasYFlask 175cm2 | Thermofisher Scientific | 159910 | |
| Falcon 50 mL Conical Centrifuge Tubes | Thermofisher Scientific | 14-432-22 | |
| 25 mL Serological Pipette | Sigma-Aldrich | SIAL1250 | |
| 10 mL Serological Pipette | Sigma-Aldrich | SIAL1100 | |
| DNA lobind tubes | Eppendorf | 30108051 | |
| Cold Centrifuge 5810 R | Eppendorf | EP022628188 | for 50 mL tubes |
| Orbital Shaking Incubator | Ratek | OM11 | |
| Frezco 17 Microcentrifuge | Thermofisher Scientific | 75002402 | |
| Eppendorf DNA lo-bind tubes | Merck/Sigma-Aldrich | EP0030108051 | |
| Eppendorf® Protein LoBind tubes | Merck/Sigma-Aldrich | EP0030108116 | |
| SW 41 Ti Swinging bucket rotor | Beckman-Coulter | 331362 | |
| Heracell™ 150i CO2 Incubator, 150 L | Thermofisher Scientific | 51026282 | |
| 0,3 mL ultra-fine II short insulin syringe | BD Medical | 328822 | |
| 3 mL syringe with Luer Lok tip | BD Medical | 302113 | |
| 25 G x 16 mm Hypodermic Needle | Terumo | TUAN2516R1 |
Referenzen
- Janapala, Y., Preiss, T., Shirokikh, N. E. Control of translation at the initiation phase during glucose starvation in yeast. International Journal of Molecular Sciences. 20 (16), 4043 (2019).
- Masvidal, L., Hulea, L., Furic, L., Topisirovic, I., Larsson, O. mTOR-sensitive translation: Cleared fog reveals more trees. RNA Biology. 14 (10), 1299-1305 (2017).
- Ashe, M. P., De Long, S. K., Sachs, A. B. Glucose depletion rapidly inhibits translation initiation in yeast. Molecular Biology of the Cell. 11 (3), 833-848 (2000).
- Crawford, R. A., Pavitt, G. D. Translational regulation in response to stress in Saccharomyces cerevisiae. Yeast. 36 (1), 5-21 (2019).
- Melamed, D., Pnueli, L., Arava, Y. Yeast translational response to high salinity: global analysis reveals regulation at multiple levels. RNA. 14 (7), 1337-1351 (2008).
- Hershey, J. W., Sonenberg, N., Mathews, M. B. Principles of translational control: An overview. Cold Spring Harbor Perspectives in Biology. 4 (12), 011528 (2012).
- Mata, J., Marguerat, S., Bähler, J. Post-transcriptional control of gene expression: a genome-wide perspective. Trends in Biochemical Sciences. 30 (9), 506-514 (2005).
- Spriggs, K. A., Bushell, M., Willis, A. E. Translational regulation of gene expression during conditions of cell stress. Molecular Cell. 40 (2), 228-237 (2010).
- Liu, B., Qian, S. B. Translational reprogramming in cellular stress response. Wiley Interdisciplinary Reviews RNA. 5 (3), 301-315 (2014).
- Archer, S. K., Shirokikh, N. E., Beilharz, T. H., Preiss, T. Dynamics of ribosome scanning and recycling revealed by translation complex profiling. Nature. 535 (7613), 570-574 (2016).
- Hinnebusch, A. G., Ivanov, I. P., Sonenberg, N. Translational control by 5'-untranslated regions of eukaryotic mRNAs. Science. 352 (6292), 1413-1416 (2016).
- Dever, T. E., Green, R. The elongation, termination, and recycling phases of translation in eukaryotes. Cold Spring Harbor Perspectives in Biology. 4 (7), 013706 (2012).
- Shirokikh, N. E., Preiss, T. Translation initiation by cap-dependent ribosome recruitment: Recent insights and open questions. Wiley Interdisciplinary Reviews RNA. 9 (4), 1473 (2018).
- Jiménez-Díaz, A., Remacha, M., Ballesta, J. P., Berlanga, J. J. Phosphorylation of initiation factor eIF2 in response to stress conditions is mediated by acidic ribosomal P1/P2 proteins in Saccharomyces cerevisiae. PLoS One. 8 (12), 84219 (2013).
- Sonenberg, N., Hinnebusch, A. G. Regulation of translation initiation in eukaryotes: mechanisms and biological targets. Cell. 136 (4), 731-745 (2009).
- Majmundar, A. J., Wong, W. J., Simon, M. C. Hypoxia-inducible factors and the response to hypoxic stress. Molecular Cell. 40 (2), 294-309 (2010).
- Barraza, C. E., et al. The role of PKA in the translational response to heat stress in Saccharomyces cerevisiae. PLoS One. 12 (10), 0185416 (2017).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: Life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Jamar, N. H., Kritsiligkou, P., Grant, C. M. The non-stop decay mRNA surveillance pathway is required for oxidative stress tolerance. Nucleic Acids Research. 45 (11), 6881-6893 (2017).
- Chen, Z., et al. The complete pathway for thiosulfate utilization in Saccharomyces cerevisiae. Applied and Environmental Microbiology. 84 (22), (2018).
- Marzluf, G. A. Molecular genetics of sulfur assimilation in filamentous fungi and yeast. Annual Review of Microbiology. 51, 73-96 (1997).
- Miller, D., Brandt, N., Gresham, D. Systematic identification of factors mediating accelerated mRNA degradation in response to changes in environmental nitrogen. PLoS Genetics. 14 (5), 1007406 (2018).
- Zhang, W., Du, G., Zhou, J., Chen, J. Regulation of sensing, transportation, and catabolism of nitrogen sources in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 82 (1), (2018).
- Tokpohozin, S. E., Fischer, S., Becker, T. Selection of a new Saccharomyces yeast to enhance relevant sorghum beer aroma components, higher alcohols, and esters. Food Microbiology. 83, 181-186 (2019).
- Walker, G. M., Stewart, G. G. Saccharomyces cerevisiae in the production of fermented beverages. Beverages. 2 (4), 30 (2016).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Jin, H. Y., Xiao, C. An integrated polysome profiling and ribosome profiling method to investigate in vivo translatome. Methods in Molecular Biology. 1712, 1-18 (2018).
- Arava, Y., et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences of the United States of America. 100 (7), 3889-3894 (2003).
- Lackner, D. H., et al. A network of multiple regulatory layers shapes gene expression in fission yeast. Molecular Cell. 26 (1), 145-155 (2007).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-Wide Analysis in Vivo of Translation with Nucleotide Resolution Using Ribosome Profiling. Science. 324 (5924), 218-223 (2009).
- Ingolia, N. T., Hussmann, J. A., Weissman, J. S. Ribosome Profiling: Global Views of Translation. Cold Spring Harbor Perspectives in Biology. 11 (5), (2019).
- Gerashchenko, M. V., Lobanov, A. V., Gladyshev, V. N. Genome-wide ribosome profiling reveals complex translational regulation in response to oxidative stress. Proceedings of the National Academy of Sciences of the United States of America. 109 (43), 17394-17399 (2012).
- Hussmann, J. A., Patchett, S., Johnson, A., Sawyer, S., Press, W. H. Understanding biases in ribosome profiling experiments reveals signatures of translation dynamics in yeast. Proceedings of the National Academy of Sciences Genetics. 11 (12), 1005732 (2015).
- Santos, D. A., Shi, L., Tu, B. P., Weissman, J. S. Cycloheximide can distort measurements of mRNA levels and translation efficiency. Nucleic Acids Research. 47 (10), 4974-4985 (2019).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nature Chemical Biology. 6 (3), 209-217 (2010).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Kage, U., Powell, J. J., Gardiner, D. M., Kazan, K. Ribosome profiling in plants: What is not lost in translation. Journal of Experimental Botany. 71 (18), 5323-5332 (2020).
- Shirokikh, N. E., Archer, S. K., Beilharz, T. H., Powell, D., Preiss, T. Translation complex profile sequencing to study the in vivo dynamics of mRNA-ribosome interactions during translation initiation, elongation and termination. Nature Protocols. 12 (4), 697-731 (2017).
- Wagner, S., et al. Selective translation complex profiling reveals staged initiation and co-translational assembly of initiation factor complexes. Molecular Cell. 79 (4), 546-560 (2020).
- Zlotorynski, E. Profiling ribosome dynamics. Nature Reviews Molecular Cell Biology. 17 (9), 535-535 (2016).
- Sen, N. D., Gupta, N., S, K. A., Preiss, T., Lorsch, J. R., Hinnebusch, A. G. Functional interplay between DEAD-box RNA helicases Ded1 and Dbp1 in preinitiation complex attachment and scanning on structured mRNAs in vivo. Nucleic Acids Research. 47 (16), 8785-8806 (2019).
- Zhao, J., Qin, B., Nikolay, R., Spahn, C. M. T., Zhang, G. Translatomics: The global view of translation. International Journal of Molecular Sciences. 20 (1), 20010212 (2019).
- Luthe, D. S. A simple technique for the preparation and storage of sucrose gradients. Analytical Biochemistry. 135 (1), 230-232 (1983).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Review Genetics. 10 (1), 57-63 (2009).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trends in Biochemical Sciences. 25 (3), 99-104 (2000).
- Schmiedeberg, L., Skene, P., Deaton, A., Bird, A. A Temporal Threshold for Formaldehyde Crosslinking and Fixation. PLoS One. 4 (2), 4636 (2009).
- Solomon, M. J., Varshavsky, A. Formaldehyde-mediated DNA-protein crosslinking: A probe for in vivo chromatin structures. Proceedings of the National Academy of Sciences. 82 (19), 6470-6474 (1985).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping proteinDNA interactions in vivo with formaldehyde: Evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Bohlen, J., Fenzl, K., Kramer, G., Bukau, B., Teleman, A. A. Selective 40S footprinting reveals cap-tethered ribosome scanning in human cells. Molecular Cell. 79 (4), 561-574 (2020).
- Shirokikh, N. E. Translation complex stabilization on messenger RNA and footprint profiling to study the RNA responses and dynamics of protein biosynthesis in the cells. Critical Reviews in Biochemistry and Molecular Biology. , (2021).
- Giess, A., et al. Profiling of small ribosomal subunits reveals modes and regulation of translation initiation. Cell Reports. 31 (3), 107534 (2020).
- Firmino, A. A. P., et al. Separation and paired proteome profiling of plant chloroplast and cytoplasmic ribosomes. Plants (Basel). 9 (7), (2020).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic Acids Research. 42 (17), 134 (2014).
- Santos, D. A., Shi, L., Tu, B. P., Weissman, J. S. Cycloheximide can distort measurements of mRNA levels and translation efficiency. Nucleic Acids Research. 47 (10), 4974-4985 (2019).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nature Chemical Biology. 6 (3), 209-217 (2010).
- Plénat, F., et al. Formaldehyde fixation in the third millennium. Annales De Pathologie. 21 (1), 29-47 (2001).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of Sciences. 105 (7), 2415-2420 (2008).
- Wang, N. S., Minassian, H. The formaldehyde-fixed and paraffin-embedded tissues for diagnostic transmission electron microscopy: A retrospective and prospective study. Human Pathology. 18 (7), 715-727 (1987).
- Grifo, J. A., et al. Characterization of eukaryotic initiation factor 4A, a protein involved in ATP-dependent binding of globin mRNA. Journal of Biological Chemistry. 257 (9), 5246-5252 (1982).
- Li, Y. Commonly used tag combinations for tandem affinity purification. Biotechnology and Applied Biochemistry. 55 (2), 73-83 (2010).
- Blum, S., et al. ATP hydrolysis by initiation factor 4A is required for translation initiation in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences. 89 (16), 7664-7668 (1992).
- Merrick, W. C. eIF4F: A Retrospective. Journal of Biological Chemistry. 290 (40), 24091-24099 (2015).
- Rogers, G. W., Komar, A. A., Merrick, W. C. eIF4A: The godfather of the DEAD box helicases. Progress in Nucleic Acid Research and Molecular Biology. 72, 307-331 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten