Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Untersuchung des Metabolismus und der Viskosität von Krebszellen mit Hilfe der Fluoreszenz-Lebensdauer-Bildgebungsmikroskopie
In diesem Artikel
Zusammenfassung
Hier demonstrieren wir den Einsatz der Fluoreszenz-Lebensdauer-Bildgebungsmikroskopie (FLIM) zur sequentiellen Abbildung des Zellstoffwechsels und der Plasmamembranviskosität in lebenden Krebszellkulturen. Metabolische Bewertungen werden durch den Nachweis endogener Fluoreszenz durchgeführt. Die Viskosität wird mit einem fluoreszierenden molekularen Rotor gemessen.
Zusammenfassung
Die Viskosität ist eine wichtige physikalische Eigenschaft einer biologischen Membran, da sie einer der Schlüsselparameter für die Regulierung des morphologischen und physiologischen Zustands lebender Zellen ist. Es ist bekannt, dass Plasmamembranen von Tumorzellen signifikante Veränderungen in ihrer Zusammensetzung, Struktur und funktionellen Eigenschaften aufweisen. Zusammen mit dem fehlregulierten Stoffwechsel von Glukose und Lipiden helfen diese spezifischen Membraneigenschaften den Tumorzellen, sich an die feindliche Mikroumgebung anzupassen und Resistenzen gegen medikamentöse Therapien zu entwickeln. Hier demonstrieren wir den Einsatz der Fluoreszenz-Lebensdauer-Bildgebungsmikroskopie (FLIM) zur sequentiellen Abbildung des Zellstoffwechsels und der Plasmamembranviskosität in lebenden Krebszellkulturen. Metabolische Bewertungen werden durch den Nachweis der Fluoreszenz endogener metabolischer Cofaktoren durchgeführt, wie z. B. reduziertes Nicotinamid-Adenin-Dinukleotid NAD(P)H und oxidierte Flavine. Die Viskosität wird mit einem fluoreszierenden molekularen Rotor gemessen, einem synthetischen viskositätsempfindlichen Farbstoff, der stark von der Viskosität der unmittelbaren Umgebung abhängig ist. In Kombination ermöglichen uns diese Techniken, die Zusammenhänge zwischen dem Membranzustand und dem metabolischen Profil von Krebszellen besser zu verstehen und die durch die Chemotherapie induzierten Veränderungen sichtbar zu machen.
Einleitung
Die maligne Transformation von Zellen geht mit mehreren Veränderungen ihres morphologischen und physiologischen Zustands einher. Das schnelle und unkontrollierte Wachstum von Krebszellen erfordert eine grundlegende Reorganisation der biochemischen Signalwege, die für die Energieproduktion und Biosynthese verantwortlich sind. Die charakteristischen Kennzeichen des Krebsstoffwechsels sind eine erhöhte Glykolyserate, auch bei normalen Sauerstoffkonzentrationen (Warburg-Effekt), die Verwendung von Aminosäuren, Fettsäuren und Laktat als alternative Brennstoffe, eine hohe ROS-Produktion in Gegenwart eines hohen Antioxidantiengehalts und eine erhöhte Biosynthese von Fettsäuren 1,2. Es wird heute anerkannt, dass der Stoffwechsel von Krebszellen sehr flexibel ist, was es ihnen ermöglicht, sich an die ungünstige und heterogene Umgebung anzupassen und einen zusätzlichen Überlebensvorteil bietet3.
Ein veränderter Stoffwechsel unterstützt die spezifische Organisation und Zusammensetzung der Membranen von Tumorzellen. Das Lipidprofil der Plasmamembran in Krebszellen unterscheidet sich quantitativ von den nicht-krebsartigen Zellen. Die wichtigsten Veränderungen im Lipidom sind der erhöhte Spiegel an Phospholipiden wie Phosphatidylinositol, Phosphatidylserin, Phosphatidylethanolamin und Phosphatidylcholin, der verringerte Sphingomyelinspiegel, ein erhöhter Cholesterinspiegel und ein geringerer Grad an Ungesättigtheit von Fettsäuren, um nur einige zu nennen 4,5,6. Daher ändern sich die physikalischen Eigenschaften der Membran, wie z. B. die Membranviskosität, der Kehrwert der Fließfähigkeit. Da die Viskosität die Durchlässigkeit biologischer Membranen bestimmt und die Aktivität von membranassoziierten Proteinen (Enzymen, Transportern, Rezeptoren) steuert, ist ihre homöostatische Regulation für die Zellfunktion von entscheidender Bedeutung. Gleichzeitig ist die Modifikation der Viskosität durch die Anpassung des Membranlipidprofils wichtig für die Zellmigration/-invasion und das Überleben bei bedingten Veränderungen.
Die Fluoreszenz-Lebensdauer-Bildgebungsmikroskopie (FLIM) hat sich als leistungsstarker Ansatz für die nicht-invasive Beurteilung mehrerer Parameter in lebenden Zellen unter Verwendung endogener Fluoreszenz- oder exogener Sonden erwiesen7. FLIM wird üblicherweise auf einem Multiphotonen-Laserscanning-Mikroskop realisiert, das eine (sub)zelluläre Auflösung bietet. Ausgestattet mit dem zeitkorrelierten Einzelphotonenzählmodul (TCSPC) ermöglicht es zeitaufgelöste Messungen der Fluoreszenz mit hoher Genauigkeit8.
Die Untersuchung des zellulären Metabolismus mittels FLIM basiert auf der Fluoreszenzmessung von endogenen Cofaktoren von Dehydrogenasen, dem reduzierten Nicotinamid-Adenin-Dinukleotid (Phosphat) NAD(P)H und oxidierten Flavinen - Flavinadenindinukleotid FAD und Flavinmononukleotid FMN, die als Elektronenträger in einer Reihe von biochemischen Reaktionen wirken 7,9,10. Die detektierte Fluoreszenz von NAD(P)H stammt von NADH und seiner phosphorylierten Form, NADPH, da sie spektral nahezu identisch sind. Typischerweise passen Fluoreszenzzerfälle von NAD(P)H und Flavinen zu einer biexponentiellen Funktion. Im Fall von NAD(P)H wird die erste Komponente (~0,3-0,5 ns, ~70%-80%) auf seinen freien Zustand zurückgeführt, der mit der Glykolyse assoziiert ist, und die zweite Komponente (~1,2-2,5 ns, ~20%-30%) auf seinen proteingebundenen Zustand, der mit der mitochondrialen Atmung assoziiert ist. Im Fall von Flavinen kann die kurze Komponente (~0,3-0,4 ns, ~75%-85%) dem gequenchten Zustand von FAD und die lange Komponente (~2,5-2,8 ns, ~15%-25%) unstillbarem FAD, FMN und Riboflavin zugeordnet werden. Veränderungen in den relativen Niveaus der Glykolyse, Glutaminolyse, oxidativen Phosphorylierung und Fettsäuresynthese führen zu Veränderungen in den Kurz- und Langzeitfraktionen der Cofaktoren. Darüber hinaus spiegelt das Fluoreszenzintensitätsverhältnis dieser Fluorophore (das Redoxverhältnis) den zellulären Redoxstatus wider und wird auch als metabolischer Indikator verwendet. Obwohl das Redoxverhältnis im Vergleich zur Fluoreszenzlebensdauer eine einfachere Metrik darstellt, ist FLIM in Bezug auf die Datenerfassung vorteilhaft für die Abschätzung von NAD(P)H und FAD, da die Fluoreszenzlebensdauer eine intrinsische Eigenschaft des Fluorophors ist und fast nicht von Faktoren wie Anregungsleistung, Photobleichung, Fokussierung, Lichtstreuung und -absorption beeinflusst wird, insbesondere in Geweben. im Gegensatz zur Emissionsintensität.
Eine der bequemen Möglichkeiten, die Viskosität in lebenden Zellen und Geweben auf mikroskopischer Ebene abzubilden, beruht auf der Verwendung von fluoreszierenden molekularen Rotoren, kleinen synthetischen viskositätsempfindlichen Farbstoffen, bei denen die Fluoreszenzparameter stark von der lokalen Viskositätabhängen 11,12. In einem viskosen Medium erhöht sich die Fluoreszenzlebensdauer des Rotors aufgrund der Verlangsamung der intramolekularen Verdrehung oder Rotation. Unter den molekularen Rotoren eignen sich die Derivate von Bordipyrromethen (BODIPY) gut für die Messung der Viskosität in biologischen Systemen, da sie einen guten dynamischen Bereich der Fluoreszenzlebensdauer im physiologischen Bereich der Viskositäten, Temperaturunabhängigkeit, monoexponentielle Fluoreszenzzerfälle, die eine einfache Dateninterpretation ermöglichen, eine ausreichende Wasserlöslichkeit und eine geringe Zytotoxizität aufweisen13,14. Die quantitative Bestimmung der Mikroviskosität mit BODIPY-basierten Rotoren und FLIM wurde bereits an Krebszellen in vitro, multizellulären Tumorsphäroiden und Maustumoren in vivo nachgewiesen 15,16.
Hier präsentieren wir eine detaillierte Beschreibung der sequentiellen Sondierungsmethoden zur Untersuchung des zellulären Stoffwechsels und der Plasmamembranviskosität in Krebszellen in vitro mittels FLIM. Um eine Kontamination der relativ schwachen endogenen Fluoreszenz mit der Fluoreszenz des BODIPY-basierten Rotors zu vermeiden, wird die Bildgebung derselben Zellschicht nacheinander durchgeführt, wobei die Fluoreszenz von NAD(P)H und FAD zuerst abgebildet wird. Die Fluoreszenzlebensdauer der Cofaktoren wird im Zytoplasma gemessen, und die Fluoreszenzlebensdauer des Rotors wird in den Plasmamembranen der Zellen durch manuelle Auswahl entsprechender Zonen als interessierende Regionen gemessen. Das Protokoll wurde angewendet, um den Stoffwechselzustand und die Viskosität für verschiedene Krebszelllinien zu korrelieren und die Veränderungen nach der Chemotherapie zu beurteilen.
Das Protokoll für die FLIM-Probenvorbereitung unterscheidet sich nicht von dem für die konfokale Fluoreszenzmikroskopie. Nach der Datenerfassung besteht die Hauptaufgabe darin, die Fluoreszenzlebensdauer aus den Rohdaten zu extrahieren. Die Leistungsfähigkeit des Protokolls wird anhand von HCT116 (humanes kolorektales Karzinom), CT26 (murines Dickdarmkarzinom), HeLa (humanes Gebärmutterhalskarzinom) und huFB (humane Hautfibroblasten) Zellen demonstriert.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Beschreibung des minimalen Setups zum Ausführen von FLIM
- Um dieses Experiment durchzuführen, stellen Sie sicher, dass der erforderliche Aufbau verfügbar ist: ein inverses konfokales Mikroskop, ein gepulster Laser, typischerweise ein ps oder fs, mit dem Synchronisationssignal, ein schneller Photonenzähldetektor (Zeitantwort 150 ps) und eine Photonenzählelektronik, verfügbare Ausgangs- und Eingangsanschlüsse für den Detektor bzw. den Laser am Mikroskop, die Scan-Taktimpulse vom Mikroskop-Scan-Controller, der Scankopf des Mikroskops mit den Laserstrahlkombinierern und den wichtigsten dichroitischen Strahlteilern, die für die Wellenlänge des für FLIM verwendeten Lasers geeignet sind.
- Wenn für FLIM eine Zwei-Photonen-Anregung verwendet wird, stellen Sie sicher, dass das Mikroskop den NDD-Anschluss enthält.
- Bei Studien an Säugetierzellen, insbesondere bei Langzeitexperimenten, ist darauf zu achten, dass ein CO2 -Inkubator auf der gewünschten Temperatur gehalten wird.
HINWEIS: Informationen zu dem in diesem Experiment verwendeten System finden Sie in der Materialtabelle.
2. Vorbereitung der Zellen für die Mikroskopie
- Züchten Sie die Zellen routinemäßig in einem Inkubator bei 37 °C mit 5 % CO2 und einer feuchten Atmosphäre.
- Für die Mikroskopie bereiten Sie die Zellsuspension in einem vollständigen Kulturmedium in einer Konzentration von 1 x 106 Zellen/ml vor.
HINWEIS: Die Zellkonzentrationen und Medienbedingungen sind zellabhängig. Die Anzahl der für die Aussaat verwendeten Zellen und die Inkubationszeit sollten angepasst werden, um eine Konfluenz von 70 % bis 80 % in der mikroskopischen Schale zu erreichen. - Die Zellen werden mit einer automatischen 200-μl-Pipette auf 35-mm-Zellkulturschalen mit Glasboden (1 x 105 Zellen in 100 μl pro Schale) ausgesät.
HINWEIS: Verwenden Sie gerasterte Glasbodenschalen für die Aussaat der Zellen, um Zellen in den gleichen mikroskopischen Sichtfeldern in der Dynamik zu überwachen.- Achten Sie bei der manuellen Aussaat darauf, dass die Pipettenspitze weder den Boden noch die Seiten der Schale zerkratzt, um eine Beschädigung des Bodens zu vermeiden.
- Stellen Sie die Schale in den CO2 -Inkubator (37 °C, 5 % CO2, feuchte Atmosphäre) und inkubieren Sie die Zellen für 24 h.
- Nach 24 h nehmen Sie die Schale aus dem Inkubator und überprüfen Sie die Morphologie und Konfluenz der Zellen unter dem Lichtmikroskop. Wenn die Zellen keine Konfluenz von etwa 80 % erreicht haben, inkubieren Sie weitere 24 Stunden.
- Entfernen Sie das alte Medium vorsichtig mit einer automatischen 1.000-μl-Pipette aus der Schale und fügen Sie 2 mL DMEM-Medium ohne Phenolrot (z. B. DMEM Life oder FluoroBrite) hinzu.
HINWEIS: Für die Bildgebung können verschiedene Nährmedien verwendet werden. Vermeiden Sie Phenolrot im Medium, wenn Sie Zellen für die Mikroskopie verwenden. - Stellen Sie die Schale für 60-120 Minuten in den Inkubator, um die Anpassung der Zellen zu ermöglichen.
3. FLIM von metabolischen Cofaktoren
- Stellen Sie die Glasbodenschale mit den Küvetten (aus Schritt 2.7) auf den Mikroskoptisch.
- Klicken Sie in der Software des Laser-Scanning-Mikroskops (z. B. ZEN - ZEISS Efficient Navigation) auf die Registerkarte "Lokalisieren " und dann auf "Transmission Light (TL"), um das Licht einzuschalten.
- Finden Sie die Fokusebene der Probe, indem Sie durch das Okular auf die mittlere Schichtebene der Zellen schauen, wo ein Quadrat maximal von Zellen eingenommen wird (bei 40-facher Vergrößerung).
- Klicken Sie auf die Schaltfläche OFF , um das Licht auszuschalten.
- Öffnen Sie die Registerkarte Akquisition . Um Bilder der Transmissions- und Autofluoreszenzintensität von endogenen NAD(P)H zu erhalten, geben Sie die folgenden Einstellungen ein: Anregungswellenlänge: Zwei-Photonen-Modus 750 nm, Registrierungsbereich: 450-490 nm, Laserleistung: 5% (~6 mW), Bildgröße: 1024 x 1024 Pixel.
HINWEIS: Die Wahl der Anregungswellenlänge und des Registrierungsbereichs basiert auf den spektralen Eigenschaften (maximale Anregung und maximale Emission) von NAD(P)H17.- Verwenden Sie für die Bildaufnahme ein Öl-Immersionsobjektiv С Plan-Apochromat 40x/1.3 NA.
- Drücken Sie die Snap-Taste und speichern Sie das Bild im ZEN-Format.
- Um die Transmissions- und Autofluoreszenzintensitätsbilder von FAD zu erhalten, ändern Sie die Anregungswellenlänge auf 900 nm. Legen Sie den Registrierungsbereich fest: 500-550 nm, die Laserleistung: 9 % (~6 mW) und die Bildgröße: 1024 x 1024 Pixel.
HINWEIS: Die Wahl der Anregungswellenlänge und des Registrierungsbereichs basiert auf den spektralen Eigenschaften (maximale Anregung und maximale Emission) von FAD18.- Verwenden Sie für die Bildaufnahme ein Öl-Immersionsobjektiv С Plan-Apochromat 40x/1,3 NA.
- Drücken Sie die Snap-Taste und speichern Sie das Bild im ZEN-Format.
- Für NAD(P)H stellen Sie die Parameter wie in Schritt 3.5 in der Software für das Laser-Scanning-Mikroskop beschrieben ein. Ändern Sie die Bildgröße in 256 x 256 Pixel.
- Geben Sie die folgenden Parameter in das Menü von SPCM (Single Photon Counting Modules) ein. Betriebssoftware des FLIM-Moduls: Sammelzeit: 60 s; TAC-Bereich: 5,00E-8; CFD-Limit-Tief: -29,41; ADC-Auflösung: 256, Bildgröße: 256 x 256 Pixel.
- Scannen Sie die Probe 60 s lang, stoppen Sie den Scanvorgang und speichern Sie das erhaltene FLIM-Bild von NAD(P)H.
- Überprüfen Sie die erhaltenen FLIM-Daten. Öffnen Sie dazu die Rohdaten in der Bildsoftware, wählen Sie ein Pixel im Zytoplasma der Zelle aus, indem Sie den Cursor darüber bewegen und analysieren Sie den Fluoreszenzzerfall in diesem Pixel. Die Pixelintensität sollte bei Binning 1 ≥3.000 Photonen pro Abklingkurve betragen.
HINWEIS: Wenn die Anzahl der Photonen unter 3.000 liegt, erhöhen Sie die Laserleistung oder die Bilderfassungszeit, während Sie die Morphologie der Zellen und die Photonenzählrate steuern. Wenn der Abfall der Zählrate 10 % des Ausgangswerts übersteigt, findet in der Regel Photobleaching statt. - Für FAD stellen Sie die Parameter wie in Schritt 3.7 beschrieben in der Software des Laser-Scanning-Mikroskops ein. Ändern Sie die Bildgröße in 256 x 256 Pixel.
- Geben Sie die folgenden Parameter in das Menü von SPCM (Single Photon Counting Modules) ein. Betriebssoftware des FLIM-Moduls: Sammelzeit: 60 s; TAC-Bereich: 5,00E-8; CFD-Limit-Tief: -29,41; ADC-Auflösung: 256, Bildgröße: 256 x 256 Pixel.
- Scannen Sie die Probe 60 s lang. Beenden Sie den Scanvorgang und speichern Sie das erhaltene FLIM-Bild von FAD.
HINWEIS: Die in den Schritten 3.10 und 3.15 angegebenen Parameter sind spezifisch für die verwendete Elektronik und den verwendeten Detektor. - Überprüfen Sie die erhaltenen Daten wie in Schritt 3.12 beschrieben.
- Wiederholen Sie die Schritte 3.5 bis 3.16, um FLIM-Bilder aus verschiedenen Sichtfeldern aufzunehmen.
4. Färbung von Zellen mit dem fluoreszierenden molekularen Rotor
HINWEIS: Die Zellen werden in der fluoreszierenden molekularen Rotorlösung abgebildet, ohne bei Raumtemperatur (~20 °C) gewaschen zu werden, um die Internalisierung des Rotors zu verlangsamen. Die Viskosität der Membran ist temperaturabhängig, wie in unseren früheren Arbeitengezeigt wurde 19,20. Der temperierte Tisch des Mikroskops sollte im Voraus, d.h. vor dem Aufsetzen des Rotors auf die Küvetten, ausgeschaltet werden. Für unseren Aufbau dauert die Abkühlung der Bühne ca. 10 min.
- Bereiten Sie eine allgemeine Stammlösung des fluoreszierenden molekularen Rotors BODIPY 2 (Stamm 1, 25,7 mM) vor.
- Öffnen Sie BODIPY 2 in einer sterilen Umgebung und wiegen Sie ca. 2 mg mit einer genauen Waage. Legen Sie es vorsichtig in ein Mikrozentrifugenröhrchen.
- Verwenden Sie eine automatische 20-μl-Pipette, um 3 μl eines geeigneten Lösungsmittels (z. B. DMSO) hinzuzufügen.
- Sobald sich die Probe vollständig in DMSO aufgelöst hat, fügen Sie 297 μl steriles PBS hinzu und mischen Sie es gründlich mit einer automatischen 200-μl-Pipette.
HINWEIS: Lagern Sie die Stammlösung in dunkler Verpackung bei +4 °C im Kühlschrank. Einmal aufgehängt, kann es mehrere Monate im Kühlschrank aufbewahrt werden.
- Bereiten Sie einen Stamm 2 (8,9 mM) vor, indem Sie 25 μl der allgemeinen Stammlösung (Stamm 1) in ein Mikrozentrifugenröhrchen geben, gefolgt von 48 μl sterilem PBS. Vorsichtig mit einer automatischen 200-μl-Pipette mischen.
HINWEIS: Verwenden Sie Rohling 2, um das endgültige Färbematerial vorzubereiten, das für die Zellfärbung verwendet wird, da eine mikromolare Konzentration erforderlich ist. - Ersetzen Sie das Nährmedium in der Schale (aus Schritt 3.1) vorsichtig durch eiskalte Hank-Lösung ohne Ca2+/Mg2+ und inkubieren Sie die Zellen 3 Minuten lang bei +4 °C.
HINWEIS: Die Verwendung einer eiskalten Lösung und die Inkubation bei +4 °C verlangsamen die Internalisierung des molekularen Rotors, und die lokale Färbung der Membran hält 20-30 Minuten an. - Bereiten Sie die endgültige Färbelösung mit 4,5 μM BODIPY 2 vor, indem Sie 1 μl des Stock 2 zu 999 μl eiskalter Hank's Lösung oder PBS hinzufügen.
HINWEIS: Die Konzentration von BODIPY 2 in der endgültigen Färbelösung kann auf ~10 μM erhöht werden, ohne dass toxische Auswirkungen auf die Zellen auftreten, was zu einer effizienteren Färbung und einer größeren Anzahl gesammelter Photonen führt. Bei höheren Konzentrationen kann es zu einer Überlastung des FLIM-Detektors kommen. - Die Hank-Lösung aus der Zellkulturschale absaugen und durch eiskalte 4,5 μM Lösung von BODIPY 2 ersetzen. Die Zellen werden in BODIPY 2-Lösung ohne Waschen abgebildet.
5. FLIM des fluoreszierenden molekularen Rotors in Zellen
HINWEIS: Führen Sie die FLIM des fluoreszierenden molekularen Rotors immer nach der FLIM der metabolischen Cofaktoren durch, da sich das Fluoreszenzspektrum von BODIPY 2 mit der Emission der endogenen Cofaktoren NAD(Р)H und FAD 12,17,18 überlappt.
- Übertragen Sie die Schale mit den gefärbten Zellen zur Bildgebung auf den Mikroskoptisch (~20 °C).
- Stellen Sie in der Software des Laser-Scanning-Mikroskops die folgenden Parameter für den Ein-Photonen-Modus ein: Anregung bei einer Wellenlänge von 488 nm mit einem Argon-Ionen-Laser, Laserleistung 1%-2%, Registrierungsbereich 500-550 nm Wellenlänge.
- Verwenden Sie für die Bildaufnahme ein Öl-Immersionsobjektiv С Plan-Apochromat 40x/1,3 NA.
- Drücken Sie die Live-Taste . Starten Sie den Scanvorgang und verwenden Sie die XY- und Z-Positionierung durch einen integrierten motorisierten Tisch, stellen Sie den Fokus ein und erhalten Sie Bilder von Transmission und Fluoreszenzintensität von Zellen in einem Vorschaufenster. Speichern Sie die erhaltenen Bilder, falls erforderlich.
- Überprüfen Sie auf dem überlappenden Transmissions- und Fluoreszenzbild, ob die Fluoreszenz des Rotors von der erwarteten Stelle (Plasmamembran der Zelle) kommt.
- Geben Sie die folgenden Parameter in das Menü der SPCM-Software des FLIM-Moduls ein: Erfassungszeit: 60 s; TAC-Bereich: 5,00E-8; CFD-Limit-Tief: -29,41; ADC-Auflösung: 256, Bildgröße: 256 x 256 Pixel.
HINWEIS: Abhängig von der Systemkonfiguration und den für FLIM verwendeten Detektoren können die Parameter der Bildaufnahme variieren. - Stellen Sie den Ti:Saphir-Laser des Mikroskops auf eine Wellenlänge von 850 nm und die Laserleistung auf 1%-2% für Zwei-Photonen-FLIM ein.
- Wählen Sie in der Software für das Laserscanning-Mikroskop die Registerkarte Kontinuierlich aus und drücken Sie dann in der SPCM-Software auf Start . Scannen Sie die Probe 60 s lang, stoppen Sie den Scanvorgang und speichern Sie das erhaltene FLIM-Bild.
- Überprüfen Sie die erhaltenen FLIM-Daten. Laden Sie dazu die Rohdaten in die FLIM-Datenanalyse SPCImage-Software, wählen Sie ein Pixel in der Zellmembran aus, indem Sie den Mauszeiger darüber bewegen, und analysieren Sie den Fluoreszenzzerfall in diesem Pixel. Die Pixelintensität sollte bei einer angemessenen Erfassungszeit (60-120 s) ≥5.000 pro Zerfall (möglicherweise einschließlich Binning) betragen.
- Wiederholen Sie die Schritte 5.4 bis 5.8, um FLIM-Bilder von Zellen aus verschiedenen Sichtfeldern aufzunehmen.
HINWEIS: FLIM-Messungen von lebenden Zellen, die mit BODIPY 2 gefärbt wurden, sollten auf ~30 min nach Zugabe von BODIPY 2 begrenzt werden.
6. Datenanalyse
- Fluoreszenzintensitätsanalyse: Redoxverhältnis
- Öffnen Sie Bilder der Fluoreszenzintensität von NAD(P)H und FAD mit ImageJ.
- Markieren Sie einen zellenfreien Bereich im NAD(P)H-Bild mit einer Kreis- oder Quadratoption. Klicken Sie auf Messen und dann auf Subtrahieren (wählen Sie Prozess im Hauptfenster und dann Mathematik und Subtrahieren), um den erhaltenen Wert des Hintergrundsignals zu subtrahieren.
- Wiederholen Sie Schritt 6.1.2 für das FAD-Bild.
- Erhalten Sie ein Bild des Redoxverhältnisses, indem Sie die FAD-Fluoreszenzintensität durch die NAD(P)H-Fluoreszenzintensität dividieren. Wählen Sie dazu im Hauptfenster Prozess und dann Bildrechner und Division aus. Aktivieren Sie das Kontrollkästchen Neues Fenster erstellen und drücken Sie dann OK.
- Speichern Sie das Bild im TIFF-Format.
- Um das Redoxverhältnis zu berechnen, wählen Sie die Region des Zytoplasmas in der jeweiligen Zelle auf dem TIFF-Bild aus und drücken Sie die Taste M . Wiederholen Sie den Vorgang für alle interessierenden Zellen.
- Importieren Sie die Messung in ein Tabellendokument.
HINWEIS: Alternativ können die Fluoreszenzintensitäten von NAD(P)H und FAD in Zellen mit der Standardsoftware des Mikroskops gemessen werden, und das Redoxverhältnis kann durch Division dieser Werte in der Tabellenkalkulationssoftware ermittelt werden.
- FLIM-Datenanalyse: Stoffwechsel
- Importieren Sie das FLIM-Image von NAD(P)H in die SPCImage-Software.
- Wenden Sie eine biexponentielle Zerfallsanpassung auf das Bild an, indem Sie 2 in den Abschnitt Komponenten einfügen.
- Korrigieren Sie den Offset-Parameter , indem Sie das entsprechende Kontrollkästchen in der SPCImage-Software aktivieren.
- Gehen Sie zu Optionen , und wählen Sie Modell aus. Verwenden Sie ein unvollständiges multiexponentielles Anpassungsmodell und eine Anpassungsmethode MLE.
- Passen Sie das Binning an, um Pixelintensitäten von ≥5000 Photonen pro Abklingkurve zu erreichen.
- Prüfen Sie den Wert χ2. χ2≤ 1,20 gibt an, dass das verwendete Modell eine angemessene Anpassung bietet.
- Berechnen Sie das Histogramm der Fluoreszenzlebensdauer in jedem Bild, indem Sie auf das obere Menü Berechnen und dann auf Zerfallsmatrix klicken.
- Wählen Sie den Bereich im Zytoplasma der spezifischen Zelle als interessierende Region aus.
- Analysieren Sie die Komponenten für die kurze und lange Lebensdauer (τ1 bzw.τ 2) und die relativen Amplituden der Lebensdauerkomponenten (a1 und a2, wobei a1 + a2 = 100 %) verwendet wird, indem Sie die Option Farbe verwenden.
- Exportieren Sie die Messungen in eine Tabellenkalkulationssoftware.
- Wiederholen Sie die Schritte 6.2.8 bis 6.2.10 für jede gewünschte Zelle.
- Wiederholen Sie die Schritte 6.2.1 bis 6.2.11 für das FAD-Image.
- FLIM-Datenanalyse: Viskosität
- Importieren Sie das FLIM-Bild in die FLIM-Datenanalyse-SPCImage-Software.
- Entfernen Sie die Markierung im Feld Streuung .
- Geben Sie 1 in den Abschnitt Komponenten ein, da der Fluoreszenzzerfall des Rotors zu einem monoexponentiellen Modell passen sollte.
- Passen Sie das Binning an, um eine Pixelintensität von ≥5000 Photonen pro Abklingkurve zu erreichen.
- Überprüfen Sie den χ2-Wert in der Plasmamembran. Ein Wert von χ2≤ 1,20 gibt an, dass das verwendete Modell eine angemessene Anpassung bietet. Im Fall von χ2≥ 1,20 ist die monoexponentielle Approximation nicht anwendbar, solche Daten können auf die Farbstoffaggregation hinweisen und sollten verworfen werden. Die Aggregation macht es unmöglich, die Kalibrierkurven zu verwenden, und führt zu falschen Viskositätsschätzungen.
HINWEIS: Biexponentielle Zerfälle können auf eine Aggregation hinweisen. An einem Mikroskop mit FLIM-Modul mit variablen Filtern kann dies durch Testen der Monomer- und aggregatspezifischen Emissionswellenlängenbereiche von 500-550 nm und 580-650 nm nachgewiesen werden, wie in Referenz21 beschrieben. - Erzeugen Sie das Histogramm der Fluoreszenzlebensdauer τ für jedes Bild, indem Sie auf das obere Menü Berechnen und dann auf Zerfallsmatrix klicken.
- Wählen Sie den Bereich der Plasmamembran einer einzelnen Zelle mit monoexponentiellem Zerfall, χ2≤ 1,20, mit der Option ROI aus.
- Exportieren Sie den Wert der Fluoreszenzlebensdauer in eine Tabellenkalkulationssoftware.
- Wiederholen Sie die Schritte 6.3.7 bis 6.3.8 für jede gewünschte Zelle.
- Umrechnung der experimentell gemessenen Lebensdauern von BODIPY 2 (in ns) in Viskositätswerte (in cP) unter Verwendung der folgenden Gleichung (die zuvor auf der Grundlage des Kalibrierdiagramms von BODIPY in Methanol/Glycerin-Gemischen erhalten wurde):
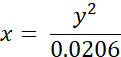
wobei x - Viskosität (in cP), y - Fluoreszenzlebensdauer τ (in ns).
HINWEIS: IRF (Instrument Response Function) ist ein wichtiger Bestandteil der FLIM-Anpassung. In SPCImage wird die IRF automatisch aus der steigenden Flanke der Fluoreszenzzerfallskurven berechnet. In der Zwischenzeit kann die IRF mit einer nicht fluoreszierenden Probe aufgezeichnet werden, z. B. Keramik, oder einer Probe, die ein SHG-Signal (Second Harmonic Generation) erzeugt, z. B. Kollagen, Harnstoffkristalle oder Zucker. Die Verwendung der aufgezeichneten IRFs wird nicht empfohlen, wenn es eine Option gibt, diese in der Software zu berechnen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Unter Verwendung des hier beschriebenen Protokolls haben wir die metabolischen Cofaktoren und die mikroskopische Membranviskosität in lebenden kultivierten Zellen mit FLIM visualisiert. Die Messungen wurden an verschiedenen Krebszelllinien durchgeführt - dem humanen kolorektalen Karzinom HCT116, dem murinen Dickdarmkarzinom CT26, dem menschlichen Gebärmutterhalskrebs HeLa Kyoto und den menschlichen Hautfibroblasten huFB.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Dieses Protokoll veranschaulicht die Möglichkeiten von FLIM für die multiparametrische, funktionelle und biophysikalische Analyse von Krebszellen. Die Kombination der optischen metabolischen Bildgebung auf Basis endogener Fluoreszenz und der Messung der Plasmamembranviskosität mittels exogener Markierung mit einem fluoreszierenden molekularen Rotor ermöglicht es uns, die Zusammenhänge zwischen diesen beiden Parametern in lebenden Krebszellen in einer Zellkultur zu charakterisieren u...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Entwicklung des Protokolls für die metabolische Bildgebung wurde vom Gesundheitsministerium der Russischen Föderation unterstützt (Regierungsauftrag, Registrierungsnummer АААА-А20-120022590098-0). Die Untersuchung der Viskosität wurde von der Russischen Wissenschaftsstiftung unterstützt (Projekt-Nr. 20-14-00111). Die Autoren danken Anton Plekhanov (PRMU) für seine Hilfe bei der Videoproduktion.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
Referenzen
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237(2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203(2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , Springer. (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671(2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. Dmitriev, R. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097(2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986(2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004(2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005(2014).
- Lee, D. -H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002(2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451(2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746(2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911(2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936(2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432(2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503(2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410(2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271(2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398(2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804(2012).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten