Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein reporterbasierter zellulärer Assay zur Überwachung der Spleißeffizienz
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen Minigene-Reporter-Assay zur Überwachung der Auswirkungen von 5'-Spleißstellenmutationen auf das Spleißen und entwickelt Suppressor U1 snRNA zur Rettung der mutationsinduzierten Spleißhemmung. Die Reporter- und Suppressor-U1-snRNA-Konstrukte werden in HeLa-Zellen exprimiert, und das Spleißen wird durch Primer-Extension oder RT-PCR analysiert.
Zusammenfassung
Während der Genexpression beinhaltet der entscheidende Schritt des Pre-mRNA-Spleißens die genaue Erkennung von Spleißstellen und die effiziente Montage von spleißosomalen Komplexen, um Exons zu verbinden und Introns vor dem zytoplasmatischen Export der reifen mRNA zu entfernen. Die Spleißeffizienz kann durch das Vorhandensein von Mutationen an Spleißstellen, den Einfluss transaktiver Spleißfaktoren oder die Aktivität von Therapeutika verändert werden. Hier beschreiben wir das Protokoll für einen zellulären Assay, der zur Überwachung der Spleißeffizienz eines bestimmten Exons angewendet werden kann. Der Assay verwendet einen anpassungsfähigen Plasmid-kodierten 3-Exon/2-Intron-Minigen-Reporter, der in Säugetierzellen durch transiente Transfektion exprimiert werden kann. Nach der Transfektion wird die gesamte zelluläre RNA isoliert, und die Effizienz des Exon-Spleißens in der Reporter-mRNA wird entweder durch Primer-Extension oder semi-quantitative Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-PCR) bestimmt. Wir beschreiben, wie die Auswirkungen von krankheitsassoziierten 5'-Spleißstellenmutationen bestimmt werden können, indem sie in den Reporter eingeführt werden; und wie die Unterdrückung dieser Mutationen durch Co-Transfektion mit einem U1-Konstrukt für kleine kernare RNA (snRNA) erreicht werden kann, das kompensatorische Mutationen in seiner 5'-Region trägt, die mit den 5'-Spleißstellen an Exon-Intron-Verbindungen in prä-mRNAs übereinstimmt. Somit kann der Reporter für das Design therapeutischer U1-Partikel verwendet werden, um die Erkennung von mutierten 5'-Spleißstellen zu verbessern. Das Einfügen von cis-wirkenden regulatorischen Stellen, wie Spleißverstärker- oder Schalldämpfersequenzen, in den Reporter kann auch verwendet werden, um die Rolle von U1 snRNP in der Regulierung zu untersuchen, die durch einen bestimmten alternativen Spleißfaktor vermittelt wird. Schließlich können Reporter-exprimierende Zellen mit kleinen Molekülen inkubiert werden, um die Wirkung potenzieller Therapeutika auf konstitutives prä-mRNA-Spleißen oder auf Exons mit mutierten 5'-Spleißstellen zu bestimmen. Insgesamt kann der Reporter-Assay angewendet werden, um die Spleißeffizienz unter einer Vielzahl von Bedingungen zu überwachen, um grundlegende Spleißmechanismen und Spleiß-assoziierte Krankheiten zu untersuchen.
Einleitung
Pre-mRNA-Spleißen ist ein wesentlicher Verarbeitungsschritt, der nicht-kodierende Introns entfernt und exakt Ligaten kodiert, die Exons kodieren, um reife mRNA zu bilden. Die Erkennung von Konsenssequenzen an Exon-Intron-Übergängen, die als 5′-Spleißstelle und 3′-Spleißstelle bezeichnet werden, durch Komponenten der Spleißmaschinerie leitet den Spleißprozess ein. Das U1 Small Nuclear Ribonucleoprotein (snRNP) erkennt die 5′-Spleißstelle durch Basenpaarung der U1-snRNA zur prä-mRNA1. Genetisch vererbte Mutationen, die 5′-Spleißstellensequenzen verändern, sind mit vielen Krankheiten assoziiert2,3. Es wird vorhergesagt, dass der Verlust des Basepairings von U1-snRNA mit den mutierten 5'-Spleißstellen ein aberrantes Spleißen verursacht, das die Translation des betroffenen Transkripts beeinträchtigen kann. Ein potenzieller therapeutischer Ansatz zur Korrektur der Spleißdefekte beinhaltet die Unterdrückung von Mutationen durch modifizierte U1-snRNA, die kompensatorische Nukleotidänderungen in ihrer 5'-Region trägt, die mit der 5'-Spleißstelle übereinstimmt. Solche modifizierten U1-snRNAs, auch als exonspezifische U1-snRNAs bezeichnet, haben sich als wirksam bei der Umkehrung von Spleißdefekten erwiesen, was zu einer erhöhten Proteinexpression aus der geretteten mRNA4,5,6,7,8 führt.
Hier beschreiben wir den U1 snRNP-Komplementierungsassay, einen reporterbasierten zellulären Spleißassay, der die Beurteilung der Wirkung von 5′-ss-Mutationen auf das Spleißen eines Exons ermöglicht und auch für die Entwicklung modifizierter U1-snRNAs verwendet werden kann, um die Rettung des Exon-Einschlusses zu ermöglichen. Wir bieten auch Protokolle zur Überwachung der gespleißten Reportertranskripte durch Primer-Erweiterung und RT-PCR und zur Bestimmung der Expression von modifizierten U1-snRNAs durch Primer-Erweiterung und RT-qPCR.
Protokoll
1. Reagenzien und Puffer
HINWEIS: Alle Sterilisationen mit Vakuumfiltern sollten mit einer 0,2 μm Polyethersulfon (PES)-Membran in einer Biosicherheitseinrichtung durchgeführt werden.
- Bereiten Sie RNase-freies Wasser vor, indem Sie 1,0 ml Diethylpyrocarbonat (DEPC) zu 1,0 l deionisiertem Wasser hinzufügen, mindestens 1 Stunde bei Raumtemperatur (RT) mischen, zweimal autoklavieren und dann vor Gebrauch zu RT abkühlen.
- Bereiten Sie Dulbeccos Modified Eagle Medium (DMEM) vor, indem Sie eine Packung DMEM-Pulver (13,4 g), 3,7 g Natriumbicarbonat, 100 ml fötales Rinderserum (FBS), Penicillin und Streptomycin zu ~ 800 ml sterilem deionisiertem Wasser mischen. Die Endkonzentration von Penicillin und Streptomycin in DMEM sollte 50 U/ml bzw. 50 μg/ml betragen. Stellen Sie den pH-Wert auf 7,4 ein und erhöhen Sie dann das Volumen mit sterilem entionisiertem Wasser auf 1,0 l. Sterilisieren durch Filtration und lagern bei 4 °C.
- 10x phosphatgepufferte Kochsalzlösung (10x PBS) durch Zugabe von 25,6 g Dinatriumhydrogenheptahydrat (Na2HPO4· 7H2O), 2 g Kaliumdihydrogenphosphat (KH2PO4), 2 g Kaliumchlorid (KCl) und 80 g Natriumchlorid (NaCl) bis 800 ml deionisiertes Wasser. Mischen, um sich aufzulösen und das Volumen auf 1,0 l aufzufüllen. Sterilisieren Sie durch Filtration und lagern Sie es bei 4 ° C.
- 0,5 M Ethylendiamintetraessigsäure (EDTA) durch Lösen von 186,1 g Na2•EDTA•2H2O in ~800 mL entionisiertes Wasser auflösen. Stellen Sie den pH-Wert auf 8,0 ein und erhöhen Sie dann das Volumen mit sterilem entionisiertem Wasser auf 1,0 l. Sterilisieren durch Filtration und lagern bei 4 °C.
- Bereiten Sie 1x Trypsin-EDTA-Lösung vor, indem Sie 100 ml 10x Trypsin (2,5%), 2 ml 0,5 M EDTA mischen und 1x PBS bis zu 1,0 l sterilisieren. Bei 4 °C für 1-2 Wochen lagern oder bei -20 °C für den Langzeitgebrauch einfrieren.
- Bereiten Sie 2x Formamid-DNA/RNA-Beladungsfarbstoff vor, indem Sie 14,4 ml Formamid und 0,6 ml 0,5 M EDTA für ein Endvolumen von 15 ml mischen. Bromphenolblau und Xylolcyanolpulver in einer Endkonzentration von 0,02 % zugeben und bei 4 °C lagern.
HINWEIS: Formamid ist giftig und ätzend. Weitere Sicherheitsempfehlungen finden Sie in den Sicherheitsdatenblättern. - Bereiten Sie 5x Tris/Borate/EDTA (5x TBE) Puffer vor, indem Sie 54,0 g Tris-Base, 27,5 g Borsäure und 20 ml 0,5 M EDTA in ~800 mL deionisiertes Wasser mischen. Mischen, um sich aufzulösen und das Volumen mit entionisiertem Wasser auf 1,0 l aufzufüllen.
- Harnstoff-Polyacrylamid-Gelelektrophorese-Lösung (Harnstoff-PAGE) durch Mischen von 200 ml 5x FSME, 250 mL 40% 19:1 Bis/Acrylamid und 450,5 g Harnstoff herstellen. Dann deionisiertes Wasser bis zu 1,0 l hinzufügen. Mischen Sie, bis sich die Zutaten vollständig aufgelöst haben, sterilisieren Sie dann durch Filtration und lagern Sie es bei 4 ° C in einer braunen Glasflasche.
HINWEIS: Bis/Acrylamid ist giftig. Lesen Sie die Sicherheitsdatenblätter für weitere Sicherheitsverfahren. - 10% Ammoniumpersulfat (APS) durch Lösen von 1 g APS in 10 ml entionisiertem Wasser herstellen und bei 4 °C lagern.
2. Cotransfektion von HeLa-Zellen mit dem Reporter und U1-snRNA-Plasmiden
HINWEIS: Die Transfektion von Hela-Zellen muss unter sterilen Bedingungen in einer biologischen Sicherheitswerkbank durchgeführt werden. Die Außenfläche aller Materialien muss mit 70% Ethanol besprüht werden, bevor sie in die biologische Sicherheitswerkbank eingebracht wird.
- Halten Sie Hela-Zellen in DMEM in einem 37 °C-Inkubator mit 5% CO2 , indem Sie alle 2-3 Tage passieren, wenn die Zellen etwa 80-90% konfluent sind.
- Zur Weitergabe von HeLa-Zellen das verbrauchte Medium absaugen und dann die Zellen mit 3 ml 0,25% Trypsin, das 1 mM EDTA bei 37 °C enthält, für 3 min inkubieren.
- Nach der Inkubation werden 7 ml frisches DMEM zugegeben. Die Zellsuspension in ein 10-ml-Röhrchen geben, 5 min lang mit 1.000 x g zentrifugieren.
- Resuspendieren Sie das Zellpellet in 10 ml frischem DMEM und schlagen Sie die Zellen dann auf eine neue Gewebekulturschale bei 20% Konfluenz.
- Für vorübergehende Transfektionen zählen Sie Hela-Zellen mit einem sauberen Hämozytometer-Objektträger und bereiten sie eine Suspension mit einer Dichte von 2,5 x 105 Zellen / ml vor.
- 1,0 ml 2,5 x 105 Zellen/ml Hela-Zellsuspension in jede Vertiefung einer 12-Well-Platte geben und über Nacht bei 37 °C inkubieren.
- Am nächsten Tag aspirieren Sie das verbrauchte Medium und fügen Sie 0,8 ml frisches DMEM mit Serum hinzu.
- Bereiten Sie Lösung I vor, indem Sie 0,2 μg Dup51- oder Dup51p-Reporterplasmid, 1,8 μg pcDNA-, pNS6U1- oder pNS6U1-5a-Plasmid und 100 μL Transfektionsmedium in ein neues 1,5-ml-Mikrozentrifugenröhrchen geben.
- Bereiten Sie eine Mastermischung aus Lösung II für alle zu transfizierenden Proben vor, indem Sie 100 μL Transfektionsmedium und 4,0 μL Transfektionsreagenz pro Probe hinzufügen.
- Bereiten Sie die Transfektionsmischung vor, indem Sie 100 μL Lösung II in jedes Mikrozentrifugenröhrchen geben, das Lösung I enthält.
- Wirbeln Sie die Transfektionsmischung für 15 s, zentrieren Sie in einer Tischmikrozentrifuge bei 3.000 x g für 10 s bei RT und inkubieren Sie dann bei RT für 5 min.
- Alle 200 μL der Transfektionsmischung in eine Vertiefung der 12-Well-HeLa-Zellplatte geben, um ein Endvolumen von 1,0 ml pro Vertiefung zu erreichen, und die Platte bei 37 °C für 48 Stunden inkubieren.
- Extrahieren Sie nach der Inkubation RNA aus den transfizierten HeLa-Zellen mit handelsüblicher Guanidinthiocyanat- und Phenollösung.
HINWEIS: Dieses Reagenz enthält Phenol und dieser Schritt sollte in einem Abzug durchgeführt werden. Die Verwendung von DEPC-behandeltem Wasser wird für die Resuspension der extrahierten RNA empfohlen.- Aspirieren Sie das verbrauchte Medium und geben Sie 500 μL des Reagenzes in jede Vertiefung. Inkubieren Sie bei RT für 5 Min.
- Homogenisieren Sie durch Pipettieren nach oben und unten. Anschließend wird die Lösung in ein neues 1,5-ml-Mikrozentrifugenröhrchen übertragen.
- 100 μL Chloroform und Wirbel für 15 s zugeben.
- Zentrifuge bei 12.000 x g für 15 min bei RT.
- Übertragen Sie 200 μL der RNA-haltigen, oberen wässrigen Schicht in ein neues 1,5 ml Mikrozentrifugenröhrchen.
- Jeder RNA-Probe werden 2 μg Glykogen und 200 μL Isopropanol zugegeben. Mischen Sie, indem Sie die Röhren umkehren.
- Sammeln Sie den RNA-Niederschlag durch Zentrifugation bei 12.000 x g für 10 min bei 4 °C.
- Entfernen und entsorgen Sie den Überstand, ohne das RNA-Pellet zu stören.
- Waschen Sie das Pellet zweimal, indem Sie 1,0 ml 70% Ethanol zugeben, die Röhrchen umkehren und die Zentrifugieren wie in Schritt 2.13.7 beschrieben durchführen.
- Trocknen Sie das Pellet für ~ 10-20 min bei RT an der Luft und resuspenieren Sie die RNA in 10-20 μL RNase-freiem Wasser.
- Bestimmen Sie die RNA-Konzentration durch Messung der Absorption bei 260 nm, wie von Desjardins und Conklin9 beschrieben.
- Fahren Sie mit der Primerverlängerung fort oder lagern Sie RNA-Proben in -20 °C. Isolierte RNA kann bei -20 °C für 6-12 Monate gelagert werden.
3. 32P-Markierung von Oligonukleotiden
HINWEIS: Schritte, die die Verwendung von 32P-ATP und 32P-markierten Oligonukleotiden beinhalten, müssen hinter einem Acrylschild von geschulten Personen mit Genehmigung von autorisierten institutionellen Stellen durchgeführt werden. Das unten beschriebene Protokoll kann für die Markierung von Oligonukleotiden, Dup3r und U17-26-R und Markern für Harnstoff-PAGE verwendet werden. Die Verwendung von DEPC-behandeltem Wasser wird zur Resuspension von Oligonukleotiden und Größenausschlussperlen empfohlen.
- Zu einem 1,5 ml Mikrozentrifugenröhrchen fügen Sie Oligonukleotid, T4-Polynukleotidkinase (T4 PNK), T4 PNK-Puffer und 32P-ATP hinzu, wie in Tabelle 1 beschrieben. 32P-ATP zuletzt zu der Mischung hinzufügen; Das ist wichtig.
HINWEIS: Für die Zugabe von radioaktiven Lösungen wird die Verwendung von Filterspitzen empfohlen. - In einem Wasserbad bei 37 °C für 30 min inkubieren.
- Während die Markierungsreaktionen inkubieren, resuspendieren Sie die Größenausschlussperlen mit einem Molekulargewicht von 25 kDa, das durch sanftes Wirbeln für ~ 10 s abgeschnitten wird.
HINWEIS: Die Perlen mit Größenausschluss sollten gemäß den Anweisungen des Herstellers hergestellt und als 50% ige Suspension in 25% Ethanol bei 4 °C gelagert werden. - Bereiten Sie die Säulen vor, indem Sie 600 μL der resuspendierten Perlen auf eine Einweg-Minisäule in einem 1,5 ml Auffangröhrchen geben und den Perlenbestand auf 4 °C zurückführen.
- Zentrifugiere bei 2.000 x g für 1 min bei RT und entsorge den Durchfluss.
- Waschen Sie die Perlen, indem Sie 300 μL RNase-freies Wasser in die Säule geben.
- Wiederholen Sie die Schritte 3.5. und 3.6. zweimal und die Minisäule in ein neues 1,5 ml Zentrifugenröhrchen überführen.
- Die Kinase-Reaktionsmischung in die Perlensäule mit Größenausschluss geben und bei 5000 x g für 1 min bei RT zentrifugieren.
- Sammeln und speichern Sie den Durchfluss, der das 32P-markierte Oligonukleotid enthält, und entsorgen Sie alle Spitzen und Röhrchen in einer Acrylabfallbox.
- Fügen Sie 20 μL RNase-freies Wasser hinzu, um 32P-markiertes Oligonukleotid auf eine Endkonzentration von 2,5 μM zu verdünnen.
HINWEIS: Verdünnen Sie die markierten Marker nach Bedarf zum Laden auf Harnstoff-PAGE-Gele. - Lagern Sie das markierte Oligonukleotid in einer Acryl-Mikroröhrchenbox bei -20 °C oder fahren Sie mit der Primerverlängerungsanalyse fort.
4. Analyse der gespleißten Reportertranskripte nach Primer-Erweiterung
HINWEIS: Es wird empfohlen, Oberflächen und Geräte vor Gebrauch mit einem RNase-inaktivierenden Reagenz zu reinigen.
- Fügen Sie 2,0 μg RNA, die aus den Hela-Zellen extrahiert wurde, in separate 200 μL-Mikrozentrifugenröhrchen und fügen Sie RNase-freies Wasser hinzu, um das Volumen auf 6,55 μL aufzufüllen.
- Master Mix I mit den verdünnten 32P-Dup3r und dNTPs wie in Tabelle 2 gezeigt vorbereiten und 0,9 μL der Mischung zu jedem PCR-Röhrchen mit RNA-Proben geben.
- Inkubieren Sie die Röhrchen, zuerst bei 65 °C für 5 min und dann auf Eis für 1 min.
- Bereiten Sie Master Mix II mit 5x First Strang Buffer, Dithiothreitol (DTT), RNase-Inhibitor und Reverse Transkriptase wie in Tabelle 2 gezeigt vor.
- 2,55 μL der Mischung in jedes Röhrchen geben, das RNA und Master Mix 1 enthält; Das Gesamtvolumen der Reaktion sollte 10 μL betragen. Halten Sie die Röhrchen für 10 min bei RT.
- Die Röhrchen in ein Trockenbad oder einen Thermocycler überführen und inkubieren, zuerst bei 50 °C für 60 min und dann bei 70 °C für 15 min.
- Nach der Inkubation 10 μL 2x Formamid-RNA-Beladungsfarbstoff zu jeder Probe geben und in einer Acrylbox bei -20 °C lagern oder mit der Trennung der Fragmente durch Harnstoff-PAGE unter Verwendung eines 14 cm langen Gels und Visualisierung des Gelbildes wie unten in Schritt 8 beschrieben fortfahren.
- Führen Sie ein densitometrisches Scannen des Gelbildes mit einer Bildanalysesoftware durch und verwenden Sie die Bandintensitäten der enthaltenen und übersprungenen Produkte, um den Prozentsatz der Exon-2-Einschließung wie unten gezeigt zu berechnen.

5. Analyse der gespleißten Reportertranskripte mittels fluoreszierender RT-PCR
HINWEIS: Die unten beschriebene RT-PCR-Analyse verwendet zufällige Hexamere für die cDNA-Synthese und eine Kombination aus nicht markierten Dup8f- und Cy5-markierten Dup3r-Oligonukleotiden zur PCR-Amplifikation der gespleißten Produkte.
- Für die cDNA-Synthese fügen Sie 2,0 μg RNA, die aus den transfizierten Hela-Zellen extrahiert wurde, in separate 200 μL-Mikrozentrifugenröhrchen hinzu und fügen Sie RNase-freies Wasser hinzu, um das Volumen auf 11,0 μL zu erhöhen.
- Bereiten Sie Master Mix I vor, der zufällige Hexamere und dNTP enthält, wie in Tabelle 3 gezeigt, und geben Sie 2,0 μL der Mischung zu jeder Probe hinzu. Inkubieren, zuerst bei 65 °C für 5 min, dann auf Eis für 1 min.
- Bereiten Sie Master Mix II vor, der First Strang Buffer, RNase-Inhibitor, DTT und Reverse Transkriptase enthält, wie in Tabelle 3 gezeigt, und geben Sie 7,0 μL der Mischung zu jedem Röhrchen, das RNA und Master Mix I enthält.
- Halten Sie die Röhrchen für 10 min bei RT und inkubieren Sie dann bei 50 ° C für 60 min und 70 ° C für 15 min.
HINWEIS: Abgeschlossene cDNA-Reaktionen können bei -20 °C gelagert werden. - Für die PCR werden 1,0 μL (100 ng/μL) jeder cDNA-Probe auf neue PCR-Röhrchen übertragen.
- Bereiten Sie eine Mastermischung aus Dup8f, Cy5-Dup3r, dNTPs, Taq-Puffer, Taq-Polymerase und Wasser wie in Tabelle 4 beschrieben vor und geben Sie 11,5 μL der Mischung zu jedem Röhrchen, das cDNA enthält.
- PCR mit einem thermischen Cycler mit einem anfänglichen Denaturierungsschritt bei 94 °C für 3 min durchführen; gefolgt von 20 Zyklen der Denaturierung (94 °C für 30 s), des Glühens (65 °C für 30 s) und der Verlängerung (72 °C für 15 s) sowie eines Abschlussschritts bei 72 °C für 5 min.
- 12,5 μL 2x Formamid-DNA-Beladungsfarbstoff in jedes Röhrchen geben und bei 95 °C für 5 min erhitzen.
- Lagern Sie die PCR-Reaktion bei -20 °C oder fahren Sie mit Harnstoff-PAGE fort, wie unten in Schritt 8.1-8.4 beschrieben.
- Entfernen Sie nach der Elektrophorese die Glasplatten aus dem Elektrophoresegerät und scannen Sie mit einem Fluoreszenzbildner, um das Gel zu visualisieren.
- Führen Sie ein densitometrisches Scannen des Gelbildes durch und verwenden Sie die Bandintensität der enthaltenen und der übersprungenen Produkte, um den Prozentsatz der Exon-2-Einschließung wie in Schritt 4.8 beschrieben zu berechnen.
6. Analyse der Varianten-U1-snRNA-Expression durch Primer-Extension
- 2,0 μg RNA, die aus den Hela-Zellen extrahiert wurde, werden in separate 200 μL-Mikrozentrifugenröhrchen gegeben und RNase-freies Wasser hinzugefügt, um das Volumen auf 4,325 μL aufzufüllen, und dann dATP hinzufügen, wie in Tabelle 5 gezeigt.
- Fügen Sie jedem Röhrchen 10.000 CPM des 32P-U17-26-R-Oligonukleotids hinzu.
HINWEIS: Um eine 10.000 cpm/μL-Lösung von 32P-U17-26-R (aus Schritt 3) herzustellen, verdünnen Sie das markierte Oligonukleotid (1:20 Verdünnung), bestimmen Sie cpm in 1,0 μL mit einem Szintillationszähler und verdünnen Sie es weiter mit deionisiertem Wasser, um eine Lösung von 10.000 cpm / μL in einem neuen 1,5 ml Mikrozentrifugenröhrchen herzustellen. - Bei 65 °C für 5 min und dann 1 min auf Eis inkubieren.
- Bereiten Sie eine Mastermischung mit 5x First Strang Buffer, RNase-Inhibitor, DTT und Reverse Transkriptase wie in Tabelle 5 gezeigt vor und geben Sie 1,8 μL der Mischung zu jeder Probe hinzu.
- Inkubieren, zuerst bei RT für 10 min und dann bei 42 °C für 10 min.
- Nach der Inkubation 10 μL 2x Formamid-RNA-Beladungsfarbstoff in jede Probe geben und in einer Acrylbox bei -20 °C lagern oder mit der Trennung der Fragmente durch Harnstoff-PAGE unter Verwendung eines 38 cm langen Gels fortfahren (siehe Schritt 8).
7. Analyse der Varianten-U1-snRNA-Expression mittels RT-qPCR
- Verdünnen Sie den wie oben in den Schritten 5.1 - 5.4 beschriebenen cDNA-Stamm auf eine Konzentration von 0,2 ng/μL.
- 5,0 μL verdünnte cDNA in einzelne Vertiefungen einer 96-Well-qPCR-Platte in dreifacher Ausfertigung pipettieren. Fügen Sie entionisiertes Wasser anstelle von cDNA für die Steuerung ohne Vorlage (NTC) hinzu.
- Bereiten Sie zwei separate Primermischungen vor, die aus den Vorwärts- und Rückwärtsprimern für U1- und U2-snRNAs und Wasser bestehen, wie in Tabelle 6 gezeigt.
HINWEIS: Sequenzen für U1- und U2-snRNA-spezifische Primer sind in Tabelle 7 aufgeführt. - Geben Sie 5,0 μL der U1- und U2-snRNA-Primermischung in die Probe und die NTC-Vertiefungen.
- Fügen Sie 10,0 μL Real-Time-PCR-Mix zu jeder Vertiefung hinzu.
- Versiegeln Sie die Platten mit einem optischen Film und zentrifugieren Sie dann bei 1.000 x g für 2 min bei RT, um die Reaktionen auf den Boden der Vertiefungen zu sammeln.
- Führen Sie eine qPCR mit einem anfänglichen Denaturierungsschritt bei 95 °C für 10 min durch, gefolgt von 40 Zyklen eines 2-stufigen Protokolls, bestehend aus Denaturierung (95 °C für 15 s) und Glühen/Extension (62 °C für 60 s), während die Schwellenwertquantifizierungszykluswerte (Cq) der Zielamplikone gesammelt werden.
- Beenden Sie die qPCR-Reaktion, indem Sie nach einem einzelnen Peak in der Dissoziationskurve für U1- und U2-snRNA-Reaktionen suchen.
- Berechnen Sie aus den Cq-Werten delta Cq (ΔCq) für U1- und U2-snRNAs im Vergleich zur pcDNA-Steuerung.
- Bestimmen Sie die snRNA-Expression der Variante U1 als relative Größe (RQ) von U1 im Vergleich zu U2 unter Verwendung des ΔΔCq-Werts, wie unten für alle Proben gezeigt.

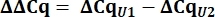
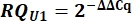
8. Einrichtung und Betrieb von Urea-PAGE Gelen
HINWEIS: Die Montage der Glasplatten und des Gellaufgeräts sollte gemäß den Anweisungen des Herstellers erfolgen. Das Gießen des 10% Harnstoff-PAGE Gels kann nach einem zuvor beschriebenen Protokoll von Summer et al.10 durchgeführt werden. Schritte zur Herstellung von Markern und Proben sowie zum Ausführen und Visualisieren von Gelen werden im Folgenden beschrieben. Um zu verhindern, dass das Gel an Glasplatten haftet, kann die innere Oberfläche optional mit Silikonlösung beschichtet werden, indem 1 ml der Lösung auf die Oberfläche gegeben und gleichmäßig über die gesamte Oberfläche mit Gewebe verteilt wird. Nach dem Trocknen sollten die Platten mit entionisiertem Wasser gewaschen und erneut getrocknet werden.
ACHTUNG: Unpolymerisiertes Acrylamid ist neurotoxisch und muss mit den im Sicherheitsdatenblatt empfohlenen Schutzmaßnahmen behandelt werden.
- Bereiten Sie die Primerverlängerungsproben und Marker vor, indem Sie 5 min bei 95 °C erhitzen und dann in einer Tischmikrozentrifuge bei 3.000 x g für 5 s bei RT zentrifugieren.
- Bevor Sie die Marker und Proben laden, spülen Sie die Vertiefungen mit 1x FSME-Puffer aus, um den abgesetzten Harnstoff zu entfernen.
- Laden Sie 10 μL / Probe / Well und lassen Sie das Gel bei 300-500V für 2-3 Stunden oder bis das Xylol-Cyanol den Boden erreicht.
HINWEIS: Etwa 1.000 cpm des 32P-markierten Markers können geladen werden. - Entfernen Sie nach der Elektrophorese die Glasplatten aus dem Elektrophoreseapparat.
- Trennen Sie die beiden Platten vorsichtig, so dass das Gel flach auf einer der beiden Glasoberflächen liegt, und übertragen Sie das Gel auf ein Filterpapier und bedecken Sie es mit Plastikfolie.
- Vakuumtrocknen Sie das Gel auf dem Filterpapier bei 80 °C für 30 min mit einem Geltrockner.
- Legen Sie das getrocknete Gel in eine Phosphor-Bildgebungskassette und bewahren Sie es über Nacht bei RT auf.
HINWEIS: Der Phosphorschirm sollte vor dem Gebrauch mit einem Leuchtkasten gelöscht werden. - Um das Gelbild zu visualisieren, entfernen Sie den Bildschirm und scannen Sie mit einem Phosphor-Imager.
Ergebnisse
Der Spleißreporter Dup51, ein Drei-Exon-Zwei-Intron-Minigen, wurde aus dem menschlichen β-Globin-Gen abgeleitet und zuvor beschrieben (Abbildung 1A)11,12 . Wir schufen einen mutierten Reporter, Dup51p, durch die Einführung von Usher-Syndrom-assoziierten 5'-Spleißstellenmutationen, die im Exon 3 des Protocadherin 15 (PCDH15) Gens13 auftreten. Die 5'-Spleißstellensequenz an der Exon-2-Intron-2-Verb...
Diskussion
Der Assay kann für die Spleißanalyse in anderen Zelllinien als HeLa angepasst werden, jedoch müssen Faktoren, die die Transfektionseffizienz beeinflussen, wie Zellkonfluenz und DNA-Menge, möglicherweise optimiert werden. Das Konstruktverhältnis von Reporter zu U1 ist ein weiterer kritischer Parameter, der möglicherweise in Abhängigkeit von den in anderen Zelltypen beobachteten Expressionsniveaus bestimmt werden muss. Die Qualität der extrahierten RNA ist entscheidend für die Spleißanalyse; Daher wird die Verwen...
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Diese Arbeit wurde durch Mittel an S.S. von den National Institutes of Health (R21CA170786 und R01GM127464) und der American Cancer Society (institutional research grant 74-001-34-IRG) sowie an S.S. und W.M. aus dem Valley Research Partnership Program (P1-4009 und VRP77) unterstützt. Der Inhalt liegt in der alleinigen Verantwortung der Autoren und stellt nicht unbedingt die offiziellen Ansichten der National Institutes of Health dar.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent Grade Deionized Water | ThermoFisher Scientific | 23-751628 | |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758-25ML | |
| Dulbecco's Modified Eagle Medium (DMEM) powder packet | Gibco | 12100-046 | |
| Sodium Bicarbonate | ThermoFisher Scientific | S233-500 | |
| Fetal Bovine Serum (FBS), Australian Source, Heat Inactivated | Omega Scientific | FB-22 | |
| Penicillin-Streptomycin (P/S) | Sigma-Aldrich | P4458-100ML | |
| Sodium Hydroxide, Standard Solution 1.0N | Sigma-Aldrich | S2567-16A | |
| Hydrochloric Acid, Certified ACS Plus, 36.5 to 38.0% | ThermoFisher Scientific | A144-500 | |
| Disposable PES Bottle Top Filters | ThermoFisher Scientific | FB12566510 | |
| EDTA Disodium Salt Dihydrate | Amresco | 0105-2.5KG | |
| 2.5% Trypsin (10x), no phenol red | ThermoFisher Scientific | 15090046 | |
| Sodium Chloride | Fisher Bioreagent | BP358-212 | |
| Potassium Chloride | Fisher Bioreagent | BP366-1 | |
| Disodium Hydrogen Phosphate Heptahydrate | Fisher Bioreagent | BP332-1 | |
| Potassium Dihydrogen Phosphate | Fisher Bioreagent | BP362-1 | |
| Transfection medium - Opti-MEM™ I Reduced Serum Medium, no phenol red | ThermoFisher Scientific | 11058021 | |
| Transfection Reagent - Lipofectamine™ 2000 | ThermoFisher Scientific | 13778150 | |
| TRIzol™ Reagent | ThermoFisher Scientific | 15596018 | |
| Chloroform (Approx. 0.75% Ethanol as Preservative/Molecular Biology) | ThermoFisher Scientific | BP1145-1 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2818-4 | |
| Isopropanol, Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2618-212 | |
| Glycogen (5 mg/ml) | ThermoFisher Scientific | AM9510 | |
| Direct-zol RNA Miniprep Kit | Zymo Research | R2052 | |
| ATP, [γ-32P]- 6000Ci/mmol 150mCi/ml Lead, 1 mCi | PerkinElmer | NEG035C001MC | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| Size exclusion beands - Sephadex® G-25 | Sigma-Aldrich | G2580-10G | |
| Size exclusion mini columns | USA Scientific | 1415-0600 | |
| pBR322 DNA-MspI Digest | New England Biolabs | N3032S | |
| Low Molecular Weight Marker, 10-100 nt | Affymetrix | 76410 100 UL | |
| Rnase inactivating reagents - RNaseZAP™ | Sigma-Aldrich | R2020-250ML | |
| dNTP Mix (10 mM ea) | ThermoFisher Scientific | 18427013 | |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | ThermoFisher Scientific | 10777019 | |
| Reverse Transcriptase - M-MLV Reverse Transcriptase | ThermoFisher Scientific | 28025013 | used for primer extension |
| Taq DNA Polymerase | ThermoFisher Scientific | 10342020 | |
| Random Hexamers (50 µM) | ThermoFisher Scientific | N8080127 | |
| Real time PCR mix - SYBR™ Select Master Mix | ThermoFisher Scientific | 4472903 | |
| SuperScript™ III Reverse Transcriptase | ThermoFisher Scientific | 18080093 | used for cDNA preparation |
| Dithiothreitol (DTT) | ThermoFisher Scientific | 18080093 | |
| 5X First-Strand Buffer | ThermoFisher Scientific | 18080093 | |
| Formamide (≥99.5%) | ThermoFisher Scientific | BP228-100 | Review Material Safety Data Sheets |
| Bromophenol Blue sodium salt | Sigma-Aldrich | 114405-5G | |
| Xylene Cyanol FF | Sigma-Aldrich | 2650-17-1 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | ThermoFisher Scientific | BP152-5 | |
| Boric Acid (Crystalline/Electrophoresis) | ThermoFisher Scientific | BP168-500 | |
| Acrylamide: Bis-Acrylamide 19:1 (40% Solution/Electrophoresis) | ThermoFisher Scientific | BP1406-1 | Review Material Safety Data Sheets |
| Urea (Colorless-to-White Crystals or Crystalline Powder/Mol. Biol.) | ThermoFisher Scientific | BP169-212 | |
| Ammonium peroxodisulphate (APS) ≥98%, Pro-Pure, Proteomics Grade | VWR | M133-25G | |
| Sigmacote | Sigma-Aldrich | SL2-100ML | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) ≥99%, Ultrapure | VWR | 0761-25ML | Review Material Safety Data Sheets |
| Adjustable Slab Gel Systems, Expedeon | VWR | ASG-400 | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 14.5cm | VWR | NGP-125NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 22.0cm | VWR | NGP-200NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 38.7cm | VWR | NGP-400NR | |
| GE Storage Phosphor Screens | Sigma-Aldrich | GE28-9564 | |
| Typhoon™ FLA 7000 Biomolecular Imager | GE Healthcare | 28-9610-73 AB | |
| Beckman Coulter LS6500 Liquid Scintillation Counter | GMI | 8043-30-1194 | |
| C1000 Touch Thermal Cycler | ThermoFisher Scientific | ||
| QuantStudio 6 Flex Real-Time PCR Systems | ThermoFisher Scientific |
Referenzen
- Zhuang, Y., Weiner, A. M. A compensatory base change in U1 snRNA suppresses a 5' splice site mutation. Cell. 46 (6), 827-835 (1986).
- Scotti, M. M., Swanson, M. S. RNA mis-splicing in disease. Nature Review Genetics. 17 (1), 19-32 (2016).
- Ward, A. J., Cooper, T. A. The pathobiology of splicing. Journal of Pathology. 220 (2), 152-163 (2010).
- Scalet, D., et al. Disease-causing variants of the conserved +2T of 5' splice sites can be rescued by engineered U1snRNAs. Human Mutatation. 40 (1), 48-52 (2019).
- Yamazaki, N., et al. Use of modified U1 small nuclear RNA for rescue from exon 7 skipping caused by 5'-splice site mutation of human cathepsin A gene. Gene. 677, 41-48 (2018).
- Yanaizu, M., Sakai, K., Tosaki, Y., Kino, Y., Satoh, J. I. Small nuclear RNA-mediated modulation of splicing reveals a therapeutic strategy for a TREM2 mutation and its post-transcriptional regulation. Science Reports. 8 (1), 6937 (2018).
- Balestra, D., et al. Splicing mutations impairing CDKL5 expression and activity can be efficiently rescued by U1snRNA-based therapy. International Journal of Molecular Sciences. 20 (17), 20174130 (2019).
- Donadon, I., et al. Exon-specific U1 snRNAs improve ELP1 exon 20 definition and rescue ELP1 protein expression in a familial dysautonomia mouse model. Human Molecular Genetics. 27 (14), 2466-2476 (2018).
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565 (2010).
- Summer, H., Gramer, R., Droge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). Journal of Visualized Experiments. (32), e1485 (2009).
- Dominski, Z., Kole, R. Selection of splice sites in pre-mRNAs with short internal exons. Molecular Cell Biology. 11 (12), 6075-6083 (1991).
- Amir-Ahmady, B., Boutz, P. L., Markovtsov, V., Phillips, M. L., Black, D. L. Exon repression by polypyrimidine tract binding protein. RNA. 11 (5), 699-716 (2005).
- Le Guedard-Mereuze, S., et al. Sequence contexts that determine the pathogenicity of base substitutions at position +3 of donor splice-sites. Human Mutation. 30 (9), 1329-1339 (2009).
- Sharma, S., Wongpalee, S. P., Vashisht, A., Wohlschlegel, J. A., Black, D. L. Stem-loop 4 of U1 snRNA is essential for splicing and interacts with the U2 snRNP-specific SF3A1 protein during spliceosome assembly. Genes and Development. 28 (22), 2518-2531 (2014).
- Steitz, J. A., et al. . Functions of the abundant U-snRNPs. Structure and function of major and minor small nuclear ribonucleoprotein particles. , 115-154 (1988).
- Fortes, P., et al. Inhibiting expression of specific genes in mammalian cells with 5' end-mutated U1 small nuclear RNAs targeted to terminal exons of pre-mRNA. Proceedings of the National Academy of Sciences U.S.A. 100 (14), 8264-8269 (2003).
- Roca, X., et al. Widespread recognition of 5' splice sites by noncanonical base-pairing to U1 snRNA involving bulged nucleotides. Genes and Development. 26 (10), 1098-1109 (2012).
- Roca, X., Krainer, A. R. Recognition of atypical 5' splice sites by shifted base-pairing to U1 snRNA. Nature Structural Molecular Biology. 16 (2), 176-182 (2009).
- Taladriz-Sender, A., Campbell, E., Burley, G. A. Splice-switching small molecules: A new therapeutic approach to modulate gene expression. Methods. 167, 134-142 (2019).
- Hamid, F. M., Makeyev, E. V. A mechanism underlying position-specific regulation of alternative splicing. Nucleic Acids Research. 45 (21), 12455-12468 (2017).
- Martelly, W., et al. Synergistic roles for human U1 snRNA stem-loops in pre-mRNA splicing. RNA Biology. , 1-18 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten