Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Live-Bildgebung des mitochondrialen Glutathion-Redox-Zustands in primären Neuronen unter Verwendung eines ratiometrischen Indikators
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt ein Protokoll zur Bestimmung von Unterschieden im basalen Redoxzustand und in den Redoxreaktionen auf akute Störungen in primären Hippocampus- und kortikalen Neuronen unter Verwendung der konfokalen Live-Mikroskopie. Das Protokoll kann mit minimalen Modifikationen auf andere Zelltypen und Mikroskope angewendet werden.
Zusammenfassung
Die mitochondriale Redoxhomöostase ist wichtig für die neuronale Lebensfähigkeit und Funktion. Obwohl Mitochondrien mehrere Redoxsysteme enthalten, gilt der sehr häufige Thiol-Disulfid-Redoxpuffer Glutathion als zentraler Akteur in der antioxidativen Abwehr. Daher liefert die Messung des mitochondrialen Glutathion-Redoxpotentials nützliche Informationen über den mitochondrialen Redoxstatus und oxidativen Stress. Glutaredoxin1-roGFP2 (Grx1-roGFP2) ist ein genetisch kodierter, auf grün fluoreszierendem Protein (GFP) basierender ratiometrischer Indikator des Glutathion-Redoxpotentials, der zwei Redox-Zustands-sensitive Anregungspeaks bei 400 nm und 490 nm mit einem einzigen Emissionspeak bei 510 nm aufweist. Dieser Artikel beschreibt, wie man konfokale Live-Mikroskopie von Mitochondrien-gerichtetem Grx1-roGFP2 in primären Hippocampus- und kortikalen Neuronen durchführt. Es beschreibt, wie das mitochondriale Glutathion-Redoxpotenzial im Steady-State-Stadium beurteilt werden kann (z. B. zum Vergleich von Krankheitszuständen oder Langzeitbehandlungen) und wie Redoxveränderungen bei akuten Behandlungen gemessen werden können (am Beispiel des exzitoxischen Arzneimittels N-Methyl-D-Aspartat (NMDA). Darüber hinaus stellt der Artikel die Co-Imaging von Grx1-roGFP2 und dem mitochondrialen Membranpotentialindikator Tetramethylrhodamin, Ethylester (TMRE) vor, um zu demonstrieren, wie Grx1-roGPF2 mit zusätzlichen Indikatoren für multiparametrische Analysen gemultiplext werden kann. Dieses Protokoll enthält eine detaillierte Beschreibung, wie (i) konfokale Laser-Scanning-Mikroskopeinstellungen optimiert, (ii) Medikamente zur Stimulation mit anschließender Sensorkalibrierung mit Diamid und Dithiothreitol angewendet und (iii) Daten mit ImageJ/FIJI analysiert werden.
Einleitung
Mehrere wichtige mitochondriale Enzyme und Signalmoleküle unterliegen der Thiol-Redox-Regulation1. Darüber hinaus sind Mitochondrien eine wichtige zelluläre Quelle reaktiver Sauerstoffspezies und selektiv anfällig für oxidative Schäden2. Dementsprechend wirkt sich das mitochondriale Redoxpotential direkt auf die Bioenergetik, die Zellsignalisierung, die mitochondriale Funktion und letztendlich auf die Lebensfähigkeit der Zellen aus3,4. Die mitochondriale Matrix enthält hohe Mengen (1-15 mM) des Thiol-Disulfid-Redoxpuffers Glutathion (GSH), um die Redoxhomöostase aufrechtzuerhalten und die antioxidative Abwehr aufzubauen5,6. GSH kann kovalent an Zielproteine gebunden werden (S-Glutathionylierung), um ihren Redoxstatus und ihre Aktivität zu kontrollieren, und wird von einer Reihe von entgiftenden Enzymen verwendet, die oxidierte Proteine reduzieren. Daher ist das mitochondriale Glutathion-Redoxpotential ein sehr informativer Parameter bei der Untersuchung der mitochondrialen Funktion und Pathophysiologie.
roGFP2 ist eine Variante von GFP, die durch Zugabe von zwei oberflächenexponierten Cysteinen, die ein künstliches Dithiol-Disulfid-Paar bilden, redoxempfindlich gemacht wurde7,8. Es hat einen einzelnen Emissionspeak bei ~510 nm und zwei Anregungsspitzen bei ~400 nm und 490 nm. Wichtig ist, dass die relativen Amplituden der beiden Anregungsspitzen vom Redoxzustand von roGFP2 abhängen (Abbildung 1), was dieses Protein zu einem ratiometrischen Sensor macht. Im Grx1-roGFP2-Sensor wurde humanes Glutaredoxin-1 (Grx1) mit dem N-Terminus von roGFP29,10 verschmolzen. Die kovalente Bindung des Grx1-Enzyms an roGFP2 ermöglicht zwei wesentliche Verbesserungen des Sensors: Es macht die Sensorantwort spezifisch für das GSH/GSSG-Glutathion-Redox-Paar (Abbildung 1) und beschleunigt das Gleichgewicht zwischen GSSG und roGFP2 um einen Faktor von mindestens 100.0009. Daher ermöglicht Grx1-roGFP2 eine spezifische und dynamische Abbildung des zellulären Glutathion-Redoxpotentials.
Grx1-roGFP2-Bildgebung kann auf einer Vielzahl von Mikroskopen durchgeführt werden, einschließlich Weitfeldfluoreszenzmikroskopen, rotierenden konfokalen Scheibenmikroskopen und konfokalen Laserscanning-Mikroskopen. Die Expression des Sensors in primären Neuronen kann durch verschiedene Methoden erreicht werden, darunter Lipofektion11, DNA/Calcium-Phosphat-Kopräzipitation12, virusvermittelter Gentransfer oder die Verwendung transgener Tiere als Zellquelle (Abbildung 2). Pseudotypisierte rekombinante adeno-assoziierte Viren (rAAV), die ein 1:1-Verhältnis von AAV1- und AAV2-Kapsidproteinen 13,14 enthielten, wurden für die Experimente in diesem Artikel verwendet. Mit diesem Vektor wird die maximale Sensorexpression typischerweise 4-5 Tage nach der Infektion erreicht und bleibt mindestens zwei Wochen lang stabil. Wir haben Grx1-roGFP2 erfolgreich in primären Hippocampus- und kortikalen Neuronen von Mäusen und Ratten eingesetzt.
In diesem Artikel wird die rAAV-vermittelte Expression von Mitochondrien-gerichtetem Grx1-roGFP2 in primären Hippocampus- und kortikalen Neuronen der Ratte verwendet, um den basalen mitochondrialen Glutathion-Redoxzustand und seine akute Störung zu beurteilen. Für die konfokale Live-Bildgebung wird ein Protokoll mit detaillierten Anweisungen bereitgestellt, wie (i) die Einstellungen für konfokale Laserscanning-Mikroskope optimiert, (ii) ein Live-Imaging-Experiment durchgeführt und (iii) Daten mit FIJI analysiert werden können.
Protokoll
Alle Tierversuche entsprachen den nationalen und institutionellen Richtlinien, einschließlich der Richtlinie 2010/63/EU des Europäischen Parlaments des Rates, und verfügten über eine vollständige ethische Genehmigung des Innenministeriums (Tierwohlamt der Universität Heidelberg und Regierungspraesidium Karlsruhe, Lizenzen T14/21 und T13/21). Primäre Hippocampus- und kortikale Neuronen wurden aus neugeborenen Maus- oder Rattenwelpen nach Standardverfahren hergestellt und wie zuvor beschrieben für 12-14 Tage aufrechterhalten13.
1. Herstellung von Lösungen
- Standardlösungen für Imaging-Puffer
- Bereiten Sie jede Stammlösung gemäß Tabelle 1 vor und bewahren Sie sie bei 4 °C auf. Für die Langzeitlagerung (>3 Monate) die Aliquots bei -20 °C aufbewahren.
| Bestandteil | MW | Konzentration (M) | Betrag (g) | Volumen (ml) |
| NaCl | 58.44 | 5 | 14.61 | 50 |
| Kcl | 74.55 | 3 | 1.12 | 5 |
| MgCl2· 6H2O | 203.3 | 1.9 | 2 | 5 |
| CaCl2·2H2O | 147.01 | 1 | 1.47 | 10 |
| Glycin | 75.07 | 0.1 | 0.375 | 50 |
| Saccharose | 342.3 | 1.5 | 25.67 | 50 |
| Natriumpyruvat | 110.04 | 0.1 | 0.55 | 50 |
| HEPES | 238.3 | 1 | 11.9 | 50 |
| Traubenzucker | 180.15 | 2.5 | 45 | 100 |
Tabelle 1: Lagerlösungen für den Imaging-Puffer.
- Stammlösungen von Arzneimitteln und Farbstoffen
- Diamid (DA; wird zur Kalibrierung im Verhältnis maximal 405:488 verwendet) in Wasser gelöst, um eine Stammlösung von 0,5 M (z. B. 1 g in 11,615 ml Wasser) zu erhalten. Aliquot und bei -20 °C lagern.
- Lösen Sie Dithiothreitol (DTT; zur Kalibrierung mit einem Mindestverhältnis von 405:488) in Wasser auf, um eine Stammlösung von 1 M (z. B. 5 g in 32,425 ml Wasser) zu erhalten. Aliquot und bei -20 °C für maximal 3 Monate lagern.
- Lösen Sie N-Methyl-D-Aspartat (NMDA; wird verwendet, um Exzitotoxizität und mitochondriale Oxidation zu induzieren) in Wasser, um eine 10 mM Stammlösung (z. B. 25 mg in 16,991 ml Wasser) zu erhalten. Lagern Sie die Aliquots bei -20 °C. Für die Langzeitlagerung (>6 Monate) halten Sie die Aliquots bei -80 °C.
- Tetramethylrhodaminethylesterperchlorat (TMRE; ein niedermolekularer Indikator für das mitochondriale Membranpotential)
- TMRE-Pulver in Methanol auflösen, um einen 20 mM-Bestand zu erhalten (z. B. 25 mg in 2,427 ml Methanol).
- Verdünnen Sie den 20 mM-Bestand 1:1.000 in Methanol, um einen 20 μM-Stamm zu erhalten.
- Aliquot die 20 mM und 20 μM Lagerlösungen, mit Parafolie versiegeln und lichtgeschützt bei -20 °C lagern.
HINWEIS: Beide Lagerlösungen sind über mehrere Jahre stabil. Verwenden Sie die 1.000-fache Stammlösung (20 μM) für Experimente.
- Imaging-Puffer
- Bereiten Sie 100 ml Bildpuffer vor, indem Sie alle Komponenten aus Tabelle 2 bis 80 mL sterilem Wasser in einen Messzylinder geben. Bringen Sie das Volumen mit sterilem Wasser auf bis zu 100 ml. Mischen Sie durch vorsichtiges Schütteln des Messzylinders, bis die Lösung homogen erscheint.
HINWEIS: Es wird empfohlen, ein Osmometer zu verwenden, um die Osmolarität des Puffers zu überprüfen. Es sollte so nah wie möglich am Wachstumsmedium der Zellen sein. Hier sind dies 315 mOsmol / L. Erhöhen oder verringern Sie die Saccharosekonzentration nach Bedarf, um die Osmolarität des Bildgebungspuffers und des Wachstumsmediums anzupassen. - Stellen Sie den pH-Wert auf 7,4 ein. Aliquots herstellen und bis zu zwei Wochen bei 4 °C aufbewahren. Für die Langzeitlagerung halten Sie die Aliquots bei -20 °C. Lassen Sie den Bildpuffer vor Gebrauch Raumtemperatur erreichen.
- Bereiten Sie 100 ml Bildpuffer vor, indem Sie alle Komponenten aus Tabelle 2 bis 80 mL sterilem Wasser in einen Messzylinder geben. Bringen Sie das Volumen mit sterilem Wasser auf bis zu 100 ml. Mischen Sie durch vorsichtiges Schütteln des Messzylinders, bis die Lösung homogen erscheint.
| Bestandteil | Stammlösung (M) | Endkonzentration (mM) | Volumen (ml) |
| NaCl | 5 | 114 | 2.3 |
| Kcl | 3 | 5.29 | 0.176 |
| MgCl2 | 1.9 | 1 | 0.053 |
| CaCl2 | 1 | 2 | 0.2 |
| Glycin | 0.1 | 0.005 | 0.005 |
| Saccharose | 1.5 | 52 | 3.5 |
| Natriumpyruvat | 0.1 | 0.5 | 0.5 |
| HEPES | 1 | 10 | 1 |
| Traubenzucker | 2.5 | 5 | 0.2 |
Tabelle 2: Zusammensetzung des Abbildungspuffers. Die angegebenen Volumina werden für die Herstellung von 100 ml Bildgebungspuffer verwendet.
- Lösungen für Stimulation und Kalibrierung
HINWEIS: Bereiten Sie immer frische Stimulationslösungen vor, indem Sie kurz vor dem Experiment Standardlösungen der angegebenen Medikamente in den Bildgebungspuffer geben. Lösungen für Stimulation und Kalibrierung werden der Bildgebungskammer nacheinander während eines Experiments hinzugefügt (siehe Abschnitte 3-5). Je nach Art des Experiments werden unterschiedliche Lösungen benötigt, um die gleiche Endkonzentration im jeweiligen Endvolumen in der Bildgebungskammer zu erreichen.- 3x NMDA-Lösung (90 μM; Endkonzentration in der Kammer: 30 μM) durch Zugabe von 63 μL eines 10 mM NMDA-Materials zu 6,937 ml Bildpuffer herstellen. 500 μL der resultierenden Lösung in die Kammer geben (Endvolumen: 1,5 mL).
- Bereiten Sie 2x DA-Lösung für die Schritte 3 und 4 (1 mM; Endkonzentration in der Kammer: 0,5 mM) vor, indem Sie 14 μL eines 0,5 M DA-Stammes zu 6,986 mL Bildgebungspuffer hinzufügen. 1 mL in die Kammer geben (Endvolumen: 2 mL).
- Bereiten Sie 4x DA-Lösung für Schritt 5 (2 mM; Endkonzentration in der Kammer: 0,5 mM) vor, indem Sie 28 μL eines 0,5 M DA-Materials zu 6,972 ml Bildgebungspuffer hinzufügen. 500 μL in die Kammer geben (Endvolumen: 2 mL).
- 1x DTT-Lösung (5 mM; Endkonzentration in der Kammer: 5 mM) durch Zugabe von 45 μL 1 M DTT-Stamm zu 8955 μL Bildgebungspuffer herstellen. 1 mL dieser Lösung nach Dem Absaugen des Bildgebungspuffers in die Kammer geben (Endvolumen: 1 mL).
2. Beladung von Zellen mit TMRE
HINWEIS: In diesem Protokoll wird TMRE im Nicht-Quench-Modus15 bei einer Endkonzentration von 20 nM verwendet. Im Allgemeinen sollte die niedrigstmögliche Konzentration von TMRE verwendet werden, die auf dem Mikroskop der Wahl immer noch eine ausreichende Signalintensität liefert. Aufgrund der ungleichmäßigen Verdunstung kann das Volumen des Mediums in verschiedenen Vertiefungen in langfristigen Primärkulturen unterschiedlich sein. Um eine konsistente TMRE-Konzentration in allen Bohrlöchern zu gewährleisten, fügen Sie TMRE nicht direkt zu den Bohrlöchern hinzu. Ersetzen Sie stattdessen das Medium in jeder Vertiefung durch die gleiche Menge an TMRE-haltigem Medium. Das folgende Protokoll ist für primäre Neuronen in 24-Well-Platten konzipiert, die ~ 1 ml Medium pro Vertiefung enthalten.
- Wenn Sie in einer Gewebekultur-Laminar-Flow-Haube arbeiten, sammeln Sie 500 μL Medium aus jeder Vertiefung in einem einzigen konischen Rohr.
- Pro Vertiefung 0,5 μL 20 μM TMRE-Material in das konische Rohr geben (z. B. 12 μL für 24 Vertiefungen).
- Das verbleibende Medium aus der ersten Vertiefung vorsichtig absaugen und durch 500 μL TMRE-haltiges Medium ersetzen. Fahren Sie mit den verbleibenden Bohrlöchern fort.
HINWEIS: Achten Sie darauf, die Zellen nicht austrocknen zu lassen und die Zellen nicht zu stören. - Bringen Sie die Zellen in den Inkubator zurück und warten Sie mindestens 60 Minuten auf das Farbstoffgleichgewicht.
HINWEIS: Die Ladezeit kann ohne nachteilige Auswirkungen auf mehrere Stunden verlängert werden. - Um konsistente TMRE-Konzentrationen und Gleichgewichte während des gesamten Bildgebungsexperiments zu gewährleisten, stellen Sie sicher, dass eine Endkonzentration von 20 nM TMRE in den Bildgebungspuffer und alle Stimulationslösungen aufgenommen wird.
3. Optimierung der Einstellungen des konfokalen Rastermikroskops
HINWEIS: Dieser Schritt zielt darauf ab, den besten Kompromiss zwischen Bildqualität und Zelllebensfähigkeit während der Live-Bildgebung zu finden. In diesem Abschnitt wird die Optimierung der Einstellungen für das roGFP-Imaging beschrieben. Wenn eine multiparametrische Bildgebung durchgeführt wird, muss für die zusätzlichen Indikatoren eine ähnliche Optimierung durchgeführt werden, einschließlich der Überprüfung auf eine stabile Baseline ohne Anzeichen von Bleichung oder Phototoxizität.
- Starten Sie das konfokale Mikroskop und laden Sie die Standardeinstellungen für die GFP-Bildgebung (488 nm Anregung, 505 - 550 nm Emission).
- Stellen Sie den Detektor auf 12 Bit oder 16 Bit ein.
HINWEIS: Normalerweise reichen 8 Bit für die quantitative Bildgebung nicht aus. - Aktivieren Sie den sequenziellen Scan-Modus und fügen Sie eine zweite Sequenz/Spur hinzu (405 nm Anregung, 505 - 550 nm Emission).
- Wählen Sie für beide Kanäle eine Pseudofarb-Nachschlagetabelle aus, die über- und unterbelichtete Pixel angibt (z. B. GLOW OU).
- Wählen Sie ein Ziel aus, das für das Objekt von Interesse geeignet ist.
HINWEIS: 10x-40x sind für die Einzelzellanalyse geeignet, 63x-100x sind für die Einzel-Mitochondrien-Analyse geeignet. - Montieren Sie einen Deckglas mit Zellen in die Bildgebungskammer, fügen Sie 1 ml Bildgebungspuffer hinzu und legen Sie die Kammer auf das Mikroskop.
- Verwenden Sie das Okular und das durchgelassene Licht, um die Zellen zu fokussieren.
HINWEIS: Verwenden Sie kein Epifluoreszenzlicht, um Zellen zu lokalisieren und zu fokussieren. Selbst bei geringer Leistung wirkt sich dies nachteilig auf die Zellen aus. - Nehmen Sie Bilder mit verschiedenen Pixelformaten auf. Wählen Sie anhand dieser Bilder die niedrigste Pixelzahl aus, die eine akzeptable Auflösung der interessierenden Struktur ergibt.
HINWEIS: Typischerweise eignen sich 512 x 512 Pixel gut für Einzelzellenaufnahmen mit 20-fachen und 40-fachen Objektiven, und 1024 x 1024 oder 2048 x 2048 Pixel eignen sich in der Regel gut für Einzel-Mitochondrien-Aufnahmen mit einem 63-fachen Objektiv. - Nehmen Sie Bilder mit unterschiedlichen Lochgrößen auf. Wählen Sie anhand dieser Bilder die größte Lochgröße aus, die eine akzeptable Auflösung der interessierenden Struktur ergibt.
HINWEIS: Typischerweise funktionieren 3-7 luftige Einheiten gut. - Nehmen Sie Bilder mit unterschiedlichen Laserintensitäten auf.
- Passen Sie die Detektorverstärkung und den Schwellenwert entsprechend an. Wählen Sie anhand dieser Bilder die niedrigste Laserintensität aus, die eine akzeptable Signalintensität und ein Signal-Hintergrund-Verhältnis ergibt.
- Um das Signal-zu-Hintergrund-Verhältnis zu bestimmen, messen Sie die Signalintensität in einer Region of Interest (ROI), die Zellen oder Mitochondrien enthält (ROI1) und in einem ROI ohne Zellen oder Mitochondrien (ROI2). Teilen Sie dann die Intensität von ROI1 durch die Intensität von ROI2.
HINWEIS: Streben Sie ein Signal-zu-Hintergrund-Verhältnis von >3 und Signalintensitäten einzelner ROIs von 200-1.000 für 405 nm Anregung mit 1-3% Laserleistung und Intensitäten einzelner ROIs von 300-1.500 für 488 nm Anregung mit 1% Laserleistung an.
- Um das Signal-zu-Hintergrund-Verhältnis zu bestimmen, messen Sie die Signalintensität in einer Region of Interest (ROI), die Zellen oder Mitochondrien enthält (ROI1) und in einem ROI ohne Zellen oder Mitochondrien (ROI2). Teilen Sie dann die Intensität von ROI1 durch die Intensität von ROI2.
- Passen Sie die Detektorverstärkung und den Schwellenwert entsprechend an. Wählen Sie anhand dieser Bilder die niedrigste Laserintensität aus, die eine akzeptable Signalintensität und ein Signal-Hintergrund-Verhältnis ergibt.
- Zeichnen Sie Bilder mit unterschiedlichen Scangeschwindigkeiten und Der Anzahl der Frame-Durchschnittswerte auf. Nehmen Sie 4-5 Bilder für jede Kombination von Einstellungen auf. Wählen Sie basierend auf diesen Bildserien die Einstellungen für die höchste Geschwindigkeit und den niedrigsten Durchschnitt aus, die ein akzeptables Bildrauschen und eine Bild-zu-Bild-Variabilität ergeben.
HINWEIS: Eine Scangeschwindigkeit von 600 Hz und 1-2 Frames für die Mittelwertbildung funktionieren in den meisten Fällen gut. - Nehmen Sie mit einem neuen Deckglas eine Zeitrafferserie mit den optimierten Einstellungen auf.
HINWEIS: Die Dauer und das Bildintervall der Serie sollten denen der geplanten Experimente ähneln. - Am Ende der Zeitraffer-Serie fügen Sie 1 ml 2x DA-Lösung in die Aufnahmekammer hinzu. Bild für weitere 2 min.
- Aspirieren Sie den Bildpuffer mit einer Peristaltikpumpe oder einer Handpipette. Fügen Sie 1 ml 1x DTT-Lösung hinzu. Bild für weitere 5 min.
- Analysieren Sie das Zeitrafferexperiment (siehe Abschnitt 5).
- Stellen Sie mit den optimierten Einstellungen sicher, dass keiner der beiden Kanäle während der DA- und DVB-T-Behandlung über- oder unterbelichtet wird.
- Stellen Sie sicher, dass keiner der beiden Kanäle während der Zeitrafferaufnahme eine erhebliche Bleiche aufweist. <2% Intensitätsverlust zwischen dem ersten und dem letzten Bild anstreben.
- Stellen Sie sicher, dass sich das Verhältnis 405:488 während der Bildgebung nicht wesentlich ändert.
- Wiederholen Sie den gesamten Vorgang iterativ mit mehreren Deckgläsern, bis Einstellungen definiert sind, die konsistent akzeptable Ergebnisse liefern.
4. Beurteilung des basalen Redoxstatus
- Starten Sie das Mikroskop und laden Sie die optimierten Einstellungen aus Abschnitt 3.
- Legen Sie den Frame-Durchschnitt auf 3-5 fest.
- Montieren Sie einen Deckglas mit Zellen in die Bildgebungskammer, fügen Sie 1 ml Bildgebungspuffer hinzu und legen Sie die Kammer auf das Mikroskop.
- Verwenden Sie das Okular und das durchgelassene Licht, um die Zellen zu fokussieren.
HINWEIS: Verwenden Sie kein Epifluoreszenzlicht, um Zellen zu lokalisieren und zu fokussieren. Selbst bei geringer Leistung wirkt sich dies nachteilig auf die Zellen aus. - Wechseln Sie in den Scan-Modus und verwenden Sie den 488-nm-Kanal in der Live-Ansicht , um Zellen für die Bildgebung zu fokussieren und zu lokalisieren.
- Verwenden Sie die Multipoint-Funktion , um 3-5 Sichtfelder auf dem Deckglas auszuwählen.
- Zeichnen Sie ein Baselinebild auf.
- 1 ml 2x DA-Lösung in die Kammer geben.
- Verwenden Sie nach 1, 2 und 3 Minuten die Live-Ansicht , um den Fokus zu bestätigen / anzupassen, und nehmen Sie dann ein Bild auf.
HINWEIS: Die Zellen werden typischerweise nach 2 min vollständig oxidiert. - Ersetzen Sie den Puffer in der Bildgebungskammer durch 1 ml 1x DTT-Lösung.
- Verwenden Sie nach 3 und 5 Minuten die Live-Ansicht , um den Fokus zu bestätigen / anzupassen und dann ein Bild aufzunehmen.
HINWEIS: Die Zellen sind in der Regel nach 4-5 Minuten vollständig reduziert.
5. Live-Bildgebung von Akutbehandlungen
HINWEIS: Das folgende Protokoll beschreibt die Bildgebung des mitochondrialen Redox-Ansprechens auf die NMDA-Behandlung. Bildintervalle und Dauer des Experiments müssen möglicherweise für andere Behandlungen angepasst werden.
- Starten Sie das Mikroskop und laden Sie die optimierten Einstellungen aus Abschnitt 3.
- Stellen Sie das Zeitrafferintervall auf 30 s und die Dauer auf 25 min ein.
- Montieren Sie einen Deckglas mit Zellen in die Bildgebungskammer, fügen Sie 1 ml Bildgebungspuffer hinzu und legen Sie die Kammer auf das Mikroskop.
HINWEIS: Um eine thermische Fokusdrift zu vermeiden, lassen Sie die Zellen 10-15 Minuten auf dem Mikroskoptisch, bevor Sie mit der Zeitrafferaufnahme beginnen. - Verwenden Sie das Okular und das durchgelassene Licht, um die Zellen zu fokussieren.
HINWEIS: Verwenden Sie kein Epifluoreszenzlicht, um Zellen zu lokalisieren und zu fokussieren. Selbst bei geringer Leistung wirkt sich dies nachteilig auf die Zellen aus. - Wechseln Sie in den Scan-Modus und verwenden Sie den 488-nm-Kanal in der Live-Ansicht , um Zellen für die Bildgebung zu fokussieren und zu lokalisieren.
- Optional: Um die Anzahl der aufgezeichneten Zellen pro Durchlauf zu erhöhen, verwenden Sie die Multipoint-Funktion , um 2-3 Sichtfelder pro Deckglas abzubilden.
- Starten Sie die Zeitrafferaufnahme und nehmen Sie 5 Bilder als 2-minütige Baseline-Aufnahme auf.
- Geben Sie 500 μL 3x NMDA-Lösung in die Kammer (Endkonzentration 30 μM) und zeichnen Sie weitere 20 Bilder als 10-minütige NMDA-Antwort auf.
HINWEIS: Neuronen reagieren sehr empfindlich auf Veränderungen der Osmolarität. Stellen Sie daher sicher, dass die Verdunstung des Bildpuffers minimiert wird. Bei längeren Behandlungen sollte die Bildgebungskammer mit einem Deckel abgedeckt werden. - Geben Sie 500 μL 4x DA-Lösung in die Kammer und nehmen Sie 6 weitere Bilder auf (maximale Kalibrierung 3 Minuten).
- Saugen Sie den Puffer aus der Bildgebungskammer ab und ersetzen Sie ihn durch 1 ml 1x DTT-Lösung. Nehmen Sie 10 weitere Bilder auf (mindestens 5 Minuten Kalibrierung).
- Beenden Sie die Aufnahme und speichern Sie die Bildserie.
6. Datenanalyse
- Datenimport und Bildvorverarbeitung in FIDSCHI
- Verwenden Sie den Bioformat-Importer , um eine Gruppe von Bildern aus Schritt 4 oder eine Bilddatei aus Schritt 5 zu öffnen. Klicken Sie auf Plugins | Bio-Formate | Bioformat-Importer. Verwenden Sie im Dialogfeld View stack with: Hyperstack, legen Sie Color mode: default fest, wählen Sie Autoscale aus, und teilen Sie sich nicht in separate Fenster auf.
HINWEIS: Die automatische Skalierung optimiert die Anzeige der Daten auf dem Computerbildschirm. Die Pixelintensitäten werden nicht geändert. - Wenn einzelne Bilder aus Schritt 4 geöffnet wurden, klicken Sie auf Bild | Stapel | Werkzeuge | Verketten Sie sie, um sie zu einem Einzelbildstapel zusammenzuführen.
- Wenn es während der Bildserie zu einer XY-Drift kommt, klicken Sie auf Plugins | StackReg , um die Bilder zu registrieren. Wählen Sie im Dialogfenster Starrkörper (Rigid Body ) oder Übersetzung (Translation).
- Ändern Sie das Bildformat auf 32 Bit, indem Sie auf Bild | Typ | 32-Bit.
- Teilen Sie die Farbkanäle in separate Fenster auf, indem Sie auf Bild | | Geteilte Kanäle.
- Wählen Sie Kanal 1 (405 nm) und passen Sie den Schwellenwert an, um die Mitochondrien für die Analyse auszuwählen, indem Sie auf Bild | | anpassen Schwellenwert. Wählen Sie im Dialogfeld Standard, Rot, Dunkler Hintergrund und Histogramm stapeln aus, und warten Sie, bis die ausgewählten Pixel rot angezeigt werden. Klicken Sie auf Übernehmen. Wählen Sie Hintergrundpixel auf NaN festlegen und Alle Bilder verarbeiten aus.
HINWEIS: Um potenzielle Beobachterverzerrungen zu vermeiden, sollte eine automatisierte Schwellenwertbestimmung verwendet werden. FIJI bietet mehrere automatisierte Methoden (z. B. Default, Huang, Intermodes, Otsu), die aus einem Dropdown-Menü im Dialogfeld "Schwellenwert" ausgewählt werden können. In der Regel liefert die Default-Methode ein gutes Ergebnis. Es wird empfohlen, während der ersten Analyse mehrere Methoden zu vergleichen, um die beste Schwellenwertmethode für den angegebenen Bildsatz zu finden. Sobald eine Methode ausgewählt wurde, muss sie auf alle Bilder angewendet werden. - Wiederholen Sie Schritt 6.1.6 für Kanal 2 (488 nm).
- Erstellen Sie ein Verhältnisbild, um das Verhältnis 405:488 nm zu visualisieren, indem Sie auf Process | Bildrechner. Wählen Sie im Dialogfeld Bild 1: Kanal 1, Operation: Teilen, Bild 2: Kanal 2, Neues Fenster erstellen, Alle Bilder verarbeiten.
- Ändern Sie die Nachschlagetabelle des Verhältnisbilds in pseudocolor. Um beispielsweise zu Fire zu wechseln, klicken Sie auf Image | Nachschlagetabellen | Feuer.
- Verwenden Sie den Bioformat-Importer , um eine Gruppe von Bildern aus Schritt 4 oder eine Bilddatei aus Schritt 5 zu öffnen. Klicken Sie auf Plugins | Bio-Formate | Bioformat-Importer. Verwenden Sie im Dialogfeld View stack with: Hyperstack, legen Sie Color mode: default fest, wählen Sie Autoscale aus, und teilen Sie sich nicht in separate Fenster auf.
- Bildanalyse
- Zeichnen Sie auf dem Verhältnisbild ROIs um einzelne Zellen oder Mitochondrien. Nachdem Sie jeden ROI gezeichnet haben, fügen Sie ihn dem ROI-Manager hinzu. analysieren | Werkzeuge | ROI Manager | Hinzufügen. (Tastenkombination: 'T') Wählen Sie Alle anzeigen aus.
HINWEIS: Da Hintergrundpixel in den Schritten 6.1.6 und 6.1.7 auf "keine Zahl" (NaN) festgelegt wurden, wirken sie sich nicht auf das Ergebnis der Messung aus. Daher ist es akzeptabel, einige Hintergrundpixel in den ROI einzubeziehen. - Messen Sie die 405:488-Verhältnisse einzelner Zellen, indem Sie auf ROI Manager | Strg+A klicken, um alle ROIs | Weitere | Multi-Maßnahme. Wählen Sie im Dialogfeld Alle Segmente messen und Eine Zeile pro Segment aus.
- Exportieren Sie die Messungen in eine Tabellenkalkulationssoftware.
- Wählen Sie das 405-nm-Bild aus. Messen Sie die Intensitäten aller ROIs wie in Schritt 6.2.2. mit den ROIs, die im ROI-Manager gespeichert sind.
- Exportieren Sie die Messungen in eine Tabellenkalkulationssoftware.
- Wählen Sie das 488-nm-Bild aus. Messen Sie die Intensitäten aller ROIs wie in Schritt 6.2.2. mit den ROIs, die im ROI-Manager gespeichert sind.
- Exportieren Sie die Messungen in eine Tabellenkalkulationssoftware.
- Speichern Sie ROIs zur späteren Bezugnahme, indem Sie auf ROI Manager | Strg+A klicken, um alle ROIs | Weitere | Speichern.
- Empfohlen: Generieren Sie Intensitäts- und Zeitdiagramme der 405- und 488-nm-Spuren. Stellen Sie sicher, dass in keinem der Kanäle eine ausgeprägte Bleiche auftritt (die Signalintensität am Ende der Bildgebungsserie sollte ≥98 % des ersten Bildes betragen) und dass sich die beiden Spuren während der Sensorreaktionen in entgegengesetzte Richtungen bewegen (z. B. sollte die 405-nm-Spur während der Oxidation zunehmen, während die 488-nm-Spur abnehmen sollte).
- Zeichnen Sie auf dem Verhältnisbild ROIs um einzelne Zellen oder Mitochondrien. Nachdem Sie jeden ROI gezeichnet haben, fügen Sie ihn dem ROI-Manager hinzu. analysieren | Werkzeuge | ROI Manager | Hinzufügen. (Tastenkombination: 'T') Wählen Sie Alle anzeigen aus.
- Datennormalisierung
- Bestimmen Sie für jeden ROI aus dem Verhältnisbild den Maximalwert während der DA-Behandlung (Rmax) und den Minimalwert während der DTT-Behandlung (Rmin).
- Berechnen Sie das normalisierte Verhältnis wie folgt:
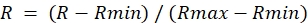
HINWEIS: Dadurch wird das maximale Verhältnis auf 1,0 und das minimale Verhältnis auf 0 festgelegt.
- Analyse der mitochondrialen Morphologie
- Um Messungen der mitochondrialen Morphologie parallel zu den roGFP-Intensitäten in Schritt 6.2.6 zu erhalten, gehen Sie zu analysieren | Stellen Sie Maße ein und aktivieren Sie Formdeskriptoren und Ellipse anpassen.
HINWEIS: Zusätzlich zur mittleren Intensität umfassen die Messungen im Ergebnisfenster die Länge der Hauptachse (Major), die Länge der Nebenachse (Minor), das Seitenverhältnis (AR; Hauptachse geteilt durch die Nebenachse; runde Mitochondrien haben einen AR ~ 1, längliche Mitochondrien haben eine größere AR) sowie Messungen der Zirkularität (Circ.) und Rundheit (Round).
- Um Messungen der mitochondrialen Morphologie parallel zu den roGFP-Intensitäten in Schritt 6.2.6 zu erhalten, gehen Sie zu analysieren | Stellen Sie Maße ein und aktivieren Sie Formdeskriptoren und Ellipse anpassen.
Ergebnisse
Quantifizierung von Unterschieden im stationären mitochondrialen Redoxzustand nach Wachstumsfaktorentzug
Um die Quantifizierung von Steady-State-Unterschieden im mitochondrialen Redoxzustand zu demonstrieren, wurden primäre Neuronen, die in einem Standardmedium gezüchtet wurden, mit Neuronen verglichen, die 48 Stunden vor der Bildgebung ohne Wachstumsfaktoren kultiviert wurden. Der Entzug des Wachstumsfaktors führt nach 72 h16 zum apoptotischen neuronalen Zelltod. Die Zelle...
Diskussion
Quantitative und dynamische Messungen des mitochondrialen Redoxzustandes liefern wichtige Informationen über die mitochondriale und zelluläre Physiologie. Es stehen mehrere fluorogene chemische Sonden zur Verfügung, die reaktive Sauerstoffspezies, "Redoxstress" oder "oxidativen Stress" erkennen. Die letztgenannten Begriffe sind jedoch nicht klar definiert und haben oft keine Spezifität9,17,18. Im Vergleich zu chemischen Farb...
Offenlegungen
Die Autoren erklären, dass sie keinen Interessenkonflikt haben.
Danksagungen
Diese Arbeit wurde von der Deutschen Forschungsgemeinschaft (BA 3679/5-1; FÜR 2289: BA 3679/4-2). A.K. wird durch ein ERASMUS+ Stipendium unterstützt. Wir danken Iris Bünzli-Ehret, Rita Rosner und Andrea Schlicksupp für die Präparation primärer Neuronen. Wir danken Dr. Tobias Dick für die Bereitstellung von pLPCX-mito-Grx1-roGFP2. Die in Abbildung 4 gezeigten Experimente wurden am Nikon Imaging Center der Universität Heidelberg durchgeführt. Abbildung 2 wurde mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| reagents | |||
| Calcium chloride (CaCl2·2H2O) | Sigma-Aldrich | C3306 | |
| Diamide (DA) | Sigma-Aldrich | D3648 | |
| Dithiothreitol (DTT) | Carl Roth GmbH | 6908.1 | |
| Glucose (2.5 M stock solution) | Sigma-Aldrich | G8769 | |
| Glucose | Sigma-Aldrich | G7528 | |
| Glycine | neoFroxx GmbH | LC-4522.2 | |
| HEPES (1 M stock solution) | Sigma-Aldrich | 15630-080 | |
| HEPES | Sigma-Aldrich | H4034 | |
| Magnesium chloride (MgCl2·6H2O) | Sigma-Aldrich | 442611-M | |
| N-methyl-D-aspartate (NMDA) | Sigma-Aldrich | M3262 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P3911 | |
| Sodium chloride (NaCl) | neoFroxx GmbH | LC-5932.1 | |
| Sodium pyruvate (0.1 M stock solution) | Sigma-Aldrich | S8636 | |
| Sodium pyruvate | Sigma-Aldrich | P8574 | |
| Sucrose | Carl Roth GmbH | 4621.1 | |
| Tetramethylrhodamine ethyl ester perchlorate (TMRE) | Sigma-Aldrich | 87917 | |
| equipment | |||
| imaging chamber | Life Imaging Services (Basel, Switzerland) | 10920 | Ludin Chamber Type 3 for Ø12mm coverslips |
| laser scanning confocal microscope, microscope | Leica | DMI6000 | |
| laser scanning confocal microscope, scanning unit | Leica | SP8 | |
| peristaltic pump | VWR | PP1080 181-4001 | |
| spinning disc confocal microscope, camera | Hamamatsu | C9100-02 EMCCD | |
| spinning disc confocal microscope, incubationsystem | TokaiHit | INU-ZILCF-F1 | |
| spinning disc confocal microscope, microscope | Nikon | Ti microscope | |
| spinning disc confocal microscope, scanning unit | Yokagawa | CSU-X1 | |
| software | |||
| FIJI | https://fiji.sc | ||
| StackReg plugin | https://github.com/fiji-BIG/StackReg/blob/master/src/main/java/StackReg_.java | ||
| TurboReg plugin | https://github.com/fiji-BIG/TurboReg/blob/master/src/main/java/TurboReg_.java |
Referenzen
- Roede, J. R., Go, Y. M., Jones, D. P. Redox equivalents and mitochondrial bioenergetics. Methods in Molecular Biology. 810, 249-280 (2012).
- Turrens, J. F. Mitochondrial formation of reactive oxygen species. Journal of Physiology. 552, 335-344 (2003).
- Lin, M. T., Beal, M. F. Mitochondrial dysfunction and oxidative stress in neurodegenerative diseases. Nature. 443 (7113), 787-795 (2006).
- Manfredi, G., Beal, M. F. The role of mitochondria in the pathogenesis of neurodegenerative diseases. Brain Pathology. 10 (3), 462-472 (2000).
- Mari, M., Morales, A., Colell, A., Garcia-Ruiz, C., Fernandez-Checa, J. C. Mitochondrial glutathione, a key survival antioxidant. Antioxidants & Redox Signaling. 11 (11), 2685-2700 (2009).
- Murphy, M. P. Mitochondrial thiols in antioxidant protection and redox signaling: distinct roles for glutathionylation and other thiol modifications. Antioxidants & Redox Signaling. 16 (6), 476-495 (2012).
- Dooley, C. T., et al. Imaging dynamic redox changes in mammalian cells with green fluorescent protein indicators. Journal of Biological Chemistry. 279 (21), 22284-22293 (2004).
- Hanson, G. T., et al. Investigating mitochondrial redox potential with redox-sensitive green fluorescent protein indicators. Journal of Biological Chemistry. 279 (13), 13044-13053 (2004).
- Gutscher, M., et al. Real-time imaging of the intracellular glutathione redox potential. Nature Methods. 5 (6), 553-559 (2008).
- Morgan, B., Sobotta, M. C., Dick, T. P. Measuring E(GSH) and H2O2 with roGFP2-based redox probes. Free Radical Biology & Medicine. 51 (11), 1943-1951 (2011).
- Marwick, K. F. M., Hardingham, G. E. Transfection in primary cultured neuronal cells. Methods in Molecular Biology. 1677, 137-144 (2017).
- Kohrmann, M., et al. convenient, and effective method to transiently transfect primary hippocampal neurons. Journal of Neuroscience Research. 58 (6), 831-835 (1999).
- Depp, C., Bas-Orth, C., Schroeder, L., Hellwig, A., Bading, H. Synaptic activity protects neurons against calcium-mediated oxidation and contraction of mitochondria during excitotoxicity. Antioxidants & Redox Signaling. 29 (12), 1109-1124 (2018).
- Hauck, B., Chen, L., Xiao, W. Generation and characterization of chimeric recombinant AAV vectors. Molecular Therapy. 7 (3), 419-425 (2003).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. Biochemical Journal. 435 (2), 297-312 (2011).
- Zhang, S. J., et al. Nuclear calcium signaling controls expression of a large gene pool: identification of a gene program for acquired neuroprotection induced by synaptic activity. PLoS Genetics. 5 (8), 1000604 (2009).
- Winterbourn, C. C. The challenges of using fluorescent probes to detect and quantify specific reactive oxygen species in living cells. Biochimica et Biophysica Acta. 1840 (2), 730-738 (2014).
- Sies, H. Oxidative stress: a concept in redox biology and medicine. Redox Biology. 4, 180-183 (2015).
- Lukyanov, K. A., Belousov, V. V. Genetically encoded fluorescent redox sensors. Biochimica et Biophysica Acta. 1840 (2), 745-756 (2014).
- Nietzel, T., et al. Redox-mediated kick-start of mitochondrial energy metabolism drives resource-efficient seed germination. Proceedings of the National Academy of Sciences of the United States of America. 117 (1), 741-751 (2020).
- Albrecht, S. C., et al. Redesign of genetically encoded biosensors for monitoring mitochondrial redox status in a broad range of model eukaryotes. Journal of Biomolecular Screening. 19 (3), 379-386 (2014).
- Albrecht, S. C., Barata, A. G., Grosshans, J., Teleman, A. A., Dick, T. P. In vivo mapping of hydrogen peroxide and oxidized glutathione reveals chemical and regional specificity of redox homeostasis. Cell Metabolism. 14 (6), 819-829 (2011).
- Breckwoldt, M. O., et al. Multiparametric optical analysis of mitochondrial redox signals during neuronal physiology and pathology in vivo. Nature Medicine. 20 (5), 555-560 (2014).
- Ricke, K. M., et al. Mitochondrial dysfunction combined with high calcium load leads to impaired antioxidant defense underlying the selective loss of nigral dopaminergic neurons. Journal of Neuroscience. 40 (9), 1975-1986 (2020).
- Bjornberg, O., Ostergaard, H., Winther, J. R. Mechanistic insight provided by glutaredoxin within a fusion to redox-sensitive yellow fluorescent protein. Biochemistry. 45 (7), 2362-2371 (2006).
- Shokhina, A. G., et al. Red fluorescent redox-sensitive biosensor Grx1-roCherry. Redox Biology. 21, 101071 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten