Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verwendung eines Echtzeit-Zellstoffwechselflussanalysators zur Überwachung der Bioenergetik des Osteoblast
In diesem Artikel
Zusammenfassung
Der Echtzeit-Zellstoffwechselfluss-Assay misst die Sauerstoffverbrauchsrate und die extrazelluläre Säuerungsrate, die der mitochondrialen und glykolytischen Adenosintriphosphatproduktion entspricht, unter Verwendung von pH- und Sauerstoffsensoren. Das Manuskript erklärt eine Methode zum Verständnis des Energiestatus von Osteoblasten und die Charakterisierung und Interpretation des zellulären bioenergetischen Status.
Zusammenfassung
Die Knochenbildung durch Osteoblasten ist ein wesentlicher Prozess für den richtigen Knochenerwerb und Knochenumsatz, um die Skeletthomöostase aufrechtzuerhalten und letztendlich Frakturen zu verhindern. In dem Interesse, sowohl die maximale Knochenmasse zu optimieren als auch verschiedene Muskel-Skelett-Erkrankungen (d.h. postmenopausale Osteoporose, Anorexia nervosa, Typ-1- und Typ-2-Diabetes mellitus) zu bekämpfen, wurden auf dem Gebiet der Knochenbiologie unglaubliche Anstrengungen unternommen, um Osteoblasten während ihres gesamten Differenzierungsprozesses vollständig zu charakterisieren. Angesichts der primären Rolle reifer Osteoblasten bei der Sekretion von Matrixproteinen und Mineralisierungsvesikeln wurde festgestellt, dass diese Prozesse eine unglaubliche Menge an zellulärer Energie oder Adenosintriphosphat (ATP) aufnehmen. Der gesamte zelluläre Energiestatus wird oft als zelluläre Bioenergetik bezeichnet und umfasst eine Reihe von Stoffwechselreaktionen, die die Substratverfügbarkeit erfassen, um ATP abzuleiten, um den zellulären Bedarf zu decken. Daher beschreibt die aktuelle Methode den Prozess der Isolierung primärer, muriner Knochenmarkstromazellen (BMSCs) und der Überwachung ihres bioenergetischen Status mit dem Echtzeit-Zellstoffwechselflussanalysator in verschiedenen Stadien der Osteoblastendifferenzierung. Wichtig ist, dass diese Daten gezeigt haben, dass sich das metabolische Profil während der Osteoblastendifferenzierung dramatisch ändert. Daher ist die Verwendung dieses physiologisch relevanten Zelltyps erforderlich, um vollständig zu verstehen, wie der bioenergetische Status einer Zelle die Gesamtfunktion regulieren kann.
Einleitung
Die Knochenbildung durch den Osteoblasten geht mit einer koordinierten Zerstörung oder Resorption der Knochen durch Osteoklasten einher. Das Gleichgewicht zwischen osteoblastischer Knochenbildung und Osteoklastenresorption ist ein gekoppelter Prozess, der den Knochenumsatz oder -umbau beschreibt, der für die Skeletthomöostase unerlässlich ist. Osteoblast-Dysfunktion führt zu einer gestörten Knochenbildung und führt zu verschiedenen Krankheiten, einschließlich Osteoporose 1,2,3. Die Ex-vivo/In-vitro-Differenzierung von Knochenmark-Stroma-Stammzellen (BMSCs) zu Osteoblasten-Vorläufern und reifen Osteoblasten führt im Laufe der Zeitzur Bildung und Ablagerung der mineralisierten Knochenmatrix im Kulturgefäß 4,5,6. Diese Knochenbildung durch das Osteoblast erfordert eine erhebliche Menge an zellulärer Energie. Insbesondere wurde gezeigt, dass Kollagensynthese und -sekretion stark von zellulärem ATP abhängen: ADP-Verhältnisse, und vermutlich erfordern der Transport und die Sekretion von mineralisierten Vesikeln zusätzliches ATP 7,8,9,10,11. Viele Forscher haben gezeigt, dass der Prozess der Osteoblastogenese und der Osteoblastenfunktion eine ausreichende Energiezufuhr erfordert, um den metabolischen Bedarf an Knochenbildung zu decken 12,13,14,15,16. Daher ist es das Ziel dieser Methode, den bioenergetischen Status von primären, murinen Stromazellen während der Osteoblastendifferenzierung mit dem Echtzeit-Zellstoffwechselflussanalysator zu charakterisieren. Diese Techniken tragen dazu bei, ein besseres Verständnis der Skeletthomöostase zu entwickeln, was letztendlich zur Entwicklung neuartiger therapeutischer Optionen führen kann, die in der Lage sind, Skeletterkrankungen zu verbessern.
Der Echtzeit-Zellstoffwechselflussanalysator kann verwendet werden, um die Sauerstoffverbrauchsrate (OCR) und die extrazelluläre Versauerungsrate (ECAR) lebender Osteoblasten zu messen, was der mitochondrialen bzw. glykolytischen ATP-Produktion entspricht. Grundlegend für diese Methodik ist die Tatsache, dass ein H + -Ion pro Laktat während der Glykolyse bei der Umwandlung von Glukose in Laktat freigesetzt wird, was den in den ECAR-Werten widergespiegelten Medien-pH-Wert verändert. Umgekehrt erzeugt die oxidative Phosphorylierung über die Mitochondrien während des TCA-Zyklus (Tricarbonsäure) CO 2 durch die Verwendung oder den Verbrauch von Sauerstoff, und daher spiegelt die Überwachung der OCR diesen Stoffwechselprozess wider. Der Analysator misst sowohl OCR als auch ECAR in der extrazellulären Mikroumgebung gleichzeitig und in Echtzeit, was ein enormes Potenzial bei der Untersuchung der zellulären Bioenergetik ermöglicht 6,17. Darüber hinaus ist die Durchführung dieser Assays relativ einfach und je nach experimentellem Ziel leicht anpassbar. Ähnliche Techniken wurden angewendet, um die metabolische Regulation des Immunsystems durch T-Zellen18,19, die Krebsinitiation und -progression 20 sowie mehrere andere Zelltypen, die zu metabolischen Syndromen beitragen, besser zu verstehen 21,22.
Zu den Vorteilen des Echtzeit-metabolischen Flussanalysators gegenüber alternativen Techniken gehören (1) die Fähigkeit, die zelluläre Bioenergetik lebender Zellen in Echtzeit zu messen, (2) die Fähigkeit, Assays mit einer relativ kleinen Anzahl von Zellen durchzuführen (erfordert nur 5.000 Zellen), (3) Injektionsanschlüsse zur parallelen Manipulation mehrerer Behandlungen in einem Hochdurchsatz-96-Well-System, (4) Verwendung eines radioaktiven markierungsfreien automatisierten Zellbildgebungsgeräts zur Normalisierung18, 23,24 Die folgenden Methoden zielen darauf ab, eine verallgemeinerte, aber detaillierte Beschreibung der Überwachung der zellulären Bioenergetik in murinen BMSCs während der Osteoblastendifferenzierung mit dem Analysator zu liefern. Es wird routinemäßig durchgeführte Assays umfassen; Wie bei vielen Techniken und Methoden wird jedoch dringend empfohlen, dass einzelne Labore spezifische Details für ihre Experimente festlegen.
Auswahl an Assays und verschiedenen Arten von Assays verfügbar: Eine Vielzahl von Assay-Kits und Reagenzien steht zur Verfügung, um die Bioenergetik von Zellen zu untersuchen und gleichzeitig die Zuverlässigkeit und Konsistenz der experimentellen Ergebnisse zu gewährleisten. Darüber hinaus bietet die Desktop-Software auch Assay-Vorlagen, die leicht angepasst werden können. Der Assay kann basierend auf den Bedürfnissen des Benutzers definiert werden, um verschiedene metabolische Parameter zu messen. Diese Assays können auf verschiedene Weise modifiziert werden, basierend auf dem experimentellen Ziel und / oder der wissenschaftlichen Fragestellung. Zum Beispiel können mit vier Injektionsports mehrere Verbindungen in die Assay-Medien injiziert werden, um die zelluläre Reaktion zu analysieren, die für jeden Stoffwechselweg spezifisch ist.
Zellenergie-Phänotyp-Test: Dieser Assay misst den metabolischen Phänotyp und das metabolische Potenzial der lebenden Zellen. Dieser Assay wird auch als erster Schritt empfohlen, um eine verallgemeinerte Vorstellung vom pfadspezifischen Stoffwechsel zu erhalten. Eine Mischung aus Oligomycin-A-an-Inhibitor der ATP-Synthase und Carbonylcyanid 4-(Trifluormethoxy)-phenylhydrazon (FCCP)-einem mitochondrialen Entkopplungsmittel wird injiziert, um das Zellenergiepotential zu verstehen. Die Injektion von Oligomycin A hemmt die Synthese von ATP, was zu einer Erhöhung der Glykolyserate (ECAR) führt, damit die Zellen ihren Energiebedarf decken können; Auf der anderen Seite führt die Injektion von FCCP zu einer höheren OCR aufgrund der Depolarisation der mitochondrialen Membran. Im Wesentlichen zeigt dieser Assay die Grundstoffwechselatmung und nach den doppelten Injektionen, Pushes oder Stress die metabolische Reaktion. Basierend auf diesen Parametern zeichnet die Software dann OCR und ECAR der Zellen auf, indem sie die Zellen im Laufe der Zeit als aeroben, ruhenden, glykolytischen oder energetischen Zustand klassifiziert25,26.
ATP-Echtzeit-Analyse der Produktionsrate: Dieser misst die zelluläre ATP-Produktion gleichzeitig aus Glykolyse und mitochondrialer Atmung. Dieser Assay misst quantitativ die metabolischen Verschiebungen aus den beiden Energiebahnen und liefert Daten über die mitochondrialen und glykolytischen ATP-Produktionsraten im Laufe der Zeit. Der Assay erhält basale OCR- und ECAR-Daten, gefolgt von der Berechnung der mitochondrialen ATP-Produktionsrate durch Injektion von Oligomycin A und der glykolytischen ATP-Produktionsrate durch Injektion von Rotenon + Antimycin A-Mischung (totale Hemmung der mitochondrialen Funktion), was zu einer mitochondrialen Versauerung führt17,27.
Zellmitochondrien-Stresstest (oder Zell-Mito-Stresstest): Dies misst die mitochondriale Funktion durch ATP-verknüpfte Atmung, quantifiziert die zelluläre Bioenergetik, identifiziert mitochondriale Dysfunktion und misst die Reaktion der Zellen auf Stress. Verschiedene Parameter, einschließlich basale und freie Atemkapazität, ATP-gebundene Atmung, maximale Atmung und nicht-mitochondrialer Sauerstoffverbrauch, können in einem Assay erhalten werden. Dieser Assay beinhaltet sequentielle Injektionen von Oligomycin A, FCCP (mitochondriales Entkopplungsmittel), einer Mischung aus Rotenon/Antimycin-A-Hemmern, um deren Wirkung auf die mitochondriale Funktion effizient zu analysieren28.
Flexibilität mito fuel flex test: Dies misst die mitochondriale Atmungsrate durch die Oxidation der drei primären mitochondrialen Brennstoffe durch das Vorhandensein und Fehlen ihrer Inhibitoren. Die sequentielle Hemmung von Glukose, Glutamin und Fettsäuren hilft bei der Messung der Abhängigkeit, Kapazität und Flexibilität von Zellen und der Abhängigkeit der Zellen in verschiedenen zellulären Wegen, um den Energiebedarf zu decken. Wenn die Mitochondrien die Anforderungen des blockierten Weges von Interesse nicht durch Oxidation anderer Brennstoffe erfüllen können, treten die Zellen in einen Abhängigkeitszustand ein. Die Kapazität der Zellen wird durch Hemmung der beiden anderen alternativen Wege berechnet, gefolgt von der Hemmung des interessierenden Signalwegs. Die Flexibilität der Zellen hilft beim Verständnis der Fähigkeit der Mitochondrien, den Brennstoffbedarf des gehemmten Weges zu kompensieren und zu decken. Es wird berechnet, indem die Abhängigkeit von Zellen von der Kapazität der Zellen subtrahiert wird. Drei verschiedene Inhibitoren werden unabhängig voneinander oder als Mischung aus zwei verwendet, um die Assay-Parameter effektiv zu berechnen. 2-Cyano-3-(1-phenyl-1H-indol-3-yl)-2-propensäure (UK5099) hemmt die Oxidation von Glukose, indem sie den Pyruvatträger in der Glykolyse blockiert. Bis-2-(5-phenylacetamido-1,3,4-thiadiazol-2-yl) (BPTES)-ethylsulfid hemmt den Glutaminoxidationsweg und Etomoxir hemmt die Oxidation langkettiger Fettsäuren29.
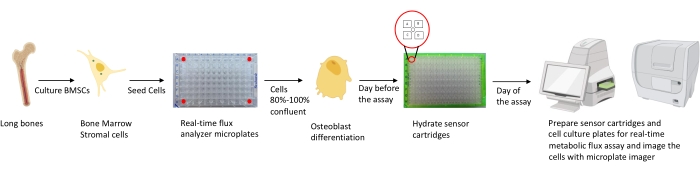
Abbildung 1: Schematische Darstellung der Methodik zur Kultivierung und Vorbereitung von Osteoblasten für die Analyse. Murine BMSCs werden aus langen Knochen isoliert, kultiviert und in 96-Well-Platten bei 25.000 Zellen / Well-Dichte ausgesät. Die Kultivierung dieser Zellen in Osteoblast-spezifischen Medien beginnt, wenn sie eine 80% -100% ige Konfluenz erreichen, um ihre Differenzierung zu beginnen. Die Assays werden in verschiedenen Stadien der Differenzierung durchgeführt. Die Kartuschenplatten werden einen Tag vor dem Assay mit Feuchtigkeit versorgt. Am Tag des Assays werden verschiedene Inhibitoren in die Ports der Sensorkartuschen injiziert, basierend auf den Assay-Anforderungen, und der 96-Well-Kalibrierplatte wird ein Kalibrierpuffer hinzugefügt. Nach der Kalibrierung wird der Echtzeit-Zellstoffwechselfluss-Assay durchgeführt, gefolgt von der Abbildung der Zellkultur-Mikroplatte mit dem Mikroplatten-Imager, um Echtzeit-Daten des Zellstoffwechselflussanalysators mit der Zellzahl zu normalisieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protokoll
Alle Verfahren basierten auf den Richtlinien und der Genehmigung des Institutional Animal Care and Use Committee am Vanderbilt University Medical Center.
1. Vorbereitung von Reagenzien und Assay-Setup
- Isolierung und Kultivierung von Stromazellen des Knochenmarks (siehe auch den vorherigen Artikel30).
- Bereiten Sie vollständige Alpha-Minimum-Essential-Medien (αMEM) Zellkulturmedien vor, indem Sie minimale essentielle Medien mit Alpha-Modifikation mit 10% FBS (fetales Rinderserum), 100 U / ml Penicillin und 100 μg / ml Streptomycin ergänzen.
- Bereiten Sie das Knochenmarksammelröhrchen vor, indem Sie das Ende eines 0,6-ml-Mikrozentrifugenröhrchens so beschneiden, dass die Zellen hindurchgehen können, und es in ein 1,5-ml-Mikrozentrifugenröhrchen einführen, das 100 μL vollständiges αMEM enthält.
- Euthanasieren Sie die Mäuse mit CO2-Behandlung wie folgt. Legen Sie das Tier für 2-3 min oder bis zur Einstellung der Atmung in die CO2 -Kammer. Warten Sie mindestens 1 Minute, nachdem das Tier bewusstlos geworden ist, um die Mäuse aus der Kammer zu entfernen und zervikal zu verrenken.
- Schneiden Sie mit einer sterilen Pinzette und einer Schere den Unterbauch der eingeschläferten Mäuse auf, um einen kleinen Schnitt zu machen. Isolieren Sie die langen Knochen (Femur, Tibia und Beckenkamm) der Mäuse.
- Schneiden Sie die langen Knochen ab, um das gesamte Weichgewebe zu entfernen. Sobald der Knochen gereinigt ist, schneiden Sie ~ 1-2 mm von den distalen und proximalen Enden, um eine Öffnung für das Knochenmark zu schaffen, um durchzuspülen.
HINWEIS: Diese Öffnung sollte konservativ sein, um zu vermeiden, dass das Mark verloren geht, während es ausgespült werden kann. - Legen Sie die Knochen in ein Auffangröhrchen, das 100 μL 1x steriles PBS (phosphatgepufferte Kochsalzlösung) enthält, um das gesamte Knochenmark zu isolieren.
- Das Mark durch Zentrifugation bei 10.000 x g für 15-20 s bei Raumtemperatur spülen. Markzellenpellet am Boden des Rohres.
- Wiederholen Sie die Zentrifugation, bis die Knochenhöhle weiß und frei von den meisten Knochenmarkelementen erscheint. Resuspendieren Sie die gemischte Population von Knochenmark, indem Sie vorsichtig auf und ab pipettieren.
- Die Zellen eines Tieres (sowohl Femur als auch Tibia) in einem 75 cm2 Zellkulturkolben in 10 ml Zellkulturmedien kultivieren und bei 37 °C in einem Zellkulturinkubator mit 5%CO2 inkubieren. Wenn Sie Zellen von 2-3 Tieren sammeln, verwenden Sie einen 150 cm2 Zellkulturkolben (empfohlen).
- Nach 24-48 h Inkubation der Mischpopulation aspirieren Sie die in den Kulturmedien enthaltene nicht-adhärente hämatopoetische Zellpopulation und waschen die adhärenten Zellen mit 1x PBS.
- Zellaussaat aus BMSCs und Osteoblastendifferenzierung
- Trypsinisieren Sie die adhärenten Zellen, indem Sie genügend 0,25% Trypsin-EDTA (ca. 3-4 ml) hinzufügen, um die Kolbenoberfläche leicht zu bedecken, gefolgt von einer 3-minütigen Inkubation bei 37 ° C.
- Fügen Sie 6-7 ml vollständiges αMEM in den Kolben/Trypsin hinzu, um die adhärenten BMSCs durch vorsichtiges Auf- und Abpipettieren wieder aufzuheben. Übertragen Sie die BMSC-Suspension auf ein konisches Zentrifugenrohr.
- Entfernen Sie ein 50 μL Aliquot der BMSC-Suspension und fügen Sie 50 μL Trypanblau (1:1-Verdünnung) hinzu. Zählen Sie die Gesamtzahl der lebensfähigen Zellen, die den Farbstoff ausschließen, indem Sie 10 μL dieser Mischung auf ein Hämozytometer pipettieren und unter dem Mikroskop beobachten. Zählen Sie keine toten oder ungesunden Zellen, die blau gefärbt erscheinen (<10% Zellen).
- Berechnen Sie basierend auf der Zellzahl das Volumen der Zellsuspension in vollständigem αMEM, das für eine Endkonzentration von 2,4 x 106 Zellen/ml für ein Gesamtvolumen von mindestens 10 ml pro Platte benötigt wird.

Abbildung 2: Die speziell für den Analysator entwickelte Zellkultur-Mikrotiterplatte . (A) Die vier Hintergrundkorrekturwellen A1, A12, H1, H12 sind hervorgehoben. Diese Vertiefungen enthalten nur Assay-Medien ohne Zellen. (B) Der Barcode an der Seite der Platte, um die Platte mit dem bildgebenden Lesegerät und dem Analysator zu scannen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Zentrifugieren Sie die Zellen im konischen Röhrchen bei 1.000 x g für 5 min und resuspendieren Sie die Zellen auf die gewünschte Endkonzentration von 2,4 x 106 Zellen/ml.
- Übertragen Sie die Zellsuspension in ein Reservoir und resuspendieren Sie die Zellen vorsichtig mit einer Mehrkanalpipette, um eine homogene Mischung von Zellen zu gewährleisten.
- Saatgut 2,5 x 104 Zellen pro Vertiefung in der 96-Well Zellkultur-Mikrotiterplatte mit 80 μL vollständigem αMEM. Samen Sie keine Zellen in den Hintergrundkorrekturvertiefungen (A1, A12, H1, H12); Fügen Sie stattdessen einfach das Medium in diese vier Vertiefungen ein.
HINWEIS: BMSCs für die Assays sind mit einer 96-Well-Zellkultur-Mikrotiterplatte plattiert, die für den Analysator in Verbindung mit den Sensorkartuschen entwickelt wurde. Die Oberfläche dieser Platten unterscheidet sich von einer normalen 96-Well-Platte. Die Oberfläche jedes Bohrlochs in der Platte beträgt 0,106 cm2, was ungefähr 40% der typischen Plattenfläche von 96 Vertiefungen entspricht. Die optimale Zellaussaatdichte wird basierend auf dem Zelltyp gewählt. Typischerweise kann der Analysator zwischen 0,5-4 x 104 Zellen pro Vertiefung erkennen. Osteoblasten müssen in Kontakt sein, um effektiv zu differenzieren; Zu diesem Zweck wurde eine Beschichtung zwischen 2,0 x 10 4-3,0 x 104 BMSCs/Well in 80 μL vollständigem αMEM gewählt. - Rühren Sie die Platte vorsichtig auf, um die gleichmäßige Verteilung der Zellen in den Vertiefungen zu gewährleisten, und inkubieren Sie bei 37 ° C, 5% CO2. Überprüfen Sie das Wachstum der Zellen und den Zellzusammenfluss unter dem Mikroskop nach 48 h. Wechseln Sie bei Bedarf das Zellkulturmedium.
- Je nach Ziel des Assays, wenn BMSCs zu 60%-80% konfluent sind (typischerweise 48-72 h), wird die Osteoblastendifferenzierung eingeleitet, indem die Zellkulturmedien in Osteoblastendifferenzierungsmedien umgewandelt werden (vollständiges αMEM ergänzt mit 5 mM β-Glycerinphosphat und 50 μg/ml L-Ascorbinsäure).
- Wenn undifferenzierte Stromazellen (Tag 0) analysiert werden sollen, halten Sie die Zellen unter vollständigem αMEM.
- Wechseln Sie die Osteoblasten-Differenzierungsmedien jeden zweiten Tag und visualisieren Sie die Zellen unter dem Mikroskop, um sicherzustellen, dass sie bis zum Tag des Assays gesund sind. Vorzugsweise 24 Stunden vor dem geplanten Assay, wechseln Sie das Medium und halten Sie einen konsistenten mittleren Wechselzeitplan ein (empfohlen).
HINWEIS: Wechseln Sie das Medium vorsichtig, indem Sie die Platten leicht in einem Winkel neigen. Dadurch wird ein versehentlicher Kontakt der Pipettenspitzen mit den Zellkulturplatten und die Störung der Monoschicht der Zellen vermieden.
2. Vorbereitung der Sensorkartusche für die Kalibrierung des extrazellulären Flusses
- Hydratisieren Sie die Sensorkartuschen aus dem extrazellulären Assay-Kit vor dem Tag des Assays. Entfernen Sie die Sensorpatronen (grüne Platte) und stellen Sie die Sensoren kopfüber auf.
- Mit einer Mehrkanalpipet 200 μLH2 Ozu jeder Vertiefung der Utility-Platte hinzufügen. Legen Sie die Sensorpatronen vorsichtig wieder auf die Gebrauchsplatte und inkubieren Sie die Platte über Nacht bei Raumtemperatur.
HINWEIS: Der Hersteller empfiehlt, die Sensorkartuschen in einem Non-CO2 37 °C Inkubator über Nacht zu inkubieren. Es kann jedoch zu einer erheblichen Verdunstung der Sensorpatronen kommen. In diesem Fall können Sensorpatronen bei Raumtemperatur inkubiert werden. Diese Platten sollten für mindestens 4 h und maximal 72 h inkubiert werden. - Entsorgen Sie am Tag des Assays dasH2 Ovon der Gebrauchsplatte und fügen Sie 200 μL Kalibrant hinzu. Inkubieren Sie die Gebrauchsplatte für mindestens 1 Stunde vor dem Assay.
3. Echtzeit-Zellstoffwechselflussanalysator Medienvorbereitung
- Verwenden Sie die phenolrotfreien DMEM-Medien mit einem voreingestellten pH-Wert von 7,4 (empfohlen), um den Assay mit BMSCs durchzuführen.
- Bereiten Sie 80 ml Assay-Medien vor, indem Sie DMEM-Medien mit 1 mM Natriumpyruvat, 2 mM Glutamin, 10 mM Glukose, 200 nM Insulin, 50-200 μM Ölsäure BSA ergänzen.
- Inkubieren Sie die kompletten Assay-Medien bei 37 °C in einem Wasserbad.
4. Herstellung von Verbindungen für die Sensorpatronen
- Tauen Sie Oligomycin A, Rotenon und Antimycin A auf Eis auf. Pipette auf und ab, um die Verbindungen vor dem Gebrauch zu lösen.
- Jedem Röhrchen werden 3 ml vorbereitetes Assay-Medium zugesetzt, gefolgt von der Zugabe des jeweiligen Verbindungsröhrchens A: 26,4 μl 2,5 mM Oligomycin A; Röhrchen B: 3,1 μL 12,67 mM Rotenon + 4,1 μL 9,4 mM Antimycin A + 30 μL Hoechst Färbung.
- Laden Sie eine 10-fache Konzentration dieser Inhibitoren in den entsprechenden Port. Die endgültige Konzentration der benötigten Injektionslösungen beträgt 2 μM Oligomycin A, 1 μM Rotenon und 4,1 μM Antimycin A.
HINWEIS: Hoechst wird dem letzten Injektionsanschluss hinzugefügt, um die Kerne für Bildgebungs- und Normalisierungszwecke fluoreszierend zu färben. Diese Konzentrationen können basierend auf dem Zelltyp optimiert werden. - Laden Sie 20 μL dieser Verbindungen in 180 μL Assay-Medien in die Zellen.
5. Zellkultur-Mikrotiterplatte für den Assay vorbereiten
- Entfernen Sie die Zellkultur-Mikrotiterplatte aus dem 37 °C Inkubator und beobachten Sie die Zellen unter dem Mikroskop.
- Entfernen Sie das Assay-Medium aus dem Wasserbad.
- Waschen Sie die Zellen vorsichtig zweimal mit 200 μL Assay-Medium und fügen Sie 200 μL Assay-Medien pro Vertiefung hinzu.
HINWEIS: Sobald die endgültigen Assay-Medien zu den Zellen hinzugefügt wurden, ist die Zeit, bis die Platten in den Analysator gelangen, entscheidend. Beginnen Sie daher erst dann mit dem Austausch des Mediums, wenn die folgenden Schritte innerhalb von 1 Stunde ausgeführt wurden. - Überprüfen Sie die Zellen unter dem Mikroskop, um sicherzustellen, dass die Zellen an den Vertiefungen haften bleiben.
- Stellen Sie sicher, dass die Zellen in D5 und E8 mit einer konsistenten Monoschicht verklebt sind und während des Waschschritts nicht weggespült wurden. Die Zellbildgebungssoftware verwendet diese beiden Vertiefungen zum Einstellen des Autofokus und der Autobelichtung.
HINWEIS: Der Hersteller empfiehlt, die Platte in einem Nicht-CO2 37 °C Inkubator für 1 h zu inkubieren; Dieser Schritt kann übersprungen werden, wenn eine automatisierte Bildgebung bevorzugt wird. Zum Beispiel hält der Mikroplatten-Imager die gleichen Bedingungen in einer geschlossenen Kammer aufrecht, und Zellen können unter einem Hellfeld abgebildet werden.
6. Einrichten des Assays und der Bildgebung
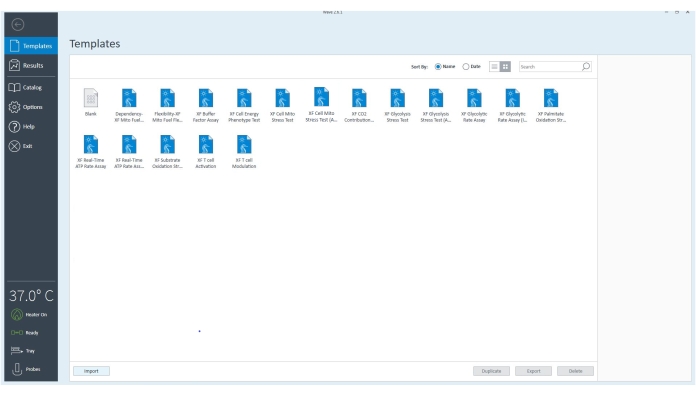
Abbildung 3: Die Controller-Software. Die Software überprüft, ob das Gerät angeschlossen und auf 37 °C eingestellt ist. Die Vorlagendateien für verschiedene Assays, die mit dem extrazellulären Flussanalysator durchgeführt werden können, können ausgewählt werden, um den Assay basierend auf den experimentellen Zielen weiter anzupassen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Öffnen Sie die Desktop-Software im Computer neben dem Gerät.
- Überprüfen Sie den Verbindungsstatus in der unteren linken Ecke der Controller-Software.
- Wechseln Sie zu Vorlagen und wählen Sie die XF ATP Rate Assay-Vorlagendatei oder die entsprechende Assay-Vorlage aus.
- Wählen Sie oben auf dem Bildschirm Gruppendefinitionen und definieren Sie die Gruppen.
- Wählen Sie das Plattenkartenlayout aus und weisen Sie die Bohrungen abhängig von den definierten Gruppen zu.
- Überprüfen Sie das Geräteprotokoll, stellen Sie sicher, dass die hinzugefügten Verbindungen korrekt aufgelistet sind, und fügen Sie die Projektinformationen für zukünftige Referenzen hinzu.
- Klicken Sie auf Test ausführen. Dadurch wird die Auswahl des Speicherorts der Ergebnisdatei angezeigt.
- Wählen Sie den Speicherort für die Ergebnisdatei aus.
- Speichern Sie die Datei mit dem Assay-Datum und klicken Sie auf Start Run.
- Legen Sie die Sensorkassette und die Gebrauchsplatte auf das Fach und klicken Sie auf Ich bin bereit , um die Kalibrierung zu starten.
- Bevor Sie mit der Kalibrierung beginnen, stellen Sie sicher, dass der Patronendeckel entfernt und die Sensorpatrone in der richtigen Ausrichtung auf der Utility-Platte platziert ist. Dieser Schritt dauert 10-20 Minuten, und sobald der Vorgang abgeschlossen ist, zeigt die Software das Dialogfeld Wägezellplatte an.
7. Brightfield-Bilder erhalten
HINWEIS: Dieser Schritt ist optional. Wenn keine bildgebenden Geräte verfügbar sind, fahren Sie mit Schritt 8 fort.

Abbildung 4: Die Zellbildgebungssoftware kommuniziert über den Computer mit dem Bildgebungsleser. Die Zellen in der Mikrotiterplatte können vor und nach dem Assay abgebildet werden, und die Zellzahl / -vertiefung wird nach dem Assay erhalten, um die Daten zu normalisieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Öffnen Sie die Zellbildgebungssoftware auf dem Computer.
- Stellen Sie sicher, dass der Microplate-Imager eingeschaltet ist und die Anschlüsse an den Computer angeschlossen sind.
- Überprüfen Sie die Statusleiste unten links auf dem Bildschirm, um sicherzustellen, dass die Temperatur auf 37 °C eingestellt ist und dass der Verbindungsstatus grün als bereit hervorgehoben werden sollte.
- Scannen Sie den Platten-Barcode, um den Bildgebungsprozess zu starten.
- Geben Sie der Zellenplatte einen Namen ein und klicken Sie auf Speichern (Dies ist der Name, bei dem sowohl helle Feld- als auch fluoreszierende Bilder gespeichert werden). Klicken Sie auf Hellfeld-Scan durchführen.
- Auf dem nächsten Bildschirm, der Platte und dem Scanmenü werden die Optionen für die Bildgebung angezeigt. Wählen Sie vor dem Assay Brightfield Scan starten aus.
- Legen Sie die Zellkultur-Mikroplatte zusammen mit der Plattenabdeckung / dem Deckel auf den Tray-Halter und richten Sie A1 gut mit dem A1 Mark aus. Klicken Sie auf Fach schließen.
- Der nächste Bildschirm, die Hellfeldbildaufnahme, mit einer Plattenkarte wird angezeigt. Klicken Sie auf Alle Vertiefungen scannen, wodurch der Systeminitialisierungsprozess gefolgt von 30 bis 35 Minuten Scan gestartet wird.
- Entfernen Sie nach dem Hellfeld-Scan die Zellkultur-Mikroplatte und legen Sie sie in den Analysator, um den Assay durchzuführen.
8. Ausführen des Assays
- Sobald die Kalibrierung abgeschlossen ist, zeigt die Software das Dialogfeld Wägezellenplatte an.
- Klicken Sie auf Open Tray, um das Utility Tray durch eine Zellkultur-Mikrotiterplatte zu ersetzen. Stellen Sie sicher, dass der Deckel entfernt wird und der A1 der Platte in die richtige Ausrichtung passt.
- Klicken Sie dann auf Wägezellenplatte , um den Assay zu starten. Die Sensorpatrone verbleibt für die Assay-Injektionen im Analysator.
- Warten Sie, bis der Assay beginnt und die geschätzte Zeit des Abschlusses anzeigt.
- Nach Abschluss des Assays zeigt die Software das Dialogfeld " Sensorkassette entladen" an. Klicken Sie auf Auswerfen und entfernen Sie die Zellkultur-Mikrotiterplatte aus dem Analysator.
- Entfernen Sie vorsichtig die Sensorpatrone und setzen Sie den Deckel der Zellenplatte wieder ein. Die Zellen sind bereit für die fluoreszierende Bildgebung und Zellzählung.
- Nach dem Entfernen der Zellenplatte und der Sensorkassette wird das Dialogfeld Assay Complete ( Assay Complete ) angezeigt.
- Klicken Sie auf Ergebnisse anzeigen , um die Assay-Ergebnisdatei zu öffnen und die Daten sofort zu normalisieren, oder klicken Sie auf Home.
9. Erhalten Sie Fluoreszenzbilder und normalisieren Sie sie
HINWEIS: Dieser Schritt ist eine optionale, aber bevorzugte Methode zur Normalisierung von BMSCs und Osteoblasten. Wenn keine bildgebenden Geräte zur Verfügung stehen, muss eine andere Normalisierungsmethode durchgeführt werden, z. B. Protein- oder DNA-Isolierung und -Quantifizierung.

Abbildung 5: Repräsentative Bilder aus der Bildgebungssoftware, die zur Normalisierung der Daten aus dem Assay verwendet werden . (A) Zusammengefügtes Hellfeldbild, das den Zellzusammenfluss im gesamten Bohrloch zeigt. (B) Zusammengefügtes Fluoreszenzbild, das Hoechst-gefärbte Kerne von Osteoblasten zeigt, die zum Zählen der Zellzahlen verwendet werden, um die Assay-Ergebnisse zu normalisieren. Dies sind Osteoblasten nach 7 Tagen Differenzierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Scannen Sie nach Abschluss des Assays den Platten-Barcode mit dem tragbaren Barcode-Lesegerät. Wenn die Platte bereits abgebildet wurde, ist kein neuer Name erforderlich.
- Wählen Sie Fluoreszenz & Zellzahl, legen Sie die Zellplatte auf den Trayhalter und klicken Sie auf Close Tray.
- Wählen Sie im Bildaufnahmefenster die Option Alle Vertiefungen scannen aus, um mit der Bildgebung zu beginnen. Die Fluoreszenzbildgebung benötigt etwa 15-20 Minuten, um die gesamte Platte zu scannen. Beachten Sie das grüne Häkchen, das angibt, dass der Scan abgeschlossen wurde.
- Überprüfen Sie die fluoreszierenden Bilder und Zellzahlen in der Bildgebungs- und Zellbildgebungsanwendung, indem Sie zufällig auf einige der Vertiefungen klicken.
HINWEIS: Es gibt eine Option zum Anzeigen der gezählten Zellen unten rechts auf dem Bildschirm. Diese Option zeigt ein maskiertes Bild an, das die gezählten Objekte hervorhebt. - Sobald die Fluoreszenzbildgebung abgeschlossen ist, exportieren Sie die Bilder für zusätzliche Referenzen.
- Sobald die Bildgebung und Zellzahl abgeschlossen ist, öffnen Sie die Ergebnisdatei und klicken Sie auf Normalisieren. Der Normalisierungsbildschirm gibt das Plattenlayout und eine Option zum Importieren der Zellenanzahl.
- Klicken Sie auf Importieren und wählen Sie Apply für die Desktop-Software, um den Assay mit der Zellzahl automatisch zu normalisieren.
Ergebnisse
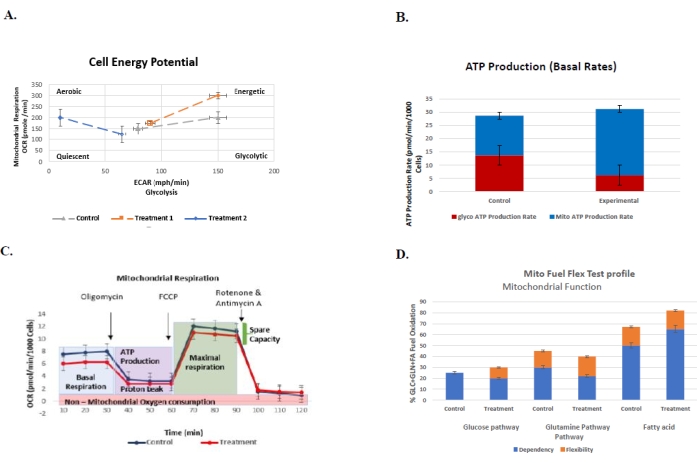
Abbildung 6: Repräsentative Diagramme für routinemäßig durchgeführte Assays, um das zelluläre bioenergetische Profil der Kontroll- vs. Behandlungsgruppe mit ihren jeweiligen Standardfehlern zu verstehen . (A) Der Zellenergie-Phänotyp-Test. Das Diagramm stellt die Glykolyse (ECAR) vs. mitochondriale Atmung (OCR) der Kon...
Diskussion
Der Echtzeit-Zellstoffwechselflussanalysator kann verwendet werden, um die zelluläre Energetik unter verschiedenen Bedingungen zu erforschen. Das Protokoll veranschaulicht die effiziente Isolierung von BMSCs, die Kultivierung von Zellen in geeigneten Zellkulturplatten und ihre Differenzierung zu reifen Osteoblasten, die für verschiedene Assays mit dem extrazellulären Flussanalysator verwendet werden können. Darüber hinaus werden die kritischen Schritte des Echtzeit-Zellstoffwechselflussassays, einschließlich der Hy...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Diese Arbeit wurde vom National Institute of Health (NIH) National Institute of Arthritis and Musculoskeletal and Skin Diseases (NIAMS) Grant AR072123 und dem National Institute on Aging (NIA) Grant AG069795 (zu ERR) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin EDTA | Sigma-Aldrich | T4049 | |
| 2-cyano-3-(1-phenyl-1H-indol-3-yl)-2-propenoic acid | Sigma - Aldrich | PZ0160 | UK5099 |
| Antimycin A | Sigma - Aldrich | A8674 | |
| Ascorbic acid | Sigma-Aldrich | A4544-100G | |
| Bis-2-(5-phenylacetamido-1,3,4-thiadiazol-2-yl)ethyl sulfide | Sigma - Aldrich | SML0601 | BPTES |
| Carbonyl cyanide 4-(trifluoromethoxy)phenylhydrazone | Sigma - Aldrich | C2920 | FCCP |
| Cytation 5 imaging reader | BioTek | N/A | Microplate imager |
| Etomoxir sodium salt hydrate | Sigma - Aldrich | E1905 | |
| Hoechst 33342 Solution (20 mM) | Thermo Scientific | 62249 | |
| Insulin | Sigma - Aldrich | I6634 | |
| Oleic Acid-Albumin from bovine serum | Sigma - Aldrich | O3008 | |
| Oligomycin A - 5 mg | Sigma - Aldrich | 75351 | |
| Rotenone | Sigma - Aldrich | R8875-1G | |
| Seahorse XF 1.0 M Glucose Solution | Agilent Technologies | 103577-100 | |
| Seahorse XF 100mM Pyruvate Solution | Agilent Technologies | 103578-100 | |
| Seahorse XF 200mM Glutamine solution | Agilent Technologies | 103579-100 | |
| Seahorse XF DMEM media | Agilent Technologies | 103575-100 | DMEM assay media eith 5mM HEPES, pH 7.4, without phenol red, sodium bicarbonate, glucose, pyruvate, and L-glutamine |
| Seahorse XFe96 Analyzer | Agilent Technologies | S7800B | Real- Time Metabolic flux analyzer |
| Seahorse XFe96 FluxPak | Agilent Technologies | 102416-100 | Includes XFe96 Sensor cartridges, Cell culture microplates, and Seahorse XF Calibrant solution |
| The Cell imaging 1.1.0.11 software | Agilent Technologies - BioTek | ||
| Wave software 2.6.1 | Agilent Technologies | ||
| β-glycerol phosphate | Sigma-Aldrich | G9422-50G |
Referenzen
- Rodan, G. A. Bone homeostasis. Proceedings of the National Academy of Sciences of the United States of America. 95 (23), 13361-13362 (1998).
- Nakahama, K. I. Cellular communications in bone homeostasis and repair. Cellular and Molecular Life Sciences. 67 (23), 4001-4009 (2010).
- Kim, J. M., Lin, C., Stavre, Z., Greenblatt, M. B., Shim, J. H. Osteoblast-osteoclast communication and bone homeostasis. Cells. 9 (9), 2073 (2020).
- Gao, J., et al. SIRT3/SOD2 maintains osteoblast differentiation and bone formation by regulating mitochondrial stress. Cell Death and Differentiation. 25 (2), 229-240 (2018).
- Baron, R. Molecular mechanisms of bone resorption by the osteoclast. The Anatomical Record. 224 (2), 317-324 (1989).
- Tian, L., Rosen, C. J., Guntur, A. R. Mitochondrial Function and Metabolism of Cultured Skeletal Cells. Methods in Molecular Biology. 2230, 437-447 (2021).
- Zanotelli, M. R., et al. Regulation of ATP utilization during metastatic cell migration by collagen architecture. Molecular Biology of the Cell. 29 (1), 1-9 (2018).
- Gonzales, S., Wang, C., Levene, H., Cheung, H. S., Huang, C. Y. C. ATP promotes extracellular matrix biosynthesis of intervertebral disc cells. Cell and Tissue Research. 359 (2), 635-642 (2015).
- Kruse, N. J., Bornstein, P. The metabolic requirements for transcellular movement and secretion of collagen. Journal of Biological Chemistry. 250 (13), 4841-4847 (1975).
- Rendina-Ruedy, E., Guntur, A. R., Rosen, C. J. Intracellular lipid droplets support osteoblast function. Adipocyte. 6 (3), 250-258 (2017).
- Sinnott-Armstrong, N., et al. A regulatory variant at 3q21.1 confers an increased pleiotropic risk for hyperglycemia and altered bone mineral density. Cell Metabolism. 33 (3), 615-628 (2021).
- Esen, E., Lee, S. Y., Wice, B. M., Long, F. PTH promotes bone anabolism by stimulating aerobic glycolysis via IGF signaling. Journal of Bone and Mineral Research. 30 (11), 1959-1968 (2015).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: I. Normal bone. Journal of Biological Chemistry. 235, 1206-1210 (1960).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: II. The metabolic patterns of accretion and resorption. Journal of Biological Chemistry. 235, 1211-1214 (1960).
- Adamek, G., Felix, R., Guenther, H. L., Fleisch, H. Fatty acid oxidation in bone tissue and bone cells in culture. Characterization and hormonal influences. The Biochemical Journal. 248 (1), 129-137 (1987).
- Frey, J. L., et al. Wnt-Lrp5 signaling regulates fatty acid metabolism in the osteoblast. Molecular and Cellular Biology. 35 (11), 1979-1991 (2015).
- Romero, N., Rogers, G., Neilson, A., Dranka, B. P. . Quantifying cellular ATP production rate using agilent seahorse XF technology. , (2018).
- vander Windt, G., Chang, C., Pearce, E. Measuring bioenergetics in T cells using a Seahorse Extracellular Flux Analyzer. Current Protocols in Immunology. 113, 1 (2016).
- Traba, J., Miozzo, P., Akkaya, B., Pierce, S. K., Akkaya, M. An optimized protocol to analyze glycolysis and mitochondrial respiration in lymphocytes. Journal of Visualized Experiments:JoVE. (117), e54918 (2016).
- Noel, P., et al. Preparation and metabolic assay of 3-dimensional spheroid co-cultures of pancreatic cancer cells and fibroblasts. Journal of Visualized Experiments:JoVE. (126), e56081 (2017).
- Nicholls, D., et al. Bioenergetic profile experiment using C2C12 myoblast cells. Journal of Visualized Experiments: JoVE. (46), e2511 (2010).
- Sakamuri, S. S. V. P., et al. Measurement of respiratory function in isolated cardiac mitochondria using Seahorse XFe24 Analyzer: applications for aging research. GeroScience. 40 (3), 347-356 (2018).
- . What are the advantages of using Seahorse XF technology Available from: https://wwwagilent.com/en/support/cell-analysis/advantages-of-using-xf-tech (2018)
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 67 (10), 1022-1035 (2012).
- . XF cell energy phenotype test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740884 (2021)
- Leung, D. T. H., Chu, S. Measurement of oxidative stress: Mitochondrial function using the seahorse system. Methods in Molecular Biology. 1710, 285-293 (2018).
- . XF ATP rate assay Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740889 (2021)
- . XF cell mito stress test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740885 (2021)
- . XF cell mito fuel flex test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740888 (2021)
- Maridas, D. E., Rendina-Ruedy, E., Le, P. T., Rosen, C. J. Isolation, culture, and differentiation of bone marrow stromal cells and osteoclast progenitors from mice. Journal of Visualized Experiments: JoVE. (131), e56750 (2018).
- Wei, J., et al. Glucose uptake and Runx2 synergize to orchestrate osteoblast differentiation and bone formation. Cell. 161 (7), 1576-1591 (2015).
- Zoch, M. L., Abou, D. S., Clemens, T. L., Thorek, D. L. J., Riddle, R. C. In vivo radiometric analysis of glucose uptake and distribution in mouse bone. Bone Research. 4, 16004 (2016).
- Divakaruni, A. S., Paradyse, A., Ferrick, D. A., Murphy, A. N., Jastroch, M. Analysis and interpretation of microplate-based oxygen consumption and pH data. Methods in Enzymology. 547, 309-354 (2014).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. . Normalization of agilent seahorse XF data by in-situ cell counting using a BioTek cytation 5 application note. , (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten