Method Article
Optimierte Knochenprobenahmeprotokolle für die Gewinnung alter DNA aus archäologischen Überresten
In diesem Artikel
Zusammenfassung
Das Protokoll präsentiert eine Reihe von Best-Practice-Protokollen für die Sammlung von Knochenpulver von acht empfohlenen anatomischen Probenahmestellen (spezifische Stellen an einem bestimmten Skelettelement) über fünf verschiedene Skelettelemente von mittelalterlichen Individuen (Radiokarbon datiert auf einen Zeitraum von ca. 1040-1400 CE, kalibrierter 2-Sigma-Bereich).
Zusammenfassung
Die hier vorgestellten Methoden zielen darauf ab, die Chancen für die Gewinnung menschlicher DNA aus antiken archäologischen Überresten zu maximieren und gleichzeitig das Eingangsprobenmaterial zu begrenzen. Dies wurde erreicht, indem anatomische Probenahmestellen gezielt wurden, die zuvor in einer vergleichenden Analyse der DNA-Erholung im gesamten Skelett die höchsten Mengen an alter DNA (aDNA) ergaben. Frühere Forschungen haben gezeigt, dass diese Protokolle die Chancen für die erfolgreiche Wiederherstellung alter menschlicher und pathogener DNA aus archäologischen Überresten maximieren. Die DNA-Ausbeute wurde zuvor von Parker et al. 2020 in einer breit angelegten Untersuchung der aDNA-Konservierung über mehrere Skelettelemente von 11 Individuen bewertet, die aus dem mittelalterlichen (Radiokarbon datiert auf einen Zeitraum von ca. 1040-1400 CE, kalibrierter 2-Sigma-Bereich) Friedhof am Krakauer Berg, einer verlassenen mittelalterlichen Siedlung in der Nähe von Peißen, Deutschland, geborgen wurden. Diese acht Probenahmestellen, die fünf Skelettelemente (Pars petrosa, permanente Molaren, Brustwirbel, distale Phalanx und Talus) umfassen, lieferten erfolgreich hochwertige alte menschliche DNA, bei der die Ausbeute signifikant über dem Gesamtdurchschnitt aller Elemente und Individuen lag. Die Erträge waren für die Verwendung in den meisten gebräuchlichsten genetischen Analysen der nachgelagerten Population ausreichend. Unsere Ergebnisse unterstützen die bevorzugte Verwendung dieser anatomischen Probenahmestellen für die meisten Studien, die die Analyse alter menschlicher DNA aus archäologischen Überresten beinhalten. Die Implementierung dieser Methoden wird dazu beitragen, die Zerstörung wertvoller archäologischer Proben zu minimieren.
Einleitung
Die Probenahme alter menschlicher Überreste zum Zwecke der DNA-Gewinnung und -Analyse ist von Natur aus zerstörerisch 1,2,3,4. Die Proben selbst sind wertvolle Exemplare und die morphologische Konservierung sollte nach Möglichkeit erhalten bleiben. Daher ist es unerlässlich, dass die Probenahmeverfahren optimiert werden, um sowohl die unnötige Zerstörung von unersetzlichem Material zu vermeiden als auch die Erfolgswahrscheinlichkeit zu maximieren. Aktuelle Best-Practice-Techniken basieren auf einer kleinen Kohorte von Studien, die sich entweder auf forensische Untersuchungen5,6, Studien an antiken Proben, bei denen die Entwicklung einer optimalen Probenahme nicht das direkte Zielder Studie 7 ist, oder spezielle Studien beschränken, die entweder nichtmenschliche Überreste8 verwenden oder auf eine sehr kleine Auswahl anatomischer Probenahmestellen abzielen (hier verwendet, um einen bestimmten Bereich eines Skelettelements zu bezeichnen, aus dem Knochenpulver, zur Verwendung in nachgeschalteten DNA-Analysen, wurde generiert)9,10. Die hier vorgestellten Probenahmeprotokolle wurden in der ersten groß angelegten systematischen Studie zur DNA-Konservierung über mehrere Skelettelemente von mehreren Individuen optimiert11. Alle Proben stammten aus Skelettelementen, die von 11 Individuen geborgen wurden, die auf dem Kirchenfriedhof der verlassenen mittelalterlichen Siedlung Krakauer Berg in der Nähe von Peißen, Sachsen-Anhalt, Deutschland ausgegraben wurden (siehe Tabelle 1 für detaillierte demografische Proben) und müssen daher möglicherweise modifiziert werden, um mit Proben außerhalb dieses geografischen/zeitlichen Bereichs verwendet zu werden.
| Individuum | Sex | Geschätztes Sterbealter | 14 C-Daten (CE, Cal 2-sigma) |
| KRA001 | Männlich | 25-35 | 1058-1219 |
| KRA002 | Weiblich | 20-22 | 1227-1283 |
| KRA003 | Männlich | 25 | 1059-1223 |
| KRA004 | Männlich | 15 | 1284-1392 |
| KRA005 | Männlich | 10-12 | 1170-1258 |
| KRA006 | Weiblich | 30-40 | 1218-1266 |
| KRA007 | Weiblich | 25-30 | 1167-1251 |
| KRA008 | Männlich | 20 | 1301-1402 |
| KRA009 | Männlich | Unbekannt | 1158-1254 |
| KRA010 | Männlich | 25 | 1276-1383 |
| KRA011 | Weiblich | 30-45 | 1040-1159 |
Tabelle 1: Genetisch bedingtes Geschlecht, archäologisch bedingtes geschätztes Sterbealter und Radiokarbondatierung (14C Cal 2-sigma) für alle 11 untersuchten Individuen. Diese Tabelle wurde angepasst von Parker, C. et al. 202011.
Diese Protokolle ermöglichen eine relativ einfache und effiziente Generierung von Knochenpulver aus acht anatomischen Probenahmestellen über fünf Skelettelemente (einschließlich der Pars petrosa) mit begrenzter laborinduzierter DNA-Kontamination. Von diesen fünf Skelettelementen wurden sieben anatomische Probenahmestellen an vier Skelettelementen als praktikable Alternativen zur zerstörerischen Probenahme der Petrouspyramide11,12 bestimmt. Dazu gehören die Zement-, Dentin- und Pulpakammer der permanenten Molaren; kortikaler Knochen aus der oberen Wirbelkerbe sowie aus dem Körper der Brustwirbel; kortikaler Knochen, der von der unteren Oberfläche des apikalen Büschels und des Schaftes der distalen Phalangen stammt; und der dichte kortikale Knochen entlang des äußeren Teils des Tali. Während es mehrere weit verbreitete Methoden für die Probenahme der Pars Petrosa 4,12,13,14, des Dentins und der Zahnpulpakammer 1,2,15 gibt, veröffentlichte Methoden, die die erfolgreiche Erzeugung von Knochenpulver aus dem Zement beschreiben 16 , Wirbelkörper, untere Wirbelkerbe und Talus können schwer zu erhalten sein. Daher demonstrieren wir hier optimierte Probenahmeprotokolle für die Petrouspyramide (Schritt 3.1); Zement (Schritt 3.2.1), Dentin (Schritt 3.2.2) und Zahnpulpa (Schritt 3.2.3) von erwachsenen Molaren; kortikaler Knochen des Wirbelkörpers (Schritt 3.3.1) und des oberen Wirbelbogens (Schritt 3.3.2); die distale Phalanx (Schritt 3.4); und den Talus (Schritt 3.5), um die effektive Nutzung dieser Skelettelemente sowohl für die aDNA- als auch für die forensische Forschung breiter zugänglich zu machen.
Protokoll
Alle hier vorgestellten Forschungen wurden in Übereinstimmung mit den Richtlinien des Max-Planck-Instituts für Menschheitsgeschichte in Jena für die Arbeit mit antiken menschlichen Überresten durchgeführt. Bevor Sie irgendwelche Schritte dieses Protokolls durchführen, stellen Sie sicher, dass Sie alle lokalen/staatlichen/bundesstaatlichen ethischen Anforderungen einhalten, die sich sowohl auf die Einholung der Genehmigung für die wissenschaftliche Untersuchung als auch auf die Verwendung menschlicher Überreste für destruktive Probenahmen in Ihrer Region beziehen. Alle Verfahren/Chemikalienlagerung sollten gemäß den individuellen institutionellen Sicherheitsrichtlinien durchgeführt werden.
1. Überlegungen vor der Probenbearbeitung
- Behandeln Sie Proben mit Vorsicht, da antike Überreste eine unwiederholbare und endliche Ressource sind (z. B. sollte die Probenahme so wenig verschwenderisch wie möglich sein und alle Überreste nach Möglichkeit an ihre jeweiligen und rechtmäßigen Lieferanten zurückgegeben werden).
- Führen Sie alle Schritte in einer Reinraumumgebung durch, vorzugsweise in einer speziellen alten DNA-Einrichtung17,18,19. Verwenden Sie persönliche Schutzausrüstung (PSA), bestehend aus sterilen mikroporösen Overalls mit Kapuze, sterilen Handschuhen (zwei Paar), chirurgischer Maske, Schutzbrille und sterilen Stiefeln oder rutschfesten Schuhen mit sterilen Bezügen (siehe Materialtabelle). Wechseln Sie häufig die Handschuhe, insbesondere zwischen den Proben.
- Reinigen und desinfizieren Sie alle Geräte und Oberflächen gründlich mit Bleichmittel/DNA-Dekontaminationslösung/Ethanol und UV-Bestrahlung (Wellenlänge: 254 nm) (z. B. Bohrer, Bohrer, Schraubstöcke/Klemmen usw.). Schließlich wird dringend empfohlen, regelmäßige ergonomische Pausen einzulegen (wenn möglich alle 2-3 Stunden), um eine Übererschöpfung aufgrund der Reinraumumgebung zu vermeiden.
HINWEIS: Alle Skelettreste sollten vor der Probenahme angemessen dokumentiert werden (z. B. fotografiert, gewogen und, wenn möglich Mikro-CT gescannt, 3D-abgebildet usw.) (Protokolle für eine geeignete Dokumentation werden in diesem Manuskript nicht behandelt). Alle Probenahmeprotokolle können zwischen den Probenahmeiterationen pausiert werden und die Proben können unbegrenzt in einer trockenen, temperaturkontrollierten (25 °C) sterilen Umgebung gelagert werden.
2. Vorbehandlung
- Dekontamination aller anatomischen Probenahmestellen vor der Knochenpulverbildung, um das Risiko einer Kontamination zu minimieren18.
HINWEIS: Die Wirksamkeit von Bleichmittel und/oder Oberflächenentfernung (siehe HINWEIS in Schritt 3.3.2 für Schritte zur Oberflächenentfernung) für die Probendekontaminierung ist unter aDNA-Forschern immer noch umstritten 8,19,20,21,22,23,24,25, da beide die Gesamt-DNA-Ausbeute beeinflussen können, insbesondere in stark abgebauten Proben. Daher werden die folgenden Schritte als optional betrachtet und sind hier enthalten, da sie in allen Stichproben verwendet wurden, um die in diesem Artikel vorgestellten repräsentativen Ergebnisse zu generieren. Es wird empfohlen, die Verwendung dieser Vorbehandlungsprotokolle von Fall zu Fall auf der Grundlage der molekularen Anwendung, des Alters, der Seltenheit und des Grads des morphologischen Abbaus jedes Probensatzes zu bestimmen.- Führen Sie alle Probenahmen in einem speziellen Reinraum unter einer mit UV-Licht ausgestatteten Polymerase-Kettenreaktions-Haube (PCR) oder einer Biosicherheitswerkbank mit ausgeschaltetem Luftstrom durch. Verteilen Sie sterile Aluminiumfolie auf der Tischplatte, um verirrtes Knochenpulver / -fragmente aufzufangen.
- Stellen Sie sicher, dass alle Knochenfragmente (für die Rückführung) geborgen sind, bevor Sie die Folie entsorgen. Wechseln Sie die Folie zwischen der Behandlung jedes Skelettelements. Entsorgen Sie gebrauchte Folien in einem autoklavierbaren Biogefahrenbeutel/-behälter.
- Entfernen Sie so viel losen Schmutz / Ablagerungen wie möglich von den anatomischen Probenahmestellen, indem Sie den Bereich vorsichtig mit einem fusselfreien trockenen sterilen Tuch abwischen (siehe Materialtabelle). Entsorgen Sie die Tücher in autoklavierbaren Biogefahrenbeuteln oder -behältern.
- Dekontamination der gereinigten Oberfläche durch Abwischen mit einem sterilen Wischtuch, das mit verdünntem handelsüblichem Bleichmittel (~0,01% v/v, verdünnt mit hochreinem DNase/RNase-freiem Wasser) angefeuchtet ist, und 5 min inkubieren lassen. Entsorgen Sie die Tücher in autoklavierbaren Biogefahrenbeuteln oder -behältern.
VORSICHT: Bleichmittel ist eine stark korrosive und reaktive Chemikalie; Daher sollten vor seiner Verwendung geeignete Sicherheitsvorkehrungen getroffen werden. - Entfernen Sie so viel Restbleichmittel wie möglich von der anatomischen Probenahmestelle mit einem sterilen, mit hochreinem DNase/RNase-freiem Wasser angefeuchteten Tuch. Entsorgen Sie die Tücher in autoklavierbaren Biogefahrenbeuteln oder -behältern.
- Alle gereinigten anatomischen Probenahmestellen 30 min (Wellenlänge: 254 nm) UV-Strahlung aussetzen und anschließend bei Raumtemperatur vollständig trocknen lassen. Stellen Sie sicher, dass die anatomischen Probenahmestellen vollständig trocken sind, bevor Sie mit der Probenahme fortfahren oder zur Lagerung zurückkehren, um nicht nur die Bildung von Knochenpulver zu erleichtern, sondern auch einen weiteren Abbau der Probe (z. B. Schimmel) zu verhindern.
ACHTUNG: Die Exposition gegenüber UV-Strahlung kann schädlich für die Augen sein. - Gehen Sie sofort zur Probenahme oder lagern Sie Skelettelemente in einer trockenen, temperaturkontrollierten (25 °C) sterilen Umgebung.
3. Knochenpulverbildung
HINWEIS: Die folgenden Protokolle sind für die DNA-Extraktion gemäß dem Protokoll26 von Dabney et al. 2019 vorgesehen.
- Probenahme von pars petrosa
HINWEIS: Dieses Protokoll basiert auf den in Pinhasi et al. 2019 beschriebenen Verfahren.4 und wird hier für die Benutzerfreundlichkeit vorgestellt. Dieses Protokoll stellt nicht die aktuelle, am wenigsten zerstörende Methode für die Probenahme von pars petrosa. Daher wird empfohlen, das von Sirak et al. 2017 beschriebene Protokoll zu verwenden.13 oder Orfanou et al. 202014 für Proben, bei denen die morphologische Konservierung von größter Bedeutung ist.- Führen Sie alle Probenahmen in einem speziellen Reinraum unter einer mit UV-Licht ausgestatteten PCR-Haube oder einer Biosicherheitswerkbank (Wellenlänge: 254 nm) mit ausgeschaltetem Luftstrom durch. Verteilen Sie sterile Aluminiumfolie auf der Tischplatte, um verirrtes Knochenpulver / -fragmente aufzufangen.
- Stellen Sie sicher, dass alle Knochenfragmente und so viel Pulver wie möglich (für die Rückführung) zurückgewonnen werden, bevor Sie die Folie entsorgen. Wechseln Sie die Folie zwischen den einzelnen Proben. Entsorgen Sie die gebrauchte Folie in einem autoklavierbaren Biogefahrenbeutel/-behälter.
- Sichern Sie das trockene, dekontaminierte Element mit einer sterilisierten Klemme oder einem Schraubstock.
- Schneiden Sie die Pars petrosa entlang des oberen Sulcus petrosus (siehe Abbildung 1) mit einer Standard-Juweliersäge mit einer 0,6-mm-Klinge (siehe Materialtabelle) bei mittlerer Geschwindigkeit in zwei Hälften, um eine Überhitzung zu vermeiden (siehe HINWEIS unten Schritt 3.1.6).
ACHTUNG: Die Pars petrosa ist sehr dicht und kann daher schwer zu schneiden sein. Achten Sie darauf, das Element sicher zu halten, um Verletzungen zu vermeiden. Entsorgen Sie zerbrochene Sägeblätter in der entsprechenden Behälter für scharfe und spitze Gegenstände. - Entfernen Sie die petrosen Teile von der Klemme. Holen Sie sich verlorenes/überschüssiges Material zurück und bewahren Sie es.
- Legen Sie das Wägepapier in ein steriles Wiegeboot
- Halten Sie den petroösen Teil über das Wägepapier, die Seite zur Waagschale geneigt. Bohren Sie in den dichten kortikalen Knochen zwischen dem Gesichtskanal und dem Warzenantrum (erscheint glänzender als das umgebende Material, siehe Abbildung 1) mit einem Zahnbohrer, der mit einem kleinen Bohrer ausgestattet ist (siehe Materialtabelle) und auf mittlere Geschwindigkeit, mittleres Drehmoment eingestellt ist, um Knochenpulver herzustellen.
HINWEIS: Das Bohren / Schneiden sollte in kurzen Ausbrüchen mit niedrigen bis mittleren Geschwindigkeiten durchgeführt werden, um eine Überhitzung des Knochens und eine mögliche Zerstörung / Beschädigung der DNA zu vermeiden. Anekdotisch, wenn der dichte Teil des Petrous zu überhitzen beginnt, kann ein Geruch beobachtet werden, der als Kochspeck beschrieben wird. Beenden Sie das Bohren / Sägen sofort und lassen Sie den Knochen ruhen, bis er ausreichend abgekühlt ist, bevor Sie ihn fortsetzen. - Wiederholen Sie das Bohren, bis etwa 50-100 mg Pulver im Wägepapier gesammelt sind, gemessen mit einer beiliegenden Waage, die auf mindestens 0,01 mg genau ist (siehe Materialtabelle).
HINWEIS: Wenn möglich, wird empfohlen, 100 mg Knochenpulver zu sammeln, um zwei replizierte DNA-Extraktionen von jeweils 50 mg zu ermöglichen. Dies ist jedoch möglicherweise nicht immer möglich, entweder aufgrund der Begrenzung der anatomischen Probenahmeorte selbst (z. B. der distalen Phalanx, der dentalen Pulpakammer) oder der Notwendigkeit einer morphologischen Konservierung. Für andere Stellen, wie z.B. den Zement, können deutlich weniger als 50 mg des Materials zur Verfügung stehen. Es wurde jedoch gezeigt, dass der Zement, die Zahnpulpakammer und die distale Phalanx trotz geringerer anfänglicher Eingabe von Knochenpulver aus dem Extraktionsprozess signifikante endogene DNA 11,27,28 ergeben. - Pulver aus dem Wägepapier zur Extraktion oder Lagerung in ein 2 ml beschriftetes Low-Binde-Safe-Lock-Röhrchen überführen. Proben auf unbestimmte Zeit bei -20 °C lagern.
- Lagern Sie verbleibende Knochen/überschüssiges Pulver in einer trockenen, temperaturkontrollierten (25 °C) sterilen Umgebung, bis die Rückgabe/Rückführung abgeschlossen werden kann.
- Entsorgen Sie alle Abfälle in autoklavierbaren Biogefahrenbeuteln oder -behältern. Sterilisieren/dekontaminieren Sie alle wiederverwendbaren Geräte (z. B. Klemmen, Bohrer, Bohrer, Sägen usw.) unter Verwendung von Bleichmittel-/DNA-Dekontaminationslösung/Ethanol und UV-Exposition (Wellenlänge: 254 nm) zwischen den einzelnen Proben.
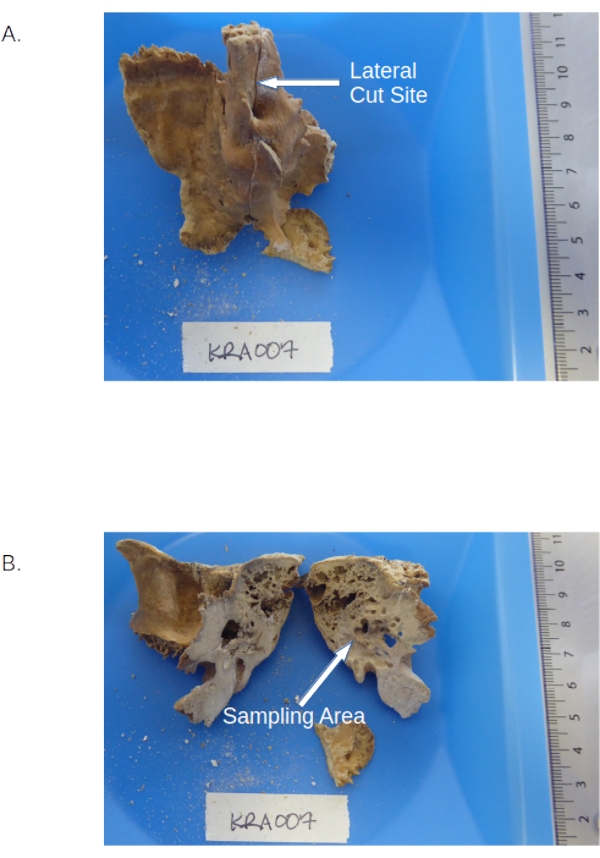
Abbildung 1: Schläfenbein einschließlich Pars petrosa. (A) Mustervorschnitt, aus dem die Standorte der Petrouspyramide und des Sulcus petrosa hervorgehen. (B) Petroiger Teil nach dem Schneiden mit Hervorhebung der dichten zu bohrenden Bereiche. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Probenahme von bleibenden Molaren
HINWEIS: Für die Probenahme von permanenten Backenzähnen wählen Sie voraus in situ Backenzähne mit verschmolzenen Wurzeln und idealerweise frei von Karies, Rissen im Zahnschmelz oder übermäßigem Verschleiß für beste Ergebnisse. Entfernen Sie alle Zahnsteinproben und lagern Sie sie bei -20 °C für mögliche zukünftige Analysen des oralen Mikrobioms (Verfahren, das hier nicht behandelt wird).- Probenahme des Zements
- Führen Sie alle Probenahmen in einem speziellen Reinraum unter einer mit UV-Licht ausgestatteten PCR-Haube oder einer Biosicherheitswerkbank (Wellenlänge: 254 nm) mit ausgeschaltetem Luftstrom durch. Verteilen Sie sterile Aluminiumfolie auf der Tischplatte, um verirrtes Knochenpulver / -fragmente aufzufangen.
- Stellen Sie sicher, dass alle Knochenfragmente und so viel Pulver wie möglich (für die Rückführung) wiederhergestellt werden, bevor Sie die Folie entsorgen. Wechseln Sie die Folie zwischen den einzelnen Proben. Entsorgen Sie gebrauchte Folien in einem autoklavierbaren Biogefahrenbeutel/-behälter.
- Legen Sie ein Blatt Wiegepapier in eine sterile Waagale.
- Halten / sichern Sie den dekontaminierten Backenzahn am Zahnschmelz, wurzeln, über einer Wiegeschale mit einer Handklemme, z. B. einem verstellbaren Schraubenschlüssel (siehe Materialtabelle).
- Statten Sie einen Dentalbohrer mit einem diamantumrandeten Kreisschneidrad aus. Wenn der Bohrer auf mittlere Drehzahl/Drehmomenteinstellung eingestellt ist, berühren Sie leicht die Kante des Meißels zur Wurzel in einem Winkel von ca. -20°.
- Kratzen Sie nach unten in die Schale, um das gelbe, äußerste Material von der Wurzel (Zement) zu entfernen/zu sammeln. Stoppen Sie die Ansammlung, wenn das hellere (weiße) Material des Dentins sichtbar wird.
HINWEIS: Es ist wichtig, die Drehrichtung des Schneidmeißels in Bezug auf die Auffangschale abzustimmen, um zu vermeiden, dass das Pulver aerosolisiert wird und die Probe möglicherweise verschwendet wird, indem die Schale vollständig fehlt. Der Zement ist besonders reich an DNA; Typische Materialausbeuten sind jedoch viel kleiner als an anderen anatomischen Probenahmestellen (~7-20 mg)11,27,28. - Erfassen Sie die Masse des in Wiegepapier gesammelten Pulvers mit einer beiliegenden Waage, die auf mindestens 0,01 mg genau ist (siehe Materialtabelle).
- Pulver aus dem Wägepapier zur Extraktion in ein 2-ml-Schlauch mit niedrigem Bindemittel, sicherem Schloss überführen. Bei -20 °C auf unbestimmte Zeit lagern.
- Probenahme der Zellstoffkammer
- Nachdem der Zement gesammelt wurde (falls gewünscht), schneiden Sie den Backenzahn entlang des Zement-Schmelz-Übergangs mit einer Juweliersäge, um die Krone zu entfernen (siehe Abbildung 2).
- Legen Sie ein neues Blatt Wägepapier in ein neues Wiegefach.
- Befestigen Sie den Kronenabschnitt in einer Handklemme oder einem Schraubstock über der Waagale. Halten Sie die geschnittene Seite nach unten geneigt und bohren/kratzen Sie das Material als ersten Durchgang mit einem Zahnbohrer, der mit einem kleinen Bohrer (siehe Materialtabelle) entlang der Kanten der Pulpakammer innerhalb des Kronenteils ausgestattet ist (siehe Abbildung 2).
HINWEIS: Nur der erste Durchgang des Inneren der Pulpakammer muss gesammelt und als Pulpamaterial (5-15 mg typische Ausbeute) gekennzeichnet werden, alles, was tiefer in den Zahn eindringt, gilt als Dentin. - Drehen Sie den Zahn mit dem unteren Teil nach unten, klopfen Sie mit einem Hammer auf die Klemme und sammeln Sie das freigesetzte Pulver auf dem Wiegepapier.
- Erfassen Sie das Gewicht des im Wiegepapier gesammelten Pulvers mit einer beiliegenden Waage, die auf mindestens 0,01 mg genau ist (siehe Materialtabelle).
- Füllen Sie das Pulver vom Wägepapier zur Extraktion in ein 2-ml-Schlauch mit niedriger Binde, Sicherheitsverschluss. Bei -20 °C auf unbestimmte Zeit lagern.
- Probenahme des Dentins
- Legen Sie ein neues Blatt Wägepapier in ein neues Wiegefach.
- Halten Sie den Kronenabschnitt über die Waagschale (gemäß Schritt 3.2.2.3), bohren Sie weitere 50-100 mg Dentin, gemessen mit einer geschlossenen Waage mit einer Genauigkeit von 0,01 mg (siehe Materialtabelle), aus dem Inneren der Pulpakammer auf die gleiche Weise für weitere Dentinproben (siehe Abbildung 2).
- Übertragen Sie Knochenpulver aus dem Wägepapier zur Extraktion in ein 2 ml Low-Bind-Safe-Lock-Röhrchen. Bei -20 °C auf unbestimmte Zeit lagern.
- Lagern Sie die restlichen Zahnstücke/überschüssiges Pulver in einer trockenen, temperaturkontrollierten (25 °C) sterilen Umgebung, bis die Rückgabe/Rückführung abgeschlossen werden kann.
- Entsorgen Sie alle Abfälle in autoklavierbaren Biogefahrenbeuteln oder -behältern. Sterilisieren/dekontaminieren Sie alle wiederverwendbaren Geräte (z. B. Klemmen, Bohrer, Bohrer, Sägen usw.) unter Verwendung von Bleichmittel-/DNA-Dekontaminationslösung/Ethanol und UV-Belichtungen (Wellenlänge: 254 nm) zwischen jeder Probenahme.
- Probenahme des Zements
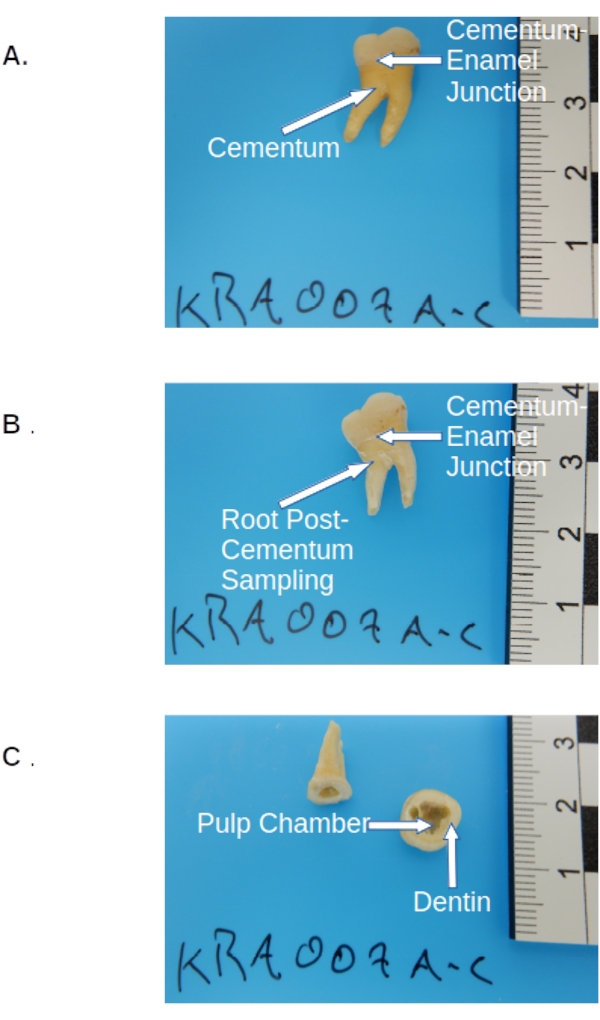
Abbildung 2: Permanente molare Vorprobenahme . (A) Vorbehandelter Molar vor der Probenahme mit Krone, Zement (gelbliche Schicht der Wurzel) und der Schnittstelle am Zementschmelzübergang. (B) Die gleiche molare Postzementsammlung, die die Schnittstelle am Zement-Schmelz-Übergang zeigt. (C) Molare Nachschneiden und Probenahme mit anatomischen Probenahmestellen für die Zahnpulpakammer und das Dentin innerhalb der Krone. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Probenahme der Brustwirbel
- Probenahme des Wirbelkörpers
- Führen Sie alle Probenahmen in einem speziellen Reinraum unter einer mit UV-Licht ausgestatteten PCR-Haube oder einer Biosicherheitswerkbank (Wellenlänge: 254 nm) mit ausgeschaltetem Luftstrom durch. Verteilen Sie sterile Aluminiumfolie auf der Tischplatte, um verirrtes Knochenpulver / -fragmente aufzufangen.
- Stellen Sie sicher, dass alle Knochenfragmente und so viel Pulver wie möglich (für die Rückführung) wiederhergestellt werden, bevor Sie die Folie entsorgen. Wechseln Sie die Folie zwischen den einzelnen Proben. Entsorgen Sie gebrauchte Folien in einem autoklavierbaren Biogefahrenbeutel/-behälter.
- Legen Sie ein kleines Blatt Wägepapier in eine Standardwägeablage.
- Sichern Sie die Wirbel mit einer Klemme oder einem Handschraubstock, wobei der Wirbelkörper nach außen gerichtet ist.
- Halten Sie die Wirbel mit nach unten geneigtem Wirbelkörper über die Waagale. Bohren Sie mit einem Dentalbohrer, der mit einem kleinen Bohrer (siehe Materialtabelle) ausgestattet ist, der auf niedriges hohes Drehmoment eingestellt ist, entlang des äußersten Randes (unterer und oberer Rand) des kortikalen Knochens, der das spongiöse innere Gewebe des Wirbelkörpers umgibt (siehe Abbildung 3).
- Schaben Sie den Meißel gegen die kortikale Schicht über eine Standard-Wiegeschale, bis 50-100 mg Material gesammelt sind, gemessen mit einer beiliegenden Waage, die auf 0,01 mg genau ist (siehe Materialtabelle).
- Übertragen Sie Knochenpulver aus dem Wägepapier zur Extraktion in ein 2-ml-Schlauch mit niedrigem Bindemittel, sicherem Schloss. Bei -20 °C auf unbestimmte Zeit lagern.
- Probenahme des oberen Wirbelbogens
HINWEIS: Dieser Schritt ist optional. Die äußerste Schicht des kortikalen Knochens des oberen Wirbelbogens wird mit einem Zahnbohrer, der mit einem kleinen Bohrer ausgestattet ist (siehe Materialtabelle), entfernt und entsorgt, indem man ihn entlang der Oberflächekratzt 19. Dies wird nicht für die Probenahme aus dem Wirbelkörper empfohlen, da die kortikale Knochenschicht im Allgemeinen sehr dünn ist und durch diesen Prozess wahrscheinlich vollständig erschöpft ist (siehe HINWEIS in Abschnitt 2).- Legen Sie ein kleines Blatt Wägepapier in eine Standardwägeablage.
- Befestigen Sie die Wirbel in einer Handklemme/einem Schraubstock mit dem Wirbelfortsatz nach außen, oberer Aspekt nach unten.
- Während Sie die Wirbel, oberen Aspekt nach unten, über einer Wiegeschale halten, bohren Sie nach oben in die Mitte der V-förmigen Kerbe, die durch die Verschmelzung des Dornfortsatzes mit den Lamellen (siehe Abbildung 3) gebildet wird, wobei ein Zahnbohrer mit einem kleinen Messmeißel (siehe Materialtabelle) verwendet wird, der auf niedrige Drehzahl und hohes Drehmoment eingestellt ist.
- Beenden Sie das Bohren, wenn der Widerstand merklich abfällt. Ändern Sie die Bohrposition leicht und wiederholen Sie den Vorgang, bis 50-100 mg Knochenpulver gesammelt sind, gemessen mit einer beiliegenden Waage, die auf 0,01 mg genau ist (siehe Materialtabelle).
- Übertragen Sie Knochenpulver aus dem Wiegepapier zur Extraktion in ein 2-ml-Low-Bind-Röhrchen. Bei -20 °C auf unbestimmte Zeit lagern.
- Lagern Sie verbleibende Knochen/überschüssiges Pulver in einer trockenen, temperaturkontrollierten (25 °C) sterilen Umgebung bis zur Rückgabe/Rückführung.
- Entsorgen Sie alle Abfälle in autoklavierbaren Biogefahrenbeuteln oder -behältern. Sterilisieren/dekontaminieren Sie alle wiederverwendbaren Geräte (z. B. Klemmen, Bohrer, Bohrer, Sägen usw.) unter Verwendung von Bleichmittel-/DNA-Dekontaminationslösung/Ethanol und UV-Exposition (Wellenlänge: 254 nm) zwischen den einzelnen Proben.
- Probenahme des Wirbelkörpers

Abbildung 3: Anatomische Probenahmestellen des Brustwirbels und des kortikalen Knochens am oberen Wirbelbogen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Probenahme der distalen Phalanx
HINWEIS: Dieser Schritt ist optional. Entfernen und entsorgen Sie die äußerste Schicht des kortikalen Knochens des Schaftes und/oder des apikalen Büschels mit einem Zahnbohrer, der mit einem kleinen Bohrer ausgestattet ist, indem Sie ihn entlang der Oberflächeabkratzen 19. Dies ist bei Proben mit übermäßig dünnen kortikalen Knochen- oder juvenilen Überresten möglicherweise nicht möglich (siehe HINWEIS in Abschnitt 2).- Führen Sie alle Probenahmen in einem speziellen Reinraum unter einer mit UV-Licht ausgestatteten PCR-Haube oder einer Biosicherheitswerkbank (UV-Wellenlänge: 254 nm) mit ausgeschaltetem Luftstrom durch. Verteilen Sie sterile Aluminiumfolie auf der Tischplatte, um verirrtes Knochenpulver / -fragmente aufzufangen.
- Stellen Sie sicher, dass alle Knochenfragmente und so viel Pulver wie möglich (für die Rückführung) wiederhergestellt werden, bevor Sie die Folie entsorgen. Wechseln Sie die Folie zwischen den einzelnen Proben. Entsorgen Sie gebrauchte Folien in einem autoklavierbaren Biogefahrenbeutel/-behälter.
- Legen Sie ein kleines Blatt Wägepapier in eine Standardwägeablage.
- Befestigen Sie die Probe in einer Handklemme/einem Schraubstock, obere Seite nach oben.
- Halten Sie die Probe über die Waagale, sammeln Sie Knochenpulver aus dem kortikalen Knochen von der unteren Seite des apikalen Büschels und des Schaftes, indem Sie durch die äußersten dichten Schichten (siehe Abbildung 4) mit einem Zahnbohrer bohren, der mit einem kleinen Bohrer ausgestattet ist (siehe Materialtabelle).
- Beenden Sie das Bohren, wenn der Widerstand deutlich abnimmt, da dies leichteres, spongiöses Material bedeutet. Wiederholen Sie diesen Vorgang, indem Sie von der ersten Bohrung nach außen strahlen, bis mindestens 50-100 mg Knochenpulver gesammelt sind, gemessen mit einer geschlossenen Waage, die auf 0,01 mg genau ist (siehe Materialtabelle).
- Übertragen Sie Knochenpulver aus dem Wägepapier zur Extraktion in ein 2 ml Low-Bind-Safe-Lock-Röhrchen. Bei -20 °C auf unbestimmte Zeit lagern.
- Lagern Sie den verbleibenden Knochen/überschüssigen Pulver in einer trockenen, temperaturkontrollierten (25 °C) sterilen Umgebung bis zur Rückgabe/Rückführung.
- Entsorgen Sie alle Abfälle in autoklavierbaren Biogefahrenbeuteln oder -behältern. Sterilisieren/dekontaminieren Sie alle wiederverwendbaren Geräte (z. B. Klemmen, Bohrer, Bohrer, Sägen usw.) unter Verwendung von Bleichmittel / DNA-Dekontaminationslösung / Ethanol und UV-Exposition, soweit zutreffend, zwischen jeder Probenahme.
HINWEIS: Für kleinere Proben (z. B. juvenile Proben) können deutlich weniger als die empfohlenen 50-100 mg kortikaler Knochen zur Probe zur Verfügung stehen. Doch selbst in geringen Mengen hat sich gezeigt, dass dieser anatomische Probenahmeort besonders reich an DNA11 ist.

Abbildung 4: Distale Phalanx, die die Positionen des dichten kortikalen Knochens entlang des Schaftes und der unteren Seite des apikalen Büschels zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Probenahme des Talus
- Führen Sie alle Probenahmen in einem speziellen Reinraum unter einer mit UV-Licht ausgestatteten PCR-Haube oder einer Biosicherheitswerkbank (Wellenlänge: 254 nm) mit ausgeschaltetem Luftstrom durch. Verteilen Sie sterile Aluminiumfolie auf der Tischplatte, um verirrtes Knochenpulver / -fragmente aufzufangen.
- Stellen Sie sicher, dass alle Knochenfragmente und so viel Pulver wie möglich (für die Rückführung) wiederhergestellt werden, bevor Sie die Folie entsorgen. Wechseln Sie die Folie zwischen den einzelnen Proben. Entsorgen Sie gebrauchte Folien in einem autoklavierbaren Biogefahrenbeutel/-behälter.
- Legen Sie ein kleines Blatt Wägepapier in eine Standardwägeablage.
- Befestigen Sie die Probe in einer Handklemme/einem Schraubstock, Kuppel nach oben.
- Halten Sie Talus, Kuppel nach oben und mediale Oberfläche in Richtung des Kollektors über die Waagale. Schaben Sie kortikalen Knochen vom Hals des Talus bis zu einer Tiefe von ~1 mm ab (siehe Abbildung 5) mit einem Zahnbohrer mit einem niedrigen Meißel (siehe Materialtabelle), der auf niedrige Drehzahl und hohes Drehmoment eingestellt ist.
- Ändern Sie die Bohrposition leicht und wiederholen Sie den Vorgang, bis etwa 50-100 mg Knochenpulver gesammelt sind, gemessen mit einer beiliegenden Waage, die auf 0,01 mg genau ist (siehe Materialtabelle).
- Übertragen Sie Knochenpulver aus dem Wiegepapier zur Extraktion in ein 2-ml-Low-Bind-Röhrchen. Bei -20 °C auf unbestimmte Zeit lagern.
- Lagern Sie den verbleibenden Knochen/überschüssigen Pulver in einer trockenen, temperaturkontrollierten (25 °C) sterilen Umgebung, bis die Rückgabe/Rückführung abgeschlossen werden kann.
- Entsorgen Sie alle Abfälle in autoklavierbaren Biogefahrenbeuteln oder -behältern. Sterilisieren/dekontaminieren Sie alle wiederverwendbaren Geräte (z. B. Klemmen, Bohrer, Bohrer, Sägen usw.) unter Verwendung von Bleichmittel-/DNA-Dekontaminationslösung/Ethanol und UV-Exposition (Wellenlänge: 254 nm) zwischen den einzelnen Proben.
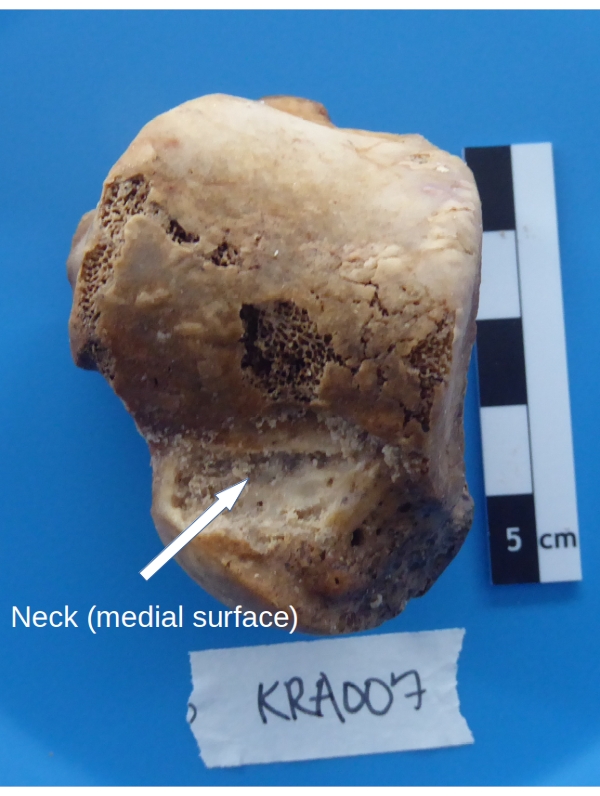
Abbildung 5: Probenahmebereich des Talus für die kortikale Knochenerholung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
HINWEIS: Der Talus hat sehr wenig kortikalen Knochen (eine dünne äußere Schicht). Das Material sollte nicht nur von der Oberfläche gesammelt werden, sondern auch von der darunter liegenden dichten Schicht aus spongiösem Knochen.
Ergebnisse
In einer separaten Studie 11 wurde DNA aus Knochenpulver extrahiert, das von jedem anatomischen Probenahmeort bei11 Personen erzeugt wurde, wobei ein Standard-DNA-Extraktionsprotokoll verwendet wurde, das für kurze Fragmente aus verkalktem Gewebe optimiert war2. Einzelsträngige Bibliotheken wurden dann28 produziert und auf einem HiSeq 4000 (75 bp paired-end) bis zu einer Tiefe von ~20.000.000 Lesevorgängen pro Sample sequenziert. Die resultierenden Sequenzdaten wurden dann mit Hilfe der EAGER-Pipeline29 auf endogenen menschlichen DNA-Gehalt ausgewertet (BWA-Einstellungen: Seedlänge von 32, 0,1 Mismatch-Strafe, Mapping-Qualitätsfilter von 37). Alle repräsentativen Ergebnisse werden aus Gründen der Konsistenz unter Verwendung derselben Metriken wie Parker et al. 202011 berichtet. Bibliotheken aus den pulverisierten Teilen der Pars petrosa ergaben im Durchschnitt eine höhere endogene DNA als alle anderen 23 untersuchten anatomischen Probenahmestellen (Abbildung 6A-B). Die sieben zusätzlichen anatomischen Probenahmestellen, die in diesem Protokoll vorgestellt werden (Zement, erster Durchgang der Zahnpulpakammer und Dentin der permanenten Molaren; kortikaler Knochen aus dem Wirbelkörper und oberer Wirbelbogen des Brustwirbels; kortikaler Knochen aus dem apikalen Büschel der distalen Phalanx; und kortikaler Knochen aus dem Hals des Talus) ergaben die nächsthöheren Erträge (ohne statistische Signifikanz zwischen diesen anatomischen Probenahmestellen; Abbildung 6A-B; Ergänzende Datei 1: EndogenousDNAPreCap). Diese alternativen Standorte produzierten alle konsistent DNA-Ausbeuten, die für populationsgenetische Standardanalysen wie mitochondriale Analysen und Einzelnukleotid-Polymorphismus-Analysen (SNP) geeignet sind. Die Duplikationsraten in Bibliotheken, die von allen anatomischen Stichprobenorten stammten, waren niedrig (Clusterfaktoren < 1,2 im Durchschnitt, berechnet als Verhältnis aller Mapping-Lesevorgänge zu eindeutigen Mapping-Lesevorgängen, Tabelle 2; Supplemental File 1: ClusterFactor), was darauf hinweist, dass alle untersuchten Bibliotheken von sehr hoher Komplexität waren. In ähnlicher Weise waren die durchschnittlichen Schätzungen der exogenen DNA-Kontamination beim Menschen niedrig und betrugen durchschnittlich < 2% (X-Chromosomenkontamination bei Männern, n = 7, wie von der ANGSD30-Pipeline berichtet) an allen anatomischen Probenahmestellen mit Ausnahme des oberen Wirbelbogens (durchschnittliche geschätzte Kontamination: 2,11%, wobei eine Probe als Ausreißer entfernt wurde; KRA005: 19,52%, siehe Tabelle 2; Ergänzende Datei 1: Xkontamination). Die durchschnittliche Fragmentlänge (nach Filterung, um alle Messwerte < 30 bp zu entfernen) war am niedrigsten in dem Material, das aus der Zahnpulpakammer und dem Dentin gesammelt wurde, ohne signifikante Unterschiede zwischen anderen anatomischen Probenahmestellen (55,14 bp bzw. 60,22 bp im Vergleich zu einem durchschnittlichen Median von 62,87, paarweise p-Werte < 0,019, Tabelle 2; Ergänzende Datei 1: AvgFragLength). Darüber hinaus enthalten die Zähne und Brustwirbel jeweils mehrere anatomische Probenahmestellen, an denen eine hohe endogene DNA-Erholung beobachtet wurde, was sie besonders als Alternative zur Pars petrosa geeignet macht.
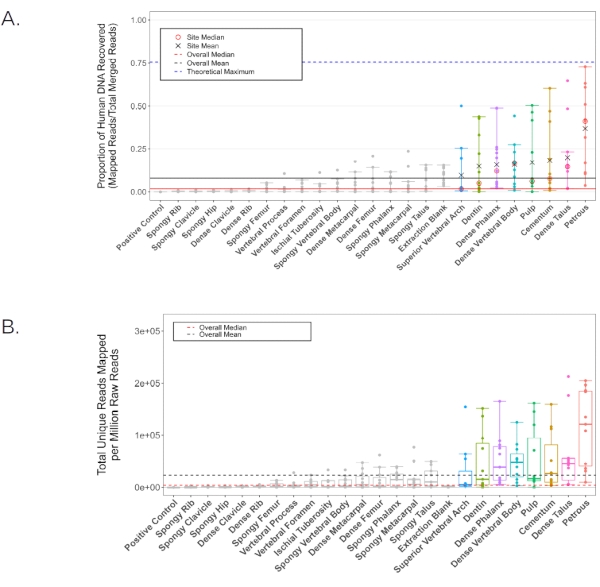
Abbildung 6: Menschlicher DNA-Gehalt für alle untersuchten Proben. Schwarze Linien stellen den Gesamtmittelwert dar, während rote Linien den Median darstellen (durchgehend: menschlicher DNA-Anteil, gestrichelt: abgebildete menschliche Lesevorgänge pro Million generierter Lesevorgänge). Einzelne anatomische Probenahmestellen mit einem durchschnittlichen menschlichen DNA-Anteil über dem Gesamtmittelwert (8,16%) werden in allen Analysen eingefärbt. (A) Der Anteil der Lesevorgänge, die auf das hg19-Referenzgenom abgebildet sind. Die blaue gestrichelte Linie stellt das theoretische Maximum unter Berücksichtigung der Kartierungsparameter der Pipeline dar (generiert mit Gargammel31 , um eine zufällige Verteilung von 5.000.000 Lesevorgängen aus dem hg19-Referenzgenom mit simulierten Schäden zu simulieren). Individuelle Mittelwerte (schwarzes X) und Mediane (roter Kreis) werden für diejenigen Proben mit einem höheren durchschnittlichen menschlichen DNA-Anteil als der Gesamtmittelwert angegeben. Konfidenzintervalle geben obere und untere Grenzen ohne statistische Ausreißer an. (B) Die Anzahl der eindeutigen Lesevorgänge, die dem hg19-Referenzgenom zugeordnet werden, pro Million Lesungen des Sequenzierungsaufwands (75 bp gepaartes Ende). Konfidenzintervalle geben obere und untere Grenzen ohne statistische Ausreißer an. Diese Abbildung wurde von Parker, C. et al. 202011 angepasst. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Tabelle 2: Durchschnittliche Duplikationsgrade (Mapping-Lesevorgänge/eindeutige Lesevorgänge), durchschnittliche und mediane Fragmentlängen und Schätzungen der X-Chromosomenkontamination für alle anatomischen Probenahmestellen. Fehler, der als Standardfehler des Mittelwerts gemeldet wird. Diese Tabelle wurde angepasst von Parker, C. et al. 202011.
| Probenahmeort | Durchschnittlicher Duplizierungsfaktor (# zugeordnete Lesevorgänge /# eindeutige zugeordnete Lesevorgänge) | Durchschnittliche Fragmentlänge in bp | Durchschnittlicher geschätzter Anteil der X-Chromosom-Kontamination |
| Petrous-Pyramide | 1,188 ± 0,006 | 65,40 ± 1,36 | 0,000 ± 0,003 |
| Zement | 1.197 ± 0.028 | 67,28 ± 1,76 | 0,011 ± 0,003 |
| Dentin | 1.188 ± 0.061 | 60,22 ± 2,37 | 0,002 ± 0,007 |
| Fruchtfleisch | 1,179 ± 0,024 | 55,14 ± 2,90 | 0,013 ± 0,006 |
| Endglied | 1,191 ± 0,049 | 65,95 ± 1,08 | 0,013 ± 0,005 |
| Wirbelkörper | 1,194 ± 0,037 | 66,14 ± 1,03 | 0,008 ± 0,003 |
| Oberer Wirbelbogen | 1,19 ± 0,017 | 63,02 ± 1,23 | 0,021 ± 0,009* |
| Sprungbein | 1.198 ± 0.010 | 68,20 ± 1,24 | 0,011 ± 0,003 |
| *Probe KRA005 als Ausreißer bei 0,1952 entfernt |
Code-Verfügbarkeit
Alle Analyseprogramme und R-Module, die in den Analysen dieses Manuskripts verwendet werden, sind bei ihren jeweiligen Autoren frei erhältlich. Alle benutzerdefinierten R-Codes sind auf Anfrage erhältlich.
Datenverfügbarkeit
Alle Rohdaten, die für die Berechnung repräsentativer Ergebnisse verwendet werden, sind im European Nucleotide Archive ENA Data Repository (Zugangsnummer PRJ-EB36983) oder ergänzenden Materialien von Parker, C. et al.11 frei verfügbar.
Ergänzende Datei 1. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die derzeitige Praxis in der alten menschlichen Populationsgenetik besteht darin, wann immer möglich bevorzugt aus der Pars petrosa (Schritt 2.1) zu entnehmen. Die Pars petrosa kann jedoch eine schwer zu erhaltende Probe sein, da sie für eine Vielzahl von Skelettbewertungen sehr geschätzt wird (z. B. Bevölkerungsgeschichte32, die Schätzung des fetalen Alters bei Tod 33 und Geschlechtsbestimmung34), und historisch gesehen kann die Probenahme der Pars petrosa für die DNA-Analyse sehr destruktiv sein3,4 (einschließlich des hier vorgestellten Protokolls, Obwohl neue, minimalinvasive Protokolle13,14 inzwischen weitgehend übernommen wurden, um diese Bedenken zu zerstreuen). Dies wird durch die Tatsache verstärkt, dass bis vor kurzem keine groß angelegte, systematische Untersuchung der menschlichen DNA-Erholung im gesamten Skelett versucht wurde11, was die Suche nach einer geeigneten Probenahmestrategie erschwert, wenn die Petrous-Pyramide nicht verfügbar ist.
Die hier vorgestellten Protokolle helfen, diese Herausforderung zu lindern, indem sie eine Reihe optimierter Verfahren für die DNA-Probenahme aus archäologischen / forensischen Skelettresten einschließlich der Pars Petrosa sowie sieben alternative anatomische Probenahmestellen für vier zusätzliche Skelettelemente bereitstellen. Die enthaltenen kritischen Schritte zielen alle darauf ab, die Möglichkeit eines DNA-Verlusts/-Schadens aufgrund einer ineffizienten Probenahme (Schritte 2.1.6 und 3.2.1.3) oder einer Überhitzung der Proben während des Bohrens/Schneidens (Schritt 3.1.6) zu minimieren. Darüber hinaus wurde im gesamten Protokoll darauf hingewiesen, dass es notwendig sein kann, die Vorbehandlungsschritte zu modifizieren / wegzulassen, um die beste Leistung bei stark abgebauten Proben zu gewährleisten. Es sollte auch darauf hingewiesen werden, dass selbst unter den hier vorgestellten ausgewählten Elementen mehrere mögliche alternative Probenahmetechniken (insbesondere für die Pars Petrosa13,14) sowie noch viel Raum für eine weitere Optimierung der hier vorgestellten nicht ausgebeuteten anatomischen Probenahmestellen (d. h. Talus: Schritt 2.5 und die Wirbel: Schritt 2.3) verbleiben.
Es ist auch wichtig zu bedenken, dass diese Protokolle mit alten juvenil-adulten Überresten von hoher Qualität (gute morphologische Konservierung) für die Zwecke endogener menschlicher DNA-Analysen entworfen und getestet wurden. Die vorgestellten Ergebnisse erstrecken sich möglicherweise nicht auf stärker abgebaute Materialien, andere Konservierungskontexte, Säuglingsreste, nichtmenschliche Überreste oder Studien zu Krankheitserregern oder dem Mikrobiom, da eine stärkere Erforschung der Verwendung dieser Protokolle in zusätzlichen Kontexten noch erforderlich ist. Darüber hinaus können die hier vorgestellten alternativen Skelettelemente (die Zähne, Wirbel, distale Phalanx und Tali) schwierig sein, einem einzelnen Individuum unter vermischten Überresten zuzuordnen, so dass eine Probenahme aus mehreren Elementen erforderlich ist, um einen einzigen Ursprung zu gewährleisten. Trotz dieser Einschränkungen kann die breite Verfügbarkeit dieser Protokolle dazu beitragen, einen Teil der Heterogenität bei der Probenauswahl und -verarbeitung zu verringern, indem ein generalisierter und quantitativ optimierter Rahmen für die Verwendung in einer Vielzahl zukünftiger aDNA/forensischer Studien an menschlichen Überresten bereitgestellt wird.
Offenlegungen
Die Autoren haben keine Interessenkonflikte zu melden.
Danksagungen
Die Autoren danken den Labormitarbeitern des Max-Planck-Instituts für Menschheitsgeschichte für ihre Hilfe bei der Entwicklung und Umsetzung dieser Protokolle. Diese Arbeit wäre ohne den Einsatz und die harte Arbeit von Dr. Guido Brandt, Dr. Elizabeth Nelson, Antje Wissegot und Franziska Aron nicht möglich gewesen. Diese Studie wurde von der Max-Planck-Gesellschaft, dem Europäischen Forschungsrat (ERC) im Rahmen des Forschungs- und Innovationsprogramms Horizon 2020 der Europäischen Union im Rahmen der Finanzhilfevereinbarungen Nr. 771234 - PALEoRIDER (WH, ABR) und Starting Grant Nr. 805268 CoDisEASe (an KIB) gefördert.
Materialien
| Name | Company | Catalog Number | Comments |
| #16 Dental Drill Bit | NTI | H1-016-HP | example drilling bit |
| 0.6 mm scroll saw blade | Fisher Scientific | 50-949-097 | blade for Jewellers Saw |
| 22mm diamond cutting wheel | Kahla | SKU 806 104 358 514 220 | Dremel cutting attachment |
| Commercial Bleach | Fisher Scientific | NC1818018 | |
| Control Company Ultra-Clean Supreme Aluminum Foil | Fisher Scientific | 15-078-29X | |
| DNA LoBind Tubes (2 mL) | Eppendorf | 22431048 | |
| Dremel 225-01 Flex Shaft Attachment | Dremel | 225-01 | Dremel flexible extension |
| Dremel 4300 Rotary Tool | Dremel | 4300 | Example drill |
| Dremel collet and nut kit | Dremel | 4485 | Adapters for various Dremel tool attachments/bits |
| Eagle 33 Gallon Red Biohazard Waste Bag | Fisher Scientific | 17-988-501 | |
| Eppendorf DNA LoBind 2 mL microcentrifuge tube | Fisher Scientific | 13-698-792 | |
| Ethanol (Molecular Biology Grade) | Millipore Sigma | 1.08543 | |
| FDA approved level 2 Surgical Mask | Fisher Scientific | 50-206-0397 | PPE |
| Fisherbrand Comfort Nitrile Gloves | Fisher Scientific | 19-041-171X | PPE |
| Fisherbrand Safety Glasses | Fisher Scientific | 19-130-208X | PPE |
| Granger Stationary Vise | Fisher Scientific | NC1336173 | benchtop vise |
| Invitrogen UltraPure DNase/Rnase free distilled water | Fisher Scientific | 10-977-023 | |
| Jewellers Saw | Fisher Scientific | 50-949-231 | |
| Kimwipes | Sigma-Aldritch | Z188956 | |
| Labconco Purifier Logic Biosafety cabinet | Fisher Scientific | 30-368-1101 | |
| LookOut DNA Erase | Millipore Sigma | L9042-1L | |
| Medium weighing boat | Heathrow Scientific | HS120223 | |
| MSC 10pc plier/clamp set | Fisher Scientific | 50-129-5352 | Miscellaneous clamps/vise grips for securely holding samples while drilling/cutting |
| Sartorius Quintix Semi-Micro Balance | Fisher Scientific | 14-560-019 | enclosed balance |
| Tyvek coveralls with hood | Fisher Scientific | 01-361-7X | PPE |
| Weigh paper | Heathrow Scientific | HS120116 |
Referenzen
- Adler, C. J., Haak, W., Donlon, D., Cooper, A. Survival and recovery of DNA from ancient teeth and bones. Journal of Archaeological Science. 38 (5), 956-964 (2011).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Ancient DNA: Methods and Protocols. , 25-29 (2019).
- Palsdottir, A. H., Bläuer, A., Rannamäe, E., Boessenkool, S., Hallsson, J. Not a limitless resource: ethics and guidelines for destructive sampling of archaeofaunal remains. Royal Society Open Science. 6 (10), 191059 (2019).
- Pinhasi, R., Fernandes, D. M., Sirak, K., Cheronet, O. Isolating the human cochlea to generate bone powder for ancient DNA analysis. Nature Protocols. 14 (4), 1194-1205 (2019).
- Latham, K. E., Miller, J. J. DNA recovery and analysis from skeletal material in modern forensic contexts. Forensic Sciences Research. 4 (1), 51-59 (2019).
- Mundorff, A. Z., Bartelink, E. J., Mar-Cash, E. DNA preservation in skeletal elements from the World Trade Center disaster: Recommendations for mass fatality management. Journal of Forensic Sciences. 54 (4), 739-745 (2009).
- Gamba, C., et al. Genome flux and stasis in a five millennium transect of European prehistory. Nature Communications. 5 (1), 1-9 (2014).
- Alberti, F., et al. Optimized DNA sampling of ancient bones using Computed Tomography scans. Molecular Ecology Resources. 18 (6), 1196-1208 (2018).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940 (2017).
- Sirak, K., et al. Human auditory ossicles as an alternative optimal source of ancient DNA. Genome Research. 30 (3), 427-436 (2020).
- Parker, C., et al. A systematic investigation of human DNA preservation in medieval skeletons. Scientific Reports. 10 (1), 18225 (2020).
- Pinhasi, R., et al. Optimal ancient DNA yields from the inner ear part of the human petrous bone. PLoS ONE. 10 (6), 0129102 (2015).
- Sirak, K. A., et al. A minimally-invasive method for sampling human petrous bones from the cranial base for ancient DNA analysis. BioTechniques. 62 (6), 283-289 (2017).
- . Minimally-invasive sampling of pars petrosa (os temporale) for ancient DNA extraction. protocols.io Available from: https://www.protocols.io/view/minimally-invasive-sampling-of-pars-petrosa-os-tem-bqd8ms9w (2020)
- Damgaard, P. B., et al. Improving access to endogenous DNA in ancient bones and teeth. Scientific Reports. 5 (1), 1-12 (2015).
- Harney, &. #. 2. 0. 1. ;., et al. A minimally destructive protocol for DNA extraction from ancient teeth. Genome Research. 31 (3), 472-483 (2021).
- Cooper, A., Poinar, H. N. Ancient DNA: Do it right or not at all. Science. 289 (5482), 1139 (2000).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Boessenkool, S., et al. Combining bleach and mild predigestion improves ancient DNA recovery from bones. Molecular Ecology Resources. 17 (4), 742-751 (2017).
- García-Garcerà, M., et al. Fragmentation of contaminant and endogenous DNA in ancient samples determined by shotgun sequencing; Prospects for human palaeogenomics. PLoS ONE. 6 (8), 24161 (2011).
- Malmström, H., et al. More on contamination: The use of asymmetric molecular behavior to identify authentic ancient human DNA. Molecular Biology and Evolution. 24 (4), 998-1004 (2007).
- Basler, N., et al. Reduction of the contaminant fraction of DNA obtained from an ancient giant panda bone. BMC Research Notes. 10, 754 (2017).
- Kemp, B. M., Smith, D. G. Use of bleach to eliminate contaminating DNA from the surface of bones and teeth. Forensic Science International. 154 (1), 53-61 (2005).
- Korlević, P., et al. Reducing microbial and human contamination in DNA extractions from ancient bones and teeth. BioTechniques. 59 (2), 87-93 (2015).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Methods in Molecular Biology. 1963, 25-29 (2019).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940 (2017).
- Gansauge, M. -. T., et al. Single-stranded DNA library preparation from highly degraded DNA using T4 DNA ligase. Nucleic Acids Research. 45 (10), 79 (2017).
- Peltzer, A., et al. EAGER: efficient ancient genome reconstruction. Genome Biology. 17 (1), 60 (2016).
- Korneliussen, T. S., Albrechtsen, A., Nielsen, R. ANGSD: analysis of next generation sequencing data. BMC Bioinformatics. 15, 356 (2014).
- Renaud, G., Hanghøj, K., Willerslev, E., Orlando, L. Gargammel: A sequence simulator for ancient DNA. Bioinformatics. 33 (4), 577-579 (2017).
- Ponce de León, M. S., et al. Human bony labyrinth is an indicator of population history and dispersal from Africa. Proceedings of the National Academy of Sciences of the United States of America. 115 (16), 4128-4133 (2018).
- Nagaoka, T., Kawakubo, Y. Using the petrous part of the temporal bone to estimate fetal age at death. Forensic Science International. 248, 188 (2015).
- Norén, A., Lynnerup, N., Czarnetzki, A., Graw, M. Lateral angle: A method for sexing using the petrous bone. American Journal of Physical Anthropology. 128 (2), 318-323 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten