Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Erforschung der biomolekularen Interaktion zwischen dem molekularen Chaperon Hsp90 und seiner Client-Proteinkinase Cdc37 unter Verwendung der Feldeffekt-Biosensortechnologie
In diesem Artikel
Zusammenfassung
Field-Effect Biosensing (FEB) ist eine markierungsfreie Technik zum Nachweis biomolekularer Wechselwirkungen. Es misst den elektrischen Strom durch den Graphen-Biosensor, an den die Bindungsziele immobilisiert werden. Die FEB-Technologie wurde verwendet, um biomolekulare Wechselwirkungen zwischen Hsp90 und Cdc37 zu bewerten und eine starke Wechselwirkung zwischen den beiden Proteinen wurde nachgewiesen.
Zusammenfassung
Biomolekulare Wechselwirkungen spielen in zahlreichen zellulären Prozessen eine vielseitige Rolle, indem sie funktionell relevante biologische Ereignisse regulieren und koordinieren. Biomoleküle wie Proteine, Kohlenhydrate, Vitamine, Fettsäuren, Nukleinsäuren und Enzyme sind grundlegende Bausteine von Lebewesen; Sie fügen sich zu komplexen Netzwerken in Biosystemen zusammen, um eine Vielzahl von Lebensereignissen zu synchronisieren. Proteine nutzen typischerweise komplexe Interaktomnetzwerke, um ihre Funktionen auszuführen; Daher ist es zwingend erforderlich, solche Wechselwirkungen zu bewerten, um ihre Bedeutung in Zellen sowohl auf zellulärer als auch auf organismischer Ebene zu entschlüsseln. Um dieses Ziel zu erreichen, führen wir eine schnell aufstrebende Technologie ein, die Feldeffekt-Biosensorik (FEB), um spezifische biomolekulare Wechselwirkungen zu bestimmen. FEB ist eine Benchtop-, markierungsfreie und zuverlässige biomolekulare Detektionstechnik zur Bestimmung spezifischer Wechselwirkungen und verwendet hochwertige elektronisch-basierte Biosensoren. Die FEB-Technologie kann aufgrund der biokompatiblen Nanomaterialien, die auf ihrer Biosensoroberfläche verwendet werden, Wechselwirkungen im nanomolaren Bereich überwachen. Als Proof of Concept wurde die Protein-Protein-Interaktion (PPI) zwischen Hitzeschockprotein 90 (Hsp90) und Zellteilungszyklus 37 (Cdc37) aufgeklärt. Hsp90 ist ein ATP-abhängiges molekulares Chaperon, das eine wesentliche Rolle bei der Faltung, Stabilität, Reifung und Qualitätskontrolle vieler Proteine spielt und dadurch mehrere lebenswichtige Zellfunktionen reguliert. Cdc37 gilt als Proteinkinase-spezifisches molekulares Chaperon, da es spezifisch Proteinkinasen zu Hsp90 erkennt und rekrutiert, um ihre nachgeschalteten Signaltransduktionswege zu regulieren. Daher gilt Cdc37 als Co-Chaperone von Hsp90. Der Chaperon-Kinase-Signalweg (Hsp90/Cdc37-Komplex) ist bei mehreren Malignomen, die das Zellwachstum fördern, hyperaktiviert; Daher ist es ein potenzielles Ziel für die Krebstherapie. Die vorliegende Studie demonstriert die Effizienz der FEB-Technologie anhand des Modellsystems Hsp90/Cdc37. FEB wies einen starken PPI zwischen den beiden Proteinen nach (K-D-Werte von 0,014 μM, 0,053 μM und 0,072 μM in drei unabhängigen Experimenten). Zusammenfassend lässt sich sagen, dass FEB eine markierungsfreie und kostengünstige PPI-Erkennungsplattform ist, die schnelle und genaue Messungen ermöglicht.
Einleitung
Biomolekulare Wechselwirkungen:
Proteine sind wesentliche Bestandteile von Organismen und an zahlreichen molekularen Signalwegen wie Zellstoffwechsel, Zellstruktur, Zellsignalisierung, Immunantwort, Zelladhäsion und mehr beteiligt. Während einige Proteine ihre Funktion(en) unabhängig voneinander ausführen, interagieren die meisten Proteine mit anderen Proteinen über eine Bindungsgrenzfläche, um die richtige biologische Aktivität zu koordinieren1.
Biomolekulare Wechselwirkungen können hauptsächlich auf der Grundlage der unterschiedlichen strukturellen und funktionellen Eigenschaften der beteiligten Proteineklassifiziert werden 2, beispielsweise basierend auf den Proteinoberflächen, der komplexen Stabilität oder der Persistenz von Wechselwirkungen3. Die Identifizierung essentieller Proteine und ihrer Rolle in biomolekularen Wechselwirkungen ist entscheidend für das Verständnis biochemischer Mechanismen auf molekularer Ebene4. Derzeit gibt es verschiedene Ansätze, um diese Wechselwirkungen5 zu erkennen: in vitro 6, in silico7, in lebenden Zellen8, ex vivo 9 und in vivo10, wobei jede ihre eigenen Stärken und Schwächen hat.
Die In-vivo-Assays werden unter Verwendung des gesamten Tieres als experimentelles Werkzeugdurchgeführt 11, unddie Ex-vivo-Assays werden an Gewebeextrakten oder ganzen Organen (z. B. Herz, Gehirn, Leber) in einer kontrollierten äußeren Umgebung durchgeführt, wobei die natürlichen Bedingungen nur minimal verändert werden. Die häufigste Anwendung von In-vivo- und Ex-vivo-Studien besteht darin, die pharmakokinetischen, pharmakodynamischen und toxischen Wirkungen potenzieller pharmakologischer Wirkstoffe vor Studien am Menschen zu bewerten, indem ihre allgemeine Sicherheit und Wirksamkeit sichergestelltwird 12.
Biomolekulare Wechselwirkungen können auch innerhalb lebender Zellen nachgewiesen werden. Die Bildgebung lebender Zellen ermöglicht es uns, dynamische Wechselwirkungen zu beobachten, während sie die Reaktionen eines bestimmten biochemischen Weges ausführen13. Darüber hinaus können Detektionstechniken wie Biolumineszenz oder Fluoreszenzresonanz-Energietransfer Informationen darüber liefern, wo und wann diese Wechselwirkungen innerhalb der Zelle auftreten14. Obwohl der Nachweis in lebenden Zellen entscheidende Details bietet, stützen sich diese Nachweismethoden auf Optik und Markierungen, die möglicherweise nicht die native Biologie widerspiegeln. Sie sind auch weniger kontrolliert als In-vitro-Methoden und erfordern spezielles Fachwissen, um15 durchzuführen.
Die In-silico-Berechnungsmethoden werden in erster Linie für das groß angelegte Screening von Zielmolekülen vor den In-vitro-Experimenten eingesetzt. Computergestützte Vorhersagemethoden, computergestützte Datenbanken, molekulares Docking, quantitative Struktur-Aktivitäts-Beziehungen und andere molekulardynamische Simulationsansätze gehören zu den etablierten In-silico-Werkzeugen 16. Im Vergleich zu mühsamen experimentellen Techniken können die In-silico-Werkzeuge leicht Vorhersagen mit hoher Empfindlichkeit, aber mit reduzierter Genauigkeit in der Vorhersageleistungtreffen 17.
In-vitro-Assays werden mit Mikroorganismen oder biologischen Molekülen außerhalb ihres biologischen Standardkontexts durchgeführt. Die Darstellung biomolekularer Interaktionen durch In-vitro-Methoden ist entscheidend für das Verständnis von Proteinfunktionen und der Biologie hinter dem komplexen Netzwerk der Zellfunktion. Die bevorzugte Assay-Methodik wird entsprechend den intrinsischen Eigenschaften des Proteins, den kinetischen Werten und dem Modus und der Intensität der Wechselwirkungenausgewählt 18,19.
Die Hsp90/Cdc37-Interaktion:
Der Chaperon-Kinase-Signalweg, der Hsp90 und Cdc37 verbindet, ist ein vielversprechendes therapeutisches Ziel in der Tumorbiologie20. Hsp90 spielt eine zentrale Rolle bei der Kontrolle des Zellzyklus, der Proteinzusammenordnung, dem Zellüberleben und den Signalwegen. Proteine, die für ihre Funktionen auf Hsp90 angewiesen sind, werden zur Komplexierung durch ein Co-Chaperon wie Cdc37 an Hsp90 abgegeben. Der Hsp90/Cdc37-Komplex steuert die Faltung der meisten Proteinkinasen und dient als Drehscheibe für eine Vielzahl von intrazellulären Signalnetzwerken21. Es ist ein vielversprechendes Anti-Tumor-Ziel aufgrund seiner erhöhten Expression bei verschiedenen Malignomen, einschließlich akuter myeloblastischer Leukämie, multiplem Myelom und hepatozellulärem Karzinom22,23.
Häufig verwendete biomolekulare In-vitro-Interaktionsnachweistechniken
Die Co-Immunpräzipitation (Co-IP) ist eine Technik, die sich auf die Spezifität von Antigen-Antikörpern stützt, um biologisch relevante Wechselwirkungen zu identifizieren24. Der Hauptnachteil dieser Methode ist ihre Unfähigkeit, Wechselwirkungen mit geringer Affinität und kinetische Werte zu erkennen24. Biophysikalische Methoden wie die isotherme Titrationkalorimetrie (ITC), die Oberflächenplasmonenresonanz (SPR), die Biolayer-Interferometrie (BLI) und die FEB-Technologie werden bevorzugt, um die kinetischen Werte zu bestimmen.
ITC ist eine biophysikalische Nachweismethode, die auf der Bestimmung der Bindungsenergie zusammen mit einer vollständigen thermodynamischen Analyse zur Charakterisierung biomolekularer Wechselwirkungenbasiert 25. Der Hauptvorteil von ITC besteht darin, dass keine Markierung oder Fixierung des Zielproteins erforderlich ist. Die Hauptschwierigkeiten, mit denen ITC konfrontiert ist, sind die hohe Konzentration des Zielproteins, die für ein Experiment erforderlich ist, und die Schwierigkeit, nicht-kovalente Komplexe aufgrund kleiner Bindungsenthalpienzu analysieren 26. Sowohl SPR als auch BLI sind markierungsfreie biophysikalische Techniken, die auf der Immobilisierung des Zielmoleküls auf der Sensoroberfläche beruhen, gefolgt von nachfolgenden Injektionen des Analyten über das immobilisierte Ziel27,28. Bei SPR werden Veränderungen des Brechungsindex während biomolekularer Wechselwirkungen gemessen27; In BLI wird die Interferenz im reflektierten Licht in Echtzeit als Änderung der Wellenlänge als Funktion der Zeit28 aufgezeichnet. Sowohl SPR als auch BLI haben gemeinsame Vorteile durch hohe Spezifität, Sensitivität und Erkennungsfunktionen29. Bei beiden Methoden wird das Zielprotein auf Biosensoroberflächen immobilisiert, und daher kann es zu einem gewissen Verlust der nativen Konformation des Ziels kommen, was es schwierig macht, zwischen spezifischen und unspezifischen Wechselwirkungenzu unterscheiden 30. BLI verwendet teure faseroptische Einweg-Biosensoren, um das Ziel zu immobilisieren, und ist daher eine kostspielige Technik31. Im Vergleich zu diesen etablierten biomolekularen Detektionswerkzeugen bietet die FEB-Technologie eine zuverlässige und markierungsfreie Plattform, indem sie niedrige nanomolare Konzentrationen für den biomolekularen Nachweis in Echtzeit mit kinetischer Charakterisierung verwendet. Die FEB-Technologie überwindet auch die sprudelnden Herausforderungen in der ITC und ist im Vergleich zu SPR oder BLI kostengünstiger.
Die auf dem Feldeffekttransistor (FET) basierenden Biosensoren sind ein aufstrebendes Gebiet zur Erkennung biomolekularer Wechselwirkungen, indem sie vielfältige biomedizinische Anwendungen anbieten. Im FET-System werden Ziele für die Biosensorchips immobilisiert und Wechselwirkungen durch Änderungen der Leitfähigkeit32 erkannt. Das Alleinstellungsmerkmal, das bei der Entwicklung eines effizienten elektronischen Biosensors zu berücksichtigen ist, sind die physikalisch-chemischen Eigenschaften wie die halbleitende Natur und die chemische Stabilität des Beschichtungsmaterials, das zur Herstellung der Sensoroberflächeverwendet wird 33. Herkömmliche Materialien wie Silizium, die für FET verwendet werden, haben die Empfindlichkeit von Sensoren eingeschränkt, da Oxidschichten zwischen dem Transistorkanal und einer spezifischen Umgebung für ein ordnungsgemäßes Funktionierenerforderlich sind 34. Darüber hinaus sind Siliziumtransistoren empfindlich gegenüber salzreichen Umgebungen, was es schwierig macht, biologische Wechselwirkungen in ihrer natürlichen Umgebung zu messen. Der graphenbasierte Biosensor wird als Alternative vorgestellt, da er eine hervorragende chemische Stabilität und ein elektrisches Feld bietet. Da Graphen eine einzelne Atomschicht aus Kohlenstoff ist, ist es sowohl als Halbleiter extrem empfindlich als auch chemisch mit biologischen Lösungen kompatibel; Beide Eigenschaften sind wünschenswert, um kompatible elektronische Biosensoren35 zu erzeugen. Das bemerkenswerte ultrahohe Belastungspotenzial von Biomolekülen, das graphenbeschichtete Biosensoren bieten, führte zur Entwicklung von graphenbasierten Biosensoren FEB-Technologie.
Prinzip der FEB-Technologie: FEB ist eine markierungsfreie biomolekulare Detektionstechnik, die den elektrischen Strom durch den Graphen-Biosensor misst, an den die Bindungsziele immobilisiert werden. Wechselwirkungen zwischen dem immobilisierten Protein und dem Analyten führen zu Stromänderungen, die in Echtzeit überwacht werden, was genaue kinetische Messungenermöglicht 36.
Instrumentierung: Das FEB-System besteht aus einem Graphen-Feldeffekttransistor (gFET)-Sensorchip und einem elektronischen Lesegerät, das während des gesamten Experiments eine konstante Spannung anlegt (Abbildung 1). Der Analyt wird in Lösung auf das Zielprotein aufgetragen, das auf der Biosensoroberfläche immobilisiert ist. Wenn eine Wechselwirkung auftritt, wird eine Veränderung des Stroms gemessen und in Echtzeit aufgezeichnet. Wenn die Analytkonzentration zunimmt, nimmt auch der Anteil des gebundenen Analyten zu, was zu höheren Veränderungen in der Strömung führt. Mit der mit dem Gerät gelieferten automatisierten Analysesoftware (Table of Materials) wird I-Response in Form von Biosensing-Einheiten (BU)37 gemessen und aufgezeichnet. I-Response ist definiert als die Veränderung des Stroms (I) durch den Biosensorchip, die in Echtzeit bei der Interaktion des immobilisierten Ziels mit dem Analyten gemessen wird. Die automatisierte Analysesoftware FEB kann sowohl die I-Response als auch die C-Response auf dynamische Interaktionsereignisse analysieren, wobei die C-Response die Änderungen der Kapazität (C) aufzeichnet. Die Variationen sowohl der I-Response als auch der C-Response entsprechen direkt dem Anteil des gebundenen Analyten und können weiter analysiert werden, um KD-Werte zu erzeugen. Die Standardeinstellung der automatisierten Analysesoftware ist I-Response.
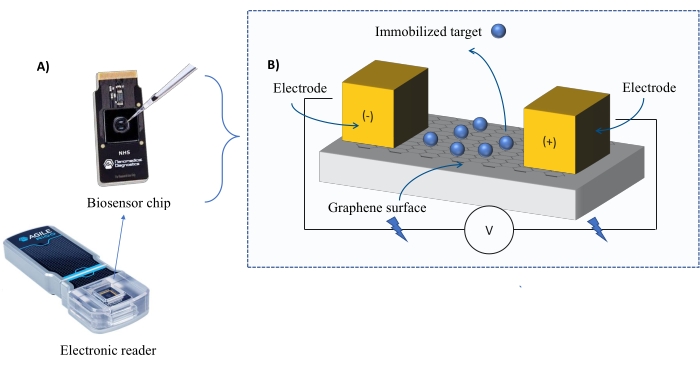
Abbildung 1: Überblick über den Versuchsaufbau . (A) Chip auf Graphenbasis und ein elektronisches Lesegerät. (B) Eine Übersicht über die Chipkomponenten. Der Chip ist an zwei Elektroden befestigt, die das System mit Strom versorgen. Die Oberfläche des Chips ist mit Graphen bedeckt, das bei Aktivierung das Ziel binden kann. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Methodologie:
Zunächst wird der aktivierte Biosensorchip in das FEB-Gerät eingesetzt (Abbildung 1), gefolgt von der Ausführung der folgenden Schritte: (1) Kalibrierung: Das Experiment beginnt mit der Systemkalibrierung unter Verwendung von 1x phosphatgepufferter Kochsalzlösung (PBS; pH = 7,4), um die Ausgangsgleichgewichtsantwort zu erzeugen. (2) Assoziation: Der Analyt wird in den Chip eingeführt und die I-Antwort wird überwacht, bis die Bindungssättigung erreicht ist. (3) Dissoziation: Der Analyt wird mit 1x PBS dissoziiert. (4) Regeneration: Reste des Analyten werden mit 1x PBS entfernt. (5) Waschen: Insgesamt werden fünf Waschungen mit 1x PBS durchgeführt, um die gebundenen und ungebundenen Analyten gründlich vom Chip zu entfernen.
Analyse:
Die Datenanalyse erfolgt mit der vollautomatischen Software, die mit dem Gerät geliefert wird. Die automatisierte Analysesoftware generiert ein Hill-Fit-Diagramm mit einem KD-Wert . Das Hill-Fit-Diagramm beschreibt die Assoziation eines Analyten mit dem Zielprotein als Funktion der Analytkonzentrationen. Die Konzentration, bei der ein halbmaximales Ansprechen erreicht wird, ist proportional zum KD-Wert . Ein niedriger KD-Wert steht für eine hohe Bindungsaffinität und umgekehrt.
Um die aus dem FEB-Experiment gewonnenen Daten zu validieren, werden I-Responses von jedem Auslesepunkt für jede Analytkonzentration mit der Datenüberprüfungs-/Exportsoftware extrahiert und können in andere statistische Analysesoftware (siehe Tabelle der Materialien) exportiert werden, wie unten erläutert.
Access restricted. Please log in or start a trial to view this content.
Protokoll
HINWEIS: Die in dieser Studie verwendeten rekombinanten Proteine, Hsp90 und Cdc37, wurden kommerziell gewonnen (siehe Materialtabelle).
1. Chip-Aktivierung
HINWEIS: Alle Materialien, die im Experiment verwendet werden sollen, sind in der Tabelle der Materialien aufgeführt. Filtern Sie alle vorbereiteten Lösungen durch einen sterilen 0,2-μm-Filter.
- Es wird eine 1-Ethyl-3-(3-dimethylaminopropyl)carbodiimid (EDC)-Lösung hergestellt, indem 2 mg EDC zu 2,5 ml 1 M2-(N-morpholino)-Ethansulfonsäure (MES)-Puffer (pH = 6,0) in einem 15-ml-Röhrchen zugegeben werden. Herstellung von N-Hydroxysulfosuccinimid (Sulfo-NHS)-Lösungen durch Zugabe von 6 mg Sulfo-NHS zu 2,5 ml 1 M MES-Puffer (pH = 6,0) in einem separaten 15-ml-Röhrchen. Aliquot 50 μL jeder Lösung in unabhängigen Röhrchen und bei -20 °C für die zukünftige Verwendung lagern.
- Mischen Sie gleiche Volumina EDC-Sandsulfo-NHS-Lösung (50 μL EDC + 50 μL Sulfo-NHS) durch Pipettieren auf und ab (nicht vortexen).
HINWEIS: Die gemischte Lösung von EDC / Sulfo-NHS muss innerhalb von 30 Minuten verwendet werden, um eine effektive Vernetzung für eine ordnungsgemäße Chipfunktionalisierung aufrechtzuerhalten. - Legen Sie den vom Unternehmen gelieferten Biosensorchip (5,7 cm x 2,4 cm; siehe Materialtabelle) in eine Glas-Petrischale mit montiertem Deckel. Es wird empfohlen, alle Funktionalisierungsschritte bei der Chipaktivierung in der Petrischale durchzuführen. Tragen Sie 50 μL 1 M MES-Puffer (pH = 6,0) auf den Biosensorchip auf, inkubieren Sie für 1 Minute bei Raumtemperatur und saugen Sie dann den Puffer ab.
- Tragen Sie 50 μL EDC/Sulfo-NHS-Lösung sofort auf den Sensorchip auf. Die Petrischale abdecken und 15 min bei Raumtemperatur inkubieren. Saugen Sie die EDC/Sulfo-NHS-Lösung aus dem Chip ab.
- Spülen Sie den Chip einmal mit 50 μL 1 M MES-Puffer (pH = 6,0); den MES-Puffer aspirieren.
2. Deaktivierung des Zielproteins
- Spülen Sie den Chip 2x mit 50 μL 1x PBS (pH = 7,4) ab. Saugen Sie das PBS vom Chip ab und fügen Sie das Zielmolekül Hsp90 (50 μL; 500 nM) hinzu.
HINWEIS: Pufferkonflikte können das gesamte Experiment ruinieren. Daher ist es vor dem Experiment wichtig sicherzustellen, dass sich das Zielmolekül im selben Puffer befindet wie das für die Kalibrierung verwendete (z. B. 1x PBS (pH = 7,4)). Führen Sie bei Bedarf vor dem Experiment einen Pufferaustausch durch Nachtdialyse durch. In diesem Experiment wurde über Nacht eine Dialyse für beide rekombinanten Proteine, Hsp90 und Cdc37, gegen 1x PBS (pH = 7,4; siehe Materialtabelle) mit korrektem Pufferaustausch bei 4 °C durchgeführt. Die Konzentration des Zielmaterials (in diesem Fall Hsp90) kann je nach experimentellen Protokollen und der Art der Zielmaterialien (Protein/Peptid/Liganden) variieren. - Die Glas-Petrischale abdecken und 30 min bei Raumtemperatur inkubieren. Die Lösung, die das Zielmolekül enthält, abpumpen und 3x mit 50 μL 1x PBS (pH = 7,4) abspülen. Saugen Sie die 1x PBS (pH = 7,4) Lösung vom Chip ab.
- 50 μL Quench 1 (3,9 mM Amino-PEG5-Alkohol in 1x PBS (pH = 7,4)) Lösung in den Chip geben. Die Glas-Petrischale abdecken und 15 min bei Raumtemperatur inkubieren. Saugen Sie die Quench 1-Lösung vom Chip ab.
- 50 μL Quench 2 (1 M Ethanolamin (pH = 8,5)) Lösung in den Chip geben. Die Glas-Petrischale abdecken und 15 min bei Raumtemperatur inkubieren. Aspirieren Sie die Quench 2-Lösung aus dem Chip und spülen Sie den Chip 5x mit 50 μL 1x PBS ab, wobei der letzte PBS-Tropfen auf dem Sensor verbleibt.
3. Vorbereitung von Analytproben
- Bereiten Sie Analytverdünnungsreihen für Cdc37 im gewünschten Konzentrationsbereich vor. Für das erste Experiment wurden die folgenden Konzentrationen verwendet: 25 nM, 50 nM, 100 nM, 200 nM, 400 nM, 800 nM, 1.000 nM, 2.000 nM, 3.000 nM und 5.000 nM. Für das zweite Experiment wurde ein anderer Satz von Konzentrationen im Bereich von 0,4 nM bis 200 nM verwendet.
- Entwerfen Sie das Experiment so, dass es mindestens acht verschiedene Analytkonzentrationen umfasst, um einen zuverlässigen KD-Wert zu erhalten. Bereiten Sie die verschiedenen Verdünnungen des Analytproteins im selben Puffer vor, der für die Kalibrierung und das Zielprotein verwendet wird. hier ist es 1x PBS (pH = 7,4).
4. Laden des aktivierten Biosensor-Chips in das FEB-Gerät
HINWEIS: Das FEB-Gerät besteht aus einem Lesegerät mit LED-Lichtanzeigen und einer Kartusche zum Einsetzen des Biosensorchips.
- Setzen Sie nach der Immobilisierung des Zielproteins den aktivierten Chip in die Kartusche des Geräts ein, die über USB mit einem Computer verbunden ist. Nach dem Einsetzen des Chips wird ein grünes LED-Licht am Lesegerät angezeigt, das anzeigt, dass das FEB-Gerät für das Experiment bereit ist. Installieren Sie die vom Unternehmen bereitgestellte automatisierte Software (siehe Materialtabelle) auf dem Computer, an den das FEB-Gerät angeschlossen ist, um das Experiment Schritt für Schritt wie unten beschrieben zu überwachen.
5. Führen Sie das Experiment aus
- Drücken Sie das Modul Run Experiment in der automatisierten Software und wählen Sie 10 Punkte mit Regeneration oder ein anderes gewünschtes Protokoll. Geben Sie die folgenden Details ein: Operatorname, Experimentname, Datum (z. B. Yana, Hsp90 + Cdc37, 14.03.2021); Regenerationspuffer (z. B. PBS-Puffer); immobilisiertes Ziel (z. B. Hsp90); Analyt in Lösung (z. B. Cdc37). Siehe ergänzende Abbildung S1 für Details.
- Klicken Sie auf die Schaltfläche Experiment starten , die in der Software angezeigt wird, und folgen Sie den Anweisungen der automatisierten Software wie unten beschrieben.
HINWEIS: Die Software ist voll automatisiert, benutzerfreundlich und führt den Benutzer Schritt für Schritt durch das Experiment. Auf dem Bildschirm erscheint ein Popup-Fenster mit Anweisungen, bei jedem Schritt des Experiments fortzufahren. Die Software liefert Anweisungen für jeden sich wiederholenden Schritt nacheinander aus Kalibrierung, Analytassoziation, Dissoziation, Regeneration und Waschen (5x) für jede Analytkonzentration während des gesamten Experiments. - Führen Sie eine Gerätekalibrierung durch. Saugen Sie dazu die restliche PBS-Lösung vom Chip ab und legen Sie 50 μL Kalibrierpuffer (1x PBS; pH = 7,4) auf. Drücken Sie die Continue-Taste und warten Sie 5 Minuten, bis der Kalibrierungsschritt abgeschlossen ist. Die Software zeigt den für den Kalibrierungsschritt ermittelten Endpunkt (5 min) mit einem Warnalarm zur Nachverfolgung an.
- Führen Sie als Nächstes eine Analytzuordnung durch. Saugen Sie dazu den Kalibrierpuffer vom Chip ab und legen Sie 50 μL der niedrigsten Analytkonzentration (25 nM Cdc37) auf. Drücken Sie die Schaltfläche Weiter und warten Sie 5 Minuten, bis der Zuordnungsschritt abgeschlossen ist. Die Software zeigt den Endpunkt für den Assoziationsschritt (5 min) mit einem Warnalarm an, um fortzufahren.
- Führen Sie eine Analytdissoziation durch. Saugen Sie dazu die Analytlösung vom Chip ab und tragen Sie 50 μL des Dissoziationspuffers (1x PBS; pH = 7,4) auf. Drücken Sie die Continue-Taste und warten Sie 5 Minuten, bis die Dissoziationsschrittdauer (5 min) beendet ist. Die Software zeigt den Endpunkt für den Dissoziationsschritt (5 min) mit einem Warnalarm zur Nachverfolgung an.
- Führen Sie als Nächstes eine Chip-Regeneration durch. Die Dissoziationslösung wird vom Chip abgesaugt und 50 μL Regenerationspuffer (1x PBS; pH = 7,4) aufgetragen. Drücken Sie die Weiter-Taste und warten Sie 30 s, bis die Regenerationsschrittdauer (30 s) beendet ist. Die Software zeigt den Endpunkt für den Regenerationsschritt (30 s) mit einem Warnalarm zur Nachverfolgung an.
- Zum Schluss den Chip waschen. Die Regenerationslösung aus dem Chip abpumpen und 50 μL Waschpuffer (1x PBS; pH = 7,4) auf den Chip auftragen. Saugen Sie die Lösung aus dem Chip und wiederholen Sie dies 5x. Lassen Sie den letzten Tropfen Waschpuffer auf dem Chip und drücken Sie die Weiter-Taste und warten Sie 30 Sekunden, bis die Waschschrittdauer in der Softwareanzeige abgeschlossen ist.
HINWEIS: Die Software zeigt den Endpunkt für den Waschschritt (30 s) mit einem Warnalarm an, um mit dem nächsten Zyklus des Experiments fortzufahren. - Wiederholen Sie die Schritte für jede verwendete Analytkonzentration. Die fünf Schritte Kalibrierung, Analytassoziation, Dissoziation, Regeneration und Waschung (5x) bilden einen Zyklus. Für das hier gezeigte Experiment führten wir 10 Zyklen für 10 Analytkonzentrationen durch (von 25 nM bis 5.000 nM oder 0,4 nM bis 200 nM; Abbildung 2).
6. Analyse
- Drücken Sie die Analysetaste, die sich am Ende des Experiments oben in der automatisierten Analysesoftware befindet. Es erscheint ein Anzeigefenster mit allen experimentellen Punkten. Stellen Sie im Fenster sicher, dass die für das vorgeschriebene Protokoll verwendeten Analytkonzentrationen korrekt sind.
- Klicken Sie auf die Schaltfläche Analyse ausführen , um den KD-Wert automatisch zu generieren. Die Software generiert ein Hill-Fit-Diagramm, indem sie die Analytkonzentrationen gegen die entsprechenden I-Antworten aufzeichnet, aus denen die Dissoziationskonstante im Gleichgewicht, KD-Wert , berechnet wird.
- Exportieren Sie die Rohdaten zur Analyse in andere statistische Analysesoftware, indem Sie die Datenüberprüfungs-/Exportsoftware wie unten beschrieben verwenden.
- Kopieren Sie die am Ende des Experiments automatisch erstellte R1R-Datei (z. B. Hsp90 + Cdc37 14.03.2021) in einen neuen Ordner auf dem Desktop. Öffnen Sie die vom Unternehmen bereitgestellte Datenüberprüfungs-/Exportsoftware (siehe Ergänzende Abbildung S2A).
- Klicken Sie auf Datenverarbeitung > R1R-Dateien verarbeiten > auf dem Startbildschirm der Datenüberprüfungs-/Exportsoftware auf OK.
- Wählen Sie den Ordner mit R1R-Dateien aus, die in Schritt 6.3.1 auf dem Desktop erstellt wurden. und drücken Sie die OK-Taste . Dadurch wird eine Kopie der ursprünglichen R1R-Daten erstellt, um die Daten zu überprüfen und zu bearbeiten, ohne die Originaldatei zu überschreiben.
- Drücken Sie auf das Datenverarbeitungssymbol, das auf dem Startbildschirm der Datenüberprüfungs-/Exportsoftware angezeigt wird. Klicken Sie auf Processed R1R-Dateien laden, > OK. Wählen Sie denselben Ordner aus, der in Schritt 6.3.1 erstellt wurde und verarbeitete R1R-Dateien enthält. Drücken Sie OK. In diesem Schritt kann der Ordner, der die Testdateien enthält, überprüft werden.
- Drücken Sie auf dem Startbildschirm der Datenüberprüfungs-/Exportsoftware auf Datenanalyse. Wählen Sie Kalibrierung > Bearbeiten von R1R-Dateien kalibrieren > OK (siehe Ergänzende Abbildung S2B). In diesem Schritt werden alle Datenpunkte gemäß dem ersten Kalibrierungsschritt kalibriert, um eine Baseline zu erstellen.
- Drücken Sie auf dem Startbildschirm der Datenüberprüfungs-/Exportsoftware auf Datenanalyse. Wählen Sie R1R-Dateien überprüfen und bearbeiten > OK aus. Überprüfen Sie die Datenpunkte, löschen Sie Punkte oder fügen Sie Schritte hinzu, indem Sie die Schaltfläche Löschen/Schritt hinzufügen verwenden. Stellen Sie sicher, dass sich alle Schritte an der richtigen Stelle befinden, z. B. der Kalibrierungsschritt auf der Basislinie, der Zuordnungsschritt auf dem Höhepunkt (siehe Ergänzende Abbildung S3), und drücken Sie dann die Schaltfläche Speichern , um alle vorgenommenen Änderungen zu speichern.
- Drücken Sie auf dem Startbildschirm der Datenüberprüfungs-/Exportsoftware auf Datenanalyse. Wählen Sie Daten analysieren/plotten, wählen Sie die zu exportierenden Schritte (siehe Ergänzende Abbildung S4). Fügen Sie jeden Zuordnungsschritt (z. B. Schritt 2, Schritt 7 usw.) hinzu, nachdem Sie den Kalibrierungsschritt (z. B. Schritt 1, Schritt 6 usw.) vor dem Exportieren der Daten subtrahiert haben.
- Klicken Sie auf Diese Daten exportieren. Die Software generiert eine Tabellenkalkulationsdatei, die die I-Antwort für jeden Analytkonzentrationsdatenpunkt von jedem Transistor enthält (wenn alle Transistoren funktionieren, haben wir drei verschiedene I-Response-Werte für jeden Konzentrationspunkt). Verwenden Sie diese Tabellenkalkulationsdatei, um die Daten in der Statistiksoftware weiter zu analysieren.
- Öffnen Sie die statistische Analysesoftware. Erstellen Sie eine XY-Tabelle mit drei Y-Werten. Klicken Sie auf Erstellen. Die Daten für diese Tabelle (X-, Y-Werte) werden aus der in Schritt 6.3.8 erstellten Tabellenkalkulationsdatei kopiert. Die x-Achse entspricht der Analytkonzentration (abhängig von der verwendeten Konzentration), und die drei Y-Werte entsprechen den I-Responses, die von der vom Unternehmen gelieferten Data Review/Export-Software erhalten wurden.
- Klicken Sie auf Diese Daten analysieren. Wählen Sie XY-Analysen > Nichtlineare Regression (Kurvenanpassung) > Bindungssättigung > einer Stelle > Summe. Die Software analysiert automatisch die Daten, generiert den KD-Wert und erstellt ein Diagramm der Datenpunkte.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Ergebnisse aus Experiment 1:
Das Zielprotein Hsp90 (500 nM) wurde nach dem oben beschriebenen Zielimmobilisierungsprotokoll auf den Chip immobilisiert. Für das erste Experiment wurden 10 Konzentrationen des Analytproteins Cdc37 im Bereich von 25 nM bis 5.000 nM auf der Grundlage der in der Literatur verfügbaren Daten hergestellt (siehe Tabelle 1).
Die Schritte des Experiments können in Echtzeit überwacht werden, indem die in der I-Antwort auftretenden V...
Access restricted. Please log in or start a trial to view this content.
Diskussion
In dieser Studie wurde die Machbarkeit der Verwendung der FEB-Technologie (ein kinetischer Charakterisierungsansatz in Echtzeit) bewertet, um die biomolekulare Wechselwirkung zwischen Hsp90 und Cdc37 zu bestimmen. Das erste explorative Experiment (erstes Experiment) schlug vor, dass die Auswahl der richtigen Analytkonzentrationen ein kritischer Teil des Experiments ist und dass das Experiment durch Einbeziehung von Konzentrationspunkten über und unter dem KD-Wert entworfen werden sollte, die auf der Grundlage...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte, weder finanziell noch anderweitig.
Danksagungen
Diese Forschung wurde durch einen Zuschuss der Binational Science Foundation (BSF) an S.K.S. und N.Q. unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA | Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA | T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 | White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA | |
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 | White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA | Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 | Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA | Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA | Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA | Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA | |
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA | |
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA | |
| Statistical software | GraphPad, Prism | NA CAS number: NA | Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 | White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA | Tip 1-200 µl, in bulk, 1,000 pcs |
Referenzen
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083(2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438(2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213(2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), San Diego, Calif. 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , Springer International Publishing. 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882(2014).
- Vu, C. -A., Chen, W. -Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214(2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434(2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era...? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten