Method Article
Generierung, Hochdurchsatz-Screening und Biobanking von human-induzierten pluripotenten Stammzell-abgeleiteten Herzsphäroiden
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier wird eine Reihe von Protokollen für die Erzeugung und Kryokonservierung von kardialen Sphäroiden (CSs) aus humaninduzierten pluripotenten Stammzell-abgeleiteten Kardiomyozyten vorgestellt, die in einem multidimensionalen Hochdurchsatzformat kultiviert wurden. Dieses dreidimensionale Modell fungiert als robuste Plattform für die Modellierung von Krankheiten, Hochdurchsatz-Screenings und behält seine Funktionalität nach der Kryokonservierung bei.
Zusammenfassung
Vom Menschen induzierte pluripotente Stammzell-abgeleitete Kardiomyozyten (hiPSC-CMs) sind von größter Bedeutung für die Modellierung und Therapie menschlicher Herzerkrankungen. Wir haben kürzlich eine kostengünstige Strategie für den massiven Ausbau von hiPSC-CMs in zwei Dimensionen (2D) veröffentlicht. Zwei wesentliche Einschränkungen sind die Unreife der Zellen und die mangelnde dreidimensionale (3D) Anordnung und Skalierbarkeit in Hochdurchsatz-Screening-Plattformen (HTS). Um diese Einschränkungen zu überwinden, bilden die expandierten Kardiomyozyten eine ideale Zellquelle für die Erzeugung von 3D-Herzzellkulturen und Tissue-Engineering-Techniken. Letzteres birgt ein großes Potenzial im kardiovaskulären Bereich und bietet fortschrittlichere und physiologisch relevantere HTS. Hier beschreiben wir einen HTS-kompatiblen Workflow mit einfacher Skalierbarkeit für die Generierung, Wartung und optische Analyse von kardialen Sphäroiden (CSs) in einem 96-Well-Format. Diese kleinen CSs sind unerlässlich, um die Lücke zu schließen, die in aktuellen In-vitro-Krankheitsmodellen und/oder -generierungen für 3D-Tissue-Engineering-Plattformen vorhanden ist. Die CSs weisen eine stark strukturierte Morphologie, Größe und zelluläre Zusammensetzung auf. Darüber hinaus zeigen hiPSC-CMs, die als CSs kultiviert werden, eine erhöhte Reifung und mehrere funktionelle Merkmale des menschlichen Herzens, wie z. B. spontane Kalziumbehandlung und kontraktile Aktivität. Durch die Automatisierung des gesamten Arbeitsablaufs, von der Erstellung der CSs bis zur Funktionsanalyse, erhöhen wir die Reproduzierbarkeit innerhalb und zwischen den Chargen, wie die Hochdurchsatz-Bildgebung (HT) und die Analyse des Kalziumhandlings zeigen. Das beschriebene Protokoll ermöglicht die Modellierung von Herzerkrankungen und die Bewertung von Arzneimittel-/Therapiewirkungen auf Einzelzellebene innerhalb einer komplexen 3D-Zellumgebung in einem vollautomatischen HTS-Workflow. Darüber hinaus beschreibt die Studie ein einfaches Verfahren zur Langzeitkonservierung und zum Biobanking von ganzen Sphäroiden, das Forschern die Möglichkeit bietet, funktionelle Gewebespeicher der nächsten Generation zu schaffen. HTS wird in Kombination mit der Langzeitlagerung einen wesentlichen Beitrag zur translationalen Forschung in einer Vielzahl von Bereichen leisten, darunter die Entdeckung und Erprobung von Arzneimitteln, die regenerative Medizin und die Entwicklung personalisierter Therapien.
Einleitung
Die Entdeckung von human-induzierten pluripotenten Stammzellen (hiPS-Zellen) bot beispiellose Möglichkeiten, die menschliche Entwicklung und Krankheit auf zellulärer Ebene zu untersuchen. In den letzten zehn Jahren wurden unter Verwendung von Entwicklungslektionen verschiedene Protokolle etabliert, um die effiziente Differenzierung von hiPS-Zellen in Kardiomyozyten (CMs) sicherzustellen1,2,3,4. hiPSC-abgeleitete Kardiomyozyten (hiPSC-CMs) können als Ressource für die Modellierung genetisch vererbbarer kardiovaskulärer Erkrankungen (CVDs), die Prüfung der kardialen Sicherheit für neue Medikamente und kardiale regenerative Strategien dienen 5,6,7,8. Trotz der gezielten kardialen Differenzierung von hiPS-Zellen bleiben unbestimmte CM-Zahlen eine Herausforderung im kardialen Bereich, da gereifte hiPSC-CMs im Allgemeinen nicht proliferativ sind und primäre menschliche Zellen nicht in großen Mengen verfügbar sind.
Kürzlich haben wir beschrieben, dass die gleichzeitige Aktivierung des Wnt-Signalwegs mit einer Kultur mit niedriger Zelldichte zu einer massiven proliferativen Reaktion (bis zu 250-fach) von hiPSC-CMs 9,10 führte. Diese kostengünstige Strategie zur massiven Expansion von hiPSC-CMs durch serielles Passaging im Kulturkolbenformat erleichtert die Standardisierung und Qualitätskontrolle einer großen Anzahl funktionaler hiPSC-CMs. Um mit der Nachfrage nach großen Chargen von hiPSC-CMs von verschiedenen Spendern Schritt zu halten, wurde außerdem das Biobanking von hiPSC-CMs beschrieben10. Kardiomyozyten-Monoschichten, die in diesen Standardkulturschalen ausgesät werden, sind jedoch nicht repräsentativ für die komplexe 3D-Struktur im Herzen. Darüber hinaus ist die Unreife von hiPSC-CMs nach wie vor ein Hindernis, so dass der biologische und physiologische Phänotyp der kardiovaskulären Umgebung in vivo nicht nachgeahmt werden kann.
Es wurden neuartige 3D-In-vitro-Modelle entwickelt, bei denen hiPSC-CMs ein näheres physiologisches Verhalten zeigen, wie z. B. Selbstorganisation 11,12, Umbau der extrazellulären Matrix (EZM) 13, verbesserte Reifung 14,15,16 und synchronisierte Kontraktion17,18,19 . 3D-Modelle wurden für die Arzneimittelforschung, Kardiotoxizitätstests von Arzneimitteln, die Modellierung von Krankheiten, regenerative Therapien und sogar die ersten klinischen Studien verwendet 20,21,22,23,24. Eines der am häufigsten verwendeten Modelle ist das Fibrin-basierte technische Herzgewebe (EHT), das eine gewebeähnliche Anordnung und kardiale Kontraktilität aufweist13,17,25. Zuvor haben wir gezeigt, dass EHTs, die aus expandierten hiPSC-CMs generiert wurden, eine vergleichbare Kontraktilität aufwiesen wie solche aus nicht expandierten hiPSC-CMs, was eine unbeeinträchtigte zelluläre Funktionalität nach der Expansion9 zeigte. Obwohl die Generierung von EHTs aus hiPSC-CMs gut etabliert ist, werden weitere Entwicklungen im Hinblick auf die Etablierung einer HT-Bewertungsplattform erwartet. Hier ermöglicht die schnelle Erzeugung einer großen Anzahl von selbstaggregierenden kardialen Sphäroiden (CSs) im 96-Well-Format eine Verbesserung der 3D-Bedingungen für Hochdurchsatz-Screening-Zwecke (HTS).
Insgesamt liegt der Vorteil von CSs als 3D-Zellkultur in ihrer hohen Reproduzierbarkeit und Skalierbarkeit. Insbesondere können CSs in Kombination mit der robotergestützten Probenhandhabung die CS-Kultur, die Arzneimittelbehandlung und die High-Content-Analyse standardisieren und automatisieren20. Hier beschreiben wir optimierte Protokolle zur Erzeugung hochreiner und qualitativ hochwertiger CSs, die effizient kryokonserviert und auf Herzfunktion untersucht werden können, indem Ca2+ transiente Messungen mit einem optischen Kalziumerfassungs- und -analysesystem durchgeführt werden. Dieses Modell bietet ein einfaches, aber leistungsstarkes Werkzeug zur Durchführung von Hochdurchsatz-Sieben auf Hunderten bis Tausenden von Sphäroiden17,18.
Protokoll
HINWEIS: Die in dieser Studie verwendeten hiPSC-CMs wurden gemäß den zuvor beschriebenen hiPSC-Kultivierungs- und CM-Differenzierungsprotokollen26,27 generiert. Optional können die hiPSC-CMs erweitert und kryokonserviert werden, wie kürzlich veröffentlicht, bevor das CS-Protokoll gestartet wird (Abschnitt 4)10.
1. Herstellung von Zellkulturmedien, Lösungen und Aliquoten
- Bereiten Sie das Basalmedium vor
- Penicillin-Streptomycin und das Medium (RPMI 1640) auf Raumtemperatur (RT) ausbalancieren und sicherstellen, dass es vollständig aufgetaut ist. Mischen Sie 500 ml des Mediums und 5 ml Pen/Streptokokken. Bei 4 °C bis zu 8 Wochen lagern; Vor Gebrauch auf 37 °C äquilibrieren.
- RPMI + B27 vorbereiten
- Gleichen Sie das B27-Präparat und das Basalmedium zu RT aus. Stellen Sie sicher, dass das Präparat vollständig aufgetaut ist. Mischen Sie 490 ml des Basalmediums und 10 ml des 50x B27-Supplements. Bei 4 °C bis zu 2 Wochen lagern; Vor Gebrauch auf 37 °C äquilibrieren.
- Vorbereiten des hiPSC-CM-Beschichtungsmediums
- Fügen Sie Rho-assoziierten, Coiled-Coil-haltigen Proteinkinase (ROCK)-Inhibitor (2 μM Endkonzentration) und 10% Knockout-Serumersatz (KSR) zu RPMI + B27-Medien hinzu. Fügen Sie den ROCK-Inhibitor bei Bedarf direkt zu den RM-Medien hinzu. Bewahren Sie keine Nährmedien auf, sobald sie ergänzt wurden.
- CM-Auftaumedien vorbereiten
- Fügen Sie eine Konzentration von 1:100 des Zellüberlebenspräparats (z. B. Revitacell) und 20 % KSR zu RPMI + B27-Medien hinzu und äquilibrieren Sie es vor Gebrauch auf 37 °C.
- Bereiten Sie die Reifungsergänzung vor
- Das zuvor beschriebene ReifemediumFormel 28 besteht aus: 3 mM Glucose, 10 mM L-Lactat, 5 mg/mL Vitamin B12, 0,82 mM Biotin, 5 mM Kreatin-Monohydrat, 2 mM Taurin, 2 mM L-Carnitin, 0,5 mM Ascorbinsäure, 1x NEAA, 0,5% (w/v) albumax, 1x B27 und 1% KOSR. Um eine volle Flasche (500 ml) Reifungspräparat zuzubereiten, entfernen Sie 65 ml aus einer Flasche DMEM ohne Glukose und ergänzen Sie es mit 2,7 g Glukose, 5,6 g L-Laktat, 0,025 mg Vitamin B12, 1 mg Biotin, 3,73 g Kreatinmonohydrat, 1,25 g Taurin, 1,975 g L-Carnitin, 0,7125 g Ascorbinsäure, 50 ml NEAA, 12,5 g Albumax und 5 ml Penicillin-Streptomycin.
- Filtern Sie durch eine sterile Einwegfiltereinheit mit einer 0,22-μm-Porenmembran aus Polyethersulfon (PES).
- Aliquot in 45 ml (zur Herstellung von 500 ml Reifungsmedium) oder 4,5 ml (zur Herstellung von 50 ml Reifungsmedium). Bei 20 °C bis zu 6 Monate lagern.
- Reifungsmedien vorbereiten
- Gleichen Sie das B27-Präparat, Knockout-SR, Penicillin-Streptomycin, das Reifungspräparat28 und das DMEM-No-Glucose-Medium bei RT aus. Stellen Sie sicher, dass das Präparat vollständig aufgetaut ist. Mischen Sie 435 ml des DMEM-No-Glucose-Mediums mit 10 ml des 50x B27-Präparats, 5 ml Penicillin-Streptomycin, 5 ml Knockout-SR und 45 ml Reifungspräparat. Bei 4 °C bis zu 2 Wochen lagern; Vor Gebrauch bei 37 °C äquilibrieren.
- Bereiten Sie fluorhelles Medium vor
- Penicillin-Streptomycin und DMEM-Fluorobrit-Medium bei RT ausbalancieren. Stellen Sie sicher, dass das Präparat vollständig aufgetaut ist. Mischen Sie 500 ml des DMEM-Fluorbrit-Mediums mit 5 ml Penicillin-Streptomycin. Bei 4 °C bis zu 1 Monat lagern; Vor Gebrauch bei 37 °C äquilibrieren.
- Bereiten Sie die nichtionische Detergenzienlösung vor
- Mischen Sie 20% w/v nichtionisches Waschmittelpulver (z. B. F-127) mit PBS. Mit einem 0,22-μm-Filter filtrieren und bis zu 6 Monate bei 4 °C lagern; vor Gebrauch bei RT äquilibrieren.
- Bereiten Sie Calciumfarbstoff vor
- Mischen Sie die nichtionische Detergenzienlösung (Endkonzentration von 0,04 % v/v) und 0,1x des Calciumfarbstoffs (z. B. Cal520 AM) in fluorhellem Medium. In einem konischen 50-ml-Röhrchen werden 10 μl Cal520 und 20 μl der nichtionischen Waschmittellösung zugegeben. Mischen, bis es vollständig aufgelöst ist. Halten Sie die Lösung im Dunkeln, bevor Sie sie zu den Zellen hinzufügen.
2. Vorbereitung von Puffern
- Permeabilisierungs- und Blockierungspuffer vorbereiten: Dieser Puffer enthält 10 ml PBS, 5 Gew.-% BSA und 0,3 % v/v Triton-X-100.
- Bereiten Sie den Durchflusszytometriepuffer vor: Dieser Puffer enthält 50 ml PBS, 1 Gew.-% BSA und 0,3 % v/v Triton-X-100.
- Durchflusszytometrie-Waschpuffer: Dieser Puffer enthält 50 ml PBS und 1 Gew.-% BSA.
- Sphäroidwaschpuffer (SWB): Dieser Puffer enthält 1 ml Triton-X-100, 2 ml 10% (w/v in DPBS) SDS und 2 g BSA in 1 l PBS.
HINWEIS: SWB kann bis zu 2 Wochen bei 4 °C gelagert werden. - Bereiten Sie die Einbettungslösung (ES) vor: Um 100 ml der Einbettungslösung herzustellen, mischen Sie 50 ml Glycerin mit 9,09 ml dH2O, 1 ml Tris-Puffer (1 M, pH 8,0) und 200 μl EDTA (0,5 M). Fügen Sie 22,7 g Fructose hinzu und mischen Sie bei RT im Dunkeln, bis es sich aufgelöst hat. Wenn es klar ist, fügen Sie 22,2 g Fructose hinzu und mischen Sie, bis es sich aufgelöst hat. Dann 15 g Harnstoff zugeben und bis er sich aufgelöst hat (bei 4 °C im Dunkeln lagern).
- Bereiten Sie den PBT-Puffer (PBS mit Tween-20) vor. Dieser Puffer enthält PBS/Tween-20 (0,1 % v/v). Fügen Sie für 1 l PBS 1 ml Tween-20 hinzu.
3. Herstellung kleiner Moleküle
- Rekonstituieren Sie Thiazovivin-Pulver (ROCK-Inhibitor) in 10 mM Aliquots von 50 μl in DMSO und lagern Sie es bis zu 6 Monate bei -20 °C. Vor Licht schützen.
- Bereiten Sie 2,5 mM Aliquote von je 10 μl Cal-520 AM in DMSO vor und lagern Sie sie bei -20 °C für bis zu 6 Monate. Vor Licht schützen.
4. Kardiale Sphäroiderzeugung
HINWEIS: Für größere Mengen an CSs säen Sie bis zu 1 Million CMs in eine 6-Well-Platte mit extrem niedrigem Aufsatz und 2 ml hiPSC-CM-Nachplattiermedium. In dieser Studie wurden mindestens 2.500 (2,5 k CS) bis 20.000 (20 k CS) hiPSC-CMs pro Well einer 96-Well-Platte verwendet.
- Bereiten Sie für eine 96-Well-Platte eine Zellkultur vor, die mindestens 2 x 106 vom Menschen induzierte pluripotente Stammzell-abgeleitete Kardiomyozyten (hiPSC-CMs)10 enthält.
- Wenn die kultivierten hiPSC-CMs die Konfluenz erreichen, geben Sie 0,1 ml/cm2 sterile Herzablösungslösung (z. B. Tryple) in jede Vertiefung. Die Platte wird bei 37 °C 15 min inkubiert.
- Trennen Sie die Zellen mit einer 5-ml-Pipette mechanisch, indem Sie sie mit 2 ml warmem Basalmedium spülen, um eine Einzelzellsuspension herzustellen. Bestätigen Sie die Ablösung mit einem Hellfeldmikroskop (4-fache Vergrößerung); Die Zellen sehen weiß aus und haben eine runde Form.
- Die Zellsuspension wird in ein konisches 15-ml-Röhrchen überführt und 3 Minuten lang bei 300 x g zentrifugiert.
- Aspirieren Sie den Überstand und resuspendieren Sie die Zellen in 1 ml hiPSC-CM-Beschichtungsmedium.
- Mit einer 1.000-μl-Pipettenspitze das Zellpellet mechanisch dissoziieren. Die Lösung erscheint nach drei oder vier Mischungen homogen. Zählen Sie die Zellen. Übertragen Sie die entsprechende Anzahl von Zellen in 100 μl des Beschichtungsmediums in jede 96-Well-Vertiefung mit extrem niedrigem Aufsatz.
- Legen Sie die CS-Platte für 24 Stunden bei 70 U/min auf einen Orbitalschüttler im Inkubator. Stellen Sie die Inkubatorbedingungen auf 37 °C, 5 % CO 2, 21 % O2 und 90 % Luftfeuchtigkeit ein.
- Saugen Sie 50 μl Medium aus jeder Vertiefung ab und fügen Sie 100 μl RPMI + B27 Medium pro Vertiefung für die ersten 48 Stunden hinzu.
HINWEIS: Bewahren Sie immer 50 μl des Mediums in der 96-Well-Platte auf, um eine versehentliche Aspiration und einen Sphäroidbruch zu vermeiden. - Saugen Sie 100 μl des Mediums aus jeder Vertiefung ab und fügen Sie 100 μl des Reifemediums pro Vertiefung hinzu. Halten Sie die Zellen im Reifemedium und erfrischen Sie das Medium alle 2-3 Tage.
5. Kryokonservierung von CSs
HINWEIS: CSs können für die Langzeitlagerung kryokonserviert werden. Die Kryokonservierung kann ab Tag 3 nach der Generierung von CSs durchgeführt werden. CSs können direkt in den Vertiefungen einer 96-Well-Platte oder als CS-Suspension in Kryozellen kryokonserviert werden.
- Kühlen Sie den Teller vor, indem Sie ihn 10 Minuten lang auf Eis legen.
- Zentrifugieren Sie die Sphäroidplatte für 3 min bei 70 x g.
- Entfernen Sie den Überstand, bis 50 μl übrig geblieben sind, und fügen Sie 200 μl eiskaltes hiPSC-Gefriermedium pro Vertiefung hinzu.
HINWEIS: Bewahren Sie die CS-Suspension während der gesamten Dauer des Eingriffs auf Eis auf. Im Falle einer 6-Well-Platte mit Sphäroiden wird eine Vertiefung in einem 500-μl-Gefriermedium eingefroren. - Versiegeln Sie die Platte mit Plattendichtungsfolie.
HINWEIS: Die 96-Well-Platte muss in einer Styroporbox aufbewahrt werden, oder, falls nicht verfügbar, kann eine Silikonform hergestellt werden, wie in Schritt 5.5.1 beschrieben. - Um einen gleichmäßigen Wärmeaustausch zwischen der Wellplatte und dem Gefrierschrank zu gewährleisten, legen Sie die Platte vorsichtig in eine Styroporbox oder in eine Silikonform.
- So bereiten Sie die Silikonform vor: Mischen Sie zwei Komponenten des Siliziumelastomer-Kits kräftig im Verhältnis 10:1. Entblasen Sie die Lösung mit einer Vakuumpumpe für 15-20 Minuten. Anschließend wird die Lösung in den unteren Teil der Vertiefungsplatte gegossen und mit einer Vakuumpumpe 10 Minuten lang entblasen. Die Form wird in einen Ofen gestellt und bei 60 °C 8 h lang ausgehärtet, um ein halbflexibles Elastomer zu erhalten, das von der Platte abgezogen wird.
- Frieren Sie die Platte bei -80 °C für mindestens 4 h in der Styroporbox oder der vorbereiteten Silikonform ein.
- Übertragen Sie die Platte zur Langzeitlagerung in einen Flüssigstickstofftank oder einen Gefrierschrank mit -150 °C.
6. Auftauen von Herzsphäroiden
HINWEIS: Tauen Sie nicht mehr als eine Platte gleichzeitig auf, um einen schnellen Auftauvorgang zu gewährleisten.
- Bereiten Sie 20 ml 37 °C vorgewärmtes Basalmedium in einem konischen 50-ml-Röhrchen vor.
- Sammeln Sie die Zellplatte mit CSs aus dem flüssigen Stickstoff und legen Sie sie für 15 Minuten in den Inkubator. Stellen Sie die Inkubatorbedingungen auf 37 °C, 5 % CO 2, 21 % O2 und 90 % Luftfeuchtigkeit ein.
- Entfernen Sie den Überstand und die Zellpelletreste und resuspendieren Sie jede Vertiefung in einem warmen basalen Medium. Verwenden Sie 200 μl Medium pro Vertiefung.
- 3 min bei 70 x g zentrifugieren.
- Wiederholen Sie die Schritte 6.3 und 6.4.
- Entfernen Sie den Überstand, bis das Zellpellet übrig bleibt, und geben Sie 200 μl CM-Auftaumedium in jede Vertiefung.
- Legen Sie die CS-Platte für 24 Stunden bei 70 U / min auf einen Orbitalschüttler in einem Inkubator. Stellen Sie die Inkubatorbedingungen auf 37 °C, 5 % CO 2, 21 % O2 und 90 % Luftfeuchtigkeit ein.
- Saugen Sie 50 μl des Mediums aus jeder Vertiefung ab und fügen Sie 100 μl RPMI + B27 Medium pro Vertiefung für die ersten 48 Stunden hinzu.
- Saugen Sie 100 μl des Mediums aus jeder Vertiefung ab und fügen Sie 100 μl Reifemedium pro Vertiefung hinzu. Halten Sie die Zellen im Reifemedium und erfrischen Sie das Medium alle 2-3 Tage.
7. Beurteilung intrazellulärerCa2+ -Transienten
HINWEIS: CSs sind insgesamt 3 Wochen in Kultur; 2 Wochen vor dem Einfrieren und 1 Woche nach dem Auftauen. Die "frischen" Kontrollen sind altersgerecht.
- Nach 1 Woche Kultur sind die aufgetauten CSs optimal für die optische Bildgebung im Umgang mit Kalzium. Verwenden Sie einen Calciumfarbstoff (z. B. Cal520AM), um die Aufnahme und Freisetzung von Ca2+ aus den Zellen zu beurteilen.
- Behandeln Sie sie mit 100 μl Calciumfarbstoff pro Vertiefung und inkubieren Sie sie 60 Minuten lang im Inkubator. Stellen Sie die Inkubatorbedingungen auf 37 °C, 5 % CO 2, 21 % O2 und 90 % Luftfeuchtigkeit ein.
HINWEIS: Cal520AM ist lichtempfindlich. Führen Sie alle Ladevorgänge und Experimente im Dunkeln durch. - Bereiten Sie das Kalziumerfassungs- und Analysesystem vor.
- Schalten Sie das Mikroskop ein und stellen Sie sicher, dass die Umgebungskontrolloption aktiviert ist.
- Passen Sie die Abmessungen der Kamera und der Blende an, um den Hintergrundbereich zu minimieren.
HINWEIS: Hier kam das Mikroskop Leica Thunder DMi8 zum Einsatz; Andere Mikroskopsysteme sind ebenfalls anwendbar, bis sie eine Abtastrate von über 30 Bildern/Sekunde (fps) ermöglichen.
- Nehmen Sie ein Video mit einem konsistenten Strom von 2-10 Peaks innerhalb von 10 s auf und scannen Sie über die 96-Well-Platte, wobei Sie sich zunächst nach links und dann im Zickzack nach unten bewegen, um die gesamte Platte abzudecken. Messen Sie das Kalziumsignal mit einem 488-nm-Laser; Stellen Sie den Kontrast während der Kalziumfreisetzung auf einen schwarzen Hintergrund mit einem hellgrünen Signal ein.
- Analysieren Sie nach der Erfassung der Ca2+ -Transienten die Daten mit der Fluoreszenzspurenanalyse-Software (z. B. CyteSeer, Vala Sciences) gemäß den Anweisungen des Herstellers.
8. Durchflusszytometrische Analysen von dissoziierten kardialen Sphäroiden
HINWEIS: In dieser Studie wurde die Durchflusszytometrie verwendet, um die Lebensfähigkeit der CSs vor und nach dem Auftauprozess zu bestimmen.
- Sammeln Sie die CSs in einem konischen 15-ml-Röhrchen mit einer 5-ml-Pipette, um eine Beschädigung des Sphäroids zu vermeiden, und zentrifugieren Sie sie 3 Minuten lang bei 70 x g. Saugen Sie den Überstand ab und fügen Sie 1 ml PBS hinzu.
- 3 min bei 200 x g zentrifugieren. Aspirieren Sie den Überstand und dissoziieren Sie die CSs durch Zugabe von 1 ml Herzablösungslösung (z. B. Tryple). Inkubieren Sie das Röhrchen bei 37 °C für 15 min.
- Dissoziieren Sie die Zellen mit einer 5-ml-Pipette mechanisch, indem Sie sie mit 2 ml Basalmedium spülen, bis einzelne Zellen unter dem Mikroskop zu sehen sind.
- 3 min bei 200 x g zentrifugieren.
- Saugen Sie den Überstand ab und fixieren Sie die CMs mit 200 μl 4%iger Paraformaldehyd (PFA)-Lösung in 1x PBS. 10 Minuten bei RT inkubieren.
- 3 min bei 200 x g zentrifugieren. Saugen Sie den Überstand ab und fügen Sie 1 ml PBS hinzu.
HINWEIS: Pausenpunkt: Die fixierten hiPSC-CMs können bis zu 4 Wochen bei 4 °C gelagert werden. - Die Zellsuspension wird in ein FACS-Röhrchen überführt und 3 min bei 200 x g zentrifugiert. Der Überstand wird abgesaugt und 1 x 105 Zellen in 50 μl des Permeabilisierungspuffers resuspendiert.
- Die Zellen werden 30 Minuten bei 4 °C inkubiert.
- Führen Sie für die Analyse der Immunfluoreszenz-Durchflusszytometrie die Schritte 8.9.1 bis 8.9.4 aus.
- Resuspendieren Sie die Zellen im Durchflusszytometriepuffer (50 μl), der den α-Actinin-Antikörper in einer Verdünnung von 1:300 enthält. In einem anderen FACS-Röhrchen werden 1 x 105 Zellen im Durchflusszytometriepuffer (50 μL) mit der entsprechenden Isotypkontrolle (z. B. FITC-Maus-IgM, κ-Isotyp [1:200-Verdünnung]) resuspendiert. In ähnlicher Weise resuspendieren 1 x 105 Zellen in 50 μl Durchflusszytometriepuffer zur Negativkontrolle.
- Die Zellen werden 30 Minuten bei 4 °C inkubiert.
- 2,5 ml Durchflusszytometriepuffer zugeben und die Zellen bei 200 x g 3 min bei 4 °C zentrifugieren; Entsorgen Sie den Überstand und wiederholen Sie diesen Waschschritt zweimal.
- Resuspendieren Sie die Zellen in 50 μl Durchflusszytometriepuffer mit dem Sekundärantikörper Ziegen-Anti-Maus (1:300 Verdünnung).
HINWEIS: Stellen Sie das Röhrchen in die Dunkelheit, da die Sekundärantikörperlösung lichtempfindlich ist.
- Zur Überprüfung der Viabilität mit Propidiumiodid (PI) 150 μl PI pro Probe (1:1.000) zugeben und 15 Minuten inkubieren lassen.
HINWEIS: Stellen Sie das Röhrchen in die Dunkelheit, da die PI-Lösung lichtempfindlich ist. - Stellen Sie die Gates gemäß der Standard-Gating-Strategie ein, wie in der ergänzenden Abbildung 1 gezeigt, und analysieren Sie die Zellen mit einem Durchflusszytometer.
9. Immunfluoreszenzfärbung ganzer 3D-Sphäroide
HINWEIS: Dieses Protokoll basiert auf dem Protokoll für die hochauflösende 3D-Bildgebung ganzer Organoide bei Immunfluoreszenzmarkierung, das zuvorveröffentlicht wurde 29 und für kardiale Sphäroide angepasst wurde. Während des Eingriffs können alle Pipettenspitzen und konischen Röhrchen mit 1 Gew.-% BSA-PBS beschichtet werden, um ein Anhaften der Sphäroide an Kunststoffen zu verhindern. Um die Materialien zu beschichten, tauchen Sie in das 1% BSA-PBS ein. Achten Sie darauf, die Sphäroide nicht zu beschädigen, indem Sie die 5-ml-Pipette verwenden, um mechanische Störungen zu vermeiden.
- Sammeln Sie die CSs in einem 15-ml-beschichteten Röhrchen mit einer 5-ml-Pipette. Die Sphäroide sind für das Auge sichtbar. Sammeln Sie ~20-50 Sphäroide pro Antikörperkombination. 3 min bei 70 x g zentrifugieren und den Überstand absaugen.
- Resuspendieren Sie die Sphäroide vorsichtig in 1 ml eiskalter 4%iger Paraformaldehyd (PFA)-Lösung in 1x PBS mit einer beschichteten 1-ml-Spitze.
- 45 Min. bei 4 °C fixieren. Nach 20 Minuten resuspendieren Sie die Sphäroide vorsichtig mit einer beschichteten 1-ml-Spitze. Dies gleicht die Fixierung zwischen allen Sphäroiden aus.
- Geben Sie 10 ml eiskaltes PBS in das Röhrchen und mischen Sie es vorsichtig, indem Sie das Röhrchen umdrehen. 10 min bei 4 °C inkubieren und 3 min bei 70 x g herunterschleudern.
HINWEIS: Ab diesem Schritt ist eine Beschichtung von Spitzen und konischen Röhrchen in der Regel nicht erforderlich, da CSs nach der Fixierung nicht an der Spitze haften bleiben. - Blockieren Sie die CSs, indem Sie das Pellet in eiskaltem SWB (200 μl SWB pro Well) resuspendieren und die Sphäroide auf eine 24-Well-Suspensionskulturplatte übertragen.
HINWEIS: CSs aus einem großen Pellet können auf mehrere Vertiefungen aufgeteilt werden, um unterschiedliche Färbungen durchzuführen. Verwenden Sie ~20-50 CSs pro Antikörperkombination. - Bei 4 °C mindestens 15 min inkubieren.
- Geben Sie 200 μl des SWB in eine leere Vertiefung, um als Referenzvertiefung zu dienen.
HINWEIS: Für die Immunfluoreszenzfärbung können auch 48- oder 96-Well-Platten verwendet werden, um den Antikörperverbrauch zu reduzieren. Die Färbe- und Waschergebnisse können jedoch aufgrund des geringeren Volumens pro Vertiefung reduziert werden. - Lassen Sie die Sphäroide am Boden der Platte absetzen, indem Sie die Platte 5 Minuten lang in einem Winkel von 45° geneigt lassen.
- Entfernen Sie die SWB und belassen Sie die CSs in 200 μl der SWB (verwenden Sie die Referenzvertiefung, um das minimale Volumen von 200 μl abzuschätzen).
- 200 μl des SWB mit den Primärantikörpern 2x konzentriert (z. B. ɑ-Actinin [1:200] und Troponin T [1:200]) zugeben und über Nacht bei 4 °C unter Schaukeln/Schütteln (40 U/min auf einem horizontalen Shaker) inkubieren.
- Geben Sie am nächsten Tag 1 ml SWB in jede Vertiefung.
- Lassen Sie die Sphäroide am Boden der Platte absetzen, indem Sie die Platte 5 Minuten lang in einem Winkel von 45° stehen lassen.
- Entfernen Sie die SWB und lassen Sie 200 μl in der Platte. Fügen Sie 1 ml SWB hinzu und waschen Sie es 2 Stunden lang unter langsamem Schaukeln/Schütteln.
- Wiederholen Sie die Schritte 9.12 und 9.13 noch zweimal.
- Lassen Sie die CSs an der Unterseite der Platte absetzen, indem Sie die Platte 5 Minuten lang um 45° geneigt lassen. Entfernen Sie die SWB und lassen Sie 200 μl in jeder Vertiefung
- 200 μl des SWB mit sekundären Antikörpern, konjugierten Antikörpern und Farbstoffen 2x konzentriert (z. B. DAPI [1 μg/ml], Maus-AF488 [1:500], Kaninchen-AF568 [1:500]) zugeben und über Nacht bei 4 °C im Dunkeln inkubieren, dabei langsam schaukeln/schütteln.
- Wiederholen Sie die Schritte 9.12 und 9.13 am nächsten Tag noch zweimal.
- Übertragen Sie die CSs vorsichtig in ein 1,5-ml-Röhrchen und schleudern Sie sie 3 Minuten lang bei 70 x g .
- Entfernen Sie so viel SWB wie möglich durch Pipettieren, ohne die CSs zu stören.
- Fügen Sie die Einbettungslösung (ES; mindestens 50 μl, bei RT) mit einer 200-μl-Spitze hinzu, wobei das Ende abgeschnitten ist, und resuspendieren Sie sie vorsichtig, um eine Blasenbildung zu verhindern, und inkubieren Sie sie bei RT für 20 Minuten.
- Erstellen Sie in der Zwischenzeit einen quadratischen Behälter auf einem Objektträger mit Nagellack oder Silikondichtmittel.
- Schneiden Sie das Ende einer 200-μl-Spitze ab und übertragen Sie die CSs in ES in die Mitte des quadratischen Behälters.
- Legen Sie ein quadratisches Deckglas darauf. Um Luftblasen zu reduzieren, legen Sie zuerst eine Seite des Deckglases auf, senken Sie dann das Deckglas langsam von einer Seite zur anderen, bis sich keine Luft mehr unter der Oberfläche befindet, und lassen Sie dann das Deckglas los.
- Drücken Sie vorsichtig auf alle Kanten des Deckglases, um es mit dem Nagellack oder Silikonversiegelungsmittel zu versiegeln.
- Lassen Sie die Folie über Nacht bei RT. Am nächsten Tag ist die Folie bereit für die Bildgebung.
HINWEIS: Eine optische Reinigung durch den ES kann zu einer geringfügigen Gewebeschrumpfung führen. Dies kann jedoch keinen Einfluss auf die Gesamtmorphologie der CSs haben. Der Färbevorgang kann hier pausiert werden, indem die Objektträger bei 4 °C (für mindestens 1 Woche) oder bei -20 °C (für mindestens 6 Monate) gelagert werden.
Ergebnisse
Das in Abbildung 1A gezeigte Protokoll beschreibt die Generierung von CSs aus zuvor erweiterten hiPSC-CMs. Die CSs erhalten am Tag 1 nach der Aussaat eine 3D-Struktur in Rundbodenplatten mit extrem niedriger Befestigung und können bis zu 6 Wochen lang kultiviert werden (Abbildung 1B). Wie durch Immunfluoreszenzfärbung beurteilt, exprimierte die Mehrheit der Zellen in 3 Wochen alten CSs sarkomere Proteine wie α-Actinin und Troponin T und zeigte eine regelmäßige Sarkomerorganisation (Abbildung 1C). Für die Quantifizierung von α-Actinin-positiven Zellen wurde eine Durchflusszytometrie-Analyse durchgeführt. In Übereinstimmung mit den Ergebnissen der Immunfluoreszenz zeigten die Durchflusszytometriedaten vergleichbar hohe α-Actinin-Spiegel sowohl an Tag 0 (76,9 % ± 16,6 %) als auch bei 3 Wochen alten CSs (71,1 % ± 22,7 %) (Abbildung 1D), was auf eine konstante und hochreine Zellzusammensetzung während der Kultivierung hinweist. Es gab eine erhöhte Expression der kardialen Gene für Verbindungen (GJA1, JPH2 und PKP2), Desmosomen (DES) und Mitochondrien (ATP5A) in hiPSC-CM-abgeleiteten Sphäroiden (Tag 42) im Vergleich zu hiPSC-CMs, die 90 Tage lang in 2D kultiviert wurden (Abbildung 1E). Die Expression dieser Gene ist ein Kennzeichen der Zell-Zell-Interaktion und -Reifung30.
Anschließend wurden die funktionellen Eigenschaften von CSs, nämlich die Schlagrate und das Ca2+ Handling, zu verschiedenen Zeitpunkten bewertet (Abbildung 2). Calcium-Transientenparameter wie Anstiegszeit, Spitzenzeit, Abklingzeit und Calcium-Transientendauer (CTD90) wurden wie in Abbildung 2A, B dargestellt bewertet. Der Prozentsatz der schlagenden CSs ist in den ersten 3 Wochen nach der Generierung ähnlich, fiel jedoch in Woche 6 (Wk6) CSs signifikant ab (Abbildung 2C). Die Schlagrate war bei Wk3 im Vergleich zu Wk1 signifikant reduziert und fiel, ähnlich wie der Prozentsatz der schlagenden CSs, bei Wk6 dramatisch ab (Abbildung 2D). Bei Wk6 wurde eine Verschlechterung der CS beobachtet, was den Rückgang sowohl der Schlagrate als auch der Anzahl der schlagenden CSs erklären kann. Die Messung der transienten Calciumparameter zeigte einen signifikant höheren Spitzenwert bei Wk2 (Abbildung 2E), während die Anstiegszeit, die Abklingzeit und CTD90 bei Wk3 im Vergleich zu Wk1 signifikant erhöht waren (Abbildung 2F-H ). Zusammengenommen zeigen diese Ergebnisse, dass hiPSC-CM-abgeleitete Sphäroide etwa 2 und 3 Wochen nach der Generierung funktionell optimal sind.
Abbildung 3 zeigt den Einfluss der Sphäroidgröße auf die Schlagrate und den Umgang mit Kalzium. CSs wurden durch Aussaat von 2,5 x 10 4, 5 x 10 4, 10 x 10 4 und 20 x 10 4 hiPSC-CMs in einer Vertiefung einer 96-Well-Platte für insgesamt 24 CSs/Vertiefungen pro Bedingung erzeugt (Abbildung 3A). Wie erwartet, nahm die Sphäroidgröße mit zunehmender Anzahl der verwendeten Zellen zu und reichte von 178 ± 36 μm bis 351 ± 65 μm (Abbildung 3A, rechtes Bild). Ca2+ Transienten wurden in 3 Wochen alten CSs bei den vier verschiedenen Seedingdichten gemessen (Abbildung 3B). Messungen der schlagenden CSs zeigten, dass nur etwa 50% der kleineren Größen-CSs (2,5K- und 5K-CSs) schlugen, während der Prozentsatz der größeren CSs (10K- und 20K-CSs) signifikant höher war (etwa 85%) (Abbildung 3C). Eine ähnliche Schlagrate (ca. 28 bpm) wurde von 5K-, 10K- und 20K-CSs gezeigt, die im Vergleich zu 2,5K-CSs signifikant höher war (Abbildung 3D). Die Spitzenwerte der Kalziumbilder waren unter allen getesteten Bedingungen ähnlich (Abbildung 3E), jedoch waren die Anstiegszeit (Abbildung 3F), die Abklingzeit (Abbildung 3G) und CTD90 (Abbildung 3H) bei größeren CSs (10K- und 20K-CSs) im Vergleich zu den kleineren (2,5K- und 5K-CSs) signifikant erhöht. Zusammengenommen zeigen diese Ergebnisse, dass hiPSC-CM-abgeleitete Sphäroide optimal für das Calcium-Handling-Screening sind, wenn eine Seeding-Dichte zwischen 10K- und 20K-hiPSC-CMs/Well verwendet wird.
Als nächstes bewerteten wir die Auswirkungen der Kryokonservierung auf die Lebensfähigkeit und Funktion von CS. Vor der Analyse wurden aufgetaute CSs 1 Woche lang in Kultur gehalten (Abbildung 4A). Wie sowohl die Durchflusszytometrie (Abbildung 4B) als auch die Calcein-AM (Abbildung 4C) Zellviabilitätstests zeigten, hatte die Kryokonservierung keinen Einfluss auf die Zellviabilität innerhalb der CSs. Darüber hinaus zeigten aufgetaute CSs ähnliche Expressionsniveaus von sarkomeren Proteinen im Vergleich zu den frischen, altersangepassten CSs (Abbildung 4D). Diese Daten deuten darauf hin, dass CSs für die anschließende Herzfunktionsanalyse und das Hochdurchsatz-Screening effizient kryokonserviert werden können.
Schließlich wurden die Schlagaktivität und dasCa2+-Handling sowohl in frischen als auch in kryokonservierten CSs gemessen (Abbildung 5). Der Prozentsatz der schlagenden CSs wurde zu verschiedenen Zeitpunkten nach dem Auftauen nach 2, 5 bzw. 7 Tagen gemessen. Während die meisten frischen CSs im Laufe der Zeit eine schlagende Aktivität zeigten, benötigten die kryokonservierten CSs eindeutig bis zu 1 Woche Kultivierung, um ihre schlagende Aktivität wiederherzustellen (Abbildung 5B). Es gab keine signifikante Veränderung in der Schlagrate von aufgetauten CSs im Vergleich zu frischen; Bei einigen eingefrorenen CSs wurde jedoch keine spontane Schlagaktivität beobachtet (Abbildung 5C). Obwohl die Spitzenwerte in gefrorenen/aufgetauten CSs im Vergleich zu frischen signifikant reduziert waren (Abbildung 5D), wurden keine signifikanten Veränderungen in der Anstiegszeit, der Abklingzeit und dem CTD90 von gefrorenen/aufgetauten CSs im Vergleich zu frischen (Abbildung 5E-G) beobachtet. Diese Daten deuten darauf hin, dass es wichtig ist, die CSs nach dem Auftauen mindestens 1 Woche im Inkubator erholen zu lassen, bevor die Schlagaktivität und der vorübergehende Ca2+ gemessen werden.
Zusammengenommen zeigen diese Ergebnisse, dass die Kryokonservierung von hiPSC-CM-abgeleiteten Sphäroiden die Lebensfähigkeit der Kardiomyozyten, die sarkomere Struktur und ihre funktionellen Eigenschaften wie spontane Schlagaktivität und Kalziumhandhabung bewahrt. Daher stellen hiPSC-CM-abgeleitete Sphäroide ein geeignetes Modell dar, um die kardiale Elektrophysiologie in vitro genau zu rekapitulieren.
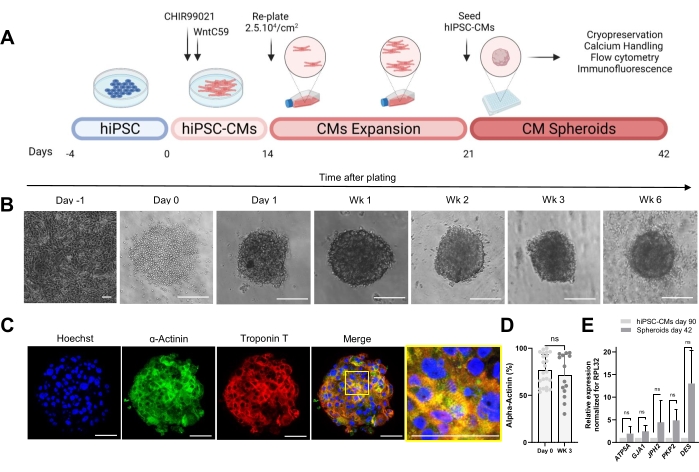
Abbildung 1: Erzeugung von kardialen Sphäroiden . (A) Schematische Darstellung der Wnt-basierten gerichteten kardialen Differenzierung, der anschließenden Expansion von hiPSC-CMs und der Generierung von CSs. Erstellt mit biorender.com. (B) Hellfeldbilder zu verschiedenen Zeitpunkten der CS-Kultivierung. Maßstabsleiste, 200 μm. Wk steht für Woche. (C) Repräsentative Immunfluoreszenzbilder für kardiale Sarkomerproteine α-Actinin und Troponin T in 3 Wochen alten CSs. Immunfluoreszenz: Hoechst (blau), α-Actinin (grün) und Troponin T (rot). Maßstabsleiste, 200 μm. Das vergrößerte zusammengeführte Bild auf der rechten Seite zeigt die Sarkomer-Organisation. Maßstabsbalken, 50 μm. (D) Durchflusszytometrische Quantifizierung von α-Actinin-positiven Zellen vor (Tag 0) und 3 Wochen nach der Bildung von CSs. (n = 14-23 pro Bedingung. (E) RT-qPCR, die an hiPSC-CMs durchgeführt wurde, die 90 Tage lang kultiviert wurden (2D), und an Sphäroidproben, die 42 Tage lang kultiviert wurden, um die Expressionsniveaus verschiedener kardialer Gene zu bestimmen, die mit Zellverbindungen, intermediären Filamenten und Mitochondrien in Verbindung stehen. (n = 1-3 Chargen). Die Daten werden als Mittelwert ± SD dargestellt. NS (nicht signifikant), berechnet durch einen ungepaarten t-Test. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
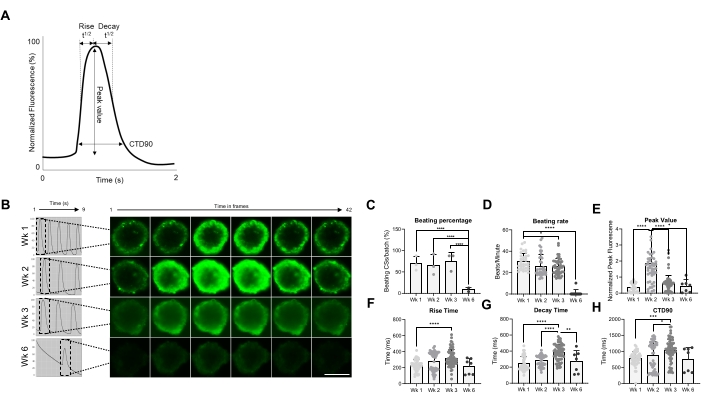
Abbildung 2: Schlagrate und Calcium-Handling in CSs in verschiedenen Wochen nach der Generierung. (A) Beispiele für transiente Calcium-Parameter, die vom Vala Sciences-Analysealgorithmus in der Cyteseer-Software berechnet wurden. (B) Repräsentative Calcium-Transientenspuren und Zeitrafferbilder der CSs zu verschiedenen Zeitpunkten (Wochen) nach der Erzeugung. Maßstabsbalken, 200 μm. (C) Die zeitliche Quantifizierung der spontanen Schlagaktivität wird als Prozentsatz der schlagenden CSs ausgedrückt. (D) Die Taktrate der CSs während der Kultivierungszeit. (E-H) Quantifizierung der Calcium-Transienten mit Spitzenwert, Anstiegszeit, Abklingzeit und CTD90. Bei den angegebenen Daten handelt es sich um Mittelwerte ± SD. Biologische Replikate = drei, technische Replikate = 38, 50, 66 bzw. 7. *p < 0,05, ****p < 0,001; Einweg-ANOVA, gefolgt von Tukeys Post-hoc-Mehrfachvergleichstest. Abkürzungen; CTD = Calcium-Transientendauer, Wk = Woche, CSs = humane Herzsphäroide. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
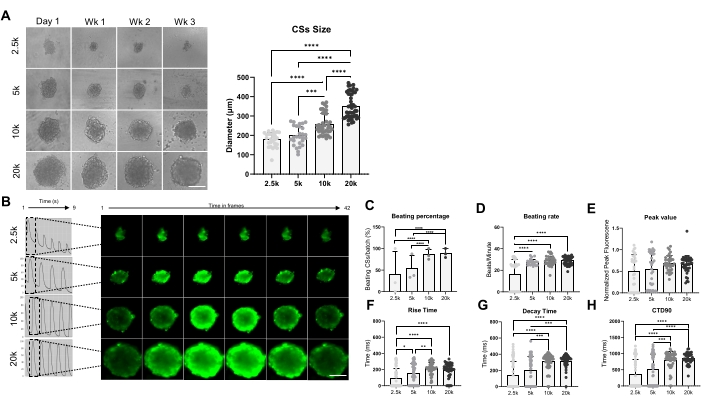
Abbildung 3: Schlagrate und Calcium-Handling in CSs, die mit unterschiedlichen Zellimpfdichten erzeugt wurden. (A) Hellfeld-Bildgebung (links) und Größenmessungen (rechts) von CSs, die mit einer unterschiedlichen Anzahl von hiPSC-CMs erzeugt wurden. Maßstabsbalken, 200 μm. (B) Repräsentative Calcium-Transientenspuren und Zeitrafferaufnahmen der 2,5K-20K-CSs. (C,D) Schlagprozentsatz und Schlagrate von 2.5K-20K-CSs. (E-H) Spitzenwert, Anstiegszeit, Abklingzeit und CTD90 in 2,5K-20K-CSs. Die Daten sind Mittelwerte ± SD. Biologische Replikate = drei, technische Replikate = 28-39. *p < 0,05, ****p < 0,001; Einweg-ANOVA, gefolgt von Tukeys Post-hoc-Mehrfachvergleichstest. Abkürzungen: CTD = Calcium Transient Duration, Wk = Woche, k = x 1.000 Zellen, CSs = kardiale Sphäroide. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
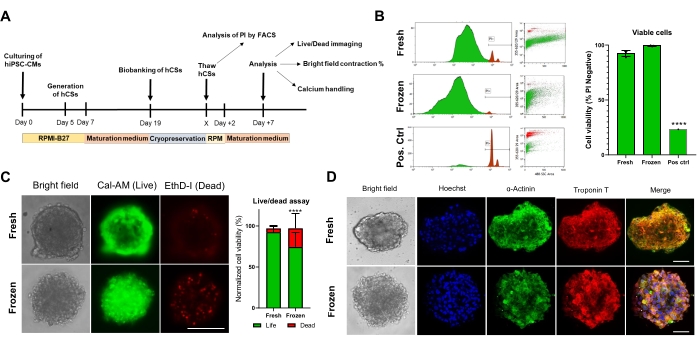
Abbildung 4. Wirkung der Kryokonservierung auf die Lebensfähigkeit und Struktur von kardialen Sphäroiden. (A) Schematische Darstellung der CS-Generierung, des anschließenden Biobankings und des Auftauens. (B) Durchflusszytometrie-Zellviabilitätstest sowohl in frischen als auch in kryokonservierten CSs. Als Positivkontrolle wurde eine Behandlung mit 10% Triton-X-Lösung für 5 min verwendet. (n = 4 pro Bedingung). Die Daten werden als Mittelwert ± SD dargestellt. ** **p < 0,001; Einweg-ANOVA, gefolgt von Tukeys Post-hoc-Mehrfachvergleichstest. (C) Calcein-AM-Zellviabilitätstest in frischen versus aufgetauten CSs nach 7 Tagen Kultivierung (n = 15-17 pro Bedingung, ** **p < 0,001, durch gepaarten t-Test; Maßstabsbalken, 200 μm). (D) Repräsentative Hellfeld- (links) und Immunfluoreszenzfärbung für die α-Actinin- und Troponin-T-Expression in frischen und aufgetauten CSs. Immunfluoreszenz: Hoechst (blau), α-Actinin (grün) und Troponin T (rot). Die zusammengeführten Bilder auf der rechten Seite zeigen Sarkomer-Streifen in den CSs. Maßstabsleiste, 50 μm. Abkürzungen: X = Wunschtautag, PI = Propidiumiodid, Cal-AM = Calcall-AM, EthD-I = Ethidium Homodimer I. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
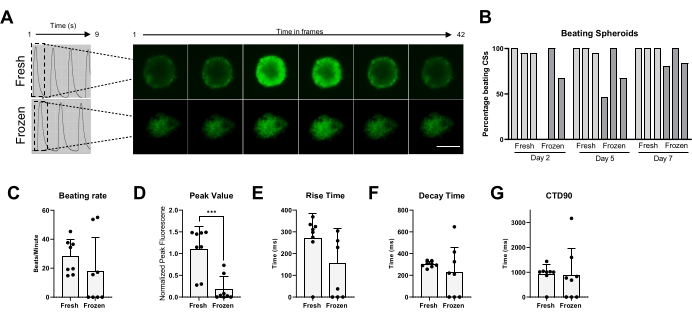
Abbildung 5: Calcium-Transienten in frischen versus aufgetauten CSs. (A) Repräsentative Calcium-Transientenspuren und Zeitrafferbilder der CSs vor der Kryokonservierung und 1 Woche nach dem Auftauen. (B) Schlagprozentsatz von frischen und gefrorenen/aufgetauten Herzsphäroiden. Balken stellen einzelne Experimente dar. (C) Schlagrate von frischen und gefrorenen/aufgetauten Herzsphäroiden. (D-G) Quantifizierung von transienten Calciumparametern: Spitzenwert, Anstiegszeit, Abklingzeit und CTD90. Die Daten sind Mittelwert ± SD. *p < 0,05, ****p < 0,001; Einweg-ANOVA, gefolgt von Tukeys Post-hoc-Mehrfachvergleichstest. Abkürzungen; CTD = Calcium-Transientendauer, CSs = kardiale Sphäroide. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzende Abbildung 1: Repräsentative Gating-Strategien für die Durchflusszytometrie-Analyse. (A) Repräsentative Gating-Strategie für α-Actinin-positive hiPSC-CMs in einer reinen Population versus Negativkontrolle und Isotypkontrolle. Die Anzahl der α-Actinin-positiv analysierten Zellen beträgt 25 x 105. Abkürzungen; SSC = Seitenstreuung, PI+ = Propidiumiodid positiv. (B) Repräsentative Gating-Strategie für die Viabilitätsanalyse sowohl in der frischen, aufgetauten, der Positivkontrolle (Triton-X) als auch in der Negativkontrolle (ungefärbt). Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die Entdeckung von Herzmedikamenten wird durch die Abhängigkeit von nicht-menschlichen Tier- und Zellmodellen mit unzureichendem Durchsatz und physiologischer Genauigkeit behindert, um genaue Messwerte durchzuführen. Die hiPSC-CM-Biologie in Verbindung mit HT-Instrumenten und physiologischen Sonden hat das Potenzial, menschliche Modelle wieder in die frühesten Stadien der Modellierung von Herzerkrankungen und der Wirkstoffforschung einzuführen. Wir haben eine 3D-Methode zur Generierung von Herzgewebe entwickelt, die qualitativ hochwertige und funktionelle CSs für eine optimale Plattform zur Modellierung von Herzerkrankungen und zum Screening von Medikamenten erzeugt. Darüber hinaus ermöglicht die Kombination der Sphäroidtechnologie in 3D-Bioreaktorsystemen für die industrielle EV-Produktion einen notwendigen Schritt in Richtung klinischer Translation der EV-basierten Therapie. Das hier beschriebene Verfahren beruht auf mehreren entscheidenden Faktoren und ist eine Variante der bestehenden Protokolle 9,10,28,29. Zu diesen Methoden gehören: 1) die Erzeugung von 3D-Gewebekonstrukten, 2) die optimale Zellzahl und das optimale Timing vor dem Screening, 3) die Verbesserung der Empfindlichkeit und der Hochdurchsatzkapazität der Instrumente und 4) die Möglichkeit, die Sphäroide vor jeder Funktionsanalyse einzufrieren. Im Gegensatz zu zuvor beschriebenen Protokollen beschreibt das vorgeschlagene Protokoll die Erzeugung von bis zu 1.500 Sphäroiden pro Tag und die Eignung für HTS. Die konventionelle Analyse von hundert Verbindungen über 6 x 0,5 logarithmische Dosen für 10 Replikate unter Verwendung bestehender 96-Well-Calcium-Bildgebungssysteme oder 24-Well-Multiplex-Engineering-Herzgewebe erfordert etwa 500 Millionen bis 3 Milliarden hiPSC-CMs31,32. Die vorgeschlagene Anwendung macht Herzscreenings im Vergleich zu den herkömmlichen Systemen kostengünstiger und zeitsparender, da die 96-Well-Platten im Vergleich zur beschriebenen Methode nur 10% der Aussaatdichte benötigten. Darüber hinaus ermöglicht die Erzeugung von Sphäroiden durch Selbstaggregation in ultraniedrigen Befestigungsplatten im Vergleich zu bisherigen Protokollen, wie z. B. der Hanging-Drop-Methode, eine qualitativ hochwertige automatisierte Bildgebung einzelner Mikrogewebe33.
Dieses kleine 3D-Modell ahmt den biologischen und physiologischen Phänotyp der kardiovaskulären Umgebung in vivo nach. Wie bereits gezeigt, nehmen Calcium-Transienten in 3D-Herzgewebekonstrukten im Vergleich zu 2D-Monolayer-Zellkulturen dramatisch zu34.
Als nächstes stellten wir fest, dass die Aussaatdichte und die richtige Kultivierungszeit ebenfalls kritische Faktoren für ein erfolgreiches CS-Screening sind. Die Dichten von 10K-20K hiPSC-CMs pro Sphäroid und das Screening zwischen den Wochen 2-3 nach der Generierung waren optimal, während zu kleine oder zu alte Sphäroide einen gestörten Umgang mit Kalzium zeigen (Abbildung 2 und Abbildung 3). Daher ist es wichtig, die Aussaatdichte so konstant wie möglich zu halten, da die Größe die Funktionsparameter beeinflusst. Auch wenn diese optische Methode gute Ergebnisse für lebende 3D-Kulturen als ganzes Gewebe liefert, ist es eine Herausforderung, Daten innerhalb größerer Sphäroide auf (sub-)zellulärer Ebene zu erhalten, ohne auf zeitaufwändige histologische Methoden angewiesen zu sein. In jüngster Zeit wurden mehrere Ansätze veröffentlicht, die "optisches Clearing" verwenden, das die Erfassung ganzer 3D-Sphäroide mit der Möglichkeit der Einzelzellquantifizierung von Markern ermöglicht. Hier haben wir ein 3-tägiges Protokoll vom CS-Harvesting auf die Bildanalyse angepasst, das für die 3D-Bildgebung mittels konfokaler Mikroskopie29 optimiert ist (Abbildung 1C und Abbildung 4D).
Schließlich steigt mit der Zunahme von 3D-Herzgewebeanwendungen und kommerziellen Anwendungen die Nachfrage nach Langzeitlagerung und patientenspezifischem Biobanking von verschiedenen Spendern. Die Kryokonservierung ist eine effektive Strategie, um HTS-Platten aus mehreren Chargen im Laufe der Zeit zu erzeugen. Das Einfrieren von hiPSC-CMs wurde bereits beschrieben und unterscheidet sich nicht von anderen kultivierten Zelltypen 10,35,36. Kürzlich wurden Ansätze zum Einfrieren von Platten mit 2D-Zellen beschrieben37. Hier stellten wir fest, dass das PSC-Kryokonservierungskit im Vergleich zu drei anderen (Daten nicht gezeigt) den optimalsten Zustand aufweist und dieses Medium für das effiziente Einfrieren von Sphäroiden verwendete. Nach der Kryokonservierung bleibt die Lebensfähigkeit hoch (Abbildung 4B, C), aber die elektrophysiologischen Eigenschaften der CSs werden beeinträchtigt und eine Inkubationszeit nach dem Auftauen ist erforderlich. Tatsächlich zeigten die CSs 1 Woche nach dem Auftauen eine spontane Schlagaktivität und einen Umgang mit Kalzium. Es wurde jedoch beschrieben, dass frische und wiedergewonnene hiPSC-CMs nicht immer identische molekulare und physiologische Eigenschaften aufweisen38. Diese Einschränkung muss berücksichtigt werden, wenn kryokonservierte hiPSC-CMs zur Beurteilung arzneimittelinduzierter kardialer Messwerte verwendet werden. Obwohl wir die Anzahl der Zellen pro Sphäroid und das optimale Timing der transienten Calcium-Bildgebung effektiv modulieren, könnten die kardialen Sphäroide verbessert werden, indem hiPSC-abgeleitete Kardiomyozytenzellen mit Endothelzellen, Fibroblasten, Zell-Zell-Verbindungen und extrazellulären Matrizen wie Chitosan, Kollagen IV, Fibronektin, Matrigel oder Laminin gemischt werden, die die in vivo Herzumgebung nachahmen39. 40. Sonstiges Insgesamt schlagen wir ein Schritt-für-Schritt-Protokoll zur effizienten Generierung von CSs vor, die für nachgelagerte Anwendungen wie Krankheitsmodellierung und HT-Wirkstoffscreening geeignet sind.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Wir bedanken uns bei VALA sciences für das Cyteseer-Softwarepaket und die Optimierung der automatisierten 3D-Calciumanalyse. Wir bedanken uns für die Unterstützung durch die PLN-Stiftung (RM). P.A.D. und F.S. werden von CUREPLaN Leducq unterstützt. J.P.G.S. wird gefördert durch H2020-EVICARE (#725229) des Europäischen Forschungsrats (ERC). J.W.B. wird durch das UMC Utrecht Clinical Fellowship, das Netherlands Heart Institute Fellowship und das CVON-Dosis Young Talent Grant unterstützt. Niederländische Herzstiftung (CVON-Dosis 2014-40). N.C. wird durch das Gravitationsprogramm "Materials Driven Regeneration" der Niederländischen Organisation für wissenschaftliche Forschung (RegmedXB #024.003.013) und die Marie-Skłodowska-Curie-Maßnahmen (Finanzhilfevereinbarung RESCUE #801540) unterstützt. V.S.-P. wird aus dem Allianzfonds (UMCU, UU, TU/e) gefördert. A.v.M. wird gefördert durch das EU-geförderte Projekt BRAVE (H2020, ID:874827)
Materialien
| Name | Company | Catalog Number | Comments |
| 24 wells suspenion plate | Corning | 3738 | |
| 96 wells Ultra-Low Attachment Multiple Well Plate | Corning | CLS3474-24EA | |
| Albumax | Thermo Fisher Scientific | 11020021 | |
| Anti-α-Actinin (Sarcomeric) antibody | Sigma-Aldrich | A7811 | Dilution: 1:200 |
| Anti-Cardiac Troponin T antibody (ab45932) | Abcam | ab45932 | Dilution: 1:200 |
| Ascorbic acid | Sigma-Aldrich | A8960 | |
| B-27 supplement | Thermo Fisher Scientific | 17504-044 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Bovine serum albumin fraction V (BSA) | Roche | 10735086001 | |
| Cal-520, AM | Abcam | ab171868 | |
| Confocal microscope | Leica | DMi8 | |
| Confocal microscope software | Leica | Las X | |
| Conical tubes 15 mL | Greiner Bio-One | 5618-8271 | |
| Creatine monohydrate | Sigma-Aldrich | C3630 | |
| DAPI | Thermo Fisher Scientific | D3571 | Concentration: 1 µg/mL |
| DMEM no glucose | Thermo Fisher Scientific | 11966025 | |
| EDTA | Thermo Fisher Scientific | 15575020 | |
| Fructose | Sigma-Aldrich | 76050771.05 | |
| Glucose | Sigma-Aldrich | G7021 | |
| Glycerol | Boom | 76050771.05 | |
| Goat anti-mouse Alexa Fluor 488 | Invitrogen | A11029 | Dilution: 1:500 |
| Goat anti-rabbit Alexa Fluor 568 | Invitrogen | A11011 | Dilution: 1:500 |
| Horizontal shaker | IKA | 4003000 | |
| Human induced pluripotent stem cell lines | (Stanford Cardiovascular Institute (S-CVI) Biobank) | CVI-273 (control 1) | |
| Human induced pluripotent stem cell lines | Germany | 141 (control 2) 144 (control 3) | |
| Hydrochloric acid (HCl) | Ajax Firechem | 265.2.5L-PL | 10 M stock solution, corrosive |
| Isotype control, FITC mouse IgM κ isotype | BD | 556652 | |
| KnockOut Serum Replacement | Thermo Fisher Scientific | 10828 | Protect from light |
| L-carnitine | Sigma-Aldrich | C0283 | |
| Myocyte calcium and contractility system | Leica | Thunder, DMi8 | |
| Non essential amino acids (NEAA) | Thermo Fisher Scientific | 11140 | |
| Paraformaldehyde solution 4% in 1x PBS, pH 7.0–7.6 | Santa Cruz | SC281692 | Hazardous |
| PBS, pH 7.4 | Thermo Fisher Scientific | 10010023 | |
| Penicillin/streptomycin | Thermo Fisher Scientific | 15140 | |
| PES Membrane Vacuum Filter system | Corning | 431097 | |
| PI/RNase Staining Solution | Invitrogen | F10797 | Dilution: 1:1000 |
| Pluronic F-127 | Sigma-Aldrich | P2443 | |
| PSC Cryopreservation Kit | Thermo Fisher Scientific | A2644601 | |
| RevitaCell | Thermo Fisher Scientific | A2644501 | |
| RPMI 1640 medium | Thermo Fisher Scientific | 11875 | |
| Silicone Elastomer Kit | SYLGARD | 184 | |
| Sodium dodecyl sulfate solution (10%) | Sigma-Aldrich | 71736 | |
| Sodium L-Lactate | Sigma-Aldrich | 71718 | |
| Taurine | Sigma-Aldrich | T0625 | |
| Tris Fisher | Scientific | 11486631 | |
| Triton X-100 | Merck | X100-1L | Hazardous |
| Trypan blue solution, 0.4% | Thermo Fisher Scientific | 15250061 | |
| TrypLE Select Enzyme (10x) | Thermo Fisher Scientific | A1217701 | |
| Tween-20 | Sigma-Aldrich | P1379 | |
| Urea | Sigma-Aldrich | 51456 | |
| Vitamin B12 | Sigma-Aldrich | V6629 | |
| Y-27632 dihydrochloride (Rho-kinase inhibitor) | Tocris | 1254 | Protect from light |
Referenzen
- Burridge, P. W., et al. Chemically defined and small molecule-based generation of human cardiomyocytes. Nature Methods. 11 (8), 855-860 (2014).
- Kattman, S. J., et al. Stage-specific optimization of activin/nodal and BMP signaling promotes cardiac differentiation of mouse and human pluripotent stem cell lines. Cell Stem Cell. 8 (2), 228-240 (2011).
- Lian, X., et al. Robust cardiomyocyte differentiation from human pluripotent stem cells via temporal modulation of canonical Wnt signaling. Proceedings of the National Academy of Sciences. 109 (27), 1848-1857 (2012).
- Paige, S. L., et al. Endogenous Wnt/beta-catenin signaling is required for cardiac differentiation in human embryonic stem cells. PLoS One. 5 (6), 11134 (2010).
- Gintant, G., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes in preclinical cancer drug cardiotoxicity testing: A scientific statement from the American Heart Association. Circulation Research. 125 (10), 75-92 (2019).
- Ahmed, R. E., et al. A brief review of current maturation methods for human induced pluripotent stem cells-derived cardiomyocytes. Frontiers in Cell and Developmental Biology. 19 (8), 178 (2020).
- Liu, C., et al. Generating 3D human cardiac constructs from pluripotent stem cells. EBioMedicine. 76, 103813 (2022).
- Musunuru, K., et al. Induced pluripotent stem cells for cardiovascular disease modeling and precision medicine: A scientific statement from the American Heart Association. Circulation: Genomic and Precision Medicine. 11 (1), 000043 (2018).
- Buikema, J. W., et al. Wnt activation and reduced cell-cell contact synergistically induce massive expansion of functional human iPSC-derived cardiomyocytes. Cell Stem Cell. 27 (1), 50-63 (2020).
- Maas, R. G. C., et al. Massive expansion and cryopreservation of functional human induced pluripotent stem cell-derived cardiomyocytes. Cell STAR Protocols. 2 (1), 100334 (2021).
- Tremblay, C., et al. A new construction technique for tissue-engineered heart valves using the self-assembly method. Tissue Engineering Part C: Methods. 20 (11), 905-915 (2014).
- Lewis-Israeli, Y. R., et al. Self-assembling human heart organoids for the modeling of cardiac development and congenital heart disease. Nature Communications. 12 (1), 5142 (2021).
- Goldfracht, I., et al. Engineered heart tissue models from hiPSC-derived cardiomyocytes and cardiac ECM for disease modeling and drug testing applications. Acta Biomaterialia. 1 (92), 145-159 (2019).
- Fleischer, S., et al. Comprehensive human stem cell differentiation in a 2D and 3D mode to cardiomyocytes for long-term cultivation and multiparametric monitoring on a multimodal microelectrode array setup. Biosensors and Bioelectronics. 126, 624-631 (2019).
- Branco, M. A., et al. Transcriptomic analysis of 3D cardiac differentiation of human induced pluripotent stem cells reveals faster cardiomyocyte maturation compared to 2D culture. Science Reports. 9 (1), 9229 (2019).
- Ergir, E., et al. Generation and maturation of human iPSC-derived cardiac organoids in long term culture. bioRxiv. , (2022).
- Lemoine, M. D., et al. Human iPSC-derived cardiomyocytes cultured in 3D engineered heart tissue show physiological upstroke velocity and sodium current density. Scienctific Reports. 7 (1), 5464 (2017).
- Kofron, C. M., et al. A predictive in vitro risk assessment platform for pro-arrhythmic toxicity using human 3D cardiac microtissues. Science Reports. 11 (1), 10228 (2021).
- Giacomelli, E., et al. Human-iPSC-derived cardiac stromal cells enhance maturation in 3D cardiac microtissues and reveal non-cardiomyocyte contributions to heart disease. Cell Stem Cell. 26 (6), 862-879 (2020).
- Richards, D. J., et al. Human cardiac organoids for the modelling of myocardial infarction and drug cardiotoxicity. Nature Biomedical Engineering. 4 (4), 446-462 (2020).
- Tenreiro, M. F., et al. Next generation of heart regenerative therapies: progress and promise of cardiac tissue engineering. npj Regenerative Medicine. 6 (1), 30 (2021).
- Hansen, A., et al. Development of a drug screening platform based on engineered heart tissue. Circulation Research. 107 (1), 35-44 (2010).
- McDermott-Roe, C., et al. Investigation of a dilated cardiomyopathy-associated variant in BAG3 using genome-edited iPSC-derived cardiomyocytes. Journal of Clinical Investigation Insight. 4 (22), 128799 (2019).
- National Library of Medicine (U.S.). Safety and efficacy of induced pluripotent stem cell-derived engineered human myocardium as biological ventricular assist tissue in terminal heart failure. National Library of Medicine. , (2020).
- Ronaldson-Bouchard, K., et al. Advanced maturation of human cardiac tissue grown from pluripotent stem cells. Nature. 556 (7700), 239-243 (2018).
- Oh, J. G., et al. Generation of ventricular-like HiPSC-derived cardiomyocytes and high-quality cell preparations for calcium handling characterization. Journal of Visualized Experiments. 155, 60135 (2020).
- Lian, X., et al. Directed cardiomyocyte differentiation from human pluripotent stem cells by modulating Wnt/β-catenin signaling under fully defined conditions. Nature Protocols. 8 (1), 162-175 (2013).
- Feyen, D. A. M., et al. Metabolic maturation media improve physiological function of human iPSC-derived cardiomyocytes. Cell Reports. 32 (3), 107925 (2020).
- van Ineveld, R. L., et al. Single-cell resolution three-dimensional imaging of intact organoids. Journal of Visualized Experiments. (160), e60709 (2020).
- Guo, Y., Pu, W. T. Cardiomyocyte maturation: New phase in development. Circulation Research. 126 (8), 1086-1106 (2020).
- Ding, B., et al. Three-dimensional renal organoids from whole kidney cells: Generation, optimization, and potential application in nephrotoxicology in vitro. Cell Transplantation. 29, 963689719897066 (2020).
- Denning, C., et al. Cardiomyocytes from human pluripotent stem cells: From laboratory curiosity to industrial biomedical platform. Biochimica Biophysica Acta. 1863, 1728-1748 (2016).
- Amaral, R. L. F., et al. Comparative analysis of 3D bladder tumor spheroids obtained by forced floating and hanging drop methods for drug screening. Frontiers in Physiology. 8, 605 (2017).
- Daily, N. J., et al. Improving cardiac action potential measurements: 2D and 3D cell culture. Journal of Bioengineering and Biomedical Science. 5 (3), 168 (2015).
- Preininger, M. K., et al. Cryopreservation of human pluripotent stem cell-derived cardiomyocytes: Strategies, challenges, and future directions. Advances in Experimental Medicine and Biology. 951, 123-135 (2016).
- Kim, Y. Y., et al. Cryopreservation of human embryonic stem cells derived-cardiomyocytes induced by BMP2 in serum-free condition. Reproductive Science. 18 (3), 252-360 (2011).
- Daily, M. I., et al. Cryopreservation of primary cultures of mammalian somatic cells in 96-well plates benefits from control of ice nucleation. Cryobiology. 93, 62-69 (2020).
- Zhang, J. Z., et al. Effects of cryopreservation on human induced pluripotent stem cell-derived cardiomyocytes for assessing drug safety response profiles. Stem Cell Reports. 16 (1), 168-181 (2021).
- Yeh, H. -. Y., et al. The calcium-dependent regulation of spheroid formation and cardiomyogenic differentiation for MSCs on chitosan membranes. Biomaterials. 33 (35), 8943-8954 (2012).
- Scalise, M., et al. From spheroids to organoids: The next generation of model systems of human cardiac regeneration in a dish. International Journal of Molecular Sciences. 22 (24), 13180 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten