Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolierung von Gentherapievektoren der nächsten Generation durch Engineering, Barcoding und Screening von Kapsidvarianten des Adeno-assoziierten Virus (AAV)
In diesem Artikel
Zusammenfassung
Generierung von AAV-Peptid-Display-Bibliotheken und anschließende Validierung durch Barcoding von Kandidaten mit neuartigen Eigenschaften für die Erstellung von AAVs der nächsten Generation.
Zusammenfassung
Genübertragungsvektoren, die vom Adeno-assoziierten Virus (AAV) abgeleitet sind, sind eines der vielversprechendsten Werkzeuge für die Behandlung genetischer Krankheiten, was durch ermutigende klinische Daten und die Zulassung mehrerer AAV-Gentherapien belegt wird. Zwei Hauptgründe für den Erfolg von AAV-Vektoren sind (i) die vorherige Isolierung verschiedener natürlich vorkommender viraler Serotypen mit unterschiedlichen Eigenschaften und (ii) die anschließende Etablierung leistungsfähiger Technologien für ihr molekulares Engineering und ihre Wiederverwendung im Hochdurchsatz. Das Potenzial dieser Techniken wird durch kürzlich implementierte Strategien zur Barcodierung ausgewählter AAV-Kapside auf DNA- und RNA-Ebene weiter gesteigert, die eine umfassende und parallele In-vivo-Stratifizierung in allen wichtigen Organen und Zelltypen eines einzigen Tieres ermöglichen. Hier stellen wir eine grundlegende Pipeline vor, die diese Reihe komplementärer Wege umfasst und die AAV-Peptidanzeige verwendet, um das vielfältige Arsenal der verfügbaren Kapsid-Engineering-Technologien darzustellen. Dementsprechend beschreiben wir zunächst die entscheidenden Schritte für die Generierung einer AAV-Peptid-Display-Bibliothek für die In-vivo-Auswahl von Kandidaten mit den gewünschten Eigenschaften, gefolgt von einer Demonstration, wie die interessantesten Kapsidvarianten für das sekundäre In-vivo-Screening mit einem Barcode versehen werden können. Als nächstes veranschaulichen wir die Methodik für die Erstellung von Bibliotheken für Next-Generation-Sequencing (NGS), einschließlich Barcode-Amplifikation und Adapterligation, bevor wir mit einem Überblick über die kritischsten Schritte während der NGS-Datenanalyse schließen. Da die hier berichteten Protokolle vielseitig und anpassungsfähig sind, können Forscher sie leicht nutzen, um die optimalen AAV-Kapsidvarianten in ihrem bevorzugten Krankheitsmodell und für Gentherapieanwendungen anzureichern.
Einleitung
Gentransfertherapie ist die Einführung von genetischem Material in Zellen, um das zelluläre genetische Material zu reparieren, zu ersetzen oder zu verändern, um Krankheiten vorzubeugen, zu behandeln, zu heilen oder zu verbessern. Der Gentransfer, sowohl in vivo als auch ex vivo, beruht auf unterschiedlichen Verabreichungssystemen, nicht-viralen und viralen. Viren haben sich auf natürliche Weise entwickelt, um ihre Zielzellen effizient zu transduzieren, und können als Liefervektoren verwendet werden. Unter den verschiedenen Arten von viralen Vektoren, die in der Gentherapie eingesetzt werden, werden zunehmend Adeno-assoziierte Viren verwendet, da sie nicht pathogen sind, Sicherheit, geringe Immunogenität und vor allem ihre Fähigkeit, eine langfristige, nicht integrierende Expression aufrechtzuerhalten 1,2,3. Die AAV-Gentherapie hat in den letzten zehn Jahren beachtliche Erfolge erzielt. drei Therapien wurden von der Europäischen Arzneimittel-Agentur und der US-amerikanischen Food and Drug Administration für die Anwendung beim Menschen zugelassen 3,4. Mehrere klinische Studien sind auch im Gange, um eine Vielzahl von Krankheiten wie Hämophilie, Muskel-, Herz- und neurologische Erkrankungen zu behandeln, wie an anderer Stelle beschrieben3. Trotz jahrzehntelanger Fortschritte hat der Bereich der Gentherapie in den letzten Jahren eine Reihe von Rückschlägen erlitten4, vor allem Todesfälle in klinischen Studien5, die aufgrund dosislimitierender Toxizitäten auf Eis gelegt wurden, insbesondere für Gewebe, die massiv sind, wie z. B. Muskeln, oder schwer zu erreichen, wie z. B. Gehirn6.
Die AAV-Vektoren, die derzeit in klinischen Studien verwendet werden, gehören mit wenigen Ausnahmen zu den natürlichen Serotypen1. AAV-Engineering bietet die Möglichkeit, Vektoren mit überlegener Organ- oder Zellspezifität und Effizienz zu entwickeln. In den letzten zwei Jahrzehnten wurden verschiedene Ansätze erfolgreich angewendet, wie z. B. Peptid-Display, Loop-Swap, Kapsid-DNA-Shuffling, fehleranfällige PCR und gezieltes Design, um einzelne AAV-Varianten oder Bibliotheken davon mit unterschiedlichen Eigenschaften zu generieren7. Diese werden dann mehreren Runden der gerichteten Evolution unterzogen, um die darin enthaltenen Varianten mit den gewünschten Eigenschaften auszuwählen, wie an anderer Stelle 1,3 beschrieben. Von allen Kapsid-Evolutionsstrategien sind Peptid-Display-AAV-Bibliotheken aufgrund einiger einzigartiger Eigenschaften am weitesten verbreitet: Sie sind relativ einfach zu generieren und können eine hohe Diversität und eine Hochdurchsatz-Sequenzierung erreichen, die es ermöglicht, ihre Evolution zu verfolgen.
Die ersten erfolgreichen Peptidinsertions-AAV-Bibliotheken wurden vor fast 20 Jahren beschrieben. In einem der ersten konstruierten Perabo et al.8 eine Bibliothek modifizierter AAV2-Kapside, in der ein Pool zufällig generierter Oligonukleotide in ein Plasmid an einer Position eingefügt wurde, die der Aminosäure 587 des VP1-Kapsidproteins entspricht, und zwar in der dreifachen Achse, die aus dem Kapsid herausragt. Unter Verwendung einer Adenovirus-Koinfektion wurde die AAV-Bibliothek durch mehrere Selektionsrunden entwickelt, und es wurde gezeigt, dass die endgültigen Re-Targeting-Varianten in der Lage sind, Zelllinien zu transduzieren, die refraktär auf das elterliche AAV28 sind. Kurz darauf führten Müller et al.9 das zweistufige System für die Bibliotheksproduktion ein, eine deutliche Verbesserung des Protokolls. Zunächst wird die Plasmidbibliothek zusammen mit einem adenoviralen Helferplasmid verwendet, um eine AAV-Bibliothek zu erzeugen, die chimäre Kapside enthält. Diese AAV-Shuttle-Bibliothek wird verwendet, um Zellen mit geringer Infektionsvielfalt (MOI) zu infizieren, mit dem Ziel, ein virales Genom pro Zelle einzuführen. Die Koinfektion mit dem Adenovirus gewährleistet die Produktion von AAVs mit einem übereinstimmenden Genom und Kapsid9. Etwa ein Jahrzehnt später nutzte Dalkara10 die in vivo gerichtete Evolution, um die 7m8-Variante zu schaffen. Diese Variante hat eine 10-Aminosäure-Insertion (LALGETTRPA), von denen drei als Linker fungieren, und zielt nach intravitrealer Injektion effizient auf die äußere Netzhautab 10. Dieses technisch hergestellte Kapsid ist eine außergewöhnliche Erfolgsgeschichte, da es eines der wenigen künstlich hergestellten Kapside ist, das es bisher in die Klinik geschafft hat11.
Einen zweiten Schub erlebte das Feld mit der Einführung von Next-Generation-Sequencing-Techniken (NGS). Zwei Veröffentlichungen von Adachi et al.12 aus dem Jahr 2014 und von Marsic et al.13 aus dem Jahr 2015 zeigten die Leistungsfähigkeit von NGS, die Verteilung von barcodierten AAV-Kapsidbibliotheken mit hoher Genauigkeit zu verfolgen. Einige Jahre später wurde das NGS der barcodierten Regionen an die Peptidinsertionsregion angepasst, um die Kapsidentwicklung zu verfolgen. Körbelin et al.14 führten ein NGS-gesteuertes Screening durch, um ein pulmonales AAV2-basiertes Kapsid zu identifizieren. Die NGS-Analyse half bei der Berechnung von drei Bewertungswerten: dem Anreicherungswert zwischen den Selektionsrunden, dem allgemeinen Spezifitätswert zur Bestimmung der Gewebespezifität und schließlich dem kombinierten Wert14. Das Gradinaru-Labor15 veröffentlichte im selben Jahr das Cre-Rekombinations-basierte AAV-Targeted-Evolutionssystem (CREATE), das eine zelltypspezifische Selektion ermöglicht. In diesem System trägt die Kapsidbibliothek einen Cre-invertierbaren Schalter, da das polyA-Signal von zwei loxP-Stellen flankiert wird. Die AAV-Bibliothek wird dann in Cre-Mäuse injiziert, wobei das polyA-Signal nur in Cre+-Zellen invertiert wird, was die Vorlage für die Bindung eines Reverse-PCR-Primers an den Forward-Primer innerhalb des Kapsidgens liefert. Diese hochspezifische PCR-Rettung ermöglichte die Identifizierung des AAV-PHP. B-Variante, die die Blut-Hirn-Schranke überwinden kann15. Dieses System wurde zu M-CREATE (Multiplexed-CREATE) weiterentwickelt, bei dem NGS und die Generierung synthetischer Bibliotheken in die Pipeline16 integriert wurden.
Eine verbesserte RNA-basierte Version dieses Systems aus dem Maguire-Labor17, iTransduce, ermöglicht die Selektion auf DNA-Ebene von Kapsiden, die Zellen funktionell transduzieren und ihre Genome exprimieren. Das virale Genom der Peptid-Display-Bibliothek besteht aus einem Cre-Gen unter der Kontrolle eines ubiquitären Promotors und dem Kapsidgen unter der Kontrolle des p41-Promotors. Die Bibliothek wird Mäusen injiziert, die eine loxP-STOP-loxP-Kassette stromaufwärts von tdTomato haben. Zellen, die mit AAV-Varianten transduziert wurden, die das virale Genom und damit Cre exprimieren, exprimieren tdTomato und können in Kombination mit Zellmarkern sortiert und selektiert werden17. In ähnlicher Weise stellten Nonnenmacher et al.18 und Tabebordbar et al.19 die Kapsidgenbibliothek unter die Kontrolle gewebespezifischer Promotoren. Nach der Injektion in verschiedene Tiermodelle wurde virale RNA verwendet, um die Kapsidvarianten zu isolieren.
Ein alternativer Ansatz ist die Verwendung von Barcoding zum Markieren von Kapsidbibliotheken. Das Björklund-Labor20 nutzte diesen Ansatz für Barcode-Peptid-Insertionskapsidbibliotheken und entwickelte die barcoded rationale AAV-Vektorevolution (BRAVE). In einem Plasmid wird die Rep2Cap-Kassette neben einem ITR-flankierten, gelb fluoreszierenden Protein (YFP)-exprimierenden, Barcode-markierten Transgen kloniert. Unter Verwendung von loxP-Stellen zwischen dem Ende der Kappe und dem Anfang des Barcodes erzeugt eine in vitro Cre-Rekombination ein Fragment, das klein genug für NGS ist, wodurch die Assoziation der Peptidinsertion mit dem eindeutigen Barcode ermöglicht wird (Look-up-Tabelle, LUT). Die AAV-Produktion wird unter Verwendung der Plasmidbibliothek durchgeführt, und die in der mRNA exprimierten Barcodes werden nach der In-vivo-Anwendung erneut mit NGS20 gescreent. Wenn die Kapsidbibliotheken Varianten des gesamten Kapsidgens enthalten (d. h. gemischte Bibliotheken), muss eine Long-Read-Sequenzierung verwendet werden. Mehrere Gruppen haben Barcodes verwendet, um diese verschiedenen Bibliotheken zu kennzeichnen, was NGS eine höhere Lesetiefe ermöglicht. Das Kay Lab21 markierte sehr unterschiedliche Kapsid-gemischte Bibliotheken mit Barcodes , die dem Cap-PolyA-Signal nachgeschaltet waren. In einem ersten Schritt wurde eine barcodierte Plasmidbibliothek generiert und die gemischte Kapsidgenbibliothek in diese kloniert. Dann wurde eine Kombination aus MiSeq (Short Read, höhere Lesetiefe) und PacBio (Long Read, niedrigere Lesetiefe) NGS sowie Sanger-Sequenzierung verwendet, um ihre LUT21 zu generieren. Im Jahr 2019 beschrieben Ogden und seine Kollegen aus dem Church-Labor22 die AAV2-Kapsideignung für mehrere Funktionen mithilfe von Bibliotheken, die in jeder Position Einzelpunktmutationen, Insertionen und Deletionen aufwiesen, was letztendlich ein maschinengeführtes Design ermöglichte. Für die Generierung der Bibliothek wurden kleinere Fragmente des Kapsidgens synthetisiert, mit einem Barcode markiert, sequenziert und dann in das vollständige Kapsidgen kloniert. Die NGS-Daten wurden verwendet, um eine LUT zu generieren. Die Bibliothek wurde dann nur mit den Barcodes und der Short-Read-Sequenzierung gescreent, was wiederum eine höhere Lesetiefe22 ermöglicht.
Barcode-Bibliotheken wurden überwiegend verwendet, um einen Pool bekannter, natürlicher und technisch entwickelter Varianten nach mehreren Auswahlrunden von Kapsidbibliotheken oder unabhängig von einer Kapsidentwicklungsstudie zu überprüfen. Der Vorteil solcher Bibliotheken ist die Möglichkeit, mehrere Kapside zu screenen und gleichzeitig die Anzahl der Tiere zu reduzieren und die Variation zwischen den Tieren zu minimieren. Die ersten Studien, die diese Technologie in den AAV-Bereich einführten, wurden vor fast einem Jahrzehnt veröffentlicht. Das Nakai-Labor 12 markierte 191 Doppel-Alanin-Mutanten, die die Aminosäuren 356 bis 736 auf dem VP1 von AAV9 mit einem Paar 12-Nukleotid-Barcodes abdecken. Unter Verwendung von NGS wurde die Bibliothek in vivo auf Galaktosebindung und andere Eigenschaften untersucht12. Marsic und Kollegen beschrieben die Bioverteilung von AAV-Varianten mit Hilfe einer Doppel-Barcord-Analyse 1 Jahr später13. Eine neuere Studie an nichtmenschlichen Primaten verglich die Bioverteilung von 29 Kapsiden im Zentralnervensystem auf verschiedenen Verabreichungswegen23. Unser Labor hat kürzlich barcodierte AAV-Bibliotheksbildschirme von 183 Varianten veröffentlicht, die natürliche und technisch hergestellte AAVs enthielten. Diese Screenings auf DNA- und RNA-Ebene führten zur Identifizierung einer hochgradig myotropen AAV-Variante24 bei Mäusen sowie anderen, die eine hohe Zelltypspezifität im Mäusegehirn aufwiesen25.
Hier beschreiben wir die in dieser Arbeit verwendete Methodik und erweitern sie um das Screening von AAV-Peptid-Display-Bibliotheken. Dies umfasst die Generierung von AAV2-Peptid-Display-Bibliotheken, eine digitale Tröpfchen-PCR-Methode (dd-PCR) zur Quantifizierung und schließlich eine NGS-Pipeline zur Analyse der AAV-Varianten, die zum Teil auf der Arbeit von Weinmann und Kollegen24 basiert. Schließlich wird eine Beschreibung der Generierung von barcodierten AAV-Bibliotheken und der NGS-Pipeline bereitgestellt, die in derselben Publikation verwendet wird.
Protokoll
1. Vorbereitung der AAV2-Random-7-Mer-Peptid-Display-Bibliothek
HINWEIS: Für die Herstellung einer AAV2-Random-Peptid-Display-Bibliothek synthetisieren Sie die entarteten Oligonukleotide als einzelsträngige DNA, wandeln Sie sie in doppelsträngige DNA um, verdauen Sie, ligieren Sie sich an das Akzeptorplasmid und elektroporat.
- Design von entarteten Oligonukleotiden
- Ordnen Sie die entarteten Oligonukleotide und vermeiden Sie Codon-Bias. Im Oligonukleotid 5' CAGTCGGCCAG AG W GGC (X01)7 GCCCAGGCGGCTGACGAG 3' entspricht X01 20 Codons, die jeweils für eine der 20 Aminosäuren kodieren. Das W kann A oder T sein und die Codons AGA oder AGT erzeugen, die für die Aminosäuren Arginin (R) oder Serin (S) kodieren.
- Bestellen Sie den Amplifikationsprimer: 5' CTCGTCAGCCGCCTGG 3' (siehe Abbildung 1 für Details). Dabei entsteht folgender Proteineinsatz: R/S GX 7. Die theoretische Diversität wird wie folgt berechnet: 1 x 2 x 207 = 2,56 x 109 eindeutige Varianten.
HINWEIS: Es ist zu beachten, dass diese Vielfalt durch die Transformationseffizienz eingeschränkt sein kann.
- Synthese des zweiten Strangs
- Resuspendieren Sie beide Oligonukleotide (entartete Oligonukleotide und Amplifikationsprimer) mit TE-Puffer auf eine Endkonzentration von 100 μM.
- Für die PCR-Reaktion wird eine 50-μl-Reaktion mit 1 μl jedes Primers, 10 μl des Puffers, 1,5 μl DMSO, 0,5 μl dNTPs (10 mM), 0,5 μl Hi-Fidelity-Hot-Start-Polymerase II und 35,5 μl nukleasefreiem Wasser eingerichtet.
- Übertragen Sie die Reaktion auf einen Thermocycler und führen Sie einen Vorinkubationsschritt für 10 s bei 98 °C durch, gefolgt von drei Zyklen von 10 s bei 98 °C, 30 s bei 59 °C und 10 s bei 72 °C, dann 5 min bei 72 °C und einem abschließenden Abkühlschritt.
- Reinigen Sie die Reaktion mit einem Nukleotidentfernungskit und eluieren Sie sie in 100 μl nukleasefreiem Wasser.
- Bestätigen Sie die Effizienz der Zweitstrangsynthese durch Analyse auf einem Bioanalysator (siehe Abbildung 2). Analysieren Sie die Größe und Reinheit des doppelsträngigen Einsatzes, indem Sie 1 μl der Reaktion auf einen mikrofluidischen Chip aus einem DNA 1000-Reagenzien-Kit gemäß den Anweisungen des Herstellers laden. Dieses Kit ist optimiert, um die Größe und Konzentration von doppelsträngigen DNA-Fragmenten von 25-1.000 bps zu messen.
- Resuspendieren Sie beide Oligonukleotide (entartete Oligonukleotide und Amplifikationsprimer) mit TE-Puffer auf eine Endkonzentration von 100 μM.
- Aufschluss von Insert und Plasmidvektor
- Aufschluss von 85 μl des gereinigten Einsatzes mit 10 μl 10-fachem Puffer und 5 μl BglI-Enzym in einem abschließenden Reaktionsvolumen von 100 μl (siehe Abbildung 1 für Details). Über Nacht bei 37 °C inkubieren. Mit einem Nukleotidentfernungskit reinigen, in 50 μl nukleasefreiem Wasser eluieren und mit dem Typ "Oligo-DNA" in einem Spektralphotometer quantifizieren.
- Verdauen Sie 10 μg eines replikationskompetenten AAV-Plasmids (pRep2Cap2_PIS)26 (ITR-flankiertes virales Genom) mit 20 μl 10-fachem Puffer und 10 μl SfiI-Enzym in einem abschließenden 200-μl-Reaktionsvolumen (siehe Abbildung 1 für Details). Über Nacht bei 50 °C inkubieren. Reinigen Sie den Vektor auf einem 1%igen Agarosegel unter Verwendung des Gelextraktionskits, gefolgt von einem zusätzlichen Reinigungsschritt unter Verwendung eines DNA-Reinigungskits. Quantifizieren Sie die Konzentration in einem Spektralphotometer.
- Ligation des Inserts an den Vektor
- Ligat 955 ng Plasmidvektor mit 45 ng Insert mit 2 μl Puffer und 2 μl Ligase in einer 20 μL Ligationsreaktion. Inkubation bei 16 °C über Nacht, gefolgt von 10 min bei 70 °C, um die Ligase hitzeinaktiv zu machen.
- Transformation, Komplexitätsberechnung und Vorbereitung von Plasmidbibliotheken
- Reinigen Sie die Reaktion mit einem DNA-Aufreinigungskit gemäß den Anweisungen des Herstellers. Die Reaktion wird in etwa 80% des Ausgangsvolumens von nukleasefreiem Wasser eluiert und zur anschließenden Umwandlung auf Eis gelagert.
- Transformieren Sie elektrokompetente Zellen: Tauen Sie ein Fläschchen mit elektrokompetenten Zellen 10 Minuten lang auf Eis auf. Geben Sie dann 1-2 μl der gereinigten Ligationsreaktion in 30 μl (eine Durchstechflasche) elektrokompetente Zellen und mischen Sie durch leichtes Klopfen. Anschließend pipettieren Sie das Zell/DNA-Gemisch vorsichtig in eine vorgekühlte Elektroporationsküvette mit 1 mm Spalt, ohne Luftblasen einzuführen.
- Elektroporat mit den folgenden Einstellungen: 1800 V, 600 Ω und 10 μF. Innerhalb von 10 s nach dem Elektroporationsimpuls werden 970 μl vorgewärmtes Rückgewinnungsmedium (mit den elektrokompetenten Zellen geliefert) in die Küvette gegeben und durch Pipettieren gemischt. Zum Schluss werden die Zellen in ein Mikrozentrifugenröhrchen überführt und 1 h bei 37 °C bei 250 U/min inkubiert. Um eine gewünschte Vielfalt zu erreichen, führen Sie 10-100 Reaktionen durch und fassen Sie nach der Inkubation alle Reaktionen in einem Röhrchen zusammen.
- Berechnen Sie die Diversität, indem Sie 10 μl der gepoolten Transformationen 10-, 100- oder 1.000-fach in PBS verdünnen und 100 μl auf Nähr-Agar-Platten verteilen, die das entsprechende Antibiotikum (75 mg/ml Ampicillin) enthalten. Inkubieren Sie die Agarplatten über Nacht bei 37 °C und zählen Sie dann die Kolonien auf den Agarplatten.
- Berechnen Sie die theoretische Diversität wie folgt:
Theoretische maximale Diversität = 10 x Verdünnungsfaktor x Anzahl der Kolonien x Anzahl der Elektroporationsreaktionen.
HINWEIS: Um die Qualität der Bibliothek zu bestätigen, sequenzieren Sie mindestens 20 Kolonien durch Sanger-Sequenzierung. Die meisten Klone sollten eine Einfügung enthalten, und alle sollten eindeutig sein. - Inokulieren Sie 400-1.000 ml LB-Medium, das das geeignete Antibiotikum enthält, mit dem Rest der gepoolten Transformationen und inkubieren Sie über Nacht bei 37 °C, 180 U / min.
- Vorbereitung der Plasmidbibliothek
- Bereiten Sie aus der Nachtkultur einen Glycerinvorrat vor (mischen Sie gleiche Volumina der Bakterienkultur und 50% Glycerinlösung in nukleasefreiem Wasser und frieren Sie ihn bei -80 °C ein) und reinigen Sie die Plasmidbibliothek mit einem Plasmid-Maxi-Kit.
- Produktion einer viralen AAV-Bibliothek
- Bereiten Sie die virale Bibliothek wie zuvor beschrieben vor27. Transfektieren Sie die Plasmidbibliothek (pRep2Cap2_PI, Peptidinsert) zusammen mit einem Adeno-Helfer-Plasmid in HEK293T-Zellen unter Verwendung eines Transfektionsreagenzes wie Polyethylenimin (PEI).
- Sammeln Sie die Zellen nach 3 Tagen und unterziehen Sie sie drei Zyklen des Einfrierens und Auftauens. Reinigen Sie das Viruslysat mit Cäsiumchlorid-Gradienten-Ultrazentrifugation, gefolgt von einem Pufferaustausch zu PBS, und konzentrieren Sie schließlich die Viruspartikel.
- AAV-Vektortitration mittels dd-PCR
- 2 μl des AAV-Vektorstamms werden seriell in 198 μl nukleasefreiem Wasser verdünnt, um eine Endverdünnung von 1:106 zu erhalten. Mischen Sie jedes Mal gründlich mit einer 200-μl-Pipette. Fügen Sie ein No-Template-Steuerelement (NTC) als Negativkontrolle hinzu.
ANMERKUNG: Es können zusätzliche niedrigere oder höhere Verdünnungen ermittelt werden (1:105-1:107). - Bereiten Sie eine 20-fache Primer-Sonden-Mischung vor. Geben Sie 3,6 μl von jedem der 100-μM-Primer (vorwärts und rückwärts, Rep2 und ITR), jeweils 1 μl der 100-μM-dd-PCR-Sonden (Rep2 und ITR) und 3,6 μl nukleasefreies Wasser in ein 1,5-ml-Zentrifugenröhrchen.
HINWEIS: Die AAV-Bibliothek wird mit einem transgen-gezielten Primer-Sonden-Set (Rep2) gemessen, das mit einer FAM-markierten Sonde detektiert wurde, und einem ITR-gezielten Primer-Sonden-Set, das mit einer HEX-markierten Sonde detektiert wurde. - Bereiten Sie eine 22-μl-PCR-Reaktion vor, indem Sie 5,5 μl Probe, 1,1 μl 20-fache Primer-Sonden-Mischung, 11 μl dd-PCR-Supermix für Sonden (kein dUTP) und 4,4 μl nukleasefreies Wasser hinzufügen. Dies führt zu Konzentrationen von 900 nM bzw. 250 nM für die Primer bzw. die Sonde.
- Erzeugen Sie die Tröpfchen mit einem Tröpfchengenerator, übertragen Sie die Reaktion auf eine 96-Well-Platte, legen Sie die Platte in einen Thermocycler und führen Sie einen Denaturierungsschritt für 10 min bei 94 °C durch, gefolgt von 40 Zyklen von 30 s bei 94 °C und 1 min bei 58 °C. Anschließend wird die Polymerase für 10 min bei 98 °C hitzeinaktiviert und ein letzter Kühlschritt hinzugefügt. Lesen Sie die Reaktionen in einem Tröpfchenleser ab und fahren Sie mit der Analysefort 28.
- Öffnen Sie die gespeicherte dd-PCR-Plattendatei mit der Analysesoftware. Verwenden Sie das Schwellenwertwerkzeug auf der Registerkarte 1D-Amplitude (Fluoreszenzamplitude vs. Ereignisnummer), um die negativen und positiven Tröpfchen für jeden Kanal zu trennen, wobei Sie den NTC als Leitfaden verwenden, und exportieren Sie die Daten in eine CSV-Datei.
- Um die Vektorkonzentration zu berechnen, berechnen Sie zunächst den Korrekturfaktor CF mit der Formel:
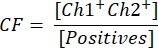
Mukoviszidose bestimmt den Anteil der Tröpfchen, die positiv für das Transgen sind [Positive], die sowohl für das Transgen als auch für das ITR [Ch1+ Ch2+] positiv sind, um den Nachweis funktioneller Vektorpartikel zu gewährleisten. Die endgültige Vektorkonzentration c kann nun mit der folgenden Gleichung berechnet werden: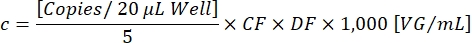
DF ist der Verdünnungsfaktor (1:10,5-1:10,7 , wie zuvor bestimmt). Die Kopien pro 20 μl/Well-Reaktion entsprechen 5 μl der verdünnten Probe. Der Faktor 1.000 korrigiert die Skala auf VG/ml (virales Genom/ml). Ein beispielhaftes Titrationsergebnis ist in Tabelle 1 und Abbildung 3 dargestellt.
- 2 μl des AAV-Vektorstamms werden seriell in 198 μl nukleasefreiem Wasser verdünnt, um eine Endverdünnung von 1:106 zu erhalten. Mischen Sie jedes Mal gründlich mit einer 200-μl-Pipette. Fügen Sie ein No-Template-Steuerelement (NTC) als Negativkontrolle hinzu.
- Analyse der AAV-Virusbibliothek durch NGS
- Amplifizieren Sie das 96-Nukleotid-Peptid-Insertionsfragment, indem Sie eine 20-μl-PCR-Reaktion unter Verwendung eines Korrekturlese-Polymerase-Kits einrichten (2x; siehe Abbildung 4). 1 μl AAV-Brühe, die 1 x 108 vg, 0,5 μl je 100 μM Primer (NGS_forward und NGS_reverse) enthält, und 10 μl der Enzymmischung zur Reaktion geben. Stellen Sie das Endvolumen mit nukleasefreiem Wasser auf 20 μl ein.
- Übertragen Sie die Reaktion auf einen Thermocycler und führen Sie einen Denaturierungsschritt für 3 min bei 98 °C durch, gefolgt von 30-35 Zyklen von 10 s bei 98 °C, 10 s bei 59 °C und 20 s bei 72 °C, gefolgt von 5 min bei 72 °C und einem abschließenden Abkühlschritt.
- Reinigen Sie die Proben mit einem PCR-Aufreinigungskit. Quantifizieren Sie die Konzentration in einem Spektralphotometer und führen Sie ein 3%iges Agarosegel durch, um die Reinheit und Fragmentgröße zu überprüfen.
- Verarbeiten Sie die PCR-Fragmente mit dem Bibliothekssystem für Proben mit geringer Komplexität gemäß den Anweisungen des Herstellers für die Erstellung einer NGS-Bibliothek. Führen Sie die Endreparaturreaktion mit 30 ng PCR-Fragment durch, gefolgt von einer Adapterligation und PCR-Amplifikation für 10 Zyklen. Verwenden Sie das PCR-Aufreinigungskit für die Aufreinigung der Reaktionen.
- Verarbeiten Sie die Endprodukte auf einem Bioanalysator, um die Größe und Reinheit zu überprüfen, indem Sie ein DNA-Reagenzien-Kit gemäß den Anweisungen des Herstellers verwenden.
- Quantifizieren Sie die Amplikons mit einem Fluorometer und fassen Sie sie zusammen. Quantifizieren Sie die endgültige gepoolte NGS-Bibliothek erneut auf einem Fluorometer (gemäß den Anweisungen des Herstellers) und überprüfen Sie die Qualität auf einem Bioanalysator.
- Sequenzieren Sie die NGS-Bibliotheken in einem Single-End-Modus (SE) mit einem 75-Takt-High-Output-Kit mit einer Leselänge von 84 und einem Index 1 von 8.
HINWEIS: Die Sequenzierung der Beispiele in diesem Artikel wurde in der GeneCore-Einrichtung des EMBL Heidelberg (http://www.genecore.embl.de/) durchgeführt. - Analysieren Sie die NGS-Sequenzierungsdaten mit Python 3 und Biopython. Die Dateien finden Sie unter https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 (alternativ unter https://doi.org/10.5281/zenodo.7032215). Die NGS-Analyse besteht aus zwei Schritten.
- Im ersten Schritt durchsuchen Sie die Sequenzdateien nach Sequenzen, die bestimmte Kriterien erfüllen (Vorhandensein von Erkennungssequenzen flankiert die Einfügestelle) (siehe Abbildung 4, Schritt 1.9.8.5.). Dies geschieht mithilfe eines Skripts (Script#1) und einer Konfigurationsdatei, die die erforderlichen Informationen bereitstellt. Sobald die richtige Sequenz identifiziert ist, extrahiert und speichert das Programm die Sequenz in der Ausgabedatei, bei der es sich um eine txt-Datei mit demselben Namen wie die Sequenzierungsdatei handelt.
- Der zweite Schritt ist die Analyse der Ausgabedateien. Die Sequenzen in der Bibliothek beginnen mit einem von sechs Nukleotiden (AGWggc, W = A/T) im Insert mit neun Aminosäuren. Basierend auf dieser Startsequenz wird das Peptid übersetzt. Dadurch werden die Ausgabedateien generiert, die die Peptidvarianten (PVs) enthalten.
- Bereiten Sie zwei Ordner vor: Skript und Daten. Kopieren Sie die gzip-komprimierten Dateien, die sich aus der Sequenzierung ergeben, in den Ordner Daten. Kopieren Sie in den Skriptordner die folgenden Dateien, Python-Datei: Skript#1_DetectionExtraction_JoVE_Py3.py; Python-Datei: Skript#2_PV_extraction_and_ranking_Py3.py; Konfigurationsdatei: Barcode_Script_JoVE.conf; und Look-up-Tabelle (LUT) Datei: Zuordnung.txt.
- Bearbeiten Sie vor dem Ausführen der Skripts die folgenden Dateien im Skriptordner. Öffnen Sie die Datei "Zuordnung.txt" und fügen Sie in zwei tabulatorgetrennten Spalten die Namen der gzip-Dateien (Spalte 1) und den gewünschten endgültigen Namen (Spalte 2; tabulatorgetrennte Werte) hinzu.
HINWEIS: Beispiel-TXT-Dateien befinden sich im GitHub-Ordner "PV_analysis_script". Die im GitHub-Ordner bereitgestellten Dateien sind für die Analyse von drei Beispieldaten aus der obigen Bibliothek vorbereitet: xaa.txt.gz, xab.txt.gz und xac.txt.gz. Die Ausgabedateien werden ebenfalls bereitgestellt. - Ändern Sie die folgenden Variablen in der Konfigurationsdatei "Barcode_Script_JoVE.conf":
my_dir = "~/Daten/"
filename_sample_file = "~/Script/Zuordnung.txt"
Die sequenzspezifischen Variablen: BCV_size = 27, BCVlinks = TCCAGGGCCAG, BCVrechts = GCCCAGG, BCVloc = 30, BCVmargin = 8, BCVleft_revcomp = GCCGCCTGGGC, BCVright_revcomp = CTGGCCC und BCVloc_revcomp = 41 (siehe Abbildung 4 für Details). - Verwenden Sie den folgenden Befehl, um die Variantensequenzerkennung und -extraktion aufzurufen:
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
HINWEIS: Die Ausgabe sind txt-Dateien mit den extrahierten DNA-Sequenzen und deren Anzahl der Lesevorgänge. Der Header dieser Datei enthält statistische Daten (d.h. die Gesamtzahl der Lesevorgänge und die extrahierten Lesevorgänge). Diese Daten werden in die nächsten Dateien übertragen. Diese txt-Daten sind die Eingabedateien für Script#2, in denen die DNA-Sequenzen übersetzt, eingestuft und analysiert werden. - Führen Sie die PV-Extraktion und -Analyse mit dem folgenden Befehl durch:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - Analysieren Sie die Textausgabedateien von Script#2. Die Ausgabedateien von Script#2 werden über die zweite Spalte der LUT in "Zuordnung.txt" mit Erweiterungen basierend auf der Art der Analyse benannt.
HINWEIS: Stellen Sie sicher, dass die drei Ausgabedateien statistische Daten in den ersten Zeilen ("# der gültigen PV-Lesevorgänge", "# der ungültigen PV-Lesevorgänge" und "# der eindeutigen PV-Lesevorgänge"), einer ersten Spalte mit dem Index jeder DNA-Sequenz aus den Eingabe-TXT-Dateien (Ausgabe von Skript #1) und den folgenden Spalten enthalten: (1) "... analyzed_all.csv": "Probe:" (DNA-Sequenz), "#" (Anzahl der Lesevorgänge), "Frw oder Rev" (Vorwärts- oder Rückwärtslesen) und "PVs" (übersetzte Peptidsequenz). Die ungültigen Sequenzen haben "NA" und "nicht gültig" in den letzten beiden Spalten. (2) "... analyzed_validSeq.csv": identisch mit der vorherigen Datei, gefiltert nach gültigen Sequenzen. (3) "... analyzed_PV.csv": "PVs" (übersetzte Peptidsequenz), "#" (Anzahl der Lesevorgänge) und "count" (die FRW- und Rev-Zählungen in den vorherigen Dateien werden zusammengeführt und die Anzahl wird mit 1 oder 2 angegeben). - Visualisieren Sie die Ausgabedateien mit verfügbarer Software basierend auf den Bedürfnissen des Benutzers.
2. Auswahl der zufälligen 7-meren Peptid-Display-Bibliothek für AAV2
- Verwenden Sie die AAV-Bibliothek nach der Quantifizierung und Qualitätskontrolle (Abschnitt 1) für die gerichtete Entwicklung in einem Modell Ihrer Wahl, um iterativ Kandidaten mit den gewünschten Eigenschaften auszuwählen (siehe Abbildung 5)16,18,21.
HINWEIS: Diese Kandidaten werden dann für die Generierung einer Barcode-Bibliothek verwendet, wie unten in Abschnitt 3 beschrieben.
3. Vorbereitung und Analyse der AAV-Kapsidbibliothek mit Barcode
HINWEIS: Überprüfen Sie nach der Identifizierung einer Reihe potenziell spezifischer und effizienter AAV-Kapside auf dem Peptidbildschirm die Funktionalität der identifizierten Peptidsequenzen und vergleichen Sie sie mit einer Reihe häufig verwendeter oder gut beschriebener Referenz-AAV-Kapsidvarianten. Dazu wird die Kapsidsequenz in ein Rep/Cap-Hilfskonstrukt ohne ITRs eingefügt.
- Herstellung einer AAV-Bibliothek mit Barcode
- Führen Sie eine rekombinante AAV-Produktion für jede Kapsidvariante unter Verwendung des Drei-Plasmid-Systems durch, wie zuvor beschrieben24.
HINWEIS: Zur Unterscheidung der verschiedenen Kapsidvarianten enthält das ITR-flankierte Reporter-Transgen-Plasmid einen eindeutigen Barcode mit einer Länge von 15 Nukleotiden. Der Barcode befindet sich in der 3'-UTR (untranslatierte Region) zwischen dem verstärkten gelb fluoreszierenden Protein (EYFP) und dem polyA-Signal (siehe Abbildung 6A). Die EYFP-Expression wird durch einen starken ubiquitären Cytomegalievirus (CMV)-Promotor gesteuert, der ausreichende Mengen an RNA-Transkripten bereitstellt. - Entwerfen Sie Barcodes mit einer Länge von 15 Nukleotiden mit Homopolymeren von weniger als drei Nukleotiden, einem GC-Gehalt von <65 %29 und einem Hamming-Abstand von mehr als vier Nukleotiden24.
- Stellen Sie jedes Kapsid separat in Kombination mit einem Transgenplasmid her, das einen eindeutigen Barcode trägt. Auf diese Weise wird jede Kapsidvariante mit einem eindeutigen Barcode versehen, der eine spezifische Nachverfolgung ermöglicht (siehe Abbildung 6B).
- Führen Sie eine rekombinante AAV-Produktion für jede Kapsidvariante unter Verwendung des Drei-Plasmid-Systems durch, wie zuvor beschrieben24.
- AAV-Vektortitration mittels dd-PCR
- Führen Sie die AAV-Titration wie zuvor in Abschnitt 1.8 beschrieben durch, indem Sie das Rep2-Primerpaar durch das YFP-Primerpaar ersetzen.
- Quantifizieren Sie die einzelnen AAV-Produktionen und fassen Sie gleiche Mengen jeder Produktion zusammen, um die endgültige Barcode-Bibliothek zu generieren.
- Quantifizieren Sie die endgültige Bibliothek erneut, um die endgültige Konzentration und Qualität zu überprüfen (siehe Abbildung 7).
- In-vivo-Anwendung der Barcode-AAV-Bibliothek
- Wenden Sie die barcodierte AAV-Bibliothek systemisch auf das Modellsystem Ihrer Wahl an (z. B. systemisch in Mäusen24).
- Sammeln Sie ON- und OFF-Zielgewebe (z. B. Leber, Lunge, Herz, Zwerchfell, glatte Muskulatur, Zwölffingerdarm, Bauchspeicheldrüse, Dickdarm, Bizeps, Eierstöcke, Magen, Innenohr, Niere, Bauchaorta, thorakale Aorta, Gehirn, braunes und weißes Fett und Milz) oder Zelltypen basierend auf dem Experiment. Frieren Sie sie bei -80 °C ein, extrahieren Sie die DNA/RNA und wenden Sie die NGS-Quantifizierungsanalyse an, wie im nächsten Abschnitt beschrieben.
- DNA/RNA-Extraktion
- Extrahieren Sie die DNA und RNA aus den interessierenden Geweben mit dem DNA/RNA Mini Kit.
- Legen Sie ein kleines Stück des interessierenden Gewebes (1 mm3, ca. 5 mg) in ein 2 ml Reaktionsröhrchen.
- 350 μl Lysepuffer, gemischt mit β-Mercaptoethanol (1%) und 5 mm Stahlkügelchen, in das Gewebe geben (Proben mit β-Mercaptoethanol unter einem Abzug behandeln).
- Homogenisieren Sie das Gewebe in einem TissueLyser für 45 s bei 40 Hz.
- 10 μl Proteinase K (10 mg/ml) zugeben und 15 min bei 55 °C unter Schütteln bei 400 U/min inkubieren.
- Zentrifugieren Sie bei 20.000 x g für 3 min bei Raumtemperatur, sammeln Sie den Überstand und fahren Sie mit dem Protokoll des Herstellers des DNA/RNA-Kits fort.
- Teilen Sie den Waschschritt in zwei Schritte mit 350 μl Waschpuffer in jedem Schritt auf. Zwischen diesen Waschschritten wird die restliche DNA auf der Säule mit RNase-freier DNase I aufgeschlossen. 80 μl der DNase I-Lösung, die gemäß den Anweisungen des Herstellers hergestellt wurde, auf die Säule geben und 15 Minuten bei Raumtemperatur inkubieren.
- Eluieren Sie RNA/DNA aus der Säule mit nukleasefreiem Wasser. Lagern Sie die isolierte RNA bei -80 °C und die gDNA bei -20 °C.
- cDNA-Synthese
- Unterziehen Sie die RNA-Proben vor der Reverse-Transkriptionsreaktion einer weiteren DNase I-Behandlung von 15-30 Minuten (zur vollständigen Entfernung der kontaminierenden DNA aus den RNA-Proben). Fügen Sie 1 μl der DNase I-Lösung, 4 μl Puffer (im Lieferumfang des Kits enthalten) und nukleasefreies Wasser zu einem Endvolumen von 40 μl bis 212 ng RNA hinzu. 30 min bei Raumtemperatur inkubieren und bei 70 °C 10 min inaktivieren.
- Synthetisieren Sie cDNA unter Verwendung von 150 ng RNA unter Verwendung eines Kits gemäß den Anweisungen des Herstellers. Fügen Sie Kontrollen ohne die reverse Transkriptase hinzu, um sicherzustellen, dass keine kontaminierende virale DNA aus der Probe stammt. Die cDNA wird bei -20 °C gelagert.
HINWEIS: Die Menge der Eingangs-RNA für eine optimale reverse Transkription kann je nach Gewebetyp und der erwarteten Transduktionseffizienz im jeweiligen Gewebe variieren.
- Analyse der AAV-Virusbibliothek (in-vivo) von NGS
- Um eine hohe Sequenzierungstiefe zu geringen Kosten zu erreichen, führen Sie die NGS-Sequenzierung über Illumina durch, wie zuvor beschrieben (Abschnitt 1.9). Verstärken Sie die Barcode-Sequenz und verbinden Sie dann die Sequenzieradapter mit dem Amplikon.
- Aufgrund der kurzen Leselänge und der Ligatur der Sequenzierungsadapter auf beiden Seiten des Amplikons ist beim Entwerfen darauf zu achten, dass das Amplikon ausreichend klein ist, um das Vorhandensein der Barcode-Sequenz innerhalb des gelesenen NGS sicherzustellen. Für die Sequenzierung der Barcodes innerhalb der viralen Genome und der viralen Transkripte ist das PCR-Amplikon auf eine Länge von 113 bp ausgelegt (siehe Abbildung 8).
- Verstärken Sie den Barcode-Bereich mit den Primern BC-seq vorwärts und BC-seq rückwärts. Bereiten Sie die folgende PCR-Reaktion vor: 0,5 μl Hi-Fidelity-DNA-Polymerase, 10 μl 5x Puffer, 0,25 μl jedes 100-μM-Primers (BC-seq fw/BC-seq rv) und 1 μl 10 mM dNTPs. Verwenden Sie 25 ng der cDNA oder DNA/Reaktion als Vorlage und stellen Sie das Endvolumen mit nukleasefreiem Wasser auf 50 μl ein.
- Bereiten Sie den PCR-Mastermix unter einer sauberen PCR-Haube vor, um eine Kontamination zu vermeiden. Verwenden Sie die folgenden Zyklusbedingungen: 30 s bei 98 °C, gefolgt von 40 Zyklen bei 98 °C für 10 s und 72 °C für 20 s und einem letzten 5-Minuten-Schritt bei 72 °C.
- Fügen Sie PCR-Kontrollen hinzu, um das Fehlen kontaminierender DNA im PCR-Mastermix zu bestätigen. Für die cDNA-Proben sind die Kontrollen ohne reverse Transkriptase einzuschließen. Fügen Sie schließlich ein Beispiel mit der AAV-Eingabebibliothek hinzu. Diese Informationen werden verwendet, um die Normalization_Variant.txt Datei zu generieren, die in der Analyse verwendet wird.
- Überprüfen Sie die Größe des PCR-Fragments jeder Probe durch Gelelektrophorese vor der PCR-Aufreinigung. Letzteres wird entweder durch die Verwendung von kommerziell erhältlichen Magnetkügelchen oder säulenbasierten DNA-Aufreinigungssystemen erreicht (siehe Materialtabelle).
- Bereiten Sie die NGS-Bibliothek mit dem Bibliothekssystem für Stichproben geringer Komplexität gemäß den Anweisungen des Herstellers vor, wie zuvor in Abschnitt 1.9 beschrieben.
- Bestimmen Sie die DNA-Konzentration über das dsDNA HS Kit und analysieren Sie die Qualität der Bibliothek wie zuvor beschrieben (Abschnitt 1.9.6), gefolgt von Pooling. Quantifizieren Sie die gepoolte Bibliothek auf einem Fluorometer und bewerten Sie die Qualität auf einem Bioanalysator.
- Führen Sie die NGS-Sequenzierung wie in Abschnitt 1.9.7 beschrieben durch.
- Quantifizieren Sie mittels qPCR die Kopienzahl des Transgens (virale Genome) und des Housekeeping-Gens, um die Verteilung der gepoolten Bibliothek zwischen Geweben oder Organen auf der DNA zu beurteilen.
- Richten Sie eine 30 μL qPCR-Reaktion wie folgt ein, um die Kopienzahl von EYFP (Transgen) und GAPDH (Glycerinaldehyd-3-phosphat-Dehydrogenase, Housekeeping-Gen) zu bestimmen:
- Bereiten Sie eine 60-fache Primer-Sonden-Mischung für EYFP vor (1,5 μM YFP_fw, 1,5 μM YFP_rv und 0,6 μM YFP_probe; siehe Materialtabelle). Verwenden Sie die GAPDH-Primer-Sonden-Mischung (siehe Materialtabelle), um die Kopienzahl des Haushälter-Gens zu bestimmen. Richten Sie die Reaktion auf Eis ein.
- Bereiten Sie einen PCR-Mastermix (15 μl, siehe Materialtabelle) vor und fügen Sie 60x Primer/Sonden-Mix (0,5 μl) für alle Proben und Standards hinzu (um die Kopienzahlen für die Standards zu berechnen, verwenden Sie den folgenden Link: http://cels.uri.edu/gsc/cndna.html). Richten Sie die Reaktion auf Eis ein.
- Übertragen Sie 15,5 μl des Mastermixes in eine 96-Well-Platte und geben Sie 14,5 μl Probe (75 ng Gesamt-DNA-Konzentration) oder Standard in die jeweilige Vertiefung. Die 96-Well-Platte mit Folie versiegeln, wirbeln und kurz schleudern.
- Übertragen Sie 10 μl jeder Probe in Duplikaten in eine 384-Well-Platte. Die Platte mit Folie verschließen und bei 800 x g 5 min bei 4 °C schleudern.
- Das Reaktionsgemisch wird in einem Thermocycler bei einer Anfangstemperatur von 50 °C für 2 min inkubiert, gefolgt von einem ersten Aktivierungsschritt von 10 min bei 95 °C. Durchführung von 40 Denaturierungszyklen bei 95 °C für 15 s und Glühen/Verlängern bei 60 °C für 1 min24.
- Um die Anzahl der diploiden Genome (dg) zu erhalten, verwenden Sie die GAPDH-Kopienzahl und teilen Sie sie durch zwei. Nehmen Sie dann den Wert der EYFP-Kopienzahl und dividieren Sie ihn durch die Anzahl der dg, was zu Vektorgenomen pro diploidem Genom (vg/dg) führt. Verwenden Sie diesen Wert, um die Normalization_Organ.txt Datei für die bioinformatische Analyse zu generieren.
- Führen Sie die Analyse der NGS-Sequenzierungsdaten durch, wie Weinmann et al.24, mit benutzerdefiniertem Code in Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Der Workflow umfasst die Erkennung von Barcode-Sequenzen, die durch flankierende Sequenzen, deren Länge und Position geführt werden (Script#1_BarcodeDetection.py), sowie die Analyse der Barcode-Anreicherung und -Verteilung über den Satz von Geweben (Script#2_BarcodeAnalysis.py).
- Barcodes erkennen und AAV-Varianten zuordnen. Legen Sie die Sequenzierungsdaten als archivierte fastq-Dateien in einem Verzeichnis ab (z.B. "Data_to_analyze"). Die Sequenzierungsdatendatei für die Eingabebibliothek ist in diesem Verzeichnis enthalten und wird nur zum Berechnen der Kapsidanteile in der Eingabebibliothek verwendet.
- Erstellen Sie vor dem Ausführen des Skripts zwei tabulatorgetrennte Textdateien: die Kapsidvariantendatei (siehe Beispieldatei "Varianten.txt) mit den Barcode-Sequenzen, die den AAV-Kapsidvariantennamen zugewiesen sind, und die Kontaminationsdatei (siehe "Kontaminationen.txt") mit Barcode-Sequenzen, die von einer möglichen Kontamination stammen (andere im Labor verfügbare Barcodes, die zur Kontamination beitragen).
- Bearbeiten Sie abschließend die Konfigurationsdatei "Barcode_Script.conf" so, dass sie die folgenden Informationen enthält: Pfad zum Ordner mit Sequenzierungsdaten (z. B. "Data_to_analyze"), Reihenfolge der flankierenden Bereiche der Barcodes, deren Position und Fenstergröße für die Barcode-Erkennung (ähnlich wie bei 1.9.8.5, siehe Abbildung 8).
- Verwenden Sie den folgenden Befehl, um die Barcode-Erkennung mit den angegebenen Pfaden zu Skript#1_BarcodeDetection.py und Konfigurationsdateien aufzurufen:
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
HINWEIS: Die Ausgabe der Script#1_BarcodeDetection.py Ausführung besteht aus Textdateien mit Lesezahlen pro Kapsidvariante sowie der Gesamtzahl der aus den Rohdaten wiederhergestellten Lesevorgänge. - Bewerten Sie die Verteilung von barcodierten AAV-Kapsiden zwischen Geweben oder Organen, indem Sie Script#2_BarcodeAnalysis.py zusammen mit den folgenden txt-Dateien ausführen:
- Ordnen Sie in der Datei "Zuordnung.txt jeder txt-Datei, die aus dem Barcode-Erkennungslauf gewonnen wird, einen Gewebe-/Organnamen zu: Namen der txt-Dateien in der ersten Spalte und entsprechende Gewebe-/Organnamen in tabulatorgetrennter Zuordnung.
HINWEIS: Ein Beispiel finden Sie im Ordner "Example" (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). Bemerkenswert ist, dass der Gewebe-/Organname Zeichen enthalten kann, die die cDNA- oder gDNA-Messung und die Anzahl der biologischen Replikate (M1, M2 usw.) definieren. - Erstellen Sie eine "Organs.txt"-Textdatei mit der Namensliste für ON- und OFF-Zielorgane, die den Namen entsprechen, die in der Zuordnungsdatei "Zuordnung.txt" angegeben sind (siehe Ordner "Beispiel": https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022).
- Erstellen Sie tabulatorgetrennte Textdateien mit "Normalization_Organ.txt" und "Normalization_Variant.txt" mit normalisierten Werten für alle Kapsidvarianten und alle Organe/Gewebe. Schreiben Sie in die erste Spalte der Datei "Normalization_Organ.txt" die Namen der einzelnen Organe (wie in der Zuordnung.txt) und in die zweite Spalte die Normalisierungswerte für die entsprechenden Gewebe, die in Abschnitt 3.6.11 generiert wurden.
- Füllen Sie die erste Spalte der Datei "Normalization_Variant.txt" mit der Liste der Kapsidnamen und die zweite Spalte mit den normalisierten Werten der Leseanzahl für jedes Kapsid in der gepoolten Bibliothek (die Normalisierung kann basierend auf der txt-Ausgabedatei für die Eingabebibliothek berechnet werden, die sich aus dem ersten Skript ergibt).
- Bearbeiten Sie die Konfigurationsdatei, indem Sie die vollständigen Pfade zu allen oben genannten zusätzlichen Dateien angeben. Führen Sie Skript#2_BarcodeAnalysis.py wie folgt aus:
>python3 /script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
HINWEIS: Das Barcode-Analyseskript gibt mehrere Dateien aus: Textdateien mit relativen Konzentrationswerten (RC) der Kapsidverteilung in verschiedenen Geweben basierend auf mehreren zuvor beschriebenen Normalisierungsschritten und die Tabellenkalkulationsdatei, die Textdateidaten zu zusammengeführten Matrixdaten kombiniert. Letzteres kann für die Clusteranalyse und Visualisierung verwendet werden. - Visualisieren Sie die Daten und führen Sie eine Clusteranalyse der Matrixdaten durch, um Kapsideigenschaften zu unterscheiden und ihre Ähnlichkeiten basierend auf RC-Profilen über Gewebe hinweg zu bewerten. Verwenden Sie das zusätzliche Skript PCA_heatmap_plot. R im Repository abgelegt:
>Rscript --vanilla ~/PCA. R ~/relative Konzentration.xls
HINWEIS: Das Skript verwendet relative concentration.xls Dateien als Eingabe und generiert zwei Diagramme der hierarchischen Cluster-Heatmap und der Hauptkomponentenanalyse (PCA). - Um Diagramme (Achsen der Heatmap, Hauptkomponenten von PCA) oder PNG-Parameter (Farbe, Größe, Beschriftung) zu ändern, öffnen Sie das R-Skript und befolgen Sie die Anweisungen in den kommentierten Abschnitten.
- Ordnen Sie in der Datei "Zuordnung.txt jeder txt-Datei, die aus dem Barcode-Erkennungslauf gewonnen wird, einen Gewebe-/Organnamen zu: Namen der txt-Dateien in der ersten Spalte und entsprechende Gewebe-/Organnamen in tabulatorgetrennter Zuordnung.
Ergebnisse
Generierung einer AAV2-Peptid-Display-Bibliothek. Als erster Schritt zur Selektion von künstlich hergestellten AAVs wird die Generierung einer Plasmidbibliothek beschrieben. Der Peptideinsatz wird unter Verwendung von entarteten Primern hergestellt. Die Reduzierung der Kombination von Codons bei 64 auf 20 hat den Vorteil, dass Stoppcodons eliminiert und die NGS-Analyse erleichtert werden, indem die Bibliotheksvielfalt auf DNA-, aber nicht auf Proteinebene verringert wird. Das Oligonukleotid-Insert wird...
Diskussion
In diesem Protokoll werden die Schritte beschrieben, die für das AAV-Kapsid-Engineering für die Peptidanzeige und für das Screening von AAV-Bibliotheken mit Barcodes sowie für die bioinformatische Analyse der Bibliothekszusammensetzung und der Kapsidleistung erforderlich sind. Dieses Protokoll konzentriert sich auf die Schritte, die die bioinformatische Analyse dieser Art von Bibliotheken erleichtern, da die meisten virologischen Laboratorien in ihren Programmierkenntnissen hinterherhinken, um ihren Kenntnissen in mo...
Offenlegungen
D.G. ist Mitbegründer der AaviGen GmbH. D.G. und K.R. sind Erfinder einer anhängigen Patentanmeldung im Zusammenhang mit der Erzeugung von immunumgehenden AAV-Kapsidvarianten. Der Rest der Autoren hat nichts zu offenbaren.
Danksagungen
D.G. freut sich sehr über die Unterstützung durch die Deutsche Forschungsgemeinschaft (DFG) durch die DFG-Sonderforschungsbereiche SFB1129 (Projektnummer 240245660) und TRR179 (Projektnummer 272983813) sowie durch das Deutsche Zentrum für Infektionsforschung (DZIF, BMBF; TTU-HIV 04.819).
Materialien
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
Referenzen
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621 (2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756 (2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075 (2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041 (2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432 (2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819 (2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899 (2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272 (2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200 (2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten