Method Article
Ein Gut-on-a-Chip-Modell zur Untersuchung der Achse Darmmikrobiom und Nervensystem
In diesem Artikel
Zusammenfassung
neuroHuMiX ist ein fortschrittliches Gut-on-a-Chip-Modell zur Untersuchung der Interaktionen von bakteriellen, epithelialen und neuronalen Zellen unter proximalen und repräsentativen Co-Kulturbedingungen. Dieses Modell ermöglicht es, die molekularen Mechanismen zu entschlüsseln, die der Kommunikation zwischen dem Darmmikrobiom und dem Nervensystem zugrunde liegen.
Zusammenfassung
Der menschliche Körper wird von mindestens der gleichen Anzahl von mikrobiellen Zellen besiedelt, wie er aus menschlichen Zellen besteht, und die meisten dieser Mikroorganismen befinden sich im Darm. Obwohl das Zusammenspiel zwischen dem Darmmikrobiom und dem Wirt ausgiebig untersucht wurde, ist die Interaktion des Darmmikrobioms mit dem enterischen Nervensystem weitgehend unbekannt. Bis heute gibt es kein physiologisch repräsentatives In-vitro-Modell zur Untersuchung der Interaktionen zwischen Darmmikrobiom und Nervensystem.
Um diese Lücke zu schließen, haben wir das Human-Microbial Crosstalk (HuMiX) Gut-on-Chip-Modell weiterentwickelt, indem wir induzierte pluripotente Stammzellen aus enterischen Neuronen in das Gerät eingeführt haben. Das daraus resultierende Modell "neuroHuMiX" ermöglicht die Co-Kultur von Bakterien-, Epithel- und neuronalen Zellen über mikrofluidische Kanäle hinweg, die durch semipermeable Membranen getrennt sind. Trotz der Trennung der verschiedenen Zelltypen können die Zellen über lösliche Faktoren miteinander kommunizieren und gleichzeitig die Möglichkeit bieten, jeden Zelltyp separat zu untersuchen. Dieser Aufbau ermöglicht erste Einblicke, wie sich das Darmmikrobiom auf die enterischen neuronalen Zellen auswirkt. Dies ist ein wichtiger erster Schritt, um die Achse zwischen Darmmikrobiom und Nervensystem zu untersuchen und zu verstehen.
Einleitung
Das menschliche Darmmikrobiom spielt eine entscheidende Rolle für die menschliche Gesundheit und Krankheit. Es wurde in den letzten anderthalb Jahrzehnten ausgiebig untersucht, und seine potenzielle Rolle bei der Modulation von Gesundheit und Krankheit ist inzwischen erwiesen1. Es wurde postuliert, dass eine Störung des Mikrobioms, die zu einer unausgewogenen mikrobiellen Gemeinschaft (Dysbiose) führt, an der Pathogenese vieler chronischer Erkrankungen wie Fettleibigkeit, entzündlicher Darmerkrankungen und Darmkrebs oder sogar neurodegenerativer Erkrankungen wie Parkinson beteiligt ist 2,3.
Obwohl das menschliche Darmmikrobiom mit neurologischen Erkrankungen in Verbindung gebracht wurde, ist immer noch unklar, wie das Darmmikrobiom mit dem enterischen Nervensystem kommuniziert und es beeinflusst. Da das menschliche enterische Nervensystem für eine unmittelbare Untersuchung nicht leicht zugänglich ist, wurden bisher Tiermodelle in Experimenten verwendet4. Angesichts der offensichtlichen Unterschiede zwischen Tiermodellen und Menschen5 ist die Entwicklung von In-vitro-Modellen, die den menschlichen Darm nachahmen, jedoch von unmittelbarem Interesse. In diesem Zusammenhang hat uns das aufkeimende und fortschreitende Feld der humanen induzierten pluripotenten Stammzellen (iPSCs) ermöglicht, repräsentative enterische Neuronen (ENs) zu erhalten6. iPSC-abgeleitete ENs ermöglichen die Untersuchung des enterischen Nervensystems in In-vitro-Kulturmodellen, wie z. B. Zellkultureinsätzen, Organoiden oder Organs-on-a-Chip 7,8.
Das Human-Microbial Crosstalk (HuMiX)-Modell ist ein Gut-on-a-Chip-Modell, das den menschlichen Darm nachahmt9. Das ursprüngliche HuMiX-Modell (im Folgenden als Ausgangsgerät bezeichnet) nahm Epithelzellen (Caco-2) und Bakterienzellenauf 10,11. Um jedoch die Verbindung zwischen Darmmikrobiom und Nervensystem zu untersuchen, wurden auch iPSC-abgeleitete ENs6 in das System eingeführt (Abbildung 1). Die proximale Kokultur von neuronalen, epithelialen und bakteriellen Zellen ermöglicht die individuelle Analyse der verschiedenen Zelltypen und die Untersuchung der Interaktionen zwischen den verschiedenen Zelltypen in einer Umgebung, die die des menschlichen Darms nachahmt.
In den letzten Jahren wurden Fortschritte bei der Entwicklung von Modellen gemacht, um Organe auf physiologisch repräsentativere Weise zu untersuchen, indem Organs-on-a-Chip-Modelle (z. B. Gut-on-a-Chip) verwendet werden. Diese Modelle sind repräsentativer für die menschliche Darmumgebung, da sie eine konstante Nährstoffzufuhr und Abfallentsorgung sowie eine Echtzeitüberwachung von z. B. Sauerstoffgehalt oder Barriereintegrität ermöglichen 8,12. Diese Modelle ermöglichen es insbesondere, die Auswirkungen von Darmbakterien auf Wirtszellen zu untersuchen. Um jedoch mit Organs-on-a-Chip die Zusammenhänge zwischen dem Darmmikrobiom und dem Nervensystem untersuchen zu können, müssen neuronale Zellen in solche Systeme integriert werden. Ziel der Weiterentwicklung von HuMiX und der Etablierung des neuroHuMiX-Systems (im Folgenden als Gerät bezeichnet) war es daher, ein Gut-on-a-Chip-Modell zu entwickeln, das enterische neuronale Zellen in proximaler Co-Kultur mit Darmepithelzellen und Bakterien umfasst.
Protokoll
1. Zellkultur und Sortierung
- Induzierte pluripotente Stammzell-abgeleitete enterische Neuronen
HINWEIS: Kultur-iPS-Zellen 6 Wochen vor Beginn eines Laufs. Das Differenzierungsprotokoll für die ENs wurde von Fattahi et al.6 übernommen.- Kultivieren Sie die iPS-Zellen auf einer mit Matrixgel beschichteten 6-Well-Platte in 2 ml/Well iPSC-Kulturmedium, das mit 1% Penicillin-Streptomycin (P/S) ergänzt ist.
- Bei einer Konfluenz von 80 % bis 90 % werden die Zellen in einer gelbeschichteten Matrixplatte mit demselben Medium durchgelassen und bei 37 °C, 5 % CO2 und 90 % relativer Luftfeuchtigkeit (RH) inkubiert.
- Für die iPSC-Ableitung werden die Zellen nach zwei Durchgängen nach dem Auftauen bei 80%-90% Konfluenz in eine Matrixgel-beschichtete 6-Well-Platte mit einer Dichte von 100.000 Zellen/Well ausgesät. In dem oben beschriebenen Medium + ROCK-Inhibitor Y-27632 (1:2.000) für 24 h bei 37 °C inkubieren.
- Nach 24 Stunden ist das überstehende Medium gemäß Tabelle 1 durch das Medium Tag 0 zu ersetzen. Fügen Sie eine Kontrollvertiefung hinzu, die als Negativkontrolle während der Zellsortierung für den Ableitungsprozess verwendet werden soll. Behalten Sie die Kontrolle über die Medienzusammensetzung von Tag 0 für den gesamten Ableitungszeitraum. Wechseln Sie das Medium jeden zweiten Tag von den Tagen 2 bis 10 gemäß Tabelle 1.
- Sortieren Sie die Zellen an Tag 11 nach CD49d-positiven Zellen, die in den folgenden Schritten dieses Protokolls verwendet werden.
- Gruppieren Sie die zu sortierenden Zellen basierend auf CD49d-positiven Zellen sowie die Kontrollvertiefung. Die Zellen werden 3 min bei 300 × g zentrifugiert. Resuspendieren Sie das Zellpellet in 2 % Rinderserumalbumin (BSA) + 1 % P/S in 1x phosphatgepufferter Kochsalzlösung (PBS).
- Teilen Sie jede Zellcharge (gepoolt und Kontrollen) in zwei Teile: Färben Sie eine Fraktion mit einem Anti-Human-CD49d-Antikörper und die andere Fraktion mit einem Isotyp-Kontrollantikörper.
- Die Hauptzellpopulation wird mit einem Gate versehen und dann mit einem Gate für einzelne Zellen, auf deren Grundlage die Zellen ausgewählt werden, die CD49d auf ihrer Oberfläche präsentieren. Sammeln Sie diese Zellen für den nächsten Schritt der Differenzierung.
HINWEIS: Normalerweise sind 30%-40% der sortierten Zellen CD49d-positiv.
- Übertragen Sie zwei bis vier Millionen der sortierten Zellen für 4 Tage auf eine 6-Well-Platte einer Platte mit extrem niedrigem Anhangsmaterial in N2B27-Medien (siehe Tabelle 2) + FGF2 + CHIR, um die Bildung von Sphäroiden zu ermöglichen.
- Am 15. Tag werden die Sphäroide auf einer mit Poly-L-Ornithin/Laminin/Fibronektin (P/L/F)-beschichteten 6-Well-Platte in 2 ml/Well N2B27-Medien mit GDNF und Ascorbinsäure (AA) neu beschichtet. Tauschen Sie das Medium alle 2-3 Tage aus.
HINWEIS: Beschichtungsverhältnis: Poly-L-Ornithin, 15 μg/ml; Laminin, 2 μg/ml; Fibronektin, 2 μg/ml in 1x PBS. - Nach 3 Wochen verwenden Sie die Zellen zur Inokulation im Gerät.
- Caco-2
HINWEIS: Tauen Sie Caco-2-Zellen mindestens 1 Woche vor einem Lauf auf.- Einen T75-Kolben mit 1 × 106 Caco-2-Zellen in RPMI 1640 Glutaminpräparat + HEPES + 10% fötales Kälberserum (FBS) aussäen; bei 37 °C, 5 % CO2 und 90 % rF inkubieren.
HINWEIS: Verwenden Sie für diese Experimente idealerweise Caco-2 beim ersten oder zweiten Durchgang nach dem Auftauen.
- Einen T75-Kolben mit 1 × 106 Caco-2-Zellen in RPMI 1640 Glutaminpräparat + HEPES + 10% fötales Kälberserum (FBS) aussäen; bei 37 °C, 5 % CO2 und 90 % rF inkubieren.
2. Vorbereitung des HuMiX-Laufs
- Vorbereitung und Beschichtung von Polycarbonat-Deckeln
- Autoklavieren Sie ein Paar Deckel aus Polycarbonat (PC) (Abbildung 2) zusammen mit vier Schrauben unter Verwendung des anfänglichen Instrumentenzyklus des Geräts (siehe Tabelle 3 für Details).
- Setzen Sie in einer Biosicherheitswerkbank vier Schrauben in jede Ecke des unteren PC-Deckels ein (siehe Abbildung 3) und geben Sie sie für einen zweistufigen Beschichtungsprozess in eine sterile quadratische Petrischale.
- Geben Sie am ersten Tag 2 ml 1,5 % Poly-L-Ornithin in PBS (1x) in die Mitte des PC-Deckels. Über Nacht bei 37 °C, 5 % CO2 und 90 % relativer Luftfeuchtigkeit inkubieren.
- Entfernen Sie am nächsten Tag die Lösung und ersetzen Sie sie durch eine Lösung, die 0,2 % Laminin und 0,2 % Fibronektin in PBS (1x) enthält, um die Oberfläche der neuronalen Zellkammer zu bedecken. Den Deckel über Nacht bei 37 °C, 5 % CO2 und 90 % relativer Luftfeuchtigkeit inkubieren.
- Bewahren Sie nach der Inkubation die Beschichtungslösung auf dem PC-Deckel auf und verschließen Sie die Petrischale mit einer Siegelfolie. Bewahren Sie sie bis zur Verwendung bei 4 °C auf.
- Entfernen Sie die Beschichtungslösung vor Gebrauch und lassen Sie sie 30 Minuten lang in einer Biosicherheitswerkbank an der Luft trocknen, bis die Beschichtung vollständig getrocknet ist.
- Dichtungsvorbereitung und -beschichtung
HINWEIS: In diesem Abschnitt wird beschrieben, wie Dichtungen vorbereitet werden, indem semipermeable Membranen auf Silikondichtungen geklebt werden. Nach einem ersten Autoklavierschritt werden die Dichtungen mit Kollagen oder Muzin beschichtet, um die Adhäsion von Darmepithelzellen oder Bakterien zu ermöglichen. Die mit Schleim beschichtete Membran ahmt die Schleimbarriere nach, die im menschlichen Darm vorhanden ist.- Befestigen Sie eine porengroße Polycarbonatmembran von 1 μm an der Kollagendichtung und eine porengroße Polycarbonatmembran von 50 nm zwischen den unteren und oberen Sandwichdichtungen. Autoklavieren Sie die Dichtungen mit den angebrachten Membranen unter Verwendung des Instrumentenzyklus des Anfangsgeräts (siehe Tabelle 3).
- Um die Membranen zu beschichten, legen Sie jede Dichtung in eine sterile quadratische Schale.
HINWEIS: Die Beschichtung erfolgt unter sterilen Bedingungen in einer Biosicherheitswerkbank.- Für die Kollagendichtung 3 ml Kollagen (50 μg/ml) in die Membran geben und 3 h bei 37 °C inkubieren.
HINWEIS: Achten Sie darauf, die Membran nicht mit der Pipettenspitze zu berühren, wenn Sie das Kollagen hinzufügen, um eine Beschädigung der Membran zu vermeiden. - Für die Muzinbeschichtung legen Sie die Sandwichdichtung mit der oberen Dichtungsseite nach oben in eine Petrischale, um die Oberseite der Membran mit 3 ml Mucin (0,025 mg/ml) zu beschichten. Die Dichtungen 1 h bei 37 °C inkubieren.
- Für die Kollagendichtung 3 ml Kollagen (50 μg/ml) in die Membran geben und 3 h bei 37 °C inkubieren.
- Nach der Inkubationszeit werden die Kollagen- und Muzinlösungen aus den jeweiligen Dichtungen abgesaugt und die Membran 30 Minuten lang in einer Biosicherheitswerkbank an der Luft getrocknet. Verschließen Sie die Schalen, verschließen Sie sie mit Laborsiegelfolie und lagern Sie die Dichtungen bis zur Verwendung bei 4 °C.
- Schlauch-Montage
HINWEIS: In diesem Abschnitt wird beschrieben, wie der Schlauch für die Perfusion des Geräts montiert wird. Autoklavieren Sie vor dem Zusammenbau alle Teile und/oder kaufen Sie diejenigen, die einzeln in einer sterilen Verpackung verpackt sind. Legen Sie alle Komponenten in eine Biosicherheitswerkbank.- Verwenden Sie drei Schlauchleitungen für jedes Gerät und für jede Schlauchleitung, ein Stück Pumpenschlauchleitung, zwei Stück lange Marprene-Schläuche (20 cm), zwei Stück kurze Marprene-Schläuche (8 cm), eine 40-mm-Nadel für die Ausflussflasche, eine 120-mm-Nadel für die 250-ml-Zuflussflaschen oder eine 80-mm-Nadel für kleinere Zuflussflaschen. drei männliche sowie sieben weibliche Luer-zu-Widerhaken-Anschlüsse, drei Luer-Adapter und zwei Dreiwege-Absperrventile.
- Montieren Sie die Schlauchleitungen von links nach rechts mit den oben aufgeführten autoklavierten und sterilisierten Komponenten (Abbildung 4).
HINWEIS: Besprühen Sie jede Komponente zwischen den einzelnen Verbindungsschritten mit 70 % Ethanol.
- Medienaufbereitung
HINWEIS: In diesem Abschnitt wird beschrieben, wie das Medium für die verschiedenen Zelltypen im Gerät vorbereitet wird und wie das Medium steril in Serumflaschen überführt wird.- Ergänzen Sie RPMI 1640 mit 10% 0,22 μm gefiltertem, hitzeinaktiviertem FBS. Ergänzen Sie die N2B27-Medien frisch mit GDNF und AA.
- Füllen Sie das Medium unter sterilen Bedingungen in Serumflaschen um. Übertragen Sie 200 ml RPMI 1640-Medien in 250-ml-Flaschen und 30 ml N2B27-Medien in 50-ml-Flaschen.
- Bakterielles Medium (RPMI 1640 + 10 % FBS + 5 % De Man-, Rogosa- und Shapre-Kulturmedium [MRS]) zu einem späteren Zeitpunkt vorbereiten, da dies nur in den letzten 24 Stunden des Experiments erforderlich ist.
- Verschließen Sie die Flaschen mit einem Septum, quetschen Sie sie aus Aluminium und autoklavieren Sie sie.
- Um das Medium zu übertragen, entfernen Sie die Aluminium-Crimpung mit einem geeigneten Decapper (Durchmesser = 20 mm). Entferne auch die Nasenscheidewand. Gießen Sie das vorbereitete Medium vorsichtig in die Serumflaschen, ohne die Flasche zu berühren.
- Zum Sterilisieren die Öffnung der Flasche mit einem tragbaren Bunsenbrenner anzünden. Verschließen Sie die Flaschen, indem Sie das Septum mit 70%igem Ethanol besprühen, es in die Flasche geben und mit einer Aluminiumquetschung mit einer Dichtungszange verschließen.
- Stellen Sie die geschlossenen Flaschen 24 Stunden lang bei 37 °C, 5 % CO2 in den Inkubator, um das Medium auf 37 °C zu erwärmen und sicherzustellen, dass keine sichtbaren Anzeichen einer Kontamination vorhanden sind, bevor Sie Medienflaschen für das Gerät verwenden.
3. HuMiX-Start
- NeuroHuMiX-Baugruppe
HINWEIS: In diesem Abschnitt wird erläutert, wie das Gerät zusammengebaut wird. Kurz gesagt, das Klemmsystem wird sterilisiert und nachgezogen, woraufhin der untere PC-Deckel auf die Basis der Klemme gesetzt wird. Dann werden die beschichteten Dichtungen und die oberen PC-Deckel aufeinander gestapelt, gefolgt vom oberen Teil der Klemme. Zum Schluss wird die Klemme angezogen, um die Dichtungen zu komprimieren und das System leckagefrei und gasdicht zu machen. Abbildung 4 zeigt die verschiedenen Teile, die für die Baugruppe benötigt werden.- Autoklavieren Sie die Klemmen und die beiden PC-Deckel (oben und unten) vor der Montage.
- Ziehen Sie die Schrauben fest, mit denen der obere und untere Teil der Klemme zusammengebaut wird (Abbildung 4C,D). Öffnen Sie die Schrauben vor dem Autoklaviervorgang und ziehen Sie sie später wieder fest.
- Übertragen Sie den beschichteten und getrockneten (Schritt 2.2.3) unteren PC-Deckel (Abbildung 4 A1) von der quadratischen Petrischale auf die Oberseite der Klemmbasis, indem Sie die vier Schrauben in den Ecken des PC-Deckels festhalten.
HINWEIS: Vermeiden Sie es, den PC-Deckel zu berühren, um das Risiko einer Kontamination zu verringern. - Legen Sie die Epithelkammerdichtung mit einer sterilen Pinzette nach oben (d. h. mit der Membran nach oben) auf den PC-Deckel. Verwenden Sie die Schrauben, um die Dichtung und den PC-Deckel auszurichten, um sicherzustellen, dass die Einlass- und Auslassöffnungen in den Ecken des Deckels mit den Öffnungen in der Dichtung ausgerichtet sind (Abbildung 3).
- Legen Sie die Sandwichdichtung mit der sterilen Pinzette mit der Oberseite nach oben auf die Kollagendichtung.
- Setzen Sie den oberen PC-Deckel auf die Sandwichdichtung. Um das Risiko einer Kontamination zu verringern, berühren Sie dazu nur die Ränder des Deckels (berühren Sie nicht den oberen oder unteren Teil des Deckels). Stellen Sie sicher, dass die Widerhaken am Klemmdeckel mit der Einlass- und Auslassöffnung an der Membranbaugruppe ausgerichtet sind und dass die Schrauben des unteren PC-Deckels in die Öffnung des oberen PC-Deckels passen.
- Um das Gerät zu schließen, legen Sie den Klemmdeckel (den oberen Teil der Klemme) auf den PC-Deckel. Stellen Sie sicher, dass die Öffnungen des Deckels mit den Einlass- und Auslasswiderhaken des oberen PC-Deckels übereinstimmen. Schließen Sie die Verriegelungen. Bewahren Sie das geschlossene Gerät (Abbildung 5) unter der Haube auf, um es an die grundierten Schlauchleitungen anzuschließen (Schritt 3.2.6).
- Grundierung von Schlauchleitungen
HINWEIS: In diesem Abschnitt wird beschrieben, wie Sie den Schlauch an die Ein- und Auslaufflaschen sowie an die Pumpe anschließen, den Schlauch anschließend mit Medium anfüllen, um alle bei der Reinigung und Sterilisation verwendeten Produktreste zu entfernen, und sicherstellen, dass keine Blasen im Schlauch vorhanden sind.- Die Grundierung der Schlauchleitungen erfolgt in der Biosicherheitswerkbank. Belüftungsnadeln mit Filtern in das Septum jeder Zu- und Abflussflasche einsetzen. Führen Sie die 120-mm-Nadeln mit einer sauberen, sterilen Pinzette in die 250-ml-Serumflaschen ein. Führen Sie die 80-mm-Nadeln in die 50-ml-Serumflaschen ein.
- Führen Sie die 40-mm-Nadeln am Ende jeder Schlauchleitung in eine Ausfluss-Serumflasche ein. Pro Gerät gibt es zwei Ausflussflaschen für die drei Schlauchleitungen. Die 40-mm-Nadeln der epithelialen und neuronalen Schlauchleitungen sind mit derselben Ausflussflasche verbunden, um das Medium zu entsorgen. Die Bakterienschlauchleitung führt zur zweiten Ausflussflasche.
- Führen Sie die Pumpenschlauchleitungen in die Pumpenkassetten ein. Stellen Sie sicher, dass die Dreiwegehähne der Schlauchleitungen alle geöffnet sind.
- Stellen Sie die Schlauchpumpe zu Beginn so ein, dass sie das Medium mit einer Drehzahl von 5 U/min vom Zufluss zur Auslaufflasche leitet (siehe Tabelle 4).
HINWEIS: Während der Grundierung der Schlauchleitung kann die Drehzahl auf bis zu 10 U/min erhöht werden, um den Prozess zu beschleunigen. - Starten Sie die Pumpe, indem Sie den Startknopf drücken, und stellen Sie sicher, dass die Richtung des Pumpvorgangs im Uhrzeigersinn verläuft. Sobald das Medium in die Auslaufflaschen tropft, stellen Sie sicher, dass keine Leckagen vorhanden sind und keine Luftblasen in den Schlauchleitungen und Verbindungsstellen zurückbleiben. Entfernen Sie verbleibende Blasen, indem Sie auf die Schläuche und Anschlüsse klopfen oder die Pumpendrehzahl erhöhen.
- Wenn alle Schlauchleitungen in die Ausflussflaschen fallen, stellen Sie die Durchflussrate für den Anschluss des Erstgeräts auf 2 U/min ein.
- Grundierung des Gerätes
HINWEIS: In diesem Abschnitt wird beschrieben, wie das Gerät mit Zellkulturmedium grundiert wird, um sicherzustellen, dass alle Kammern mit Medium gefüllt und vorbeschichtet sind, damit keine Blasen im System verbleiben und die Zellen während der Inokulation leicht anhaften können.- Die Grundierung des Gerätes erfolgt in der Biosicherheitswerkbank unter sterilen Bedingungen. Um das Gerät anzuschließen, verbinden Sie zunächst die neuronale Leitung, die untere Kammer des Geräts.
- Um eine Leitung anzuschließen, schließen Sie die Dreiwege-Absperrventile (Abbildung 5C) und beginnen Sie zunächst mit der Auslassseite, wo der kurze Schlauch von der Luer-Buchse getrennt und mit der Auslassöffnung des Geräts verbunden wird, indem Sie den Schlauch über den Widerhaken schieben. Um eine ordnungsgemäße Verbindung sicherzustellen und die Wahrscheinlichkeit von Undichtigkeiten und/oder Verunreinigungen zu verringern, stellen Sie sicher, dass der Schlauch mit dem Deckel in Kontakt ist, indem Sie ihn ganz nach unten über den Stecker drücken. Schließen Sie den Zulaufschlauch auf die gleiche Weise an. Sobald eine Leitung vollständig an das Gerät angeschlossen ist (Zu- und Abfluss), öffnen Sie die Drei-Wege-Absperrhähne.
- Wiederholen Sie den vorherigen Schritt mit der Epithellinie und dann mit der Bakterienlinie. Halten Sie die Pumpe auf einer Durchflussrate von 2 U/min. Erhöhen Sie die Pumpendrehzahl auf 2,5 U/min, jedoch nicht höher, um Leckagen durch Druckaufbau zu vermeiden. Lassen Sie die Pumpe die Kammern im Gerät entleeren.
- Überwachen Sie die Serumflaschen mit dem Abfluss jederzeit. Wenn Luftblasen in den Kammern, Leitungen oder Anschlüssen stecken bleiben, fällt eine Schlauchleitung mit einer Luftblase im Inneren nicht mehr in die Ausströmkammer. Um Luftblasen loszuwerden, lassen Sie die Geräte zunächst einige Minuten laufen. Wenn das Problem dadurch nicht behoben wird, schließen Sie eine der anderen Leitungen, die für kurze Zeit abfallen, indem Sie den Drei-Wege-Absperrhahn des Abflusses schließen.
- Sobald alle Kammern des Geräts vollständig entleert sind - alle Kammern sind mit Zellkulturmedium gefüllt und keine Blasen mehr im Gerät - reduzieren Sie die Pumpendrehzahl auf 0,5 U/min. Das Gerät ist nun bereit für die Zellinokulation.
4. Zellvorbereitung und Inokulation
HINWEIS: In diesem Abschnitt wird beschrieben, wie die verschiedenen Zelltypen, die für die Inokulation des Produkts erforderlich sind, vorbereitet werden und wie sie steril und ohne das Einbringen von Luftblasen in das Gerät inokuliert werden. Darüber hinaus wird beschrieben, wie eine Mediumserfrischung für die neuronalen Zellen durchgeführt wird und wie das Medium für die Bakterienkultur im Gerät vorbereitet wird.
- Epithelzellen
- Die Caco-2-Zellen werden mit Trypsin-Ethylendiamintetraessigsäure (EDTA) aus dem Kolben gelöst, in RPMI 1640 + 10% FBS resuspendiert und in einem Neubauer-Zellzähler mit dem Trypanblau-Ausschlussassay gezählt. Zentrifugieren Sie die Caco-2-Zellsuspension (3 min, 300 × g) und verwerfen Sie den Überstand, um das verbleibende Trypsin-EDTA zu entfernen. Resuspendieren Sie die Caco-2-Zellen in RMPI 1640 + 10% FBS, um eine Suspension von 350.000 Zellen/ml zu erhalten. Für jedes Gerät wird ein Volumen von 1,5 ml benötigt.
- 1,5 ml der Caco-2-Zellsuspension in eine sterile 2-ml-Spritze überführen und Luft oder Blasen, die in der Spritze verbleiben, entfernen.
- Schließen Sie die Dreiwege-Absperrventile der Schläuche der Bakterien- und Nervenkammern. Trennen Sie die Schläuche der Bakterien- und Nervenkammern von der Pumpe, indem Sie die Kassetten mit den entsprechenden Schläuchen vom Rotor entfernen.
- Öffnen Sie die Kappe des Dreiwege-Absperrventils des Zuflussschlauchs, der zur Epithelkammer führt, und drehen Sie das Dreiwege-Absperrventil, um den Mediumstrom vom Gerät zum "offenen Anschluss" umzuleiten (Abbildung 5C). Lassen Sie das Medium fließen, bis ein Tropfen Medium am offenen Ende des Dreiwege-Absperrhahns erscheint.
- Führen Sie die Spritze mit den Epithelzellen in den "offenen Konnektor" ein, indem Sie die Tropfen-Tropfen-Verbindungsmethode verwenden, um das Einführen der Spritze in den Konnektor ohne das Eindringen von Luftblasen zu ermöglichen. Drehen Sie das Ventil des Dreiwegehahns, um den Durchfluss des Mediums aus der Zulaufflasche (über die Pumpe) zu stoppen und den Durchfluss von der angeschlossenen Spritze zum Ausgangsgerät zu ermöglichen. Trennen Sie den Epithelkanal von der Pumpe.
- Drücken Sie langsam auf die Spritze, um die Epithelkammer mit der Zellsuspension zu impfen. Stellen Sie sicher, dass etwa ein Tropfen pro 3 s in die Auslaufflasche tropft.
- Fügen Sie 1,5 ml der Zellsuspension hinzu und schließen Sie dann das Ventil des Dreiwegehahns für den Auslass. Trennen Sie die Spritze und verschließen Sie das offene Ende des Dreiwegehahns mit der Kappe. Halten Sie die Kammer mindestens 2 h geschlossen. Impfen Sie in der Zwischenzeit die neuronalen Zellen.
- Neuronale Zellen
- Lösen Sie die neuronalen Zellen aus der Vertiefung, indem Sie die Zellen im Medium mit einer Pipette aus den jeweiligen Vertiefungen resuspendieren. Beimpfen Sie jedes Gerät mit vollständig konfluenten Zellen aus einer Vertiefung (9,6cm2) einer 6-Well-Platte.
- Resuspendieren Sie das Zellpellet im entsprechenden N2B27-Mediumvolumen (1,5 ml Medien pro Gerät) + 1 μl Fibronektin/ml Zellsuspension. 1,5 ml der resuspendierten Zellsuspension in eine 2-ml-Spritze überführen und alle in der Spritze verbliebenen Luftblasen entfernen. Geben Sie die gefüllte Spritze in ein steriles konisches 50-ml-Röhrchen und geben Sie sie in die Biosicherheitswerkbank, in der sich das grundierte HuMiX-Gerät befindet.
- Befolgen Sie den gleichen Inokulationsprozess wie zuvor für die Epithelkammer-Inokulation beschrieben, mit Ausnahme des Wechsels der Schlauchleitung. Trennen Sie hier die Bakterien- und Epithelschlauchleitungen von der Pumpe.
- Schließen Sie nach der Inokulation der neuronalen Zellen alle Dreiwegehähne der Schlauchleitungen und trennen Sie sie von der Pumpe. Stellen Sie das Gerät bei 37 °C und 5 % CO2 in den Inkubator. Halten Sie alle Kanäle 2 h lang geschlossen, damit sich die Zellen anheften können.
- Schließen Sie nach 2 h die Bakterien- und Epithelkanäle an die Pumpe an und öffnen Sie die Ein- und Auslauf-Dreiwegehähne beider Leitungen. Halten Sie die neuronale Kammer während der kommenden 14 Tage nach dem ersten Start des Geräts geschlossen, außer während des Mediumwechsels.
- Wechseln Sie während des 14-tägigen Laufs alle 3-4 Tage das Medium der neuronalen Kammer. Für eine mittlere Auffrischung 3 ml frisches N2B27-Medium pro Gerät zubereiten und in eine sterile 20-ml-Serumflasche umfüllen. Verschließen Sie die Serumflasche mit Medium mit einem sterilen Septum und einem Aluminium-Bördelverschluss.
- Führen Sie eine Belüftungs- und 80-mm-Nadel in das Septum der neuen Flasche ein. Ersetzen Sie die alte Medienflasche durch die neue im Inkubator, indem Sie den männlichen Luer von der Nadel der alten Flasche zur Nadel der neuen Flasche trennen.
- Verbinden Sie die Kassette mit dem Pumpenschlauch der neuronalen Kammer mit der Pumpe und öffnen Sie die Drei-Wege-Absperrhähne. Lassen Sie das Medium 2 h lang mit 0,5 U/min fließen, bevor Sie die Dreiwegehähne des neuronalen Schlauchs schließen und bis zum nächsten Medienwechsel von der Pumpe trennen.
5. Bakterienkultur und Inokulation
HINWEIS: In dieser Studie wurde an Tag 12 eine Flüssigkultur des Limosilactobacillus reuteri-Stammes F275 aus einem Glycerinstamm reanimiert. Je nach Bedarf oder Studiendesign können auch andere Bakterienarten eingesetzt werden.
- Bereiten Sie drei Röhrchen mit 5 ml MRS-Brühe vor, ein steriles Kontrollröhrchen und zwei für die Inokulation von L. reuteri. Mit einer Impfschleife kratzen Sie die Oberseite der Glycerinbrühe ab und geben Sie sie in ein Röhrchen. Wiederholen Sie den Vorgang mit einem zweiten Impfröhrchen. Inkubieren Sie die Röhrchen bei 37 °C und 170 U/min über Nacht.
HINWEIS: Lassen Sie die Glycerinbrühe nicht auftauen. - Bereiten Sie frisches Medium für die ersten Geräte vor, indem Sie Roswell Park Memorial Institute (RPMI) 1640 + 10 % FBS mit 5 % MRS-Brühe mischen. Bereiten Sie 25 ml pro Gerät vor und füllen Sie sie unter sterilen Bedingungen in 100-ml-Serumflaschen um. Verschließen Sie die Flaschen mit einem sterilen Septum und einer Aluminiumquetschung. Stellen Sie die Flaschen über Nacht bei 37 °C, 5 % CO2 in den Inkubator.
- Bevor Sie die Flaschen an die Bakterienschlauchleitung anschließen, fügen Sie jeder Flasche RPMI 1640/MRS-Medien eine Belüftungsnadel und eine 80-mm-Nadel hinzu.
- Bereiten Sie neue Röhrchen mit jeweils 3 ml RPMI 1640 + 10 % FBS + 5 % MRS-Brühe vor. Bereiten Sie mindestens zwei Röhrchen vor, ein Kontrollröhrchen und ein Röhrchen pro Gerät. Inimpfen Sie 15 μl der L. reuteri-Übernachtkultur (optische Dichte [OD] > 2) in zwei der neu präparierten Röhrchen.
- Die Röhrchen werden 1 h lang bei 37 °C und 170 U/min inkubiert, danach wird ein OD von 0,05-0,10 erreicht, was ~1 × 107 koloniebildenden Einheiten (KBE)/ml entspricht. 1,5 ml in eine 2-ml-Spritze (eine pro Gerät) überführen. Verwenden Sie den Rest der bakteriellen Suspension für die KBE-Beschichtung und die Lebend-/Totfärbung.
- Für die bakterielle Inokulation des Produkts ist das gleiche Verfahren wie in Schritt 4.1 beschrieben, mit der Ausnahme, dass hier die neuronalen und epithelialen Schlauchleitungen geschlossen sind.
- Schließen Sie nach der Impfung auch die Bakterienschlauchleitung, indem Sie die Ventile schließen und die Pumpe für 30 Minuten von der Pumpe trennen. Verbinden Sie die epithelialen und bakteriellen Schlauchleitungen und öffnen Sie sie wieder. Lassen Sie die Geräte weitere 24 h bei 37 °C und 0,5 U/min laufen.
6. HuMiX-Öffnung und Probenahme
HINWEIS: Im folgenden Abschnitt wird die Probenahme verschiedener Zelltypen beschrieben. Zum Beispiel werden neuronale Zellpellets für die RNA-Extraktion und die anschließende quantitative Polymerase-Kettenreaktion (qPCR), Bakterienpellets für die DNA-Extraktion und die 16S-rRNA-Gensequenzierung sowie Überstände für Enzyme-Linked Immunosorbent Assays (ELISAs) und andere Assays (z. B. Laktat-Assay) verwendet.
- Schließen Sie am 14. Tag, dem Eröffnungstag, alle Dreiwegehähne, trennen Sie die Schläuche von der Pumpe und bringen Sie das Gerät des Inkubators zum Labortisch.
HINWEIS: Achten Sie beim Bewegen des Geräts darauf, dass das eigentliche Gerät waagerecht bleibt. - Entfernen Sie die mit dem Gerät verbundenen Schlauchleitungen, bevor Sie die Klemme langsam öffnen, und entfernen Sie den Klemmdeckel. Entfernen Sie vorsichtig den oberen PC-Deckel, sammeln Sie das Medium in einem 1,5-ml-Mikrozentrifugenröhrchen und legen Sie es auf Eis.
- Entfernen Sie vorsichtig die Sandwichdichtung, während Sie das Medium aus der Epithelkammer auffangen. Achten Sie darauf, die Zellschicht nicht zu berühren. Legen Sie die Sandwichdichtung in eine quadratische Petrischale und geben Sie sterile 0,9%ige NaCl-Lösung inH2Oin die Bakterienkammer, bis die Kammer vollständig bedeckt ist (ca. 1 ml).
- Entfernen Sie langsam die Kollagendichtung, während Sie das Medium aus der neuronalen Kammer sammeln und in ein Mikrozentrifugenröhrchen überführen. Lege alle Medienröhrchen auf Eis. Legen Sie die Kollagendichtung in eine quadratische Schale und geben Sie vorsichtig einige Milliliter 1x PBS in die Caco-2-Schicht, bis die Zellschicht vollständig bedeckt ist.
- Legen Sie den unteren PC-Deckel in eine quadratische Petrischale und geben Sie vorsichtig ca. 2 ml 1x PBS auf die neuronalen Zellen, damit sie während des Probenahmeprozesses nicht austrocknen.
- Zentrifugieren Sie das bakterielle Medienröhrchen bei 5.000 × g für 5 min bei 4 °C. Die Epithel- und neuronalen Medienröhrchen werden bei 300 × g für 5 min bei 4 °C zentrifugiert. Nach der Zentrifugation wird der Überstand jedes Röhrchens in ein neues Mikrozentrifugenröhrchen überführt und sofort auf Trockeneis gestellt.
- Epithelzellen
- Entfernen Sie das PBS vorsichtig aus der Dichtung und sammeln Sie es in einem konischen 15-ml-Röhrchen. Geben Sie 2 ml Trypsin in die Zellen und inkubieren Sie die Dichtung 5 Minuten lang bei 37 °C, 5 % CO2. Nach der Inkubation 2 ml RPMI 1640 in die Dichtung geben, die Zellen resuspendieren und in einem weiteren konischen 15-ml-Röhrchen sammeln.
- Beide Röhrchen 5 min bei 300 × g zentrifugieren. Verwerfen Sie den Überstand. Resuspendieren Sie das Pellet aus der PBS-Wäsche in 300 μl PBS und das Zellpellet in 1 ml PBS.
- Übertragen Sie 50 μl von jedem Röhrchen in ein 0,5-ml-Mikrozentrifugenröhrchen, um den automatisierten Zellzähler und den Trypanblau-Ausschluss-Assay-Zellzähler zu erhalten. Das resuspendierte Zellpellet (1 ml) in ein 1,5-ml-Röhrchen überführen und 5 Minuten lang bei 300 × g zentrifugieren.
- Entfernen Sie den Überstand und resuspendieren Sie das Zellpellet in 250 μl Lysepuffer + 1 % Beta-Mercaptoethanol und stellen Sie das Röhrchen auf Trockeneis.
- Neuronale Zellen
- Nehmen Sie die quadratische Petrischale mit dem unteren, durchsichtigen PC-Deckel mit den neuronalen Zellen aus dem vorherigen Schritt (6.5) in ein inverses Phasenkontrastmikroskop.
HINWEIS: Seien Sie beim Bewegen des PC-Deckels mit PBS sehr vorsichtig, da sich das neuronale Netzwerk sehr leicht löst. - Führen Sie vor der Inokulation eine letzte Qualitätskontrolle durch, indem Sie die Morphologie und Zelldichte der neuronalen Zellen mit Hilfe der Hellfeldmikroskopie beobachten. Vergewissern Sie sich, dass die Zellen aus den Sphäroiden gewandert sind und sich ein neuronales Netzwerk gebildet hat. Das neuronale Netzwerk sollte zu etwa 90 % konfluent sein. Weitere Informationen finden Sie bei Fattahi et al.6.
- Am Labortisch resuspendieren Sie die Zellen in PBS und sammeln sie in einem 1,5-ml-Röhrchen. Zentrifugieren Sie das Röhrchen bei 300 × g für 3 Minuten. Verwerfen Sie den Überstand, resuspendieren Sie das Zellpellet in 250 μl Lysepuffer + 1 % Beta-Mercaptoethanol und legen Sie es auf Trockeneis.
HINWEIS: Die Immunfluoreszenzfärbung (IF) kann an den neuronalen Zellen auf dem PC-Deckel durchgeführt werden. Wenn die IF-Färbung der bevorzugte Assay ist, um das neuronale Netzwerk nicht zu zerstören, sollten die Zellen mit 4% Paraformaldehyd (PFA) auf dem PC-Deckel fixiert werden, was eine Wiederverwendung des PC-Deckels in zukünftigen Experimenten verhindert.
- Nehmen Sie die quadratische Petrischale mit dem unteren, durchsichtigen PC-Deckel mit den neuronalen Zellen aus dem vorherigen Schritt (6.5) in ein inverses Phasenkontrastmikroskop.
- Bakterielle Zellen
- Resuspendieren Sie die an der Membran befestigten Bakterienzellen in 0,9%iger NaCl-Lösung. Wenn die Zellen fest anhaften, verwenden Sie vorsichtig einen Zellschaber, um die Bakterienzellen von der Membran zu lösen.
HINWEIS: Die Verwendung eines Schabers kann die Zellen beschädigen und die Wahrscheinlichkeit erhöhen, dass mehr abgestorbene Zellen zu sehen sind. - Sammeln Sie die Zellsuspension in einem konischen 15-ml-Röhrchen. Geben Sie das Pellet, das von den zuvor gesammelten Medien übrig geblieben ist, in das konische 15-ml-Röhrchen. Bei 5.000 × g 5 min bei 4 °C zentrifugieren. Verwerfen Sie den Überstand und resuspendieren Sie das Zellpellet in 1 ml 0,9%iger NaCl-Lösung.
- Teilen Sie dieses Volumen in drei Teile auf: einen zum Einfrieren eines Bakterienpellets für die Nukleinsäureextraktion (650 μl), einen für die KBE-Beschichtung (50 μl) auf MRS-Platten und einen für die Lebend-/Totfärbung (300 μl). Zur Vorbereitung des Pellets für die Nukleinsäureextraktion wird die Zellsuspension in ein Mikrozentrifugenröhrchen überführt, bei 5.000 × g für 5 min bei 4 °C zentrifugiert und der Überstand verworfen. Legen Sie das Zellpellet auf Trockeneis.
- Am Ende der Probenahme werden alle Röhrchen auf Trockeneis in einen Gefrierschrank bei -80 °C überführt, um sie für spätere nachgelagerte Analysen zu lagern.
HINWEIS: Der Überstand kann auch für die Analyse der Gaschromatographie-Massenspektrometrie (GC-MS) verwendet werden. Darüber hinaus kann die kollagenbeschichtete Membran während des Öffnens für verschiedene Analysen in verschiedene Teile geteilt werden - eine Hälfte für die IF-Occludin-Färbung, ein anderer Teil für die RNA-Extraktion für die weitere Genexpressionsanalyse und ein weiterer Teil für die Zellzählung.
- Resuspendieren Sie die an der Membran befestigten Bakterienzellen in 0,9%iger NaCl-Lösung. Wenn die Zellen fest anhaften, verwenden Sie vorsichtig einen Zellschaber, um die Bakterienzellen von der Membran zu lösen.
Ergebnisse
In neuroHuMiX haben wir drei verschiedene Zelltypen gemeinsam kultiviert: bakterielle, epitheliale und neuronale Zellen (Abbildung 1). Um sicherzustellen, dass die Zellen alle lebensfähig waren, führten wir verschiedene Assays an den verschiedenen Zelltypen durch. So führten wir beispielsweise KBE-Zählungen an Bakterienzellen, Zellzahl- und Zellviabilitätsassays an den Epithelzellen durch, während die neuronalen Zellen mittels mikroskopischer Analysen beurteilt wurden.
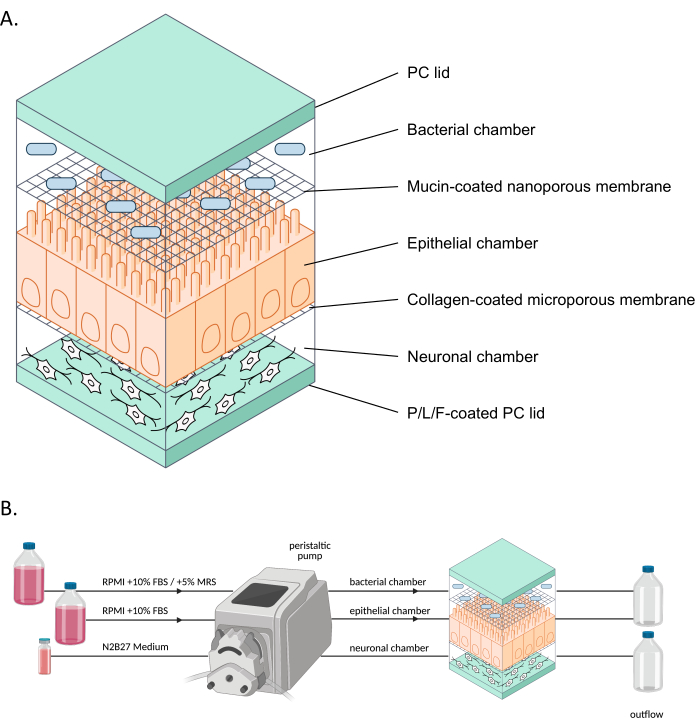
Abbildung 1: Schematische Darstellung von neuroHuMiX und seinem Versuchsaufbau . (A) Die drei Kammern werden zwischen zwei PC-Deckeln gehalten, um sie geschlossen zu halten. Jede Kammer ist mit einem spezifischen Medium für die darin gezüchteten Zellen gefüllt. Die verschiedenen Kammern sind durch semipermeable Membranen getrennt, die eine Zellkommunikation über lösliche Faktoren ermöglichen, die die Membranen passieren. (B) Darstellung des neuroHuMiX-Aufbaus. Jede Kammer ist mit unterschiedlichen Medienflaschen verbunden. Die Bakterienkammer ist in den ersten 12,5 Tagen mit RPMI + 10% FBS verbunden, bevor sie in den letzten 36 Stunden auf RPMI + 10% FBS + 5% MRS umgestellt wird. Abkürzungen: PC = Polycarbonat; P/L/F = Poly-L-Ornithin/Laminin/Fibronektin; RPMI = Zellkulturmedium des Roswell Park Memorial Institute; MRS = De Man, Rogosa und Shapre-Nährmedium. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Um festzustellen, ob die Zellen angemessen befestigt waren, untersuchten wir beim Öffnen der Geräte die Bildung einer Zellschicht auf der kollagenbeschichteten Membran (Abbildung 6A). Um sicherzustellen, dass die Zellen im Gerät lebensfähig waren, wurde eine automatische Zellzählerzählung (Abbildung 6B) und eine Zellzählung mit Trypanblau-Ausschlussassay durchgeführt (Abbildung 6C). Die Assays wurden an Caco-2-Zellen aus drei verschiedenen HuMiX-Setups durchgeführt: (i) Caco-2 in Kultur mit ENs, (ii) Caco-2 in Kultur mit L. reuteri und (iii) das Gerät mit Co-Kultur aller drei Zelltypen. Statistische Tests mit einer Einweg-ANOVA ergaben keine signifikanten Unterschiede zwischen den Zelltypen, was darauf hindeutet, dass die Caco-2-Zellen in all diesen anfänglichen Geräteaufbauten und -bedingungen, die in dieser Studie getestet wurden, lebensfähig blieben. Dies unterstreicht die Tatsache, dass die Bakteriendichte, die während der Co-Kultur von L. reuteri und den beiden menschlichen Zelltypen erreicht wurde, keine zytotoxischen Wirkungen auf die menschlichen Zellen hat.
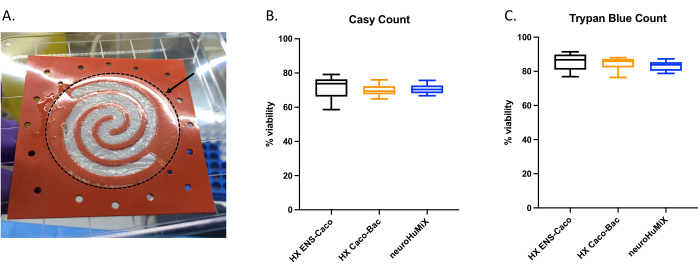
Abbildung 6: Beurteilung von Caco-2-Zellen auf der kollagenbeschichteten Membran. (A) Schicht von Caco-2-Zellen auf der kollagenbeschichteten Membran nach dem Öffnen. Der Pfeil zeigt die kollagenbeschichtete Membran an, die von einem gestrichelten Kreis umgeben ist. Die Caco-2-Zellen wuchsen auf der Spiralform der Membran. Zelllebensfähigkeit von Caco-2-Zellen nach 14 Tagen in HuMiX. Die Zellzahlen wurden mit (B) dem automatisierten Zellzähler und (C) der Zellzahl des Trypanblau-Ausschlusstests ermittelt. Die Anzahl der Caco-2-Zellen wurde aus verschiedenen Kulturaufbauten im Ausgangsgerät bestimmt: Co-Kultur mit enterischen Neuronen (ENs) (schwarz), Co-Kultur mit L. reuteri (orange) und im Gerät (ENs und L. reuteri) (blau). Es wurde eine unidirektionale ANOVA durchgeführt, die zeigte, dass es keinen signifikanten Unterschied zwischen den verschiedenen Kulturaufbauten gibt (unidirektionale ANOVA, p = 0,1234 [ns]; Fehlerbalken zeigen Standardfehler an). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Um L. reuteri mit Säugetierzellen kultivieren zu können, haben wir zunächst die Nährmedien für den Einsatz im Gerät optimiert und angepasst. Wir fanden heraus, dass eine 5%ige Mischung von MRS in RPMI 1640 (ergänzt mit 10% FBS) optimal für das Wachstum von L. reuteri geeignet war, während sie für die in diesen Assays verwendeten Säugetierzellen nicht zytotoxisch war. Anschließend wurde eine KBE-Zählung durchgeführt, um das Wachstum von L. reuteri zu beurteilen, wenn es 24 Stunden lang im Gerät kultiviert wurde. Die KBE-Anzahl wurde für zwei verschiedene anfängliche Geräteeinstellungen bestimmt (Abbildung 7) - L. reuteri kokultiviert mit Caco-2 und L. reuteri im Gerät. In beiden Setups unterschieden sich die KBE-Zählungen signifikant von denen des HuMiX-Inokulums und der geernteten Zellen (Einweg-ANOVA, p = 0,0002), was auf ein Wachstum der Bakterienzellen im Inneren des ursprünglichen Geräts hindeutet.
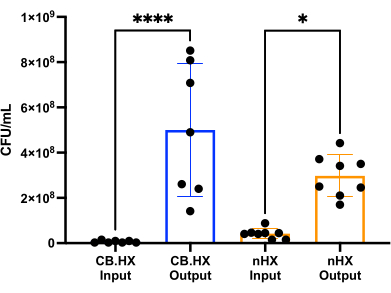
Abbildung 7: Limosilactobacillus reuteri KBE-Zählung des Inokulums (verdünnt 1:100.000) und nach 24 h in HuMiX. Zwei verschiedene Setups: Caco-2-Zellen in Co-Kultur mit L. reuteri und dem Gerät. Eine Einweg-ANOVA zeigt einen signifikanten Unterschied (p = 0,0002 [***]) zwischen dem Inokulum und den geernteten Zellen, was bedeutet, dass die Bakterien in HuMiX wachsen. Fehlerbalken zeigen den Standardfehler an. Abkürzungen: CB. HX = Caco-2-Bakterien HuMiX; nHX = neuroHuMiX. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Um zu beurteilen, ob die Kultivierung der ENs im Gerät den Phänotyp der Zellen verändern würde, wurde die grobe Morphologie der ENs mit einem inversen Phasenkontrastmikroskop beobachtet. In diesem Schritt wurden sowohl die Konfluenz als auch die EN-Morphologie untersucht. Die Etablierung eines konfluierenden neuronalen Netzwerks deutete darauf hin, dass die Zellen gut auf dem PC-Deckel des beschichteten Geräts hafteten. Wichtig ist, dass dies die Vorstellung unterstreicht, dass sie in Co-Kultur mit Caco-2 und L. reuteri gewachsen sind. Die Kante zwischen dem konfluierenden neuronalen Netzwerk und der durch Dichtungen abgegrenzten Spirale war deutlich erkennbar (Abbildung 8).
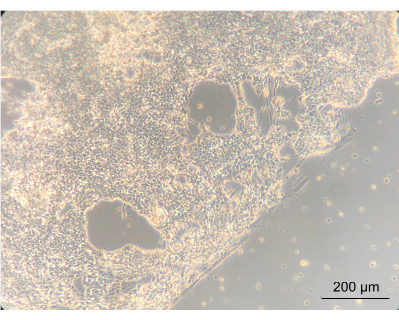
Abbildung 8: Enterische Neuronen nach 14 Tagen Kultur im Gerät. Auf der linken Seite des Bildes sind die Neuronen zu einer konfluierenden Schicht auf der Spirale gewachsen. Die Kante zwischen der neuronalen Schicht und dem Raum ohne Zellen ist die Kante der Spirale; 10-fache Vergrößerung, Maßstabsbalken = 200 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Im Gerät verwendete Deckel. Die Bilder zeigen die oberen (links) und unteren (rechts) PC-Deckel. Jede Seite des PC-Deckels ist 6,4 cm lang. Abkürzung: PC = Polycarbonat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Epithelkammerdichtung am unteren PC-Deckel. Draufsicht auf die Epithelkammerdichtung auf dem unteren PC-Deckel (links) und Unteransicht (rechts) zeigt die Ausrichtung der Epithelkammerdichtung mit den Ein- und Auslässen des unteren PC-Deckels. Jede Seite der Dichtungen sowie des PC-Deckels misst 6,4 cm. Abkürzung: PC = Polycarbonat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Montage des Gerätes. (A) Verschiedene Teile für die Montage von HuMiX: (1) unterer PC-Deckel; (2) Dichtung mit kollagenbeschichteter mikroporöser Membran, die auf (1) aufgesetzt wird; (3) Sandwichdichtung mit einer mucinbeschichteten nanoporösen Membran dazwischen, die auf (2) aufgesetzt ist; (4) Oberer PC-Deckel auf (3) aufgesetzt. Jede Seite der Dichtungen und PC-Deckel misst 6,4 cm. (B) Alle Teile aus (A) werden zusammengefügt. (C,D) Zusammengebaute Geräteansicht von oben (links) und von der Seite (rechts). B wird in das Spannsystem eingesetzt, um das System zu schließen. (C) Jede Seite der oberen Klemme misst 8 cm. Abkürzung: PC = Polycarbonat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
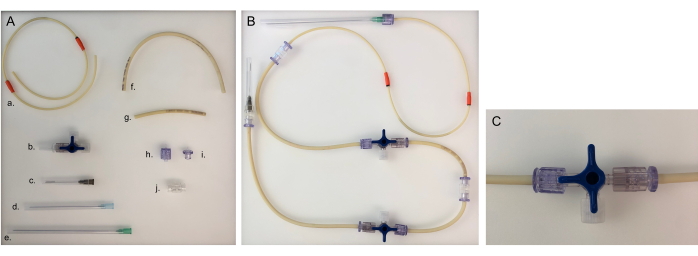
Abbildung 5: Benötigte Teile für die Schlauchleitung und die montierte Schlauchleitung für eine Kammer. (A) Verschiedene Teile zum Aufbau einer Schlauchleitung: a. Pumpenschlauchleitung; b) Dreiwegehahn, ca. 40 mm Nadel; d. 80 mm Nadel; e. 120 mm Nadel; f. lange Schlauchleitung (20 cm); g. kurze Schlauchleitung (8 cm); h. männlicher Luer; i. weiblicher Luer; J. Adapter. (B) Konfektionierte Schlauchleitung für die Bakterien- oder Epithelkammer. Für die neuronale Kammer müsste die 120-mm-Nadel gegen eine 80-mm-Nadel ausgetauscht werden. (C) Dreiwege-Absperrhahn gedreht, um den Mediumstrom vom Gerät zum "offenen Anschluss" umzuleiten und die Kammer zu schließen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Tag | 0 | 2 | 4 | 6 | 8 | 10 |
| Medien-Komposition | 100% E6 | 100% E6 | 75% E6 | 50% E6 | 25% E6 | 100% N2 |
| + LDN | + LDN | 25% N2 | 50% N2 | 75% N2 | + LDN | |
| + SB | + SB | + LDN | + LDN | + LDN | + SB | |
| + CHIR | + SB | + SB | + SB | + CHIR | ||
| + CHIR | + CHIR | + CHIR | + RA | |||
| + RA | + RA | |||||
| Molekül | [Konzentration] | |||||
| LDN | 100 nM | |||||
| SB | 10 μM | |||||
| CHIR | 3 μM | |||||
| Retinsäure (RA) | 1 μM |
Tabelle 1: Zusammensetzung der Medien.
| Medien | Bestandteile (Konzentrationen, die in der Materialtabelle aufgeführt sind) | Volumen (ml) |
| N2-Medien (50 ml) | DMEM-F12-KARTON | 48 |
| N2 Zuschlag | 0.5 | |
| L-Glutamin | 0.5 | |
| Penicillin/Streptomycin | 0.5 | |
| NEAA | 0.5 | |
| N2B27/ENS-Medien (50 ml) | Neurobasal | 48 |
| N2 Zuschlag | 0.5 | |
| L-Glutamin | 0.5 | |
| Penicillin/Streptomycin | 0.5 | |
| B27-A | 0.5 |
Tabelle 2: Medienrezepte.
| Sterilisationstemperatur (°C) | 116 |
| Sterilisationszeit (min) | 20 |
| Trocknungszeit (min) | 10 |
| Hülsenfrüchte | 3 |
| Endtemperatur (°C) | 99 |
Tabelle 3: HuMiX-Autoklavlauf.
| Umdrehungen pro Minute (U/min) | Durchschnittliche Durchflussrate (μl/min) |
| 0.5 | 13 |
| 2 | 79 |
| 5 | 180 |
Tabelle 4: Durchflussmengen der Schlauchpumpe.
Diskussion
Inzwischen ist bekannt, dass das menschliche Darmmikrobiom die Gesundheit und Krankheit des Wirts beeinflusst. Trotz des Wissens, das auf die Bedeutung unseres Mikrobioms hindeutet, insbesondere bei neurologischen Erkrankungen wie Alzheimer oder Parkinson 3,13, ist weitgehend unbekannt, wie das Darmmikrobiom mit dem enterischen Nervensystem und anschließend mit dem Gehirn interagiert.
Ein repräsentatives Modell, um die Wechselwirkungen zwischen dem Darmmikrobiom und dem Nervensystem zu untersuchen, war bisher nicht verfügbar. Studien zur Darm-Hirn-Achse wurden traditionell mit murinen Modellen durchgeführt13. Mäuse und Menschen haben 85 % ihrer Genomsequenzen gemeinsam14, aber es gibt erhebliche Unterschiede, die beim Vergleich von Mäusen und Menschen zu berücksichtigen sind. In Bezug auf den Darm ist es wichtig zu beachten, dass Mäuse im Vergleich zum Menschen ausschließlich Pflanzenfresser sind. Infolgedessen unterscheidet sich ihr Magen-Darm-Trakt in Länge und Merkmalen, wie z. B. der "Magenentleerung"14. Auch die Gehirne von Mäusen zeigen wichtige Unterschiede, wobei die Gesamtstruktur zwischen Mäusen und Menschen unterschiedlich ist15. Wichtig ist, dass Menschen längere Zellzykluszeiten von neuronalen Vorläuferzellen haben15. Daher ist es wichtig, repräsentative Modelle zu entwickeln, die menschliche Zellen, einschließlich Darm- und neuronaler Zellen, einbeziehen5. In diesem Zusammenhang verringert die Entwicklung reproduzierbarerer Forschungsarbeiten anhand von In-vitro-Modellen die Notwendigkeit der Verwendung von Tiermodellen und verbessert die Reproduzierbarkeit.
neuroHuMiX ist eine weiterentwickelte Version des bisherigen HuMiX-Modells9. HuMiX ist ein Gut-on-a-Chip-Modell, das proximale und repräsentative Co-Kulturen von Epithel- und Bakterienzellen ermöglicht. Die Zell-Zell-Kommunikation ist durch die proximale Co-Kultur und Diffusion von sezernierten Faktoren und Metaboliten über semipermeable Membranen möglich. Um jedoch den Nutzen des ersten Geräts zur Untersuchung der menschlichen Darmumgebung zu erweitern, ist die Einführung eines zusätzlichen Zelltyps erforderlich. Um dieses Problem zu lösen, ermöglicht neuroHuMiX, das mit der Einführung von iPSC-abgeleiteten ENs entwickelt wurde, eine proximale Co-Kultur von Bakterien, Darmepithelzellen und ENs. Das daraus resultierende In-vitro-Modell ermöglicht es uns, Fragen zum menschlichen Darmmikrobiom in Bezug auf das menschliche Nervensystem zu beantworten. Die Co-Kultivierung verschiedener Zelltypen, insbesondere von Co-Kulturen von Säugetierzellen und Bakterien, bringt mehrere Herausforderungen mit sich, darunter der Verlust der Lebensfähigkeit, die schlechte Adhäsion und der allgemeine Verlust des Konfluens16. Hier haben wir gezeigt, dass wir mit diesem Gerät in der Lage sind, drei verschiedene Zelltypen innerhalb desselben Systems zu co-kultivieren und gleichzeitig die Zelllebensfähigkeit hoch zu halten.
Ein kritischer Schritt im Protokoll besteht darin, die Konfluenz der neuronalen Zellen – 80 % bis 90 % Zellkonfluenz und Lebensfähigkeit – sicherzustellen, bevor sie in das Gerät inokuliert werden. Da es nicht möglich ist, das Zellwachstum während des Laufs zu beurteilen, ist es von größter Bedeutung, sicherzustellen, dass die Zellen konfluieren und gut wachsen, bevor sie in das Modell eingeführt werden. Obwohl dies ein limitierender Faktor sein kann, ist die allgemeine Lebensfähigkeit und Konfluenz, die innerhalb des Geräts beobachtet wird, im Allgemeinen hoch.
Das Gerät ist über Schlauchleitungen mit einer Schlauchpumpe verbunden. Jede Zellkammer hat ihre eigene Schlauchleitung. Der Schlauch umfasst einen Pumpenschlauch, der die Verwendung einer Peristaltikpumpe für die Perfusion von Medium ermöglicht, sowie einen Schlauch, der den Pumpenschlauch mit dem Gerät verbindet, und einen Schlauch, der das Gerät mit den Ausfluss-/Abfallflaschen verbindet. Vor und nach dem Gerät befinden sich Probenahmeöffnungen, um die Inokulation und Probenahme des Abflussmediums zu ermöglichen. Jede Kammer kann mit einem anderen Medium verbunden werden, was die besten Kulturbedingungen für jeden einzelnen Zelltyp ermöglicht. Jede Kammer kann je nach den spezifischen Anforderungen an die Medienversorgung geöffnet oder geschlossen werden. In dem Gerät bleibt die neuronale Kammer während des größten Teils des Experiments geschlossen, während die Bakterien- und Epithelkammern die ganze Zeit geöffnet sind, was bedeutet, dass sie während des gesamten Versuchslaufs frisches Medium erhalten. Um sicherzustellen, dass das Medium ohne Unterbrechung fließt, ist es wichtig, dass sich keine Luft mehr in den Schläuchen, Anschlüssen oder im Gerät befindet. Daher ist es wichtig, die Geräte beim Grundieren zunächst einige Minuten laufen zu lassen. Dadurch wird das Problem oft behoben. Ist dies nicht der Fall, kann eine der anderen Leitungen, die fallen, für kurze Zeit geschlossen werden, indem der Drei-Wege-Absperrhahn des Abflusses geschlossen wird. Dadurch wird das Medium auf die Leitung mit der Luftblase umgeleitet und so das Problem gelöst, indem die Blase durch den Schlauch nach außen gedrückt wird.
Bei jedem Zellkulturexperiment ist das Medium eine Schlüsselkomponente, wobei jeder Zelltyp sein jeweiliges Medium hat. In einem Co-Kultur-Setup muss das Medium nicht nur für den darin wachsenden Zelltyp, sondern auch für die anderen Zelltypen innerhalb der Co-Kultur kompatibel sein. Dies ist bei dem Gerät nicht anders, was eine zusätzliche Herausforderung darstellt, da wir drei verschiedene Kompartimente mit drei verschiedenen Zelltypen im Inneren haben - bakterielle, epitheliale und neuronale Zellen. Wir haben jedoch gezeigt, dass durch Modifikation des bakteriellen Mediums - mit der Zugabe von 5% MRS zu RPMI 1640 mit 10% FBS - alle Zelltypen, insbesondere Bakterien- und Epithelzellen, erfolgreich innerhalb des Systems co-kultiviert werden können. In dem Gerät werden jedoch verschiedene Zelltypen in unmittelbarer Nähe kokultiviert und stehen daher nicht in direktem Kontakt miteinander. Auch wenn dies nicht vollständig repräsentativ für den direkten Kontakt zwischen Zellen im menschlichen Darm und daher eine Einschränkung darstellt, ist die proximale und repräsentative Co-Kultur-Bedingung eine Stärke für nachgeschaltete Analysen. Austausch löslicher Faktoren zwischen den verschiedenen Kammern und Zelltypen; Daher interagieren die Zellen immer noch miteinander. Die Tatsache, dass die Zelltypen separat geerntet und analysiert werden können, ermöglicht es uns außerdem, die Wirkung eines gesunden und/oder kranken Mikrobioms auf verschiedene Zelltypen (einschließlich neuronaler Zellen) zu untersuchen und dadurch zelltypspezifische Messwerte zu bestimmen/abzurufen. Eine weitere Einschränkung besteht darin, dass die Morphologie der Zellen während des Versuchslaufs nicht verfolgt werden kann, da das Gerät nur am Ende eines jeden Experiments geöffnet und die Zellen überprüft werden können.
Unseres Wissens nach ist neuroHuMiX das erste Gut-on-a-Chip-Modell mit ENs. Dies ist ein Schritt zur Aufklärung der Kommunikation zwischen der Darmmikrobiota und dem enterischen Nervensystem. Es handelt sich um ein Modell, mit dem das Zusammenspiel zwischen einer Bakterienart, einer Epithelschicht und ENs untersucht werden kann. Sein Design ermöglicht es uns, den Austausch von löslichen Faktoren, die von den verschiedenen Zelltypen abgesondert werden, und ihre Wirkung aufeinander zu untersuchen. Für die Zukunft wäre es wichtig, nicht nur iPSC-abgeleitete ENs, sondern auch iPSC-abgeleitete Epithelzellen im Gerät zu haben, um das Gerät in ein personalisiertes Modell zu überführen. Wichtig ist, dass dieses personalisierte Modell verwendet werden könnte, um Prä-, Pro- und Synbiotika zu testen 10,11 und möglicherweise personalisierte Screening- und Therapieansätze zu entwickeln17. Personalisiertes neuroHuMiX könnte schließlich Licht auf die "dunkle Materie" des menschlichen Darmmikrobioms und seine Wechselwirkungen mit dem Nervensystem entlang der Achse Darmmikrobiom-Nervensystem werfen und den Weg für therapeutische Untersuchungen und Interventionen ebnen.
Wir können schlussfolgern, dass die Möglichkeit, einen Darm-on-a-Chip zu haben, der das enterische neuronale System einschließt, entscheidend ist, um Fortschritte bei der Untersuchung und dem Verständnis der Wechselwirkungen entlang der Achse zwischen Darmmikrobiom und Nervensystem zu erzielen. NeuroHuMiX ermöglicht es uns, die Auswirkungen von Bakterienarten auf Wirtszellen zu untersuchen und liefert uns eine gute Grundlage, um das Modell noch physiologisch repräsentativer zu verbessern.
Offenlegungen
P.W. erklärt, in den Patenten PCT/EP2013/056607, PCT/EP2016/062024, PCT/US2017/061602 und PCT/EP2019/081424 als Erfinder aufgeführt zu sein. P.W., C.S. und L.G. erklären, dass sie als Erfinder im Patent LU503075 aufgeführt sind.
Danksagungen
Die Autoren danken Dr. Jared Sterneckert für die Bereitstellung der Zellen aus der K7-Linie. Wir möchten uns auch bei den langjährigen Mitarbeitern Dr. Frederic Zenhausern und Matthew W. Barret von der University of Arizona für ihre Unterstützung bei den technischen Aspekten bedanken. Wir möchten uns auch bei Dr. Valentina Galata für ihre Hilfe bei der Gestaltung der schematischen Darstellung von neuroHuMiX bedanken. Dieses Projekt wurde vom Europäischen Forschungsrat (ERC) im Rahmen des Forschungs- und Innovationsprogramms Horizon 2020 der Europäischen Union gefördert (Grant Agreement 863664). Abbildung 1 wurde teilweise mit Biorender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma Aldrich | 10712 | |
| Aeration cannula (length: 1.10 diameter: 30 mm) | VWR (B.Braun) | BRAU4190050 | |
| Agar-agar | Merck Millipore | 1.01614.1000 | |
| Aluminium Crimp | Glasgerätebau Ochs | 102050 | |
| Ascorbic acid | Sigma Aldrich | A4544 | |
| B-27 Supplement Minus Vitamin A (50x) | Gibco | 12587-010 | |
| Bacterial Cell Membrane, pore size: 1 µm | VWR (Whatman) | 515-2084 | |
| Caco-2 cells | DSMZ | ACC169 | |
| Cell Counter & Analyzer CASY | OMNI Life Sceince | ||
| CHIR | Axon Mechem BV | CT99021 | |
| Collagen I, Rat Tail | Invitrogen | A1048301 | |
| Costar 6-well Clear Flat Bottom Ultra-Low Attachment Plates | Corning | 3471 | |
| Difco Lactobacilli MRS Broth | BD Biosciences | 288130 | |
| Discofix 3-way stopcock | B. Braun | BRAU40951111 | |
| DMEM/F12, no glutamine | Thermofisher Scientific | 21331020 | |
| Dulbecco's Phosphate-Buffered Saline, D-PBS | Sigma Aldrich | 14190-169 | |
| Essential 6 Medium | Thermofisher Scientific | A1516401 | |
| Essential 8 Medium | Thermofisher Scientific | A1517001 | |
| Female Luer Lock to Barb Connector | Qosina | 11733 | |
| FGF2 | R&D Systems | 233-FB | |
| Fibronectin | Sigma Aldrich | F1141 | |
| Foetal Bovine Serum, FBS | Thermofisher Scientific | 10500-064 | |
| GDNF | PeproTech | 450-10 | |
| Human Cell Membrane, pore size: 50 nm | Sigma Aldrich (GE Healthcare) | WHA111703 | |
| HuMiX Gasket Collagen | Auer Precision | 216891-003 | |
| HuMiX Gasket Sandwich Bottom | Auer Precision | 216891-002 | |
| HuMiX Gasket Sandwich Top | Auer Precision | 216891-001 | |
| iPSC | Max Planck Institute for Molecular Biomedicine | K7 line | |
| L-Glutamine (200 mM) | Gibco | 25030081 | |
| Laminin from Engelbreth-Holmswarm | Sigma Aldrich | L2020 | |
| LDN193189 | Sigma Aldrich | SML0559 | |
| Limosilactobacillus reuteri | ATCC | 23272 | |
| Live/Dead BacLight Bacterial Viability kit | Thermofisher Scientific | L7012 | |
| Male Luer with Spin Lock to Barb | Qosina | 11735 | |
| Marprene tubing (0.8 mm x 1.6 mm) | Watson-Marlow | 902.0008.J16 | |
| Matrigel hESC-qualified matrix | Corning | 354277 | |
| Mucin, from porcine stomach | Sigma Aldrich | T3924 | |
| N2 Supplement (100x) | Gibco | 17502048 | |
| NEAA | Thermofisher Scientific | 11140050 | |
| Needle (length: 120 mm; diameter: 0.80 mm) | B.Braun (color code: green) | 466 5643 | |
| Needle (length: 40 mm; diameter: 0.70 mm) | Henke Sass Wolf (color code: black) | 4710007040 | |
| Needle (length: 80 mm; diameter: 0.60 mm) | B.Braun (color code: blue) | 466 5635 | |
| Neurobasal Medium | Gibco | 21103049 | |
| PE/Cy7 anti-human CD49d antibody | Biolegend | 304314 | |
| Penicillin-Streptomycin | Sigma Aldrich | P0781 | |
| Peristaltic pump | Watson-Marlow | 205CA | |
| Poly-L-ornithine Hydrobromide | Sigma Aldrich | P3655 | |
| Polycarbonate lids (HuMiX) | University of Arizona | HuMiX 1.0 / 2.0 | |
| Retinoic Acid | Sigma Aldrich | R2625 | |
| RLT Buffer (RNeasy Minikit) | Qiagen | 74104 | |
| RPMI 1640 Medium | Thermofisher Scientific | 72400-021 | |
| SB431542, ALK inhibitor | Abcam | ab120163 | |
| Serum bottles | Glasgerätebau Ochs | 102091 | |
| Syringe | BD Biosciences | 309110 | |
| Trypsin-EDTA solution | Sigma Aldrich | T3924 | |
| Y-27632 Dihydrochloride | R&D Systems | 1254 |
Referenzen
- Heintz-Buschart, A., Wilmes, P. Human gut microbiome: function matters. Trends in Microbiology. 26 (7), 563-574 (2018).
- Toor, D., et al. Dysbiosis disrupts gut immune homeostasis and promotes gastric diseases. International Journal of Molecular Sciences. 20 (10), 2432 (2019).
- Braak, H., de Vos, R. A. I., Bohl, J., Del Tredici, K. Gastric α-synuclein immunoreactive inclusions in Meissner's and Auerbach's plexuses in cases staged for Parkinson's disease-related brain pathology. Neuroscience Letters. 396 (1), 67-72 (2006).
- Schmit, K. J., et al. Dietary fibre deprivation and bacterial curli exposure shift gut microbiome and exacerbate Parkinson's disease-like pathologies in an alpha-synuclein-overexpressing mouse. bioRxiv. , (2022).
- Fritz, J. V., Desai, M. S., Shah, P., Schneider, J. G., Wilmes, P. From meta-omics to causality: experimental models for human microbiome research. Microbiome. 1 (1), 14 (2013).
- Fattahi, F., et al. Deriving human ENS lineages for cell therapy and drug discovery in Hirschsprung disease. Nature. 531 (7592), 105-109 (2016).
- Wu, Q., et al. Organ-on-a-chip: Recent breakthroughs and future prospects. BioMedical Engineering Online. 19 (1), 9 (2020).
- May, S., Evans, S., Parry, L. Organoids, organs-on-chips and other systems, and microbiota. Emerging Topics in Life Sciences. 1 (4), 385-400 (2017).
- Shah, P., et al. A microfluidics-based in vitro model of the gastrointestinal human-microbe interface. Nature Communications. 7, 11535 (2016).
- Greenhalgh, K., et al. Integrated in vitro and in silico modeling delineates the molecular effects of a synbiotic regimen on colorectal-cancer-derived cells. Cell Reports. 27 (5), 1621-1632 (2019).
- Mao, J. H., et al. Genetic and metabolic links between the murine microbiome and memory. Microbiome. 8 (1), 53 (2020).
- Moysidou, C. M., Owens, R. M. Advances in modelling the human microbiome-gut-brain axis in vitro. Biochemical Society Transactions. 49 (1), 187-201 (2021).
- Kim, S., et al. Transneuronal propagation of pathologic α-synuclein from the gut to the brain models Parkinson's disease. Neuron. 103 (4), 627-641 (2019).
- Hugenholtz, F., de Vos, W. M. Mouse models for human intestinal microbiota research: a critical evaluation. Cellular and Molecular Life Sciences. 75 (1), 149-160 (2018).
- Marshall, J. J., Mason, J. O. Mouse vs man: Organoid models of brain development & disease. Brain Research. 1724, 146427 (2019).
- Goers, L., Freemont, P., Polizzi, K. M. Co-culture systems and technologies: taking synthetic biology to the next level. Journal of the Royal Society Interface. 11 (96), 20140065 (2014).
- Sedrani, C., Wilmes, P. Toward hypothesis-driven, personalized microbiome screening. Cell Reports Methods. 2 (1), 100139 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten