Method Article
Einsatz minimal-invasiver Methoden zur Beurteilung des Kraftstoffverbrauchs und des zirkadianen Rhythmus bei älteren Erwachsenen
In diesem Artikel
Zusammenfassung
Es wird ein neuartiges und minimal-invasives Protokoll vorgestellt, um die synergistischen Auswirkungen der Brennstoffnutzung und des zirkadianen Rhythmus auf alternde Personen unter Verwendung von mononukleären Zellen des peripheren Blutes zu bewerten.
Zusammenfassung
Das Altern ist mit mehreren physiologischen Veränderungen verbunden, die synergetisch und unabhängig voneinander zu körperlicher Behinderung und dem Risiko chronischer Krankheiten beitragen. Obwohl die Ätiologie der altersbedingten körperlichen Behinderung komplex und multifaktoriell ist, scheint der Rückgang der mitochondrialen Funktion mit dem Fortschreiten des funktionellen Verfalls bei vielen älteren Erwachsenen zusammenzufallen. Der Grund, warum es mit zunehmendem Alter zu einer Abnahme der mitochondrialen Funktion kommt, bleibt schwer fassbar, aber neue wissenschaftliche Erkenntnisse deuten darauf hin, dass sowohl der Kraftstoffstoffwechsel als auch der zirkadiane Rhythmus die mitochondriale Funktion beeinflussen können.
Jüngste Studien haben gezeigt, dass zirkadiane Rhythmen mit zunehmendem Alter gestört werden und dass gestörte zirkadiane Rhythmen pathologische Folgen haben, die sich auf die mitochondriale Funktion auswirken und sich mit vielen altersbedingten chronischen Krankheiten überschneiden. Die derzeitigen quantitativen Methoden zur direkten Beurteilung der mitochondrialen Funktion sind invasiv und erfordern in der Regel eine Muskelbiopsie, was angesichts des wahrgenommenen Ausmaßes potenzieller Schmerzen und Risiken Schwierigkeiten bei der Rekrutierung von Teilnehmern und der Einhaltung der Studie darstellen kann. Daher wurde ein innovatives und relativ nicht-invasives Protokoll zur Beurteilung von Veränderungen der mitochondrialen Funktion auf zellulärer Ebene und zirkadianer Muster bei älteren Erwachsenen angepasst. Konkret wird ein Echtzeit-Stoffwechselflussanalysator verwendet, um die mitochondriale bioenergetische Funktion der weißen Blutkörperchen unter unterschiedlicher Substratverfügbarkeit zu bewerten.
Die Expression von Genen der zirkadianen Uhr in weißen Blutkörperchen, die mit der mitochondrialen Bioenergetik und den zirkadianen Rhythmus korrelieren, wird ebenfalls analysiert. Es wird angenommen, dass diese innovativen methodischen Ansätze zukünftige klinische Studien unterstützen werden, indem sie minimalinvasive Methoden zur Untersuchung der mitochondrialen Substratpräferenz und des zirkadianen Rhythmus bei älteren Erwachsenen bereitstellen.
Einleitung
Die Fortschritte des letzten Jahrhunderts haben zu einem Anstieg der Lebenserwartung und der Bevölkerung alternder Erwachsener geführt. Mit Blick auf die Zukunft wird der Anteil der Erwachsenen im Alter von 65 Jahren und älter in den Vereinigten Staaten von 2020 bis 2050 voraussichtlich um 5 % steigen1. Dieser Anstieg der Lebenserwartung impliziert keine Verlängerung der Gesundheitsspanne - der Lebensspanne, die mit unabhängigem Funktionieren verbunden ist. Die Realität ist, dass das Altern von unzähligen biologischen Veränderungen begleitet wird, die den Zellstoffwechsel und die Physiologie beeinflussen und zu einer allmählichen Verschlechterung der kognitiven und körperlichen Funktionen führen 2,3. Da die Lebenserwartung der Menschen weiter steigt, besteht ein größerer Bedarf, die Funktionsfähigkeit und Unabhängigkeit im Alter von4 Jahren zu erhalten.
Es ist seit langem bekannt, dass die Abnahme der körperlichen Funktion und Unabhängigkeit mit zunehmendem Alter multifaktoriell ist, obwohl sie häufig mit dem Auftreten chronischer Erkrankungen und akuter auslösender Ereignisse verbunden ist5. Umgekehrt wurde gezeigt, dass diese Abnahme der körperlichen Leistungsfähigkeit und der Muskeleigenschaften mit der Entwicklung einer Behinderung im Alter verbunden ist, ohne dass ein eindeutiger Zusammenhang mit einer einzigen Krankheit besteht6. Angesichts der Schwierigkeiten, die genaue Ätiologie chronischer Erkrankungen und körperlicher Behinderungen zu kennen, wurde angenommen, dass Beeinträchtigungen der mitochondrialen Funktion mit dem Auftreten und Fortschreiten chronischer Erkrankungen und dem Verlust der körperlichen Funktion bei alternden Erwachsenen zusammenfallen 7,8.
Die Mitochondrien liefern den Großteil des Adenosintriphosphats (ATP), das für viele zelluläre Prozesse notwendig ist9. Hochoxidatives Gewebe ist auf Mitochondrien angewiesen, um eine angemessene Energieproduktion zu gewährleisten. mit zunehmendem Alter nehmen die oxidative Kapazität und die mitochondriale ATP-Synthese ab. Dieser Rückgang ist zum Teil auf oxidative Schäden an der mitochondrialen DNA (mtDNA) zurückzuführen, die zu einer inkrementellen Akkumulation von mtDNA-Mutationen und -Deletionen führen10. Die Akkumulation von mtDNA-Mutationen und -Deletionen führt zu einer Abnahme der Bildung von funktionellen Elektronentransportkettenproteinen, was zu einer verminderten Fähigkeit der Zellen führt, ATP zu produzieren. Der altersbedingte Rückgang der mitochondrialen Funktion ist am deutlichsten in stark oxidativen Geweben wie dem Herzen und der Skelettmuskulatur11. Studien haben gezeigt, dass die Mitochondrien des Gastrocnemius-Muskels in älteren Rattenproben im Vergleich zu jüngeren Proben eine Verringerung der ATP-Produktion und des ATP-Gehalts um etwa 50 % aufweisen12. Darüber hinaus konnte gezeigt werden, dass die Kapazität der mitochondrialen ATP-Produktion in der menschlichen Skelettmuskulatur um etwa 8% pro Lebensjahrzehnt abnimmt13. Diese Ergebnisse deuten darauf hin, dass ein altersbedingter Rückgang der mitochondrialen Funktion zu einer verminderten Energieproduktion in Organismen beitragen kann.
Es wird angenommen, dass ein Schlüsselregulator der mitochondrialen Aktivität der Peroxisom-Proliferator-aktivierte Rezeptor-γ (PPARγ)-Koaktivator-1 (PGC-1α)14 ist. Eine Verschlechterung der PGC-1α-Aktivität oder eine Abnahme seiner Häufigkeit führt zu einer verminderten mitochondrialen oxidativen Aktivität und folglich zu einer beeinträchtigten Energieproduktion. Darüber hinaus kann eine Abnahme der mitochondrialen Qualität die Qualität der Skelettmuskulatur beeinträchtigen und in der Folge zur Entwicklung oder Verschlimmerung von Sarkopenie, Dynapenie und Abnahme der Funktionsfähigkeit führen15,16. Die Evidenz für den altersbedingten gleichzeitigen Rückgang der mitochondrialen Funktion und der Skelettmuskelqualität deutet auf einen Zusammenhang zwischen mitochondrialer Beeinträchtigung und der Pathogenese des funktionellen Verfalls hin17. Kürzlich wurde dies bei funktionellen älteren Erwachsenen bestätigt, die in der Funktionsgemeinschaft leben, und zeigt, dass eine Verringerung des Mitochondrienstoffwechsels der Skelettmuskulatur einen Rückgang der Mobilität in dieser Population vorhersagt18. Obwohl der genaue Mechanismus, der zum mitochondrialen Rückgang mit zunehmendem Alter führt, unklar ist, haben neuere Erkenntnisse ein wechselseitiges Zusammenspiel zwischen der zirkadianen Uhr und der mitochondrialen Funktion hervorgehoben, mit Folgen für die mitochondriale Brennstoffverwertung und die Biogenese19.
Kraftstoffverbrauch
Die mitochondriale Funktion scheint durch den Kraftstoffstoffwechsel und die Art des Brennstoffs beeinflusst zu werden, der auf zellulärer Ebene im Skelettmuskelgewebe verwendet wird11. In Zeiten des Brennstoffmangels, insbesondere des Kohlenhydratmangels beim Menschen, ändert sich die Brennstoffpräferenz für die (mitochondriale) Energieproduktion. Bei niedrigen Glukosespiegeln verschiebt sich die Brennstoffpräferenz weg von Glukose hin zu Fettsäuren und von Säuren abgeleiteten Ketonkörpern. Dieser Stoffwechselschalter ist gekennzeichnet durch die Hochregulierung des Fettstoffwechsels in den Adipozyten, gefolgt von einer erhöhten Freisetzung von Ketonen ins Blut4. Die Umstellung der Brennstoffnutzung von Glukose auf Ketone bei einer ketogenen Diät scheint sich positiv auf die Produktion mitochondrialer reaktiver Sauerstoffspezies, die antioxidative Abwehr, die ATP-Synthese und die Biogenese auszuwirken20.
Die metabolische Umstellung vom Kohlenhydrat- auf den Fettstoffwechsel erfolgt in Zeiten geringer Nährstoffverfügbarkeit in der Umwelt und wenn die Glykogenspeicher erschöpft sind. Wenn dieser Schalter eingeleitet wird, werden gespeicherte Triglyceride in Glycerin, ein Substrat für die Glukoneogenese, und freie Fettsäuren abgebaut, die in die Leber transportiert werden, um über β-Oxidation zu Acetyl-Coenzym A (Acetyl-CoA) oxidiert zu werden. Ketonkörper werden hauptsächlich in der Leber durch eine zweistufige Kondensation von drei Acetyl-CoA-Molekülen zu β-Hydroxy-β-methylglutaryl-CoA synthetisiert, die dann zu Ketonkörpern, einschließlich Acetoacetat und 3-βeta-Hydroxybutyrat21, weiterverarbeitet werden. Diese Ketonkörper werden in Geweben im ganzen Körper verteilt, wobei der höchste Verbrauch im Herzen, im Gehirn und in der Skelettmuskulatur auftritt21. Mit zunehmendem Alter wird die Oxidation der mitochondrialen Fettsäuren beeinträchtigt, wodurch der Stoffwechselschalter beeinflusstwird 22. Es wurde vorgeschlagen, dass Beeinträchtigungen der mitochondrialen Brennstoffverwertung zu einer weiteren mitochondrialen Dysfunktion führen, die wiederum zu altersbedingten Erkrankungen und funktionellem Verfall beiträgt23.
Veränderungen des mitochondrialen Sauerstoffverbrauchs von mononukleären Zellen des peripheren Blutes (PBMCs) wurden untersucht, um die Muster zu bewerten, die mit Dysfunktion und Vaskularisation verbunden sind. Hartman et al. führten eine Studie durch, die darauf abzielte, die Korrelation zwischen Sauerstoffverbrauch und vielfältig vermittelter Dilatation zu bestimmen, was auf einen Zusammenhang zwischen mitochondrialer Dysfunktion und Dysfunktion der vaskulären glatten Muskelzellen hindeutete24. In Bezug auf andere Organe wurden PBMCs mit höheren kognitiven und Gehirnfunktionen korreliert, wie durch Respirometrie bestimmt wurde25. Somit können die PBMC-Bioenergetik und die Atmungskapazität als potenzielle Biomarker für die Beurteilung der Funktionsfähigkeit von Organen oder Geweben im gesamten Körper dienen.
Circadianer Rhythmus
Ein weiterer wichtiger Faktor, der die Gesundheit der Mitochondrien beeinflusst, ist der zirkadiane Rhythmus. Zirkadiane Rhythmen sind ~24 h Schwingungen im Verhalten und in der Physiologie, die in Abwesenheit von Umweltreizen auftreten26. Diese Rhythmen funktionieren auf prädiktive Weise, um die Homöostase des Systems und des Gewebes zu unterstützen. Der Mechanismus, der den zirkadianen Rhythmen zugrunde liegt, ist eine Transkriptions-Translations-Rückkopplungsschleife, die als zirkadiane Uhrbezeichnet wird 27. In den letzten 15 Jahren wurde gezeigt, dass der zirkadiane Uhrmechanismus in praktisch allen Zellen des Körpers vorhanden ist28. Neben der Zeitmessung trägt der molekulare Uhrmechanismus auch zu einem täglichen Programm der Genexpression bei, das als zirkadiane Uhr bezeichnet wird29. Die Clock-Output-Gene sind für jeden Gewebetyp einzigartig und funktionell mit Signalwegen verbunden, die für den Zellstoffwechsel, die Autophagie, die Reparatur und die Homöostase wichtig sind. Jüngste Erkenntnisse haben gezeigt, dass die Gesundheit der Mitochondrien von der Funktion der zirkadianen Uhr abhängt und die mitochondriale Funktion beeinflusst, einschließlich der mitochondrialen Biogenese, der Brennstoffverwertung und der Mitophagie30.
Neue Erkenntnisse sowohl in präklinischen als auch in klinischen Studien haben gezeigt, dass es im Laufe des Alterns zu Störungen des zirkadianen Rhythmus kommt31. Dazu gehören Störungen des normalen Schlaf- und Wachzyklus, eine verminderte Amplitude der Körperkerntemperaturrhythmen und eine verzögerte Fähigkeit, sich an Verschiebungen in Phase31 anzupassen. Eine Studie forderte beispielsweise das zirkadiane System von erwachsenen und alten (20+ Monaten) Mäusen heraus, indem sie den Lichtplan um 6 Stunden verschoben hat. Es zeigte sich, dass die alten Mäuse länger brauchten, um ihre Aktivitätsmuster wieder an den neuen Lichtplan anzupassen32. In Übereinstimmung mit den Veränderungen des zirkadianen Verhaltens ergab die Analyse der Gewebeuhren, dass sowohl die zentralen als auch die peripheren Gewebeuhren in der alternden Kohorte beeinträchtigt waren.
In jüngerer Zeit haben mehrere Gruppen eine transkriptomische Analyse der zirkadianen Uhr und der Uhrausgabe in verschiedenen Geweben im Alter von33 Jahren durchgeführt. Die Ergebnisse dieser Studien zeigen, dass es mit zunehmendem Alter zu einer groß angelegten Reprogrammierung der zirkadianen Uhr kommt. Das bedeutet, dass, obwohl die Kernuhr eine Timing-Funktion beibehält, die Gene, auf die die tägliche Expression abzielt, weitgehend unterschiedlich sind. Zum Beispiel haben zwei Studien Muskelbiopsien von menschlichen Probanden alle 4 Stunden für 24 Stunden gesammelt, wobei die Ergebnisse zeigten, dass der Höhepunkt und das Tief der Uhrgenexpression zwischen nachtaktiven Nagetieren und tagaktiven Menschen umgekehrt sind 34,35,36. Dies deutet darauf hin, dass, wenn die Expression des Uhrgens ausschließlich auf der Grundlage der aktiven und der Ruhephase (und nicht der hellen und dunklen Phase) verglichen wird, die Muster der Uhrgenexpression in den Muskeln zwischen den Arten praktisch gleich sind. Es wird vermutet, dass diese altersassoziierte Änderung der Uhrleistung zu Beeinträchtigungen in der Regulation von Signalwegen führt, die die bekannten Kennzeichen des Alterns wie mitochondriale Funktion, DNA-Schäden und -Reparatur sowie Autophagie umfassen37.
Begründung der Studie
Der Zusammenhang zwischen der mitochondrialen Funktion und dem Rückgang der körperlichen Funktion ist gut belegt. Die zugrunde liegende Ursache der mitochondrialen Dysfunktion bleibt jedoch umstritten. Neuere Forschungen deuten darauf hin, dass die zelluläre Brennstoffnutzung und der zirkadiane Rhythmus eine Rolle bei diesem Prozess spielen könnten. Traditionelle Methoden zur Bewertung der mitochondrialen Funktion, wie z. B. die Messung des mitochondrialen Sauerstoffverbrauchs in einer Muskelbiopsieprobe, werden oft als schmerzhaft und invasiv empfunden, was insbesondere bei Populationen mit geringer Muskelmasse, wie z. B. gebrechlichen und sarkopenischen Erwachsenen, von der Teilnahme abhaltenkann 38.
Angesichts dieser Einschränkungen besteht ein Bedarf an einer weniger invasiven Methode zur Beurteilung von Veränderungen der zellulären Brennstoffverwertung und des zirkadianen Rhythmus bei älteren Erwachsenen. Ziel dieser Studie ist es, ein neuartiges, minimal-invasives Protokoll zu evaluieren, das zur Beurteilung des Kraftstoffstoffwechsels und des zirkadianen Rhythmus in dieser Population verwendet werden kann. Die Ergebnisse dieser Studie werden zu einem besseren Verständnis der altersbedingten Veränderungen und des Ansprechens auf medizinische oder verhaltensbezogene Interventionen beitragen und als Modell für zukünftige Studien in diesem Bereich dienen.
Protokoll
Verfahren mit menschlichen Teilnehmern wurden von der Forschungsethikkommission (Florida Ethics Policy 1.0104) und dem Institutional Review Board der University of Florida genehmigt.
1. Mitochondriale Funktion
- Isolierung von mononukleären Zellen des peripheren Blutes (PBMCs)
- Sammeln Sie PBMCs mit speziellen 8-ml-Blutentnahmeröhrchen (16 mm x 125 mm; enthalten entweder 0,1 M Natriumcitrat-Antikoagulans oder Natriumheparin-Antikoagulans) (siehe Materialtabelle).
HINWEIS: Die Blutentnahmeröhrchen enthalten Bluttrennmedien, die aus einem thixotropen Polyestergel und einem Gradientenmedium bestehen (siehe Materialtabelle), um die Trennung der weißen Blutkörperchen zu verbessern. - Verarbeiten Sie die 8-ml-Zellvorbereitungsröhrchen innerhalb von 2 Stunden nach der Entnahme, um brauchbare Ergebnisse zu erzielen.
- Mischen Sie die Blutentnahmeröhrchen nach dem Befüllen mit Blut (entweder durch sanfte Inversion oder auf einem Mixer) und halten Sie sie maximal 2 h bei Raumtemperatur (RT).
- Die Blutentnahmeröhrchen werden in einem passenden Ausschwingrotor 15 min lang bei 2.000 × g (oder entsprechend 30.000 g-min; nicht mehr als 2.000 × g) bei RT zentrifugiert.
HINWEIS: Diese Röhrchen sind höher als ein normales 15-ml-Zentrifugenröhrchen; Daher muss zusätzlich auf die Verwendung des richtigen Rotors geachtet werden. - Aspirieren und entsorgen Sie ~80% der Plasmaschicht in einen Behälter für biogefährliche Abfälle und sammeln Sie die Zellschicht aus dem Blutentnahmeröhrchen (mit einer Transferpipette) in einem 15-ml-Zentrifugenröhrchen.
HINWEIS: Wenn Sie ein 15-ml-Röhrchen verwenden, fügen Sie 7 ml Zellschicht hinzu und füllen Sie 14 ml mit steriler phosphatgepufferter Kochsalzlösung (PBS) auf. Das Verhältnis von Zellen zu PBS sollte 1:1 betragen. Verwenden Sie bei Bedarf mehrere Röhrchen. Vorsichtig durch Wenden mischen. - In einem Ausschwingrotor bei 900 × g für 5 min (oder entspricht 4.500 G-min) bei RT zentrifugieren.
- Ohne das Zellpellet zu stören, aspirieren Sie die PBS-Lösung und entsorgen Sie sie in einem Behälter für biologisch gefährliche Abfälle.
- Resuspendieren Sie das Zellpellet in 1 mL Basalmedium (BM; siehe Materialtabelle) durch sanftes Zerreiben. Fügen Sie das BM zu insgesamt 10 ml hinzu und mischen Sie es vorsichtig durch Invertieren. Führen Sie eine Zellzählung durch.
- Die Zellsuspension wird in einem Ausschwingrotor bei 900 × g 5 min bei RT zentrifugiert.
HINWEIS: Wenn Zellen am selben Tag für die bioenergetische Bewertung verwendet werden, fahren Sie mit Schritt 1.1.10 fort. Wenn die Zellen für die Lagerung eingefroren sind, fahren Sie mit Schritt 1.1.13 fort. - Berechnen Sie das Volumen des Testmediums (AM; siehe Materialtabelle) für die Resuspension des nächsten Zellpellets, um die gewünschte Zellkonzentration zu erreichen.
HINWEIS: Bei einer Aussaatdichte von 150.000 Zellen/Well in 50 μl beträgt die gewünschte Konzentration der Zellsuspension beispielsweise 3 Millionen Zellen/ml. - Ohne das Zellpellet zu stören, saugen Sie den Überstand an und entsorgen Sie ihn in einen Behälter für biologisch gefährliche Abfälle.
- Resuspendieren Sie das Zellpellet vorsichtig in 1 ml oder weniger AM. Fügen Sie AM zum berechneten Gesamtvolumen für die gewünschte Zellkonzentration hinzu. Die Zellen sind nun bereit, in eine beschichtete Zellkulturplatte ausgesät zu werden (siehe Materialtabelle).
HINWEIS: Die folgenden Schritte gelten für das Einfrieren der Zellen für die Lagerung (1.1.13-1.1.16) und gelten nicht für das Verfahren am selben Tag. - Zum Einfrieren von Zellen aspirieren Sie den Überstand, ohne das Zellpellet zu stören, und entsorgen Sie ihn in einen Behälter für biologisch gefährliche Abfälle.
- Resuspendieren Sie die Zellen vorsichtig in Gefriermedien (siehe Materialtabelle) in der gewünschten Konzentration (5-10 Millionen Zellen/ml). Aliquot der gewünschten Volumina in markierten Kryofläschchen.
- Die Zellsuspension wird in einem Gefrierbehälter (siehe Materialtabelle) mindestens 4 Stunden lang oder über Nacht bei -80 °C langsam eingefroren.
- Nach 4 h oder am nächsten Morgen werden die Kryofläschchen in ein Lager für flüssigen Stickstoff überführt, wo sie sich in der Dampfphase befinden sollten.
- Beschichten Sie am Tag vor der Messung die Well-Böden einer Zellkultur-Mikroplatte (siehe Materialtabelle) mit Zellkleber (siehe Materialtabelle). Bereiten Sie den Klebstoff gemäß den Anweisungen des Herstellers mit einer empfohlenen Konzentration von 22,4 μg/μl vor, indem Sie das entsprechende Volumen der Klebstoff-Stammlösung in 0,1 M Natriumbicarbonat (pH 8,0) verdünnen. Tragen Sie 25 μl Klebstoff auf jede Vertiefung der Zellkultur-Mikroplatte auf.
- Nach 20 Minuten saugen Sie die restliche Flüssigkeit aus jeder Vertiefung ab und waschen Sie die Vertiefungen zweimal mit 200 μl sterilem, gereinigtem Wasser. Lassen Sie die Platte in der Biosicherheitswerkbank trocknen (für ~2 h).
- Wickeln Sie die Platte in Alufolie ein oder legen Sie sie in einen geschlossenen Zweitbehälter und lagern Sie sie im Kühlschrank bei 4 °C.
- Sammeln Sie PBMCs mit speziellen 8-ml-Blutentnahmeröhrchen (16 mm x 125 mm; enthalten entweder 0,1 M Natriumcitrat-Antikoagulans oder Natriumheparin-Antikoagulans) (siehe Materialtabelle).
- Bioenergetische Bewertung von PBMCs mit einem Theal-Time-Metabolic Flux Analyzer
HINWEIS: Bei Verwendung eines Flussmittelanalysators und des Multimode-Lesegeräts (siehe Tabelle der Materialien) wurde eine optimale Aussaatdichte von 150.000 PBMCs pro Well, eine endgültige Entkopplerkonzentration von Carbonylcyanid-4 (Trifluormethoxy)phenylhydrazon (FCCP) von 2 μM und eine endgültige Hoechst 33342-Konzentration von 4 μM (mit einer Inkubationszeit von mindestens 20 min nach der Injektion) bestimmt.- Um Sensorkartuschen mit Hydrobooster zu hydratisieren, nehmen Sie die Sensorkartusche mit der Utility-Platte aus der Verpackung und platzieren Sie die Kartusche "Sensor nach oben" auf dem Labortisch. Pipettieren Sie 200 μl Kalibrant (siehe Materialtabelle) in jede Vertiefung der Auffangplatte und setzen Sie den Hydrobooster fest auf die Auffangplatte, gefolgt von der Sensorpatrone. Inspizieren und entfernen Sie eingeschlossene Luftblasen und stellen Sie sie über Nacht in einen 37 °C heißen Nicht-CO 2-Inkubator.
- Schalten Sie den Flussmittelanalysator ein und öffnen Sie anschließend die Assay-Analysesoftware (siehe Materialtabelle). Stellen Sie die Temperatur auf 37 °C ein. Lassen Sie das Gerät über Nacht eingeschaltet, um eine Stabilisierung der eingestellten Temperatur zu ermöglichen.
HINWEIS: Die folgenden Programmierschritte (1.2.4-1.2.10) können am Tag vor oder am Tag des Versuchs durchgeführt werden - Um die Datei der Assay-Analysesoftware (siehe Materialtabelle) zu programmieren, öffnen Sie die Vorlage für den Substratoxidations-Stresstest. Gehen Sie zu Gruppendefinitionen, öffnen Sie die Registerkarte Injektionsstrategien , bearbeiten Sie den Inhibitor + Sub Ox Stresstest in Etomoxir + Sub Ox Stress Test und fügen Sie zwei weitere Injektionsstrategien hinzu: UK5099 + Sub Ox Stress Test und BTPES + Sub Ox Stress Test. Klicken Sie innerhalb jeder Injektionsstrategie im Fenster Injektionsbedingung auf A für Port A und geben Sie die Endkonzentration des jeweiligen Inhibitors ein (Etomoxir: 4 μM; UK5099: 2 μM; BTPES: 3 μM).
- Klicken Sie auf die Ports B, C und D und geben Sie die jeweiligen Endkonzentrationen ein (Port B: Oligomycin-Konzentration [1,50 μM], Port C: FCCP [2,0 μM]; Anschluss D: Rotenon + Antimycin A [0,5 μM]). Geben Sie Hoechst 33342, 4 μM in das Compounds-Fenster für Anschluss D.
- Öffnen Sie die Registerkarte Vorbehandlungen und wählen Sie ggf. Vorbehandlungen aus. Beispiel: Kontrolle, Experimentell oder üblich (z. B. Baseline und Follow-up). Fügen Sie bei Bedarf Beschreibungen im Fenster darunter hinzu.
- Öffnen Sie die Registerkarte Assay-Medien und wählen Sie im Pulldown-Menü Medien die Option RPMI-Medium, pH 7,4, geben Sie die Chargennummer, das Personal, das das Medium vorbereitet, den Zeitpunkt der Vorbereitung und die dem Medium hinzugefügten Zusätze ein (10 mM Glukose, 2 mM Glutamin, 1 mM Pyruvat; siehe Materialtabelle).
- Öffnen Sie die Registerkarte Zelltyp und geben Sie PBMC-Subject ID als Name, PBMC als Zellentyp, 150000 als Seeding-Dichte, Subject-ID als Quelle, Personal, das die Zellen vorbereitet hat, Datum der Vorbereitung und Tag des Auftauens der Zelle ein, falls zutreffend.
- Klicken Sie auf Gruppen generieren, gehen Sie zu Plate Map und ordnen Sie die Wells der 96-Well-Platte den jeweiligen Gruppen zu. Lassen Sie die Vertiefungen A1, A12, H1 und H12 als Hintergrund-Vertiefungen zugewiesen.
- Gehen Sie zu Protokoll, aktivieren Sie das Kontrollkästchen Äquilibrieren (Standardeinstellung) und geben Sie die folgenden Informationen ein: Ausgangswert: fünf Messzyklen: 3 min Mix, 0 min Wait und 3 min Messung; Medium oder Inhibitor (Anschluss A): sechs Messzyklen: 3 Minuten Mischen, 0 Minuten Warten und 3 Minuten Messen; Oligomycin (Port B): drei Messzyklen: 3 Minuten Mixen, 0 Minuten Warten und 3 Minuten Messen; FCCP (Port C): drei Messzyklen: 3 min Mix, 0 min Wartezeit und 3 min Messung; Rotenon + Antimycin A + Hoechst 33342 (Port D): drei Messzyklen: 3 min Mix, 0 min Wait und 3 min Messung.
- Nehmen Sie am Tag der bioenergetischen Bewertung die beschichtete Zellkultur-Mikroplatte aus dem Kühlschrank und lassen Sie sie in der Biosicherheitswerkbank auf RT erwärmen.
- Bereiten Sie das Testmedium (AM) vor, indem Sie 97 ml vorgewärmtes BM und Nahrungsergänzungsmittel mischen: 1 ml Pyruvat (Endkonzentration: 1 mM), 1 ml Glukose (Endkonzentration 10 mM) und 1 ml Glutamin (Endkonzentration: 2 mM). Stellen Sie den AM bis zur Verwendung bei 37 °C in einen Nicht-CO2 -Inkubator.
- Um die Zellen zu säen, geben Sie 50 μl Zellsuspension (aus 1.1.12) mit 150.000 PBMC in jede Vertiefung, mit Ausnahme von A1, H1, A12 und H12 (als Hintergrundvertiefungen zugewiesen).
KRITISCH: Die optimale Aussaatdichte wurde bereits ermittelt. - Decken Sie die Zellplatte mit dem Deckel ab und lassen Sie die Zellen 60 Minuten lang bei RT in der Biosicherheitswerkbank ruhen, um die gleichmäßige Verteilung der Zellen zu erleichtern.
KRITISCH: Betrachten Sie die Zellen unter einem Mikroskop, um die gewünschte homogene Zellverteilung über die Vertiefung sicherzustellen. - Schalten Sie für die Zellbildgebung den Multimodus-Reader (siehe Materialtabelle) einige Stunden vor der ersten Zellinkubation ein. Warten Sie, bis das Initiierungsprotokoll des Lesegeräts abgeschlossen ist, bevor Sie die Bildgebungssoftware öffnen (siehe Materialtabelle). Öffnen Sie die Imaging-Software und stellen Sie die Vorheiztemperatur auf 37 °C ein.
- Befolgen Sie zur Vorbereitung der Assay-Reagenzien die Anweisungen des Assay-Kits zur Rekonstitution der Reagenzien mit leichten Änderungen, wie in Tabelle 1 beschrieben.
KRITISCH: Die optimalen endgültigen FCCP- und Hoechst33342-Konzentrationen wurden bereits zuvor bestimmt. - Nach der 60-minütigen Zellruhe inspizieren Sie die Vertiefungen mit einem Zellkulturmikroskop und notieren Sie sich visuelle Anomalien, wie z. B. Zellaggregationen.
- Zentrifugieren Sie die Zellkulturplatte bei RT in einer Zentrifuge mit Ausschwingrotor für Mikrotiterplatten bei 60 × g für 1 min, mit minimaler Beschleunigung und minimaler Pause; Drehen Sie die Platte und zentrifugieren Sie erneut bei 40 × g für 1 min. Stellen Sie die Beschleunigung und Bremse auf ein Minimum ein. Inkubieren Sie die Zellen für 25-30 min bei 37 °C in einem Nicht-CO2 -Inkubator.
HINWEIS: Wenn eine Zellbildgebung durchgeführt wird, wird die Zellplatte stattdessen im vorgeheizten Multimode-Reader inkubiert (siehe Materialtabelle), und während dieser Inkubationszeit (erste Zellinkubation) wird von jeder Vertiefung ein Hellfeldbild aufgenommen. - Scannen Sie den Barcode der Zellkulturplatte mit dem Barcode-Scanner (siehe Materialtabelle), wählen Sie Hellfeld-Scan starten, legen Sie die Zellkulturplatte auf die Plattenablage und starten Sie das Zurückziehen des Tabletts. Wählen Sie Zellen aus, die Vertiefungen für die Untersuchung enthalten, und starten Sie den Scan.
- Während der ersten Zellinkubation werden die Anschlüsse der Sensorkartusche mit den Reagenzien beladen, wie in Tabelle 1 angegeben: Anschluss (A): 20 μl AM (Kontrollen), Etomoxir (Inhibitor des langkettigen Fettsäuretransports in die Mitochondrien), UK 5099 (2-Cyano-3-(1-phenyl-1H-indol-3-yl)-2-propensäure; Inhibitor des mitochondrialen Pyruvatträgers) oder BPTES (Bis-2-(5-phenylacetamido-1, 3,4-thiadiazol-2-yl)ethylsulfid; Inhibitor der Umwandlung von Glutamin in Glutamat); Port (B): 22 μl Oligomycin (Inhibitor der ATP-Synthase); Anschluss (C): 25 μl FCCP (Entkoppler Carbonylcyanid-4 (Trifluormethoxy)phenylhydrazon); Port (D): 27 μl Rotenon/Antimycin A/Hoechst 33342 (Inhibitoren des mitochondrialen Komplexes I bzw. III und Kernfarbstoff). Stellen Sie die Sensorkartusche für weitere 5 Minuten in den 37 °C heißen Nicht-CO2 -Inkubator zurück.
HINWEIS: Befolgen Sie die Anweisungen des Herstellers für das Laden der Ports. - Nehmen Sie die Zellkulturplatte nach der ersten Zellinkubation und nach Abschluss der Zellbildgebung (falls zutreffend) aus dem Inkubator oder Plattenleser und geben Sie warmes AM in jede Vertiefung bis zu einem endgültigen Volumen von 180 μl pro Vertiefung. Legen Sie die Zellplatte bei 37 °C für weitere 15-25 min in einen Nicht-CO2 -Inkubator (zweite Zellinkubation).
- Starten Sie den metabolischen Assay , indem Sie die Sensorkalibrierung während der zweiten Zellinkubation starten. Klicken Sie auf Run Assay , wenn der Assay bereit ist, mit der Kalibrierung der Sensorkartusche gestartet zu werden. Wenn Sie dazu aufgefordert werden, übertragen Sie die Sensorkartusche aus dem 37 °C Nicht-CO2 -Inkubator in das Einschubfach des Flussmittelanalysators und starten Sie die Kalibrierung. Befolgen Sie die Aufforderung der Assay-Analysesoftware und ersetzen Sie die Utility-Platte durch die Zellplatte, nachdem die Kalibrierung abgeschlossen ist.
HINWEIS: Die Kalibrierung dauert ca. 20 Minuten. - Befolgen Sie nach dem Stoffwechseltest die Eingabeaufforderung der Software und entfernen Sie die Sensorpatrone und die Zellplatte aus dem Gerät. Entfernen Sie die Sensorpatrone von der Zellplatte und decken Sie die Zellplatte mit dem Deckel ab.
- Nachdem die Inkubationszeit des Hoechst-Farbstoffs in Vorversuchen bestimmt wurde, scannen Sie den Barcode der Platte und folgen Sie den Anweisungen der Software, um mit dem Multimode-Reader ein Fluoreszenzbild jeder zellhaltigen Vertiefung aufzunehmen.
HINWEIS: Die Bildgebungs- und Assay-Analysesoftware ist miteinander verbunden und ermöglicht den Import der Zellzahldaten in die Assay-Analysedatendatei zur Normalisierung der Daten auf die Zellzahl.
2. Genexpression der zirkadianen Uhr
HINWEIS: Die Expression von Uhrengenen aus PBMCs bei den Teilnehmern wird durch Isolierung von RNA mit dem RNA-Blutkit überprüft (siehe Materialtabelle).
- Ziehen Sie 3 ml Blut des Teilnehmers direkt in ein RNA-Röhrchen (siehe Materialtabelle) mit 6 ml stabilisierenden Reagenzien. 10 s Vortex, um vollständig zu mischen.
HINWEIS: Wenn stabilisierende Reagenzien nicht gründlich mit der Blutprobe des Teilnehmers gemischt werden, besteht ein erhöhtes Risiko für Studienfehler. - Übertragen Sie 1,0 ml der stabilisierten Blutprobe in ein 15-ml-Röhrchen mit 5 ml Erythrozyten-Lysepuffer. 10-15 min auf Eis inkubieren. Wirbeln Sie das Röhrchen während der Inkubation 2x auf. Das Röhrchen bei 4 °C bei 400 × g 10 min zentrifugieren.
- Identifizieren Sie das Pellet und gießen Sie den Überstand mit den lysierten roten Blutkörperchen vorsichtig ab. 2 mL Erythrozyten-Lysepuffer zugeben (siehe Materialtabelle), die Zellen resuspendieren, kurz vortexen und bei 400 × g für 10 min bei 4 °C zentrifugieren.
- Den Überstand abgießen und die Tube umgedreht auf saugfähigem Papier 1-2 min stehen lassen. Tupfen Sie alle Flüssigkeiten am Rand der Tube mit sauberem Papier ab, bevor Sie mit dem nächsten Schritt fortfahren.
- Vorsichtig 600 μl RNA-Lysepuffer zugeben (siehe Materialtabelle). Fügen Sie gemäß dem Protokoll des Herstellers β-Mercaptoethanol hinzu und resuspendieren Sie das Pellet.
- Pipettieren Sie das Lysat direkt in eine Einweg-Zelllysat-Homogenisatorin (siehe Materialtabelle), die Spin-Säule in ein 2-ml-Sammelröhrchen gegeben und zentrifugieren Sie 2 Minuten lang bei maximaler Geschwindigkeit, um zu homogenisieren. Entsorgen Sie die Spin-Säule des Einweg-Zelllysat-Homogenisators und bewahren Sie das homogenisierte Lysat auf.
- 1 Volumen (600 μl) 70 % Ethanol zum homogenisierten Lysat geben und durch Pipettieren mischen. Pipettieren Sie die Probe einschließlich etwaiger Ausfällungen, die sich möglicherweise gebildet haben, vorsichtig in eine neue Spin-Säule in einem 2-ml-Sammelröhrchen, aber befeuchten Sie den Rand nicht. 15 s bei >8.000 × g zentrifugieren.
- Übertragen Sie die Spin-Säule (siehe Materialtabelle) in ein neues 2-ml-Entnahmeröhrchen. Geben Sie 700 μl strengen Waschpuffer (siehe Materialtabelle) auf die Schleudersäule und zentrifugieren Sie 15 s lang bei >8.000 × g zum Waschen. Verwerfen Sie den Durchfluss.
- Öffnen Sie vorsichtig die Schleudersäule und fügen Sie 500 μl milden Waschpuffer hinzu (siehe Materialtabelle). Den Deckel schließen und bei voller Geschwindigkeit (20.000 × g) 3 min zentrifugieren.
- Übertragen Sie die Spin-Säule in ein 1,5-ml-Mikrozentrifugenröhrchen und pipettieren Sie 30-50 μl RNase-freies Wasser direkt auf die Kieselsäuremembran. 1 min bei >8.000 × g zentrifugieren, um die RNA zu eluieren. Wiederholen Sie den Vorgang noch einmal.
HINWEIS: Die RNA kann bei -80 °C gelagert werden. - Führen Sie die DNase-Behandlung an einer Säule mit dem RNase-freien DNase-Set (siehe Materialtabelle) gemäß dem Protokoll des Herstellers durch.
- Erzeugen Sie cDNA mit 500 ng Gesamt-RNA und einem Echtzeit-Polymerase-Kettenreaktionssystem (PCR) (siehe Materialtabelle) gemäß dem Protokoll des Herstellers. Verdünnen Sie alle cDNA-Proben 1:25 in RNase-freiem Wasser und verwenden Sie 4 ml zur Durchführung einer quantitativen reversen Transkriptions-PCR (qRT-PCR).
- Verwenden Sie eine Primerzugabemethode (siehe Materialtabelle), um die qRT-PCR mit 10 mM jedes der in Tabelle 2 gezeigten Primer abzuschließen. Führen Sie die qRT-PCR mit einem Echtzeitsystem durch (siehe Materialtabelle).
- Normalisieren Sie die mRNA-Spiegel der ausgewählten Gene mit Hilfe der Rpl26-mRNA-Spiegel . Verwenden Sie die 2-DDCt-Methode , um die relative Quantifizierung zu berechnen.
- Um zu bestimmen, ob die Expression einer gegebenen mRNA eine zirkadiane Oszillation aufwies, verwenden Sie einen aufLR_rhythmicity 39 Wahrscheinlichkeit basierenden Test (unter Verwendung von p≤ 0,01) zum Nachweis der zirkadianen Rhythmizität in einer experimentellen Bedingung40.
3. Plan zur Datenanalyse
HINWEIS: Ein medizinisches Inventar wird verwendet, um die Teilnehmer auf der Grundlage der Einnahme von Medikamenten zu kategorisieren43.
- Verwenden Sie ein lineares Modell mit gemischten Effekten, in dem Alter, Geschlecht, Gewicht, Größe, Blutdruck, Herzfrequenz und andere Risikofaktoren als Kovariaten einbezogen werden sollten.
- Ein Random-Effect-Term sollte aufgenommen werden, um die Korrelation zwischen den Probanden zu erklären.
- Für die Modellanpassung sollte eine rückwärts gerichtete Variablenauswahl nach dem hierarchischen Prinzip implementiert werden.
- Messen Sie im angepassten Modell die Änderungen von Zeitpunkt 1 bis Zeitpunkt 2, wobei alle Kovariaten im angepassten Modell berücksichtigt werden.
Ergebnisse
Das vorgeschlagene Protokoll enthält vorläufige Daten, die als Validierung für die Methodik dienen. Das Protokoll umfasst einen Echtzeit-Metabolic-Flux-Analysator zur Untersuchung der mitochondrialen Funktion und der zellulären Brennstoffnutzung sowie RNA-Extraktion und qRT-PCR zur Analyse von Genen des zirkadianen Rhythmus (z. B. BMAL1, CLOCK, Nfil2, Nr1d1, Dbp, Cry1, Per2).
Die Sauerstoffverbrauchsrate (OCR) von isolierten humanen PBMCs von fünf Kontrollteilnehmern, 10 Tage nach einer ersten Analyse, ist in Abbildung 1 dargestellt. Die Daten werden zum Vergleich von Prä- und Post-Werten verwendet und zeigen die Durchschnittswerte für die basale Atmung, das akute Ansprechen, die maximale Atmung und die freie Kapazität nach der Injektion einer Kontrolle, Etomoxir, UK5099 und BPTES. Bemerkenswert ist, dass Abbildung 1C eine signifikante negative akute Reaktion nach der Etomoxir-Injektion zeigt, aber keine signifikanten Auswirkungen auf die basale Atmung, die maximale Atmung oder die freie Kapazität beobachtet wurden.
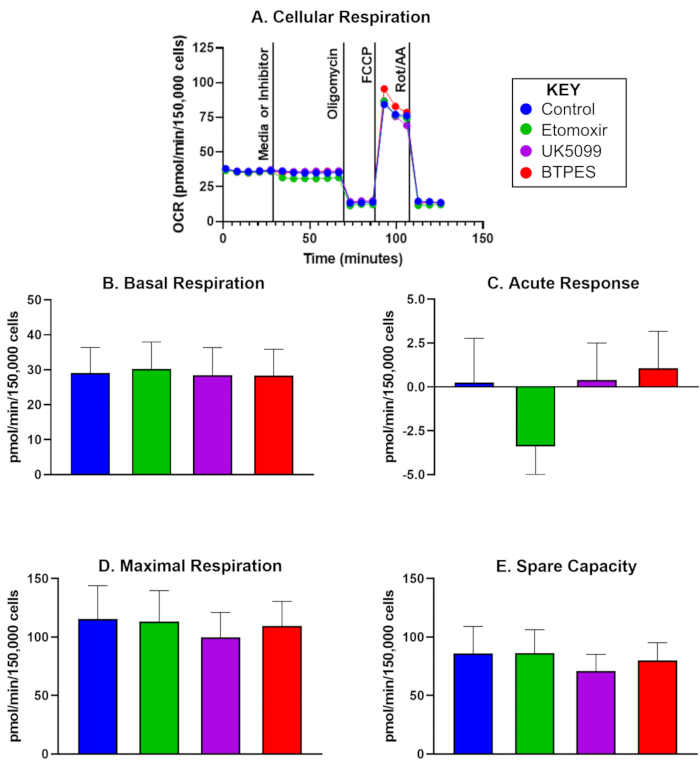
Abbildung 1: Sauerstoffverbrauch isolierter mononukleärer Zellen des peripheren Blutes (PBMCs). (A) Echtzeit-Sauerstoffverbrauchsrate (OCR; pmol/(min∙150.000 Zellen) von PBMCs, die von einer Kontrollperson isoliert wurden, gemessen mit einem Flux Analyzer und bewertet mit dem Substratoxidationsassay. Die Zellen wurden mit einer Dichte von 150.000 Zellen/Well ausgesät. Die erste Injektion erfolgte entweder als Medium (Kontrolle) oder als Inhibitor (Etomoxir, UK5099 oder BPTES; siehe Text für Details) und erfolgte nach Messung der basalen zellulären Atmungsrate. Das akute Ansprechen auf die mitochondriale Substratlimitierung wurde als Differenz der basalen OCR vor und nach der Inhibitorinjektion bestimmt. Oligomycin, der ATP-Synthase-Hemmer, hemmt die ATP-Produktions-gekoppelte Atmung und führt zu einer Protonenleckatmung. FCCP, der Entkoppler, induziert eine maximale, ungekoppelte Atmung; Rotenon und Antimycin A (Inhibitoren von Komplex I bzw. III) hemmen die gesamte Atmung außer der nicht-mitochondrialen Atmung (siehe Text für Details). (B-E) Quantifizierung der Zellatmung (n = 5; die Daten werden als Mittelwert ± SD dargestellt). (B) Basale OCR vor der Injektion des Inhibitors, (C) akutes Ansprechen auf den Inhibitor (Änderung der OCR im Verhältnis zur Basalrate vor der Injektion des Inhibitors), (D) maximale OCR und (E) freie Kapazität (Differenz zwischen maximaler OCR und basaler OCR nach der ersten Injektion). Das akute Ansprechen (C) auf die Injektion von Etomoxir könnte auf eine höhere Abhängigkeit der OCR von der Fettsäure als Energiesubstrat unter basalen Bedingungen im Vergleich zu den anderen Substratgruppen hindeuten, ohne dass ein merklicher Effekt auf die OCR bei hohem Energiebedarf (D) zu beobachten ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Verbindung | AM (μL) wird der Verbindung zugesetzt | Stamm (μM) | Rohmaterial (μL) für Arbeitsmaterial | AM (μL) für Arbeitsmaterial | Arbeitsmaterial (μM) | Arbeitsmaterial (μL) [Port] | Endkonzentration (μM) |
| Etomoxir | 700 | 160 | 500 | 1500 | 40 | 20 [A] | 4 |
| UK5099 | 700 | 80 | 500 | 1500 | 20 | 20 [A] | 2 |
| BPTES | 700 | 120 | 500 | 1500 | 30 | 20 [A] | 3 |
| Oligo | 420 | 150 | 300 | 2700 | 15 | 22 [B] | 1.5 |
| FCCP | 720 | 100 | 600 | 2400 | 20 | 25 [C] | 2 |
| Rot/AA/H | 540 | 50 | 300 | 2700 | 5 | 27 [D] | 0.5 |
Tabelle 1: Vorbereitung der Reagenzien für den Substratoxidationstest und Konzentrationen von Stoff-, Arbeits- und. Alle Reagenzien sind Teil des cell mito Stresstests oder der Substratoxidations-Stresstest-Kits. Abkürzungen: oligo = Oligomycin; FCCP = Entkoppler Carbonylcyanid-4 (Trifluormethoxy)phenylhydrazon; Rot/AA/H = Rotenon/Antimycin A/Hoechst 33342. Etomoxir, UK5099, BTPES: Inhibitoren der Fettsäure-, Glukose- bzw. Glutaminoxidation.
| Bmal1 | Vorwärts – GCACGACGTTCTTTCTTCTGT |
| Rückwärts – GCAGAAGCTTTTTCGATCTGCTTTT | |
| Uhr | Vorwärts – CGTCTCTCAGACCCTTCCTCAAC |
| Rückwärts – GTAAATGCTGCCTGGGTGGA | |
| Schrei1 | Vorwärts – ACTGCTATTGCCCTGTTGGT |
| Rückwärts – GACAGGCAAATAACGCCTGA | |
| Pro 1 | Vorwärts – ATTCGGGTTACGAAGCTCCC |
| Rückwärts – GGCAGCCCTTTCATCCACAT | |
| Per2 | Vorwärts – CATGTGCAGTGGAGCAGATTC |
| Rückwärts – GGGGTGGTAGCGGATTTCAT | |
| Rev-erb α | Vorwärts – ACAGATGTCAGCAATGTCGC |
| Rückwärts – CGACCAAACCGAACAGCATC |
Tabelle 2: Genprimer für die zirkadiane Uhr.
Diskussion
Die Abnahme der mitochondrialen Funktion und die Regulation des zirkadianen Rhythmus mit zunehmendem Alter werden zunehmend als Faktoren angesehen, die zu altersbedingten Erkrankungen beitragen. Die Veränderung des zirkadianen Rhythmus durch Änderungen des Lebensstils, wie z. B. Ernährung und körperliche Aktivität, stellt eine potenzielle Strategie dar, um ein gesundes Altern zu fördern und die mit dem Altern verbundenen Mobilitätsverluste zu reduzieren. Die derzeitigen Methoden zur direkten Beurteilung der mitochondrialen Funktion sind jedoch invasiv und erfordern häufig eine Muskelbiopsie, was aufgrund der wahrgenommenen Schmerzen und Risiken eine Herausforderung bei der Rekrutierung und Bindung der Teilnehmer darstellen kann.
Die Bewertung von Markern der zirkadianen und metabolischen Gesundheit durch weniger invasive Methoden, wie z. B. die Blutentnahme, würde wertvolle Ergebnisse für die Erforschung und Erprobung therapeutischer Ziele in zukünftigen Studien liefern. Diese minimal-invasiven Methoden haben das Potenzial, das Feld erheblich voranzubringen, indem sie neue Einblicke in das komplexe Zusammenspiel zwischen zirkadianem Rhythmus und metabolischer Gesundheit und deren Auswirkungen auf die Funktion liefern. Das Ziel dieser Studie ist es, den Zusammenhang zwischen dem zellulären Energiestoffwechsel und dem zirkadianen Rhythmus zu bewerten. Insbesondere wird die bioenergetische Flussanalyse verwendet, um die mitochondriale Funktion unter verschiedenen Substratverfügbarkeitsbedingungen zu bewerten, zusammen mit der Genexpressionsüberwachung einer Gruppe von zirkadianen Genen in den weißen Blutkörperchen der Teilnehmer. Durch den Einsatz beider Arme der Analyse, der Bioenergie und der Genexpression, kann ein umfassendes Verständnis der Beziehung zwischen diesen beiden grundlegenden Prozessen erreicht werden.
Die statistische Analyse dieser Zeitreihendaten aus zirkadianer Perspektive bietet einen Einblick in die Stärke, den Bereich und das Timing der zirkadianen Rhythmen. Zusammenfassend lässt sich sagen, dass die Integration von Genexpressionsanalyse, zellulärer Bioenergetik und metabolischen Messungen auf Organismusebene einen neuen und innovativen Ansatz darstellt, der das Zusammenspiel zwischen Energiestoffwechsel und zirkadianen Rhythmen beim Menschen beleuchten wird.
In einer Pilotstudie konnten wir eine akute Reaktion in der OCR von PBMCs auf die Einschränkung der Fettsäureverwertung (nach Injektion von Etomoxir, einem Inhibitor der Carnitinpalmitoyltransferase 1a) feststellen. Dieser Befund deutet darauf hin, dass bei PBMCs aus dieser speziellen Gruppe von Teilnehmern eine Abhängigkeit von Fettsäuren als Energiesubstrat während der basalen Atmung bestehen könnte. Die maximale Atmung wurde jedoch nicht beeinflusst, was darauf hindeutet, dass alternative Energiequellen wie Glukose und Glutamin die verminderte Verwertung von Fettsäuren bei hohem Energiebedarf kompensieren können. Zukünftige Studien sollten untersuchen, ob a) die Bioenergetik von PBMCs die Ganzkörperenergetik widerspiegelt und b) ob Interventionen wie zeitlich begrenztes Essen die Energiesubstratpräferenzen beeinflussen könnten.
Es gibt mehrere kritische Schritte bei der Flussanalyse von PBMCs. Zunächst sollte vor der Bewertung von Versuchsproben die Zellaussaatdichte (Zellen pro Vertiefung) optimiert werden, indem sichergestellt wird, dass eine kontinuierliche gleichmäßige Verteilung der Zellen in jeder Vertiefung und über jede Platte hinweg gewährleistet ist. Die endgültige FCCP-Konzentration sollte durch Durchführung von Konzentrationstestläufen mit den Konzentrationen 0, 0,125, 0,25, 0,5, 1,0 und 2,0 μM optimiert werden. Gegebenenfalls sollte die Färbung von Hoechst 33342 optimiert werden, indem die Anweisungen des Herstellers befolgt werden. Zweitens ist die Normalisierung der Stoffwechseldaten auf zelluläre Parameter entscheidend für die Vergleichbarkeit der Daten zwischen den Experimenten. In dem vorliegenden Protokoll wird die Zellzahl nach Abschluss des Flussanalysator-Assays unter Verwendung von Hoechst 33342-gefärbten Zellen und einer Zellbildgebungsvorrichtung beschrieben. Wenn ein geeignetes Gerät nicht verfügbar ist, können alternative Normalisierungsmethoden angewendet werden, wie z. B. der Gesamtgehalt an zellulärem Protein oder Kern-DNA pro Well. Es gibt eine bemerkenswerte Änderung, die innerhalb des Protokolls verwendet werden kann, verglichen mit denen, die vorgeschlagen wurden. Insbesondere kann das Protokoll mit einem individuellen Assay-Kit für jeden der drei Inhibitoren vervollständigt werden, verglichen mit nur den beiden hier vorgeschlagenen Kits (siehe Materialtabelle).
Die Verwendung von PBMCs als Surrogat zur Untersuchung des Zusammenspiels zwischen Energiestoffwechsel und zirkadianen Rhythmen bei älteren Erwachsenen wird durch die Annahme eingeschränkt, dass ihr Ansprechen auf die Behandlung das Ansprechen in anderen Geweben und Organen genau widerspiegeln kann. Obwohl dieser Ansatz neuartig und minimalinvasiv ist, ist es wichtig zu berücksichtigen, dass verschiedene Gewebe und Organe wie Gehirn, Leber und Skelettmuskulatur unter verschiedenen Bedingungen unterschiedlich reagieren können. Eine präklinische Studie zeigte, dass die Expression von Uhren-Genen bei gefütterten und nüchternen Mäusen verändert war, was zu einer teilweisen Hochregulierung von BMAL1-Zielgenen in Leber und Muskelgewebe führte, aber zur Herunterregulierung anderer Mäuse41. Diese peripheren Gewebe und Organe sind in hohem Maße repräsentativ für Stoffwechselprozesse und können durch Umwelteinflüsse beeinflusst werden, die die Expressionsmechanismen der Uhr beeinflussen42. Weitere Forschung ist erforderlich, um die Beziehung zwischen peripheren Geweben, Organen und der zentralen zirkadianen Uhr vollständig zu verstehen.
Eine weitere Einschränkung besteht darin, dass die Teilnehmer nicht für die Einnahme von Verschreibungen disqualifiziert werden, was die statistische Analyse einschränken könnte. Um dieser Einschränkung entgegenzuwirken, wird in der zukünftigen Forschung ein medizinisches Inventar verwendet, das an Populationen älterer Erwachsener, die Medikamente einnehmen, validiert wurde43. Die Teilnehmer werden auf der Grundlage der empfohlenen Medikamente kategorisiert, die im Abschnitt zur Datenüberprüfung des Protokolls aufgezeichnet sind. Es gibt insgesamt drei Kategorien in Bezug auf Medikamente, von denen gezeigt wurde, dass sie 1) den funktionellen Verfall beschleunigen, 2) den funktionellen Verfall verlangsamen und 3) die Funktion der Skelettmuskulatur beeinflussen.
Schließlich weist die mitochondriale oxidative Kapazität der menschlichen Skelettmuskulatur einen Tag-Nacht-Rhythmus auf, der zwischen 18:00 und 23:00 Uhr ihren Höhepunkt erreicht und zwischen 08:00 und 11:00 Uhr abnimmt44. Es ist noch nicht klar, ob dies auch für die mitochondriale oxidative Fähigkeit von PBMCs gilt. Vorläufige Daten deuten jedoch darauf hin, dass PBMCs und der mitochondriale Stoffwechsel zusammenhängen45. Da die Informationen über Muskelbiopsien und die Veränderungen der PBMCs nicht so eindeutig sind, ist bei der Analyse der Ergebnisse Vorsicht geboten. Angesichts dieser Einschränkung ist es wichtig, diese Informationen bei der Bewertung und Entwicklung eines Protokolls im Hinterkopf zu behalten, da sie wertvollen Kontext und Einblicke liefern können, die dazu beitragen können, die Gültigkeit und Wirksamkeit des Protokolls sicherzustellen.
Nach unserem besten Wissen haben keine früheren Studien die Muster der Brennstoffnutzung oder des zirkadianen Rhythmus mit den in diesem Projekt vorgeschlagenen Methoden bewertet. Unser Ziel ist es, die Reaktionsfähigkeit von Markern der mitochondrialen Brennstoffverwertung und der zirkadianen Gesundheit auf Veränderungen zu untersuchen. In dieser Studie wird eine minimal-invasive Methode zur Messung eines hochsensitiven Biomarkers vorgestellt, die als Alternative in zukünftigen interventionellen Studien dienen kann, bei denen eine Muskelbiopsie nicht durchführbar ist.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Studie wurde vom Older American's Independence Center (NIH/NIA P30AG028740) mit Unterstützung des Clinical and Translational Science Institute (NIH/NCRR UL1TR000064) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Agilent Technologies Cell Imaging | Agilent Technologies | Cell image software | |
| Agilent/Seahorse Wave desktop program | Agilent Technologies | 5994-0039EN | Software used to analyse data from the celluar analyser and stress test assay |
| Agilent/Seahorse XFe96 Flux Analyzer | Agilent Technologies | S7800B | Real-time cellular flux analyzer; flux analyser |
| Bar Code Reader | Agilent Technologies | G2615-90007 | |
| Seahorse Wave Desktop Software | Agilent technologies | Data acquisition software; assay analysis; wave program | |
| Seahorse XF 1.0 M Glucose solution | Agilent Technologies | 103577-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 100 mM Pyruvate solution | Agilent Technologies | 103578-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 200 mM Glutamine solution | Agilent Technologies | 103579-100 | Supplement to basal medium to make assay medium |
| Seahorse XF Cell Mito Stress Test | Agilent Technologies | 103015-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF Mito Fuel Flex Test | Agilent Technologies | 103260-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF RPMI Medium | Agilent Technologies | 103576-100 | Basal medium for PBMCs |
| Seahorse XFe96 FluxPak mini | Agilent Technologies | 102601-100 | Sensor cartridges and cell culture microplates |
| Cytation 1 Cell Imaging Multi-Mode Reader | Agilent/BioTek | Multimode reader to image cells | |
| CPT Sodium Heparin Tube, 16 x 125 mm x 8.0 mL | Becton Dickinson | 362753 | Blood collection tubes for isolation of peripheral blood mononuclear cells |
| CellTak Cell and Tissue Adhesive | Corning | 354240 | Cell adherent to coat cell culture microplate |
| Phosphate Buffered Saline | Corning | 21-040-CV | Buffer to wash blood cells |
| Ficoll Paque Plus | Cytiva | GE17-1440-02 | Gradient medium |
| Lunar Prodigy DXA scanner | General Electric | EN 60601-2-7 5.1 | Whole body lean mass and fat/lean tissue mass ratio |
| Freezing container, Nalgene Mr. Frosty | MilliporeSigma | C1562 | Freezing container used to slow-freeze cell suspension |
| Buffer EL. | Qiagen | 79217 | Erythrocyte lysis buffer |
| Buffer RLT | Qiagen | 79216 | RNA lysis buffer |
| Buffer RPE | Qiagen | 1018013 | Mild washing buffer |
| Buffer RW1 | Qiagen | 1053394 | Stringent washing buffer |
| QIAamp DNA Micro Kit | Qiagen | 56304 | DNA preps: QIAamp MinElute Columns, Proteinase K, Carrier RNA, Buffers, Collection Tubes (2 ml) |
| QIAamp RNA Blood Mini Kit | Qiagen | 52304 | RNA blood kit; Used to isolate RNA |
| QIAshredder | Qiagen | 79656 | disposable cell-lysate homogenizers |
| RNase-Free DNase Set | Qiagen | 79254 | Used to perform DNA digest |
| 2-Mercaptoethanol (Reagent) | Thermo Fisher Scientific | MFCD00004890 | |
| 2-mL collection tubes, 100 count | Thermo Fisher Scientific | AM12480 | |
| Fast SYBR Green Master Mix | Thermo Fisher Scientific | 4385612 | Primers are added to this and used to carry out qRT-PCR |
| Microcentrifuge Tubes, 1.5 mL | Thermo Fisher Scientific | 69715 | Used to hold RNA purification filter during RNA purification |
| Narrow p1000 pipette tips | Thermo Fisher Scientific | 02-707-402 | |
| QuantStudio 3 Real-Time PCR System, MiniAmp Plus Thermal Cycler, and 96-Well Plates Package | Thermo Fisher Scientific | A40393 | |
| Tempus Blood RNA Tube | Thermo Fisher Scientific | 4342792 | RNA Tube |
| Tempus Spin RNA Isolation kit | Thermo Fisher Scientific | 4380204 | RNA extraction and isolation |
Referenzen
- UN Department of Economic and Social Affairs. Population Division 2019, World Population Prospects. UN Department of Economic and Social Affairs. , (2019).
- Anton, S., Leeuwenburgh, C. Fasting or caloric restriction for healthy aging. Experimental Gerontology. 48 (10), 1003-1005 (2013).
- Dziechciaż, M., Filip, R. Biological psychological and social determinants of old age: Bio-psycho-social aspects of human aging. Annals of Agricultural and Environmental Medicine. 21 (4), 835-838 (2014).
- Anton, S. D., et al. Flipping the metabolic switch: understanding and applying the health benefits of fasting. Obesity. 26 (2), 254-268 (2018).
- Fried, L. P., Guralnik, J. M. Disability in older adults: evidence regarding significance, etiology, and risk. Journal of the American Geriatrics Society. 45 (1), 92-100 (1997).
- Manini, T. Development of physical disability in older adults. Current Aging Science. 4 (3), 184-191 (2011).
- Chung, H. Y., et al. Molecular inflammation: underpinnings of aging and age-related diseases. Ageing Research Reviews. 8 (1), 18-30 (2009).
- Sun, N., Youle, R. J., Finkel, T. The mitochondrial basis of aging. Molecular Cell. 61 (5), 654-666 (2016).
- Tarasov, A. I., Griffiths, E. J., Rutter, G. A. Regulation of ATP production by mitochondrial Ca2. Cell Calcium. 52 (1), 28-35 (2012).
- Chistiakov, D. A., Sobenin, I. A., Revin, V. V., Orekhov, A. N., Bobryshev, Y. V. Mitochondrial aging and age-related dysfunction of mitochondria. Biomed Research International. 2014, 238463 (2014).
- Boengler, K., Kosiol, M., Mayr, M., Schulz, R., Rohrbach, S. Mitochondria and ageing: role in heart, skeletal muscle and adipose tissue. Journal of Cachexia, Sarcopenia, and Muscle. 8 (3), 349-369 (2017).
- Drew, B., et al. Effects of aging and caloric restriction on mitochondrial energy production in gastrocnemius muscle and heart. American Journal of Physiology Regulatory, Integrative and Comparative Physiology. 284 (2), R474-R480 (2003).
- Short, K. R., et al. Decline in skeletal muscle mitochondrial function with aging in humans. Proceedings of the National Academy of Sciences. 102 (15), 5618-5623 (2005).
- Musci, R. V., Hamilton, K. L., Miller, B. F. Targeting mitochondrial function and proteostasis to mitigate dynapenia. European Journal of Applied Physiology. 118 (1), 1-9 (2018).
- Picca, A., et al. Targeting mitochondrial quality control for treating sarcopenia: lessons from physical exercise. Expert Opinion on Therapeutic Targets. 23 (2), 153-160 (2019).
- Fernandez-Marcos, P. J., Auwerx, J. Regulation of PGC-1α, a nodal regulator of mitochondrial biogenesis. The American Journal of Clinical Nutrition. 93 (4), 884-890 (2011).
- Kim, Y., Triolo, M., Hood, D. A. Impact of aging and exercise on mitochondrial quality control in skeletal muscle. Oxidative Medicine and Cellular Longevity. 2017, 3165396 (2017).
- Wang, H., Hiatt, W. R., Barstow, T. J., Brass, E. P. Relationships between muscle mitochondrial DNA content, mitochondrial enzyme activity and oxidative capacity in man: alterations with disease. European Journal of Applied Physiology and Occupational Physiology. 80 (1), 22-27 (1999).
- Tian, Q., et al. Muscle mitochondrial energetics predicts mobility decline in well-functioning older adults: The baltimore longitudinal study of aging. Aging Cell. 21 (2), e13552 (2022).
- Sardon Puig, L., Valera-Alberni, M., Cantó, C., Pillon, N. J. Circadian rhythms and mitochondria: connecting the dots. Frontiers in Genetics. 9, 452 (2018).
- Gano, L. B., Patel, M., Rho, J. M. Ketogenic diets, mitochondria, and neurological diseases. Journal of Lipid Research. 55 (11), 2211-2228 (2014).
- Liesa, M., Shirihai, O. S. Mitochondrial dynamics in the regulation of nutrient utilization and energy expenditure. Cell Metabolism. 17 (4), 491-506 (2013).
- Lesnefsky, E. J., Chen, Q., Hoppel, C. L. Mitochondrial metabolism in aging heart. Circulation Research. 118 (10), 1593-1611 (2016).
- Hartman, M. L., et al. Relation of mitochondrial oxygen consumption in peripheral blood mononuclear cells to vascular function in type 2 diabetes mellitus. Vascular Medicine. 19 (1), 67-74 (2014).
- Mahapatra, G., et al. Blood-based bioenergetic profiling is related to differences in brain morphology in African Americans with Type 2 diabetes. Clinical Science. 132 (23), 2509-2518 (2018).
- Moore-Ede, M. C. Physiology of the circadian timing system: predictive versus reactive homeostasis. The American Journal of Physiology. 250 (5), R737-R752 (1986).
- Young, M. W. Life's 24-hour clock: molecular control of circadian rhythms in animal cells. Trends in Biochemical Sciences. 25 (12), 601-606 (2000).
- Yoo, S. H., et al. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proceedings of the National Academy of Sciences. 101 (15), 5339-5346 (2004).
- Zhang, R., Lahens, N. F., Ballance, H. I., Hughes, M. E., Hogenesch, J. B. A circadian gene expression atlas in mammals: implications for biology and medicine. Proceedings of the National Academy of Sciences. 111 (45), 16219-16224 (2014).
- de Goede, P., Wefers, J., Brombacher, E. C., Schrauwen, P., Kalsbeek, A. Circadian rhythms in mitochondrial respiration. Journal of Molecular Endocrinology. 60 (3), R115-R130 (2018).
- Hood, S., Amir, S. The aging clock: circadian rhythms and later life. The Journal of Clinical Investigation. 127 (2), 437-446 (2017).
- Sellix, M. T., et al. Aging differentially affects the re-entrainment response of central and peripheral circadian oscillators. The Journal of Neuroscience. 32 (46), 16193-16202 (2012).
- Sato, S., et al. Circadian reprogramming in the liver identifies metabolic pathways of aging. Cell. 170 (4), 664-677 (2017).
- Lundell, L. S., et al. et al. feeding alters lipid and amino acid metabolite rhythmicity without perturbing clock gene expression. Nature Communications. 11 (1), 4643 (2020).
- Perrin, L., et al. Transcriptomic analyses reveal rhythmic and CLOCK-driven pathways in human skeletal muscle. eLife. 7, e34114 (2018).
- Gutierrez-Monreal, M. A., Harmsen, J. -. F., Schrauwen, P., Esser, K. A. Ticking for metabolic health: the skeletal-muscle clocks. Obesity. 28, S46-S54 (2020).
- Wolff, C. A., et al. Defining the age-dependent and tissue-specific circadian transcriptome in male mice. bioRxiv. 42 (1), 111982 (2023).
- Wilson, D., Breen, L., Lord, J. M., Sapey, E. The challenges of muscle biopsy in a community based geriatric population. BMC Research Notes. 11 (1), 830 (2018).
- Ding, H., et al. Likelihood-based tests for detecting circadian rhythmicity and differential circadian patterns in transcriptomic applications. Briefings in Bioinformatics. 22 (6), 224 (2021).
- Ding, Z., Lamb, T. M., Boukhris, A., Porter, R., Bell-Pedersen, D. Circadian clock control of translation initiation factor eIF2α activity requires eIF2γ-dependent recruitment of rhythmic PPP-1 phosphatase in Neurospora crassa. mBio. 12 (3), e00871 (2021).
- Di Francesco, A., Di Germanio, C., Bernier, M., de Cabo, R. A time to fast. Science. 362 (6416), 770-775 (2018).
- Kalfalah, F., et al. Crosstalk of clock gene expression and autophagy in aging. Aging. 8 (9), 1876-1895 (2016).
- Psaty, B. M., et al. Assessing the use of medications in the elderly: methods and initial experience in the cardiovascular health study. Journal of Clinical Epidemiology. 45 (6), 683-692 (1992).
- van Moorsel, D., et al. Demonstration of a day-night rhythm in human skeletal muscle oxidative capacity. Molecular Metabolism. 5 (8), 635-645 (2016).
- Janssen, J. J. E., et al. Extracellular flux analyses reveal differences in mitochondrial PBMC metabolism between high-fit and low-fit females. American Journal of Physiology. Endocrinology and Metabolism. 322 (2), E141-E153 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten