Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Anwendung von AlDeSense zur Stratifizierung von Eierstockkrebszellen basierend auf der Aktivität der Aldehyddehydrogenase 1A1
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Methoden zur Messung der Aktivität von ALDH1A1 in lebenden Zellen sind in der Krebsforschung von entscheidender Bedeutung, da es ein Biomarker für die Stammzellbildung ist. In dieser Studie verwendeten wir eine isoformselektive fluorogene Sonde, um die relativen Konzentrationen der ALDH1A1-Aktivität in einem Panel von fünf Eierstockkrebszelllinien zu bestimmen.
Zusammenfassung
Ein Rückfall nach einer Krebsbehandlung wird oft auf die Persistenz einer Subpopulation von Tumorzellen zurückgeführt, die als Krebsstammzellen (CSCs) bekannt sind und sich durch ihre bemerkenswerte Fähigkeit zur Tumorinitiierung und Selbsterneuerung auszeichnen. Abhängig von der Herkunft des Tumors (z. B. Eierstöcke) kann das Profil des CSC-Oberflächen-Biomarkers stark variieren, was die Identifizierung solcher Zellen durch immunhistochemische Färbung zu einem schwierigen Unterfangen macht. Im Gegensatz dazu hat sich die Aldehyddehydrogenase 1A1 (ALDH1A1) aufgrund ihres konservierten Expressionsprofils in fast allen Vorläuferzellen, einschließlich CSCs, als hervorragender Marker zur Identifizierung von CSCs erwiesen. Die Isoform ALDH1A1 gehört zu einer Superfamilie von 19 Enzymen, die für die Oxidation verschiedener endogener und xenobiotischer Aldehyde zu den entsprechenden Carbonsäureprodukten verantwortlich sind. Chan et al. haben kürzlich AlDeSense entwickelt, eine isoformselektive "Einschaltsonde" zum Nachweis der ALDH1A1-Aktivität, sowie ein nicht-reaktives Matching-Kontrollreagenz (Ctrl-AlDeSense), um Off-Target-Färbungen zu berücksichtigen. Dieses isoformselektive Werkzeug hat sich bereits durch den Nachweis der ALDH1A1-Aktivität in myeloischen K562-Leukämiezellen, Mammosphären und aus Melanomen gewonnenen CSC-Xenotransplantaten als vielseitiges chemisches Werkzeug erwiesen. In diesem Artikel wurde der Nutzen der Sonde durch zusätzliche Fluorimetrie-, konfokale Mikroskopie- und Durchflusszytometrie-Experimente demonstriert, bei denen die relative ALDH1A1-Aktivität in einem Panel von fünf Eierstockkrebszelllinien bestimmt wurde.
Einleitung
Krebsstammzellen (CSCs) sind eine Subpopulation von Tumorzellen, die stammzellähnliche Eigenschaften aufweisen1. Ähnlich wie ihre nicht-krebsartigen Gegenstücke besitzen CSCs die außergewöhnliche Fähigkeit, sich selbst zu erneuern und zu vermehren. Zusammen mit anderen eingebauten Mechanismen, wie z.B. der Hochregulierung von ATP-bindenden Kassettentransportern, bleiben CSCs oft von anfänglichen chirurgischen Debulking-Anstrengungen sowie einer anschließenden adjuvanten Therapie verschont2. Aufgrund ihrer entscheidenden Rolle bei der Behandlungvon Resistenz 3, Rezidiv4 und Metastasierung5 sind CSCs zu einer Priorität in der Krebsforschung geworden. Obwohl es eine Vielzahl von Zelloberflächenantigenen (z. B. CD133) gibt, die zur Identifizierung von CSCs verwendet werden können6, hat sich die Nutzung der enzymatischen Aktivität von Aldehyddehydrogenasen (ALDHs) im Zytoplasmaals attraktive Alternative 7 herausgestellt. ALDHs sind eine Superfamilie von 19 Enzymen, die für die Katalyse der Oxidation von reaktiven endogenen und xenobiotischen Aldehyden zu den entsprechenden Carbonsäureprodukten verantwortlich sind8.
Im Allgemeinen ist die Entgiftung von Aldehyden von entscheidender Bedeutung, um die Zellen vor unerwünschten Vernetzungsereignissen und oxidativem Stress zu schützen, die die Integrität der Stammzellen schädigen können9. Darüber hinaus steuert die 1A1-Isoform den Retinsäurestoffwechsel, der wiederum die Stammzellbildung über die Retinaldehyd-Signalübertragungbeeinflusst 10. AlDeSense 11,12, eine aktivitätsbasierte Sensorsonde (ABS) mit kleinen Molekülen zum selektiven Nachweis der Aktivität von ALDH1A1, wurde kürzlich entwickelt. ABS-Designs erreichen die Detektion von Analyten durch eine chemische Veränderung und nicht durch ein Bindungsereignis, was eine hohe Selektivität und verringerte Off-Target-Reaktionen ermöglicht13,14,15,16. Das Konstruktionsprinzip der isoformselektiven fluorogenen Sonde beruht auf einem Donor-Photo-induzierten Elektronentransfer (d-PeT)-Quenching-Mechanismus17, der von der funktionellen Gruppe des Aldehyds ausgeht und dazu dient, die fluoreszierende Signatur der Sonde18 zu unterdrücken. Bei der Umwandlung von ALDH1A1 in die Carbonsäure wird die Strahlungsrelaxation freigesetzt, um ein stark fluoreszierendes Produkt zu erhalten. Da die d-PeT-Abschreckung nie zu 100 % effizient ist, wurde die Restfluoreszenz, die zu möglichen falsch positiven Ergebnissen führen kann, bei der Etablierung dieses Assays durch die Entwicklung von Ctrl-AlDeSense berücksichtigt, einem nicht ansprechenden Reagenz mit übereinstimmenden photophysikalischen Eigenschaften (z. B. Quantenausbeute) und einem identischen zytoplasmatischen Färbemuster in Zellen. Bei gleichzeitiger Verwendung kann diese einzigartige Paarung mithilfe von Fluorimetrie, molekularer Bildgebung und Durchflusszytometrie zuverlässig Zellen mit hoher ALDH1A1-Aktivität von solchen unterscheiden, die niedrige Konzentrationen aufweisen. Die Verwendung von isoformselektiv aktivierbaren Farbstoffen gegenüber herkömmlichen immunhistochemischen Methoden bringt mehrere entscheidende Vorteile mit sich. Zum Beispiel wird angenommen, dass CSCs tief in einem Tumor vergraben sind und daher für ein kleines Molekül im Vergleich zu großen Antikörpern leichter zugänglich sind19. Darüber hinaus modifiziert das umgedrehte fluoreszierende Produkt keine zellulären Komponenten kovalent, was bedeutet, dass es leicht über Waschzyklen entfernt werden kann, um ein CSC in einem unveränderten Zustand zu belassen. Schließlich identifiziert die Einschaltreaktion nur lebensfähige Zellen und Funktionen, ähnlich wie beim MTT-Assay, da sie auf den NAD+-Kofaktor angewiesen ist.
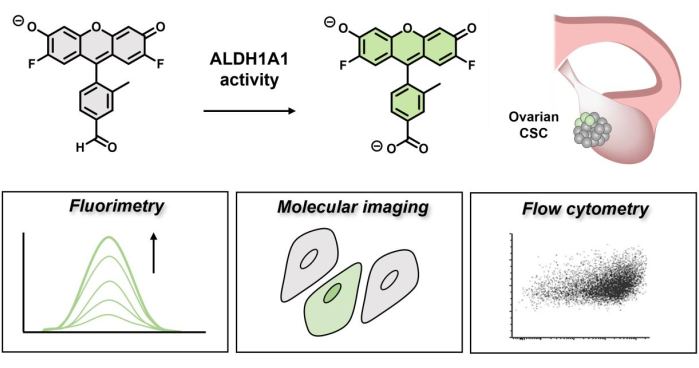
Abbildung 1: Schematische Darstellung des Fluoreszenz-Einschaltens von AlDeSense. Der isoformselektive Farbstoff wird durch ALDH1A1 aktiviert und kann verwendet werden, um eine erhöhte ALDH1A1-Aktivität in Eierstockkrebszellen mittels Fluorimetrie, molekularer Bildgebung und Durchflusszytometrie zu identifizieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
In früheren Arbeiten wurde mit dem isoformselektiven fluorogenen Sondenassay erfolgreich ALDH high (ALDH+) Zellen von ALDH low (ALDH-) Zellen in humanen K562 chronischen Leukämiezellen, MDA-MB-231 humanen Brustkrebszellen und B16F0 murinen Melanomzellen stratifiziert. Dies ist wichtig, da für viele Krebsarten eine hohe ALDH1A1-Proteinexpression eine schlechtere klinische Prognose bedeutet20. Dies setzt voraus, dass erhöhte ALDH1A1-Spiegel auf CSCs hinweisen, die sich der Behandlung entziehen, Resistenzen entwickeln und sich im ganzen Körper ausbreiten können. Im Fall von Eierstockkrebs gibt es jedoch Studien, die das Gegenteil belegen (eine hohe ALDH1A1-Expression ist mit einem verbesserten Überleben der Patientin verbunden)21,22,23,24. Auch wenn dies auf den ersten Blick widersprüchlich erscheinen mag, korreliert die Expression nicht unbedingt mit der Enzymaktivität, die durch Veränderungen der Tumormikroumgebung (z. B. pH-Fluss, Sauerstoffgradienten), die Verfügbarkeit des NAD+-Cofaktors oder der Aldehydsubstrate, den Gehalt an Carbonsäuren (Produkthemmung) und posttranslationale Modifikationen, die die Enzymaktivität verändern können, beeinflusst werden kann25 . Darüber hinaus wird das Ovarialkarzinom in fünf histologische Haupttypen unterteilt (hochgradig serös, niedriggradig serös, endometrioid, klarzellig und muzinös), von denen wir annehmen, dass sie unterschiedliche Konzentrationen der ALDH1A1-Aktivität aufweisen26. Mit dem Ziel, die Aktivität von ALDH1A1 in Ovarialtumoren zu untersuchen, wurde ein isoformselektiver fluorogener Sondenassay verwendet, um ALDH1A1+-Populationen in einem Panel von fünf Ovarialkarzinomzelllinien zu identifizieren, die zu den verschiedenen oben genannten histologischen Typen gehören. Zu den in dieser Studie getesteten Zelllinien gehören BG-1-, Caov-3-, IGROV-1-, OVCAR-3- und PEO4-Zellen, die klare Zell- und seröse Histotypen abdecken. Hier wurde die Vielseitigkeit und Generalisierbarkeit der Sonde hervorgehoben, um CSCs für die Forscher zu identifizieren, die ähnliche Studien in anderen immortalisierten Krebszelllinien sowie in Patientenproben durchführen möchten. Der Einsatz von AlDeSense wird Aufschluss über die biochemischen Signalwege geben, die an der Aufrechterhaltung von CSC in komplexen Gewebemikroumgebungen beteiligt sind, und möglicherweise als klinisches Werkzeug zur Bestimmung der Prognose und Messung der Krebsaggressivität dienen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Messung der Gesamtaktivität von ALDH1A1 in Homogenaten von Eierstockkrebszellen mittels Fluorimetrie
- Tauen Sie 1 × 106 Zellen in einem T25-Zellkulturkolben in 5 ml der folgenden Zellkulturmedien auf:
- IGROV-1 und PEO4: Roswell Park Memorial Institute (RPMI) 1640 medium mit 10 % fötalem Kälberserum (FBS) und 1 % Penicillin/Streptomycin (P/S).
- BG-1 und Caov-3: Dulbeccos Modified Eagle's Medium (DMEM) mit 10% FBS und 1% P/S.
- OVCAR-3: RPMI 1640 mit 20 % FBS, 1 % P/S und 0,01 mg/ml Insulin.
- Halten Sie die Zellen in einem Inkubator bei 37 °C und 5 % CO2 für zwei bis drei Durchgänge. Stellen Sie sicher, dass die Zellen vor dem Passieren eine Konfluenz von 80 % bis 90 % nicht überschreiten.
- Die Zellen werden 10 min lang in 0,25%igem Trypsin trypsinisiert, mit einem automatisierten Zellzähler gezählt und 1 × 107 Zellen durch Zentrifugation (180 × g) bei 25 °C für 5 min pelletiert.
- Entfernen Sie den Überstand vorsichtig durch Aspiration, waschen Sie das Pellet, indem Sie die Zellen in 1 ml 1x PBS resuspendieren, und pelletieren Sie die Zellen durch Zentrifugation unter den gleichen Bedingungen wie in Schritt 4 beschrieben.
- Resuspendieren Sie die Zellen in einer Lösung von 1x Proteaseinhibitor in 1x PBS. Verwenden Sie 1 ml dieser Lösung pro 2,5 × 10 bis6 Zellen.
- Beschallen Sie die Zellsuspension 2 Minuten lang auf Eis (1 s Impuls, 40 % Amplitude) mit einer Zellhomogenisatorsonde.
- Pellet-unlösliche/Membranfraktionen durch Zentrifugation (3.200 × g) bei 25 °C für 15 min. Entfernen und bewahren Sie den Überstand auf, da dies das Homogenat ist, das in den folgenden Schritten verwendet wird. Trennen Sie die Homogenate in drei Küvetten, um das Experiment in dreifacher Ausführung durchzuführen.
- Geben Sie die Sonde in das Homogenat, um eine endgültige Sondenkonzentration von 4 μM zu erhalten. Pipettieren Sie dreimal auf und ab, um die Lösung gut zu mischen.
- Messen Sie das Fluoreszenzsignal unmittelbar nach der Zugabe und nach den gewünschten Inkubationszeiten auf einem Fluorimeter. Die Inkubationszeit kann optimiert werden, um die maximale Falzeinschaltung zu sehen (1 h in diesem Experiment). Die Inkubationszeit muss über alle verglichenen Zelllinien hinweg konstant sein.
- Stellen Sie die Anregungswellenlänge auf 496 nm ein.
- Stellen Sie die Emission auf 510-600 nm ein.
- Stellen Sie die Schlitzbreite auf 0,5 mm ein.
- Pipettieren Sie die Lösung in eine 1-ml-Quarzküvette, legen Sie sie in das Fluorimeter und klicken Sie auf Scannen.
- Teilen Sie die endgültige Fluoreszenzintensität bei 516 nm (maximale Fluoreszenzwellenlänge) durch die Intensität bei der gleichen Wellenlänge aus dem ursprünglichen Messwert, um die Faltungsaktivierung des isoformselektiven Farbstoffs zu bestimmen.
2. Einsatz der Fluoreszenzmikroskopie zur Abbildung von Zellen mit hoher ALDH1A1-Aktivität
- Tauen Sie 1 × 106 Zellen in einem T25-Zellkulturkolben in 5 ml des entsprechenden Zellkulturmediums auf.
- Halten Sie die Zellen in einem Inkubator bei 37 °C und 5 % CO2 für zwei bis drei Durchgänge. Stellen Sie sicher, dass die Zellen vor dem Passieren eine Konfluenz von 80 % bis 90 % nicht überschreiten.
- Am Tag vor der konfokalen Bildgebung Platte 4 × 105 Zellen in einem 8-Well-Kammerobjektträger.
- Beschichten Sie den Boden jeder Vertiefung 10 Minuten lang mit Poly-L-Lysin (0,1 mg/ml, 100 μl pro Well) und saugen Sie anschließend auf.
- Waschen Sie jede Vertiefung 3x, indem Sie Wasser in Zellkulturqualität (100 μl pro Vertiefung) hinzufügen und absaugen.
- Trypsinisieren Sie die Zellen 10 Minuten lang in 0,25%igem Trypsin, zählen Sie sie mit einem automatisierten Zellzähler und legen Sie die Zellen auf 4 × 105 Zellen pro Well.
- Lassen Sie die Zellen über Nacht (12-16 h) absetzen und anheften.
- Saugen Sie das Nährmedium ab und fügen Sie serumfreie Medien (500 μl pro Well) hinzu, ergänzt mit 2 μM der Sonde oder der Kontrollsonde.
- Mit der Sonde bei Raumtemperatur 30 min inkubieren und die Zellen sofort abbilden.
- Schalten Sie das konfokale Mikroskop ein und stellen Sie es auf die Probe ein.
- Legen Sie die Probe vorsichtig mit der niedrigsten Vergrößerung ein. Suchen Sie die Zellen, um die richtige Positionierung sicherzustellen.
- Stellen Sie sicher, dass die Objektivlinse eine 10-fache Vergrößerung hat, und lokalisieren Sie die Zellen.
- Für dieses Experiment werden der FITC-Kanal (Anregung: 488 nm Laser; Emission: 516-521 nm) und der T-PMT-Kanal (Durchlicht) benötigt. Lokalisieren und fokussieren Sie die Zellen mit T-PMT, um sich während des gesamten Experiments auf dieselbe z-Ebene zu konzentrieren, um Verzerrungen zu beseitigen.
- Stellen Sie die Laserleistung und die FITC-Verstärkung auf die entsprechende Einstellung ein, bei der das Signal der Ctrl-AlDeSense AM-Samples minimal wahrnehmbar ist, während das Signal in den AlDeSense AM-Samples weiterhin sichtbar ist. Jeder Parameter kann durch Verschieben des entsprechenden Balkens angepasst werden. Die Einstellungen müssen möglicherweise einige Male angepasst werden, um die richtigen Parameter zu identifizieren. Nach der Optimierung schließen Sie den Rest des Experiments innerhalb dieser Zelllinie mit identischen Parametern ab.
- Nehmen Sie drei Bilder pro Vertiefung auf, um insgesamt drei Vertiefungen pro Behandlungsbedingung zu erhalten (insgesamt neun Bilder). Konzentrieren Sie sich mit T-PMT auf die richtige Ebene, um Verzerrungen zu vermeiden, anstatt den Fluoreszenzkanal oder das zusammengeführte Bild zu verwenden.
- Verarbeiten Sie die Bilder, um den prozentualen Anteil der ALDH1A1+-Zellen zu bestimmen.
- Teilen Sie die CCI-Datei mithilfe einer Bildbearbeitungssoftware in verschiedene Kanäle auf.
- Zählen Sie die Gesamtzahl der Zellen und die Gesamtzahl der fluoreszierenden Zellen.
- Um den prozentualen Anteil der ALDH1A1+-Zellen zu bestimmen, teilen Sie die Anzahl der fluoreszierenden Zellen durch die Gesamtzahl der Zellen in jedem Bild. Es ist zwingend erforderlich, für jedes Bild auf die gleiche Weise zu zählen, ohne die Bilder zu manipulieren, da z. B. das Anpassen der Helligkeit eine Störvariable hinzufügen kann.
3. Anwendung der Durchflusszytometrie zur Identifizierung von Zellen mit hoher ALDH1A1-Aktivität
- Tauen Sie 1 × 106 Zellen in einem T25-Zellkulturkolben in 5 ml des entsprechenden Zellkulturmediums auf.
- Halten Sie die Zellen in einem Inkubator bei 37 °C und 5 % CO2 für zwei bis drei Durchgänge. Stellen Sie sicher, dass die Zellen vor dem Passieren eine Konfluenz von 80 % bis 90 % nicht überschreiten.
- Trypsinisieren, zählen und pelletieren Sie die Zellen in einem 15-ml-Zentrifugenröhrchen durch Zentrifugation (180 × g) bei 25 °C für 5 Minuten.
- Resuspendieren Sie die Zellen in 1 ml 2 μM Sonden-/Kontrollsondenlösung in PBS. Schaukeln Sie die Zellen 60 Minuten lang bei Raumtemperatur, um sicherzustellen, dass der Farbstoff gleichmäßig ausgesetzt ist.
- Nach der Inkubationszeit pelletieren Sie die Zellen durch Zentrifugation (180 × g) bei 25 °C für 5 min. Resuspendieren Sie die Zellen in 0,5 ml PBS. Führen Sie die Zellen durch ein Zellsieb (35 μm Nylonnetz), um Zellklumpen zu entfernen, die das Durchflusszytometer verstopfen könnten. Legen Sie die Zellen sofort auf Eis.
- Schalten Sie das Gerät ein und führen Sie das Startprotokoll aus.
- Prüfen Sie, ob die Mantelflüssigkeit vorhanden ist und der Abfall entleert ist.
- Lassen Sie die Leitungen mit 10% Bleichmittel und Wasser für jeweils 5 Minuten laufen.
- Führen Sie Qualitätskontrollperlen durch, um eine ordnungsgemäße Funktion sicherzustellen.
- Wählen Sie auf der Registerkarte "Einstellungen" FSC ( Vorwärtsstreuung), SSC (Seitenstreuung) und FITC (Fluorescein-Isothiocyanat) für den Fluoreszenzfilter aus.
- Zeichnen Sie die folgenden Diagramme, um die Viabilität, die Singuletts und die Fluoreszenz zu ermitteln. Optimieren Sie die Laserleistung (spezifisch für das Gerät des Benutzers), so dass die Zellpopulationen innerhalb der angegebenen Parameter für die weitere Analyse liegen.
- FSC-A- und SSC-A-Streudiagramm: Die Hauptzellpopulation befindet sich in der Nähe der Mitte des Diagramms.
- FSC-A- und FSC-W-Streudiagramm: schmales horizontales Band, das auf Einzelstücke (und nicht auf Zellklumpen) hinweist.
- FITC-A- und FSC-A-Streudiagramm: Beobachten Sie die Verteilung der Zellen, sortiert nach der isoformselektiven fluorogenen Einschaltsonde.
- FITC-A-Histogramm: Beobachten Sie die Verschiebung der Population basierend auf FITC, um den Prozentsatz der ALDH1A1+-Zellen zu bestimmen.
- Um die FITC-Laserleistung zu optimieren, führen Sie eine Probe mit der Sonde so durch, dass sich das rechte Ende der Histogrammkurve in der Nähe des maximalen FITC-A-Signals befindet. Führen Sie anschließend eine Probe mit der Kontrollsonde durch. Eine Populationsverschiebung sollte beobachtbar sein, um den maximalen Dynamikumfang zu ermitteln. Der Schritt zur Optimierung der Laserleistung muss möglicherweise mehrmals wiederholt werden, aber die Laserleistung sollte nicht über die Proben hinweg verändert werden, sobald eine Einstellung für ein Experiment festgelegt wurde.
- Führen Sie jede Probe für 10.000 Zählungen aus (in dreifacher Ausfertigung).
- Wiederholen Sie Schritt 3.12 für jede Zelllinie, da es zu einer Variabilität der Aufnahme und der ALDH1A1-Aktivität kommt.
- Lassen Sie die Leitungen nach Abschluss der Probenentnahme mit jeweils 10 % Bleichmittel und Wasser für jeweils 5 Minuten laufen und leiten Sie dann das Herunterfahren des Geräts ein.
- Verarbeiten Sie die Daten mit der Durchflusszytometrie-Software und ermitteln Sie die gewünschte Zellpopulation. Stellen Sie die Gatter so ein, dass alle Ereignisse entweder in das ALDH1A1- oder ALDH1A1+-Gatter fallen. Stellen Sie das ALDH1A1-Gatter mithilfe der Rechteck-Gate-Auswahl so ein, dass >99,5 % der Ereignisse in den Kontrollsonden-Samples innerhalb dieses Gates auftreten. Die verbleibenden Zellen werden als ALDH1A1+ betrachtet. Dieselben Gatter können dann auf die Sondenprobe angewendet werden, um die Anzahl der Ereignisse zu quantifizieren, die als ALDH1A1- und ALDH1A1+ betrachtet werden.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Gesamtaktivität von ALDH1A1 in Homogenaten von Eierstockkrebszellen
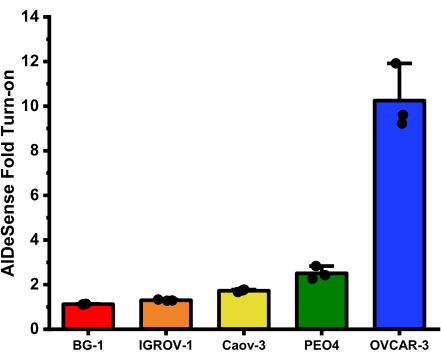
Die durchschnittlichen Fold Turn-Ons für jede Zelllinie, die aus diesem Assay erhalten wurden, sind: BG-1 (1,12 ± 0,01); IGROV-1 (1,30 ± 0,03); Caov-3 (1,72 ± 0,06); PEO4 (2,51 ± 0,29); und OVCAR-3 (10,25 ± 1,46) (Abbildung 2).
Abbildung
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Pan-Selektivität ist eine wesentliche Einschränkung vieler ALDH-Sonden. In jüngster Zeit wurden jedoch mehrere isoformselektive Beispiele berichtet (32,33,34,35,36,37,38,39,40,41)....
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Wir geben ein angemeldetes Patent (US20200199092A1) für die AlDeSense-Technologie bekannt.
Danksagungen
Diese Arbeit wurde von den National Institutes of Health (R35GM133581 an JC) und dem Cancer Center at Illinois Graduate Scholarship (vergeben an SG) unterstützt. JC dankt der Camille und Henry Dreyfus Stiftung für die Unterstützung. Die Autoren danken Dr. Thomas E. Bearrood für seinen ersten Beitrag zur Erstellung von AlDeSense- und AlDeSense-AM-Beständen. Wir danken Herrn Oliver D. Pichardo Peguero und Herrn Joseph A. Forzano für ihre Unterstützung bei der Herstellung verschiedener synthetischer Vorstufen. Wir danken Prof. Erik Nelson (Department of Molecular and Integrative Physiology, UIUC) für Caov-3-, IGROV-1- und PEO4-Zellen. Wir danken Prof. Paul Hergenrother (Department of Chemistry, UIUC) für BG-1-Zellen. Wir danken den Core Facilities des Carl R. Woese Instituts für Genomische Biologie für den Zugang zum Zeiss LSM 700 Konfokalmikroskop und der dazugehörigen Software. Wir danken der Durchflusszytometrie-Einrichtung für den Zugang zum BD LSR II CMtO-Analysator. Wir danken Dr. Sandra McMasters und der Cell Media Facility für die Unterstützung bei der Herstellung von Zellkulturmedien.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin, 0.1% EDTA in HBSS w/o Calcium, Magnesium and Sodium Bicarbonate | Corning | 25-050-CI | |
| 1x Phosphate Buffer Saline | Corning | 21-040-CMX12 | |
| AccuSpin Micro 17R | Fisher Scientific | 13-100-675 | |
| AlDeSense | Synthesized in-house | ||
| BG-1 | A gift provided by the Prof. Paul Hergenrother Lab, University of Illinois Urbana-Champaign | ||
| BioLite 25cm2 Flask | Thermo Fisher Scientific | 130189 | |
| Biosafety Cabinet 1300 series A2 | Thermo Fisher Scientific | ||
| Caov-3 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| Cell homogenizer | Fisher Scientific | ||
| Centrifuge 5180R | Eppendorf | 22627040 | |
| Contrl-AlDeSense | Synthesized in-house | ||
| DMEM, 10% FBS, 1% P/S | Prepared by UIUC cell media facility | ||
| Falcon Round-Bottom Polystyrene Test Tubes with Cell Strainer Snap Cap, 5mL | Corning | 352003 | |
| FCS Express 6 | Provided by UIUC CMtO | ||
| FL microscope | EVOS | ||
| Fluorometer | Photon Technology International | ||
| Forma Series II Water-Jacketed CO2 Incubator | Fisher Scientific | 3110 | |
| IGROV-1 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| ImageJ | NIH | ||
| Innova 42R Incubated Shaker | |||
| LSM 700 | Zeiss | ||
| LSR II | BD | ||
| Nunc Lab-Tek Chambered #1.0 Borosicilate Coverglass System | Thermo Fisher Scientific | 155383 | |
| OVCAR-3 | ATCC | HTB-161 | |
| PEO4 | A gift provided by the Prof. Erik Nelson Lab, University of Illinois Urbana-Champaign | ||
| Pierce Protease Inhibitor Tablets | Thermo Scientific | A32963 | |
| Poly-L-Lysine | Cultrex | 3438-100-01 | |
| Rocker | VWR | ||
| RPMI, 10% FBS, 1% P/S | Prepared by UIUC cell media facility | ||
| RPMI, 20% FBS, 1% P/S, 0.01 mg/mL Insulin | Prepared by UIUC cell media facility |
Referenzen
- Bonnet, D., Dick, J. E. Human acute myeloid leukaemia is organised as a heirarchy that originates from a primitive haematopoetic cell. Nature Medicine. 3 (7), 730-737 (1997).
- Begicevic, R. R., Falasca, M. ABC transporters in cancer stem cells: Beyond chemoresistance. International Journal of Molecular Sciences. 18 (11), 2362(2017).
- Cojoc, M., Mäbert, K., Muders, M. H., Dubrovska, A. A role for cancer stem cells in therapy resistance: Cellular and molecular mechanisms. Seminars in Cancer Biology. 31, 16-27 (2015).
- Islam, F., Gopalan, V., Smith, R. A., Lam, A. K. Y. Translational potential of cancer stem cells: A review of the detection of cancer stem cells and their roles in cancer recurrence and cancer treatment. Experimental Cell Research. 335 (1), 135-147 (2015).
- Li, F., Tiede, B., Massagué, J., Kang, Y. Beyond tumorigenesis: Cancer stem cells in metastasis. Cell Research. 17 (1), 3-14 (2007).
- Kim, W. T., Ryu, C. J. Cancer stem cell surface markers on normal stem cells. BMB Reports. 50 (6), 285-298 (2017).
- Pors, K., Moreb, J. S. Aldehyde dehydrogenases in cancer: An opportunity for biomarker and drug development. Drug Discovery Today. 19 (12), 1953-1963 (2014).
- Jackson, B., et al. Update on the aldehyde dehydrogenase gene (ALDH) superfamily. Human Genomics. 5 (4), 283-303 (2011).
- Vasiliou, V., Pappa, A., Petersen, D. R. Role of aldehyde dehydrogenases in endogenous and xenobiotic metabolism. Chemico-Biological Interactions. 129 (1-2), 1-19 (2000).
- Tomita, H., Tanaka, K., Tanaka, T., Hara, A. Aldehyde dehydrogenase 1A1 in stem cells and cancer. Oncotarget. 7 (10), 11018-11032 (2016).
- Anorma, C., et al. Surveillance of cancer stem cell plasticity using an isoform-selective fluorescent probe for aldehyde dehydrogenase 1A1. ACS Central Science. 4 (8), 1045-1055 (2018).
- Bearrood, T. E., Aguirre-Figueroa, G., Chan, J. Rational design of a red fluorescent sensor for ALDH1A1 displaying enhanced cellular uptake and reactivity. Bioconjugate Chemistry. 31 (2), 224-228 (2020).
- Chan, J., Dodani, S. C., Chang, C. J. Reaction-based small-molecule fluorescent probes for chemoselective bioimaging. Nature Chemistry. 4 (12), 973-984 (2012).
- East, A. K., Lucero, M. Y., Chan, J. New directions of activity-based sensing for in vivo NIR imaging. Chemical Science. 12 (10), 3393-3405 (2021).
- Yadav, A. K., et al. Activity-based NIR bioluminescence probe enables discovery of diet-induced modulation of the tumor microenvironment via nitric oxide. ACS Central Science. 8 (4), 461-472 (2022).
- Yadav, A. K., et al. An activity-based sensing approach for the detection of cyclooxygenase-2 in Live Cells. Angewandte Chemie. 59 (8), 3307-3314 (2020).
- Ueno, T., et al. Rational principles for modulating fluorescence properties of fluorescein. Journal of the American Chemical Society. 126 (43), 14079-14085 (2004).
- Tanaka, F., Mase, N., Barbas 3rd, C. F. Design and use of fluorogenic aldehydes for monitoring the progress of aldehyde transformations. Journal of the American Chemical Society. 126 (12), 3692-3693 (2004).
- Thurber, G. M., Schmidt, M. M., Wittrup, K. D. Antibody tumor penetration: transport opposed by systemic and antigen-mediated clearance. Advanced Drug Delivery Reviews. 60 (12), 1421-1434 (2008).
- Marcato, P., Dean, C. A., Giacomantonio, C. A., Lee, P. W. K. Aldehyde dehydrogenase its role as a cancer stem cell marker comes down to the specific isoform. Cell Cycle. 10 (9), 1378-1384 (2011).
- Meng, E., et al. ALDH1A1 maintains ovarian cancer stem cell-like properties by altered regulation of cell cycle checkpoint and DNA repair network signaling. PLoS One. 9 (9), e107142(2014).
- Kaipio, K., et al. ALDH1A1-related stemness in high-grade serous ovarian cancer is a negative prognostic indicator but potentially targetable by EGFR/mTOR-PI3K/aurora kinase inhibitors. The Journal of Pathology. 250 (2), 159-169 (2020).
- Deng, S., et al. Distinct expression levels and patterns of stem cell marker, aldehyde dehydrogenase isoform 1 (ALDH1), in human epithelial cancers. PLoS One. 5 (4), e10277(2010).
- Chang, B., et al. ALDH1 expression correlates with favorable prognosis in ovarian cancers. Modern Pathology. 22 (6), 817-823 (2009).
- Gardner, S. H., Reinhardt, C. J., Chan, J. Advances in activity-based sensing probes for isoform-selective imaging of enzymatic activity. Angewandte Chemie. 60 (10), 5000-5009 (2021).
- Reid, B. M., Permuth, J. B., Sellers, T. A. Epidemiology of ovarian cancer: a review. Cancer Biology and Medicine. 14 (1), 9-32 (2017).
- Tulake, W., et al. Upregulation of stem cell markers ALDH1A1 and OCT4 as potential biomarkers for the early detection of cervical carcinoma. Oncology Letters. 16 (5), 5525-5534 (2018).
- Nwani, N. G., et al. A novel ALDH1A1 inhibitor targets cells with stem cell characteristics in ovarian cancer. Cancers. 11 (4), 502(2019).
- Roy, M., Connor, J., Al-Niaimi, A., Rose, S. L., Mahajan, A. Aldehyde dehydrogenase 1A1 (ALDH1A1) expression by immunohistochemistry is associated with chemo-refractoriness in patients with high-grade ovarian serous carcinoma. Human Pathology. 73, 1-6 (2018).
- Landen Jr, C. N., et al. Targeting aldehyde dehydrogenase cancer stem cells in ovarian cancer. Molecular Cancer Therapeutics. 9 (12), 3186-3199 (2010).
- Condello, S., et al. β-catenin-regulated ALDH1A1 is a target in ovarian cancer spheroids. Oncogene. 34 (18), 2297-2308 (2015).
- Storms, R. W., et al. Isolation of primitive human hematopoietic progenitors on the basis of aldehyde dehydrogenase activity. Proceedings of the National Academy of Sciences. 96 (16), 9118-9123 (1999).
- Duellman, S. J., et al. A bioluminescence assay for aldehyde dehydrogenase activity. Analytical Biochemistry. 434 (2), 226-232 (2013).
- Minn, I., et al. A red-shifted fluorescent substrate for aldehyde dehydrogenase. Nature Communications. 5, 3662(2014).
- Dollé, L., Boulter, L., Leclercq, I. A., van Grunsven, L. A. Next generation of ALDH substrates and their potential to study maturational lineage biology in stem and progenitor cells. American Journal of Physiology. Gastrointestinal and Liver Physiology. 308 (7), 573-578 (2015).
- Yagishita, A., et al. Development of highly selective fluorescent probe enabling flow-cytometric isolation of ALDH3A1-positive viable cells. Bioconjugate Chemistry. 28 (2), 302-306 (2017).
- Maity, S., et al. Thiophene bridged aldehydes (TBAs) image ALDH activity in cells: Via modulation of intramolecular charge transfer. Chemical Science. 8 (10), 7143-7151 (2017).
- Koenders, S. T. A., et al. Development of a retinal-based probe for the profiling of retinaldehyde dehydrogenases in cancer cells. ACS Central Science. 5 (12), 1965-1974 (2019).
- Oe, M., et al. Deep-red/near-infrared turn-on fluorescence probes for aldehyde dehydrogenase 1A1 in cancer stem cells. ACS Sensors. 6 (9), 3320-3329 (2021).
- Yagishita, A., Ueno, T., Tsuchihara, K., Urano, Y. Amino BODIPY-based blue fluorescent probes for aldehyde dehydrogenase 1-expressing cells. Bioconjugate Chemistry. 32 (2), 234-238 (2021).
- Okamoto, A., et al. Identification of breast cancer stem cells using a newly developed long-acting fluorescence probe, C5S-A, targeting ALDH1A1. Anticancer Research. 42 (3), 1199-1205 (2022).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten