Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Optimierte Methoden zur Oberflächenimmobilisierung von Kollagenen und Kollagenbindungsassays
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
In dieser Arbeit wird ein optimiertes Protokoll zur reproduzierbaren Immobilisierung und Quantifizierung von Kollagen vom Typ I und III auf Mikrotiterplatten vorgestellt, gefolgt von einem verbesserten in vitro Binding Assay-Protokoll zur Untersuchung von Kollagen-Verbindungen-Wechselwirkungen unter Verwendung einer zeitaufgelösten Fluoreszenzmethode. Die anschließende Schritt-für-Schritt-Datenanalyse und Dateninterpretation wird bereitgestellt.
Zusammenfassung
Fibrose tritt in verschiedenen Geweben als reparative Reaktion auf Verletzungen oder Schäden auf. Bei übermäßiger Fibrose kann es jedoch zu Gewebevernarbungen und Organversagen kommen, was mit einer hohen Morbidität und Mortalität verbunden ist. Kollagen ist ein Haupttreiber der Fibrose, wobei Kollagen Typ I und Typ III die Haupttypen sind, die an vielen fibrotischen Erkrankungen beteiligt sind. Im Gegensatz zu herkömmlichen Protokollen, die zur Immobilisierung anderer Proteine (z. B. Elastin, Albumin, Fibronektin usw.) verwendet werden, sind umfassende Protokolle zur reproduzierbaren Immobilisierung verschiedener Arten von Kollagenen zur Herstellung stabiler Beschichtungen nicht ohne weiteres verfügbar. Die Immobilisierung von Kollagen ist eine überraschende Herausforderung, da mehrere experimentelle Bedingungen die Effizienz der Immobilisierung beeinflussen können, einschließlich der Art des Kollagens, des pH-Werts, der Temperatur und der Art der verwendeten Mikroplatte. Hier wird ein detailliertes Protokoll zur reproduzierbaren Immobilisierung und Quantifizierung von Typ-I- und Typ-III-Kollagenen zur Verfügung gestellt, das zu stabilen und reproduzierbaren Gelen/Filmen führt. Darüber hinaus zeigt diese Arbeit, wie zeitaufgelöste Fluoreszenzbindungsstudien in vitro durchgeführt, analysiert und interpretiert werden können, um die Wechselwirkungen zwischen Kollagenen und kollagenbindenden Verbindungen zu untersuchen (z. B. ein Peptid, das an ein Metallchelat konjugiert ist, das z. B. Europium [Eu(III)]) trägt). Ein solcher Ansatz kann universell auf verschiedene biomedizinische Anwendungen angewendet werden, einschließlich des Bereichs der molekularen Bildgebung zur Entwicklung gezielter Bildgebungssonden, der Arzneimittelentwicklung, Zelltoxizitätsstudien, Zellproliferationsstudien und Immunoassays.
Einleitung
Die Ansammlung von fibrösem Bindegewebe im Rahmen des natürlichen Wundheilungsprozesses nach einer Gewebsverletzung wird als Fibrose bezeichnet. Wenn die Ablagerung von fibrösem Gewebe jedoch nicht beendet wird und über das für die Gewebereparatur erforderliche Maß hinausgeht, wird die Fibrose übermäßig 1,2. Übermäßige Fibrose beeinträchtigt die Physiologie und Funktion der Organe und kann zu Organschäden und möglicherweise zu Organversagen führen 3,4,5. Zwei Haupttreiber der Fibrose sind die Proteine der extrazellulären Matrix (ECM), Kollagen Typ I und Typ III6. Kollagen ist ein Strukturprotein, das in verschiedenen Organen vorkommt und etwa ein Drittel des gesamten Proteingehalts des menschlichen Körpers ausmacht1. Es gibt 28 verschiedene Arten von Kollagenen, die durch die Sequenzierung des menschlichen Genoms identifiziert wurden, und die am häufigsten vorkommenden sind die fibrillären Kollagene7. Das primäre fibrilläre Kollagen ist Kollagen vom Typ I, das der EZM Zugfestigkeit und Verformungsbeständigkeit verleiht8. Typ-III-Kollagen ist eine strukturelle Komponente, die für Elastizität sorgt und mit Typ-I-Kollagen kolokalisiert. Es wird während der Embryogenese exprimiert und kommt natürlicherweise in geringen Mengen in der Haut, den Muskeln und den Blutgefäßen von Erwachsenen vor9.
Die In-vivo-Kollagensynthese beginnt mit einem intrazellulären Prozess, bei dem die mRNA im Zellkern abgelesen wird und dann in das Zytoplasma gelangt, wo sie translatiert wird. Nach der Translation erfährt die gebildete Kette eine posttranslationale Modifikation im endoplasmatischen Retikulum, wo Prokollagen (die Vorstufe des Kollagens) gebildet wird. Pro-Kollagen wandert dann zur endgültigen Modifikation in den Golgi-Apparat, bevor es in den extrazellulären Raum ausgeschieden wird10. Durch die proteolytische Spaltung wird Prokollagen in Tropokollagen umgewandelt. Diese wird dann entweder über einen enzymatisch vermittelten Vernetzungsweg, der durch das Enzym Lysyloxidase (LOX) katalysiert wird, oder über einen nicht-enzymatisch vermittelten Vernetzungsweg, an dem die Maillard-Reaktion beteiligt ist, vernetzt11. In-vitro-Protokolle zur Immobilisierung von Kollagen beruhen hauptsächlich auf der Fähigkeit des Kollagens, sich selbst zu organisieren. Kollagen wird aufgrund seiner Löslichkeit aus Geweben gewonnen, die weitgehend vom Ausmaß der Vernetzung einzelner Kollagenfibrillen abhängt7. Fibrilläres Kollagen wird in Essigsäure aufgelöst, und die Fibrillen können sich neu bilden, wenn der pH-Wert und die Temperatur eingestellt werden12. In vitro kann die Fibrillogenese von Kollagen als zweistufiger Prozess angesehen werden7. Die erste Phase ist die Keimbildungsphase, in der Kollagenfasern Dimere und Trimerfibrillen bilden, bevor sie zu einer dreifach helikalen Struktur neu angeordnet werden. Die zweite Phase ist die Wachstumsphase, in der die Fibrillen seitlich zu wachsen beginnen und zur charakteristischen D-Bandenbildung führen, die im Allgemeinen durch Veränderungen der Trübung beobachtet wird7. Untersuchungen der Rasterkraftmikroskopie (AFM) haben auch gezeigt, dass Kollagen Typ I und Typ III unterschiedliche Eigenschaften aufweisen (Tabelle 1)13.
Um die Bindungswechselwirkungen zwischen Kollagen und anderen Verbindungen zu untersuchen, muss Kollagen reproduzierbar in die Vertiefungen von Mikrotiterplatten immobilisiert werden. Es gibt verschiedene Protokolle zur Immobilisierung von löslichem Kollagen 14,15,16. Für die Zellkultur werden in der Regel kommerziell erhältliche Mikrotiterplatten verwendet, die mit Kollagen vorbeschichtet sind. Vorbeschichtete Mikrotiterplatten haben jedoch eine sehr dünne Schicht einer unbekannten Menge Kollagen, die auf die Vertiefungen aufgetragen ist, was sie für In-vitro-Bindungsassays ungeeignet macht. Es gibt mehrere Herausforderungen bei der Immobilisierung von Kollagen in den Plattenvertiefungen. Eine der größten Herausforderungen ist die Wahl eines geeigneten Mikroplattentyps, da verschiedene Arten von Kollagenen (z. B. Typ I und III) unterschiedliche chemische Eigenschaften haben und daher je nach Material der Mikroplatte stabiler und effektiver immobilisiert werden. Eine weitere Herausforderung sind die experimentellen Bedingungen des Immobilisierungsprotokolls, da der Prozess der Fibrillogenese von mehreren Faktoren abhängt, darunter Temperatur, pH-Wert, die Stammkonzentration von Kollagen und die Ionenkonzentration des Puffers7.
Um die Wechselwirkungen zwischen dem Kollagen (dem Ziel) und anderen Verbindungen (d. h. einem Zielpeptid) zu untersuchen, ist es auch notwendig, einen robusten Screening-Assay zu entwickeln, um die Spezifität und Selektivität der Verbindung gegenüber dem Ziel durch Messung der Dissoziationskonstante Kd zu untersuchen. Die Position des Gleichgewichts der Bildung eines bimolekularen Komplexes zwischen einem Protein (Kollagen) und einem Liganden wird durch die Assoziationskonstante Ka ausgedrückt, deren Größe proportional zur Bindungsaffinität ist. Am häufigsten drücken Biochemiker jedoch Affinitätsbeziehungen in Form der Gleichgewichtsdissoziationskonstante Kd des bimolekularen Komplexes aus, die als Kd = 1/Ka (Kd und ist der Kehrwert von Ka) definiert ist. Je niedriger der Kd-Wert, desto stärker ist die Bindungsstärke zwischen dem Protein und dem Liganden. Der Vorteil der Verwendung von Kd zum Vergleich der Bindungsaffinität verschiedener Liganden für dasselbe Protein (und umgekehrt) hängt mit der Tatsache zusammen, dass die Einheiten von Kd für einen bimolekularen Komplex mol/L (d. h. die Konzentrationseinheit) sind. Unter den meisten experimentellen Bedingungen entspricht der Kd-Wert der Ligandenkonzentration, die zu einer 50%igen Sättigung der verfügbaren Bindungsstellen auf dem Target im Gleichgewichtführt 17,18. Die Dissoziationskonstante wird typischerweise durch Analyse der Rezeptorfraktionsbelegung (FO) extrahiert, die als das Verhältnis zwischen den besetzten Bindungsstellen und den insgesamt verfügbaren Bindungsstellen in Abhängigkeit von der Ligandenkonzentration definiert ist. Dies kann unter der Voraussetzung erfolgen, dass ein analytischer Assay verfügbar ist, der in der Lage ist, die Menge des gebundenen Liganden zu unterscheiden und zu messen.
In-vitro-Ligandenbindungsassays können mit verschiedenen bioanalytischen Methoden durchgeführt werden, darunter optische Photometrie, Radioligandenmethoden, Massenspektrometrie mit induktiv gekoppeltem Plasma (ICP-MS) und Oberflächenplasmonenresonanz (SPR). Unter den photometrischen Methoden erfordern diejenigen, die auf Fluoreszenzemission basieren, typischerweise die Markierung von Liganden oder Proteinen mit Fluorophoren, um die Empfindlichkeit zu erhöhen und die Nachweisgrenze des Assays zu verbessern. Chelate bestimmter Lanthanid(III)-Ionen, wie z. B. Eu(III), sind als Fluorophore sehr attraktiv, da sie große Stokes-Verschiebungen, schmale Emissionsbanden (für ein gutes Signal-Rausch-Verhältnis), begrenztes Photobleaching und lange Emissionslebensdauern aufweisen. Wichtig ist, dass die letztere Eigenschaft die Verwendung der zeitaufgelösten Fluoreszenz (TRF) von Eu(III)-Fluorophoren ermöglicht, um die Hintergrund-Autofluoreszenzaufzuheben 19. In der dissoziationsverstärkten Lanthanid-Fluoreszenz-Immunoassay (DELFIA)-Version des Eu(III)-basierten TRF-Assays werden Liganden, die mit einem nicht-lumineszierenden Eu(III)-Chelat markiert sind, inkubiert, wobei der Rezeptor auf Mikrotiterplatten immobilisiert ist. Der markierte Liganden/Rezeptor-Komplex wird von dem ungebundenen Liganden getrennt, und die Eu(III)-Fluoreszenz wird durch Dissoziation des Eu(III)-Komplexes bei einem sauren pH-Wert aktiviert, gefolgt von einer Re-Komplexierung mit einem fluoreszenzverstärkenden Chelator, um einen in Mizellen eingebetteten, stark fluoreszierenden Eu(III)-Komplex20 zu bilden.
Der Dekomplexierungsschritt kann vernünftigerweise mit Chelatoren wie Diethylentriaminpentaacetat (DTPA) erreicht werden, die eine schnelle Dekomplexierungskinetik aufweisen. Eu(III)-Komplexe mit bestimmten makrozyklischen Chelatoren, wie z. B. DOTA (1,4,7,10-Tetraazacyclododecan1,4,7,10-Tetraessigsäure) und seine Monoamidderivate (DO3AAm), weisen jedoch eine hohe thermodynamische Stabilität und eine sehr hohe kinetische Inertheit auf. In diesem Fall müssen die Dekomplexierungsschritte genau optimiert werden, um eine ausreichende und reproduzierbare Aktivierung von Eu(III)-basiertem TRF21 zu erreichen. Es ist erwähnenswert, dass Lanthanid (Ln(III))-DOTA und Ln(III)-DO3AAm-Komplexe diejenigen sind, die am häufigsten als Kontrastmittel für die molekulare In-vivo-Bildgebung mittels Magnetresonanztomographie (MRT) eingesetzt werden22. Daher ist der Ln(III)-basierte TRF-Assay das Werkzeug der Wahl, um in vitro die Bindungsaffinität von molekularen MRT-Sonden zu ihren beabsichtigten biologischen Zielen zu untersuchen. Derzeit fehlen umfassende und reproduzierbare Protokolle für die Immobilisierung von Typ-I- und Typ-III-Kollagen und eine reproduzierbare Pipeline für die Durchführung von in vitro bindenden Eu(III)-TRF-Experimenten. Um diese Einschränkungen zu überwinden, wurden reproduzierbare Methoden zur Selbstorganisation und Immobilisierung von Typ-I- und Typ-III-Kollagen und zur Erzeugung stabiler Gele bzw. Filme mit der für In-vitro-Bindungsassays erforderlichen ausreichenden Kollagenkonzentration entwickelt. Es wird ein optimiertes Protokoll für Eu(III)-TRF von hochinerten Eu(III)-DO3Aam-basierten Komplexen vorgestellt. Schließlich wird ein optimierter in vitro Mikrotiterplatten-Eu(III)-TRF-Assay zur Messung des Kd von Eu(III)-markierten Liganden gegenüber immobilisiertem Typ-I- und Typ-III-Kollagen demonstriert (Abbildung 1).
Protokoll
HINWEIS: Alle Produktinformationen, die für diese Arbeit verwendet werden, sind in der Materialtabelle aufgeführt.
1. Kollagen-Immobilisierung
HINWEIS: Stellen Sie sicher, dass in jeder Vertiefung der Mikroplatte, die während des Bindungsassays verwendet wird, benachbarte Vertiefungen frei sind, um Kreuzfluoreszenz zu vermeiden. Führen Sie diesen Teil des Protokolls auf Eis durch, da sich Kollagen bei steigenden Temperaturen und pH-Werten selbst zusammensetzt. Führen Sie dieses Verfahren in einer Gewebekulturhaube und unter sterilen Bedingungen durch, da die Mikrotiterplatten anschließend in einem Gewebekultur-Inkubator (TC) inkubiert werden.
- Immobilisierung von Typ-I-Kollagen auf den 96-Well-Mikroplatten (Abbildung 2)
Tag 1- Bereiten Sie eine Silikonschale mit Eis vor. Legen Sie das Fläschchen mit Typ-I-Kollagen, die kalte 10-fach phosphatgepufferte Kochsalzlösung (PBS) und die Mikrotiterplatten auf Eis und besprühen Sie alles mit 70 % Ethanol. Legen Sie das Material unter die TC-Haube.
- Neutralisieren Sie das Kollagen mit gleichen Volumina von Kollagen Typ I und 10x PBS (pH 7,4).
- Drehen Sie die Lösung einige Male um und achten Sie darauf, dass sich keine Blasen bilden.
- Geben Sie 100 μl des neutralisierten Kollagens in jede zweite Vertiefung und jede zweite Reihe der Mikroplatte und inkubieren Sie es 18-20 Stunden lang bei 37 °C, um das Kollagen bis zur Trockenheit zu verdampfen.
Tag 2 - Waschen Sie die Mikrotiterplatten zweimal mit 100 μl 1x PBS, pH 7,4, um ungebundenes Kollagen zu entfernen.
- Bringen Sie die Mikrotiterplatten für weitere 2 Stunden bei 37 °C in den Inkubator, um sie zu trocknen, bevor Sie sie für weitere Bindungsexperimente verwenden.
- Immobilisierung von Kollagen Typ III auf den 96-Well-Mikroplatten (Abbildung 3)
Tag 1- Bereiten Sie eine Silikonschale mit Eis vor. Stellen Sie das Fläschchen mit Typ-III-Kollagen, das kalte 10x PBS und die Mikrotiterplatten auf Eis und besprühen Sie alles mit 70% Ethanol. Legen Sie das Material unter die TC-Haube.
- Neutralisieren Sie das Kollagen mit gleichen Volumina von Typ-III-Kollagen und 10x PBS (pH 7,4).
- Geben Sie 70 μl des neutralisierten Kollagens in jede zweite Vertiefung und jede zweite Reihe der Mikroplatte und inkubieren Sie 2 Stunden lang bei 37 °C, indem Sie die Mikroplatte unter die Gewebekulturhaube legen, um das Kollagen bis zur Trockenheit zu verdampfen.
Tag 2 - Waschen Sie die Mikrotiterplatten zweimal mit 70 μl 1x PBS, pH 7,4, um ungebundenes Kollagen zu entfernen.
- Bringen Sie die Mikrotiterplatten für 1 h bei 37 °C in den Inkubator, bringen Sie sie dann auf den Tisch und lassen Sie sie 1 h trocknen, bevor Sie sie in weiteren Bindungsexperimenten verwenden.
2. Beurteilung der Stabilität der immobilisierten Kollagengele/-filme
- Inkubation mit PBS für 1 h
HINWEIS: Inkubieren Sie während des Bindungsexperiments das immobilisierte Kollagen mit der interessierenden Verbindung. Es ist wichtig, die Stabilität des resultierenden Kollagengels oder -films zu untersuchen. Messen Sie dazu die Stabilität von drei Bedingungen: kein Waschen = misst das immobilisierte Kollagen direkt nach der Inkubation; Waschen = misst das immobilisierte Kollagen nach zweimaligem Waschen der Platte mit 100 μL PBS; und 1 h PBS mimic & wash = misst das immobilisierte Kollagen nach 1 h Inkubation mit PBS, gefolgt von zwei Wäschen mit PBS. Im Folgenden wird die PBS-Inkubationsmethode erläutert.- Geben Sie 70 μl PBS (1x) in jede der mit Kollagen beschichteten Vertiefungen und inkubieren Sie die Mikroplatte 1 h lang bei Raumtemperatur.
- Aspirieren Sie die überschüssige Flüssigkeit mit einer Pipette aus jeder Vertiefung und waschen Sie sie zweimal mit PBS (1x), bevor Sie den unten beschriebenen Proteinquantifizierungsassay durchführen.
- Quantifizierung der Menge an immobilisiertem Kollagen mittels eines Bicinchoninsäure-Assays (BCA)
HINWEIS: Verwenden Sie das Pierce BCA Protein Assay Kit (Materialtabelle) gemäß den Anweisungen des Herstellers. Stellen Sie entsprechende Kollagenstandards für diesen Assay her. Der Konzentrationsbereich für Kollagen I liegt zwischen 0-3.000 μg/ml und für Kollagen III zwischen 0 und 750 μg/ml. Machen Sie insgesamt 11 Standards pro Kollagen.- Bereiten Sie das Gesamtvolumen des benötigten Arbeitsreagenzes (WR) vor, indem Sie die Anweisungen des Herstellers befolgen.
- Geben Sie jeweils 25 μl des Kollagenstandards in die Mikrotiterplatten-Vertiefungen (in doppelter Ausführung). Diese Lösungen werden verwendet, um die Standardkurve zu zeichnen.
- Geben Sie 200 μl Arbeitsreagenzlösung in jede Vertiefung, die die Standards enthält, und in die Vertiefungen, die mit unbekannten Kollagenkonzentrationen beschichtet sind.
- Legen Sie die Mikroplatte für 30 s auf einen Plattenschüttler. Decken Sie die Mikrotiterplatten ab und inkubieren Sie sie 30 Minuten lang bei 37 °C.
- Entfernen Sie die Mikrotiterplatten und lassen Sie sie bei Raumtemperatur abkühlen. Messen Sie die Extinktion bei 560 nm mit einem Plattenleser.
- Erstellen Sie eine Kalibrierkurve, indem Sie A560 (AU) mit der Konzentration (μg/ml) der 11 Standardlösungen vergleichen, und verwenden Sie die Kalibrierkurve, um die Kollagenmenge zu berechnen.
3. Europium(III) TRF-Ligandenbindungsassay (Abbildung 1)
HINWEIS: Bei der verwendeten Verbindung handelt es sich um ein kollagenbindendes Peptid (CBP), das mit einem einzigen Eu(III)-DO3AAm-Komplex markiert ist, der als Eu(III)-DO3AAm-CBP bezeichnet wird (Abbildung 4).
- Inkubation der kollagenbeschichteten Platten mit der Eu(III)-DO3AAm-CBP-Verbindung
- Bereiten Sie Lösungen der Eu(III)-DO3AAm-CBP-Verbindung mit Konzentrationen zwischen 0,1 und 15 μM (0,1 μM, 0,5 μM, 1 μM, 3 μM, 5 μM, 7 μM, 10 μM und 15 μM) in 1x PBS her.
- Geben Sie 75 μl jeder Konzentration der Verbindung in die mit Kollagen beschichteten Vertiefungen (Platte A). Führen Sie das Experiment in dreifacher Ausführung durch, um die Menge der Verbindung zu berechnen, die an das Kollagen bindet.
- Verwenden Sie eine zweite unbeschichtete Platte (Platte B) und geben Sie 75 μl jeder Verbindung in die leeren Vertiefungen, um die unspezifische Bindung der Verbindung an die Platte zu berechnen. Verwenden Sie Triplikate für jede Konzentration.
- Inkubieren Sie die Mikrotiterplatten 1 h lang bei Raumtemperatur.
- Aspirieren und verwerfen Sie mit einer Pipette die überschüssige Lösung aus jeder Vertiefung und waschen Sie die Vertiefungen zweimal mit 1x PBS, um überschüssige, ungebundene Verbindung zu entfernen. Führen Sie diesen Schritt sowohl mit den kollagenbeschichteten als auch mit den unbeschichteten Mikrotiterplatten durch.
- Auf eine dritte unbeschichtete Platte (Platte C) werden 10 μl des gleichen Bereichs von Eu(III)-DO3AAm-CBP-Konzentrationen (in zweifacher Ausfertigung) gegeben. Verwenden Sie den Fluoreszenzmesswert des Eu(III)-DO3AAm-CBP in Lösung, um eine Kalibrierkurve zu erstellen.
HINWEIS: Waschen oder aspirieren Sie die Lösung nicht von dieser Platte.
- Säureextraktion von Europium(III) und zeitaufgelösten Fluoreszenzmesswerten (TRF)
HINWEIS: Bitte beachten Sie die ergänzenden Informationen zur Herstellung und Kalibrierung der Volumina der sauren Lösung (AS) und der Pufferlösung (BS). Die Volumina des AS und BS, die erforderlich sind, um reproduzierbar einen optimalen pH-Wert zu erreichen, betrugen in dieser Arbeit 54 μl bzw. 46 μl. Führen Sie den folgenden Vorgang an Platte A, Platte B und Platte C durch.- Geben Sie 54 μl saure Lösung (AS) in jede Vertiefung und legen Sie die Platte 90 Minuten lang bei 37 °C in den Inkubator, wobei Sie die Mikrotiterplatten mit Folie abdecken, um eine Verdunstung zu vermeiden. Die Temperatur und die Inkubationszeit müssen sorgfältig kontrolliert werden, um eine reproduzierbare Dekomplexierung zu erreichen.
- Geben Sie 46 μl Pufferlösung (BS) in jede Vertiefung und schütteln Sie die Platte vorsichtig 30 s lang.
- Fügen Sie 100 μl Verstärkungslösung (ES) hinzu und schütteln Sie die Platte 30 s lang.
- Warten Sie 30 Minuten, bevor Sie die Platte mit einem TRF-Plattenleser ablesen. Verwenden Sie die in Tabelle 2 aufgeführten Parameter.
4. Datenanalyse
- Quantifizierung der Konzentration von Kollagen, das auf den Vertiefungen immobilisiert ist
- Ermitteln Sie die Gleichung der Kalibrierkurve des A560 (AU) gegenüber der Konzentration (μg/mL) der 11 Standardlösungen.
- Verwenden Sie die Absorptionswerte, die aus den Vertiefungen mit den Kollagenstandards gewonnen wurden.
- Tabellarisch darstellen Sie die Durchschnittswerte aus den duplizierten Vertiefungen und stellen Sie die mittlere Absorption gegen die bekannten Proteinkonzentrationen (Kollagen) (μg/ml) dar, um die Gleichung für die Standardkurve zu erhalten.
- Verwenden Sie die Absorptionswerte, um die Masse (μg) und Konzentration (M) von immobilisiertem Kollagen zu berechnen.
- Berechnen Sie die durchschnittlichen Absorptionswerte für die drei Vertiefungen, die immobilisiertes Kollagen enthielten, und notieren Sie die Standardabweichung.
- Verwenden Sie die Standardkurvengleichung, die aus der Kollagen-Standardkurve (Schritt 2.2.6) erhalten wurde, um die aus den kollagenbeschichteten Vertiefungen gemessene Absorption in eine Konzertierung umzuwandeln. Berechnen Sie daraus die Konzentration des Kollagens, das in den experimentellen Vertiefungen immobilisiert wurde, in μg/ml.
- Die in Schritt 4.1.2.2 berechnete Konzentration (μg/ml) wird zunächst in Gramm/Liter und dann, basierend auf dem Molekulargewicht des Kollagens, in molaren (M) umgerechnet.
- Berechnen Sie schließlich die Masse des in jeder Vertiefung immobilisierten Kollagens, indem Sie die Konzentration durch das Volumen des Kollagens dividieren, das in die Vertiefung gegeben wurde (100 μl für Kollagen Typ I und 70 μl für Kollagen Typ III).
- Ermitteln Sie die Gleichung der Kalibrierkurve des A560 (AU) gegenüber der Konzentration (μg/mL) der 11 Standardlösungen.
- Berechnung der Dissoziationskonstante (Kd) (Abbildung 4)
- Extrahieren Sie die Fluoreszenzmesswerte.
- Exportieren Sie die Fluoreszenzmesswerte aus dem Platten-Reader in eine Tabelle.
HINWEIS: Bei Bindungsassays ist es wichtig, die mögliche unspezifische Bindung einer Verbindung an die plastische Oberfläche der Platten zu berücksichtigen. - Berechnen Sie die Mittelwerte der dreifachen Messungen von jeder Verbindungskonzentration für die drei verschiedenen Platten: die spezifischen Bindungsmesswerte aus den beschichteten Vertiefungen (Platte A), die unspezifische Bindung aus den unbeschichteten Vertiefungen (Platte B) und das Gesamt-Eu(III)-DO3AAm-CBP in Lösung in den unbeschichteten Vertiefungen (Platte C).
- Die Fluoreszenzwerte für die gebundene Verbindung werden bestimmt, indem die Fluoreszenzmesswerte der unbeschichteten Vertiefungen (Platte B) von denen der beschichteten Vertiefungen (Platte A) subtrahiert werden.
Gleichung 1: Bestimmung der gebundenen Fluoreszenz17:
Gebundene Fluoreszenz = Spezifisch (beschichtete Vertiefungen) - Unspezifisch (unbeschichtete Vertiefungen) - Erstellen Sie eine Kalibrierkurve unter Verwendung der Messwerte der Eu(III)-markierten Verbindung in Lösung (Platte C). Die erhaltenen Fluoreszenzmesswerte werden gegen die Konzentration der Eu(III)-markierten Verbindung aufgetragen. Führen Sie eine lineare Regressionsanpassung durch.
- Exportieren Sie die Fluoreszenzmesswerte aus dem Platten-Reader in eine Tabelle.
- Rechnen Sie die Fluoreszenzmesswerte in Konzentrationen um.
- Die Messwerte der gebundenen Fluoreszenz (Schritt 4.2.3) werden unter Verwendung der Standardfluoreszenzkurve aus den Daten, die unter Verwendung der Verbindungskonzentrationen in Lösung (Schritt 4.2.1.4) erzeugt wurden, in die Konzentration umgerechnet.
HINWEIS: Beim Vergleich der Bindungseigenschaften einer Verbindung zu verschiedenen Zielproteinen, die in unterschiedlichen Konzentrationen immobilisiert werden, muss letzteres bei der Berechnung der Menge der an das Ziel gebundenen Verbindung (d. h. gebundene Verbindung/Protein) berücksichtigt werden. - Die Konzentration der gebundenen Verbindung wird durch die Konzentration des in der Vertiefung immobilisierten Proteins dividiert.
HINWEIS: Für diese Berechnung wird die Konzentration an immobilisiertem Kollagen verwendet, die berechnet wurde, nachdem die Vertiefungen 1 h lang mit PBS inkubiert wurden (sogenanntes PBS-Nachahmungsexperiment; Abschnitt 2.1 oben). Dies dient dazu, mögliche Kollagenverluste während des Inkubations- und Waschschritts zu berücksichtigen, die nicht zum endgültigen Fluoreszenzsignal beitragen. - Stellen Sie die Daten mit einem Streudiagramm dar, das die Konzentrationen der Verbindung auf der x-Achse (μM) und die gebundene Verbindung/das gebundene Protein auf der y-Achse enthält.
- Die Messwerte der gebundenen Fluoreszenz (Schritt 4.2.3) werden unter Verwendung der Standardfluoreszenzkurve aus den Daten, die unter Verwendung der Verbindungskonzentrationen in Lösung (Schritt 4.2.1.4) erzeugt wurden, in die Konzentration umgerechnet.
- Rufen Sie die Kd-Werte ab.
- Passen Sie die in Schritt 4.2.2.3 erfassten Daten mit zwei möglichen kinetischen Bindungsmodellen an: Ein-Standort-Bindung und Ein-Standort-Bindung mit einer Hangneigung. Die Gleichungen für jedes Modell sind in Abbildung 6 dargestellt.
- Wählen Sie das Modell aus, das beim Anpassen der Daten eine eindeutige Anpassung mit dem höchsten R-Quadrat-Wert bietet.
- Schließen Sie die Ausreißer (s) für jeden Satz von Fluoreszenzmesswerten pro Konzentration und Platte aus.
- Berechnen Sie den endgültigen Kd-Wert und stellen Sie die Daten als Mittelwert ± Standardabweichung unabhängiger Experimente dar.
HINWEIS: Um robuste Ergebnisse zu erzielen, führen Sie dreifache Messungen innerhalb jeder Platte und mindestens drei unabhängige Experimente mit verschiedenen Mikrotiterplatten durch.
- Berechnen Sie die fraktionale Belegung (FO).
HINWEIS: Aus Gleichung 2 ist die Konzentration des Ziels unbekannt, und daher ergibt sich aus Gleichung 3 unter Verwendung von Algebra und Kd eine praktikable Gleichung zur Berechnung der fraktionellen Belegung in Form von Gleichung 4.
Gleichung 2: Definition der Teilbelegung17:
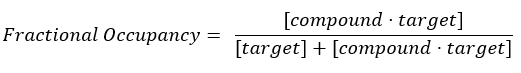
Gleichung 3: Die Dissoziationskonstante Kd, d. die Konzentration, bei der die Verbindung 50 % des Ziels im Gleichgewicht17 einnimmt:
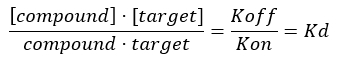
Gleichung 4: Gleichung neu angeordnet, um die FO-Gleichung17 zu berechnen:
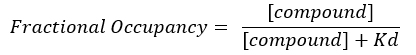
- Berechnen Sie den FO anhand der unabhängigen Kd-Werte , die für jede einzelne Platte erhalten werden. Plotten Sie die Ergebnisse, den Mittelwert und die Standardabweichungen des FO gegen die Konzentration der Verbindung.
- Melden Sie den FO mit Werten zwischen 0 und 1 oder als Prozentsatz mit Werten zwischen 0 % und 100 %.
- Extrahieren Sie die Fluoreszenzmesswerte.
Ergebnisse
Beurteilung der Stabilität und Konzentration von Typ-I- und Typ-III-Kollagen, das in Gelen/Filmen immobilisiert ist
Die Quantifizierung der immobilisierten Kollagenkonzentration pro Vertiefung erfolgte unter drei verschiedenen Bedingungen: a) in Vertiefungen ohne Waschen mit PBS nach Immobilisierung der Proteine (keine Wäsche); b) in Vertiefungen mit einem Waschschritt (zweimal mit PBS) nach der Immobilisierung, um unbeschichtetes Protein zu entfernen; c) in Vertief...
Diskussion
In dieser Arbeit wird eine reproduzierbare Methode zur Immobilisierung von Kollagen vom Typ I und Typ III vorgestellt. Es demonstriert auch ein Protokoll zur Erfassung, Analyse und Interpretation von in vitro Eu(III)-TRF-Bindungsdaten, um die Bindungseigenschaften eines Kandidatenliganden gegenüber Kollagen vom Typ I und III zu charakterisieren. Die hier vorgestellten Protokolle zur Immobilisierung von Typ-I- und Typ-III-Kollagen wurden unter Berücksichtigung zuvor veröffentl...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken den folgenden Geldgebern für die Unterstützung dieser Arbeit: (1) dem UK Medical Research Council (MR/N013700/1) und dem King's College London, Mitglied der MRC Doctoral Training Partnership in Biomedical Sciences; (2) Zuschuss des BHF-Programms RG/20/1/34802; (3) BHF-Projektzuschuss PG/2019/34897; (4) Zuschuss des King's BHF Centre for Research Excellence RE/18/2/34213; (5) das ANID Millennium Science Initiative Program - ICN2021_004; und (6) ANID Basal Grant FB210024.
Materialien
| Name | Company | Catalog Number | Comments |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| Ice | |||
| Ice | |||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH meter (sevenCompact) | Mettler Toledo | ||
| pH meter (sevenCompact) | Mettler Toledo | ||
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Tissue culture incubator (37 °C, 5% CO2) | |||
| Type I bovine collagen, 3 mg/mL | Corning | 354231 | |
| Type III human placenta collagen, 0.99 mg/mL | Advanced Biomatrix | 5021 |
Referenzen
- Distler, J. H. W., et al. Review: Frontiers of antifibrotic therapy in systemic sclerosis. Arthritis and Rheumatology. 69 (2), 257-267 (2017).
- Wynn, T. A. Fibrotic disease and the TH1/TH2 paradigm. Nature Reviews Immunology. 4 (8), 583-594 (2004).
- Saha, P., et al. Magnetic resonance T1 relaxation time of venous thrombus is determined by iron processing and predicts susceptibility to lysis. Circulation. 128 (7), 729-736 (2013).
- Mirshahi, M., et al. Defective thrombolysis due to collagen incorporation in fibrin clots. Thrombosis Research. 8, 73-80 (1988).
- Comerota, A. J. The ATTRACT trial: Rationale for early intervention for iliofemoral DVT. Perspectives in Vascular Surgery and Endovascular Therapy. 21 (4), 221-225 (2009).
- Bateman, E. D., Turner-Warwick, M., Adelmann-Grill, B. C. Immunohistochemical study of collagen types in human foetal lung and fibrotic lung disease. Thorax. 36 (9), 645-653 (1981).
- Pawelec, K. M., Best, S. M., Cameron, R. E. Collagen: A network for regenerative medicine. Journal of Materials Chemistry B. 4 (40), 6484-6496 (2016).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Copes, F., Pien, N., Van Vlierberghe, S., Boccafoschi, F., Mantovani, D. Collagen-based tissue engineering strategies for vascular medicine. Frontiers in Bioengineering and Biotechnology. 7, 166 (2019).
- Veis, A. The biochemistry of collagen. Annals of Clinical and Laboratory Science. 5 (2), 123-131 (1975).
- Bielajew, B. K., Hu, J. C., Athanasiou, K. A. Collagen: Quantification, biomechanics, and role of minor subtypes in cartilage. Nature Reviews Materials. 5, 730-747 (2020).
- Zhao, Z., et al. Structural and functional plasticity of collagen fibrils. DNA and Cell Biology. 38 (4), 367-373 (2019).
- Eryilmaz, E., Teizer, W., Hwang, W. In vitro analysis of the co-assembly of type-I and type-III collagen. Cellular and Molecular Bioengineering. 10 (1), 41-53 (2017).
- Jagnow, J., Clegg, S. Klebsiella pneumoniae MrkD-mediated biofilm formation on extracellular matrix- and collagen-coated surfaces. Microbiology. 149 (9), 2397-2405 (2003).
- O'Sullivan, D., O'Neill, L., Bourke, P. Direct plasma deposition of collagen on 96-well polystyrene plates for cell culture. ACS Omega. 5 (39), 25069-25076 (2020).
- Caravan, P., et al. Collagen-targeted MRI contrast agent for molecular imaging of fibrosis. Angewandte Chemie - International Edition. 46 (43), 8171-8173 (2007).
- Copeland, R. A. . Enzymes: A Practical Introduction to Structure, Mechanism, and Data Analysis. , (2000).
- Salahudeen, M. S., Nishtala, P. S. An overview of pharmacodynamic modelling, ligand-binding approach and its application in clinical practice. Saudi Pharmaceutical Journal. 25 (2), 165-175 (2017).
- Bünzli, J. C. G., Piguet, C. Taking advantage of luminescent lanthanide ions. Chemical Society Reviews. 34 (12), 1048-1077 (2005).
- Hemmilii, I. Luminescent lanthanide chelates - A way to more sensitive diagnostic methods. Journal of Alloys and Compounds. 225 (1-2), 480-485 (1995).
- De Silva, C. R., Vagner, J., Lynch, R., Gillies, R. J., Hruby, V. J. Optimization of time-resolved fluorescence assay for detection of europium-tetraazacyclododecyltetraacetic acid-labeled ligand-receptor interactions. Analytical Biochemistry. 398 (1), 15-23 (2010).
- Digilio, G., Lacerda, S., Lavin Plaza, B., Phinikaridou, A. Extracellular matrix targeted MRI probes. Analysis & Sensing. 3 (1), (2022).
- Phinikaridou, A., et al. Tropoelastin: A novel marker for plaque progression and instability. Circulation. Cardiovascular imaging. 11 (8), 007303 (2018).
- Guzaeva, T. V., et al. Protein A used in DELFIA for the determination of specific antibodies. Immunology Letters. 35 (3), 285-289 (1993).
- Nasiri, A. H., Nasiri, H. R. Polymerase assays for lead discovery: An overall review of methodologies and approaches. Analytical Biochemistry. 563, 40-50 (2018).
- Capuana, F., et al. Imaging of dysfunctional elastogenesis in atherosclerosis using an improved gadolinium-based tetrameric MRI probe targeted to tropoelastin. Journal of Medicinal Chemistry. 64 (20), 15250-15261 (2021).
- Drescher, D. G., Drescher, M. J., Ramakrishnan, N. A. Surface plasmon resonance (SPR) analysis of binding interactions of proteins in inner-ear sensory epithelia. Methods in Molecular Biology. 493, 323-343 (2009).
- Murali, S., Rustandi, R. R., Zheng, X., Payne, A., Shang, L. Applications of surface plasmon resonance and biolayer interferometry for virus-ligand binding. Viruses. 14 (4), 717 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten