Analyse der transgenerationalen epigenetischen Vererbung in C. elegans mittels Fluoreszenzreporter und Chromatin-Immunpräzipitation (ChIP)
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen RNA-Interferenz- und ChIP-Assay zur Untersuchung der epigenetischen Vererbung von RNAi-induziertem Silencing und damit verbundenen Chromatinmodifikationen in C. elegans.
Zusammenfassung
Die transgenerationale epigenetische Vererbung (TEI) ermöglicht die Übertragung von Informationen durch die Keimbahn, ohne die Genomsequenz zu verändern, durch Faktoren wie nicht-kodierende RNAs und Chromatinmodifikationen. Das Phänomen der RNA-Interferenz (RNAi)-Vererbung im Fadenwurm Caenorhabditis elegans ist ein effektives Modell zur Untersuchung von TEI, das sich den kurzen Lebenszyklus, die Selbstvermehrung und die Transparenz dieses Modellorganismus zunutze macht. Bei der RNAi-Vererbung führt die Exposition von Tieren gegenüber RNAi zu Gen-Silencing und veränderten Chromatinsignaturen am Zielort, die über mehrere Generationen hinweg ohne den ursprünglichen Auslöser bestehen bleiben. Dieses Protokoll beschreibt die Analyse der RNAi-Vererbung in C. elegans unter Verwendung eines Keimbahn-exprimierten kerngrünen Fluoreszenzproteins (GFP)-Reporters. Das Reporter-Silencing wird durch die Fütterung der Tiere mit Bakterien eingeleitet, die doppelsträngige RNA exprimieren, die auf GFP abzielt. Bei jeder Generation werden die Tiere durchgelassen, um eine synchronisierte Entwicklung aufrechtzuerhalten, und das Stummschalten der Reportergene wird durch Mikroskopie bestimmt. In ausgewählten Generationen werden Populationen gesammelt und für die Chromatin-Immunpräzipitation (ChIP)-quantitative Polymerase-Kettenreaktion (qPCR) aufbereitet, um die Anreicherung der Histonmodifikation am GFP-Reporterlocus zu messen. Dieses Protokoll zur Untersuchung der RNAi-Vererbung kann leicht modifiziert und mit anderen Analysen kombiniert werden, um TEI-Faktoren in kleinen RNA- und Chromatin-Signalwegen weiter zu untersuchen.
Einleitung
Epigenetische Vererbung ermöglicht die Weitergabe genregulatorischer Informationen über Generationen hinweg und kann daher dazu führen, dass die Umwelt oder die Erfahrungen der Eltern ihre Nachkommen beeinflussen. Bei C. elegans kann das durch exogene doppelsträngige RNA (dsRNA) initiierte Gen-Silencing in der Keimbahn für mehrere Generationen in Nachkommen vererbt werden, die nicht dem ursprünglichen Auslöserexponiert sind 1,2,3,4. Dieser Prozess, der als RNA-Interferenz (RNAi)-Vererbung bezeichnet wird, ist eines von mehreren verwandten epigenetischen Silencing-Phänomenen bei C. elegans, einschließlich piRNA-initiiertes mehrgenerationenübergreifendes Silencing 2,5, Paramutation/RNAe (RNA-induziertes epigenetisches Silencing)6,7,8 und Multicopy-Array-initiiertes Silencing9, die überlappende, aber unterschiedliche Anforderungen an kleine RNA- und Chromatin-Regulationsmaschinen stellen . In der exogenen RNAi von C. elegans wird dsRNA zu kleinen interferierenden RNAs (siRNAs) verarbeitet, die in einem Komplex mit primären Argonautenproteinen agieren, um ihre Ziel-mRNA zu erkennen. Dieses Targeting führt zur Amplifikation von sekundären siRNAs, die mit sekundären Argonauten assoziiert sind, um die Ziel-mRNA sowohl über zytoplasmatische als auch über nukleäre Silencing-Signalwege zum Schweigen zu bringen. Für Keimbahn-exprimierte RNAi-Targets zielen das nukleäre sekundäre Argonaute HRDE-1 und zusätzliche nukleäre RNAi-Faktoren auf naszierende Transkripte ab, was zu einer transkriptionellen Repression und Rekrutierung von Histonmethyltransferasen führt, um repressive Chromatinmarkierungen, einschließlich H3K9me310, abzuscheiden. Histon H3K9me3 fördert die Etablierung eines vererbbaren Silencings von Keimbahn-exprimierten grün fluoreszierenden Proteinen (GFP)-Transgenen durch RNAi-Vererbung11,12.
Das Ziel dieses Protokolls ist es, ein Transgen, das ein GFP-Histon-Fusionsprotein in der Keimbahn exprimiert, als Reporter für die RNAi-Vererbung zu verwenden und Veränderungen der Histonmodifikationen am Reportertransgenlocus mittels Chromatin-Immunpräzipitation und quantitativer Polymerase-Kettenreaktion (ChIP-qPCR) zu untersuchen. Dieses Protokoll beschreibt einen standardisierten plattenbasierten RNAi-Fütterungsansatz, um das Reporter-Silencing zu initiieren. Es bietet auch einen detaillierten Zeitplan für die Passage von Tieren zwischen den Generationen, indem Embryonen in utero von trächtigen Erwachsenen durch alkalische Hypochloritbehandlung ("Bleichung") isoliert werden. Methoden und repräsentative Daten zur Überwachung der Häufigkeit des GFP-Silencings in einer Untergruppe der Bevölkerung mittels Fluoreszenzmikroskopie und für die Histon H3K9me3 ChIP-qPCR werden ebenfalls beschrieben. Reporter-basierte RNAi-Vererbungsassays bieten ein hochgradig handhabbares System, um die Rolle genetischer und umweltbedingter Faktoren in der epigenetischen Regulation funktionell zu untersuchen 13,14, und genetische Screenings mit solchen Reportern haben sowohl Gene identifiziert, die für 2,3,15 erforderlich sind, als auch Gene, die 16,17 die Dauer der transgenerationalen epigenetischen Vererbung negativ regulieren.
Protokoll
HINWEIS: Abbildung 1 zeigt einen Zeitplan für den Assay.
1. Herstellung von RNAi-Nematoden-Wachstumsmedium-Platten (RNAi-NGM)
- E. coli HT115(DE3), das entweder GFP- oder Kontroll-RNAi-Vektoren aus Glycerinstämmen enthält, auf Luria-Bertani (LB)-Agarplatten auftragen, die mit 100 μg/ml Ampicillin ergänzt werden. Inkubieren Sie die Bakterien über Nacht bei 37 °C.
- Am nächsten Tag werden RNAi-NGM-Platten (1,7 % [w/v] Agar, 0,3 % [w/v] NaCl, 0,25 % [w/v] Pepton, 1 mM CaCl2, 5 μg/ml Cholesterin, 25 mM Kaliumphosphatpuffer [pH 6,0], 1 mM MgSO4, 25 μg/ml Carbenicillin und 5 mM Isopropyl-β-d-1-thiogalactopyranosid [IPTG]) gemäß dem Standardprotokoll18 hergestellt.
- Bereiten Sie für jeden Stamm im Assay mindestens sechs RNAi-NGM-Platten mit GFP-RNAi (zwei Platten für jedes der drei biologischen Replikate) und zwei RNAi-NGM-Platten mit Kontroll-RNAi (ein biologisches Replikat) vor.
- Die Platten zum Schutz vor Licht locker mit Folie abbinden und über Nacht bei Raumtemperatur trocknen lassen.
- Am selben Tag wie das Ausgießen der RNAi-NGM-Platte 4 ml Flüssigkulturen von RNAi-Bakterien vorbereiten.
- Unter sterilen Bedingungen werden 4 ml LB-Bouillon aliquot mit 10 μg/ml Tetracyclin und 100 μg/ml Ampicillin in Kulturröhrchen gegeben. Entnehmen Sie einzelne Kolonien aus den LB-Agarplatten in jedes Röhrchen und inkubieren Sie sie über Nacht unter ca. 16 h Schütteln bei 37 °C.
- Säen Sie RNAi-Bakterien auf die RNAi-NGM-Platten.
- Messen Sie nach dem Wachstum über Nacht die optische Dichte (OD600) der Kulturen mit einer 1:5-Verdünnung der Bakterien mit LB-Brühe.
- Verdünnen Sie RNAi-Bakterien auf einen relativen OD von 600 von 2 mit LB-Bouillon, ergänzt mit 10 μg/ml Tetracyclin und100 μg/ml Ampicillin.
- Geben Sie unter sterilen Bedingungen 150 μl der Kontroll- oder GFP-RNAi-Bakterien auf jede RNAi-NGM-Platte.
- Planen Sie die Platten mit Folie und lassen Sie die Bakterien vor der Verwendung mindestens 24 h bei Raumtemperatur wachsen.
HINWEIS: Unbenutzte RNAi-NGM-Platten können invertiert bis zu 1 Woche bei 4 °C in einem verschlossenen Behälter im Dunkeln gelagert werden.
2. Start des RNAi-Vererbungsassays: Bleichen und Plattieren von Embryonen für die P0-Generation
HINWEIS: Vor Beginn des RNAi-Vererbungstests sollten Würmer, die den GFP-Reporter mjIs134 [mex-5p::gfp-h2b::tbb-2 3'UTR]7 enthalten, mindestens zwei Generationen lang bei 21 °C unverhungert bleiben.
- 4 Tage (96 Stunden) vor Beginn des RNAi-Vererbungstests (Abbildung 1) werden drei oder vier schwangere Erwachsene auf jede 35-mm-Standard-NGM-Platte mit OP50-1-Bakterien gepickt.
HINWEIS: Drei NGM-Platten pro Stamm sind ausreichend. Für Stämme mit beeinträchtigter Fruchtbarkeit können mehr als vier Tiere pro Platte erforderlich sein. - Stellen Sie nach 4 Tagen sicher, dass die erwachsene Bevölkerung begonnen hat, Embryonen auf die Platte zu legen. Waschen Sie die Würmer von den Platten in 1,5-ml-Röhrchen mit 800 μl M9-Puffer, ergänzt mit Triton X-100 (TX-100) (22 mM KH 2 PO 4, 42 mM Na2HPO 4, 86 mM NaCl, 1 mM MgSO 4, 0,01 % (v/v) TX-100). Wiederholen Sie den Waschvorgang und geben Sie ihn in dieselbe Röhre.
- Isolieren Sie die Embryonen aus der Population durch Bleichen wie folgt.
- Bereiten Sie eine Bleichlösung mit Bleichmittel (6% Natriumhypochlorit) und 10 N NaOH in einem Volumenverhältnis von 1,5:1 vor. Bereiten Sie eine ausreichende Menge vor, so dass für jede Probe 250 μl verwendet werden können.
- Zentrifugieren Sie die 1,5-ml-Röhrchen mit den Würmern bei 1.000 x g für 1,5-2 Minuten.
- Saugen Sie den Überstand an und lassen Sie 100 μl übrig, ohne das "Pellet" des Wurms zu stören. Alternativ können die Röhrchen auch ca. 2 Minuten einwirken lassen, damit sich die Würmer vor dem Absaugen absetzen können.
- Geben Sie 650 μl ddH2O in jedes Röhrchen, um ein Endvolumen von 750 μl zu erreichen.
HINWEIS: Die folgenden Schritte sind zeitkritisch. - Geben Sie 250 μl der Bleichlösung in jedes Röhrchen und starten Sie einen Timer.
- Alle 1-2 Minuten die Röhrchen gründlich vortexen. Untersuchen Sie nach 5 Minuten die Würmer unter dem Stereoskop, um den Abbau der erwachsenen Tiere zu überwachen.
- Fahren Sie mit dem Wirbel fort, bis sich die Würmer vollständig aufgelöst haben und nur noch Embryonen übrig bleiben. Die Röhrchen werden sofort bei 1.000 x g für 1,5 min zentrifugiert.
HINWEIS: Das Bleaching ist in der Regel innerhalb von 6-7 Minuten abgeschlossen, sollte aber unter einem Stereoskop mit einer Vergrößerung überwacht werden, die ausreicht, um die freigesetzten Embryonen (40x) zu beobachten. Lassen Sie die Würmer nicht über einen längeren Zeitraum in Bleichmittel, da dies die Embryonen beeinträchtigt. - Nach der Zentrifugation wird der Überstand abgesaugt und ca. 50-100 μl übrig gelassen. 1 ml M9-Puffer mit TX-100 zum Waschen und Mischen durch Wirbel hinzufügen. Erneut bei 1.000 x g für 1,5-2 min zentrifugieren und mindestens zwei weitere Male waschen.
- Saugen Sie den Überstand aus der letzten Wäsche an und lassen Sie ca. 100 μl im Röhrchen. Mischen Sie durch Vortexing. Pipettieren Sie 2 μl aus jedem Röhrchen zweimal auf einen beschrifteten Objektträger. Zählen Sie die Embryonen mit einem Zählzähler, um die Konzentration der Embryonen pro Mikroliter zu schätzen.
HINWEIS: Objektträger mit mehreren mattierten Vertiefungen können hier verwendet werden, um Replikate mehrerer Stämme zu zählen. Zählobjektträger können gewaschen und wiederverwendet werden.
- Um ein halbsynchronisiertes Populationswachstum zu starten, mischen und pipettieren Sie ein Volumen mit 250 Embryonen auf jede RNAi-NGM-Platte. Verwenden Sie für jede Replikation zwei Platten. Lassen Sie die Flüssigkeit einziehen.
- Für die mit RNAi behandelte P0-Generation wird bei 21 °C für 4 Tage (96 h) inkubiert (vom Embryo bis ins Erwachsenenalter). Der Zeitpunkt kann je nach Genotyp variieren.
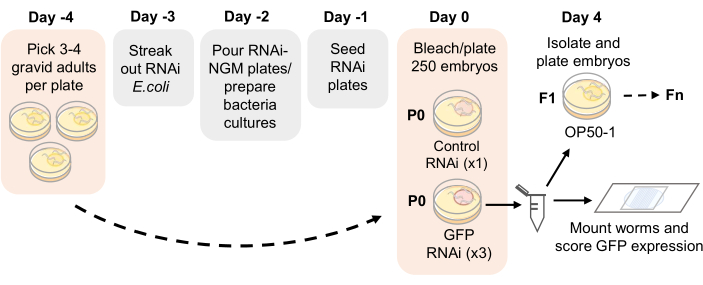
Abbildung 1: Schematische Darstellung des RNAi-Vererbungsassays. Vorgeschlagener Zeitplan für die RNAi-NGM-Plattenvorbereitung und den Aufbau des RNAi-Vererbungsassays. Trächtige adulte Tiere werden am Tag -4 auf NGM-Platten gepflückt, die mit OP50-1 besät sind. Nach 4 Tagen werden die erwachsenen Nachkommen gebleicht und die Embryonen auf RNAi-NGM-Platten plattiert. Die P0-Generation wird 4 Tage lang bei 21 °C RNAi ausgesetzt. Sobald die Würmer das Erwachsenenalter erreicht haben, werden die Replikate durch Bleiche durchlaufen und in jeder Generation für die GFP-Expression der Keimbahn bewertet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Passage und Bewertung jeder Generation auf GFP-Expression in der Keimbahn
HINWEIS: Um das Scoring zu erleichtern, verwenden Sie eine Vinyl-Schallplatte, um Agarose-Pads mit linearen Rillen zu erstellen, um die Würmer auszurichten. Diese Methode wurde von Rivera Gomez und Schvarzstein19 übernommen.
- Man bereitet 1 % (w/v) Agarose vor, indem man Agarose inddH2Oin einem kleinen Kolben durch Erhitzen in der Mikrowelle auflöst. Einen Rührstab hinzufügen und mit Folie abdecken. Beim Umschmelzen die Agarose auf einer Heizplatte bei 200 °C unter Rühren erhitzen. Nach dem Schmelzen die Hitze auf 80 °C reduzieren.
- Waschen Sie die Schnecken von den NGM-Platten mit 800 μl M9-Puffer mit TX-100 in ein 1,5-ml-Röhrchen. Waschen Sie die Teller zweimal, um alle trächtigen Erwachsenen zu entfernen. Überprüfen Sie die Platten unter dem Stereoskop, um sicherzustellen, dass die Tiere effizient gesammelt wurden.
- Zentrifugieren Sie die Röhrchen bei 1.000 x g für 1,5-2 Minuten oder lassen Sie die Würmer 2 Minuten lang ruhen. Saugen Sie den Überstand an und lassen Sie 100 μl stehen, ohne das Wurm-"Pellet" zu stören.
- Agarose-Pads für die Montage der Würmer wie folgt vorbereiten.
- Geben Sie einen Tropfen geschmolzener 1%iger Agarose (w/v) mit einer Pasteurpipette aus Glas (wobei das schmale Ende abgebrochen ist) auf die Schallplatte (siehe19).
- Richten Sie einen Objektträger so aus, dass die Linien auf der Platte entweder horizontal oder vertikal verlaufen, und legen Sie den Objektträger schnell auf den Agarosetropfen. Warten Sie ca. 30 Sekunden, bevor Sie den Objektträger aus der Schallplatte entfernen.
HINWEIS: Wenn Sie mehrere Genotypen bewerten, kann es nützlich sein, ein größeres Agarose-Pad auf einem Objektträger herzustellen und es mit einer Klinge in zwei Hälften zu schneiden.
- Montieren Sie die Würmer auf den Agarose-Pads.
- Geben Sie unter dem Stereoskop etwa 5 μl 5 mM Levamisol in das Agarose-Pad. Die Levamisol-Verdünnung sollte jede Woche frisch gemacht werden.
- Schnippen Sie vorsichtig die Röhrchen mit den Würmern, um sie zu mischen, und übertragen Sie 5-10 μl Würmer auf das Agarose-Pad. Schätzen Sie die Anzahl der Würmer mit Augenmaß, wenn sie hinzugefügt werden, so dass es ungefähr 40 Würmer pro Replikat gibt.
- Richte die Würmer mit einem Wimpernpickel in Reihen aus, um das Scoring zu erleichtern. Fügen Sie ein Deckglas hinzu.
HINWEIS: Dias mit Tieren, die auf diese Weise montiert sind, können mehrere Stunden halten, bevor sie trocknen.
- Isolieren Sie die Embryonen von den übrigen Tieren anhand des Bleichprotokolls (siehe Schritt 2.3), bevor Sie die Objektträger ritzen. Platte 250 Embryonen auf 35 mm NGM-Platten, die mit OP50-120 ausgesät sind. Sobald die Flüssigkeit aufgesogen ist, drehen Sie die Platten um und legen Sie sie zurück in den 21 °C warmen Inkubator.
- Verwenden Sie ein Fluoreszenzstereoskop mit einem GFP-Filterset. Zählen und notieren Sie die Anzahl der GFP-positiven und GFP-negativen Würmer auf den Objektträgern für jede Wiederholung mit Hilfe eines Zählzählers.
HINWEIS: GFP-positive Würmer haben eine nukleäre GFP-Expression in ihrer Keimbahn und ihren Embryonen in utero. Die Keimbahnen von GFP-negativen Würmern fluoreszieren nicht, aber wenn das GFP in späteren Generationen wieder eingeschaltet wird, kann die Fluoreszenz schwach sein. Es kann hilfreich sein, zusätzliche nicht-transgene Wurmstämme zu montieren, um die beobachtete Autofluoreszenz zu berücksichtigen. - Passage der Population durch Bleichen, wie oben beschrieben, etwa alle 4 Tage (96 h) bei 21 °C. Alternativ kann die Passage der Einfachheit halber nach einem abwechselnden 3-Tage-/4-Tage-Zeitplan durchgeführt werden. Teller mit zusätzlichen ~50 Embryonen für die kürzeren Populationen, wenn sie sich abwechseln, da es nach 72 Stunden zu einer geringeren Embryonenausbeute kommen kann.
HINWEIS: Halten Sie die Durchlaufzeit der P0-Generation konstant bei 4 Tagen nach der Beschichtung der Embryonen.
4. Tiersammlung für ChIP
HINWEIS: Die Anzahl der Tiere und der Zeitpunkt hängen vom Stamm, dem Entwicklungsstadium, dem Epitop und der Anzahl der Immunpräzipitationsziele (IP) ab. Sammeln Sie im folgenden Beispiel Tiere für drei IP-Adressen: H3K9me3, Histon H3 und IgG-Kontrolle. Das ChIP-Protokoll wurde von Askjaer et al.21 adaptiert.
- Erweitern Sie vor der ChIP-Probenentnahme die vorherige Generation des RNAi-Vererbungsassays um weitere drei NGM-Platten (auf mindestens vier Platten pro Replikation). 4 Tage lang bei 21 °C wachsen.
- Bleichen Sie die trächtigen Erwachsenen, um die Embryonen zu isolieren (siehe Schritt 2.3).
- Für jeden Stamm werden etwa 3.500 Embryonen auf 14 Platten (250 pro Platte) aufgetragen und 3 Tage lang bei 21 °C bis zum jungen Erwachsenenstadium heranwachsen.
- Waschen Sie die Tiere in 1,5-ml-Röhrchen mit phosphatgepufferter Kochsalzlösung (PBS), die 0,01 % (v/v) TX-100 (PBS/TX) enthält. Bei 1.000 x g für 2 min zentrifugieren. Die Tiere von einem Stamm in ein Röhrchen absaugen und zusammenlegen und drei weitere Male mit mindestens 1 ml PBS/TX waschen.
- Auf 1.000 μl absaugen, zum Mischen invertieren und die Anzahl der Tiere in 3 μl dreimal zählen (siehe Schritt 2.3.9).
- Berechnen Sie die Konzentration der Tiere und übertragen Sie ein Volumen, das 3.000 Tieren entspricht, zur Formaldehydvernetzung in ein neues Röhrchen.
- Achten Sie darauf, dass das Gesamtvolumen mindestens das 10-fache des Wurmpelletvolumens beträgt.
5. Formaldehyd-Vernetzung
VORSICHT: Arbeiten Sie mit Formaldehyd in einem Abzug, um eine Dampfexposition zu vermeiden.
- Formaldehyd (1,8 % Endkonzentration) in das Röhrchen mit den Würmern geben. Bei Raumtemperatur 6 Min. drehen.
- Sofort in flüssigem Stickstoff einfrieren. Das Experiment kann in diesem Schritt pausiert und die Proben bei -80 °C gelagert werden.
- Die vernetzte Probe wird in einem Wasserbad bei Raumtemperatur 3 min aufgetaut und 16 min bei Raumtemperatur gedreht.
- 1,25 M Glycin bis zu einer Endkonzentration von 125 mM zugeben und 5 min drehen.
HINWEIS: Bewahren Sie die Proben bis zur magnetischen Bead-Elution auf Eis oder bei 4 °C auf und verwenden Sie eiskalte Puffer. - Die Probe wird bei 1.000 x g für 3 min zentrifugiert. Dreimal mit jeweils 1 ml PBS/TX waschen. Zweimal mit 1 ml Resuspensionspuffer (150 mM NaCl, 50 mM HEPES-KOH [PH 7,5], 1 mM Ethylendiamintetraessigsäure [EDTA], 0,01 % TX-100, Proteaseinhibitor [eine Tablette pro 5 ml]) waschen.
- Lassen Sie nach der letzten Wäsche genügend Puffer, um sicherzustellen, dass das Gesamtvolumen mindestens das Dreifache des Pelletvolumens und mindestens ~100 μl beträgt.
6. Beschallung
HINWEIS: Die Beschallungsparameter hängen von der Art und dem Modell des Beschallungsgeräts und der Tierstufe ab. Parameter wie Probenvolumen und -konzentration, Ein-/Ausschaltintervalle, Anzahl der Zyklen und Leistungseinstellung müssen empirisch optimiert werden. Überwachen Sie beispielsweise die Wurmlyse über einen bestimmten Zeitraum mit einem Proteinassay und bestimmen Sie, wann die Konzentration ein Plateau erreicht. Wenn die durchschnittliche Schergröße der genomischen DNA etwa 200-1.000 bp beträgt, ist außerdem durch Elektrophorese der DNA, die nach der Umkehrung der Vernetzung gereinigt wurde, auf einem 1,5%igen Agarose/Trisacetat-EDTA (TAE)-Gel zu überwachen.
- Messen Sie das Volumen der Proben aus dem vorherigen Schritt. Mischen und aliquotieren Sie 90-120 μl in ein Styropor-Beschallungsröhrchen.
- Geben Sie ein gleiches Volumen Resuspensionspuffer mit 2x Detergenzien (150 mM NaCl, 50 mM HEPES-KOH [PH 7,5], 1 mM EDTA, 0,2 % Natriumdesoxycholat, 0,7 % Sarkosyl) hinzu.
- Beschallung in einem Wasserbad-Ultraschallgerät bei 50 % Leistung für 7 Minuten (20 s an/40 s aus) bei 4 °C. Durch Pipettieren vorsichtig mischen. Wiederholen Sie die Beschallung für weitere 7 Minuten.
- Überführen Sie das beschallte Lysat in ein 1,5-ml-Röhrchen. Geben Sie 0,5 Volumina Resuspensionspuffer ohne Detergenzien (150 mM NaCl, 50 mM HEPES-KOH [PH 7,5], 1 mM EDTA) hinzu (z. B. 100 μl Puffer zu 200 μl Probe hinzufügen).
- Bei 13.000 x g 15 min bei 4 °C zentrifugieren. Behalten Sie den Lysatüberstand und geben Sie ihn in ein neues Röhrchen um.
- Führen Sie optional einen Proteinassay durch, um die Konzentrationen jedes Lysats zu bestimmen. Dieser Schritt kann für große Probenmengen oder Vergleiche zwischen den Assays nützlich sein. Die Standardisierung der Anzahl der Tiere, wie oben beschrieben, funktioniert jedoch gut für die hier beschriebenen Probenmengen, da eine bessere Korrelation mit der Ausbeute an Chromatin/DNA besteht, wie sie durch qPCR bestimmt wird.
7. Immunpräzipitation
HINWEIS: Skalieren Sie die Menge der Antikörper und magnetischen Kügelchen auf das Volumen und die Konzentration des Lysats.
- Teilen Sie den Lysatüberstand in vier Portionen auf: drei gleiche Volumina für jede IP und 10 % des Volumens einer IP als Eingang (z. B. 100 μl IP #1, 100 μl IP #2, 100 μl IP #3, 10 μl Eingangs). Das IP-Lysat wird in ein 200-μl-PCR-Röhrchen überführt und bei -20 °C in einem 1,5-ml-Röhrchen gelagert.
- Geben Sie 0,5 μg Anti-H3K9me3-Antikörper, Anti-Histon-H3-Antikörper oder IgG in die entsprechende IP-Probe. Bei 4 °C über Nacht mit Rotation inkubieren.
- Am nächsten Tag werden 9 μl Protein-G-beschichtete Magnetkügelchen pro IP in ein einzelnes 1,5-ml-Röhrchen aliquotiert. Waschen Sie die Kügelchen zweimal mit 1 ml FA-150 (150 mM NaCl, 50 mM HEPES-KOH [PH 7,5], 1 mM EDTA, 1 % TX-100, 0,1 % Natriumdesoxycholat).
- Resuspendieren Sie die magnetischen Kügelchen im FA-150-Puffer auf das ursprüngliche Volumen, das in Schritt 7.3 oben aus dem Material entnommen wurde. Fügen Sie 7,5 μl zu jeder IP hinzu. Bei 4 °C 2 h mit Rotation inkubieren.
8. Waschen und Elution
HINWEIS: Um sicherzustellen, dass die magnetischen Perlen nicht austrocknen, fügen Sie jede Wäsche oder jeden Elutionspuffer schnell nach dem Absaugen der vorherigen Wäsche hinzu.
- Verwenden Sie jedes Mal 0,2-1 ml der folgenden Pufferlösungen, um die Perlen zu waschen. Sammeln Sie die Perlen auf einem magnetischen Ständer, um die Wäsche abzusaugen. Jede Wäsche bei 4 °C für 5 Minuten mit Rotation inkubieren. Befolgen Sie die folgende Reihenfolge.
- Zweimal mit FA-150 waschen.
- Einmal mit FA-1M (FA-150 mit 1 M NaCl) waschen.
- Einmal mit FA-0,5M (FA-150 mit 0,5 M NaCl) waschen. In ein neues PCR-Röhrchen umfüllen.
- Einmal mit TE-LiCl waschen (250 mM LiCl, 10 mM Tris-Cl [pH 8,0], 1 mM EDTA, 1% IGEPAL CA-630, 1% Natriumdesoxycholat).
- Zweimal mit TE+ (50 mM NaCl, 10 mM Tris-Cl [pH 8,0], 1 mM EDTA, 0,005 % IGEPAL CA-630) waschen.
HINWEIS: Setzen Sie die Probenverarbeitung bei Raumtemperatur fort, sofern nicht anders angegeben.
- Saugen Sie den letzten Waschpuffer an. Die magnetischen Kügelchen werden mit 50 μl ChIP-Elutionspuffer (200 mM NaCl, 10 mM Tris-Cl [pH 8,0], 1 mM EDTA, 1 % Natriumdodecylsulfat [SDS]) resuspendiert und in ein 1,5-ml-Röhrchen überführt.
- Bei 65 °C für 15 min in einem Thermomischer mit Mischen bei 1.000 U/min für 5 s pro Minute eluieren.
- Sammeln Sie die Perlen auf einem magnetischen Ständer und geben Sie den Überstand in ein neues Röhrchen.
- Wiederholen Sie die Elution mit weiteren 50 μl ChIP-Elutionspuffer. Fassen Sie die Überstände für insgesamt 100 μl zusammen. Fahren Sie mit der umgekehrten Vernetzung und DNA-Elution fort, wie unten beschrieben.
9. Reverse Crosslinking und DNA-Elution
- Tauen Sie die Eingangslysatprobe auf. Bis zu 100 μl mit ChIP-Elutionspuffer auffüllen.
- Fügen Sie 16,5 μg RNase A zu jeder IP- und Eingangsprobe hinzu. 1 Stunde bei 37 °C inkubieren.
- 40 μg Proteinase K zugeben und bei 55 °C für 2 h inkubieren. Anschließend über Nacht bei 65 °C inkubieren.
- Kühlen Sie die Proben auf Raumtemperatur ab und reinigen Sie die DNA mit einem Spin-Säulen-Kit.
10. Aufbau und Ablauf der qPCR-Reaktion
HINWEIS: Die Parameter des Primers, des Reaktionsaufbaus und des Thermocyclers sollten so geändert werden, dass sie den Empfehlungen des Herstellers für die verwendete qPCR-Reaktionsmischung entsprechen.
- Bereiten Sie Primer-Sets vor, die auf den GFP-RNAi-Reporter und die positiven und negativen Anreicherungskontrollregionen von H3K9me3 abzielen. Die Schmelztemperatur der Grundierung beträgt 60 °C. Siehe Materialtabelle für Primer-Sequenzen.
- Für die Eingangs-DNA werden vier vierfache serielle Verdünnungen vorgenommen (z. B. 1:5, 1:20, 1:80, 1:320).
ANMERKUNG: Die IP-DNA kann direkt oder verdünnt (z. B. 1:2 oder 1:3) verwendet werden, um mehr Reaktionen zu ermöglichen. Die Eignung der Verdünnung muss empirisch bestimmt werden, da die qPCR-Leistung beeinträchtigt werden kann. - Organisieren Sie alle Reaktionen, die einem Primer-Set entsprechen, auf einer PCR-Platte. Richten Sie technische Duplikatreaktionen für jede Eingangs-DNA-Verdünnung und jede IP-DNA-Probe ein. Jede 10-μl-Reaktion enthält 1,5 μl Eingangs- oder IP-DNA (oder Elutionspuffer als Kontrolle), qPCR-Mastermix (1-fache Endkonzentration), Vorwärtsprimer und Reverse-Primer (400 nM Endkonzentration für jeden Primer).
- Führen Sie auf einem Echtzeit-Thermocycler das folgende Programm aus: anfängliche Denaturierung: 95 °C für 4 min; Amplifikation und Fluoreszenzdetektion: 40 Zyklen von 95 °C für 10 s und 60 °C für 30 s mit einem Plate-Read; Endausdehnung: 60 °C für 5 min; Schmelzekurve: von 60 °C bis 90 °C in Schritten von 0,5 °C, 5 s pro Schritt.
11. Bestimmung der Amplifikationseffizienz und Überprüfung der Produktspezifität
- Zeichnen Sie für jeden Satz von Eingangs-DNA-Verdünnungen die vier Datenpunkte mit [log10(1/dilution)] auf der x-Achse und [Eingabe-Cq] auf der y-Achse auf. Bestimmen Sie die Steigung der Linie mit der besten Anpassung.
- Berechnen Sie die Verstärkungseffizienz. Ideale Primer-Sets sollten konstant einen Wirkungsgrad von 95 % bis 100 % aufweisen.
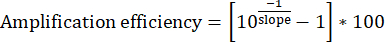
- Stellen Sie sicher, dass alle Reaktionen eine scharfe Schmelzkurvenspitze aufweisen und dass Reaktionen mit demselben Primersatz die gleiche Schmelztemperatur haben. Mehrere Peaks oder unterschiedliche Schmelztemperaturen können auf eine unspezifische Verstärkung hinweisen.
- Optional können Reaktionen auf einem Standard-Gel mit 2 % Agarose/TAE bei Raumtemperatur durchgeführt werden, um die Bandengröße des Produkts zu überprüfen.
12. Berechnung des Prozentsatzes des Inputs
- Berechnen Sie den Eingangsverdünnungsfaktor. Da 10 % des IP-Lysatvolumens als Input gespeichert wurden, beträgt der Verdünnungsfaktor für die 1:5-Verdünnung der Input-DNA 50.
- Bestimmen Sie den IP-Verdünnungsfaktor. Wenn die IP-DNA vor der qPCR nicht verdünnt wird, beträgt der Verdünnungsfaktor 1.
- Berechnen Sie die Cq-Differenz zwischen IP und Input, bereinigt um Verdünnungsfaktoren.
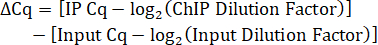
- Berechnen Sie den Prozentsatz der Eingabe.
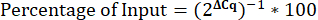
Repräsentative Ergebnisse
Tiere, die den Keimbahn-exprimierten GFP-Histon H2B [mex-5p::gfp-h2b::tbb-2 3'UTR]7 Reporter (Abbildung 2A) trugen, wurden GFP-RNAi oder Kontroll-RNAi durch Fütterung ausgesetzt und wie im Protokoll und in Abbildung 1 beschrieben durchgelassen. Das nukleäre GFP-Signal in der Keimbahn wurde manuell mit einem Fluoreszenz-Präpariermikroskop für eine Probe der Population zu jeder Generation bewertet. Die Stilllegung des Transgens war bei den bewerteten P0- und F1-Tieren, die mit GFP-RNAi behandelt wurden, vollständig penetrant (Abbildung 2B). In der F2-Generation lag der Anteil der Bevölkerung, der eine Vererbung des GFP-Silencings aufwies, bei etwa 50%. In der F5-Generation zeigte die Mehrheit der Population keine Vererbung des Silencings, und in der F10-Generation wurde keine Vererbung festgestellt, da alle Tiere GFP exprimierten.
Um die Veränderung der Histon-H3K9me3-Anreicherung zu bestimmen, die dem RNAi-induzierten Silencing entspricht, wurde eine ChIP-qPCR an Tieren der F1-Generation nach einer GFP-RNAi- oder einer Kontroll-RNAi-Behandlung durchgeführt. Wie erwartet, wies die GFP-RNAi-behandelte Population im Vergleich zu den mit RNAi behandelten Kontrolltieren höhere Histon-H3K9me3-Spiegel am GFP-Ziel und in der 1,3 kb stromabwärts gelegenen Region auf (Abbildung 2C). Die Spezifität des Histons H3K9me3 ChIP wird durch die Anreicherung an einem Positivkontrollort (clec-18) unterstützt, von dem bekannt ist, dass er in dieser Markierung angereichert ist, nicht jedoch an einem nahe gelegenen negativen Kontrollort (hrp-2). Die Histon-H3-Anreicherung und die Hintergrundanreicherung in den IgG-Kontroll-Immunpräzipitationen wurden erwartungsgemäß ebenfalls an allen qPCR-Loci nachgewiesen. Wenn die ChIP-Anreicherung am Reporter auf den clec-18-positiven Kontrolllocus normalisiert wird, zeigt sich eine höhere Histon-H3K9me3-Anreicherung auf GFP-RNAi, während die Histon-H3-Anreicherung zwischen der Kontroll- und der GFP-RNAi-Behandlung ähnlich ist (Abbildung 2D). Da nicht zu erwarten ist, dass GFP-RNAi das Histon H3K9me3 oder die Gesamtbelegung von Histon H3 am clec-18-Locus beeinflusst, mildert diese Normalisierung technische Schwankungen ab, wie z. B. Unterschiede in der ChIP-Effizienz zwischen den GFP-RNAi- und Kontroll-RNAi-Proben. Die Faltungsänderung der Histon-H3K9me3- und Histon-H3-Spiegel zwischen RNAi-Behandlungen zeigt eine GFP-Reporter-spezifische Histon-H3K9me3-Anreicherung, unabhängig von der Histonbelegung, bei GFP-RNAi-induzierter Stummschaltung (Abbildung 2E).
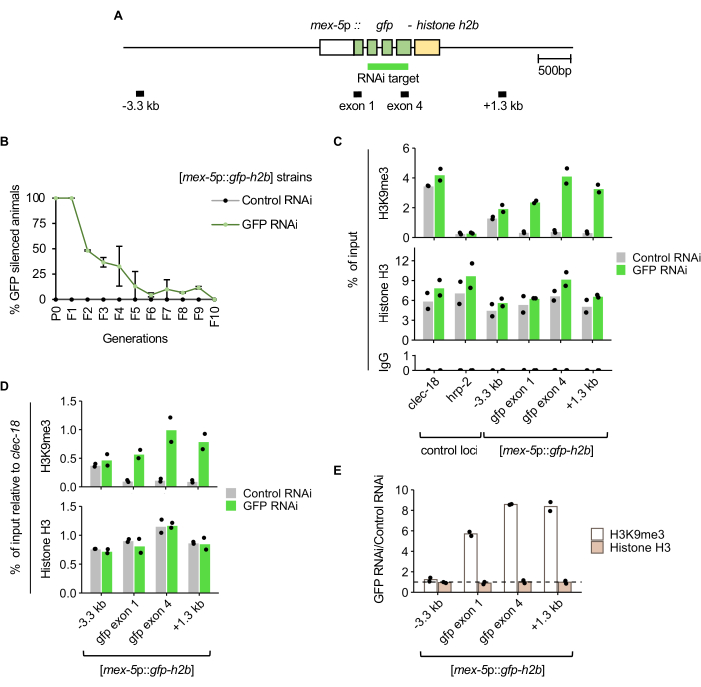
Abbildung 2: GFP-RNAi-induziertes Silencing entspricht einer erhöhten H3K9me3-Anreicherung am RNAi-Ziel. (A) Diagramm des Keimbahn-exprimierten GFP-RNAi-Reporters mjIs134[mex-5p::gfp-h2b::tbb-2 3'UTR] mit markierten qPCR-Amplikonregionen. (B) Die GFP-Expression wurde über Generationen hinweg nach RNAi-Behandlungen bei 21 °C bewertet. Fehlerbalken stellen die Standardabweichung von zwei biologischen Replikaten dar. (C) ChIP-qPCR von H3K9me3, Histon H3 und IgG-Kontrolle bei jungen F1-Erwachsenen aus zwei biologischen Replikaten. clec-18 und HRP-2 sind die positiven bzw. negativen Kontrollloci für die H3K9me3-Anreicherung. (D) Die Anreicherung von H3K9me3 und Histon H3 am GFP-RNAi-Reporter normalisierte sich auf den clec-18-Positivkontrolllocus. (E) Veränderung der H3K9me3- und Histon-H3-Anreicherung zwischen GFP-RNAi- und Kontroll-RNAi-behandelten Tieren mit Normalisierung auf clec-18. Die gestrichelte Linie stellt eine Falzänderung von 1 dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
In diesem Protokoll wird dsRNA durch Fütterung eingeführt, was bei C. elegans zu einer Standardmethode geworden ist 18. Für RNAi-Vererbungsassays bietet der Fütterungsansatz eine einfache Methode, um eine große P0-Population 2,11,12,22,23,24,25 zu erhalten. Der Zeitpunkt und die Dauer der RNAi-Exposition beeinflussen jedoch die Wirksamkeit des Transgen-Silencings26, und die Konzentration von RNAi-Bakterien beeinflusst die Persistenz des vererbbaren RNAi-Silencings1. Daher sind standardisierte RNAi-Bakterien und Wurmwachstum wichtig, um ein konsistentes Maß an GFP-Silencing und Vererbungsdauer zu erreichen. Dabei werden Embryonen auf RNAi-Platten plattiert, so dass P0-Tiere vom Schlüpfen an RNAi-Bakterien ausgesetzt sind. Bei alternativen Ansätzen wurden synchronisierte Tiere im L1 24,27- oderL4 25-Stadium auf RNAi-NGM-Platten plattiert. Da Hunger und andere Belastungen die Aufrechterhaltung der RNAi-Vererbung beeinträchtigen13, müssen die Platten überwacht werden, um eine Überfüllung und Erschöpfung des Nahrungsangebots zu verhindern. Als Alternative zur Initiierung von RNAi durch Fütterung induzierten einige der bahnbrechenden RNAi-Vererbungsstudien von C. elegans RNAi durch Gonadeninjektion, was eine bessere Kontrolle der dsRNA-Konzentration ermöglicht 1,28.
In diesem Protokoll wird jede Generation als Population mit alkalischer Hypochloritbehandlung durchlaufen, wie zuvor beschrieben 16,22,23,24. Die Hypochloritbehandlung stellt sicher, dass die F1-Generation nicht mit RNAi-Bakterien aus der elterlichen Umgebung kontaminiert wird22 und verhindert mögliche unerwünschte Engpässe in der Population. Die Massenverbringung kann jedoch auch eine Einschränkung darstellen, da einzelne Tiere unterschiedliche Vererbungsmuster aufweisen können 1,29. Eine alternative Methode, um jede Generation zu bestimmen, ist die Auswahl einzelner Tiere 1,2,9,11,25. Dieser Ansatz ermöglicht es, Phänotypen innerhalb von Abstammungslinien zu verfolgen und genetische Kreuzungen einzubauen. Die Abstammungsanalyse kann auch bei Phänotypen mit geringer Penetranz von Vorteil sein12.
Die Expression fluoreszierender Proteine ist ein leistungsfähiges und bequemes Auslesen des RNAi-induzierten Silencings 2,3,4,12,14,15,16,25,30. Wie in diesem Protokoll beschrieben, kann die GFP-Expression manuell als EIN oder AUS 11,12,15,16,25 bewertet werden. Das manuelle Scoring kann durch die Zuweisung der qualitativen Intensitätsstufen 3,4,14 weiter verfeinert werden. Alternativ kann die automatisierte Intensitätsbewertung von Mikroskopiebildern 11,12,14,25,27 oder die Durchflusszytometrie-Fluoreszenzmessung von lebenden Tieren 2 eine quantitative und Hochdurchsatz-Auslesung liefern. Da die Reporterexpression jedoch auf die Keimbahn beschränkt ist, besteht ein Vorbehalt automatisierter Ansätze darin, dass sie auch zwischen Tieren mit stillgelegter GFP-Expression und Tieren ohne Keimbahn unterscheiden müssen, insbesondere wenn die Studie Mutanten mit abnormaler Keimbahnentwicklung umfasst. Als Alternative zur GFP-Fluoreszenz kann die reverse Transkription (RT)-qPCR verwendet werden, um die RNAi-Ziel-Prä-mRNA- und mRNA-Spiegel 2,16,23,24 zu quantifizieren. Dieser Ansatz ermöglicht ein direkteres Auslesen des Silencings, das auf RNA abzielt, und ist besonders nützlich für andere RNAi-Ziele, bei denen das Silencing keinen sichtbaren Phänotyp erzeugt. Eine Einschränkung bei der Verwendung künstlicher GFP-Reporter besteht darin, dass exogene und endogene Sequenzen in transgenerationaler RNAi11 unterschiedlich reguliert werden. Studien mit endogenen Targets, wie z. B. dem temperaturempfindlichen embryonalen letalen Allel oma-1(zu405)1,11,16,25, sollten daher als komplementärer Ansatz zu fluoreszierenden transgenen Reportern in Betracht gezogen werden.
Die Analyse von ChIP im Rahmen von RNAi-Vererbungsassays erfordert einen Vergleich zwischen Behandlungen und Replikaten. Erstens, um Unterschiede im Ausgangsmaterial zwischen den Proben zu berücksichtigen, wird das ChIP-Signal auf das Eingangssignal an der gleichen Stelle wie der "Prozentsatz des Eingangs" normalisiert. Die parallele Verarbeitung eines Histon-H3-ChIP hilft zu bestimmen, ob Veränderungen der Histonmodifikation mit Änderungen der Nukleosomendichte korrespondieren. Da es sich bei ChIP um einen mehrstufigen Prozess handelt, kann die Effizienz zwischen den Proben variieren. Die Auswahl geeigneter positiver und negativer Kontrollloci ist nützlich, um das Signal-Rausch-Verhältnis zwischen Proben und Experimenten zu bewerten und zu vergleichen. Um Vergleiche zwischen Proben zu erleichtern, werden die ChIP-DNA-qPCR-Schwellenwerte am RNAi-Ziel häufig auf einen Kontrollort 3,11,15,23,30,31 normiert. Um die Auswirkungen der RNAi-Behandlung zu bewerten, wird auch das Verhältnis des ChIP-Signals bei der Behandlung mit RNAi im Vergleich zu Kontroll- oder Nicht-RNAi-Bedingungen verglichen. Eine Einschränkung des derzeitigen Ansatzes besteht darin, dass ChIP mit ganzen Tieren durchgeführt wird, während das Ansprechen auf die RNAi-Behandlung möglicherweise nur in der Keimbahn erfolgt. Ein Ansatz, um diesen Vorbehalt zu überwinden, besteht darin, ChIP mit isolierten Keimbahnkernen durchzuführen. Weitere technische Überlegungen zur Optimierung von ChIP wurden auch an anderer Stelle ausführlich diskutiert32,33.
Insgesamt bietet diese RNAi-Vererbung und das ChIP-Protokoll eine detaillierte und leicht anzupassende Grundlage, die mit anderen Techniken integriert werden kann, um die transgenerationale epigenetische Regulation weiter zu erforschen. Zum Beispiel können Hochdurchsatz-Sequenzierungsbibliotheken aus der ChIP-DNA (ChIP-seq) konstruiert werden, um eine detailliertere Sicht auf die Chromatinlandschaft sowohl proximal des RNAi-Ziels als auch auf genomweiter Ebene zu erhalten.
Offenlegungen
Alle Autoren bestätigen, dass sie keine Konflikte offenzulegen haben.
Danksagungen
Wir möchten uns bei den Laboren in der C. elegans-Gemeinschaft bedanken, die die Werkzeuge entwickelt und geteilt haben und deren Arbeit in diesem Manuskript zitiert wird. Einige Stämme wurden vom CGC zur Verfügung gestellt, das vom NIH Office of Research Infrastructure Programs (P40 OD010440) finanziert wird. Diese Arbeit wurde durch ein Projektstipendium des Canadian Institutes of Health Research (CIHR) an A.L.S. (PJT-175245) unterstützt. C.L. wird durch ein Postgraduiertenstipendium des Natural Sciences and Engineering Research Council of Canada (NSERC) (PGS-D) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Agarose | Bioshop | AGA002 | |
| Ampicillin | Bioshop | AMP201 | Make a 100 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Anti-H3K9me3 Rabbit Polyclonal Antibody | Abcam | ab8898 | Concentration is batch-dependent (0.9 - 1 mg/mL). |
| Anti-Histone H3 Rabbit Polyclonal Antibody | Abcam | ab1791 | Concentration is batch-dependent (0.7 - 1 mg/mL). |
| Bleach (6% Sodium hypochlorite) | Lavo | 02358107 | |
| C. elegans strain with GFP RNAi Reporter | NA | SX1263 | Sapetschnig et al. 2015 (ref. 7). A gift from E. Miska lab, University of Cambridge. |
| Carbenicillin | BioShop | CAR544 | Make a 25 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Dynabeads Protein G Magnetic Beads | Invitrogen | 10003D | |
| E. coli strain HT115(DE3) | Caenorhabditis Genetics Center (CGC) | HT115(DE3) | |
| E. coli strain OP50-1 | Caenorhabditis Genetics Center (CGC) | OP50-1 | |
| EDTA (0.5 M, pH 8.0) | Invitrogen | 15575020 | |
| Fluorescence Stereoscope | Zeiss | Axio Zoom.V16 | |
| Formaldehyde (37%) | Sigma | F8775 | |
| Glycine | Sigma | 50046 | Make a 1.25 M solution and store at 4 °C. |
| HEPES-KOH (1 M, pH 7.5) | Teknova | H1035 | |
| Hydrophobic Printed Slides, 10 wells | VWR | 100488-904 | |
| IGEPAL CA-630 (Octylphenol ethoxylate) | BioShop | NON999 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
| IPTG (Isopropyl-β-D-thiogalactoside) | BioShop | IPT001 | Make a 0.2 g/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725122 | |
| LB Agar Plates supplemented with 100 µg/mL Ampicillin | NA | NA | Standard lab recipe. |
| Levamisole (Tetramisole hydrochloride) | Sigma | L9756 | Make a 200 mM solution in ultrapure water. Store at -20 °C. |
| LiCl (8 M) | Sigma | L7026 | |
| M9 Buffer | NA | NA | 22 mM KH2PO4, 42 mM Na2HPO4, 86 mM NaCl, 1 mM MgSO4. |
| Magnetic Separator (1.5 mL tubes) | Applied Biosystems | A13346 | |
| Magnetic Separator (0.2 mL tubes) | Permagen | MSR812 | |
| Microscope Cover Glass | Fisher Scientific | 12541B | |
| Microscope Slide | Technologist Choice | LAB-037 | |
| NaCl (5 M) | Promega | V4221 | For ChIP buffers. |
| NaOH | Sigma | S5881 | Make a 10 M solution and store at room temperature. |
| NGM Plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 μg/mL cholesterol, 25 mM Potassium phosphate pH 6.0, 1 mM MgSO4, 50 µg/mL streptomycin. |
| Normal Rabbit IgG (1 mg/mL) | Cell Signaling Technology | 2729 | |
| Petri Dishes (35 mm x 10 mm) | Sarstedt | 82.1135.500 | |
| Phosphate Buffered Saline (10X) | Fisher BioReagents | BP3991 | |
| Plasmid - Control RNAi | Addgene | L4440 (Plasmid #1654) | |
| Plasmid - GFP-targetting RNAi | Addgene | L4417 (Plasmid #1649) | Note, alternative L4440-derived plasmids targeting GFP can be used. |
| Primer pair [-3.3 kb upstream of gfp] | Integrated DNA Technologies | NA | F: AAACCAAAGGACGAGAGATTCA, R: GGCTCGATCAAGTAAAATTTCG |
| Primer pair [+1.3 kb downstream of gfp] | Integrated DNA Technologies | NA | F: TCGACCAGTTCTAAAGTCACCG, R: ACGTGCGGGATCATTTCTTACT |
| Primer pair [clec-18] | Integrated DNA Technologies | NA | F: TGCTCCATGACCTCAACAACA, R: AGTACAGTTCACCGATCCAGA |
| Primer pair [gfp exon 1] | Integrated DNA Technologies | NA | F: CTGGAGTTGTCCCAATTCTTGT, R: GGGTAAGTTTTCCGTATGTTGC |
| Primer pair [gfp exon 4] | Integrated DNA Technologies | NA | F: GATGGCCCTGTCCTTTTACCA, R: ATGCCATGTGTAATCCCAGCA |
| Primer pair [hrp-2] | Integrated DNA Technologies | NA | F: CGTCAACAGGGAGCAGCTG, R: CCTCCGAACTTTCTCTGTCCA |
| Protease Inhibitor Cocktail Tablet | Roche | 11836170001 | |
| Proteinase K | Bioline | BIO-37084 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Real-Time PCR Detection System | Bio-Rad | CFX96 | |
| RNAi-NGM plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 µg/mL cholesterol, 25 mM Potassium phosphate buffer pH 6.0, 1 mM MgSO4, 25 µg/mL carbenicillin and 5 mM IPTG. |
| RNase A | Sigma | R4642 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma | L5777 | Make a 10% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| SDS | Sigma | 74255 | Make a 10% (w/v) solution and store at room temperature. |
| Sodium deoxycholate | Sigma | 30970 | Make a 5% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| Sonication Tube | Evergreen | 214-3721-010 | |
| Sonication Tube Cap | Evergreen | 300-2911-020 | |
| Sonicator | Qsonica | Q800R3-110 | |
| Streptomycin sulfate | Bioshop | STP101 | Make a 50 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| TAE buffer (1X) | NA | NA | 40 mM Tris, 20 mM acetate, 1 mM EDTA |
| Tally counter clicker | Uline | H-7350 | |
| Tetracycline | Bioshop | TET701 | Make a 5 mg/mL solution in ethanol and store at -20 °C. |
| Thermomixer | Eppendorf | 05-400-205 | |
| Tris-HCl (1 M, pH 8.0) | Invitrogen | 15568025 | |
| Triton X-100 | Sigma | T8787 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
Referenzen
- Alcazar, R. M., Lin, R., Fire, A. Z. Transmission dynamics of heritable silencing induced by double-stranded RNA in Caenorhabditis elegans. Genetics. 180 (3), 1275-1288 (2008).
- Ashe, A., et al. piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell. 150 (1), 88-99 (2012).
- Buckley, B. A., et al. A nuclear Argonaute promotes multigenerational epigenetic inheritance and germline immortality. Nature. 489 (7416), 447-451 (2012).
- Vastenhouw, N. L., et al. Long-term gene silencing by RNAi. Nature. 442 (7105), 882 (2006).
- Lee, H. -. C., et al. C. elegans piRNAs mediate the genome-wide surveillance of germline transcripts. Cell. 150 (1), 78-87 (2012).
- Luteijn, M. J., et al. Extremely stable Piwi-induced gene silencing in Caenorhabditis elegans. The EMBO Journal. 31 (16), 3422-3430 (2012).
- Sapetschnig, A., Sarkies, P., Lehrbach, N. J., Miska, E. A. Tertiary siRNAs mediate paramutation in C. elegans. PLoS Genetics. 11 (3), e1005078 (2015).
- Shirayama, M., et al. piRNAs initiate an epigenetic memory of nonself RNA in the C. elegans germline. Cell. 150 (1), 65-77 (2012).
- Minkina, O., Hunter, C. P. Stable heritable germline silencing directs somatic silencing at an endogenous locus. Molecular Cell. 65 (4), 659-670 (2017).
- Seroussi, U., et al. Mechanisms of epigenetic regulation by C. elegans nuclear RNA interference pathways. Seminars in Cell & Developmental Biology. 127, 142-154 (2022).
- Lev, I., Gingold, H., Rechavi, O. H3K9me3 is required for inheritance of small RNAs that target a unique subset of newly evolved genes. eLife. 8, e40448 (2019).
- Woodhouse, R. M., et al. Chromatin modifiers SET-25 and SET-32 are required for establishment but not long-term maintenance of transgenerational epigenetic inheritance. Cell Reports. 25 (8), 2259-2272 (2018).
- Houri-Zeevi, L., Teichman, G., Gingold, H., Rechavi, O. Stress resets ancestral heritable small RNA responses. eLife. 10, e65797 (2021).
- Houri-Ze'evi, L., et al. A tunable mechanism determines the duration of the transgenerational small RNA inheritance in C. elegans. Cell. 165 (1), 88-99 (2016).
- Spracklin, G., et al. The RNAi inheritance machinery of Caenorhabditis elegans. Genetics. 206 (3), 1403-1416 (2017).
- Perales, R., et al. Transgenerational epigenetic inheritance is negatively regulated by the HERI-1 chromodomain protein. Genetics. 210 (4), 1287-1299 (2018).
- Shukla, A., Perales, R., Kennedy, S. piRNAs coordinate poly(UG) tailing to prevent aberrant and perpetual gene silencing. Current Biology. 31 (20), 4473-4485 (2021).
- Ahringer, J. Reverse GeneticsWormBook: the Online Review of C. elegans Biology. WormBook. , (2006).
- Rivera Gomez, K., Schvarzstein, M. Immobilization of nematodes for live imaging using an agarose pad produced with a Vinyl Record. microPublication Biology. 2018, (2018).
- Stiernagle, T. Maintenance of C. elegans. WormBook: The Online Review of C. Elegans Biology. , 1-11 (2006).
- Askjaer, P., Ercan, S., Meister, P. Modern techniques for the analysis of chromatin and nuclear organization in C. elegans. WormBook: the Online Review of C. elegans Biology. , 1-35 (2014).
- Gu, S. G., et al. Amplification of siRNA in Caenorhabditis elegans generates a transgenerational sequence-targeted histone H3 lysine 9 methylation footprint. Nature Genetics. 44 (2), 157-164 (2012).
- Kalinava, N., Ni, J. Z., Peterman, K., Chen, E., Gu, S. G. Decoupling the downstream effects of germline nuclear RNAi reveals that H3K9me3 is dispensable for heritable RNAi and the maintenance of endogenous siRNA-mediated transcriptional silencing in Caenorhabditis elegans. Epigenetics & Chromatin. 10, 6 (2017).
- Kalinava, N., et al. elegans heterochromatin factor SET-32 plays an essential role in transgenerational establishment of nuclear RNAi-mediated epigenetic silencing. Cell Reports. 25 (8), 2273-2284 (2018).
- Lev, I., et al. MET-2-dependent H3K9 methylation suppresses transgenerational small RNA inheritance. Current Biology. 27 (8), 1138-1147 (2017).
- Kamath, R. S., Martinez-Campos, M., Zipperlen, P., Fraser, A. G., Ahringer, J. Effectiveness of specific RNA-mediated interference through ingested double-stranded RNA in Caenorhabditis elegans. Genome Biology. 2 (1), (2001).
- Xu, F., et al. A cytoplasmic Argonaute protein promotes the inheritance of RNAi. Cell Reports. 23 (8), 2482-2494 (2018).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287 (5462), 2494-2497 (2000).
- Houri-Zeevi, L., Korem Kohanim, Y., Antonova, O., Rechavi, O. Three rules explain transgenerational small RNA inheritance in C. elegans. Cell. 182 (5), 1186-1197 (2020).
- Burton, N. O., Burkhart, K. B., Kennedy, S. Nuclear RNAi maintains heritable gene silencing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences. 108 (49), 19683-19688 (2011).
- Mao, H., et al. The Nrde pathway mediates small-RNA-directed histone H3 lysine 27 trimethylation in Caenorhabditis elegans. Current Biology. 25 (18), 2398-2403 (2015).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Mukhopadhyay, A., Deplancke, B., Walhout, A. J. M., Tissenbaum, H. A. Chromatin immunoprecipitation (ChIP) coupled to detection by quantitative real-time PCR to study transcription factor binding to DNA in Caenorhabditis elegans. Nature Protocols. 3 (4), 698-709 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten