Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Unfraktionierte Massenkultur des Mausskelettmuskels zur Rekapitulation der Nischen- und Stammzellruhe
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Skelettmuskulatur besteht aus mehreren Zelltypen, einschließlich residenter Stammzellen, von denen jede einen besonderen Beitrag zur Muskelhomöostase und -regeneration leistet. Hier werden die 2D-Kultur von Muskelstammzellen und die Muskelzellnische in einer ex vivo Umgebung beschrieben, die viele der physiologischen, in vivo und Umwelteigenschaften bewahrt.
Zusammenfassung
Der Skelettmuskel ist das größte Gewebe des Körpers und erfüllt mehrere Funktionen, von der Fortbewegung bis zur Kontrolle der Körpertemperatur. Seine Funktionalität und Genesung nach Verletzungen hängt von einer Vielzahl von Zelltypen und von molekularen Signalen zwischen den Kernmuskelzellen (Myofasern, Muskelstammzellen) und ihrer Nische ab. Die meisten Versuchsumgebungen bewahren diese komplexe physiologische Mikroumgebung nicht, und sie erlauben auch nicht die Ex-vivo-Untersuchung von Muskelstammzellen in Ruhezustand, einem Zellzustand, der für sie entscheidend ist. Hier wird ein Protokoll für die ex vivo Kultur von Muskelstammzellen mit zellulären Komponenten ihrer Nische skizziert. Durch den mechanischen und enzymatischen Abbau von Muskeln erhält man eine Mischung von Zelltypen, die in eine 2D-Kultur gegeben wird. Die Immunfärbung zeigt, dass innerhalb von 1 Woche mehrere Nischenzellen in Kultur neben Myofasern und vor allem Pax7-positiven Zellen vorhanden sind, die die Eigenschaften ruhender Muskelstammzellen aufweisen. Diese einzigartigen Eigenschaften machen dieses Protokoll zu einem leistungsstarken Werkzeug für die Zellamplifikation und die Erzeugung von ruhenden Stammzellen, die zur Beantwortung grundlegender und translationaler Fragen verwendet werden können.
Einleitung
Bewegung, Atmung, Stoffwechsel, Körperhaltung und Aufrechterhaltung der Körpertemperatur hängen alle von der Skelettmuskulatur ab, und Fehlfunktionen in der Skelettmuskulatur können daher schwächende Pathologien (d. h. Myopathien, Muskeldystrophien usw.) verursachen. 1. Aufgrund seiner wesentlichen Funktionen und seines Überflusses hat die Skelettmuskulatur die Aufmerksamkeit von Forschungslabors weltweit auf sich gezogen, die sich bemühen, die Schlüsselaspekte zu verstehen, die eine normale Muskelfunktion unterstützen und als therapeutische Ziele dienen können. Darüber hinaus ist die Skelettmuskulatur ein weit verbreitetes Modell zur Untersuchung der Regeneration und Stammzellfunktion, da sich ein gesunder Muskel nach einer vollständigen Verletzung und Degeneration vollständig selbst reparieren kann, hauptsächlich aufgrund seiner ansässigen Stammzellen2; Diese werden auch Satellitenzellen genannt und sind unter der Basallamina in der Peripherie der Muskelfasern lokalisiert3.
Die Kernzellen der adulten Skelettmuskulatur sind die Myofasern (lange synzytiale multinukleäre Zellen) und die Satellitenzellen (Stammzellen mit myogenem Potenzial, die ruhen, bis eine Verletzung sie aktiviert). Die letztgenannten Zellen sind die zentralen Zellen der Muskelregeneration, und dieser Prozess kann in ihrer Abwesenheit nicht stattfinden 4,5,6,7. In ihrer unmittelbaren Mikroumgebung gibt es mehrere Zelltypen und molekulare Faktoren, die ihnen Signale geben. Diese Nische etabliert sich allmählich im Laufe der Entwicklung und bis ins Erwachsenenalter8. Der adulte Muskel enthält mehrere Zelltypen (Endothelzellen, Perizyten, Makrophagen, fibroadipogene Vorläuferzellen (FAPs), regulatorische T-Zellen usw.) 9,10 und extrazelluläre Matrixkomponenten (Laminine, Kollagene, Fibronektin, Fibrilline, Periostin, etc.) 11, die miteinander und mit den Satellitenzellen im Kontext von Gesundheit, Krankheit und Regeneration interagieren.
Die Bewahrung dieser komplexen Nische in experimentellen Umgebungen ist grundlegend, aber auch eine Herausforderung. Ebenso schwierig ist es, die Ruhe aufrechtzuerhalten oder wieder herzustellen, ein Zellzustand, der für Satellitenzellen kritisch ist9. Es wurden mehrere Methoden eingeführt, um diese Herausforderungen teilweise zu bewältigen, jede mit ihren Vor- und Nachteilen (ausführlich im Diskussionsabschnitt). Hier wird eine Methode vorgestellt, die diese beiden Barrieren teilweise überwinden kann. Muskeln werden zunächst entnommen und dann mechanisch und enzymatisch abgebaut, bevor die heterogene Zellmischung in Kultur gegeben wird. Im Verlauf der Kultur werden viele Zelltypen der Nische nachgewiesen und Satellitenzellen beobachtet, die in den Ruhezustand zurückgekehrt sind. Als letzter Schritt des Protokolls werden die Immunfluoreszenzschritte vorgestellt, die den Nachweis jedes Zelltyps durch die Verwendung allgemein anerkannter Marker ermöglichen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Alle Versuche entsprachen den französischen und EU-Tierschutzvorschriften des Institut Mondor de Recherche Biomédicale (INSERM U955), insbesondere der Richtlinie 2010/63/EU. Die Tiere wurden in einer kontrollierten und angereicherten Umgebung in den Tiereinrichtungen mit den Zertifizierungsnummern A94 028 379 und D94-028-028 gehalten. Sie wurden nur von autorisierten Forschern und Tierpflegern angefasst und vom Personal der Tierhaltung visuell auf Anzeichen von Unbehagen während ihres Lebens untersucht. Sie wurden vor der Sektion durch Gebärmutterhalsluxation euthanasiert. Zu Lebzeiten der Tiere wurden keine interventionellen Eingriffe durchgeführt; Daher war es nicht erforderlich, die Zustimmung einer Ethikkommission und des französischen Ministeriums für Hochschulbildung, Forschung und Innovation für das Verfahren einzuholen. In der Tat ist für die Euthanasie und die postmortale Sektion gemäß der Richtlinie 2010/63/EU keine Ethik-Unbedenklichkeitsbescheinigung erforderlich. Die in diesem Manuskript vorgestellten Ergebnisse stammen aus der Wildtyp-Linie C57BL/6NRj (siehe Materialtabelle) und der transgenen Tg:Pax7-nGFP-Linie 12 (gezüchtet von unserem Team). Das Protokoll wurde auf männliche und weibliche Mäuse im Alter von 8-12 Wochen angewendet.
1. Vorbereitung des Reagenzes und der Ausrüstung vor dem Verdau
- Besprühen Sie die Sezierwerkzeuge (gerade und gebogene Schere, Pinzette, siehe Materialtabelle) mit 70%igem Ethanol und trocknen Sie sie mit Papier. Beschichten Sie eine Korkplatte mit Alufolie und halten Sie 10 cm Petrischalen (eine pro Tier) in der Nähe. Halten Sie Papier und 70% Ethanol in Reichweite.
HINWEIS: Spülen Sie die Sezierwerkzeuge am Ende der Sektion mit Wasser ab, besprühen Sie sie dann mit 70%igem Ethanol und trocknen Sie sie mit Papier. - Stellen Sie ein rotierendes Wasserbad auf 37 °C ein und bereiten Sie die Aufschlussmischung (20 ml/Tier) zu, indem Sie DMEM mit 1 % Penicillin-Streptomycin, 0,5 U/ml-Kollagenase, 3 U/ml-Dispase (siehe Materialtabelle) und 0,2 % BSA in 50-ml-Röhrchen kombinieren.
- Die Aufschlussmischung wird durch einen 0,22-μm-Filter in einer Zellkulturhaube geleitet.
HINWEIS: Es wird empfohlen, die Verdauungsmischung jedes Mal frisch zuzubereiten.
2. Vorbereitung des Reagenzes und der Ausrüstung nach dem Aufschluss
- Nach der Verdauung kann die Mischung eingefroren oder kultiviert werden. Zum Einfrieren bereiten Sie 10 % DMSO: 90 % fötales Kälberserum (FBS) sowie einen Satz Kryoröhrchen (1 ml Zellsuspension pro 2 ml Kryoröhrchen) vor. Für die Kultur bereiten Sie das Nährmedium (DMEM ergänzt mit 1 % Penicillin-Streptomycin, 4 ng/ml bFGF und 20 % FBS) und einen Satz 8-Well-Platten vor. Die Platten müssen vor dem Beschichten der Zellen beschichtet werden (Details finden Sie in Schritt 7.1).
- Für die Färbung werden 4 % Paraformaldehyd (PFA) in phosphatgepufferter Kochsalzlösung (PBS) (0,15 ml/Well der 8-Well-Platte) und Blockierungslösung (5 % IgG-freies Rinderserumalbumin [BSA] in PBS; 0,15 ml/Well der 8-Well-Platte) vorbereitet.
VORSICHT: Atmen Sie das PFA-Pulver nicht ein; Bereiten Sie es vor und handhaben Sie es unter einer chemischen Haube.
3. Sezieren
- Besprühen Sie das eingeschläferte Tier mit 70%igem Ethanol. Machen Sie einen horizontalen Schnitt (linke Körperseite zur rechten Seite) mit einer großen Schere auf Höhe des Bauches und schneiden Sie um die Taille herum. Ziehen Sie die Haut von den Hintergliedmaßen ab, um die Muskeln freizulegen (Abbildung 1A).
- Lege das Tier auf die mit Aluminiumfolie überzogene Korkplatte und stecke die gegenüberliegende Vorder- und Hintergliedmaße fest. Entfernen Sie schnell alle Muskeln der Hintergliedmaßen (vorne und hinten) in eine 10 cm lange Petrischale, die auf Eis gestellt wird (Abbildung 1B,C). Achten Sie besonders darauf, das Fettgewebe aus den Bereichen um den Quadrizeps und die hinteren Muskeln zu entfernen. Zu diesem Zeitpunkt können auch Faszien, Nerven und Sehnen entfernt werden, wenn dies die Gesamtzeit für die Präparation nicht beeinträchtigt.
HINWEIS: Eine optimale Dissektionszeit für beide Hintergliedmaßen sollte etwa 15-20 Minuten betragen. Es wird empfohlen, dass die Präparierzeit 30 Minuten nicht überschreitet. - Geben Sie gelegentlich DMEM-Tropfen in die Muskeln, um sie feucht zu halten, aber nicht zu viel, da dies das Hacken erschwert. Wiederholen Sie den Vorgang für die andere Hintergliedmaße. Sobald sich alle Muskeln eines Tieres in der Petrischale befinden (Abbildung 1D), schneiden Sie sie 7-10 Minuten lang mit einer Schere fein zusammen, um ein glattes Homogenat zu erhalten (Abbildung 1E).
HINWEIS: In diesem Protokoll wird DMEM mit L-Glutamin, Pyruvat und 4,5 g/L D-Glucose ergänzt.
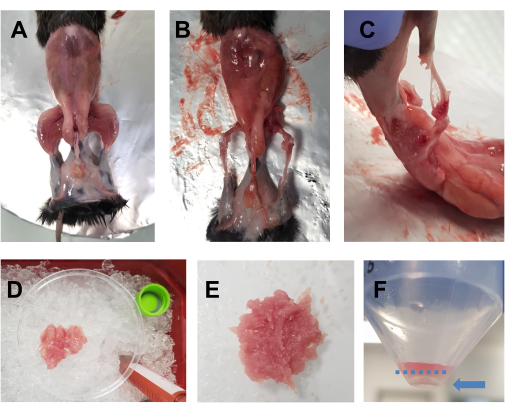
Abbildung 1: Vorbereitung der Muskeln vor der Kultur. (A) Die Haut wird entfernt, um die Muskeln der Hintergliedmaßen freizulegen, wie in Schritt 3.1 beschrieben. (B,C) Alle Muskeln der Hintergliedmaßen werden (B) um und (C) zwischen den Knochen entnommen, wie in Schritt 3.2 beschrieben. (D) Die entnommenen Muskeln werden in einer 10 cm großen Petrischale mit DMEM-Tropfen auf Eis gelegt, um sie feucht zu halten, wie in Schritt 3.3 beschrieben. (E) Die Muskeln werden mit einer Schere fein gehackt, bis eine glatte Paste mit der in diesem Bild dargestellten Konsistenz entsteht. (F) ein Bild des Pellets nach der abschließenden Zentrifugation; Der blaue Pfeil hebt das Pellet hervor, das sich gegen das Rohr befindet, unter der gestrichelten blauen Linie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Verdauung
HINWEIS: Am Ende des Aufschlusses werden eine Zentrifuge bei 4 °C, ein Eimer Eis, drei Zellsiebe (100 μm, 70 μm, 40 μm) und drei 50-ml-Röhrchen (pro Tier) für Abschnitt 5 benötigt.
- Bereiten Sie die Aufschlussmischung vor und filtern Sie sie, wie in Schritt 1.2 beschrieben. Halte die Mischung auf Eis.
- Sobald alle Muskeln zerkleinert sind, geben Sie das Homogenat in ein 50-ml-Röhrchen mit 20 ml Aufschlussmischung. Umwickeln Sie die Ränder des Deckels mit flexibler Folie, um ein Auslaufen zu verhindern, und legen Sie das Röhrchen in ein 37 °C heißes Schüttelwasserbad mit niedriger bis mittlerer Geschwindigkeit (50 U/min).
- Öffnen Sie nach 1 Stunde bei 37 °C den Deckel und mischen Sie, indem Sie mit einer 10-ml-Pipette siebenmal vorsichtig auf und ab pipettieren, um eine homogene Mischung zu erhalten. Legen Sie eine neue Folie um den Deckel und legen Sie ihn wieder in das Schüttelwasserbad. Entfernen Sie nach 1 Stunde den Schlauch und schalten Sie das Bad aus.
HINWEIS: Verwenden Sie diese Inkubationszeit für die Kultur, um die Platten wie in Schritt 7.1 beschrieben zu beschichten, bevor Sie mit Abschnitt 5 fortfahren.
5. Filtration
- Füllen Sie das Aufschlussröhrchen mit kaltem DMEM (ergänzt mit 1% Penicillin-Streptomycin) bis zu 50 ml. Mischen Sie, indem Sie die Tube dreimal umdrehen. Bewahren Sie das DMEM für die nächsten Schritte in einem Eiskübel auf.
- Legen Sie ein 100-μm-Zellsieb auf ein neues 50-ml-Röhrchen. Führen Sie die verdaute Mischung durch das Zellsieb in das neue Röhrchen. Bei 600 x g 5 min bei 4 °C zentrifugieren. Gießen Sie den Überstand in einen flüssigen Abfallbehälter.
- Resuspendieren Sie das Pellet in 1 ml kaltem DMEM (ergänzt mit 1% Penicillin-Streptomycin). Füllen Sie das Röhrchen bis zu 50 ml mit demselben DMEM. HINWEIS: Wenn die Zentrifugation übersprungen wird, ist das nächste Pellet schwieriger zu identifizieren und zu warten.
- Legen Sie ein 70-μm-Zellsieb auf ein neues 50-ml-Röhrchen. Führen Sie die zentrifugierte/resuspendierte Mischung durch das Zellsieb in das neue Röhrchen. Bei 80 x g 5 min bei 4 °C zentrifugieren.
HINWEIS: Dieser Schritt ist nicht obligatorisch, wird aber empfohlen, um Zelltrümmer zu beseitigen. - Legen Sie ein 40-μm-Zellsieb auf ein neues 50-ml-Röhrchen. Führen Sie den Überstand durch das Zellsieb in das neue Röhrchen. Bei 600 x g für 5 min bei 4 °C zentrifugieren, den Überstand in einen Behälter für flüssige Abfälle gießen und das Pellet in FBS unter der Kulturhaube resuspendieren. Das Pellet ist in diesem Schritt sehr klein (Abbildung 1F).
HINWEIS: Durch das Filtern durch das 40-μm-Sieb werden Ablagerungen entfernt, die bei der späteren Färbung der Kulturen ein unspezifisches Signal abgeben würden.
6. (Optional) Einfrieren
HINWEIS: Abschnitt 6 ist optional. Das Protokoll kann nach der Filterung pausiert werden, was jedoch das Überleben der Zellen und den Kulturerfolg verringern kann.
- Fügen Sie DMSO hinzu, um ein Verhältnis von 10 % DMSO zu 90 % FBS zu erhalten, und überführen Sie es in Kryoröhrchen (1 ml resuspendiertes Pellet pro 2 ml Kryoröhrchen).
- Legen Sie das Kryoröhrchen bei −80 °C über Nacht in eine Styroporbox. Am nächsten Tag auf −150 °C für eine langfristige Lagerung umstellen.
HINWEIS: Eine kurzzeitige Lagerung bei −80 °C ist ebenfalls möglich. - Zu Beginn der Kultur wird das Kryoröhrchen in einem 37 °C warmen Wasserbad schnell aufgetaut, bis die Zellsuspension aufgetaut ist. Mischen Sie mit 4 ml DMEM unter der Kultivierungshaube. Bei 600 x g 5 min bei 4 °C schleudern. Pipettieren Sie den Überstand heraus und fahren Sie fort, wie in Schritt 7.2 beschrieben.
7. Kultivierung
HINWEIS: Es kann davon ausgegangen werden, dass gefrorene oder frische Zellsuspensionen 24-32 Wells mit drei bis vier 8-Well-Platten füllen.
- Beschichten Sie 8-Well-Platten mit der Beschichtungslösung, die bei 4 °C oder auf Eis aufgetaut werden sollte (Standardbeschichtungslösung wird normalerweise bei −20 °C aufbewahrt). Geben Sie 0,4 ml Beschichtungslösung in eine Vertiefung und pipettieren Sie sie von Vertiefung zu Vertiefung. Nachdem die Beschichtungslösung alle Vertiefungen durchlaufen hat, kann sie für zukünftige Kulturen wieder aufgefangen und wieder eingefroren werden. Halten Sie die beschichteten Platten 30 Minuten lang bei 37 °C, bevor Sie die Zellen plattieren.
- DMEM (ergänzt mit 1% Penicillin-Streptomycin) ergänzt mit 4 ng/ml bFGF (siehe Materialtabelle) zur FBS-Zellsuspension hinzufügen, um ein FBS:80%-DMEM-Verhältnis von 20 % zu erhalten.
HINWEIS: Auch wenn die Zugabe von bFGF in primären Myoblastenkulturen und bei der satellitenähnlichen Zellproduktion in Massenkulturen von Vorteil sein kann, ist ihre Zugabe optional, da ihre Auslassung in Massenkulturen von ~7 Tagen die Zellausbeute nicht ernsthaft beeinträchtigt. - Platte 0,4 ml der Suspension pro Vertiefung (aus Schritt 7.2) in die beschichteten 8-Vertiefungsplatten.
HINWEIS: Berechnen Sie 30cm2 Kultur pro Tier für gefrorene und frische Zubereitungen. - Inkubieren Sie die Kulturen bei 37 °C mit 5 % CO2 für bis zu 10 Tage, wobei das Medium jeden Tag gewechselt wird, nachdem die Kultur beginnt, eine gelbliche Farbe anzunehmen (normalerweise 5-7 Tage).
HINWEIS: Um Zellen in der S-Phase des Zellzyklus 13 zu quantifizieren, fügen Sie 2 h vor der Fixierung10 μM EdU hinzu. Um die erste S-Phase einzufangen, fügen Sie 10 μM EdU von der Beschichtung hinzu und fixieren Sie sie bei 40 h Kultur.
8. Fixierung
HINWEIS: Die Abschnitte 8-10 sollten bei Raumtemperatur durchgeführt werden, sofern nicht anders angegeben.
- Pipettieren Sie das Kulturmedium heraus und fixieren Sie die Zellen mit 4 % PFA (0,15 ml/Well).
VORSICHT: Fügen Sie PFA unter einer chemischen Haube hinzu.
HINWEIS: Wenn alle Vertiefungen gleichzeitig fixiert sind, 10 Minuten lang mit PFA bei Raumtemperatur inkubieren. Wenn die Vertiefungen zu unterschiedlichen Zeitpunkten fixiert sind, fügen Sie PFA zu den zu fixierenden Vertiefungen hinzu und lassen Sie die Platte 5 Minuten lang bei 37 °C im Inkubator. - Pipettieren Sie die PFA heraus und fügen Sie PBS für 10 s (0,15 ml/Well) hinzu. Pipettieren Sie das PBS heraus und fügen Sie frisches PBS für 5 Minuten (0,15 ml/Well) hinzu.
HINWEIS: Wenn alle Vertiefungen gleichzeitig fixiert sind, inkubieren Sie mit PBS bei Raumtemperatur. Wenn die Vertiefungen zu unterschiedlichen Zeitpunkten fixiert sind, fügen Sie PBS zu den festen Vertiefungen hinzu und lassen Sie die Platte 5 Minuten lang bei 37 °C im Inkubator. Fügen Sie dann 0,4 ml PBS hinzu und bewahren Sie die Platte bis zu 1 Woche im Inkubator auf.
9. Permeabilisierung und Blockierung
- Wenn Sie bereit sind, das PBS zu färben, pipettieren Sie es aus und permeabilisieren Sie es mit 0,5 % TritonX 100 in PBS (0,15 ml/Well) für 8 Minuten. Pipettieren Sie den TritonX 100, spülen Sie ihn 10 s lang (0,15 ml/Well) mit PBS, pipettieren Sie den PBS heraus und waschen Sie ihn 5 Minuten lang mit PBS (0,15 ml/Well).
- Block mit 5% IgG-freiem BSA in PBS für 30-60 min (0,15 ml/Well).
10. Färbung
- Pipettieren Sie die BSA heraus und fügen Sie die primäre Antikörpermischung in PBS verdünnt (0,15 ml/Well) hinzu (siehe Materialtabelle; Verdünnungen: Anti-CD31 1:100, Anti-FOSB 1:200, Anti-GFP 1:1.000, Anti-KI67 1:1.000, Anti-MyHC 1:400, Anti-MYOD 1:200, Anti-MYOG 1:150, Anti-PAX7 1:100, Anti-PDGFRa 1:50) für die nächtliche Inkubation bei 4 °C.
HINWEIS: Nach der Antikörperinkubation die Antikörpermischung auffangen, Natriumazid hinzufügen und bei 4 °C oder −20 °C (gemäß den Anweisungen des Antikörperherstellers) für die zukünftige Wiederverwendung aufbewahren. - Pipettieren Sie die Antikörpermischung heraus, spülen Sie sie 10 s lang mit PBS (0,15 ml/Well), pipettieren Sie die PBS heraus und waschen Sie sie 5 Minuten lang mit PBS (0,15 ml/Well).
- Pipettieren Sie das Wasch-PBS heraus, fügen Sie die sekundäre Antikörpermischung (Ziegen-Anti-Maus Alexa Fluor 488, Ziegen-Anti-Kaninchen Alexa Fluor 555, Ziegen-Anti-Ratten-Alexa Fluor 647, Ziegen-Anti-Maus Alexa Fluor 555, Ziegen-Anti-Huhn Alexa Fluor 488, alle in Verdünnungen von 1:500-1.000 verwendet) und Zellmarker (z. B. DAPI) in PBS (0,15 ml/Well) verdünnt hinzu (siehe Materialtabelle), und 1 h bei Raumtemperatur lichtgeschützt inkubieren.
- Pipettieren Sie die sekundäre Antikörpermischung, spülen Sie sie 10 s lang (0,15 ml/Well) mit PBS ab, pipettieren Sie die PBS, waschen Sie sie 5 Minuten lang mit PBS (0,15 ml/Well), pipettieren Sie die PBS heraus und montieren Sie sie.
HINWEIS: Wenn 8-Well-Platten mit abnehmbaren Separatoren verwendet werden, ziehen Sie die Separatoren vor der Montage ab.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Dieses Protokoll ermöglicht die Kultivierung von Muskelzellen, während die Satellitenzellen und die meisten Zellen aus ihrer endogenen Nische geschützt werden. Abbildung 2 fasst die wichtigsten Schritte des Protokolls zusammen, während wesentliche Teile der Dissektion und des Aufschlusses in Abbildung 1 dargestellt sind. Eine Dissektion der Muskulatur der Hintergliedmaßen wird empfohlen (Abbildung 1A-C<...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Funktion der Skelettmuskulatur bei Erwachsenen wird durch eine fein orchestrierte Reihe von zellulären Interaktionen und molekularen Signalen untermauert. Hier wird eine Methode vorgestellt, die es ermöglicht, diese Parameter in einer ex vivo Umgebung zu untersuchen, die der physiologischen Mikroumgebung sehr ähnlich ist.
Mehrere Gruppen haben über In-vitro-Methoden zur Kultivierung myogener Zellen berichtet. Diese Methoden zielten darauf ab, Satellitenzellen zu isoli...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Für Abbildung 2 wurden Vorlagen von Servier Medical Art (https://smart.servier.com/) verwendet. Das FR-Labor wird von der Association Française contre les Myopathies - AFM via TRANSLAMUSCLE (Stipendien 19507 und 22946), der Fondation pour la Recherche Médicale - FRM (EQU202003010217, ENV202004011730, ECO201806006793), der Agence Nationale pour la Recherche - ANR (ANR-21-CE13-0006-02, ANR-19-CE13-0010, ANR-10-LABX-73) und der La Ligue Contre le Cancer (IP/SC-17130) unterstützt. Die oben genannten Geldgeber spielten keine Rolle bei der Konzeption, Sammlung, Analyse, Interpretation oder Berichterstattung dieser Studie oder dem Schreiben dieses Manuskripts.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| anti-CD31 | BD | 550274 | dilution 1:100 |
| anti-FOSB | Santa Cruz | sc-7203 | dilution 1:200 |
| anti-GFP | Abcam | ab13970 | dilution 1:1000 |
| anti-Ki67 | Abcam | ab16667 | dilution 1:1000 |
| anti-MyHC | DSHB | MF20-c | dilution 1:400 |
| anti-MYOD | Active Motif | 39991 | dilution 1:200 |
| anti-MYOG | Santa Cruz | sc-576 | dilution 1:150 |
| anti-Pax7 | Santa Cruz | sc-81648 | dilution 1:100 |
| anti-PDGFRα | Invitrogen | PA5-16571 | dilution 1:50 |
| b-FGF | Peprotech | 450-33 | concentration 4 ng/mL |
| Bovine serum albumin (BSA) – used for digestion | Sigma Aldrich | A7906-1006 | concentration 0.2% |
| BSA IgG-free, protease-free – used for staining | Jackson ImmunoResearch | 001-000-162 | concentration 5% |
| Cell strainer 40 um | Dominique Dutscher | 352340 | |
| Cell strainer 70 um | Dominique Dutscher | 352350 | |
| Cell strainer 100 um | Dominique Dutscher | 352360 | |
| Collagenase | Roche | 10103586001 | concentration 0.5 U/mL |
| Culture plate | Sarstedt | 94.6140.802 | |
| Dimethyl sulfoxide (DMSO) | Euromedex | UD8050-05-A | |
| Dispase | Roche | 4942078001 | concentration 3 U/mL |
| Dissection forceps size 5 | Fine Science Tools | 91150-20 | |
| Dissection forceps size 55 | Fine Science Tools | 11295-51 | |
| Dissection scissors (big, straight) | Fine Science Tools | 9146-11 | ideal for chopping |
| Dissection scissors (small, curved) | Fine Science Tools | 15017-10 | |
| Dissection scissors (small, straight) | Fine Science Tools | 14084-08 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | ThermoFisher | 41966-029 | |
| EdU Click-iT kit | ThermoFisher | C10340 | |
| Fetal bovine serum – option 1 | Eurobio | CVF00-01 | |
| Fetal bovine serum – option 2 | Gibco | 10270-106 | |
| Matrigel | Corning Life Sciences | 354234 | coating solution |
| Parafilm | Dominique Dutscher | 090261 | flexible film |
| Paraformaldehyde – option 1 | PanReac AppliChem ITW Reagents | 211511.1209 | concentration 4% |
| Paraformaldeyde – option 2 | ThermoFisher | 28908 | concentration 4% |
| Penicillin streptomycin | Gibco | 15140-122 | |
| Shaking water bath | ThermoFisher | TSSWB27 | |
| TritonX100 | Sigma Aldrich | T8532-500 ML | concentration 0.5% |
| Wild-type mice | Janvier | C57BL/6NRj |
Referenzen
- Frontera, W. R., Ochala, J. Skeletal muscle: A brief review of structure and function. Calcified Tissue International. 96 (3), 183-195 (2015).
- Forcina, L., Cosentino, M., Musarò, A. Mechanisms regulating muscle regeneration: Insights into the interrelated and time-dependent phases of tissue healing. Cells. 9 (5), 1297(2020).
- Mauro, A. Satellite cell of skeletal muscle fibers. Journal of Biophysical and Biochemical Cytology. 9 (2), 493-495 (1961).
- Lepper, C., Partridge, T. A., Fan, C. -M. An absolute requirement for Pax7-positive satellite cells in acute injury-induced skeletal muscle regeneration. Development. 138 (17), 3639-3646 (2011).
- McCarthy, J. J., et al. Effective fiber hypertrophy in satellite cell-depleted skeletal muscle. Development. 138 (17), 3657-3666 (2011).
- Murphy, M. M., Lawson, J. A., Mathew, S. J., Hutcheson, D. A., Kardon, G. Satellite cells, connective tissue fibroblasts and their interactions are crucial for muscle regeneration. Development. 138 (17), 3625-3637 (2011).
- Sambasivan, R., et al. Pax7-expressing satellite cells are indispensable for adult skeletal muscle regeneration. Development. 138 (17), 3647-3656 (2011).
- Hicks, M. R., Pyle, A. D. The emergence of the stem cell niche. Trends in Cell Biology. 33 (22), 112-123 (2022).
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nature Communications. 12 (1), 692(2021).
- Gama, J. F. G., et al. Role of regulatory T cells in skeletal muscle regeneration: A systematic review. Biomolecules. 12 (6), 817(2022).
- Loreti, M., Sacco, A. The jam session between muscle stem cells and the extracellular matrix in the tissue microenvironment. NPJ Regenerative Medicine. 7 (1), 16(2022).
- Sambasivan, R., et al. Distinct regulatory cascades govern extraocular and pharyngeal arch muscle progenitor cell fates. Developmental Cell. 16 (6), 810-821 (2009).
- Pereira, P. D., et al. Quantification of cell cycle kinetics by EdU (5-ethynyl-2'-deoxyuridine)-coupled-fluorescence-intensity analysis. Oncotarget. 8 (25), 40514-40532 (2017).
- Bismuth, K., Relaix, F. Genetic regulation of skeletal muscle development. Experimental Cell Research. 316 (18), 3081-3086 (2010).
- Yin, H., Price, F., Rudnicki, M. A. Satellite cells and the muscle stem cell niche. Physiological Reviews. 93 (1), 23-67 (2013).
- Lertkiatmongkol, P., Liao, D., Mei, H., Hu, Y., Newman, P. J. Endothelial functions of platelet/endothelial cell adhesion molecule-1 (CD31). Current Opinion in Hematology. 23 (3), 253-259 (2016).
- Scholzen, T., Gerdes, J. The Ki-67 protein: From the known and the unknown. Journal of Cellular Physiology. 182 (3), 311-322 (2000).
- Abou-Khalil, R., Le Grand, F., Chazaud, B. Human and murine skeletal muscle reserve cells. Stem Cell Niche. 1035, 165-177 (2013).
- Pasut, A., Oleynik, P., Rudnicki, M. A. Isolation of muscle stem cells by fluorescence activated cell sorting cytometry. Methods in Molecular Biology. 798, 53-64 (2011).
- Liu, L., Cheung, T. H., Charville, G. W., Rando, T. A. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting. Nature Protocols. 10 (10), 1612-1624 (2015).
- Montarras, D., et al. Direct isolation of satellite cells for skeletal muscle regeneration. Science. 309 (5743), 2064-2067 (2005).
- Qu, Y., Edwards, K., Barrow, J. Isolation, culture, and use of primary murine myoblasts in small-molecule screens. STAR Protocols. 4 (2), 102149(2023).
- Danoviz, M. E., Yablonka-Reuveni, Z. Skeletal muscle satellite cells: Background and methods for isolation and analysis in a primary culture system. Methods in Molecular Biology. 798, 21-52 (2011).
- Saclier, M., Theret, M., Mounier, R., Chazaud, B. Effects of macrophage conditioned-medium on murine and human muscle cells: analysis of proliferation, differentiation, and fusion. Methods in Molecular Biology. 1556, 317-327 (2017).
- Giordani, L., et al. High-dimensional single-cell cartography reveals novel skeletal muscle-resident cell populations. Molecular Cell. 74 (3), 609-621 (2019).
- Tabula Muris Consortium et al. Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris. Nature. 562 (7727), 367-372 (2018).
- Brunetti, J., Koenig, S., Monnier, A., Frieden, M. Nanopattern surface improves cultured human myotube maturation. Skeletal Muscle. 11 (1), 12(2021).
- Denes, L. T., et al. Culturing C2C12 myotubes on micromolded gelatin hydrogels accelerates myotube maturation. Skeletal Muscle. 9 (1), 17(2019).
- LaFramboise, W. A., et al. Effect of muscle origin and phenotype on satellite cell muscle-specific gene expression. Journal of Molecular and Cellular Cardiology. 35 (10), 1307-1318 (2003).
- Azhar, M., Wardhani, B. W. K., Renesteen, E. The regenerative potential of Pax3/Pax7 on skeletal muscle injury. Journal of Generic Engineering and Biotechnology. 20 (1), 143(2022).
- Hardy, D., et al. Comparative study of injury models for studying muscle regeneration in mice. PLoS One. 11 (1), e0147198(2016).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten