Dreidimensionale Morphogenese im Darm-on-a-Chip von Hunden unter Verwendung von Darmorganoiden, die von Patienten mit chronisch entzündlichen Darmerkrankungen stammen
In diesem Artikel
Zusammenfassung
Die Integration von caninen Darmorganoiden und einem mikrofluidischen Gut-on-a-Chip-System bietet relevante translationale Modelle für menschliche Darmerkrankungen. Die vorgestellten Protokolle ermöglichen eine 3D-Morphogenese und eine dynamische In-vitro-Modellierung des Darms und helfen so bei der Entwicklung wirksamer Behandlungen für Darmerkrankungen bei Hunden und Menschen mit One Health.
Zusammenfassung
Der Darm von Hunden weist Ähnlichkeiten in Anatomie, Mikrobiologie und Physiologie mit dem des Menschen auf, und Hunde entwickeln auf natürliche Weise spontane Darmerkrankungen, die denen des Menschen ähneln. Die Überwindung der inhärenten Begrenzung dreidimensionaler (3D) Organoide beim Zugang zur apikalen Oberfläche des Darmepithels hat zur Generierung von zweidimensionalen (2D) Monolayer-Kulturen geführt, die die zugängliche luminale Oberfläche mit Hilfe von Zellen freilegen, die von den Organoiden abgeleitet sind. Die Integration dieser Organoide und aus Organoiden gewonnenen Monolayer-Kulturen in ein mikrofluidisches Gut-on-a-Chip-System hat die Technologie weiterentwickelt und die Entwicklung physiologisch relevanterer dynamischer In-vitro-Darmmodelle ermöglicht.
In dieser Studie stellen wir ein Protokoll zur Generierung der 3D-Morphogenese des Darmepithels von Hunden unter Verwendung von primären Darmgewebeproben vor, die von Hunden gewonnen wurden, die von chronisch entzündlichen Darmerkrankungen (CED) betroffen sind. Wir skizzieren auch ein Protokoll zur Generierung und Aufrechterhaltung von 2D-Monolayer-Kulturen und Intestine-on-a-Chip-Systemen unter Verwendung von Zellen, die von den 3D-Darmorganoiden abgeleitet sind. Die in dieser Studie vorgestellten Protokolle dienen als grundlegender Rahmen für die Etablierung eines mikrofluidischen Gut-on-a-Chip-Systems, das speziell für Hunde entwickelt wurde. Indem wir den Grundstein für diesen innovativen Ansatz legen, wollen wir die Anwendung dieser Techniken in der biomedizinischen und translationalen Forschung ausweiten und uns dabei an den Prinzipien der One-Health-Initiative orientieren. Durch die Verwendung dieses Ansatzes können wir physiologisch relevantere dynamische In-vitro-Modelle für die Untersuchung der Darmphysiologie sowohl bei Hunden als auch bei Menschen entwickeln. Dies hat erhebliche Auswirkungen auf biomedizinische und pharmazeutische Anwendungen, da es bei der Entwicklung wirksamerer Behandlungen für Darmerkrankungen beider Arten helfen kann.
Einleitung
Die Morphogenese des Darmepithels wurde weitgehend anhand von Labortiermodellen untersucht, die kostspielig und zeitaufwändig sind und menschliche Entwicklungsprozesse nicht genau abbilden1. Darüber hinaus sind herkömmliche statische 2D-Zellkulturmodelle nicht in der Lage, die komplexe räumliche Organisation einer 3D-Epithelarchitektur nachzuahmen2. Infolgedessen besteht ein Bedarf an einem Protokoll zur Induktion der In-vitro-3D-Morphogenese unter Verwendung von Darmepithelzellen aus humanrelevanten Tiermodellen, um unser Verständnis der Darmepithelarchitektur zu verbessern.
Begleithunde haben eine Darmanatomie und Mikrobiomzusammensetzung entwickelt, die dem Menschen aufgrund ihrer gemeinsamen Umgebung und Ernährung während der Domestizierung bemerkenswert ähnlich ist3. Zusätzlich zu dieser Ähnlichkeit teilen sowohl Menschen als auch Hunde verschiedene chronische Krankheiten, von denen angenommen wird, dass sie auf die Darmgesundheit zurückzuführen sind. Hunde können, wie Menschen, spontan chronische Erkrankungen wie Fettleibigkeit, kognitive Dysfunktion, Diabetes mellitus, entzündliche Darmerkrankungen (CED) und kolorektales Adenokarzinom entwickeln 4,5,6,7,8,9,10. Trotz der Entwicklung und Verwendung von humanen und murinen Epithelzellen in früheren Gut-on-a-Chip-Studien 2,11,12,13,14 wurde das Darmepithel von Hunden bisher nicht verwendet. Unser neuartiger Ansatz, bei dem das intestinale Organoidepithel von Hunden in einem dynamischen Kultursystem mit einer 3D-Epithel-Morphogenese verwendet wird, hat erhebliche Auswirkungen sowohl auf die Hunde- als auch auf die Humanmedizin.
Jüngste Fortschritte in der Darmorganoidkultur haben zur Etablierung der Darmorganoidkultur bei Hunden geführt15. Dieses Kultursystem beinhaltet die Kultivierung von Darmstammzellen unter definierter morphogener Konditionierung, was zu einem 3D-Modell mit selbsterneuernden Eigenschaften führt, das aus adulten Stammzellen gewonnen wird16. Die Durchführung von Transportassays oder Wirt-Mikrobiom-Kokulturen bereitet diesem 3D-Modell jedoch aufgrund der geschlossenen Natur des Darmlumens Schwierigkeiten17. Um dieses Problem zu lösen, haben die Forscher eine 2D-Monoschicht aus Darmorganoiden erzeugt, die eine Freilegung der luminalen Oberfläche ermöglicht18,19. Sowohl 3D-Organoide als auch 2D-Monolayer werden jedoch unter statischen Bedingungen aufrechterhalten, die die In-vivo-Biomechanik der intestinalen Mikroumgebung nicht genau widerspiegeln. Die Kombination von patientenbezogener Hunde-Organoid-Technologie mit In-vitro-3D-Morphogenese bietet eine Chance für die translationale Erforschung chronischer multifaktorieller Erkrankungen. Dieser Ansatz ermöglicht es den Forschern, wirksamere Behandlungen zu entwickeln, die sowohl Menschen als auch Hunden zugute kommen, und die translationale Forschung weiter voranzutreiben, im Einklang mit der One Health Initiative, einem kollaborativen Ansatz, der die Verflechtung der Gesundheit von Mensch, Tier und Umwelt anerkennt. Es fördert die interdisziplinäre Zusammenarbeit, um komplexe gesundheitliche Herausforderungen anzugehen und optimale Gesundheitsergebnisse für alle zu erzielen. Durch das Verständnis der wechselseitigen Abhängigkeiten zwischen Menschen, Tieren und Ökosystemen zielt die Initiative darauf ab, Risiken durch neu auftretende Infektionskrankheiten, Umweltzerstörung und andere gemeinsame Gesundheitsprobleme zu mindern20,21,22.
Dieses Protokoll beschreibt umfassende Methoden zur Kultivierung von Darmepithelzellen von Hunden, die aus Patientenorganoiden gewonnen wurden, auf einem Gut-on-a-Chip-Mikrogerät mit einer porösen Membran auf Basis von Polydimethylsiloxan (PDMS). Die Etablierung der 3D-Epithelmorphogenese durch die Integration von Darmorganoiden bei Hunden und diese Gut-on-a-Chip-Technologie ermöglicht es uns, zu untersuchen, wie der Darm seine zelluläre Organisation und Stammzellnische entwickelt und aufrechterhält. Diese Plattform bietet eine wertvolle Gelegenheit, den Einfluss von Mikrobiomgemeinschaften auf die Darmgesundheit zu untersuchen und zu verstehen, wie diese Gemeinschaften mikrobielle Metaboliten erzeugen, die zur Pathophysiologie des Darms beitragen14,23. Diese Fortschritte können nun auf Darmproben von Hunden ausgeweitet werden, was den Forschern die Möglichkeit bietet, die komplizierte Beziehung zwischen dem Darmmikrobiom und der Physiologie des Wirts zu untersuchen. Dies eröffnet Möglichkeiten, wertvolle Einblicke in die zugrunde liegenden Mechanismen der intestinalen Pathophysiologie zu gewinnen und die potenzielle Rolle mikrobieller Metaboliten sowohl für die Gesundheit von Hunden als auch für Menschen sowie für verschiedene Krankheitszustände zu verstehen. Das Protokoll, das für den caninen Gut-on-a-Chip verwendet wird, ist reproduzierbar, was es zu einem geeigneten experimentellen Modell für die vergleichende Medizin macht, da dieser Ansatz die Untersuchung von Wirt-Mikrobiom-Interaktionen, pathogenen Infektionen und probiotischen therapeutischen Effekten sowohl bei Hunden als auch bei Menschen ermöglicht.
Protokoll
Die Studie wurde in Übereinstimmung mit dem Institutional Animal Care and Use Committee der Washington State University (ASAF# 6993) genehmigt und durchgeführt. In diesem Protokoll haben wir ein etabliertes mikrofluidisches Gut-on-a-Chip-Gerät aus PDMS verwendet, das im eigenen Haus hergestellt wurde2 (Abbildung 1D). Detaillierte Methoden zur Herstellung des Gut-on-a-Chip-Mikrogeräts finden sich in früheren Berichten 2,24. Dieses Protokoll demonstriert eine einzigartige Integration von Darmorganoiden und einem mikrofluidischen System (Abbildung 2).
1. Oberflächenaktivierung eines Gut-on-a-Chips aus PDMS
- Bereiten Sie 1%ige Polyethylenimin (PEI)-Lösung vor, indem Sie 1 ml 50%ige PEI-Lösung in 49 ml destilliertes (DI) Wasser in einem konischen 50-ml-Röhrchen zugeben. Drehen Sie das Röhrchen zwei- bis dreimal um, um die Lösung gut zu mischen, und filtern Sie die Lösung dann mit einem 0,2-μm-Spritzenvorsatzfilter.
HINWEIS: Bewahren Sie es bei 4 °C auf. - Bereiten Sie eine 0,1%ige Glutaraldehyd (GA)-Lösung vor, indem Sie 100 μl 50%ige GA-Lösung in 49,9 ml DI in einem konischen 50-ml-Röhrchen zugeben. Drehen Sie das Röhrchen zwei- bis dreimal um, um die Lösung gut zu mischen, und filtern Sie die Lösung dann mit einem 0,2-μm-Spritzenvorsatzfilter.
HINWEIS: Lagern Sie es bei 4 °C und schützen Sie es vor direktem Licht. - Stellen Sie das Gut-on-a-Chip-Gerät in einen trockenen Ofen bei 60 °C und inkubieren Sie es mindestens 30 Minuten lang, um die restliche Feuchtigkeit zu entfernen.
- Setzen Sie das Gut-on-a-Chip-Gerät 60 Minuten lang einer UV- und Ozonbehandlung mit einem UV/Ozon-Generator aus.
HINWEIS: Um eine optimale Aktivierung der PDMS-Oberflächen während der Behandlung zu gewährleisten, halten Sie einen Abstand von ca. 3 cm oder weniger zwischen der UV-Lampe und dem Gerät ein. Vermeiden Sie eine Überfüllung der Geräte im Generator, um eine effiziente Oberflächenaktivierung zu erreichen. - Befestigen Sie die Einlass-, Bypass- und Auslassschläuche des oberen Mikrokanals, indem Sie sie mit Binderklammern festklemmen. Klemmen Sie auch den Einlassschlauch für den unteren Mikrokanal ein.
- Trennen Sie den Auslassschlauch für den unteren Mikrokanal. Stellen Sie sicher, dass der Bypassschlauch des unteren Mikrokanals offen bleibt.
- Mit einer P100-Mikropipette 100 μl einer 1%igen PEI-Lösung durch das Auslassloch des unteren Mikrokanals einführen.
HINWEIS: Es wird empfohlen, diesen Vorgang durchzuführen, während das Gerät noch warm von der UV-/Ozonbelastung ist. Beobachten Sie, wie die PEI-Lösung durch den Mikrokanal fließt und die PEI-Lösung aus dem Bypass-Schlauch für den unteren Mikrokanal tropft. - Schließen Sie den Auslassschlauch für den unteren Mikrokanal wieder an.
- Befestigen Sie die Einlass-, Bypass- und Auslassschläuche des unteren Mikrokanals, indem Sie sie mit Binderklammern festklemmen. Klemmen Sie auch den Einlassschlauch für den oberen Mikrokanal ein.
- Trennen Sie den Auslassschlauch für den oberen Mikrokanal. Achten Sie darauf, dass der Bypassschlauch des oberen Mikrokanals offen bleibt.
- Mit einer P100-Mikropipette 100 μl einer 1%igen PEI-Lösung durch das Auslassloch des oberen Mikrokanals einführen.
HINWEIS: Beobachten Sie, wie die PEI-Lösung durch den Mikrokanal fließt und die PEI-Lösung aus dem Bypass-Schlauch für den oberen Mikrokanal tropft. - Bringen Sie den Auslassschlauch des oberen Mikrokanals wieder an.
- Sobald sowohl der obere als auch der untere Mikrokanal mit 1%iger PEI-Lösung gefüllt sind, lassen Sie das Gerät 10 Minuten lang bei Raumtemperatur (RT) inkubieren.
- Führen Sie die Schritte 1.5-1.12 mit 0,1%iger GA-Lösung durch.
- Sobald sowohl der obere als auch der untere Mikrokanal mit 0,1%iger GA-Lösung gefüllt sind, lassen Sie das Gerät 20 Minuten lang bei RT inkubieren.
- Führen Sie die Schritte 1.5-1.12 mit DI-Wasser durch, um überschüssige Oberflächenaktivierungslösung zu entfernen.
- Den Chip bei 60 °C in den trockenen Backofen geben und über Nacht trocknen lassen.
HINWEIS: Stellen Sie sicher, dass Sie die Binderklammern von allen Schläuchen entfernen, um ein Aufprallen des Schlauchs zu vermeiden. Dieser Trocknungsprozess ist entscheidend für die gleichmäßige Oberflächenaktivierung des Mikrokanals innerhalb des Gut-on-a-Chip12.
2. Beschichtung der extrazellulären Matrix (ECM) und Vorbereitung des Nährmediums für die Gut-on-a-Chip-Kultur
- Bereiten Sie eine ECM-Mischung mit organoidem Basalmedium vor, so dass die Endkonzentration von Kollagen I und Matrigel 60 μg/ml bzw. 2 % (vol/vol) beträgt.
HINWEIS: Bereiten Sie 50 μl ECM-Mischung pro Darmchip vor (d. h. 20 μl ECM-Mischung für jeden oberen und unteren Mikrokanal und 10 μl extra). Bereiten Sie diese am Tag der Anwendung vor und lagern Sie diese Lösung bei 4 °C oder legen Sie sie auf Eis, bis sie gebrauchsfertig ist. - Nehmen Sie den Gut-on-a-Chip, der mit UV/Ozon, PEI und GA behandelt wurde, aus dem Trockenofen.
HINWEIS: Unter einem Phasenkontrastmikroskop prüfen, ob Restfeuchte vorhanden ist. - Lassen Sie den Gut-on-a-Chip 10 Minuten in einer Biosicherheitswerkbank abkühlen.
- Befestigen Sie die Einlass-, Bypass- und Auslassschläuche des oberen Mikrokanals, indem Sie sie mit Binderklammern festklemmen. Klemmen Sie auch den Einlassschlauch für den unteren Mikrokanal ein.
- Trennen Sie den Auslassschlauch für den unteren Mikrokanal.
- Mit einer P100-Mikropipette 20 μl ECM-Mischung durch das Auslassloch des unteren Mikrokanals einführen.
HINWEIS: Beobachten Sie, wie das ECM-Gemisch durch den Mikrokanal fließt, ohne dass Luftblasen eingeschlossen werden. Wenn eine Luftblase vorhanden ist, führen Sie eine zusätzliche ECM-Mischung in den unteren Mikrokanal ein, bis die Blase verschwunden ist. - Bringen Sie den Auslassschlauch für den unteren Mikrokanal wieder an.
- Befestigen Sie die Einlass-, Bypass- und Auslassschläuche des unteren Mikrokanals, indem Sie sie mit Binderklammern festklemmen. Klemmen Sie auch den Einlassschlauch für den oberen Mikrokanal ein.
- Trennen Sie den Auslassschlauch für den oberen Mikrokanal.
- Mit einer P100-Mikropipette 20 μl ECM-Mischung durch das Auslassloch des oberen Mikrokanals einführen.
- Bringen Sie den Auslassschlauch für den oberen Mikrokanal wieder an.
- Legen Sie den Chip 1 h lang in einen befeuchteten CO 2 -Inkubator mit 5 % CO2 bei 37 °C, um die ECM-Schicht auf der mit PEI und GA behandelten PDMS-Membran zu bilden (Abbildung 3A).
HINWEIS: Stellen Sie sicher, dass alle Schläuche während der Inkubation eingeklemmt sind. - Während der Inkubation der ECM-Beschichtung werden zwei 1-ml-Spritzen mit kaltem Aussaatmedium vorbereitet, bei dem es sich um das organoide Kulturmedium handelt, dem A8301 fehlt, das jedoch 10 μM Y-27632 und 2,5 μM CHIR99021 enthält, wie bereits in Gut-on-a-Chip aus menschlichen Organoiden2 berichtet.
HINWEIS: A8301 wurde aus dem Medium entfernt, um die Bindung der Zellen an die beschichtete ECM zu verbessern, wie bereits berichtet2. Tag 0 der Gut-on-a-Chip-Kultur erfordert nur eine obere Mikrokanalströmung. Bereiten Sie daher eine volle Spritze für den oberen Kanal und mindestens 0,2 ml für den unteren Kanal vor. - Sobald die ECM-Beschichtung abgeschlossen ist, nehmen Sie den Chip aus dem Inkubator und lösen Sie die Klemme an dem Bypass-Schlauch, der mit dem unteren Mikrokanal verbunden ist.
- Schließen Sie eine mit Impfmedium gefüllte 1-ml-Spritze an die Nadel mit stumpfem Ende an, die mit dem unteren Mikrokanal des Gut-on-a-Chip verbunden ist. Führen Sie das Aussaatmedium (~50 μl) vorsichtig in den Bypass-Schlauch ein. Sobald der Schlauch mit dem eingebrachten Medium gefüllt ist, befestigen Sie den Bypass-Schlauch, der mit dem unteren Mikrokanal verbunden ist, mit einem Binderclip.
- Lösen Sie die Klemme des Auslassschlauchs, der mit dem unteren Mikrokanal verbunden ist. Führen Sie das Aussaatmedium vorsichtig in den unteren Mikrokanal ein, damit es reibungslos durch das System fließen kann. Befestigen Sie den Auslassschlauch, der nach der Perfusion mit dem unteren Mikrokanal verbunden ist, mit einer Bindemittelklammer.
- Öffnen Sie als Nächstes den Bypassschlauch, der mit dem oberen Mikrokanal verbunden ist.
- Verbinden Sie eine mit Impfmedium gefüllte 1-ml-Spritze mit der Nadel mit dem stumpfen Ende, die mit dem oberen Mikrokanal des Gut-on-a-Chip verbunden ist. Führen Sie das Aussaatmedium (~50 μl) vorsichtig in den Bypass-Schlauch ein. Sobald der Schlauch mit dem eingebrachten Medium gefüllt ist, befestigen Sie den Bypassschlauch, der mit dem oberen Mikrokanal verbunden ist, mit einer Binderklemme.
- Lösen Sie den Auslassschlauch, der mit dem unteren Mikrokanal verbunden ist. Führen Sie das Aussaatmedium vorsichtig in den oberen Mikrokanal ein, damit es reibungslos durch das System fließen kann. Befestigen Sie den mit dem oberen Mikrokanal verbundenen Auslassschlauch nach der Perfusion mit einer Bindemittelklammer.
3. Vorbereitung der intestinalen Organoidzellen des Hundes für die Aussaat
HINWEIS: Um das Gut-on-a-Chip-Modell zu generieren, wurden in diesem Protokoll canine Dickdarm-Organoide (als Kolonoide bezeichnet) verwendet, die von Hunden mit CED-Patienten stammen. Diese Kolonoide wurden aus drei bis fünf kleinen Fragmenten von biopsiertem Dickdarmgewebe nach einer zuvor beschriebenen Methode gewonnen15,18. Um optimale Ergebnisse zu erzielen, ist es entscheidend, canine Kolonoide zu verwenden, die mindestens drei Kulturpassagen durchlaufen haben, um stabile Organoide zu etablieren, die für In-vitro-Anwendungen geeignet sind. Es wird empfohlen, die caninen Kolonoide für eine Dauer von mindestens 3-4 Tagen zu kultivieren, um eine adäquate Differenzierung von Multilineage-Zellen innerhalb der Organoide zu ermöglichen und ihre funktionelle Reife und Eignung für nachfolgende Experimente im Gut-on-a-Chip-Modell sicherzustellen. Die maximale Grenze der Passage für diese Arbeit liegt unter 20, wie aus einer früheren Studie hervorgeht, die den unveränderten Phänotyp und Karyotyp über 20 aufeinanderfolgende Passagen hinweg demonstrierte25. Die Signalisierung dieser Spender ist in der Zusatztabelle S1 dargestellt.
- Kultivierung der Hundekolonoide in 24-Well-Platten, eingebettet in 30 μl Matrigel 15,18 mit Organoid-Kulturmedium, das in der Materialtabelle aufgeführt ist, das gegenüber dem zuvor berichteten Medium 15,26,27 modifiziert ist. Das konditionierte Medium wurde durch die Kultivierung von Rspo1-Zellen und HEK293-Zellen gewonnen, die zur Sekretion von Noggin28 entwickelt wurden.
- Entsorgen Sie das organoide Kulturmedium durch Vakuumabsaugung und geben Sie 500 μl Cell Recovery Solution bei eiskalter Temperatur in jede Vertiefung. 30 Minuten bei 4 °C inkubieren.
HINWEIS: In der Regel liefern drei Vertiefungen aus einer 24-Well-Platte reifer Darmorganoide von Hunden eine ausreichende Menge an dissoziierten Zellen, um ein einzelnes Gut-on-a-Chip-Gerät mit 40-50 Organoiden/einem Sichtfeld bei 10-facher Vergrößerung zu besiedeln. - Mechanische Unterbrechung der Matrigel-Kuppeln für 5 s mit einer P1000-Mikropipette. Anschließend wird die Organoidsuspension in einem konischen 15-ml-Röhrchen gesammelt.
- Das konische Röhrchen wird bei 200 × g und 4 °C 5 min zentrifugiert, gefolgt von der Entfernung des Überstandes.
- Geben Sie 1 ml Trypsin-ähnliche Protease bei Raumtemperatur, ergänzt mit 10 μM Y-27632, und resuspendieren Sie das Zellpellet durch Pipettieren mit einer P1000-Mikropipette.
- Die Zellsuspension wird in ein Wasserbad bei 37 °C gegeben und 10 Minuten lang inkubiert, während die Mischung regelmäßig geschüttelt wird.
- Geben Sie 5 ml warmes organoides Basalmedium hinzu und pipettieren Sie die Zellsuspension mit einer P1000-Mikropipette kräftig, bis sie trüb wird, ohne sichtbare Zellklumpen.
- Passieren Sie die Zellsuspension durch ein Zellsieb mit einem Cutoff von 70 μm, um alle Matrigel-Trümmer und großen Zellcluster zu entfernen.
- Das konische Röhrchen wird bei 200 × g und 4 °C für 5 min zentrifugiert, gefolgt von einer Resuspension des Pellets im Aussaatmedium. Verwenden Sie für die Aussaat eines Gut-on-a-Chip-Geräts für Hunde 20 μl Saatmedium, um das Zellpellet zu resuspendieren (d. h. verwenden Sie 20 μl Saatmedium bei der Aussaat eines Gut-on-a-Chip-Geräts).
- Ändern Sie die Konzentration lebensfähiger Zellen auf 1 × 107 Zellen/ml mit dem zuvor berichteten Aussaatmedium2. Führen Sie eine Viabilitätsbewertung mit einem Hämozytometer durch, indem Sie 10 μl der Zellsuspension mit 10 μl Trypanblau kombinieren und dann die Zellen unter einem Mikroskop beobachten.
HINWEIS: Es ist entscheidend, die Zellkonzentration entsprechend einzustellen, um eine optimale Zelladhäsion und die Bildung einer gleichmäßigen Monoschicht auf der Chipmembran zu gewährleisten. Wenn die anfängliche Anzahl der Zellen nicht ausreicht, kann dies zu einer verzögerten oder erfolglosen Etablierung einer konfluenten Monoschicht führen. Umgekehrt können sich bei einer zu hohen Anzahl von Zellen nicht adhärente Zellen zu Klumpen innerhalb des Kanals aggregieren, was zu unerwünschten Konzentrationseffekten führt.
4. Aussaat und Bildung einer 2D-Zellmonoschicht
- Trennen Sie den Auslassschlauch für den oberen Mikrokanal. Achten Sie darauf, dass der Bypassschlauch des oberen Mikrokanals offen bleibt. Sichern Sie sowohl den Ein- als auch den Auslass des unteren Mikrokanals, indem Sie sie mit Binderklammern festklemmen.
- Mit einer P100- oder P20-Mikropipette werden 20 μl der Zellsuspension aus Protokoll 3 in das Auslassloch des oberen Mikrokanals eingeführt (Abbildung 3B Seeding).
- Sichern Sie den Bypass- und Einlassschlauch des oberen Mikrokanals, indem Sie ihn mit Binderklammern festklemmen. Befestigen Sie dann den Auslassschlauch wieder an der Auslassöffnung des oberen Mikrokanals und stellen Sie sicher, dass der Schlauch während des gesamten Prozesses offen bleibt, um zu vermeiden, dass Druck auf den oberen Mikrokanal ausgeübt wird. Befestigen Sie nach diesem Schritt den Auslassschlauch des oberen Kanals langsam mit einer Binderklammer, um ihn festzuklemmen.
- Überprüfen Sie mit einem Mikroskop, ob die Zellen gleichmäßig über den oberen Mikrokanal verteilt sind.
HINWEIS: Es ist wichtig, den Schlauch zu klemmen, um die Bewegung des Mediums innerhalb des Kanals zu stoppen und stabile statische Bedingungen zu ermöglichen, bis die gewünschte Zellanhaftung erreicht ist. - Legen Sie den Chip in einen befeuchteten CO2 - Inkubator bei 37 °C.
HINWEIS: Es dauert ca. 3 Stunden, bis sich die Darmorganoidzellen des Hundes an die ECM-Beschichtung anheften (Abbildung 3B Anhang). - Verbinden Sie die Spritze, die am oberen Mikrokanal des Gut-on-a-Chip befestigt ist, mit einer Spritzenpumpe, die sich in einem CO2 - Inkubator befindet.
HINWEIS: Spülen Sie das Medium vorsichtig mit dem Knopf der Spritzenpumpe in den Mikrokanal und entfernen Sie ungebundene Zellen. Untersuchen Sie den Chip unter dem Mikroskop, um sicherzustellen, dass alle nicht anhaftenden Zellen effektiv weggespült wurden. - Initiieren Sie den Fluss des Nährmediums mit 30 μl/h mit dem Aussaatmedium. Diese kontinuierliche Strömung gilt zunächst nur für den oberen Mikrokanal bis zum Aufbau der 2D-Monoschicht auf einem Gut-on-a-Chip. Lassen Sie für den unteren Mikrokanal die Mikrokanäle eingespannt und das Medium ungespült.
- Wechseln Sie ab dem Tag nach der Aussaat der Zellen das Kulturmedium in ein organoides Kulturmedium, das A8301 enthält und dem 10 μM Y-27632 und 2,5 μM CHIR99021 aus dem Aussaatmedium fehlen.
- Sobald die Monoschicht aufgebaut ist, wird der kontinuierliche Mediumfluss auch in den unteren Mikrokanal eingeleitet. In der Regel dauert es 2 bis 3 Tage, bis die intestinalen Organoid-Epithelzellen des Hundes epitheliale Monoschichten gebildet haben (Abbildung 3C).
5. Etablierung der 3D-Morphogenese im caninen Gut-on-a-Chip
HINWEIS: Nachdem die konfluenten Monoschichten in Gut-on-a-Chip gebildet wurden, wurde ein Mediumfluss sowohl des oberen als auch des unteren Kanals und des Zellstamms eingeführt, um die 3D-Morphogenese zur 2D-Monoschicht zu initiieren, wie in Abbildung 2 dargestellt.
- Das organoide Kulturmedium wird sowohl in den oberen als auch in den unteren Mikrokanal eingebracht, um die Entwicklung der 3D-Morphogenese in einem Gut-on-a-Chip-System zu initiieren. Um eine Scherspannung von 0,02 dyn/cm2 im bestehenden Gut-on-a-Chip-Design (d. h. Mikrokanal mit einer Höhe von 500 μm) zu erreichen, ist die Durchflussrate auf 50 μl/hzu erhöhen 29.
- Initiieren Sie 10 % des Zellstamms und 0,15 Hz Frequenz, wie vor2 empfohlen, unter Verwendung eines computergesteuerten Bioreaktors, der zyklische Stämme auf in vitro kultivierte Zellen anwendet. Bei diesem Verfahren wird das Gut-on-a-Chip-Gerät mit einem Vakuum abgesaugt.
- Halten Sie diese Kulturbedingungen mindestens 2 bis 3 Tage lang aufrecht. Die 3D-Morphogenese der intestinalen Monoschicht des Hundes erfolgt in der Regel 2 bis 3 Tage nach Beginn des Duralkanalflusses und der Vakuumabsaugung (Abbildung 3C).
6. Charakterisierung von caninem Gut-on-a-Chip
- Bildgebung lebender Zellen
- Entfernen Sie alle Luftblasen im Gut-on-a-Chip, indem Sie das Medium vorsichtig mit der Spritzenpumpe fließen lassen.
- Trennen Sie das Gut-on-a-Chip-Gerät von der Spritzenpumpe.
HINWEIS: Vermeiden Sie alle Manöver, die Druck auf den Gut-on-a-Chip ausüben können. - Positionieren Sie das Gerät auf einem Mikroskop, um Bilder des etablierten 3D-Epithels aufzunehmen. Um die Struktur der 3D-Epithelschichten mit Phasenkontrast zu visualisieren, verwenden Sie 10x- und 20x-Objektive (Abbildung 3C).
- Immunfluoreszenz-Färbung
- Bereiten Sie die Blockierungslösung vor, indem Sie 1 g BSA in 50 ml Phosphatpuffersalzlösung (PBS) auflösen, um 2 % BSA herzustellen. Die Lösung wird zur Filtration durch einen Spritzenfilter von 0,2 μm geleitet. Bewahren Sie diese Lösung bei 4 °C auf.
- Bereiten Sie die permeabilisierende Lösung vor, indem Sie 150 μl Triton X-100 mit 50 ml blockierender Lösung kombinieren, was zu einer Endkonzentration von 0,3 % Triton X-100 führt. Die Lösung wird zur Filtration durch einen Spritzenfilter von 0,2 μm geleitet. Bewahren Sie diese Lösung bei 4 °C auf.
- Führen Sie die Fixierung der Zellen durch, indem Sie 100 μl eines 4%igen PFA sowohl in den oberen als auch in den unteren Mikrokanal injizieren, wie in den Schritten 2.4-2.11 beschrieben.
- Spülen Sie die Zellen, indem Sie 100 μl PBS in die oberen und unteren Mikrokanäle injizieren, wie in den Schritten 2.4-2.11 beschrieben.
- Permeabilisierung der Zellen durch Injektion von 100 μl 0,3%igem Triton in den oberen und unteren Mikrokanal, wie in den Schritten 2.4-2.11 beschrieben. Stellen Sie das Gerät 30 Minuten lang auf RT.
- Spülen Sie die Zellen, indem Sie 100 μl PBS in die oberen und unteren Mikrokanäle injizieren, wie in den Schritten 2.4-2.11 beschrieben.
- Führen Sie eine Blockierung der Zellen durch, um eine unspezifische Bindung zu verhindern, indem Sie 100 μl 2% BSA sowohl in den oberen als auch in den unteren Mikrokanal injizieren, wie in den Schritten 2.4-2.11 beschrieben. Stellen Sie das Gerät 1 Stunde lang auf RT.
- Injizieren Sie 20 μl der in 2 % BSA verdünnten Primärantikörperlösung und stellen Sie sie für 3 h auf RT, gefolgt von einer nächtlichen Inkubation bei 4 °C.
HINWEIS: Stellen Sie sicher, dass alle Schläuche während der Inkubation bei 4 °C für die Nacht eingeklemmt sind. Die Konzentration eines Primärantikörpers sollte 2-5 mal höher sein als die empfohlene Konzentration für die Monolayer- oder 3D-Organoid-Färbung (Ergänzende Abbildung S1). - Spülen Sie die Zellen, indem Sie 100 μl PBS sowohl in den oberen als auch in den unteren Mikrokanal injizieren, wie in den Schritten 2.4-2.11 beschrieben.
- Injizieren Sie 20 μl der in 2 % BSA verdünnten Sekundärantikörperlösung und geben Sie sie 1 h lang auf RT.
HINWEIS: Stellen Sie sicher, dass alle Schläuche während der Inkubation eingeklemmt sind. Ausgehend von diesem Schritt ist es notwendig, den Geräteaufbau mit Aluminiumfolie abzuschirmen, um ein Ausbleichen zu verhindern. - Spülen Sie die Zellen, indem Sie 100 μl PBS sowohl in den oberen als auch in den unteren Mikrokanal injizieren, wie in den Schritten 2.4-2.11 beschrieben.
- Bereiten Sie eine kombinierte Lösung für die F-Aktin- und DAPI-Gegenfärbung (Diamidino-2-phenylindol) vor. Injizieren Sie 20 μl der kombinierten Lösung in den oberen und unteren Mikrokanal, wie in den Schritten 2.4-2.11 beschrieben.
- Spülen Sie die Zellen, indem Sie 100 μl PBS in die oberen und unteren Mikrokanäle injizieren, wie in den Schritten 2.4-2.11 beschrieben.
Fluoreszenzbildgebung der Architektur der 3D-Epithelzellen mit einem Fluoreszenzmikroskop oder einem konfokalen Mikroskop durchführen.
7. Funktion der epithelialen Barriere
- Entfernen Sie alle Luftblasen im Gut-on-a-Chip, indem Sie das Medium vorsichtig mit der Spritzenpumpe fließen lassen. Stellen Sie sicher, dass alle Schläuche während der Messung geöffnet sind.
- Entfernen Sie den Gut-on-a-Chip aus der Spritzenpumpe und stellen Sie ihn für mindestens 10 Minuten auf RT.
- Entfernen Sie die Ag/AgCl-Elektroden aus der 70%igen EtOH-Lösung.
- Platzieren Sie zwei Ag/AgCl-Elektroden in den oberen bzw. unteren Einlass, um den Widerstand der Epithelschicht mit einem Multimeter zu messen.
- Platzieren Sie zwei Ag/AgCl-Elektroden in den unteren Einlass bzw. oberen Auslass. Geben Sie den Mittelwert dieser beiden Werte als Widerstandswert für den Gut-on-a-Chip an.
HINWEIS: Der Blind-TEER sollte auf einem Gut-on-a-Chip mit nur ECM-Beschichtung ohne Epithel gemessen werden. - Die Berechnung des transepithelialen elektrischen Widerstands (TEER) (kΩ × cm2) kann mit Gleichung (1) berechnet werden.
TEER = (Ωt - Ωleer) × A (1)
Dabei ist Ωt ein Widerstandswert, der zu einem bestimmten Zeitpunkt seit Beginn des Experiments gemessen wird, ΩLeerwert ist ein Widerstandswert , der zu diesem Zeitpunkt ohne das Epithel gemessen wird, und A ist der Bereich der Oberfläche, der von einer Zellschicht bedeckt ist (ungefähr 0,11cm2 für dieses Gut-on-a-Chip-Design29).- Berechnen Sie den normalisierten TEER mit Gleichung (2).
TEER =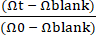 (2)
(2)
Dabei ist Ω0 ein Widerstandswert zum Zeitpunkt des ersten Ablesens des Experiments, wie zuvor berichtet30 (Abbildung 4C).
- Berechnen Sie den normalisierten TEER mit Gleichung (2).
Repräsentative Ergebnisse
Dieses Protokoll ermöglicht zuverlässig die spontane Entwicklung der 3D-Darmmorphogenese in einem Gut-on-a-Chip-System. Bei diesem Ansatz werden canine Darmepithelzellen verwendet, die aus Darmorganoiden von Hunden gewonnen werden, die von chronisch entzündlichen Darmerkrankungen (CED) betroffen sind (Abbildung 1B). Eine gelegentliche Häufung der 3D-Morphogenese von intestinalen Epithelzellen von Hunden kann nach 6-9 Tagen Mediumfluss im gesamten Mikrokanal beobachtet werden (Abbildung 3C). Diese morphologischen Veränderungen können mit Hilfe von Phasenkontrasttechniken überwacht werden. In dieser Studie verwendeten wir Organoide, die von zwei Hunden stammten, bei denen CED diagnostiziert wurde. Bemerkenswert ist, dass eine erfolgreiche 3D-Morphogenese in zwei biologischen Replikaten beobachtet wurde, die jeweils mit zwei technischen Replikaten durchgeführt wurden. Die Ergebnisse dieser Studie bilden eine Grundlage für zukünftige Untersuchungen mit Darmorganoiden, die von anderen Hundespendern stammen. Diese Ergebnisse demonstrieren die potenzielle Anwendbarkeit und Wiederholbarkeit unseres experimentellen Ansatzes, über den bereits in menschlichen Proben berichtet wurde. Diese Ergebnisse sind eine weitere Bestätigung dafür, dass die Gut-on-a-Chip-Technologie auf Darmepithelzellen von Hunden anwendbar ist, wie bereits in den Studien mit menschlichen Darmepithelzellen berichtetwurde 2.
Dieses Protokoll zeigte, dass die Immunfluoreszenzfärbung verwendet werden kann, um die 3D-Struktur von Organoid-abgeleiteten Monoschichten, die zottenartige Strukturen in mikrofluidischen Chips gebildet haben, mit konventioneller Fluoreszenzmikroskopie zu bewerten (Abbildung 4 A,B). Dieses Protokoll kann angepasst werden, um die differenzierten und räumlich organisierten zellulären Phänotypen durch Immunfluoreszenzfärbung zu validieren. Die Visualisierung einer 3D-Morphogenese innerhalb eines Gut-on-a-Chip bietet eine hervorragende Möglichkeit, die Wirtsantwort während verschiedener pathologischer Interaktionen zu untersuchen 14,23,31. In Kombination mit Epithelzellen, die von Patientenspendern stammen, wie sie zuvor beim Menschen beschrieben wurden, kann diese Technologie genutzt werden, um personalisierte Modelle von Darmerkrankungen zu erstellen13. Durch die Integration von Immunfluoreszenz-Bildgebung mit gezielten RNA-Visualisierungstechniken wie der Fluoreszenz-in-situ-Hybridisierung kann es möglich sein, die Transkriptome und Proteome des Wirts innerhalb eines Gut-on-a-Chip-Systems visuell zu analysieren.
Die Erhaltung der Integrität der Darmmembran ist für die Aufrechterhaltung der Darmhomöostase von entscheidender Bedeutung, und die Gut-on-a-Chip-Plattform bietet einen wertvollen Vorteil, indem sie eine präzise Überwachung und Quantifizierung dieser entscheidenden Funktion ermöglicht. Die Messung von TEER mit der Gut-on-a-Chip-Technologie bietet mehrere Vorteile. Zum Beispiel haben frühere Studien TEER erfolgreich während der Co-Kultivierung von Darmzellen mit nicht-pathogenen und probiotischen Bakterien32 sowie unter Leaky-Gut-Bedingungen23 untersucht. Dies ermöglicht es den Forschern, die Auswirkungen verschiedener Erkrankungen auf die Barrierefunktion des Darms zu untersuchen und potenzielle Interventionen zur Förderung der Darmgesundheit zu identifizieren.
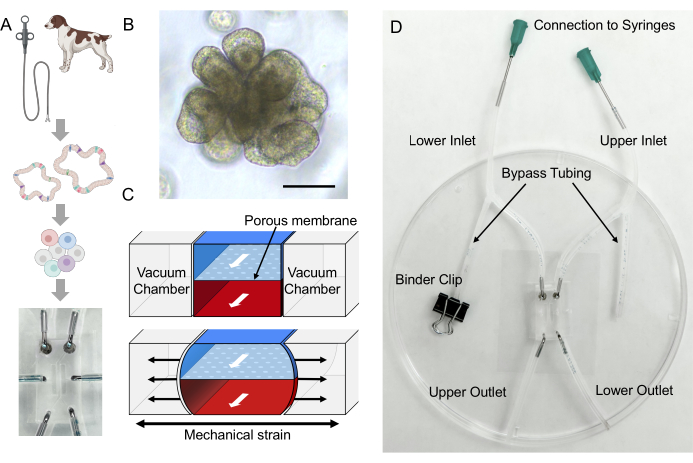
Abbildung 1: Etablierung von patienteneigenen CED Gut-on-a-Chip. (A) Integration der patienteneigenen Darmorganoide und der Gut-on-a-Chip-Plattform. Eine endoskopische Biopsie kann durchgeführt werden, um Darmkryptenzellen zu isolieren, um spenderspezifische Darmorganoide zu entwickeln. Epithelzellen können von den Organoiden in einzelne Zellen dissoziiert, dann in ein PDMS-basiertes Gut-on-a-Chip ausgesät und in einer einzigartigen dynamischen Mikroumgebung kultiviert werden. (B) Repräsentative Bilder von Kolonoiden eines CED-Hundes. Maßstabsbalken = 100 μm. (C) Dieses Schema zeigt ein Gut-on-a-Chip-Gerät, das aus einer porösen Membran besteht, die zwischen dem oberen und dem unteren Mikrokanal platziert ist. Der obere Mikrokanal wird durch den blauen Bereich angezeigt, während der untere Mikrokanal durch den roten Bereich gekennzeichnet ist. Auf jeder Seite des Mikrokanals sind Vakuumkammern vorhanden, die die poröse Membran verformen, um die peristaltische Bewegung24 nachzuahmen. (D) Ein Aufbau von Hunde-Gut-on-a-Chip umfasst einen PDMS-basierten Gut-on-a-Chip, der mit einem Schlauch zusammengebaut ist, der auf einem Deckglas 2,24 platziert ist. Der Bypass-Schlauch ist entscheidend, um zu vermeiden, dass sich während der Handhabung (d. h. beim Anschließen an Spritzen) Druck im Mikrokanal aufbaut. Zum Einklemmen der Schläuche werden Binderklammern verwendet. Volumenempfindliche Materialien können über die offenen Löcher des oberen oder unteren Auslasses infundiert werden. Organoides Kulturmedium kann infundiert werden, indem die Spritzen mit den Nadeln mit dem stumpfen Ende und dem Durchfluss durch den oberen und unteren Einlass verbunden werden. Abkürzungen: CED = entzündliche Darmerkrankung; PDMS = Polydimethylsiloxan. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
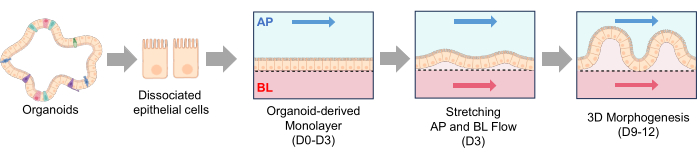
Abbildung 2: Bildung von zottenartigen Strukturen in caniner CED Gut-on-a-Chip. Die dissoziierten Epithelzellen wurden in einem ECM-beschichteten Gut-on-a-Chip ausgesät. Sobald die dissoziierten Zellen an die PDMS-Membran gebunden waren, wurde der apikale Fluss für 3 Tage eingeleitet (D0-D3). Wenn eine konfluierende 2D-Monoschicht gebildet wird (D3), wird ein basolateraler Fluss mit häufigem Dehnen eingeleitet (Stretching, AP und BL Flow). Nach 2-3 Tagen dualer Strömung und Membrandehnung beginnt die 2D-Monoschicht eine 3D-Morphogenese zu entwickeln, und nach 9 Tagen Kultur werden zottenartige Strukturen gebildet (3D-Morphogenese, D9-D12). Abkürzungen: ECM = extrazelluläre Matrix; PDMS = Polydimethylsiloxan; AP = apikal; BL = basolateral. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
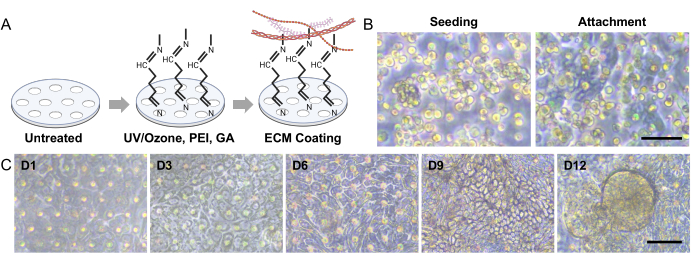
Abbildung 3: Aussaat von intestinalen Organoiden und 3D-Morphogenese in einem Gut-on-a-Chip. (A) Die experimentellen Schritte zur Oberflächenaktivierung einer porösen Membran in einem PDMS-basierten Gut-on-a-Chip. Die Verwendung von UV/Ozon-Behandlung, PEI- und GA-Behandlung in Verbindung erleichtert die Vernetzung von Aminen, die in ECM-Lösungen vorhanden sind. Dieser Prozess führt zu einer stabilen Immobilisierung von EZM-Proteinen auf der porösen Membran. (B) Die Phasenkontrastbilder zeigen die Morphologien der Zellen unmittelbar nach der Aussaat (links) und 3-5 h nach der Aussaat (rechts). Die poröse Membran zeigt nach 3 Stunden Aussaat dünnere und dunklere Bereiche, an denen sich einzelne Zellen angeheftet haben, was den Anheftungsprozess unterstreicht. (C) Die Phasenkontrastbilder zeigen die 3D-Morphogenese von Darmmonolayern innerhalb eines Gut-on-a-Chip-Systems. Diese Monoschichten stammten von Hunden, die an CED erkrankt waren, und diese Organoidzellen wurden über einen Zeitraum von 12 Tagen unter dynamischen Bedingungen kultiviert, die Flüssigkeitsfluss und Dehnungsbewegungen beinhalteten. Maßstabsbalken = 50 μm (B,C). Abkürzungen: CED = entzündliche Darmerkrankung; ECM = extrazelluläre Matrix; PDMS = Polydimethylsiloxan; PEI = Polyethylenimin; GA = Glutaraldehyd. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
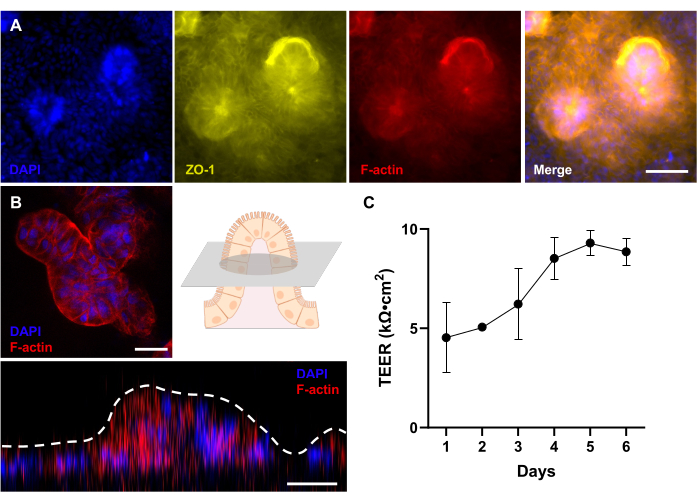
Abbildung 4: Evaluierung der 3D-morphologischen Entwicklung bei patienteneigenen Hunden Gut-on-a-Chip. (A) Immunfluoreszenz-Bildgebung eines caninen IBD-Gut-on-a-Chips, die eine Draufsicht auf ein voll entwickeltes 3D-Epithel nach 12 Tagen Kultur zeigt, das mit einem Fluoreszenzmikroskop ausgewertet wird. Das Tight-Junction-Protein (ZO-1) ist gelb dargestellt; die Pinselrandmembran (F-Aktin) erscheint rot; und die Kerne sind mit DAPI gefärbt und erscheinen blau. (B) Immunfluoreszenz-Bildgebung eines IBD-Gut-on-a-Chip bei Hunden unter Verwendung eines konfokalen Mikroskops mit einer Fernlinse. Wie im Schema gezeigt, ist ein Fluoreszenzbild eines Querschnitts einer zottenartigen Struktur eines voll entwickelten 3D-Epithels nach 12 Tagen Kultur zu sehen. Darüber hinaus zeigt das Z-Stacking eine Seitenansicht des 3D-Epithels, die die Bildung von zottenartigen Strukturen zeigt. Die Pinselrandmembran (F-Aktin) erscheint rot, und die Zellkerne sind mit DAPI gefärbt und erscheinen blau. (C) Die intestinale Barrierefunktion wurde mittels TEER in von Patienten stammenden Gut-on-a-Chips von Hunden evaluiert und gemessen. Stabile TEER-Werte wurden an Tag 5 der Kultur auf Gut-on-a-Chip erreicht. Fehlerbalken drücken das REM der Messungen aus. Der TEER-Wert wurde zwischen zwei biologischen Replikaten und einem technischen Replikat gemessen. Maßstabsbalken = 50 μm (A), 25 μm (B). Abkürzungen: CED = entzündliche Darmerkrankung; DAPI = 4',6-Diamidino-2-phenylindol; TEER = transepithelialer elektrischer Widerstand. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung S1: Charakterisierung von polyklonalen Anti-ZO-1-Antikörpern in Monolayern aus dem Dickdarm von Hunden und auf Gut-on-a-Chip-Geräten. (A) Immunfluoreszenzfärbung von ZO-1 in Gelb mit F-Aktin in Rot und deren Überlagerungsbild. Maßstabsbalken = 25 μm. (B) Die Immunfluoreszenzvisualisierung von ZO-1 in Gelb in "Leaky Gut Chips" wurde unter verschiedenen Bedingungen durchgeführt, einschließlich Stimulation mit probiotischen Bakterien (LGG + Zytokine oder VSL#3 + Zytokine) und keimfreien Kontrollen ohne probiotische Stimulation (Zytokine). Maßstabsbalken = 50 μm. Diese Abbildung ist reproduziert von Min et al.23. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S1: Eine Zusammenfassung der Informationen über Gewebespender. Eine zusammenfassende Tabelle mit dem Alter, dem Geschlecht, der Rasse, der histopathologischen Bewertung und dem CCD-Aktivitätsindex (CIBDAI) der Spender. Das CIBDAI ist ein numerisches Scoring-System, das verwendet wird, um den klinischen Schweregrad bei caniner CED33 abzuleiten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Diese Studie markiert den bahnbrechenden Nachweis der Kompatibilität von Darmorganoiden bei Hunden mit der Entwicklung eines IBD-Gut-on-a-Chip-Modells für Hunde. Die Integration von Darmorganoiden und aus Organoiden gewonnenen Monolayer-Kulturen in ein mikrofluidisches System (d. h. Gut-on-a-Chip-System) hat die Technologie weiterentwickelt und die Erstellung von In-vitro-Darmmodellen ermöglicht, die die physiologische Dynamik genau nachahmen und für biologische Bedingungen repräsentativer sind. Insbesondere, da es nur sehr wenige Berichte über Gut-on-a-Chip-Kulturen mit IBD-abgeleiteten Organoiden beim Menschen gibt, könnte die aktuelle Studie mit CED-abgeleitetem Gut-on-a-Chip bei Hunden führende Einblicke in die Untersuchung von CED beim Menschen liefern.
Die erfolgreiche Entwicklung der 3D-Morphogenese des Darmepithels bei Hunden auf einem Gut-on-a-Chip erfordert die sorgfältige Beachtung mehrerer kritischer Schritte. Erstens kann die hydrophobe Oberfläche von mikrofluidischen PDMS-Kanälen die ECM-Adhäsion und die anschließende Zellanhaftung behindern, was eine Oberflächenaktivierung des PDMS vor der ECM-Beschichtung und der Zellaussaat erforderlich macht (siehe Protokollabschnitt 1). Um eine stabile Monolayer-Kultur zu erreichen, ist die Entfernung überschüssiger ungebundener Zellen nach der Zelladhäsion entscheidend (Protokollschritte 4.6-4.7). Darüber hinaus ist für die 3D-Morphogenese des Darmepithels eine dynamische Stimulation, wie z. B. ein konstanter Medienfluss und eine peristaltikartige Vakuumbewegung, erforderlich (Protokollschritt 5.2). Eine sorgfältige Handhabung ist unerlässlich, um Luftblasen im Mikrokanal bei allen Schritten der Gut-on-a-Chip-Kultur zu vermeiden.
Wenn Sie auf eine schlechte Zellaussaat im Gut-on-a-Chip stoßen, könnte dies auf eine niedrige Zellzahl oder eine schlechte Zellanhaftung zurückzuführen sein. Um eine niedrige Zellzahl zu beheben, ist es wichtig, die Gesundheit der präparierten Darmorganoide zu überprüfen, indem man ihr Wachstum in Matrigel beobachtet. Die Zelllebensfähigkeit kann durch Trypanblau-Färbung nach der Zelldissoziation beurteilt werden, um sicherzustellen, dass nicht mehr als 20 % der Zellen tot sind. Wenn die Anzahl der lebensfähigen Zellen nicht ausreicht, kann versucht werden, die Bedingungen des organoiden Mediums zu optimieren. Eine weitere Möglichkeit ist eine unvollständige Organoid-Dissoziation, die zu einem Überschuss an Zellklumpen führt, die größer als 70 μm sind und vom Filter eingeschlossen werden. Um dieses Problem zu lösen, besteht eine Möglichkeit darin, die Dauer des Pipettierens während der Zelldissoziation zu verlängern. Alternativ kann das konische 15-ml-Röhrchen während der Behandlung mit einer Trypsin-ähnlichen Protease minütlich sanft bewegt werden. Eine schlechte Zellanhaftung am Gut-on-a-Chip kann auf eine unsachgemäße ECM-Beschichtung zurückzuführen sein. Während des Beschichtungsprozesses wird empfohlen, sorgfältig auf das Vorhandensein von Luftblasen zu prüfen und deren Bildung zu verhindern, indem bei Bedarf vorsichtig mehr Beschichtungslösung hinzugefügt wird. Eine Überfüllung der Zellen und das Versäumnis, ungebundene Zellen wegzuspülen, kann zu einer unzureichenden anfänglichen Monoschicht führen. In einem solchen Fall kann ein leichtes Pulsieren beim Drücken des Spritzenkolbens angewendet werden. Diese Schritte zur Fehlerbehebung können dazu beitragen, Probleme während des Gut-on-a-Chip-Kulturprozesses zu identifizieren und zu beheben.
Während diese Gut-on-a-Chip-Plattform die Erzeugung von wellenförmigen 3D-Epithelschichten ermöglicht, erkennen wir die Notwendigkeit zusätzlicher biologischer Komplexität, um die Darmmikroumgebung vollständig zu replizieren. Es ist von entscheidender Bedeutung, die Wechselwirkungen zwischen Epithel- und Mesenchymzellen, die Ablagerung von EZM für die 3D-Regeneration und das Vorhandensein von Kryptenzottenmerkmalen zu berücksichtigen, die eine geeignete Stammzellnische bilden. Stromazellen, wie z.B. Fibroblasten, spielen eine wichtige Rolle bei der Produktion von EZM-Proteinen und der Regulation der Darmmorphogenese34,35,36. Die Einbeziehung mesenchymaler Zellen in dieses Modell hat das Potenzial, sowohl die Morphogenese als auch die Effizienz der Zelladhäsion zu verbessern. Endothelschichten, die Kapillargefäße und Lymphgefäße umfassen, spielen eine entscheidende Rolle bei der Steuerung des molekularen Transports und der Rekrutierung von Immunzellen37,38. Die Einbeziehung von patienteneigenen Immunzellen könnte für die Modellierung von Darmerkrankungen von entscheidender Bedeutung sein, da sie den Nachweis des Zusammenspiels zwischen angeborener und adaptiver Immunität sowie die Etablierung einer gewebespezifischen Immunität ermöglicht39. Nach Abschluss der 3D-Morphogenese auf Gut-on-a-Chip kann das organoide Kulturmedium zu einem organoiden Differenzierungsmedium modifiziert werden. Dies kann ein praktikabler Ansatz sein, um eine zusätzliche zelluläre Differenzierung zu induzieren, abhängig von den experimentellen Zielen.
Die Abbildung der 3D-Mikroarchitektur in situ ist aufgrund des erforderlichen großen Arbeitsabstandes, der mit einem Fernobjektiv überwunden werden kann, eine Herausforderung. Darüber hinaus erschweren die schichtweise Mikrofabrikations- und Verklebungsmethoden den Zugang zu den oberen Schichten für die Untersuchung mit dem REM. Für das aktuelle Gut-on-a-Chip-Design wird eine Spritzenpumpe pro Gut-on-a-Chip-Mikrogerät benötigt, was Platz im CO2 -Inkubator belegt und groß angelegte Experimente verhindert. Innovationen sind erforderlich, um die Skalierbarkeit für eine benutzerfreundliche Plattform und ein Hochdurchsatz-Screening zu erhöhen.
Diese aktuellen Protokolle ermöglichen die spontane Entwicklung von 3D-Epithelschichten in vitro und übertreffen damit die Grenzen herkömmlicher 3D-Organoide, 2D-Monoschichten und statischer Mikrogeräte-Kultursysteme. Diese dynamische in-vitro-Darmmikroumgebung kann durch die Einführung von Kokulturen verschiedener Zelltypen kontrolliert werden. Frühere Studien haben Methoden zur Manipulation der Gut-on-a-Chip-Mikroumgebung untersucht, einschließlich der Kokultivierung des Darmmikrobioms14,23 und der peripheren mononukleären Zellen30. Diese rekonstituierte Mikroumgebung hat zahlreiche potenzielle Anwendungen, darunter Arzneimitteltests, grundlegende mechanistische Studien und Krankheitsmodellierung. Die rekonstruierte Mikroumgebung birgt ein erhebliches Potenzial für eine Vielzahl von Anwendungen, wie z.B. Arzneimitteltests 23,40,41 und Krankheitsmodellierung 12,13,14,30 sowie grundlegende mechanistische Untersuchungen der Darmmorphogenese 42. Eine Vielzahl von Assays kann entweder durch Sammeln von Überständen zur Bewertung von Metaboliten 43, durch Sammeln von Zellen zur genomischen Untersuchung 2,32 oder durch visuelle Untersuchung der Zellen unter Verwendung von Lebendzellfarbstoffen oder Fixierung für die anschließende Immunfluoreszenzbildgebung 23, 44 durchgeführt werden.
In dieser Studie wird ein reproduzierbares Protokoll für die Entwicklung der 3D-Morphogenese von Darmepithelschichten bei Hunden in einer Gut-on-a-Chip-Plattform vorgestellt. Die resultierende 3D-Epithelstruktur bietet eine realistischere Darstellung der Darmmikroumgebung, die ein immenses Potenzial für Anwendungen in verschiedenen biomedizinischen Studien hat. Durch die Nutzung dieser Darmarchitektur können wir mehr translationale Forschung betreiben und möglicherweise vielversprechende Ergebnisse erzielen.
Offenlegungen
Die Autoren haben keinen Interessenkonflikt zu erklären.
Danksagungen
Wir danken dem WSU Small Animal Internal Medicine Service (Dr. Jillian Haines, Dr. Sarah Guess, Shelley Ensign LVT) und der WSU VTH Clinical Studies Coordinator Valorie Wiss für ihre Unterstützung bei der Rekrutierung von Fällen und der Probenentnahme von Citizen Scientists (Patientenspendern). Diese Arbeit wurde teilweise vom Büro des Direktors, den National Institutes Of Health (K01OD030515 und R21OD031903 to Y.M.A.) und dem Japan Society for the Promotion of Science Overseas Challenge Program for Young Researchers (202280196 to I.N.) unterstützt. Abbildung 1A und Abbildung 3A wurden mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| Organoid basal medium | |||
| Advanced DMEM/F12 | Gibco | 12634-010 | |
| GlutaMAX | Gibco | 35050-061 | 2 mM, glutamine substitute |
| 1 M HEPES | VWR Life Science | J848-500ML | 10 mM |
| 100x penicillin–streptomycin | Corning | MT30009CI | 1x |
| Organoids and organoid medium | |||
| A-83-01 | PeproTech | 9094360 | 500 nM |
| B27 supplement | Gibco | 17504-044 | 1x |
| CHIR99021 | Reprocell | 04-0004-base | 2.5 µM |
| HEK293 cells engineered to secrete Noggin | Baylor College of Medicine | ||
| Murine EGF | PeproTech | 315-09-1MG | 50 ng/mL |
| Murine Wnt-3a | PeproTech | 315-20-10UG | 100 ng/mL |
| N-Acetyl-L-cysteine | Sigma | A9165-25G | 1 mM |
| N2 MAX Media supplement | Gibco | 17502-048 | 1x |
| Nicotinamide | Sigma | N0636-100G | 10 mM |
| Noggin Conditioned Medium | NA | NA | 10% vol/vol |
| Primocin | InvivoGen | ant-pm-1 | 100 µg/ml |
| R-spondin1 (Rspo1) cells | Trevigen | 3710-001-01 | Rspo1 cells |
| R-Spondin-1 Conditioned Medium | NA | NA | 20% vol/vol |
| SB202190 | Sigma-Aldrich | S7067-25MG | 10 µM |
| Y-27632 | StemCellTechnologies | 72308 | 10 µM |
| [Leu15 ]-Gastrin I human | Sigma-Aldrich | G9145-.5MG | 10 nM |
| Reagents | |||
| 4% Paraformaldehyde solution | Fisher Scientific | AAJ19943K2 | |
| Alexa Fluor 647 Phalloidin | Thermo Fisher Scientific | A22287 | x250 dilution |
| Anti-Rabbit IgG H&L labeled with Alexa Fluor 555 | Abcam | ab150078 | x1,000 dilution |
| Anti-ZO-1 polyclonal antibody | Thermo Fisher Scientific | 61-7300 | x50 dilution |
| Cell Recovery Solution | Corning | 354253 | |
| Collagen I, Rat Tail 3 mg/mL | Gibco | A10483-01 | |
| Diamidino-2-phenylindole (DAPI) | Thermo Fisher Scientific | 62248 | x1,000 dilution |
| EMS Glutaraldehyde Aqueous 50% | Electron Microscopy Sciences | 16320 | |
| Matrigel Matrix | Corning | 356255 | |
| Poly(ethyleneimine) solution | Sigma | 408700-250ML | |
| TrypLE Express | Gibco | 12604-021 | |
| Materials and Equipment | |||
| 24-well culture plates | Corning | 3524 | |
| 87V Industrial Multimeter | Fluke Corporation | ||
| Centrifuge | Eppendorf | 5910R | |
| CO2 incubator | Eppendorf | C170i | |
| DMi8 fluorescence microscope | Leica microsystems | DMi8 | |
| Dry oven | Fisher Scientific | 15-103-0519 | |
| FlexCell FX-5000 Tension system | Flexcell International Corporation | ||
| Inverted phase-contrast microscope | Leica microsystems | DMi1 | |
| SP8-X inverted confocal microscope | Leica microsystems | SP8-X | |
| Syringe pump | Braintree Scientific | model no. BS-8000 120V | |
| Syringe, 3 mL sterile | BD Biosciences | 14-823-435 | |
| Syringes, 1 mL sterile | BD Biosciences | 14-823-434 | |
| UV/ozone generator | Jelight Company | model no. 30 | |
| Software | |||
| LAS X imaging software | Leica microsystems |
Referenzen
- Shanks, N., Greek, R., Greek, J. Are animal models predictive for humans. Philosophy, ethics, and humanities in medicine: PEHM. 4, 2 (2009).
- Shin, W., Kim, H. J. 3D in vitro morphogenesis of human intestinal epithelium in a gut-on-a-chip or a hybrid chip with a cell culture insert. Nature protocols. 17 (3), 910-939 (2022).
- Coelho, L. P., et al. Similarity of the dog and human gut microbiomes in gene content and response to diet. BMC Biome. 6 (72), 1-11 (2018).
- German, A. J. The growing problem of obesity in dogs and cats. The Journal of Nutrition. 136, 1940-1946 (1940).
- Patronek, G. J., Waters, D. J., Glickman, L. T. Comparative longevity of pet dogs and humans: Implications for gerontology research. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 52 (3), B171-B178 (1997).
- Lutz, T. A. Mammalian models of diabetes mellitus, with a focus on type 2 diabetes mellitus. Nature reviews. Endocrinology. 19 (6), 350-360 (2023).
- Allenspach, K., Culverwell, C., Chan, D. Long-term outcome in dogs with chronic enteropathies: 203 cases. Veterinary Record. 178 (15), 368 (2016).
- Patnaik, A. K., Hurvitz, A. I., Johnson, G. F. Canine gastrointestinal neoplasms. Veterinary Pathology. 14 (6), 547-555 (1977).
- Saito, T., et al. Immunohistochemical analysis of beta-catenin, e-cadherin and p53 in canine gastrointestinal epithelial tumors. Journal of Veterinary Medical Science. 82 (9), 1277-1286 (2020).
- Kopper, J. J., et al. Harnessing the biology of canine intestinal organoids to heighten understanding of inflammatory bowel disease pathogenesis and accelerate drug discovery: A One Health approach. Frontiers in Toxicology. 3, 1-13 (2021).
- Jalili-firoozinezhad, S., et al. A complex human gut microbiome cultured in an anaerobic intestine-on-a-chip. Nature Biomedical Engineering. 3 (7), 520-531 (2019).
- Shin, W., et al. Robust formation of an epithelial layer of human intestinal organoids in a polydimethylsiloxane-based Gut-on-a-Chip microdevice. Frontiers in Medical Technology. 2, (2020).
- Shin, Y. C., et al. Three-dimensional regeneration of patient-derived intestinal organoid epithelium in a physiodynamic mucosal Interface-on-a-Chip. Micromachines. 11 (7), 663 (2020).
- Tovaglieri, A., et al. Species-specific enhancement of enterohemorrhagic E. coli pathogenesis mediated by microbiome metabolites. Microbiome. 7 (1), 43 (2019).
- Chandra, L., et al. Derivation of adult canine intestinal organoids for translational research in gastroenterology. BMC Biology. 17 (1), 1-21 (2019).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Wilson, S. S., Tocchi, A., Holly, M. K., Parks, W. C., Smith, J. G. A small intestinal organoid model of non-invasive enteric pathogen-epithelial cell interactions. Mucosal Immunology. 8 (2), 352-361 (2015).
- Ambrosini, Y. M., et al. Recapitulation of the accessible interface of biopsy-derived canine intestinal organoids to study epithelial-luminal interactions. PLoS ONE. 15 (4), 1-17 (2020).
- Gabriel, V., et al. Canine intestinal organoids in a dual-chamber permeable support system. Journal of Visualized Experiments. 2 (181), 1-24 (2022).
- Yamada, A., et al. Confronting emerging zoonoses: the one health paradigm. Confronting Emerging Zoonoses: The One Health Paradigm. , 1 (2014).
- Lerner, H., Berg, C. The concept of health in One Health and some practical implications for research and education: what is One Health. Infection Ecology & Epidemiology. 5 (1), 25300 (2015).
- Garcia, S. N., Osburn, B. I., Jay-Russell, M. T. One Health for food safety, food security, and sustainable food production. Frontiers in Sustainable Food Systems. 4, 1-9 (2020).
- Min, S., et al. Live probiotic bacteria administered in a pathomimetic Leaky Gut Chip ameliorate impaired epithelial barrier and mucosal inflammation. Scientific Reports. 12 (1), 22641 (2022).
- Kim, H. J., Huh, D., Hamilton, G., Ingber, D. E. Human gut-on-a-chip inhabited by microbial flora that experiences intestinal peristalsis-like motions and flow. Lab on a Chip. 12 (12), 2165-2174 (2012).
- Qu, M., et al. Establishment of intestinal organoid cultures modeling injury-associated epithelial regeneration. Cell Research. 31 (3), 259-271 (2021).
- Sahoo, D. K., et al. Differential transcriptomic profiles following stimulation with lipopolysaccharide in intestinal organoids from dogs with inflammatory bowel disease and intestinal mast cell tumor. Cancers. 14 (14), 3525 (2022).
- Gabriel, V., et al. Standardization and maintenance of 3D canine hepatic and intestinal organoid cultures for use in biomedical research. Journal of Visualized Experiments. (179), 1-28 (2022).
- Heijmans, J., et al. ER stress causes rapid loss of intestinal epithelial stemness through activation of the unfolded protein response. Cell reports. 3 (4), 1128-1139 (2013).
- Shin, W., et al. A robust longitudinal co-culture of obligate anaerobic gut microbiome with human intestinal epithelium in an anoxic-oxic interface-on-a-chip. Frontiers in Bioengineering and Biotechnology. 7, 1-13 (2019).
- Shin, W., Kim, H. J. Intestinal barrier dysfunction orchestrates the onset of inflammatory host-microbiome cross-talk in a human gut inflammation-on-a-chip. Proceedings of the National Academy of Sciences of the United States of America. 115 (45), E10539-E10547 (2018).
- Park, G. S., et al. Emulating host-microbiome ecosystem of human gastrointestinal tract in vitro. Stem Cell Reviews and Reports. 13 (3), 321-334 (2017).
- Kim, H. J., Li, H., Collins, J. J., Ingber, D. E. Contributions of microbiome and mechanical deformation to intestinal bacterial overgrowth and inflammation in a human gut-on-a-chip. Proceedings of the National Academy of Sciences of the United States of America. 113 (1), E7-E15 (2016).
- Jergens, A. E., et al. A scoring index for disease activity in canine inflammatory bowel disease. Journal of Veterinary Internal Medicine. 17 (3), 291-297 (2003).
- Roulis, M., Flavell, R. A. Fibroblasts and myofibroblasts of the intestinal lamina propria in physiology and disease. Differentiation; research in biological diversity. 92 (3), 116-131 (2016).
- Göke, M., Kanai, M., Podolsky, D. K. Intestinal fibroblasts regulate intestinal epithelial cell proliferation via hepatocyte growth factor. The American Journal of Physiology. 274 (5), G809-G818 (1998).
- Powell, D. W., Pinchuk, I. V., Saada, J. I., Chen, X., Mifflin, R. C. Mesenchymal cells of the intestinal lamina propria. Annual Review of Physiology. 73, 213-237 (2011).
- Pappenheimer, J. R., Michel, C. C. Role of villus microcirculation in intestinal absorption of glucose: coupling of epithelial with endothelial transport. The Journal of Physiology. 553, 561-574 (2003).
- Agace, W. W. T-cell recruitment to the intestinal mucosa. Trends in Immunology. 29 (11), 514-522 (2008).
- Ambrosini, Y. M., Shin, W., Min, S., Kim, H. J. Microphysiological engineering of immune responses in intestinal inflammation. Immune Network. 20 (2), 13 (2020).
- Kasendra, M., et al. Development of a primary human Small Intestine-on-a-Chip using biopsy-derived organoids. Scientific Reports. 8 (1), 1-14 (2018).
- Sontheimer-Phelps, A., et al. Human Colon-on-a-Chip enables continuous in vitro analysis of colon mucus layer accumulation and physiology. Cellular and Molecular Gastroenterology and Hepatology. 9 (3), 507-526 (2020).
- Shin, W., et al. Human intestinal morphogenesis controlled by transepithelial morphogen gradient and flow- dependent physical cues in a microengineered Gut-on-a-Chip. iScience. 15, 391-406 (2019).
- Gijzen, L., et al. An Intestine-on-a-Chip model of plug-and-play modularity to study inflammatory processes. SLAS Technology. 25 (6), 585-597 (2020).
- Kim, H. J., Lee, J., Choi, J. H., Bahinski, A., Ingber, D. E. Co-culture of living microbiome with microengineered human intestinal villi in a gut-on-a-chip microfluidic device. Journal of Visualized Experiments. (114), 3-9 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten