Kristallisations- und In-situ-Raumtemperatur-Datenerfassung mit der Kristallisationsanlage in Harwell und der Beamline VMXi, Diamantlichtquelle
In diesem Artikel
Zusammenfassung
Wir präsentieren ein Protokoll für die Kristallisation von Proteinen unter Verwendung der Kristallisationsanlage im Forschungskomplex in Harwell und die anschließende in-situ-Röntgenkristallographie-Datenerfassung von Kristallen innerhalb der Platten an der Versatile Macromolecular Crystallography in situ (VMXi) Beamline von Diamond. Wir beschreiben Probenanforderungen, Kristallisationsprotokolle und Richtlinien für die Datenerfassung.
Zusammenfassung
Es werden Protokolle für die robotergestützte Proteinkristallisation mit der Crystallization Facility in Harwell und die In-situ-Datenerfassung bei Raumtemperatur von Kristallisationsplatten an der Diamond Light Source-Beamline VMXi beschrieben. Dieser Ansatz ermöglicht es, qualitativ hochwertige Kristallstrukturen bei Raumtemperatur aus mehreren Kristallen auf einfache Weise zu bestimmen und liefert eine sehr schnelle Rückmeldung über die Ergebnisse von Kristallisationsversuchen sowie eine serielle Kristallographie. Der Wert von Strukturen bei Raumtemperatur für das Verständnis der Proteinstruktur, Ligandenbindung und -dynamik wird in der Strukturbiologie zunehmend anerkannt. Diese Pipeline ist für Benutzer aus der ganzen Welt mit mehreren verfügbaren Zugriffsmodi zugänglich. Aufgebaute Kristallisationsexperimente können abgebildet und aus der Ferne betrachtet werden, wobei Kristalle mithilfe eines maschinellen Lerntools automatisch identifiziert werden. Die Daten werden in einem warteschlangenbasierten System mit bis zu 60°-Rotationsdatensätzen aus benutzerdefinierten Kristallen in einer Platte gemessen. Die Daten aller Kristalle innerhalb einer bestimmten Vertiefung oder Probengruppe werden automatisch mit xia2.multiplex zusammengeführt, wobei die Ausgaben direkt über eine Webbrowser-Schnittstelle abgerufen werden können.
Einleitung
Die Röntgenkristallographie ist nach wie vor ein wichtiges Werkzeug zum Verständnis der Struktur und Funktion von Proteinen, da sie hochauflösende Strukturen von Proteinen oder deren Komplexen liefert, z. B. mit Substraten oder Wirkstoffkandidaten. In vielen Fällen bleibt jedoch die Gewinnung von Kristallen mit wünschenswerten Eigenschaften - stark beugend, Kristallform, die für das Einweichen geeignet ist und ohne Kristallpathologien wie Zwillinge - ein erheblicherEngpass 1. Da geeignete chemische Bedingungen zur Herstellung von Proteinkristallen im Allgemeinen nicht vorhergesagt werden können, ist das Kristallisations-Screening, bei dem Tausende potenzieller chemischer Mischungen untersucht werden, Standard, oft unterstützt durch Automatisierung/Robotik bei der Einstellung von Bildschirmen und Kristallhotels zur Überwachung der aufgezeichneten Kristallisationstropfenbilder, oft aus der Ferne.
Wenn Kristalle auftreten, müssen sie in der Regel mit einer Nylon- oder Kaptonschleife aus der Kristallisationsumgebung geerntet und dann in ein Tröpfchen überführt werden, das ein Kryoschutzmittel enthält (dessen Suche eine zusätzliche Variable ist), bevor sie in flüssigen Stickstoff gefriert werden. Diese zusätzlichen Schritte zwischen der Kristallisation und der Röntgendatenerfassung können unter anderem eine Dehydrierung des Kristallisationstropfens beinhalten, wenn seine versiegelte Umgebung gebrochen wird, mechanische Belastungen des Kristalls bei der Handhabung und eine Beschädigung des Kristallgitters durch die Kryoschutzmittel (was typischerweise zu einer erhöhten Mosaikausbreitung führt)2. Darüber hinaus ist die Kristallernte zeit- und arbeitsintensiv und kann zu Inhomogenitäten zwischen den Proben führen, insbesondere wenn sich während des Ernteprozesses Haut auf Tropfen bildet. Die VMXi-Beamline ermöglicht den Zugriff auf nutzbare Daten von Kristallen, die auf der Platte kleben und sonst für die Datenerfassung verworfen würden.
Die überwiegende Mehrheit der Röntgenkristallstrukturen wird mit dem oben genannten Ansatz bei 100 K bestimmt, was einen einfachen Kristalltransport und eine einfache Handhabung ermöglicht und die Lebensdauer der Kristalle im Röntgenstrahl um Größenordnungen erhöht. Es besteht jedoch ein zunehmendes Interesse an der Bestimmung von Strukturen unter nicht-kryogenen Bedingungen, d.h. viel näher an den physiologischen Bedingungen, die für die Proteinfunktion relevant sind 2,3,4. Dies ermöglicht ein viel besseres Verständnis der dynamischen Struktur von Proteinen, vermeidet, dass Aminosäurekonformationen oder Schleifen in funktionell nicht relevanten Zuständen eingefroren werden5, und ermöglicht es, die Ligandenbindung unter Bedingungen zu untersuchen, die denen in der natürlichen Umgebung des Proteins innerhalb der Zelle und des Organismus viel näher kommen6.
Ein alternativer Ansatz, der an der Beamline Versatile Macromolecular Crystallography in situ (VMXi) am Synchrotron Diamond Light Source, Großbritannien, implementiert wurde, besteht darin, die Beugungsdaten direkt von Kristallen in der Umgebung, in der sie gewachsen sind (d. h. innerhalb der Kristallisationsplatte), unter Umgebungsbedingungen und ohne Störung zu messen 7,8. Dies ermöglicht eine sehr schnelle Rückmeldung von Kristallisationsbildschirmen und Optimierungen, um den Anwender zu einer optimalen Kristallform für seine Anforderungen zu führen. Sie ermöglicht auch die automatisierte Herstellung hochwertiger Strukturen bei Raumtemperatur9.
Dieses Protokoll geht davon aus, dass ein Benutzer eine hochreine Proteinprobe zur Kristallisation bereitsteht. Wir beschreiben die Benutzererfahrung beim Zugriff auf die Kristallisationsanlage in Harwell, um Proteinkristalle herzustellen und dann die Beamline VMXi für die Datenerfassung zu verwenden (Abbildung 1).
Die Kristallisationsanlage in Harwell
Die Kristallisationsanlage in Harwell (CF) befindet sich im Forschungskomplex in Harwell (RCaH) neben der Diamond Light Source. Die Anlage bietet Anwendern ein automatisiertes Hochdurchsatzlabor für makromolekulare Kristallisation, das Robotik für das Kristallisationsscreening, die Kristalloptimierung, die Kristallbildgebung und die Charakterisierung einsetzt. Durch die enge Integration mit der hochautomatisierten VMXi-Beamline hat sich das Tempo bei der Bestimmung von Strukturen bei Raumtemperatur stark beschleunigt und ermöglicht die Charakterisierung neuartiger Proteinstrukturen, Protein-Ligand- und DNA-Liganden-Komplexe sowie ein automatisiertes Fragment-Screening (Abbildung 1) unter nicht-kryogenen Bedingungen.
Die CF-Pipeline besteht aus einer Reihe von Instrumenten, die Nanoliter-Kristallisationsroboter9 für die Kristallisation von löslichen Proteinen und Membranproteinen, Liquid-Handling-Roboter zur Herstellung kommerzieller Kristallisationssiebe und komplexer kundenspezifischer Optimierungsbildschirme sowie vier Bildgebungsinstrumente (eines bei 4 °C und drei bei 20 °C für die Abbildung von Kristallisationsplatten (siehe Materialtabelle) umfassen). Ein Imager ist in der Lage, Lipid-Cubic-Phase-Glasplatten (LCP) abzubilden, und ein Imager ist mit einer Multifluoreszenzoptik ausgestattet (beide bei 20 °C).
Die Anlage wird heute von einem breiten Spektrum akademischer und industrieller Nutzer genutzt, darunter das Membrane Protein Laboratory (MPL; https://www.diamond.ac.uk/Instruments/Mx/MPL.html), die XChem Fragment Screening Facility 10, MX Beamlines, der XFEL-Hub sowie das Rosalind Franklin Institute (RFI). Diese gut etablierte und optimierte Pipeline hat es ermöglicht, Kristallisationsexperimente in einem breiten Spektrum von strukturbiologischen Projekten durchzuführen. Dieses Dokument beschreibt die Pipeline für Kristalle, die für die Datenerfassung bei VMXi bestimmt sind, obwohl Kristalle auch geerntet und kryogekühlt oder an die XChem-Pipeline geleitet werden können.
Der Benutzerzugang wird über das Diamond MX Proposal System (https://www.diamond.ac.uk/Instruments/Mx/Synchrotron-Access.html) zugewiesen und industrielle Benutzer werden durch die Diamond Industry Liaison Group unterstützt. Alle Benutzer können mit ihrer Probe(n) oder Platten, die von Hand transportiert werden können, auf die Baustelle kommen. Es wird nicht empfohlen, Platten per Kurier zu versenden, da sich die Tropfen erfahrungsgemäß von der Stelle, an der sie ausgegeben wurden, entfernen oder durch das Kristallisationsreservoir beschädigt werden können. Alternativ können Anwender ihre Proteinproben nach Absprache an die CF schicken, wo Mitarbeiter in ihrem Auftrag Kristallisationsexperimente durchführen. Die Experimente können vom Benutzer aus der Ferne überwacht werden, indem er sich im Falle von CF entweder bei Rock Maker Web oder im Falle von VMXi über ISPyB anmeldet. Der Zugang zur Mukoviszidose kann iterativ auf der Grundlage der bei Diamond gesammelten Röntgenbeugungsergebnisse erfolgen.
Beamline VMXi an Diamantlichtquelle
Die Beamline VMXi (im Folgenden als "Beamline" bezeichnet) ist ein einzigartiges und kürzlich entwickeltes Instrument, das vollständig für die hochautomatisierte Röntgenkristallographie bei Raumtemperatur bestimmt ist, wobei der Schwerpunkt auf der Messung von Daten von Kristallen in geeigneten Kristallisationsplatten liegt. Die Beamline bietet einen Mikrofokus (10 x 10 μm), rosa Strahl (Bandpass von <5 × 10-2ΔE/E) mit einem hohen Fluss von ~2 × 1013 Photonen/s (bei 16 KeV)7. Dieser High-Flux-Strahl, gekoppelt mit einem schnellen Detektor, ermöglicht einen sehr hohen Durchsatz von Proben und die Erfassung von Daten von Proben mit einer Größe von mehr als 10 μm.
Kristallisationsplatten gelangen in die Beamline, indem sie in einem Probenspeichersystem gespeichert und auf der Grundlage des Zeitplans abgebildet werden, den der Benutzer bei der Registrierung der Platten über die ISPyB11-Schnittstelle SynchWeb12 angegeben hat. In der Regel wird dem Benutzer empfohlen, eine Fibonacci-Folge von Zeitpunkten für die Bildgebung auszuwählen (0, 12, 24, 36, 60... 7.320 h ab Eingabe der Platte in das System). Der Benutzer wird per E-Mail informiert, sobald eine Platte belichtet wurde. Sowohl die Bildgebung mit sichtbarem Licht als auch mit UV-Licht steht den Anwendern auf Anfrage zur Verfügung. Die vom Probenspeichersystem aufgenommenen Bilder werden von einem Algorithmus für maschinelles Lernen analysiert. Dadurch werden automatisch Points of Interest von Objekten, die Kristallen ähneln, lokalisiert und definiert, und die Points of Interest werden registriert, damit der Benutzer sie einer Warteschlange für die Datenerfassung hinzufügen kann. Benutzer können auch manuell auf die Bilder des sichtbaren Lichts klicken, um Points of Interest zu registrieren, oder sie können auf eine Region klicken und ziehen, die per Raster-Scan analysiert werden soll. Diese Punkte stehen den Benutzern zur Verfügung, um sie der Warteschlange zusammen mit den automatisch gefundenen Punkten hinzuzufügen.
Sobald alle Proben die entsprechenden Parameter für die Datenerfassung aufweisen, kommt die Platte in eine Warteschlange. Wenn die Platte die Spitze der Warteschlange erreicht, wird sie automatisch an die Beamline abgegeben. Die Kristallisationsplatten werden von einem Roboterarm automatisch von den Kristallhotels in die Beamline geladen, und nach dem Bildabgleich werden kristallographische Datensätze mit einer Drehung von bis zu 60° von jedem ausgewählten Kristall gemäß benutzerdefinierten Anweisungen gemessen. Alle Tropfen innerhalb einer Platte können für diese Experimente an der Beamline verwendet werden. Daten aus mehreren Kristallen werden automatisiert zusammengeführt, um isomorphe, optimal zusammengeführte Datensätze zu erzeugen 7,9. Sobald alle in der Warteschlange befindlichen Datensätze erfasst sind, erhält der Benutzer eine E-Mail mit einem Link, dem er folgen muss, um die Datensätze in ISPyB11 wie in anderen Diamond MX-Beamlines anzuzeigen. Die Nutzer werden auch auf die Beamline-Webseite (https://www.diamond.ac.uk/Instruments/Mx/VMXi.html) weitergeleitet.
Protokoll
1. Herstellung von Kristallen in In-situ-Platten mit der Kristallisationsanlage in Harwell
HINWEIS: Der Zugriff auf die CF wird über eine Reihe verschiedener Wege unterstützt und hängt von der Anwendung des Projekts und dem Benutzertyp (akademisch oder industriell) ab. XChem- und MPL-Projekte verfügen über ein eigenes Antragssystem über das User Administration System (UAS) und können entweder über den Standardzugang (u.a. iNEXT Discovery und EUbOPEN) oder über den BAG Access eingereicht werden. Das folgende Protokoll ist spezifisch für VMXi-Benutzer.
- Antragstellung und Vorbereitung des Besuchs
- Stellen Sie Informationen über das Projekt in einem BAG-Antrag bereit oder fügen Sie sie einem aktiven BAG-Antrag hinzu. In der Regel gibt es einen BAG-Koordinator, der den Papierkram organisiert. Alternativ können Sie auch einen Rapid Access Proposal für den Zugang zur Beamline einreichen.
- Stellen Sie sicher, dass die Probe vor der Ankunft vor Ort entweder per Kurier oder persönlich in UAS registriert und sicherheitsvalidiert wurde.
- Stellen Sie sicher, dass der Benutzer registriert ist (mit FedID und Passwort).
- Stellen Sie sicher, dass der Benutzer vom BAG-Koordinator als Associate in UAS zu einem MX-Vorschlag hinzugefügt wurde.
- Füllen Sie das Formular mit den Details der Beamline-Kristallisationsprobe aus und senden Sie es an VMXi@diamond.ac.uk.
- Kommunizieren Sie mit dem Beamline-Personal über die Anforderungen an das Experiment und die Verfügbarkeit der Beamline.
- Wenn Proteinproben versendet werden, senden Sie Proben nur nach vorheriger Absprache. Weitere Informationen finden Sie in Abschnitt 1.2.
- Wenn der Benutzer auf die Baustelle kommen soll, um Kristallisationsplatten im CF aufzustellen, erkundigen Sie sich beim Personal der Einrichtung nach der Verfügbarkeit eines Zeitfensters für die Nutzung der Anlageninstrumentierung und befolgen Sie Abschnitt 1.2.1.
- Wenn der Benutzer Platten mit auf die Baustelle bringt, stellen Sie sicher, dass die Probe in den richtigen Plattentyp abgegeben wird, und platzieren Sie die Kristallisationstropfen an der richtigen Stelle und in der richtigen Menge. Befolgen Sie Abschnitt 1.2.2. Die Beamline akzeptiert nur spezifische In-situ-Kristallisationsplatten (Greiner CrystalQuickX und MiTeGen In Situ-1); Stellen Sie sicher, dass die Tropfen nicht größer als 200 nL sind.
- Kristallisationsexperiment bei Mukoviszidose durchgeführt
HINWEIS: Die Anlage bietet eine Reihe von makromolekularen Kristallisationsmethoden mit hohem Durchsatz, wie z. B. Dampfdiffusion sowie Batch-Kristallisation unter Öl und LCP. Es wird empfohlen, mit 70-100 μl reinem Protein zu beginnen und Dampfdiffusionsexperimente für lösliche Proteine mit drei Screenings unter Verwendung eines Verhältnisses von 100 nL Proteinlösung und 100 nL Kristallisationsreservoirlösung durchzuführen und die Platten bei 20 °C zu inkubieren. In der Anlage stehen eine Reihe von kommerziellen Bildschirmen zur Verfügung. Feuchtigkeits- und Temperaturregelung sind mit 4 °C und 20 °C am häufigsten erhältlich. Benutzer, die das CF besuchen, erhalten eine standardisierte Schulung und Unterstützung bei der Bedienung der Kristallisationsinstrumente und verwenden die hier beschriebenen Einstellungen.- Versand von Mustern für den Aufbau am CF
HINWEIS: Vor der Ankunft vor Ort muss die Proteinprobe auf Vorschlag innerhalb des UAS-Systems validiert worden sein. Sobald die Proteinprobe vor Ort eingetroffen ist, richten die Mitarbeiter Kristallisationsexperimente ein, wie sie in der vorherigen Kommunikation mit dem Benutzer angewiesen wurden. Die Bestätigung erfolgt per E-Mail mit Barcode-Informationen für die experimentellen Kristallisationsplatten. Der Benutzer wird aufgefordert, die Kristallisationsplatten als Behälter zum entsprechenden Vorschlag hinzuzufügen. Ist dies geschehen, können die Platten in automatisierten Imagern in der Kristallisationsanlage oder an der Beamline gelagert werden. ISPyB wird die Schnittstelle sein, die für die Interaktion an der Beamline verwendet wird.- Stellen Sie die Proteinprobenlösung in der Konzentration für die Kristallisation in Vielfachen von 25 μl Aliquoten bereit. Beschriften Sie die Probenröhrchen mit der Proteinprobe deutlich.
- Stellen Sie bei Bedarf eine Proteinpufferlösung, eine Ligandenlösung oder eine Reservoirlösung zur Verfügung.
- Informieren Sie das Personal der Einrichtung, welche Bildschirme und Fallverhältnisse verwendet werden sollten.
- Kristallisationsplatten-Einstellungen
HINWEIS: Wir verlangen, dass sich die Kristallisationstropfen in der Greiner CrystalQuickX- und MiTeGen In Situ-1-Platte an einer bestimmten Stelle befinden. Platten, die an anderer Stelle aufgestellt werden, sollten die folgenden Mosquito13-Einstellungen verwenden, die hier beschrieben sind.- Um die Plattendefinition für MiTeGen In Situ-1 anzupassen, öffnen Sie die Mosquito SPT-Software und klicken Sie auf die Standard-MiTeGen In Situ-1-Plattendefinitionsseite, indem Sie auf Einstellungen | 96 Well | MiTeGen In Situ-1 (96 x 2 Tropfen) (Abbildung 2A). Klicken Sie auf die Schaltfläche "Bearbeiten" und ändern Sie die Werte für die Position der Unterschacht 2: X-Versatz auf - 1,2 und Y-Versatz auf 1,8 und für die Position der Unterschacht 3: X-Versatz auf 1,3 und Y-Versatz auf 1,8 (Abbildung 2B,C).
- Um die Plattendefinition für Greiner CrystalQuickX anzupassen, öffnen Sie die Mosquito SPT-Software und klicken Sie auf die Standard-Greiner CrystalQuickX-Plattendefinitionsseite, indem Sie auf Einstellungen | 96 Well | Greiner CrystalQuickX (Abbildung 2D). Klicken Sie auf die Schaltfläche "Bearbeiten " und ändern Sie die Werte für die Position der Unterschacht 1: X-Versatz auf - 1,95 und Y-Versatz auf 1,45 und für die Position der Unterschacht 2: X-Versatz auf 1,95 und Y-Versatz auf 1,45 (Abbildung 2E,F).
- Versand von Mustern für den Aufbau am CF
2. Nutzung der Beamline an der Diamond Light Source
HINWEIS: Die gesamte Interaktion der Benutzer mit der Beamline erfolgt aus der Ferne über die ISPyB11-Schnittstelle . Es ist keine physische Anwesenheit an der Beamline erforderlich, und die Daten werden über ein warteschlangenbasiertes System gesammelt, anstatt zu einem bestimmten Zeitpunkt geplant zu werden. Benutzer erhalten einen Vorschlag, der mit ihrem Zugang zur Diamantlichtquelle verknüpft ist. An der Beamline wird jeder Kristallisationsplatte ein eindeutiger Besuch zugewiesen und sie wird als "Behälter" innerhalb von ISPyB11 definiert, analog zu einem Puck, der Proben bei 100 K enthält. Optimierungsbildschirme können nicht über die SynchWeb-Schnittstelle erstellt werden, daher werden Informationen in der Regel in den Kommentarbereich eingefügt (siehe Schritt 2.1.4.). Die Person, die die Platte registriert, muss die E-Mail-Adresse überprüfen, da der Plattenbesitzer E-Mails bezüglich der Bildgebung sowie Benachrichtigungen über die Fertigstellung der Platte erhält.
- Kennzeichen registrieren
- Melden Sie sich mit einer entsprechenden Diamond-FedID bei ISPyB an und wählen Sie Vorschläge aus. Suchen Sie nach dem gewünschten Vorschlag, indem Sie scrollen oder die Angebotsnummer in die Suchleiste eingeben. Wählen Sie im Dropdown-Menü unter der Vorschlagsnummer (Abbildung 3A) die Option Sendung aus, um das Fenster Sendungen mit Sendungen in diesem Vorschlag zu öffnen. Klicken Sie oben rechts auf + Sendung hinzufügen (Abbildung 3B), um das Fenster "Neue Sendung hinzufügen" zu öffnen, geben Sie der Sendung einen Namen, klicken Sie auf "Automatisiert/Imager" und dann unten links auf die Schaltfläche "Sendung hinzufügen" (Abbildung 3C).
- Klicken Sie im Sendungsfenster (Abbildung 3D) auf +Container hinzufügen, um dann die Seitenansicht Container hinzufügen anzuzeigen (Abbildung 3E). Wählen Sie im Dropdown-Menü Behältertyp einen der entsprechenden Plattentypen aus. Die Seite ändert sich, um den ausgewählten Containertyp widerzuspiegeln. Geben Sie einen Barcode und einen Containernamen gemäß den E-Mail-Anweisungen des Beamline-Personals ein, die für die Versuchsplatten spezifisch sind. Beachten Sie, dass die Groß-/Kleinschreibung beachtet wird.
- Wählen Sie VMXi 20 °C Imager aus dem Dropdown-Menü "Requested Imager ", den Fibonacci-Imaging-Zeitplan aus dem Dropdown-Menü "Imaging-Zeitplan ", den Kristallisationsbildschirm aus dem Dropdown-Menü "Kristallisationsbildschirm " und den Benutzernamen aus dem Dropdown-Menü "Besitzer " aus, klicken Sie auf die Schaltfläche "Ansicht " und geben Sie die richtige Kontakt-E-Mail-Adresse in das Feld "E-Mail" ein (Abbildung 3F).
- Geben Sie weitere Details zur Platte in das Feld Kommentare ein. Wählen Sie die entsprechende Probe aus dem Dropdown-Menü "Protein " aus und verwenden Sie das in UAS registrierte und von Diamond genehmigte Akronym innerhalb des experimentellen Vorschlags. Geben Sie denselben Namen in das Feld Beispielname ein. Lassen Sie die restlichen Kästchen leer.
- Klicken Sie auf das Symbol +Platte , um die Probe über die gesamte Platte zu replizieren und den gesamten Behälter mit grünen Quadraten zu füllen. Klicken Sie unten auf der Seite auf +Behälter hinzufügen , um die Platte zu registrieren. Bitten Sie einen Mitarbeiter an der Beamline, die Platte in den entsprechenden Imager zu übertragen, wo sie gespeichert und belichtet wird. Ein Besuch wird generiert, wenn der Behälter in den Imagern gespeichert ist, und der Benutzer erhält eine E-Mail mit einem Link zur Platte und ihren Bildern.
- Anzeigen von Imaging-Ergebnissen
- Navigieren Sie zu dem gewünschten Vorschlag (Schritte 2.1.1), wählen Sie Container aus dem Dropdown-Menü unter der Vorschlagsnummer aus, und beobachten Sie die Liste der verfügbaren Container für den Vorschlag. Wählen Sie den Filter Platten aus, wenn andere Probenhaltertypen vorhanden sind. Um die Suche weiter einzugrenzen, aktivieren Sie das Kontrollkästchen Meine Container, um nur die relevantesten Container anzuzeigen, die mit der aktuell angemeldeten Benutzer-ID verknüpft sind. Klicken Sie auf den entsprechenden Container, indem Sie den Mauszeiger über die einzelne Zeile bewegen und mit der linken Maustaste klicken.
- Nach der Auswahl des Behälters wird eine neue Ansicht angezeigt, die eine Übersicht über die Platte zeigt (Abbildung 4A). Klicken Sie auf einen Tropfen in der Plattendarstellung auf der linken Seite des Displays, um das neueste Bild des jeweiligen Tropfens anzuzeigen. Verwenden Sie die Pfeiltasten, um zwischen den Tropfen zu navigieren, oder wählen Sie einzelne Tropfen mit einer Maus/einem Cursor aus.
- Um historische Bilder eines Tropfens anzuzeigen, klicken Sie auf die Schaltfläche H und warten Sie, bis eine Popup-Galerie mit Bildern über dem aktuellen Brunnentropfenbild erscheint. Bewegen Sie den Mauszeiger über die einzelnen Bilder, um das Hauptablagebild zu aktualisieren.
- Bewerten Sie Bilder, um den Status jedes Tropfens anzuzeigen, indem Sie die Tasten 0 - 9 drücken. Um die einzelnen Kategorien zu sehen, öffnen Sie das Dropdown-Menü Score oben links im Drop-Bild. Suchen Sie nach blauen Kreuzen in jedem der Tropfenbilder, die das Ergebnis eines Algorithmus (CHiMP) sind, der darauf trainiert wurde, nach "Kristallen" in den Bildern zu suchen.
- Klicken Sie auf die dritte Symbolschaltfläche namens Messen oben rechts im Tropfenbild, um auf ein Messwerkzeug zuzugreifen. Um dieses Werkzeug zu verwenden, klicken und ziehen Sie eine Linie, und ein Lineal verlängert und gibt den Abstand in μm an.
- Um eine zusätzliche Imaging-Sitzung anzufordern, klicken Sie im Dropdown-Feld neben der Überschrift "Aktionen" unten auf der Seite entweder auf "Sichtbar" oder "UV". Klicken Sie dann auf die Schaltfläche Plattenbelichtung anfordern.
- Kristallauswahl/CHiMP
- Um Punkte für die Datenerfassung manuell hinzuzufügen, klicken Sie auf die Schaltfläche +Punkt markieren . Bewegen Sie den Mauszeiger über den gewünschten Point of Interest und wählen Sie ihn aus. Warten Sie, bis ein rotes Kreuz erscheint.
HINWEIS: Pro Drop können bis zu 100 Objekte erstellt werden. - Wenn alle Punkte markiert sind, klicken Sie auf die Schaltfläche +Fertig stellen . Denken Sie daran, auch auf die Schaltfläche +Fertig stellen zu klicken, bevor Sie versuchen, Objekte zu messen. Um Regionen für die Datenerfassung über Rasterscans hinzuzufügen, klicken Sie auf die Schaltfläche +Region markieren . Klicken Sie auf den oberen linken Punkt und ziehen Sie nach unten und rechts, um einen Bereich zu erstellen, der auf der Beamline gerastert wird. Klicken Sie wie bei Punkten auf die Schaltfläche +Fertig stellen , wenn alle gewünschten Regionen erstellt wurden.
HINWEIS: Es ist besser, eine größere Region als viele kleine Regionen zu erstellen. - Beobachten Sie die blauen Kreuze bereits auf den Tropfenbildern, die das Ergebnis eines Algorithmus sind, der zur automatischen Lokalisierung kristalliner Objekte (CHiMP) entwickelt wurde. Um die CHiMP-Bewertung von Kristallisationstropfen zu visualisieren, klicken Sie auf das Kontrollkästchen Automatische Bewertungen anzeigen und ändern Sie dann das Dropdown-Menü für Klasse. In der Regel ist die nützlichste Einstellung hier die Kristalloption (Abbildung 4B).
HINWEIS: Dies ist eine neue Funktion und es ist nicht garantiert, dass alle Kristalle gefunden werden, und es können auch andere Objekte gefunden werden, die keine Kristalle sind. - Wenn alle Punkte und Regionen in den jeweiligen Tropfen markiert wurden, klicken Sie unten auf der Seite auf die Schaltfläche Für die Datenerfassung vorbereiten .
- Um Punkte für die Datenerfassung manuell hinzuzufügen, klicken Sie auf die Schaltfläche +Punkt markieren . Bewegen Sie den Mauszeiger über den gewünschten Point of Interest und wählen Sie ihn aus. Warten Sie, bis ein rotes Kreuz erscheint.
- Vorbereiten von Proben für die Datenerfassung
- Beobachten Sie die Liste der Samples, die die im vorherigen Schritt ausgewählten oder automatisch lokalisierten Punkte oder Bereiche enthalten (Abbildung 4C). Fügen Sie einzelne Punkte oder Regionen hinzu, indem Sie auf die Schaltfläche + klicken, oder fügen Sie alle angezeigten Beispiele hinzu, indem Sie auf die Schaltfläche Aktuelle Seite zur Warteschlange hinzufügen klicken .
- Es stehen Filter zur Verfügung, um nur Punkt-, Regions-, Auto- oder manuelle Punkte anzuzeigen. Um nur die Proben anzuzeigen, die nicht aufgenommen wurden (d. h. Röntgenstrahlen ausgesetzt wurden), klicken Sie auf die Optionen "Ohne Daten " und "Nicht abgeschlossen " über den Filterschaltflächen.
- Wählen Sie einzelne Proben aus, indem Sie auf die jeweilige Zeile klicken, und aktualisieren Sie das Bild auf der rechten Seite des Bildschirms, um den richtigen Tropfen und den richtigen Einzelpunkt anzuzeigen. Wenn viele Samples in der Liste enthalten sind, erhöhen Sie die Anzahl der pro Seite angezeigten Samples , indem Sie das Dropdown-Menü mit 10 als Standard und bis zu 100 als maximale Anzahl der angezeigten Samples auswählen.
- Nachdem alle Punkte und Regionen zur Warteschlange hinzugefügt wurden, stellen Sie sicher, dass alle Parameter für die experimentelle Datenerfassung mit jedem Experiment verknüpft sind.
- Verwenden Sie Filter für Punkt, Region, Manuell und Auto. Klicken Sie auf den Punktfilter und dann auf das Kontrollkästchen Alle auswählen unter den Filterschaltflächen, um gleichzeitig Parameter auf alle Samples anzuwenden, die in der aktuellen Liste der Samples in der Warteschlange angezeigt werden.
- Wählen Sie experimentelle Parameter aus dem Dropdown-Menü auf der rechten Seite des Bildschirms unter dem Tropfenfoto aus (Abbildung 4D). Wählen Sie für Regionen die Option Grid Scan DMM 10 Mikrometerschritte, 100 Prozent Übertragung. Wählen Sie für alle anderen Punktexperimente nach Bedarf andere Optionen aus dem Dropdown-Menü aus.
- Klicken Sie für Oszillationsdatenerfassungen auf die Omega Scan DMM 60 Grad 5 Prozent Übertragungsoption , um die maximale Datenmenge aus einer einzelnen Probe zu erfassen. Wenden Sie kleine Rotationen für sehr kleine Kristalle oder strahlungsempfindliche Proben an und variieren Sie die Transmission basierend auf früheren Erfahrungen mit einer bestimmten Kristallform. Nachdem alle experimentellen Parameter korrekt angewendet wurden, klicken Sie unten auf der Seite auf die Schaltfläche Warteschlangencontainer .
- Sobald die Platte die Spitze der Warteschlange erreicht hat, wird sie der Beamline präsentiert, Datensätze werden gesammelt und kehrt dann wieder in das Probenlager innerhalb der Beamline zurück. Sobald die Datenerfassung von einer Platte abgeschlossen ist, suchen Sie nach einer E-Mail mit einem Link, dem Sie folgen können, um auf die relevanten Daten zuzugreifen.
- Erstellen von Beispielgruppen
HINWEIS: Probengruppen können erstellt werden, um ähnliche Proben über mehrere Tropfen oder Platten hinweg zu gruppieren. Alle Datensätze innerhalb dieser Stichprobengruppen werden nach der Verarbeitung durch DIALS mit der xia2.multiplex14-Pipeline verarbeitet. Dies kann nützlich sein, wenn viele sehr kleine Datenkeile gesammelt werden, und kann auch nützlich sein, um das Signal-Rausch-Verhältnis für Ligandenbindungsexperimente zu erhöhen.- Wählen Sie im Dropdown-Menü unter der Vorschlagsnummer die Option Beispielgruppenverwaltung aus. Suchen Sie nach einer Liste von Gruppen, wenn diese bereits von anderen Benutzern erstellt wurden. Um eine neue Gruppe zu generieren, klicken Sie auf die Schaltfläche +Beispielgruppe erstellen. Klicken Sie im Dropdown-Menü auf der Seite "Beispielgruppe erstellen" auf eine Sendung, um den Beispiel-Viewer anzuzeigen (Abbildung 5A). Klicken Sie in der aufgefüllten Liste auf den Container mit den relevanten Beispielen.
- Wenn ein Behälter angeklickt wurde, suchen Sie nach einer Grafik, die die Plattenübersicht zeigt.
- Klicken Sie einzeln auf Drops, indem Sie auf den einzelnen Drop klicken (Abbildung 5B), oder klicken Sie auf Drops in Zeilen oder Spalten, indem Sie auf den entsprechenden Zeilenbuchstaben oder die entsprechende Spaltennummer klicken. Wenn alle Wells, die einer einzelnen Gruppe zugeordnet sind, ausgewählt wurden, geben Sie einen Namen für die Gruppe in das Feld Probengruppenname ein und klicken Sie auf die Schaltfläche Probengruppe speichern . Klicken Sie auf dieser Seite auf die Schaltfläche Stichprobengruppen anzeigen , um zur Liste der bereits generierten Stichprobengruppen zurückzukehren, die dem Vorschlag zugeordnet sind (Abbildung 5C).
- Bearbeiten von Beispielgruppen
- Klicken Sie in der Liste der Gruppen auf der Seite "Beispielgruppenverwaltung " auf eine Beispielgruppe.
- Klicken Sie auf die Schaltfläche +Beispielgruppe bearbeiten neben den Containern , die unter den Gruppeninformationen angezeigt werden (Abbildung 5C).
- Beobachten Sie die Tropfen, die bereits einer Probengruppe zugeordnet sind und auf der Plattenübersicht hervorgehoben sind.
- Fügen Sie der Beispielgruppe weitere Tropfen hinzu, indem Sie wie zuvor auf Tropfen, Vertiefungen oder Spalten klicken.
HINWEIS: Tropfen können nicht aus einer Probengruppe entnommen werden. - Sobald zusätzliche Tropfen hinzugefügt wurden, bearbeiten Sie den Namen der Probengruppe und speichern Sie ihn dann, oder speichern Sie ihn einfach, indem Sie auf die Schaltfläche Probengruppe speichern klicken .
- Visualisieren und Analysieren der Ausgabe von Probengruppen
- Klicken Sie in der Liste der Probengruppen auf eine einzelne Gruppe, um die Plattenübersicht des Behälters oder der Behälter anzuzeigen, die der Gruppe zugeordnet sind. Die in der Gruppe enthaltenen Tropfen werden auf dieser Anzeige hervorgehoben (Abbildung 5D).
- Suchen Sie nach einer Liste mit den chronologischen letzten drei Multiplex-Jobs, wenn in dieser Gruppe Daten gesammelt wurden.
- Klicken Sie auf die Zeile für einen Multiplexlauf, um die Verarbeitungsergebnisse unten zu aktualisieren.
- Beachten Sie die Schaltfläche für den Quicklink , die die Anzahl der mit der Gruppe verknüpften Datensätze anzeigt. Klicken Sie auf diese Schaltfläche, um eine neue Seite "Datensammlungen " zu öffnen, auf der die einzelnen Datensatzsammlungen angezeigt werden.
3. Zugriff auf die automatische Datenverarbeitung
HINWEIS: Sobald die Daten erfasst wurden, durchlaufen sie mehrere automatische Datenverarbeitungspipelines. Die vier Standard-Pipelines, die über die MX-Beamlines bei Diamond verwendet werden, werden ebenfalls mit Daten betrieben, die an der Beamline gesammelt werden. Es handelt sich um 'fast_dp', 'xia2 dials', 'xia2 3dii' und 'autoPROC'15. "fast_dp" ermöglicht eine schnelle Datenreduktion, um die Qualität schnell zu bewerten. Die anderen drei Pipelines benötigen mehr Rechenzeit und führen eine Vielzahl verschiedener Softwarepakete zur Datenreduzierung zum Vergleich aus. Dementsprechend ist die Ausgabe in der Regel von höherer Qualität als die "fast_dp"-Ausgabe. Die an der Beamline gesammelten Datensätze durchlaufen auch die automatische Multikristall-Merging-Software "xia2.multiplex"14, die alle Datensätze innerhalb einer definierten Gruppe zusammenführt. Beachten Sie, dass Rasterscans derzeit zwar nicht automatisch verarbeitet werden, die Daten jedoch manuell mit der Pipeline "xia2.ssx" verarbeitet werden können. Die Ergebnisse der automatischen Verarbeitungspipelines finden Sie in ISPyB11 unter Verwendung des folgenden Protokolls.
- Auffinden der Datasets
- Melden Sie sich wie oben beschrieben bei ISPyB an und wählen Sie Vorschläge aus.
- Suchen Sie nach dem gewünschten Vorschlag , indem Sie scrollen oder die Angebotsnummer in die Suchleiste eingeben.
- Klicken Sie in der Liste, die auf dem Bildschirm angezeigt wird, auf den gewünschten Besuch , um das Fenster Datensammlungen für diesen Besuch aufzurufen.
- Wenden Sie die gewünschten Filter an.
HINWEIS: Ein beliebter Filter ist der Filter "Automatisch integriert", der nur Datensätze anzeigt, die erfolgreich eine oder mehrere Verarbeitungspipelines durchlaufen haben. Dadurch werden Grid-Scans ausgeschlossen, da diese derzeit nicht automatisch über ISPyB verarbeitet werden. - Scrollen Sie auf der Seite nach unten, um das gewünschte Dataset zu finden.
HINWEIS: Jeder Datensatz zeigt die Proben-ID, die verwendeten experimentellen Parameter, einen Beugungsbildbetrachter, einen Kristallbildbetrachter und ein Analysediagramm pro Bild zur schnellen Beobachtung der Datenqualität an.
- So greifen Sie auf die Ergebnisse der automatischen Verarbeitung zu
- Klicken Sie auf die Registerkarte Automatische Verarbeitung unter der Datenzusammenfassung eines bestimmten Experiments, um die Ergebnisse der automatischen Datenreduktion zu überprüfen (Abbildung 6A).
- Klicken Sie auf die verschiedenen Registerkarten, die den verschiedenen Pipelines entsprechen, um eine detaillierte Zusammenfassung der einzelnen Ausgaben anzuzeigen.
HINWEIS: Wenn Sample-Gruppen definiert wurden, gibt es zwei Registerkarten, die Multiplex-Jobs entsprechen. Die eine entspricht der Zusammenführung aller Datensätze in der Gruppe bis zu diesem Punkt, während die andere nur der Zusammenführung von Datensätzen innerhalb dieser Ablage entspricht. - Klicken Sie auf die Schaltfläche Protokolle & Dateien , um die resultierenden MTZ-Dateien herunterzuladen, wenn die Verarbeitung erfolgreich war, und alle zugehörigen Protokolldateien. Klicken Sie auf die Registerkarte Downstream-Verarbeitung unter dem Abschnitt Automatische Verarbeitung , um die Ausgabe von DIMPLE anzuzeigen.
HINWEIS: DIMPLE wird nur ausgeführt, wenn bei der Übermittlung der Probe eine PDB-Datei bereitgestellt wurde. - Klicken Sie auf die Schaltfläche Logs & Files , um alle resultierenden Ausgaben von DIMPLE herunterzuladen.
- Um auf die Ergebnisse des Gruppenmultiplex zuzugreifen, öffnen Sie das Dropdown-Menü am oberen Bildschirmrand mit der Vorschlagsnummer und klicken Sie auf Sample Group Management. Klicken Sie auf die Zeile , die der gewünschten Gruppe innerhalb des richtigen Containers entspricht. Scrollen Sie nach unten, um die Liste der Multiplex-Ausgänge zu finden, die der Gruppe entsprechen, wie sie visuell durch ein Diagramm der Platte dargestellt wird.
- Klicken Sie auf den gewünschten Multiplex-Ausgang aus der angegebenen Liste. Klicken Sie auf die Schaltfläche xxx Datensätze , wobei xxx die Anzahl der zusammengeführten Datensätze ist (Abbildung 6B).
HINWEIS: Dadurch wird der Bildschirm Datensammlungen geöffnet, aber es werden nur die Datensätze aus dem ausgewählten Multiplex-Auftrag angezeigt. - Klicken Sie auf die Registerkarte Automatische Verarbeitung des obersten Experiments.
- Klicken Sie auf die Registerkarte Multiplex-Verarbeitung , die der korrekten Anzahl der zusammengeführten Datensätze entspricht.
- Klicken Sie auf die Schaltfläche Protokolle & Dateien , um die .mtz-Datei und die entsprechenden Protokolldateien herunterzuladen (wie in Schritt 3.2.3.)
- Klicken Sie auf den gewünschten Multiplex-Ausgang aus der angegebenen Liste. Klicken Sie auf die Schaltfläche xxx Datensätze , wobei xxx die Anzahl der zusammengeführten Datensätze ist (Abbildung 6B).
- So greifen Sie auf Rasterscan-Ergebnisse zu
- Navigieren Sie zum Bildschirm Datensammlungen für den gewünschten Besuch. Die Ergebnisse der Rasterscandaten werden zusammen mit den erfassten Rotationsdaten angezeigt.
HINWEIS: Es werden keine Ergebnisse der automatischen Verarbeitung angezeigt. - Im Bild des Kristalltropfens wird das Gitter mit einer Heatmap überlagert, die das Vorhandensein von Beugung darstellt. Klicken Sie auf ein Quadrat , um das Beugungsbild für diese Position im Raster anzuzeigen. Klicken Sie auf das Dropdown-Menü am oberen Rand des Kristallbrunnens, um die Darstellung der Heatmap zu ändern. Die Standardeinstellung ist die Gesamtintensität der Beugung, kann aber in Gesamtpunkte, geschätzte Auflösung oder Bilder ohne Eis geändert werden.
- Navigieren Sie zum Bildschirm Datensammlungen für den gewünschten Besuch. Die Ergebnisse der Rasterscandaten werden zusammen mit den erfassten Rotationsdaten angezeigt.
4. Datenaufbereitung
HINWEIS: Ausgewählte Datensätze können über die ISPyB11-Schnittstelle mit denselben Verarbeitungspipelines erneut verarbeitet werden, die automatisch mit geänderten Einstellungen ausgeführt werden, die vom Benutzer definiert werden. Es kann ein Auflösungsgrenzwert angewendet werden. Wenn die Symmetrie/Zelle des Kristalls bekannt ist, dann kann dies auch definiert werden, um sicherzustellen, dass die Verarbeitungspipelines in der richtigen Einstellung laufen. Ausgewählte Bildbereiche in bestimmten Datensätzen können auch mithilfe verfügbarer mehrkristalliner Pipelines zusammengeführt werden. Dies kann sich als vorteilhaft erweisen, wenn systematische Strahlenschäden dazu führen, dass der letzte Teil der Beugungsbilder von schlechter Qualität ist. Es ist auch eine Option für den Benutzer, seine Datensätze mit dem oben beschriebenen Protokoll herunterzuladen und die gewünschte Aufbereitungssoftware lokal auszuführen, für die Tutorials an anderer Stelle frei verfügbar sind (https://dials.github.io/documentation/tutorials/index.html# ).
- So verarbeiten Sie mehrere einzelne Datasets erneut
- Melden Sie sich bei ISPyB an und navigieren Sie zu den gewünschten Datensätzen (Schritt 3.1).
- Klicken Sie auf einen Datensatz und dann auf das Zahnradsymbol in der Titelleiste des Datensatzes (Abbildung 6), um das Fenster für die erneute Verarbeitung zu öffnen.
- Konfigurieren Sie die gewünschten Einstellungen und wählen Sie aus, welche Frames in die erneute Verarbeitung einbezogen werden sollen.
Hinweis: Der Bildbereich kann entweder durch Eingabe eines Bereichs in die beschrifteten Felder oder durch Klicken und Ziehen des gewünschten Bereichs in der Analysezeichnung pro Bild definiert werden (Abbildung 7A). - OPTIONAL: Um einen weiteren Datensatz für die individuelle Wiederaufbereitung hinzuzufügen, klicken Sie auf das Zahnradsymbol und es wird im Wiederaufbereitungsfenster unter dem ersten Datensatz angezeigt. Markieren Sie das Kästchen Einzeln verarbeiten .
- Klicken Sie auf die Schaltfläche Integrieren .
- So verarbeiten Sie mehrkristalline Daten erneut
- Öffnen Sie das Fenster für die erneute Verarbeitung in einem beliebigen Dataset.
- Klicken Sie auf die Schaltfläche Multikristall, um einen neuen Bildschirm zu öffnen.
- Scrollen Sie nach unten, um eine Reihe von Analysediagrammen pro Bild aus Experimenten während des Besuchs zu finden.
- Wählen Sie eine Verarbeitungspipeline aus dem Dropdown-Menü aus.
- OPTIONAL: Definieren Sie alle Auflösungsgrenzen oder bekannten Einheitszellenparameter.
- Klicken und ziehen Sie, um Bildbereiche zu definieren, die in die mehrkristalline Verarbeitung einbezogen werden sollen (Abbildung 7).
HINWEIS: Dies sollte über mehrere verschiedene Diagramme erfolgen, damit Datensätze aus mehreren verschiedenen Kristallen zusammengeführt werden. - Klicken Sie auf die Schaltfläche Integrieren (Abbildung 7B).
- So greifen Sie auf wiederverarbeitete Daten zu
- Navigieren Sie zur Seite Datensammlungen für den jeweiligen Besuch (Schritte 3.1.1-3.1.3).
- Klicken Sie auf die Schaltfläche Nachbearbeitung am oberen Rand des Bildschirms.
- Scrollen Sie nach unten, um den gewünschten Auftrag zu finden.
- Klicken Sie auf den Dateipfad in der rechten Spalte, um den Bildschirm Datensammlungen für die wiederverarbeiteten Daten zu öffnen.
- Öffnen Sie die Registerkarte Automatische Verarbeitung und laden Sie die Daten wie zuvor beschrieben herunter (Schritt 3.2).
HINWEIS: Alle neu verarbeiteten Aufträge sind an dem kreisförmigen Pfeilsymbol neben dem Pipelinenamen zu erkennen.
Repräsentative Ergebnisse
Die Kristallisationsanlage und die VMXi-Beamline wurden für eine Vielzahl von Projekttypen und Anwendungsfällen eingesetzt. Hier sind einige Beispiele, um zu veranschaulichen, was Benutzer möglicherweise verfolgen möchten.
Fallstudie 1: Standard-Datenerhebung
Die Beamline ermöglicht die schnelle Bestimmung von Kristallstrukturen bei Raumtemperatur aus einer kleinen Anzahl von Kristallen innerhalb einer Kristallisationsplatte. Die minimale Anzahl von Kristallen hängt von der Raumgruppe und der Kristallorientierung ab, liegt aber häufig bei 1-4, obwohl eine verbesserte Datenqualität durch die Zusammenführung von Daten von mehreren Dutzend Kristallen erreicht werden kann. Ein aktuelles Beispiel ist einer der Beamline-Standards, Thaumatin. Mehrere Kristalle, die in Abbildung 8A gezeigt sind, wurden für die Datenerfassung manuell markiert, wie in Protokollabschnitt 2.3 beschrieben. Diese Kristalle wurden der Warteschlange hinzugefügt, wie in Protokollabschnitt 2.4 beschrieben, und experimentelle Parameter wurden aus der Dropdown-Liste ausgewählt. Nachdem die experimentellen Parameter angewendet worden waren, wurde die Platte für die Datenerfassung in die Warteschlange gestellt. Die Datensätze wurden mithilfe der xia2.multiplex-Pipeline gesammelt, automatisch skaliert und zusammengeführt, wie in Protokollabschnitt 3 beschrieben. Eine Beispielausgabe von SynchWeb ist in Abbildung 8A in der Mitte dargestellt. Aus fünf zusammengeführten Datensätzen ergab sich ein Datensatz mit einer Auflösung von 1,66 Å. Für die Standarddatenerfassung von etwa fünf Kristallen in einer Vertiefung wurden die Datensätze innerhalb von 2,5 Minuten gesammelt.
Fallstudie 2: Ligandenbindung – Fragmentexperiment mit dem Mac1-Protein
Die Herstellung von Strukturen von Protein-Ligand-Komplexen bei Raumtemperatur kann mit Hilfe der Beamline einfach erreicht werden. Liganden können zu Tropfen auf Kristallisationsplatten hinzugefügt werden (entweder manuell oder durch akustische Tropfeninjektion) und Daten können nach einer geeigneten Inkubationszeit gemessen werden. In dem hier beschriebenen Beispiel wurde eine Reihe von Fragmenten in Vertiefungen abgegeben, die Kristalle der ersten SARS-CoV-2-Makrodomäne des Proteins nsp3 (Mac-1) in einer Kristallisationsplatte enthielten. Zwei der Vertiefungen, die das gleiche Fragment enthielten, wurden als Gruppe zugeordnet, wie in Protokollschritt 2.5 beschrieben. Mehrere Kristalle (42) wurden für die Datenerfassung markiert, wie in den Protokollschritten 2.3 und 2.4 beschrieben, und die Datensätze wurden unter Verwendung von Standardparametern (60° Drehung, 0,1° Schritt, 0,00178 s Exposition, 5% Transmission, 16 KeV - pro Kristall) gesammelt (Abbildung 8B). Die Datensätze aus den beiden Bohrlöchern wurden automatisch mit der xia2.dials-Pipeline verarbeitet, und anschließend wurde die xia2.multiplex-Pipeline initiiert, um 22 dieser Datensätze automatisch zusammenzuführen. DIMPLE wurde dann mit dem Ausgang dieser Pipelines ausgeführt und lieferte Karten, die eindeutig Hinweise auf das gebundene Fragment zeigten. Das Fragmentmodell wurde in die unbesetzte Dichte eingebaut und weiter verfeinert (Abbildung 8B rechts). Ligandengebundene Strukturen bei Raumtemperatur können mit dieser Reihe von Schritten leicht bestimmt werden, um unschätzbare Informationen und Feedback für den strukturbasierten Wirkstoffdesignprozess zu liefern. Für diese Datensammlung von 42 Kristallen in einer Reihe von Bohrlöchern wurden Datensätze innerhalb von 10 Minuten gesammelt.
Fallstudie 3: Strukturlösung mit einer Raumgruppe mit geringer Symmetrie und bevorzugten Orientierungen Ein Stapel von Mehrfachkristallen mit plattenartiger Morphologie wurde aus Kristallisationsexperimenten mit einem gasbindenden Cytochrom vom C-Typ hergestellt (Abbildung 8C). Durch die Auswahl mehrerer Positionen am Rand des Stapels, an denen sich nur ein einziger Kristall im Röntgenstrahl befand, war es möglich, einen Datensatz von guter Qualität mit einer Auflösung von 1,75 Å zu erhalten, indem Keile von vier Kristallen zusammengeführt wurden, trotz einer monoklinen (C2) Raumgruppe. Dies ermöglichte einen schnellen Projektfortschritt, ohne dass die Kristallisationsbedingungen weiter optimiert werden mussten. Dieses Ergebnis wurde bereits beschrieben9. Für diese Datensammlung von vier Kristallen in einem Bohrloch wurden Datensätze innerhalb von 2 Minuten gesammelt.
Fallstudie 4: Gewinnung von Informationen und Raumtemperaturstruktur aus Mikrokristallen in einer Platte mittels serieller Kristallographie
Wenn Mikrokristalle in einem Tropfen auftreten oder wenn Anwender versuchen, Mikrokristallisationsprotokolle im Batch als Vorstufe zu seriellen Kristallographie-Experimenten an Synchrotron- oder XFEL-Quellen zu optimieren, ist es oft sehr hilfreich, eine schnelle Rückmeldung über die Beugungseigenschaften und Einheitszellabmessungen verschiedener Versuche mit minimalem Material zu erhalten. In diesem Anwendungsfall wurden Mikrokristalle aus Lysozym, die im Batch wachsen, in eine Kristallisationsplatte (200 nL Volumen pro Tropfen) pipettiert und Daten aus acht Tropfen mit einem Rasterscan mit einer Schrittweite von 10 μm gesammelt (Abbildung 9). Die resultierenden 25.906 Standbilder wurden mit einer seriellen Kristallographie-Software verarbeitet, was zu einem Datensatz führte, in dem 9.891 Beugungsmuster indiziert und zusammengeführt wurden, wodurch ein Datensatz mit einer Auflösung von 2,0 Å entstand, der sich gut mit der veröffentlichten Raumtemperaturstruktur verfeinerte (R-Arbeit = 19,6 %,R-frei = 23,6 % unter Verwendung von PDB 8A9D) (Tabelle 1). Dies ermöglichte eine detaillierte Analyse der Einheitszellverteilung und eine mikrokristalline Raumtemperaturstrukturbestimmung, die in komplexe serielle Kristallographieexperimente einschließlich zeitaufgelöster Studien einfließen konnte. Das Gesamtvolumen der erforderlichen Mikrokristallsuspension betrug 1,6 μl. Für diese Datenerfassung von Mikrokristallen über acht Wells mittels Grid-Scans wurden die Datensätze innerhalb von 40 Minuten gesammelt.
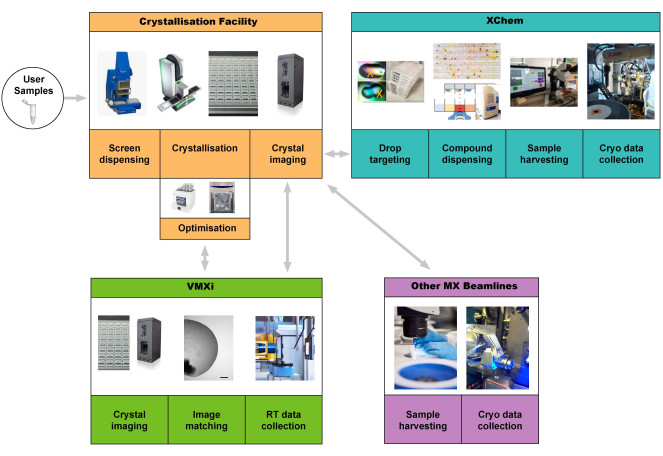
Abbildung 1: Schematische Darstellung der Protein-zu-Struktur-Pipeline, die das Kristallisations-Screening, die Optimierung an der Kristallisationsanlage, die automatisierte Datenerfassung und -verarbeitung bei Raumtemperatur ohne Probenentnahme bei VMXi, das XChem-Fragment-Screening und die Datenerfassung an anderen MX-Beamlines integriert. Benutzer können die Pipeline starten, indem sie eine Probe bereitstellen oder Platten zur VMXi-Beamline bringen. Abkürzung: Vielseitige makromolekulare Kristallographie in situ. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Die Mosquito SPT Labtech-Schnittstelle zum Einrichten von Kristallisationsplatten. (A) (1) Die MiTeGen In Situ-1 Setup-Ansicht. Wählen Sie die MiTeGen 2-Drop-Standardplatte aus, indem Sie zu (2) dem 96-Well-Plattentyp gehen und (3) die MiTeGen-Plate 2-Drop-Platte auswählen. Um die Definitionsparameter für Drop 1 und Drop 2 zu ändern, was für VMXi erforderlich ist, klicken Sie auf das Bearbeitungssymbol (4). Dadurch wird ein neues Fenster (B) geöffnet, in dem (5) die X- und Y-Offsets wie gezeigt geändert werden müssen. Wählen Sie (B) die Unterschacht 2 und (C) die Untervertiefung 3 und ändern Sie die Werte entsprechend. (D) Die CrystalQuickX-Setup-Ansicht . Wählen Sie die CrystalQuickX 2-Drop-Standardplatte , indem Sie zum 96-Well-Plattentyp gehen und die MiTeGen-Plate 2-Drop-Platte auswählen. Um die Definitionsparameter für Drop 1 und Drop 2 zu ändern, die für VMXi erforderlich sind, klicken Sie auf das Bearbeitungssymbol wie oben. Dadurch öffnet sich ein neues Fenster, in dem (E,F) die X- und Y-Offsets wie gezeigt geändert werden müssen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
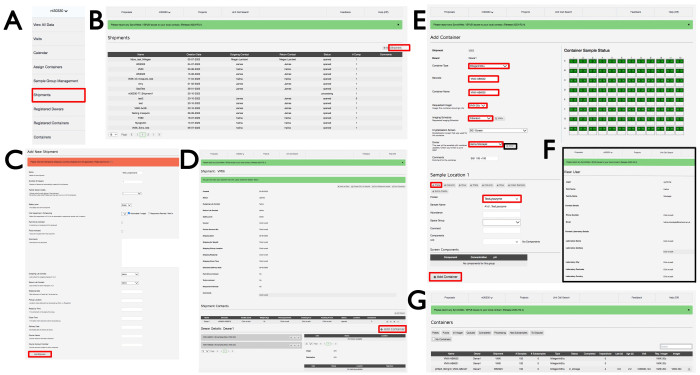
Abbildung 3: Die SynchWeb-Schnittstelle zeigt, wie Sie eine VMXi-Sendung erstellen, ein Kennzeichen registrieren und Kontaktdaten überprüfen. Screenshots der verschiedenen Phasen des Hochladens von Informationen in die SynchWeb-Oberfläche werden aus (A) dem Dropdown-Menü, (B,C) der Registrierung einer neuen Sendung, (D) der Registrierung eines neuen Containers, (E) der Eingabe der Kennzeicheninformationen, (F) der Überprüfung der Kontaktdaten und (G) einer Liste der registrierten Container innerhalb eines Angebots angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
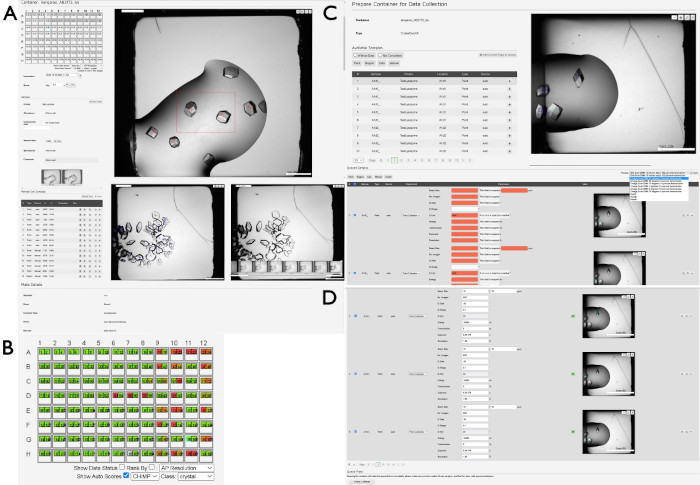
Abbildung 4: Auswahl und Vorbereitung von Proben für die Datenerfassung mit SynchWeb. Es wird eine Reihe von Screenshots angezeigt, die die verschiedenen Phasen der Vorbereitung von Proben für die Datenerfassung über die SynchWeb-Schnittstelle zeigen. (A) Points und Regions of Interest werden aus der Drop-Übersicht ausgewählt. Im unteren Teil dieser Tafel befindet sich eine chronologische Serie von Fotografien eines Tropfens. (B) Ein Beispiel für die CHiMP-Ausgabe für eine Platte, bei der die Ergebnisse für die Kategorie "Kristall" hervorgehoben werden. (C) Hinzufügen von Stichproben zur Warteschlange aus der Liste der ausgewählten Punkte und Regionen und (D) Anwenden von Parametern für die Datenerfassung auf die in der Warteschlange befindlichen Proben aus der Dropdown-Liste der von der Beamline erstellten Experimenteinstellungen. Beachten Sie den Unterschied zwischen Proben ohne experimentelle Parameter (rot) und solchen, bei denen die Parameter korrekt angewendet wurden (oben und unten). Am unteren Rand dieses Fensters befindet sich die Schaltfläche Warteschlangencontainer , mit der die zu sammelnde Platte an der Beamline in die Warteschlange gestellt wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
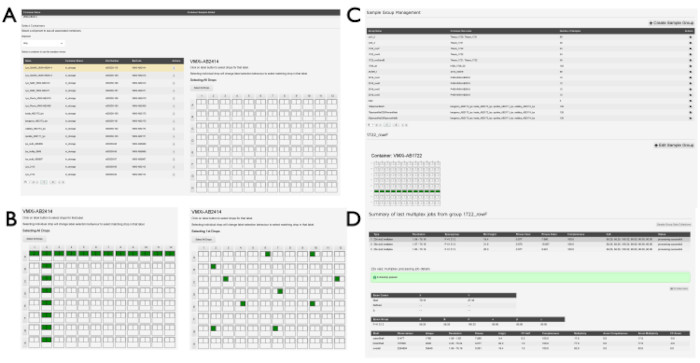
Abbildung 5: Beispielgruppenerstellung in SynchWeb. Eine Reihe von Screenshots, die die verschiedenen Phasen der Erstellung von Beispielgruppen zeigen. (A) Die Platte(n) mit den Proben werden aus der entsprechenden Sendung ausgewählt und (B) die Tropfen innerhalb der Platte werden ausgewählt. Dabei kann es sich um einzelne Tropfen handeln oder es kann zeilen- und/oder spaltenweise ausgewählt werden. (C) Eine Liste der Stichprobengruppen, die bereits erstellt wurden. (D) Die Ausgaben der letzten drei Multiplex-Verarbeitungsaufträge werden aufgelistet und können ausgewählt werden, um Statistiken aus der Verarbeitungspipeline anzuzeigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
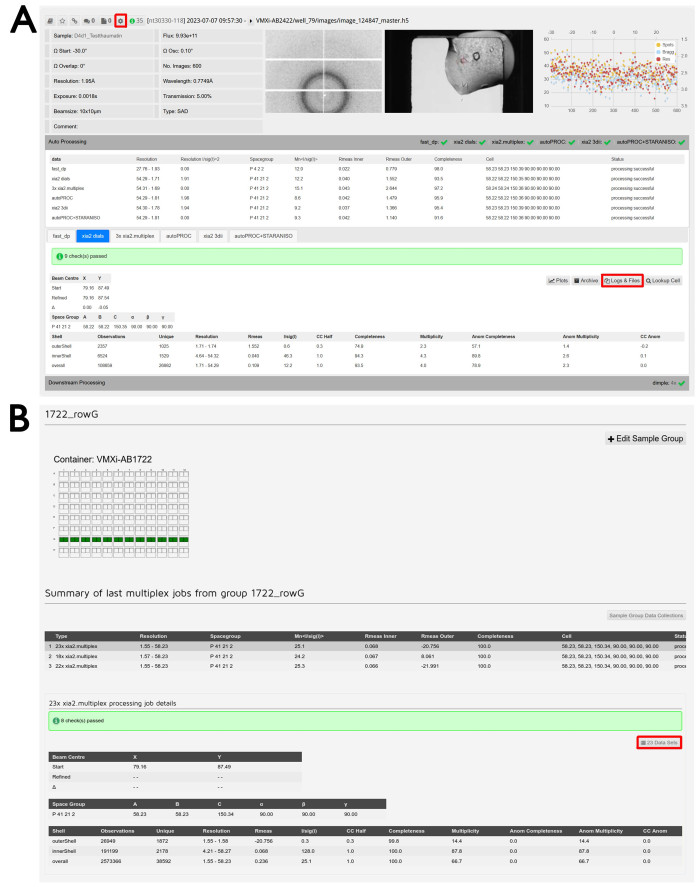
Abbildung 6: Datenverarbeitung und Datenreduktion. (A) Screenshot eines verarbeiteten Datensatzes in ISPyB11. Die Schaltfläche für den Zugriff auf die Aufbereitungsfunktionen ist hervorgehoben. Die Proben-ID und die experimentellen Parameter sind oben links und der Beugungsbildbetrachter in der Mitte dargestellt. Wenn Sie auf dieses Bild klicken, öffnet sich ein interaktives Fenster, in dem Sie verschiedene Bilder untersuchen können. Der Crystal Image Viewer wird auf der rechten Seite angezeigt und ein Klick auf dieses Bild öffnet auch ein interaktives Fenster, in dem Sie Beamline- und Formulatrix-Speicherbilder vergleichen können. Das Analysediagramm pro Bild wird ganz rechts angezeigt, und wenn Sie auf dieses Bild klicken, wird eine vergrößerte Version dieser Ausgabe geöffnet. Wenn Sie auf die Registerkarte Automatische Verarbeitung klicken, wird die automatische Verarbeitung sichtbar und der Vergleich zwischen den Ergebnissen der verschiedenen Pipelines wird erleichtert. Klicken Sie auf die Registerkarten, um zwischen den verschiedenen Verarbeitungspipelines zu wechseln und die detaillierte Ausgabe der ausgewählten Pipeline anzuzeigen. Die Schaltfläche Protokolle und Dateien zum Herunterladen von Daten ist hervorgehoben. Wenn Sie auf die Registerkarte "Downstream-Verarbeitung " klicken, werden die Ergebnisse für alle Datensätze angezeigt, die gegebenenfalls durch Pipelines nach der Datenreduzierung ausgeführt werden. (B) Screenshot aus dem Bildschirm " Sample Group Management ". Der benutzerdefinierte Gruppenname befindet sich oben und die visuelle Beschreibung der enthaltenen Wells ist unten zu sehen. Eine grüne Vertiefung zeigt an, dass alle Kristalle, die von diesem Tropfen gemessen werden, in die Gruppe aufgenommen werden. Eine Zusammenfassung der verschiedenen Multiplex-Jobs, die auf dieser Gruppe ausgeführt wurden, ist zu sehen, und darunter befindet sich die detaillierte Ausgabe von Multiplex. Die Schaltfläche Datensätze zum Untersuchen der enthaltenen Experimente ist hervorgehoben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
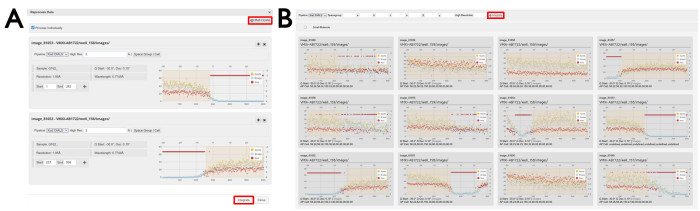
Abbildung 7: Fenster für die Datenwiederverarbeitung. (A) Individuelle und (B) multikristalline Datensätze. Es werden zwei einzelne Datensätze angezeigt, in denen Datenbereiche ausgewählt wurden. Wenn das Kontrollkästchen Einzeln bearbeiten aktiviert ist, werden die ausgewählten Beugungsbilder einzeln bearbeitet, indem Sie auf die Schaltfläche Integrieren klicken. Wenn Sie auf die Schaltfläche Multikristall klicken, wird eine Anzeige der einzelnen Datensätze geöffnet. Um Beugungsbilder aus mehreren Datensätzen erneut zu verarbeiten, werden Bildbereiche wie angezeigt ausgewählt, und die erneute Verarbeitung wird durch Klicken auf die Schaltfläche Integrieren wie hervorgehoben eingeleitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 8: Repräsentative Ergebnisse aus der VMXi-Pipeline. (A) Markierte Kristalle für das Protein Thaumatin innerhalb eines Kristallisationstropfens (linkes Bild), Datenverarbeitungsergebnisse (mittleres Bild) und Elektronendichte (rechtes Bild). (B) Sammlung an mehreren Kristallen, um die Bindung des Fragments an die SARS-CoV-2-Makrodomäne zu bestimmen. Die Datensätze wurden an mehreren Kristallen in Gegenwart eines Fragments aus dem EU-OPENSCREEN-Fragmentbildschirm unter Verwendung von Standard-Versuchseinstellungen gesammelt. Beispiele für diese Datensammlungen sind in diesem Auszug aus SynchWeb dargestellt. Das Fragment wurde in die entsprechende Dichte eingebaut und weiter verfeinert, wie am weitesten rechts zu sehen ist. (C) Markierte monokline Kristalle in einem Stapel aus einem schwierigen Kristallisationstreffer, der für die Datenerfassung verwendet wird. Grüne Kreuze und rote Zahlen zeigen an, wo die Daten mit einem 10-μm-Strahl und einer 60°-Drehung gemessen wurden. Vier der resultierenden Keile wurden zu einem Datensatz mit einer Auflösung von 1,75 Å zusammengeführt. Die Elektronendichte um die Häm-Gruppe wird auf der rechten Seite angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 9: Serielle Kristallographie in der Kristallisationsplatte. (A) Optisches Bild des Kristallisationstropfens mit einem weißen Kasten, der den interessierenden Bereich darstellt. (B) Definition von Raster-Scan-Punkten. (C) Heatmap, die die Beugung anzeigt. (D) Elektronendichtekarte, die sich aus einem seriellen Kristallographie-Datensatz von mehr als 9.000 Beugungsmustern ergibt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Auflösung (Å) | Vollständigkeit (%) | Multiplizität | I/σ(I) | Rgeteilt | CC1/2 | Einzigartige Beobachtungen |
| Insgesamt | 100 | 95.5 | 20.8 | 0.063 | 0.998 | 8422 |
| niedrig (55.55 - 5.43) | 100 | 147.1 | 81.7 | 0.028 | 0.999 | 488 |
| Hoch (2.03 -2.00) | 100 | 75.3 | 1.2 | 1.092 | 0.410 | 411 |
Tabelle 1: Datenstatistiken für den seriellen VMXi RT-Datensatz. Abkürzungen: I = mittlere Intensität der skalierten Beobachtungen; Rsplit = ein Maß für die Diskrepanz der gemessenen Intensitäten; CC 1/2 = Korrelationskoeffizient zwischen zwei zufälligen Hälften des Datensatzes.
Diskussion
Wir haben den gesamten Ablauf von der Ankunft einer Proteinprobe in der Mukoviszidose bis zum Herunterladen der finalen Daten durch den Anwender für weitere Anwendungen beschrieben. Kritische Schritte sind die Herstellung einer qualitativ hochwertigen Proteinprobe und geeigneter Kristallsiebe, entweder unter Verwendung kommerzieller dünnbesetzter Matrixsiebe oder Optimierungssiebungen auf der Grundlage etablierter Bedingungen. Dieser Prozess kann im CF stattfinden, oder Anwender können die Kristallisationsvorgänge in den Heimlaboren durchführen und geeignete Kristallisationsplatten an die Beamline bringen. Die Identifizierung geeigneter Datenerfassungsparameter kann für bestimmte Proben wichtig sein, insbesondere wenn Strahlenschäden ein Problem darstellen. In den meisten Fällen ist die automatisierte Datenverarbeitung völlig ausreichend, um die wissenschaftliche Fragestellung zu beantworten, obwohl die Benutzer weiterhin die Möglichkeit haben, mit den Beamline-Werkzeugen nachzuverarbeiten, z. B. wenn die Raumgruppe mehrdeutig ist oder nur der erste Teil der gesammelten Daten verwendet wird, um die Auswirkungen von Strahlenschäden zu minimieren.
Wenn aus anfänglichen Kristallisationsversuchen keine geeigneten Kristalle hergestellt werden, können Änderungen der Proteinkonzentration, der Reinheit oder der Kristallisationssiebe untersucht werden, ebenso wie die Verwendung von Kristallimpfungen. Wenn Kristalle an der Beamline nicht auf eine brauchbare Auflösung gebeugt werden, können Gitterscans mit einem nicht abgeschwächten Strahl verwendet werden, um die inhärente Beugungsgrenze und die Elementarzelle der Kristalle zu beurteilen und die Optimierungsbemühungen zu steuern. Kristalle, die für die Datenerfassung innerhalb von Platten zu klein sind (z. B. <10 μm), können stattdessen für serielle Kristallographie oder Nanofokusexperimente (z. B. an der Diamant-Beamline VMXm) geeignet sein. Das Lösen von Strukturen unter Verwendung von VMXi-Daten ist im Allgemeinen einfach durch molekularen Ersatz, insbesondere seit dem Aufkommen von Alphafold16 , um effektive Suchmodelle zu erhalten. Wenn dies nicht erfolgreich ist, können Kristalle geerntet und von Platten kryogekühlt werden, um konventionelle Experimente mit anomaler Beugung einzelner Wellenlängen, anomaler Beugung mit mehreren Wellenlängen oder Experimenten mit langen Wellenlängenphasen zu ermöglichen.
Zu den Vorteilen dieser Methode gehört die Möglichkeit, schnelle, qualitativ hochwertige Datensätze und Rückmeldungen direkt von Kristallisationsplatten zu erhalten, ohne dass Kristalle aus den Umgebungen, in denen sie gewachsen sind, gestört werden müssen. Die sogenannte "Raumtemperatur-Renaissance" in der Strukturbiologie legt Wert auf Strukturen, die unter nicht-kryogenen Bedingungen gewonnen wurden, um mehr physiologische Relevanz und Proteindynamik zu erforschen2. In der Regel wird eine etwas geringere Auflösung als bei einem optimierten kryogekühlten Kristall erreicht, jedoch nur, wenn geeignete Kryobedingungen hergestellt wurden und wenn die Kristalle robust gegenüber mechanischer Handhabung und Öffnung des Kristallisationstropfens3 sind. Eine bevorstehende Anwendung, für die sich diese Pipeline sehr gut eignet, ist ein großflächiges Screening von Protein-Liganden-Komplexen oder Fragmentkampagnen bei Raumtemperatur in der Wirkstoffforschung. Liganden oder Fragmente können entweder kokristallisiert oder durch Pipette oder akustisches Tropfenauswerfen vor der Datenerfassung bei Raumtemperatur hinzugefügt werden. Eine weitere Anwendung besteht darin, Daten von vielen Hunderten oder Tausenden von Kristallen schnell auf hocheffiziente Weise zu messen und dann die Software DIALS17 Multiplex14 zu verwenden, um isomorphe Cluster zu extrahieren, die verschiedene biologische Einheiten darstellen können, oder um statistisch signifikante Unterschiede zwischen Populationen von Kristallen festzustellen, die auf unterschiedliche Weise behandelt oder anderen Liganden oder Signalen ausgesetzt wurden.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Wir danken den vielen Wissenschaftlern der Diamond Light Source und den Mitgliedern des Support-Teams, die zur Planung, zum Bau und zum Betrieb der VMXi-Beamline beigetragen haben. Wir danken den Beamline-Nutzern, die später Ideen zur Entwicklung der Kristallisations- und Datensammelpipelines beigetragen haben. Die Kristallisationsanlage in Harwell wird von Diamond Light Source Ltd., dem Rosalind Franklin Institute und dem Medical Research Council unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Formulator | Formulatrix | on request | Liquid handling robot |
| Formulatrix imager | Formulatrix | on request | Crystallisation plate imager |
| Greiner CrystalQuick X | Greiner | Z617644 | Crystallisation plate |
| Gryphon | Art Robbins Instruments | 620-1000-10 | Crystalisation robot |
| MiTeGen Insitu-1 | Mitegen | InSitu-01CL-40 | Crystallisation plate |
| Mosquito LCP | (SPT Labtech) | on request | Crystallisation robot |
| Rock Imager & Maker | Formualtrix | on request |
Software for Imager [1] https://formulatrix.com/protein-crystallization-systems/rock-maker-crystallization-software/ |
| Scorpion | Art Robbins Instruments | 640-1000-10 |
Liquid handling robot https://www.artrobbins.com/scorpion |
Referenzen
- Lynch, M. L., Snell, M. E., Potter, S. A., Snell, E. H., Bowman, S. E. J. 20 years of crystal hits: Progress and promise in ultrahigh-throughput crystallization screening. Acta Crystallographica Section D Structural Biology. 79 (Pt 3), 198-205 (2023).
- Fischer, M. Macromolecular room temperature crystallography. Quarterly Reviews of Biophysics. 54, 1 (2021).
- Helliwell, J. R. What is the structural chemistry of the living organism at its temperature and pressure. Acta Crystallographica Section D Structural Biology. 76 (Pt 2), 87-93 (2020).
- Thorne, R. E. Determining biomolecular structures near room temperature using x-ray crystallography: Concepts, methods and future optimization. Acta Crystallographica Section D Structural Biology. 79 (Pt 1), 78-94 (2023).
- Keedy, D. A., et al. Crystal cryocooling distorts conformational heterogeneity in a model michaelis complex of dhfr. Structure. 22 (6), 899-910 (2014).
- Huang, C. Y., et al. Probing ligand binding of endothiapepsin by 'temperature-resolved' macromolecular crystallography. Acta Crystallographica Section D Structural Biology. 78 (Pt 8), 964-974 (2022).
- Sanchez-Weatherby, J., et al. Vmxi: A fully automated, fully remote, high-flux in situ macromolecular crystallography beamline. Journal of Synchrotron Radiation. 26 (Pt 1), 291-301 (2019).
- Jacquamet, L., et al. Automated analysis of vapor diffusion crystallization drops with an x-ray beam. Structure. 12 (7), 1219-1225 (2004).
- Mikolajek, H., et al. Protein-to-structure pipeline for ambient-temperature in situ crystallography at vmxi. IUCrJ. 10, 420-429 (2023).
- Douangamath, A., et al. Achieving efficient fragment screening at xchem facility at diamond light source. Journal of Visualised Experiments. (171), (2021).
- Delageniere, S., et al. Ispyb: An information management system for synchrotron macromolecular crystallography. Bioinformatics. 27 (22), 3186-3192 (2011).
- Fisher, S. J., Levik, K. E., Williams, M. A., Ashton, A. W., Mcauley, K. E. Synchweb: A modern interface for ispyb. Journal of Applied Crystallography. 48 (Pt 3), 927-932 (2015).
- Jenkins, J., Cook, M. Mosquito®: An accurate nanoliter dispensing technology. JALA: Journal of the Association for Laboratory Automation. 9 (4), 257-261 (2016).
- Gildea, R. J., et al. Xia2.Multiplex: A multi-crystal data-analysis pipeline. Acta Crystallographica Section D Structural Biology. 78 (Pt 6), 752-769 (2022).
- Winter, G., Mcauley, K. E. Automated data collection for macromolecular crystallography. Methods. 55 (1), 81-93 (2011).
- Jumper, J., et al. Highly accurate protein structure prediction with alphafold. Nature. 596 (7873), 583-589 (2021).
- Winter, G., et al. Dials as a toolkit. Protein Science. 31 (1), 232-250 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten