Method Article
PyDesigner v1.0: Eine pythonische Implementierung der DESIGNER-Pipeline für die diffusionsmagnetische Resonanztomographie
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier stellen wir PyDesigner vor, eine Python-basierte Verarbeitungspipeline für die Diffusionsmagnetresonanztomographie (dMRT), die in der Lage ist, typische dMRT-Artefakte zu korrigieren und Diffusions-Tensor-Imaging (DTI), Diffusions-Kurtosis-Imaging (DKI), Faserkugel-Imaging (FBI), Mikrostrukturmodellierung (Integrität der weißen Substanz [WMTI] und Faserkugel-weiße Substanz [FBWM]) und Traktographie-Ausgaben zu erzeugen.
Zusammenfassung
PyDesigner ist ein Python-basiertes Softwarepaket, das auf dem ursprünglichen Diffusionsparameter EStImation with Gibbs and NoisE Removal (DESIGNER) Pipeline (Dv1) für die dMRT-Vorverarbeitung und Tensorschätzung basiert. Diese Software wird offen für nicht-kommerzielle Forschung zur Verfügung gestellt und darf nicht für die klinische Versorgung verwendet werden. PyDesigner kombiniert Werkzeuge von FSL und MRtrix3 zur Entrauschung, Gibbs-Ringing-Korrektur, Wirbelstrom-Bewegungskorrektur, Gehirnmaskierung, Bildglättung und Rician-Bias-Korrektur, um die Schätzung mehrerer Diffusionsmaße zu optimieren. Es kann plattformübergreifend unter Windows, Mac und Linux verwendet werden, um häufig verwendete Metriken aus DKI-, DTI-, WMTI-, FBI- und FBWM-Datensätzen sowie Traktographie-ODFs und .fib-Dateien genau abzuleiten. Es ist auch dateiformatunabhängig und akzeptiert Eingaben in Form der Formate .nii, .nii.gz, .mif und dicom. Diese Software ist benutzerfreundlich und einfach zu installieren und gibt auch Qualitätskontrollmetriken aus, die Signal-Rausch-Verhältnisdiagramme, Ausreißervoxel und Kopfbewegungen veranschaulichen, um die Datenintegrität zu bewerten. Darüber hinaus unterstützt diese dMRT-Verarbeitungspipeline die Verarbeitung mehrerer Echozeit-Datensätze und bietet eine Pipeline-Anpassung, die es dem Benutzer ermöglicht, anzugeben, welche Prozesse eingesetzt und welche Ausgaben erzeugt werden, um eine Vielzahl von Benutzeranforderungen zu erfüllen.
Einleitung
Die Diffusions-MRT (dMRT) wird häufig für die nicht-invasive Untersuchung der mikrostrukturellen Eigenschaften im Gehirn eingesetzt. Während viele dMRT-Methoden vorgeschlagen wurden, sind zwei häufig verwendete die Diffusions-Tensor-Bildgebung (DTI) und die Diffusionskurtosis-Bildgebung (DKI). Diese Techniken sind eng miteinander verwandt, wobei DKI eine Erweiterung von DTI ist, die die Quantifizierung der diffusionsbedingten Nicht-Gaußianität1 umfasst. Beide bieten eine Vielzahl von skalaren Diffusionsmaßen und ermöglichen die Konstruktion der Fasertraktographie der weißen Substanz. Ein wichtiger Vorteil von DTI und DKI besteht darin, dass sie eine solide Grundlage in der Diffusionsphysik haben, so dass ihre Gültigkeit nicht von detaillierten Annahmen über die Gewebemikrostruktur abhängt 2,3. Dies ermöglicht die Anwendung von DTI und DKI im gesamten Gehirn und Körper für Teilnehmer jeden Alters und Krankheitsstatus.
Da diffusionsgewichtete Rohbilder (DWIs) durch mehrere Faktoren beeinträchtigt werden, darunter Signalrauschen, Bewegung, Gibbs-Ringing und Wirbelstromverzerrung, sollte vor der Berechnung von Diffusionsgrößen eine Vorverarbeitung durchgeführt werden4. Die Vorverarbeitung von DWIs ist ein aktives Forschungsfeld, das mittlerweile hoch entwickelt ist. Die Kombination der notwendigen Verarbeitungsschritte in einer einzigen Pipeline, die konsistente Ergebnisse liefert, ist jedoch eine Herausforderung, da je nach den Details der dMRT-Aufnahme mehrere benutzerdefinierte Einstellungen angepasst werden müssen und die Reihenfolge, in der die Vorverarbeitungsschritte durchgeführt werden, das Ergebnis beeinflusst. Aus diesem Grund wurde die Diffusionsparameter-EStImation mit Gibbs und NoisE Removal (DESIGNER, GitHub: NYU-DiffusionMRI/DESIGNER) ursprünglich im Jahr 2016 vorgeschlagen, um die Vorverarbeitung für DWIs zu optimieren, zu standardisieren und zu rationalisieren5. DESIGNER Dv1 stützt sich auf Software-Tools, die in FSL, MRtrix3, MATLAB und Python eingebettet sind, um einen nahtlosen und vollständigen DWI-Prozess zu erstellen, der die Bildkorrektur durch Vorverarbeitung und die Schätzung von Diffusions-/Kurtosis-Tensoren umfasst5. Mit Steuerflags zum Ein- oder Ausschalten von Vorverarbeitungsschritten können DWI-Korrekturen selektiv durchgeführt werden. DESIGNER verarbeitet in einer bestimmten Reihenfolge vor: (i) Marchenko-Patur-Hauptkomponentenanalyse (MP-PCA) Rauschunterdrückung6, (ii) Gibbs-Ringing-Korrektur7, (iii) Verzerrungskorrektur der echoplanaren Bildgebung (EPI)8, Wirbelstromkorrektur9, Bewegungskorrektur10 und Ausreißersetzung11, (iv) B1-Bias-Feldkorrektur, (v) Erzeugung von Gehirnmasken, (vi) Glättung, (vii) Korrektur der ricianischen Rauschverzerrung und (viii) b0-Normalisierung. Die Vorverarbeitung in dieser speziellen Reihenfolge verbessert sowohl die Genauigkeit als auch das effektive Signal-Rausch-Verhältnis (SNR)5. Es ist erwähnenswert, dass jeder Schritt von PyDesigner optional ist und je nach Benutzerpräferenz verwendet oder übersprungen werden kann. Das Glätten ist z. B. für einige Datasets möglicherweise kein notwendiger Vorverarbeitungsschritt. Obwohl es hilft, Filterfehler zu verringern, ist es möglicherweise nicht erforderlich, wenn es sich um Datasets mit extrem hoher Qualität handelt. Auf diese Weise können Benutzer auswählen, ob sie nur die Schritte ausführen möchten, die für ihre Daten erforderlich sind.
Die plattformübergreifende Implementierung von DESIGNER ist aufgrund von Unterschieden bei Betriebssystemen und Umgebungseinstellungen eine Herausforderung. Insbesondere die Tatsache, dass DESIGNER Dv1 hauptsächlich in MATLAB geschrieben ist, führt zu erheblichen Portabilitätsproblemen, die sich aus komplizierten Konfigurationsanforderungen ergeben, die für die Aktivierung der Python-MATLAB-Schnittstelle erforderlich sind. Darüber hinaus beeinträchtigen unterschiedliche Kombinationen von MATLAB-, Python- und Abhängigkeitsversionen die Reproduzierbarkeit dieser Pipeline. Aus diesem Grund haben wir den PyDesigner entwickelt, der vollständig auf Python basiert. Diese dMRT-Verarbeitungspipeline ermöglicht nicht nur eine nahtlose Vorverarbeitung, sondern ermöglicht auch die Integration der Pipeline in einen Docker-Container, was die Portabilität und Reproduzierbarkeit erheblich verbessert. Darüber hinaus vermeidet PyDesigner durch den Ersatz des MATLAB-Codes alle Lizenzgebühren und verbessert die Zugänglichkeit.
Diese dMRT-Verarbeitungspipeline erweitert den mit DESIGNER eingeführten freihändigen Ansatz, fügt mehrere neue Funktionen hinzu und enthält Tools von FSL und MRtrix3 zur Durchführung der Vorverarbeitung. Standard-mathematische Python-Bibliotheken wie Numpy12, SciPy13 und CVXPY14,15 wurden verwendet, um die MATLAB-Teile von DESIGNER durch Python-Code zu ersetzen. Diese Software wird offen zur Verfügung gestellt und ist auf Github16 verfügbar. Das Ziel dieses Dokuments ist es nicht, unsere Software zu validieren oder mit ähnlicher Software zu vergleichen, sondern den Benutzern eine Schritt-für-Schritt-Anleitung für die Verarbeitung ihrer Daten mit PyDesigner zu geben, falls sie sich dafür entscheiden.
Protokoll
Alle Daten, die für die Entwicklung und den Test der Software verwendet wurden, wurden im Rahmen eines von der institutionellen Prüfkommission genehmigten Studienprotokolls gesammelt.
HINWEIS: PyDesigner wurde unter MacOS 10.14 erstellt und erfordert ein Windows-Subsystem für Linux (WSL), um unter Windows ausgeführt zu werden. Die gleichen Befehle können für Linux/Mac-Systeme verwendet werden.
1. Kompatibilität mit dem Betriebssystem
- Für Windows-Systeme folgen Sie dem Link zum Installieren von WSL17 , und fahren Sie dann mit Schritt 2.6 fort.
- Für Unix-basierte Systeme (wie Linux und Mac) fahren Sie mit Abschnitt 2 fort.
HINWEIS: Unix-basierte Systeme können PyDesigner und seine Abhängigkeiten nativ installieren.
2. FSL installieren
HINWEIS: Alle PyDesigner-Tests wurden mit FSL Version 6.0.2 durchgeführt, und die Verwendung von v6.0.2 oder höher wird empfohlen.
- Gehen Sie zum FSL-Wiki18 und wählen Sie Download/Installieren. Für Windows-Benutzer fahren Sie mit Schritt 2.6 fort.
- Wählen Sie Download FSL unter der Überschrift Installing FSL aus. Wählen Sie dann die FSL-Installationssoftwarelizenz aus, lesen Sie die Vereinbarung, und wählen Sie Zustimmen aus.
- Füllen Sie die erforderlichen Felder aus, und wählen Sie Registrieren aus.
- Wählen Sie den Link FSL herunterladen (erster Link auf der Seite). Wählen Sie dann den Link Installationsanleitung (zweiter Link auf der Seite). Befolgen Sie diese Anweisungen, um FSL für jedes Betriebssystem herunterzuladen.
HINWEIS: Die Installation von FSL dauert ca. 10 Minuten. - Um die erfolgreiche Installation zu überprüfen, geben Sie Folgendes in ein neues Terminalfenster ein:
$ flirt -version. Dies sollte die Versionsnummer zurückgeben. Fahren Sie mit Schritt 3 fort. - Windows-Benutzer befolgen die WSL-Installationsanleitung für FSL19.
3. Erstellen Sie eine Conda-Umgebung
- Installieren Sie ein Conda-Paket, um Python-Umgebungen zu verwalten. Der Benutzer kann zwischen Miniconda und Anaconda wählen.
- Laden Sie Miniconda/Anaconda20 herunter. Mac- und Linux-Benutzer fahren mit Schritt 3.2 fort.
- Richten Sie unter Windows conda in WSL21 ein.
HINWEIS: Die folgenden Anweisungen gelten für Miniconda, aber die gleichen Befehle funktionieren auch für Anaconda. Obwohl conda Python 3.7 nicht mehr unterstützt, ist es immer noch möglich, mit Python 3.7 eine conda-Umgebung zu erstellen, so dass dieses Protokoll genau wie beschrieben implementiert werden kann.
- Geben Sie folgendes in das Terminal ein, um die Conda-Umgebung zu erstellen:
$ conda create -n mri python=3.7. Verwenden Sie diese Conda-Umgebung für alle PyDesigner-bezogenen Aktivitäten.- Wenn Sie mit
Proceed ([y]/n)?Entery.
HINWEIS: -n Flag: Verwenden Sie dieses Flag, um den Namen der Conda-Umgebung anzugeben. Im Video wird die Conda-Umgebung als mrt bezeichnet.
HINWEIS:python=: Version 3.7 wird empfohlen, da PyDesigner auf dieser Version erstellt und getestet wurde.
- Wenn Sie mit
4. Installieren Sie MRtrix3
- Aktivieren Sie die Conda-Umgebung, indem Sie den folgenden Befehl eingeben:
$ conda activate mri - Öffnen Sie die Download-Seite für MRtrix322 und folgen Sie den Download-Anweisungen für das entsprechende Betriebssystem. Der Installationscode fragt nach der Bestätigung bestimmter Abhängigkeiten. Geben Sie die Eingabetaste
yein, um den Download-Vorgang fortzusetzen.- Drucken Sie die MRtrix3-Dokumentation aus, um die Installation zu bestätigen, indem Sie Folgendes in das Terminal eingeben:
$ mrinfo -h
- Drucken Sie die MRtrix3-Dokumentation aus, um die Installation zu bestätigen, indem Sie Folgendes in das Terminal eingeben:
5. Installieren Sie PyDesigner
- Öffnen Sie ein neues Terminalfenster und aktivieren Sie die Conda-Umgebung, indem Sie Folgendes in das Terminal eingeben:
$ conda activate mri - Führen Sie den folgenden Befehl aus:
$ pip install PyDesigner-DWI - Um die erfolgreiche Installation zu überprüfen, drucken Sie die Versionsnummer aus, indem Sie Folgendes in das Terminal eingeben:
$ pydesigner -v- Wenn der Fehler "Modul nicht gefunden" angezeigt wird, lesen Sie bitte den Abschnitt "Fehlerbehebung" am Ende des Protokolls (Abschnitt 10)
HINWEIS: $ pydesigner -h gibt eine Hilfedokumentation aus, die die Befehle und die Syntax für die Software erklärt. PyDesigner kann auch über Github heruntergeladen werden. Siehe die Installationsanleitung auf der Website23.
- Wenn der Fehler "Modul nicht gefunden" angezeigt wird, lesen Sie bitte den Abschnitt "Fehlerbehebung" am Ende des Protokolls (Abschnitt 10)
6. Datenaufbereitung
HINWEIS: PyDesigner funktioniert mit mehreren Dateitypen wie .nii, .nii.gz, .mif und DICOM. Im Video werden DICOMs sortiert und in NIfTI konvertiert, bevor diese dMRT-Verarbeitungspipeline verwendet wird. NIfTI-Dateien sind einfacher zu bearbeiten, da sie nicht identifiziert werden können und es eine Datei pro Sequenz gibt.
- Um die DICOMs zu sortieren, verwenden Sie das DicomSort-Werkzeug24.
HINWEIS: Jedes andere verfügbare DICOM-Sortierwerkzeug, das die gleiche Aufgabe erfüllt, ist zulässig. - Nachdem Sie die DICOMs sortiert haben, konvertieren Sie sie mit dcm2niix25 in NIfTI-Dateien.
- Installieren Sie dcm2niix, indem Sie den folgenden Befehl in das Terminal eingeben:
$ sudo apt installdcm2niix - Geben Sie nach dem Herunterladen Folgendes in das Terminal ein, um die Installation zu überprüfen, indem Sie die Dokumentation für dcm2nii drucken:
$ dcm2niix -h
- Installieren Sie dcm2niix, indem Sie den folgenden Befehl in das Terminal eingeben:
- Um dcm2niix auszuführen, geben Sie den folgenden Befehl in das Terminal ein:
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
HINWEIS: -f: Um die Dateinamensstruktur für die Ausgabedatei anzugeben, fügen Sie diesem Tag eine beliebige Dateinamensstruktur hinzu. Das Video verwendet: -f %s_%p_%d . %s, %p und %d sind Notationen für Reihe, Protokollnummer bzw. Beschreibung. Jede Dateistruktur funktioniert. Erwartete Ausgaben von dcm2niix sind .bval-, .bvec-, .json- und .nii-Dateien. Wenn TOPUP-Daten erfasst wurden, gibt es .json- und .nii TOPUP-Dateien.
7. Grundlegende Verwendung von PyDesigner
HINWEIS: PyDesigner kann je nach Verarbeitungsparametern und Computerfunktionen bis zu 1,5 Stunden für einen einzelnen Datensatz benötigen.
- Um die Standardverarbeitung auszuführen, verwenden Sie das Flag
-soder-standard. Mit diesem Befehl wird die empfohlene Vorverarbeitungspipeline in der folgenden Reihenfolge ausgeführt: Rauschunterdrückung6, Gibbs-Ringing-Korrektur7, EPI-Verzerrungskorrektur8, Wirbelstrombewegungskorrektur9, Erstellung von Gehirn- und Liquormasken, Glättung, Rician-Bias-Korrektur, Ausreißererkennung11, Tensorschätzung und Erstellung parametrischer Karten. - Um den grundlegenden PyDesigner-Befehl auszuführen, aktivieren Sie zunächst die Conda-Umgebung, indem Sie den folgenden Befehl in das Terminal eingeben:
$ conda activate mri - Drucken Sie die Hilfedokumentation für die Software aus, indem Sie den folgenden Befehl eingeben:
$ pydesigner -h. In dieser Dokumentation werden alle möglichen Befehle und die Syntax für PyDesigner erläutert. - Verwenden Sie das folgende Beispiel, um den Befehl zu strukturieren.
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - Beachten Sie die folgenden wichtigen Flags:
--verbose: Verwenden Sie diese Option, um die PyDesigner-Konsole anzuzeigen, während sie Daten verarbeitet (nicht erforderlich).--rpe pairs #: Verwenden Sie diese Option, um Benutzern mit einer TOPUP-Sequenz zu ermöglichen, den Prozess der EPI-Verzerrungskorrektur8 mithilfe von TOPUP zu beschleunigen. Das # bezieht sich darauf, wie viele B0-Datenträger aus der TOPUP-Sequenz verwendet werden. Die Verwendung von #=1 verhindert eine Überschätzung des Verzerrungsfeldes und reduziert die Zeit, die zum Erstellen dieses Feldes benötigt wird. Benutzer haben routinemäßig #=1 mit zuverlässigen Ergebnissen verwendet. Wenn Sie #=1 verwenden, nimmt PyDesigner standardmäßig das erste Volume.
8. Erweiterte PyDesigner-Nutzung: Anpassung der Software an die individuellen Bedürfnisse der Benutzer
HINWEIS: Lassen Sie beim Anpassen von PyDesigner das -s Flag weg; dieses Flag ist der Befehl für die automatische Vorverarbeitung.
- Aktivieren Sie die Conda-Umgebung, indem Sie den folgenden Befehl in das Terminal eingeben:
$ conda activate mri - Auf der Websiteder Software 23 finden Sie eine Liste der Flags; Jedes Flag ermöglicht es dem Benutzer, jeden Teil von PyDesigner einzeln auszuführen.
- Strukturieren Sie den Befehl. Beginnen Sie mit den Flags für jede Sequenz, die der Benutzer ausführen möchte. Leiten Sie dann PyDesigner zu den Eingabe- und Ausgabedateien. Zum Beispiel:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - Beispiel 1: Führen Sie die Tensoranpassung alleine durch (keine zusätzliche Vorverarbeitung).
- Aktivieren Sie die Conda-Umgebung, indem Sie den folgenden Befehl in das Terminal eingeben:
$ conda activate mri - Auf der PyDesigner-Website23 finden Sie eine Liste der Flags. In diesem Beispiel wird
-m. -m: Fügen Sie dieses Flag hinzu, um die Tensoranpassung nur innerhalb von Gehirnvoxeln zu begrenzen. Dies beschleunigt die Tensoranpassung + Ausreißererkennung.- Verwenden Sie das folgende Beispiel, um den Befehl zu strukturieren:
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
HINWEIS: Die Eingabe für die Tensoranpassung ist eine vorverarbeitete .nii-Datei.
- Aktivieren Sie die Conda-Umgebung, indem Sie den folgenden Befehl in das Terminal eingeben:
- Beispiel 2: Rauschunterdrückung allein ausführen (keine zusätzliche Vorverarbeitung).
- Aktivieren Sie die conda-Umgebung, indem Sie Folgendes in das Terminal eingeben:
$ conda activate mri - Auf der PyDesigner-Website23 finden Sie eine Liste der Flags. In diesem Beispiel wird
-n. - -n: Fügen Sie dieses Flag hinzu, um den Rauschunterdrückungsschritt abzuschließen. Geben Sie die zuvor vorverarbeitete DWI-Datei mit der Bezeichnung DWI_preproccessed.nii ein, die sich im Ausgabeordner jedes Betreffs befindet.
- Verwenden Sie das folgende Beispiel, um den Befehl zu strukturieren:
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- Aktivieren Sie die conda-Umgebung, indem Sie Folgendes in das Terminal eingeben:
9. Zukünftige PyDesigner-Updates
HINWEIS: Aktualisierungen finden Sie auf der Website23.
- Um PyDesigner zu aktualisieren, geben Sie Folgendes in ein neues Terminalfenster ein:
$ pip install --upgrade PyDesigner-DWI
HINWEIS: Wenn PyDesigner über Github16 installiert wurde, muss es auch mit git aktualisiert werden.
10. Fehlerbehebung bei der Installation
- Beim Herunterladen von PyDesigner wird der Fehlercode "Modul nicht gefunden" angezeigt, wenn ein Python-Modul fehlt.
- Beheben Sie ein fehlendes Modul, indem Sie Folgendes in das Terminal eingeben:
$ pip install nameofmodule - Wiederholen Sie dann den ersten Installationsbefehl, indem Sie Folgendes in das Terminal eingeben:
$ pip install PyDesigner-DWI - PyDesigner sollte jetzt vollständig installiert sein. Bitte kehren Sie zu Schritt 5.3 zurück. Wenn derselbe Fehler auftritt, wiederholen Sie diesen Vorgang, bis alle fehlenden Python-Module installiert sind.
- Beheben Sie ein fehlendes Modul, indem Sie Folgendes in das Terminal eingeben:
11. Fehlerbehebung bei Ausgabefehlern
- Wenn Probleme auftreten, überprüfen Sie die log_command.json Datei, um Vorverarbeitungsfehler zu identifizieren. Überprüfen Sie die Zwischendateien, um den spezifischen Fehler zu ermitteln.
- Wenn Sie Hilfe bei der Problembehandlung benötigen, senden Sie eine Anfrage auf der GitHub-Seite16.
12. Ausführen von PyDesigner mit einem Beispieldatensatz
- Laden Sie den Beispieldatensatz von OpenNeuro hierherunter 26.
HINWEIS: Dieser Beispieldatensatz enthält sowohl Rohdaten als auch vorverarbeitete Daten. Die Rohdaten sind als Eingabe für die PyDesigner-Beispielverarbeitung gedacht. Die vorverarbeiteten Daten bestehen aus den Ausgaben, die generiert wurden, nachdem die Rohdaten mit der dMRI-Verarbeitungspipeline auf einem iMac OS 12.4 verarbeitet wurden. Benutzer können die Rohdaten herunterladen, diese Daten mit PyDesigner verarbeiten und ihre Ausgaben mit den bereitgestellten Beispielausgaben vergleichen, um sicherzustellen, dass PyDesigner ordnungsgemäß ausgeführt wird. Die Rohdaten sind im Hauptordner (/PyDesigner Example Dataset/sub-01) enthalten, und die vorverarbeiteten Daten sind im Ordner derivatives (/PyDesigner Example Dataset/derivatives/sub-01) enthalten. Sofern nicht anders angegeben, wird der OpenNeuro-Datensatz in den Standardbenutzerordner heruntergeladen. - Öffnen Sie ein neues Terminal unter Mac/Linux oder die WSL unter Windows.
HINWEIS: Um die standardmäßige PyDesigner-Verarbeitungspipeline auszuführen, befolgen Sie die folgende Beispielbefehlsstruktur. Geben Sie die folgenden Befehle in das Terminal/WSL ein. In diesem Beispiel gibt " ./user_download_folder/" den Pfad für den Beispieldatenordner an. Dieser Pfad ist spezifisch für den Ort, an dem die Beispieldaten auf dem Computer der einzelnen Benutzer gespeichert sind. - Um einen neuen Ordner für Ausgaben zu erstellen, geben Sie folgendes in das Terminal ein:
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - Um die Conda-Umgebung zu aktivieren, geben Sie folgendes in das Terminal ein:
$ conda activate mri - Um PyDesigner für das Beispieldataset auszuführen, geben Sie Folgendes in das Terminal ein:
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
HINWEIS: In diesem Beispiel gibt ./user_download_folder/ den Pfad für den Beispieldatenordner an. Dieser Pfad ist spezifisch für den Computer jedes Benutzers. Die Befehle in den Schritten 12.3 bis 12.5 folgen der Struktur von Schritt 8.3. Dieser Befehl verwendet das Flag -s, das Standardverarbeitungsflag von PyDesigner, das die folgenden Verarbeitungsschritte in dieser Reihenfolge ausführt: Rauschunterdrückung6, Gibbs-Ringkorrektur7, Unverzerrung 8,9, Gehirnmaskierung, Glättung und Rician-Bias-Korrektur. - Wenn die Software korrekt installiert ist, werden alle Ausgaben in den neu erstellten Ordner PyDesigner_Outputs eingefügt. Vergleichen Sie die von PyDesigner erzeugten Vorverarbeitungsdateien mit dem vorverarbeiteten Beispielordner für einen Datensatz, der sich im von OpenNeuro heruntergeladenen Ordner "derivatives" befindet.
- Konsultieren Sie den Abschnitt "Repräsentative Ergebnisse", um ihre Metrikzuordnungen mit denen in den Beispiel-Dataset-Ergebnissen zu vergleichen.
Ergebnisse
Die PyDesigner-Software wendet mehrere Bildkorrekturschritte auf Rohdiffusionsdaten an und generiert Ausgaben, die zur Verbesserung der Genauigkeit der Rohdateien bei der Durchführung von Analysen verwendet werden. Jeder in der Pipeline verfügbare Schritt wurde zuvor durch Peer-Review-Publikationenvalidiert 5,6,7,8,9,10,11, wie in der Einleitung erläutert. Die Ausgaben der Software können in Analysen wie Traktographieprofilen, Konnektivitätsmatrizen, voxelweisen Analysen, ROI-Analysen, fODF-Analysen, TBSS und Fixel-basierten Analysen verwendet werden.
Die Software-Website23 listet alle Ausgabedateien auf, die während der Vorverarbeitungspipeline erzeugt werden. Nachdem jede Eingabeaufforderung ausgeführt wurde, gibt die Konsole eine Beschreibung aller abgeschlossenen Prozesse aus. Es gibt 3 Arten von Ausgabedateien: Verarbeitungsdateien, Metriken und Qualitätskontrolle. Die Struktur des Ausgabeverzeichnisses ist in Abbildung 1 dargestellt. Diese Dateien sind verfügbar, wenn die Standardvorverarbeitung verwendet wird (siehe Abschnitt 7 des Protokolls). Wenn der Benutzer eine fortgeschrittenere Nutzung wünscht (siehe Abschnitt 8 des Protokolls), hängen die verfügbaren Ausgabedateien davon ab, welche Prozesse abgeschlossen wurden.
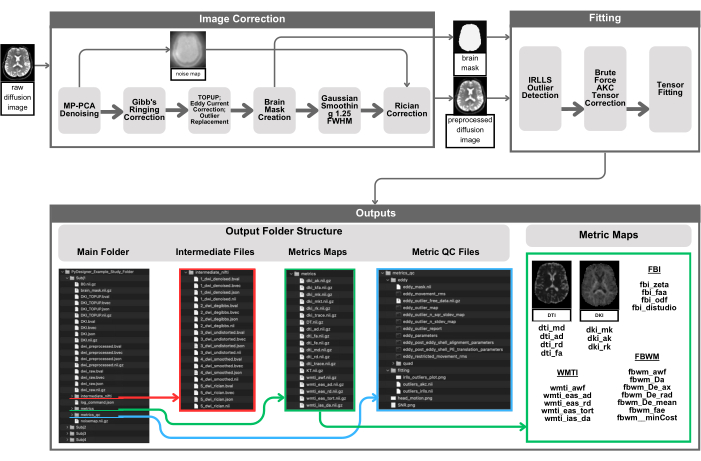
Abbildung 1: Visuelle Darstellung der PyDesigner-Pipeline. Die Vorverarbeitung beginnt mit der Bereitstellung einer 4D-DWI-Eingabe für PyDesigner (oben links), die dann einer MP-PCA-Rauschunterdrückung unterzogen wird, um eine entrauschte 4D-DWI und eine 3D-Rauschkarte zu erhalten. Das entrauschte 4D-DWI wird dann einer Gibbs-Ringing-Korrektur, einer Rician-Bias-Korrektur, TOPUP, einer Wirbelstromkorrektur und einer Ausreißerkorrektur unterzogen. Eine Gehirnmaske wird dann für nachfolgende Schritte, die Ausreißererkennung und die Tensoranpassung berechnet, um die Berechnungen zu beschleunigen, indem sie nur innerhalb der Gehirnmaske durchgeführt werden. Die Ausgaben befinden sich in Unterverzeichnissen im Hauptordner für die Betreffverarbeitung - intermediate_nifti, Metriken und metrics_qc. Beachten Sie, dass die Screenshots der PyDesigner-Ausgaben nicht als vollständige Liste aller möglichen Ausgaben gedacht sind, sondern vielmehr als schematisches visuelles Beispiel dessen dienen, was Benutzer erwarten können. Die Ausgaben, sowohl die endgültigen Ausgaben als auch die Zwischendateien, variieren je nach den Benutzereingabedaten und den verwendeten Verarbeitungsflags. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Verarbeitung
Verarbeitungsdateien werden während der PyDesigner-Pipeline verwendet und im Stammausgabeverzeichnis gespeichert. Für jeden Vorverarbeitungsschritt werden DWI-Zwischendateien im Ausgabeordner "intermediate_nifti" gespeichert, wie in Abbildung 1 dargestellt. Auf diese Dateien sollte verwiesen werden, wenn Probleme mit der Verarbeitung oder den Ausgaben behoben werden, um jeden Schritt der Pipeline separat auszuwerten.
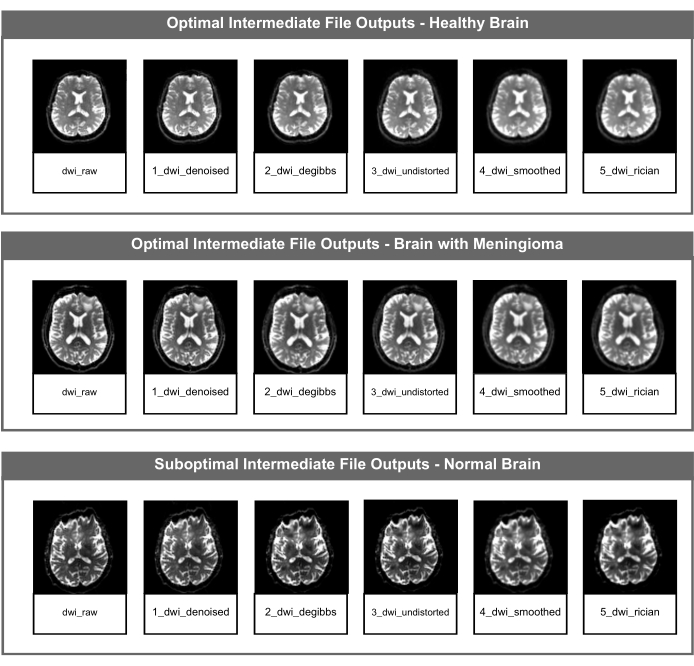
Abbildung 2: Optimale und suboptimale DWI-NifTI-Zwischendateien. Die Abbildung zeigt die NIfTI-Zwischendatei für jeden Bildkorrekturschritt der PyDesigner-Pipeline. Die obere Zeile ist ein Beispiel für optimale Zwischendateiausgaben mit Daten aus einem gesunden erwachsenen Gehirn, die mittlere Zeile ist ein Beispiel für optimale Zwischendateiausgaben mit Daten aus einem pathologischen Gehirn (Meningeom), und die untere Zeile zeigt suboptimale Zwischendateiausgaben mit Daten aus einem gesunden erwachsenen Gehirn mit einem Suszeptibilitätsartefakt, das nichts mit der Gehirnstruktur oder -gesundheit zu tun hat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Metrik
Dieser Ordner enthält alle parametrischen Karten, die PyDesigner berechnet (siehe Abbildung 1). Dazu gehören parametrische Karten für DTI/DKI, Fiber Ball Imaging (FBI)/Fiber Ball White Substance (FBWM) und White Matter Tract Integrity (WMTI) Metriken (Tabelle 1)16.

Tabelle 1: Erwarteter Wertebereich für DTI/DKI- und FBI/FBWM-Metriken. Die Tabelle enthält eine Liste der robusten DTI-, DKI-, FBI- und FBWM-Metriken, die von PyDesigner generiert wurden, und deren erwartete Wertebereiche. Die erforderlichen Sequenzen und b-Werte (s/mm2) zur Ableitung jeder Metrik sind ebenfalls aufgeführt. Die aufgeführten DTI-Metriken sind FA, MD, AD und RD. Die aufgeführten DKI-Metriken sind MK, AK, RK und KFA. Die aufgeführte FBI-Metrik ist FAA. Die aufgeführten FBWM-Metriken sind AWF, DA, DE_AX, DE_RAD und FAE.
Benutzer können eine visuelle und wertmäßige Qualitätskontrolle (QC) der Metriken für die mittlere Diffusivität (MD), die fraktionale Anisotropie (FA) und die mittlere Kurtosis (MK) durchführen, um suboptimale Ergebnisse zu identifizieren. Wenn diese Metriken nach den unten beschriebenen Standards suboptimal sind, sollten Benutzer sich jede in Abbildung 2 beschriebene Zwischendatei ansehen, um festzustellen, welcher Vorverarbeitungsschritt nicht erfolgreich war.
Die visuelle Qualitätskontrolle wird verwendet, um suboptimale Ergebnisse zu identifizieren (z. B. Probleme mit der Tensoranpassung und Artefakte). Wir empfehlen die Verwendung von ImageJ für die visuelle Qualitätskontrolle, um sicherzustellen, dass das Image nicht über Software-Standardeinstellungen manipuliert wird. Die obere Reihe von Abbildung 3 zeigt typische metrische MD-, FA- und MK-Karten mit einem biologisch plausiblen Schwellenwert von 0-3 μm2/ms, 0-1 μm2/ms bzw. 0-2 μm2/ms (Abbildung 3 [Obere Reihe]). Die MD-Karte sollte die höchsten Werte in den Ventrikeln und hohe Werte in der kortikalen grauen Substanz aufweisen (Abbildung 3A [obere Reihe]). Die FA-Karte sollte sichtbare Bahnen der weißen Substanz enthalten, die im gesamten Gehirn klar sind (Abbildung 3B [obere Reihe]). Die MK-Karte sollte hohe Werte in WM und niedrigere Werte in der grauen Substanz und im Liquor aufweisen (Abbildung 3C [obere Reihe]). Abbildung 3D [Obere Reihe] ist ein Beispiel für eine Metrikkarte mit Problemen bei der Tensoranpassung, die zu Clustern von Voxeln mit null Werten führt. Wenn Probleme auftreten, überprüfen Sie die log_command.json Datei, um nach Vorverarbeitungsfehlern zu suchen. Überprüfen Sie die Zwischendateien, um den spezifischen Fehler zu ermitteln. Wenn Sie Hilfe bei der Problembehandlung benötigen, senden Sie eine Anfrage auf der GitHub-Seite von PyDesigner.
Die Qualitätskontrolle des Wertes wird verwendet, um zu ermitteln, ob die Voxel einer bestimmten Metrikkarte zwischen den Subjekten für jeden Datensatz relativ einheitlich sind. Der erwartete Wertebereich für jede Karte und jedes Dataset hängt von den Daten und den PyDesigner-Parametern ab. In unserem Testdataset hatten wir konsistente Spitzenwerte in den Bereichen 8000 bis 10.000, 2500 bis 4000 bzw. 5000 bis 13.000 für MD, FA und MK unter Verwendung des Standard-Histogramm-Binning von FSLeyes. Die untere Reihe von Abbildung 3 zeigt Beispiele für die Variabilität des Histogramms. Tabelle 1 enthält die erwarteten X-Achsen-Werte für diese Metriken. Höhere oder niedrigere Voxel sind charakteristisch für den Datensatz oder weisen auf ein Artefakt oder ein Vorverarbeitungsproblem hin (Abbildung 3D [untere Reihe]).
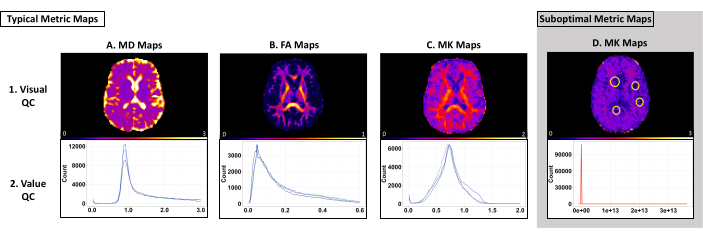
Abbildung 3: Beispiel-Metrikkarten und Histogramme aus PyDesigner mit optimalen und suboptimalen Ergebnissen. In der oberen Zeile werden Beispiele für MD-, FA- und MK-Maps mit einem Motiv angezeigt, die für die visuelle Qualitätskontrolle verwendet werden. In der unteren Zeile werden Histogramme mit mehreren Themen angezeigt, die für die Qualitätskontrolle verwendet werden. (A-C) Typische Beispiele für metrische Zuordnungen und Histogramme, die die visuelle Qualitätskontrolle und die Wertqualitätskontrolle bestehen. Jede blaue Linie in den Histogrammen für jeden Metriktyp stellt einen einzelnen Datensatz dar. Beachten Sie, dass jedes Dataset einer ähnlichen Kurve folgt und in einen ähnlichen Bereich fällt. (D) Ein Beispiel für eine Metrikzuordnung, die die visuelle Qualitätskontrolle oder die Qualitätskontrolle nicht besteht. Beachten Sie, dass die rote Linie im Histogramm eine Kurve zeigt, die sich von der von A-C unterscheidet. Die Nullvoxel, die auf dieser metrischen Karte eingekreist sind, sind auf Probleme bei der Tensoranpassung während der Vorverarbeitung zurückzuführen (Feld D, obere Reihe). Dieses Histogramm ist ein Beispiel dafür, dass in einem Datensatz im Allgemeinen höhere oder niedrigere Voxel als erwartet auftreten (Feld D, untere Reihe). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Qualitätskontrolle
Nachdem PyDesigner die Daten verarbeitet hat, sollte der Ordner metrics_qc (siehe Abbildung 1) verwendet werden, um suboptimale Datensätze zu identifizieren. Für jeden Datensatz gibt PyDesigner drei Diagramme aus, die für die Qualitätskontrolle verwendet werden.
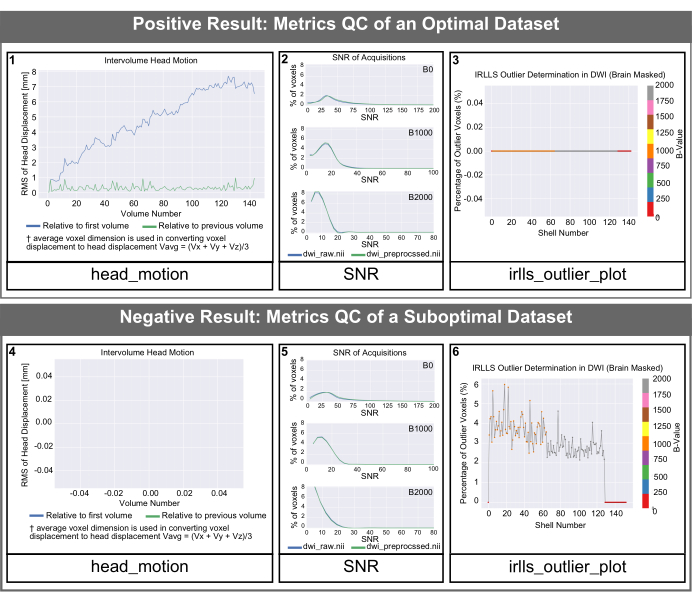
Abbildung 4: Vom PyDesigner generierte QC-Histogramme für optimale und suboptimale Datensätze. SNR-, Intervolume-Kopfbewegungs- und Ausreißer-Histogramme, die von PyDesigner generiert werden. Beide Zeilen stellen Daten aus einem gesunden erwachsenen Gehirn dar. Die oberste Zeile ist ein Beispiel für Histogramme der Qualitätskontrolle für ein optimales Dataset. Die untere Zeile zeigt die Ergebnisse der Qualitätskontrolle eines suboptimalen Datensatzes mit einem Suszeptibilitätsartefakt, das nichts mit der Gehirnstruktur oder der Gesundheit zu tun hat. Bitte beachten Sie, dass die Textgröße der Beschriftungen in den Standardausgaben von PyDesigner kleiner ist, als sie in dieser Abbildung angezeigt werden. Wir haben die Textgröße in dieser Abbildung zur besseren Lesbarkeit erhöht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die head_motion Diagramme in Abbildung 4 zeigen die Kopfverschiebung relativ zum ersten Volumen und zum vorherigen Volumen. Wie in Abbildung 4 (Bild 1) zu sehen ist, ist die Kopfverschiebung in der Regel gering, und PyDesigner passt diese Bewegungsartefakte in der Standardverarbeitungspipeline mit dem FSL-Programm Eddy zusammen mit TOPUP für die Bewegungs- und Wirbelstrombewegungskorrektur9 an. Bei suboptimalen Datensätzen kann das head_motion Diagramm leer erscheinen, wie in Abbildung 4 (Feld 4) dargestellt. Dies deutet darauf hin, dass die Wirbelstrom-Bewegungskorrektur nicht erfolgreich war und der PyDesigner daher kein Diagramm ausgeben konnte. Die Protokolldateien für die aktuelle Korrektur von Wirbeln befinden sich im Unterordner eddy innerhalb des Ordners metrics_qc (siehe Abbildung 1). Das Diagramm des Signal-Rausch-Verhältnisses (SNR) zeigt 3 Diagramme an. Jedes Diagramm bezieht sich auf einen anderen b-Wert und zeigt sowohl die vorverarbeiteten als auch die Rohdaten. Für einen optimalen Datensatz sollte der SNR-Peak der Rohdaten ≥5 betragen (Abbildung 4 [Panel 2]). Suboptimale Datensätze haben einen SNR-Peak der Rohdaten von ≤3 (Abbildung 4 [Feld 5]). Im Idealfall sollten Benutzer sehen, dass die SNR-Spitze für alle b-Werte leicht, aber nicht dramatisch ansteigt. Das Ausreißerdiagramm befindet sich im Anpassungsordner innerhalb von metrics_qc und zeigt den Prozentsatz der Ausreißer im Datensatz (Abbildung 4 [Panel 3 und 6]). Ein guter Datensatz sollte einen geringen Prozentsatz an Ausreißern aufweisen, in der Regel weniger als 5 % (Abbildung 4 [Feld 3]). Ein suboptimaler Datensatz weist einen hohen Prozentsatz an Ausreißern auf, wie in Abbildung 4 (Feld 6) dargestellt.
Beispiele für Datensatzergebnisse
Sobald PyDesigner die Verarbeitung des Beispieldatensatzes abgeschlossen hat, sollten alle Ausgaben im Ordner "PyDesigner_Outputs" enthalten sein. Diese Ausgaben können mit denen verglichen werden, die im Ordner "derivatives" zu finden sind, der mit dem von OpenNeuro heruntergeladenen Beispieldatensatz (verarbeitet unter MacOS 12.4) verpackt ist. Wenn die Software korrekt läuft, ist die Dateistruktur von "PyDesigner_Outputs" und "derivatives" genau gleich. Ebenso sollten die SNR-, Kopfbewegungs- und Ausreißerdiagramme im Unterordner "metrics_qc" mit denen in Abbildung 5A übereinstimmen. Metrische Karten (zu finden im Unterordner "metrics") können mit Bildgebungssoftware wie FSLeyes, MRIcron, ImageJ usw. verglichen werden. Histogramme von FA-, MD- und MK-Werten aus vorverarbeiteten Metriken/Metriken sind in Abbildung 5B zu sehen. Beachten Sie, dass alle in Abbildung 5B gezeigten Metrik-Histogramme basierend auf den vorgeschlagenen Metrikwertskalen in Tabelle 1 skaliert werden.
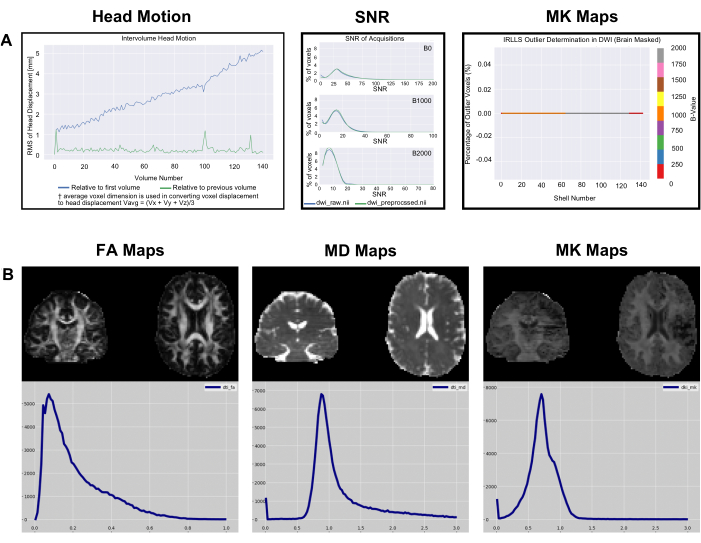
Abbildung 5: Beispiel-Datenmetrik und metrische QC-Histogramme. (A) SNR-, Intervolume-Kopfbewegungs- und Ausreißer-Histogramme, die von PyDesigner für die von OpenNeuro heruntergeladenen Beispieldaten generiert wurden. Beachten Sie, dass die Textgröße in den Diagrammen in dieser Abbildung zur besseren Lesbarkeit erhöht wurde. (B) Die Metrik für ein einzelnes Subjekt bildet Histogramme der Voxelwertzahlen für FA, MD und MK für denselben Datensatz ab, der mit FSLeyes v6.0 visualisiert wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
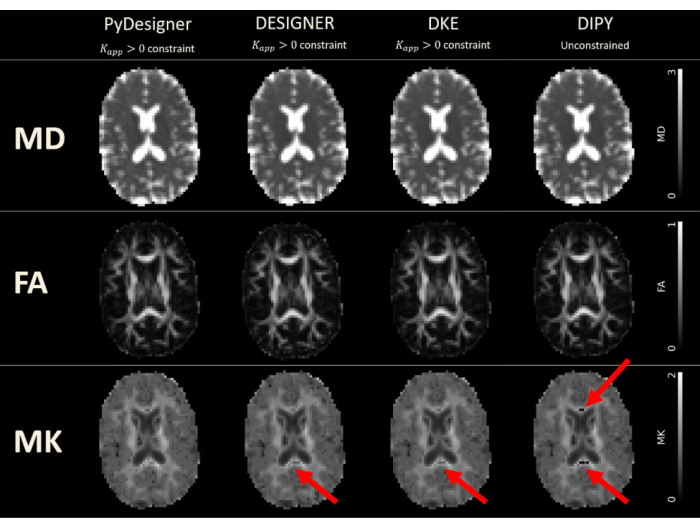
Abbildung 6: DTI- und DKI-Zuordnungen, die von PyDesigner, DESIGNER, DKE und DIPY abgeleitet wurden. Die Tensoranpassung wurde mit der Einschränkung Kapp > 0 in PyDesigner, DESIGNER5 und Diffusional Kurtosis Estimator (DKE)27 durchgeführt, während die uneingeschränkte Anpassung aufgrund von Softwareeinschränkungen in Diffusion Imaging in Python (DIPY)28 verwendet wurde. Die Einheiten für MD sind quadrierte Mikrometer pro Millisekunde (μm2/ms), während die anderen Metriken dimensionslos sind. Aussetzer aufgrund eines generischen Problems bei der Gibbs-Ringing-Korrektur sind in MK-Maps zu sehen, die von DESIGNER, DKE und DIPY erstellt wurden. Diese Abbildung wurde mit Genehmigung von Dhiman et al.29 reproduziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
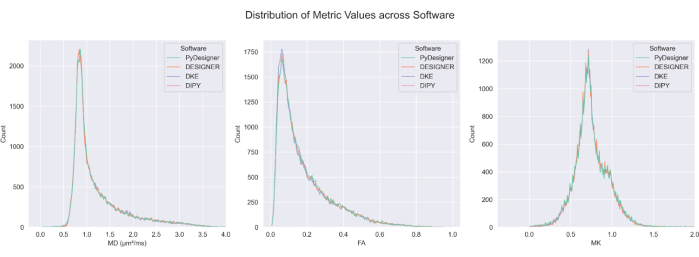
Abbildung 7: Vergleich von FA, MD und MK über Pipelines hinweg. Die Verteilung der berechneten Werte für FA, MD und MK aus PyDesigner, DESIGNER5, DKE27 und DIPY28 in CSF-ausgeschlossenen Gehirnen ist bei den meisten Voxeln ähnlich. Die Berechnung der Metrikzuordnung über alle Methoden hinweg ist vergleichbar. Diese Abbildung wurde mit Genehmigung von Dhiman et al.29 reproduziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Hauptmotivation für die Entwicklung von PyDesigner bestand darin, die Schlüsselelemente von DESIGNER zu implementieren und gleichzeitig den gesamten MATLAB-Code durch Python zu ersetzen, um so eine größere Portabilität und Zugänglichkeit zu ermöglichen. PyDesigner und DESIGNER Dv1 liefern nahezu identische Ausgaben29. Nichtsdestotrotz gibt es einige zusätzliche Optionen, Standardeinstellungen und kleinere Fehlerbehebungen, die in PyDesigner enthalten sind. Die Online-Dokumentation zum PyDesigner16 beschreibt diese im Detail.
PyDesigner liefert auch ähnliche Ergebnisse wie der häufig verwendete Diffusional Kurtosis Estimator (DKE)27 und Diffusion Imaging in Python (DIPY)28 DKI-Analysetools29 (siehe Abbildung 6 und Abbildung 7), aber die Karten, die auf der Grundlage von DESIGNER-vorverarbeiteten dMRT erstellt wurden, gelten aufgrund ihres Anpassungsalgorithmus als genauer, wie Ades-Aron et al.5 gezeigt haben. In Abbildung 6 finden Sie einen Vergleich der Metrikzuordnungen MD, FA und MK aus PyDesigner, DESIGNER5, DKE27 und DIPY28. Abbildung 7 zeigt den Vergleich der MD-, FA- und MK-Histogramme aus den einzelnen Rohrleitungen. Die Kombination von eingeschränkter Tensoranpassung, Ausreißererkennung und scheinbarer Kurtosiskoeffizientenkorrektur führt zu einer robusteren und genaueren Tensoranpassung, wie sie in PyDesigner und DESIGNER5 zu sehen ist.
Ein Vorteil von PyDesigner gegenüber dem DESIGNER Dv1 ist, dass er über den NeuroDock Docker Container30 verfügbar ist, was die Portabilität erheblich verbessert und die Installation vereinfacht. Dieser Container wird auf allen wichtigen Betriebssystemplattformen ausgeführt, die mit Docker kompatibel sind, einschließlich Windows, Mac OS und verschiedenen Linux-Distributionen. Basierend auf dem Feedback der Benutzer wird PyDesigner v2.0 Updates für den PyDesigner Docker Container enthalten. Die Verbesserungen, die durch die neue Version von PyDesigner und die Einführung eines mehrstufigen Dockerfiles eingeführt wurden, werden alle bestehenden Probleme lösen, mit denen Benutzer konfrontiert sind. Wenn Benutzer Probleme mit der Installation haben, empfehlen wir, Fragen an die PyDesigner-Diskussionsseite16 zu senden. Die Container-Technologie von Docker ermöglicht auch eine unkomplizierte Bereitstellung auf Hochleistungsclustern (HPCs) für die schnelle Batch-Verarbeitung von DWIs auf Docker-kompatiblen lokalen Clustern.
PyDesigner enthält auch mikrostrukturelle Modellierungsberechnungen, die über DKI hinausgehen, einschließlich WMTI3, FBI und FBWM. Für WMTI ist ein Standard-DKI-Datensatz ausreichend, und die zugehörigen mikrostrukturellen Parameter werden standardmäßig berechnet. Es sollte jedoch betont werden, dass die Validität von WMTI auf Regionen der weißen Substanz mit hoher FA (d.h. FA ≥ 0,4) beschränkt ist. Einige WMTI-Metriken haben eine begrenzte Genauigkeit, da von einer parallelen Ausrichtung der Axone in einem gegebenen Voxel31 ausgegangen wird. FBI 32,33,34 ist eine eigenständige dMRT-Methode, die in der gesamten weißen Substanz des Gehirns anwendbar ist und einen hohen b-Wert (d. h. b ≥ 4000 s/mm 2) und dMRT-Daten erfordert, die mit mindestens 64 Diffusionskodierungsrichtungen abgetastet wurden (zusammen mit Daten für b = 0). Die Hauptausgaben von FBI sind die Faserorientierungsdichtefunktion (fODF) für jedes Voxel der weißen Substanz, die für die Traktographie der weißen Substanz verwendet werden kann und als Eingabe für FBWM dient, sowie die intraaxonale fraktionelle Anisotropie (FAA). FBWM verwendet die dMRT-Daten von DKI und FBI, um die gleichen Parameter wie WMTI zu schätzen, jedoch mit verbesserter Genauigkeit, und es kann unabhängig vom FA-Wert auf die gesamte weiße Substanz angewendet werden. Wenn also diese zusätzlichen Daten verfügbar sind, werden FBWM-Schätzungen gegenüber denen von WMTI35 bevorzugt. Wie beim FBI wurde FBWM nur bei erwachsenen zerebralen weißen Substanzen validiert.
Zusätzlich zu den von PyDesigner bereitgestellten Rotationsinvarianten (RAS-Ausrichtung) werden DSIstudio-spezifische .fib-Dateien (LPS-Ausrichtung) separat für DTI, DKI und FBI erstellt. Die .fib-Dateien enthalten ODF-Richtungsinformationen zum Generieren von Traktographieprofilen für jede Methode. Die resultierenden Traktographieprofile können verwendet werden, um Konnektivitätsmatrizen auf Subjekt- und Gruppenebene zu erstellen. Die DKI- und FBI-FIB-Dateien enthalten multidirektionale Informationen, die Crossing Fiber-Informationen weiterleiten, die bei der DTI-Traktographie nicht verfügbar sind. Darüber hinaus wurden in jeder .fib-Datei die verschiedenen Rotationsinvarianten jeder Methode eingeschlossen, die in verschiedenen Kombinationen als Kriterien für das Seeding, die Durchführung und das Stoppen der Traktographie verwendet werden können. Weitere Informationen zu DSIstudio finden Sie auf der Website36.
Ein weiteres bemerkenswertes Merkmal von PyDesigner ist die Eingabe mehrerer Dateien, die es ermöglicht, verschiedene Dateieingaben zu verarbeiten - NifTi (.nii), komprimiertes NifTi (.nii.gz), DICOM (.dcm) und MRtrix Dateiformat (.mif). PyDesigner kann Erfassungsinformationen aus Header-Metadaten unabhängig vom Eingabeformat automatisch identifizieren und entsprechende Korrekturen vornehmen, wodurch ein Hands-Off-Ansatz unterstützt wird. Unabhängig von Unterschieden in den Protokollen kann derselbe Befehl (siehe oben) verwendet werden, um eine Vielzahl von DWIs zu verarbeiten. Diese Software spart somit Zeit und Aufwand, indem sie manuelle Vorverarbeitungsschritte und Befehle minimiert. In einem kürzlich veröffentlichten Update (v1.0-RC10) wurde dies durch die Einführung der Kompatibilität für mehrere Echo-Time-Datensätze (Multi-TE) verbessert. Auf diese Weise kann PyDesigner Bildvorverarbeitungsschritte, die weitgehend unabhängig von TE sind, auf einem Multi-TE-DWI ausführen, um ein Bild mit minimalem Rauschen und Artefakten zu erhalten. TE-abhängige Tensorberechnungen werden dann für jeden TE separat durchgeführt, um Diffusions- oder Kurtosismetriken zu erstellen.
Wir weisen darauf hin, dass PyDesigner v1.0 keinen Endpunkt beim Entwurf von Bildvorverarbeitungspipelines darstellt. Die Entwicklung und Validierung von Bildverarbeitungswerkzeugen ist ein aktives Forschungsgebiet. Insbesondere während der Vorbereitung dieses Manuskripts wurden neue Entwicklungen in der Bildrauschunterdrückung und der Entfernung von Gibbs-Ringing vorgeschlagen, die zur Veröffentlichung einer überarbeiteten Designer-Pipeline, Designer Dv237, führten, die eine verbesserte Rauschunterdrückung und Korrektur von Gibbs-Artefakten für dMRT enthält, die mit partiellen Fourier-Aufnahmen35 erfasst wurden, und auf dem DESIGNER Github38 verfügbar ist. Ebenso ist die organisatorische Compliance von BIDS39 ein wichtiger Aspekt jeder MRT-Analyse-Pipeline. Das BIDS-Format ermöglicht eine effizientere gemeinsame Nutzung von Daten und die Implementierung von Pipelines, indem eine spezifische Set-Struktur für alle MRT-Datentypen festgelegt wird. Obwohl PyDesigner derzeit nicht BIDS-konform ist, wird eine kommende Version von PyDesigner BIDS-konforme Ausgaben enthalten. Darüber hinaus wurde PyDesigner ursprünglich mit Python Version 3.7 geschrieben. Zum Zeitpunkt dieser Veröffentlichung gilt Python 3.7 nun als veraltet. Die in diesem Manuskript enthaltenen Informationen sind jedoch unabhängig von der Python-Version. Das heißt, die hier beschriebenen Verfahren, die wichtige Vorverarbeitungsfunktionen darstellen (z. B. Rauschunterdrückung, Gibbs-Ringing-Korrektur usw.), folgen dem gleichen betrieblichen und konzeptionellen Arbeitsablauf, unabhängig von Änderungen in den Installationsverfahren. Wichtig ist, dass zukünftige PyDesigner-Versionen, wie bei jeder Python-basierten Software, auf eine neue Version von Python aktualisiert werden. Wir sind bestrebt, solche Entwicklungen im Laufe der Zeit weiter zu validieren und zu integrieren.
Für die aktuellste Version von PyDesigner, einschließlich aller neuen Dokumentationen, die für Python-Versionsupdates relevant sind, werden die Leser ermutigt, die Website zu konsultieren, bevor sie mit einer neuen Analyse beginnen, und sich am Diskussionsforum zu beteiligen, in dem Fragen zu PyDesigner eingereicht werden können16. Die Docker-Implementierung für die Portabilität heißt NeuroDock30 und enthält PyDesigner und seine Abhängigkeiten, um die Verarbeitung auf einer Vielzahl von Plattformen zu ermöglichen.
Offenlegungen
Nichts.
Danksagungen
Wir bedanken uns für die hilfreichen Gespräche mit Olivia Horn, Daniel Lench und Graham Warner.
Die in dieser Veröffentlichung berichtete Forschung wurde teilweise durch Zuschüsse der National Institutes of Health R01AG054159, R01AG057602, R01AG055132, R01DC014021, R01NS110347, R21DA050085, F31NS108623, P20GM109040, P50DC000422, T32GM008716 und T32DC014435 unterstützt. Der Inhalt liegt ausschließlich in der Verantwortung der Autoren und gibt nicht unbedingt die offizielle Meinung der National Institutes of Health wieder. Zusätzliche Mittel wurden von der Litwin Stiftung zur Verfügung gestellt.
Materialien
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
Referenzen
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten