Einzelmolekül-Echtzeit-Visualisierung der DNA-Abwicklung durch CMG-Helikase
In diesem Artikel
Zusammenfassung
Dieses Protokoll demonstriert die Durchführung eines Einzelmolekül-Assays zur Live-Visualisierung der DNA-Abwicklung durch CMG-Helikase. Sie beschreibt (1) die Herstellung eines DNA-Substrats, (2) die Reinigung der fluoreszenzmarkierten Drosophila melanogaster CMG-Helikase, (3) die Vorbereitung einer mikrofluidischen Durchflusszelle für die Totalreflexionsfluoreszenzmikroskopie (TIRF) und (4) den Einzelmolekül-DNA-Entwindungsassay.
Zusammenfassung
Eine originalgetreue Genomduplikation ist unerlässlich, um die genetische Stabilität der sich teilenden Zellen zu erhalten. Die DNA-Replikation wird während der S-Phase durch einen dynamischen Komplex von Proteinen durchgeführt, der als Replisom bezeichnet wird. Das Herzstück des Replisoms ist die Helikase CDC45-MCM2-7-GINS (CMG), die die beiden Stränge der DNA-Doppelhelix so trennt, dass DNA-Polymerasen jeden Strang kopieren können. Bei der Genomduplikation müssen Replisomen eine Vielzahl von Hindernissen und Herausforderungen überwinden. Jede dieser Faktoren gefährdet die Stabilität des Genoms, da das Versäumnis, die DNA vollständig und genau zu replizieren, zu Mutationen, Krankheiten oder Zelltod führen kann. Daher ist es von großem Interesse zu verstehen, wie CMG im Replisom sowohl während der normalen Replikation als auch unter Replikationsstress funktioniert. Hier beschreiben wir einen Totalreflexionsfluoreszenzmikroskopie-Assay (TIRF) unter Verwendung rekombinanter gereinigter Proteine, der eine Echtzeit-Visualisierung von oberflächengebundenen, gestreckten DNA-Molekülen durch einzelne CMG-Komplexe ermöglicht. Dieser Assay bietet eine leistungsstarke Plattform zur Untersuchung des CMG-Verhaltens auf Einzelmolekülebene, so dass die Dynamik der Helikase direkt beobachtet werden kann und die Reaktionsbedingungen in Echtzeit gesteuert werden können.
Einleitung
Die DNA-Replikation ist streng reguliert, da eine Zelle ihr Genom genau duplizieren muss, um Mutationen, Krankheiten oder Tod zu verhindern. Die Replikation der eukaryotischen DNA erfolgt durch den Replisomenkomplex, der die elterliche DNA abwickelt und einzelsträngige DNA (ssDNA) als Vorlage für die Synthese neuer DNA verwendet. In der G1-Phase werden katalytisch inaktive Doppelhexamere von MCM2-7 an den Replikationsursprüngen1 auf doppelsträngige DNA (dsDNA) geladen. In der S-Phase werden MCM2-7-Komplexe durch die Bindung von CDC45 und GINS2 aktiviert, um CMG-Komplexe mit 11 Untereinheiten (CDC45, MCM2-7, GINS) zu bilden. Jedes CMG initiiert die DNA-Entfaltung in entgegengesetzte Richtungen und bildet die Kerneinheit, um die sich das Replisom herum anordnet3.
Vor zwei Jahrzehnten wurde die CMG-Helikase erstmals als ein Komplex aus 11 Untereinheiten identifiziert, der für die DNA-Replikation unerlässlich ist4. Seitdem hat sich unser Verständnis von CMG erheblich weiterentwickelt, von der Beladung und Aktivierung 5,6 bis hin zur DNA-Entwindung und -Beendigung7. Traditionelle biochemische und strukturbiologische Techniken waren für viele dieser Entdeckungen von entscheidender Bedeutung. Diese Methoden waren jedoch oft nur begrenzt in ihrer Fähigkeit, die dynamischeren Aspekte der CMG zu untersuchen. Einzelmolekülmethoden nutzen die physikalische Manipulation einzelner Biomoleküle, um ihre Aktivität Molekül für Molekül zu messen oder zu visualisieren. Dies kann genutzt werden, um Einblicke in die Echtzeitdynamik von Proteinen zu erhalten, die mit anderen Techniken oft übersehen oder nicht nachweisbar sind 8,9.
Hier beschreiben wir einen Totalreflexionsfluoreszenz-Mikroskopie-Assay (TIRF), um die DNA-Abwicklung durch CMG-Helikase in Echtzeit zu visualisieren. Gereinigtes, fluoreszenzmarkiertes CMG wird auf das freie 3'-Ende der langen DNA geladen, die eine vorgefertigte DNA-Gabelstruktur enthält. Lineare DNA wird auf einem Biotin-PEG-Deckglas in einer mikrofluidischen Durchflusszelle gedehnt, indem jedes Ende der DNA nacheinander an die Oberfläche gebunden wird. Dieser Ansatz ermöglicht ein einheitlicheres DNA-Tethering, wodurch die Variation, die bei der Datenanalyse berücksichtigt werden muss, erheblich reduziert wird. In Gegenwart von ATP-γ-s wird CMG auf die einzelsträngige DNA am 3'-Ende der Gabel geladen. ATP-γ-s ist ein langsam hydrolysierbares ATP-Analogon, das die Bindung von CMG an die DNA ermöglicht, sich aber nicht abwickelt. Die anschließende Zugabe von ATP zusammen mit gereinigtem, fluoreszenzmarkiertem RPA aktiviert CMG und leitet eine umfangreiche DNA-Entwindung ein. Visuell transloziert CMG entlang der DNA und hinterlässt einen wachsenden Trakt RPA-gebundener ssDNA. Das ungebundene DNA-Ende bewegt sich mit CMG und bildet aufgrund der durch RPA-Bindung verursachten Verdichtung einen "engen Ball". Das Design der Durchflusszelle ermöglicht es, den Puffer während des Abwickelns jederzeit auszutauschen, was eine hervorragende Kontrolle während und über jedes Experiment bietet.
Dieses Protokoll ist in vier Methoden unterteilt, die unabhängig voneinander durchgeführt werden können. Abschnitt 1 beschreibt die Herstellung eines 20 kb linear gegabelten DNA-Substrats für Einzelmolekül-Assays. Abschnitt 2 beschreibt die Aufreinigung und Fluoreszenzmarkierung von Drosophila melanogaster CMG (DmCMG). Wichtige Informationen über die Ausprägung von DmCMG finden Sie im Abschnitt "Hinweise". Abschnitt 3 befasst sich mit der Vorbereitung einer mikrofluidischen Durchflusszelle, die an einem TIRF-Mikroskop verwendet werden kann. Abschnitt 4 beschreibt, wie der Einzelmolekül-DNA-Entwindungsassay durchzuführen ist.
Protokoll
1. Präparation von 20 kb linear gegabelter DNA, die in Einzelmolekül-Assays verwendet wird (Abbildung 1)
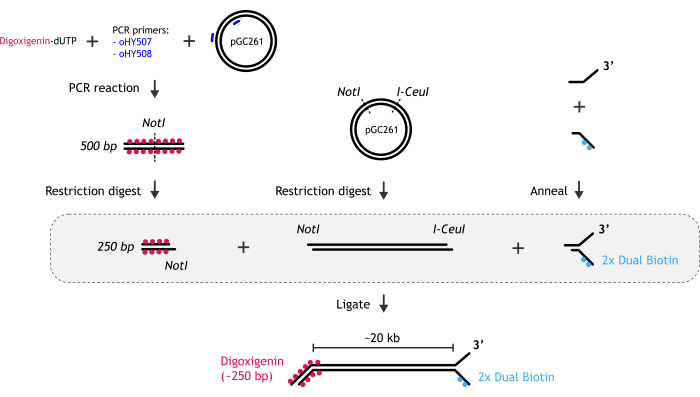
Abbildung 1: Grafische Darstellung der DNA-Substratvorbereitung. (A) Das biotinylierte DNA-Gabelende wird durch Annealing von zwei teilweise komplementären Oligonukleotiden hergestellt: biotinyliertem und nicht-biotinyliertem. (B) Das Haupt-dsDNA-Fragment (~20 kb) wird durch Restriktionsverdau des pGC261-Plasmids mit zwei Enzymen erzeugt, um eine lineare DNA mit unterschiedlichen Überhängen an jedem Ende zu erzeugen. (C) Das Digoxigenin-Duplex-DNA-Ende wird durch eine PCR-Reaktion erhalten, die in Gegenwart von Digoxigenin-dUTP durchgeführt wird, gefolgt von einem Restriktionsverdau. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Erzeugung von biotinyliertem DNA-Gabelende.
- Glühen Sie die DNA der Gabel.
- Mischen Sie zwei Oligonukleotide (oHY502 und oHYbio85) in 1x STE-Puffer (100 mM NaCl, 10 mM Tris, pH 8,0; 1 mM Ethylendiamintetraessigsäure [EDTA]). Die endgültige Oligonukleotidkonzentration beträgt 10 μM und das endgültige Reaktionsvolumen 100 μl.
- Die Reaktion wird bei 80 °C 5 Minuten lang auf einem Heizblock inkubiert. Schalten Sie den Heizblock aus und lassen Sie die Reaktion allmählich abkühlen, bis die Temperatur unter 30 °C fällt.
- Validieren Sie die Glüheffizienz an einem Tris-Borat-EDTA (TBE)-Polyacrylamid-Gelelektrophorese (PAGE).
- Bereiten Sie 8% Acrylamid - FSME-Gel vor (für 4 Gele: 32,8 mL H2O; 4,8 mL 10x TBE-Puffer (1 M Tris, 1 M Borsäure, 20 mM EDTA), 9,6 mL 40% Acrylamid/Bis-Lösung, 37,5:1; 0,8 mL 10% Ammoniumpersulfat [APS], 40 μL Tetramethylethylendiamin [TEMED]).
ACHTUNG: FSME, Acrylamid, APS, TEMED sind giftig/gesundheitsschädlich. - Ein kleines Volumen (1 μl) geglühter Oligonukleotide wird zusammen mit den einzelnen Oligonukleotiden, verdünnt mit 6x Ladefarbstoff, auf das 8%ige Acrylamid - FSME-Gel bei 80 V geführt, 1 h lang bei RT.
- Das Gel in 1x FSME, ergänzt mit einer geeigneten Nukleinsäurefärbung, für 30 min auf einem Wippschüttler einweichen, dann das Gel mit einem Gel-Imaging-System scannen.
- Bereiten Sie 8% Acrylamid - FSME-Gel vor (für 4 Gele: 32,8 mL H2O; 4,8 mL 10x TBE-Puffer (1 M Tris, 1 M Borsäure, 20 mM EDTA), 9,6 mL 40% Acrylamid/Bis-Lösung, 37,5:1; 0,8 mL 10% Ammoniumpersulfat [APS], 40 μL Tetramethylethylendiamin [TEMED]).
- Gelöste DNA auf TBE-PAGE-Gel für die Gelexzision.
- Wenn die Oligonukleotide ordnungsgemäß geglüht sind, lösen Sie den Rest der Probe in Gegenwart von BSA auf (~100 μl der geglühten Probe, 25 μl des 6x Ladefarbstoffs, 2 μl von 33 mg/ml BSA). Um das größere Volumen auf das Gel zu laden, kombinieren Sie mehrere Vertiefungen mit einem sterilen Skalpell.
- Lassen Sie das Gel in 1x FSME, ergänzt mit geeigneter Nukleinsäurefärbung, 30 min auf einem Wippschüttler ziehen.
- Füllen Sie das Gel in eine Glasschale und visualisieren Sie die DNA in einem dunklen Raum unter blauem Licht. Verwenden Sie bei der Verwendung von blauem Licht einen geeigneten Schutzschirm oder eine Schutzbrille.
- Schneiden Sie das gewünschte Band mit einem sterilen Skalpell aus. Überschüssiges Gel abschneiden.
- Reinigen Sie die geglühte Gabel durch Elektroelution.
- Schneiden Sie ein Stück Dialyseschlauch lang genug ab, um das Gelstück einzuführen, befeuchten Sie es mit 1x FSME und klemmen Sie ein Ende zu.
- BSA auf 0,3 mg/ml in 500 μl 1x FSME verdünnen und das gesamte Volumen in den Schlauch pipettieren.
- Schiebe das Gelstück mit dem Metallspatel hinein. Bewegen Sie das Gelstück an eine Seite des Schlauchs, um Pufferraum zu schaffen, in den die DNA während der Elektroelution wandern kann.
- Drücken Sie den überschüssigen Puffer heraus und klemmen Sie das zweite Ende des Schlauchs ab.
HINWEIS: Das verbleibende Volumen bestimmt das endgültige Volumen und die Konzentration. - Legen Sie den Dialyseschlauch in den Agarose-Elektrophorese-Tank in 1x FSME-Puffer und stellen Sie sicher, dass der Schlauch vollständig eingetaucht ist.
HINWEIS: Stellen Sie sicher, dass das Gelstück auf der Seite platziert wird, die der negativen Elektrode am nächsten liegt, damit die DNA aus dem Gel in den Dialysebeutel wandern kann. - Lassen Sie die Elektroelution bei 80 V für 1-2 h laufen.
- Dialysieren Sie die elektroeluierte DNA in 10 mM Tris-Puffer (pH 8).
- Nehmen Sie nach der Elektroelution den Dialysebeutel aus dem Tank und trocknen Sie ein Ende mit Taschentuch ab. Vermeiden Sie es, den mittleren Teil des Schlauchs mit dem Gewebe zu berühren, wo sich das Gel und die Probe befinden.
- Entfernen Sie den Clip vom getrockneten Ende und pipettieren Sie die Probe vorsichtig im Schlauch auf und ab, um die DNA in den Puffer zu mischen.
- Tauchen Sie einen sauberen Spatel in 1x FSME-Puffer und schöpfen Sie das Gelstück aus dem Schlauch.
HINWEIS: Entfernen Sie beim Entfernen des Gelstücks so wenig Puffer wie möglich. - Klemmen Sie das offene Ende des Dialysebeutels wieder zu und entfernen Sie so jegliche Luft aus dem Inneren.
- Legen Sie den Dialysebeutel in ein 2-l-Becherglas, das mit 1,5 l 10 mM Tris-Puffer (pH 8), 20 mM NaCl und 2 mM MgCl2 gefüllt ist. Wickeln Sie die Clips mit Parafilm ein, um den Schlauch an den Rand des Becherglases zu kleben, während Sie den Schlauch vollständig in den Puffer eintauchen.
HINWEIS: Salz stabilisiert kurze Oligoduplexe. - Rühren Sie den Puffer mit einem Magnetstab vorsichtig um und dialysieren Sie die Probe mindestens 3-4 h lang bei Raumtemperatur (RT) oder über Nacht bei 4 °C.
- Trocknen Sie nach der Dialyse ein Ende des Dialysebeutels mit einem Taschentuch ab (um die DNA nicht zu verdünnen) und lösen Sie dieses Ende.
- Pipettieren Sie die Probe vorsichtig im Schlauch auf und ab und geben Sie sie in ein sauberes 1,5-ml-Röhrchen.
- Messen Sie die Konzentration der DNA mit einem Mikrovolumen-Spektrophotometer.
- Glühen Sie die DNA der Gabel.
- Generierung eines 20-KB-Fragments
- Durchführung des Restriktionsverdaus des pGC261-Plasmids18 unter Verwendung von NotI/I-CeuI-Enzymen.
- Mischen Sie vorsichtig 8 μl NotI-HF (20.000 U/ml), 8 μl I-CeuI (5.000 U/ml) und das pGC261-Plasmid (endgültige Plasmidkonzentration ~40 ng/μl in einer 200-μl-Reaktion) in einem Puffer, der von den Herstellern von Restriktionsenzymen empfohlen wird.
- Die Reaktion wird über Nacht bei 37 °C inkubiert.
- Lösen Sie verdaute DNA auf Agarosegel für die Gelexzision.
- OPTIONAL: Führen Sie zuerst eine kleine Menge der Reaktion auf einem 0,6%igen Agarosegel durch, um die Verdauungseffizienz zu testen, bevor Sie die gesamte Reaktion laden.
- Bereiten Sie 0,6% Agarosegel nach den Schritten 1.2.2.3-1.2.2.4 vor.
- Mischen Sie 0,48 g Agarosepulver mit 80 mL 1x FSME-Puffer. Erhitzen Sie die Lösung in der Mikrowelle, bis sie kocht. Schwenken, um sicherzustellen, dass die Agarose vollständig geschmolzen ist. Einige Minuten abkühlen lassen und dann in den dafür vorgesehenen Tank gießen.
- Um die Vertiefungen so zu kombinieren, dass sie das größere Volumen der Probe aufnehmen können, kleben Sie vorher ein Stück Klebeband über mehrere Vertiefungen des Kamms.
- Sobald das Gel fest geworden ist, den Kamm vorsichtig entfernen und den Tank mit 1x FSME-Puffer auffüllen. Mischen Sie die Probe vorsichtig mit dem DNA-Ladefarbstoff und lassen Sie das Gel 1 h lang bei 120 V laufen.
- Lassen Sie das Gel in 1x FSME, ergänzt mit einer geeigneten Nukleinsäurefärbung, auf einem Wippschüttler, bis es gefärbt ist.
- Übertragen Sie das gefärbte Gel auf eine Glasschale und visualisieren Sie die DNA in einem dunklen Raum unter blauem Licht.
- Schneiden Sie das gewünschte Band mit einem sterilen Skalpell aus. Überschüssiges Gel abschneiden.
- Optional: Reinigen Sie das gewünschte Fragment durch Elektroelution, wie in den Schritten 1.1.4 und 1.1.5 beschrieben. Lassen Sie hier das Salz aus dem Dialysepuffer weg, da dieses nur zur Stabilisierung des kurzen Oligo-Duplex benötigt wird.
- Durchführung des Restriktionsverdaus des pGC261-Plasmids18 unter Verwendung von NotI/I-CeuI-Enzymen.
- Erzeugung von Digoxigenin-Duplex-DNA-Enden
- Führen Sie eine PCR-Reaktion mit Digoxigenin-dUTP (dig-dUTP) durch, wobei pGC261-DNA als Template verwendet wird.
- Mix: 400 μl Wasser, 8 μl pGC261 (0,8 ng/μl), 3,5 μl Primer oHY507 (100 μM), 3,5 μl Primer oHY508 (100 μM), 8 μl dig-dUTP (1 mM), 400 μl 2x PCR-Mix (hergestellt aus DNA-Polymerase (20 μl), 10 mM dNTPs (40 μl), 5x High-Fidelity (HF)-Puffer (400 μl) und Wasser (540 μl).
- Führen Sie die folgende Reaktion in einem Thermocycler durch:
98 °C - 1 min
30x: 98 °C - 20 s; 65 °C - 20 s; 72 °C - 30 s;
72 °C - 10 min
4 °C - Halten
- Verdau und Reinigung von Digoxigenin-markiertem PCR-Produkt
- Aufreinigung des PCR-Produkts mit einem kommerziellen DNA-Aufreinigungskit.
- Mischen Sie 10 μL NotI-HF (20.000 U/ml) mit dem PCR-Produkt in einem vom Enzymlieferanten empfohlenen Puffer (DNA-Endkonzentration von ~50 ng/μL in einer ~200 μL-Reaktion). Die Reaktion wird über Nacht bei 37 °C inkubiert.
- Reinigen Sie die verdaute DNA mit einem kommerziellen DNA-Aufreinigungskit.
- Führen Sie eine PCR-Reaktion mit Digoxigenin-dUTP (dig-dUTP) durch, wobei pGC261-DNA als Template verwendet wird.
- Setze Komponenten zusammen, um das DNA-Substrat herzustellen.
- Mischen Sie das biotinylierte Gabelende, das 20-kb-DNA-Fragment und das mit Digoxigenin markierte Nicht-I-behandelte PCR-Fragment vorsichtig mit 5 μl T4-DNA-Ligase (400.000 U/ml) in einem vom Enzymlieferanten empfohlenen Puffer in einer 200-μl-Reaktion. Basierend auf dem hinzugefügten 20-kb-DNA-Fragment (1-5 μg) wird jedoch etwa ein 100-facher molarer Überschuss sowohl des biotinylierten Gabelendes als auch des PCR-Fragments hinzugefügt.
- Aliquotieren Sie die Reaktion in PCR-Röhrchen (je 50 μl) und inkubieren Sie sie über Nacht bei 16 °C in einem Thermocycler.
- Die ligierte Probe wird auf einem 0,6%igen Agarosegel aufgelöst und die ligierte DNA durch Elektroelution aufgereinigt, wie in den Schritten 1.2.2 und 1.2.3 beschrieben.
- Lagern Sie schockgefrorene DNA bei -80 °C.
2. Aufreinigung von Drosophila melanogaster CMG (Abbildung 2)

Abbildung 2: Aufreinigung von Drosophila melanogaster CMG aus 4 L Hi Five-Zellen. Die Proteine wurden auf 4%-12% Bis-Tris-Polyacrylamid-Gel unter 200 V in Gegenwart von MOPS-Puffer aufgelöst. Die Probe wird in jeder Phase der Aufreinigung gezeigt (Zelllysat - 2 μl, FLAG-Elution - 10 μl, nach der ersten Ionenaustauschersäule - 10 μl und nach der Markierung und der zweiten Ionenaustauschersäule - 1 μl. (A) Die Coomassie-Färbung bestätigt das Vorhandensein aller 11 Untereinheiten des CMG-Komplexes vor (10 μl) und nach (1 μl) Fluoreszenzmarkierung. (B) Die Markierungseffizienz der MCM3-Untereinheit wurde durch Scannen nach Cy5 mit einem Fluoreszenzbildanalysator unter Verwendung eines LPR-Filters (Long Pass Red) validiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
HINWEIS: Zur Herstellung von fluoreszenzmarkiertem Drosophila melanogaster CMG wurde eine TEV-Spaltstelle (ENLYFQG) gefolgt von vier Gly-Resten stromabwärts des N-terminalen FLAG-Tags auf der MCM3-Untereinheit (im pFastBac1-Vektor) eingeführt10. Um den Komplex zu exprimieren, wurde das Baculovirus-Expressionssystem verwendet. Für die initiale Transfektion wurden Sf21-Zellen für jede CMG-Untereinheit (P1-Virusstadium) separat verwendet. Um die Viren weiter zu amplifizieren, wurden Sf9-Zellen verwendet (P2-Virusstadium). Anschließend wurden Sf9-Zellkulturen (100 ml für jede CMG-Untereinheit; 0,5 x 106 Zellen/ml) mit 0,5 ml P2-Virus infiziert, das mit 10 % fötalem Kälberserum (P3-Virusstadium) ergänzt wurde. Um den gesamten CMG-Komplex in 4 L Hi-Five-Zellen (1 x 106 Zellen/ml) zu exprimieren, wurden 200 ml P3-Viren für jede der Untereinheiten verwendet. Nach der Ernte der Hi Five-Zellen, die den CMG-Komplex exprimieren, kann das Zellpellet in flüssigem Stickstoff schockgefroren und bei -80 °C gelagert werden. Die gesamte Reinigung wird auf Eis oder bei 4 °C durchgeführt. Die Puffer können im Voraus hergestellt werden, sofern die Reduktionsmittel (DTT oder 2-Mercaptoethanol) und Proteaseinhibitoren (CAUTION) erst kurz vor der Verwendung zugegeben werden. Stellen Sie sicher, dass alle Puffer im Voraus vorgekühlt, gefiltert und entgast werden.
- Bereiten Sie die folgenden Puffer vor.
- Bereiten Sie den Resuspensionspuffer A vor, indem Sie 25 mM 4-(2-Hydroxyethyl)-1-piperazinethensulfonsäure (HEPES) pH 7,5, 0,02 % Tween-20, 10 % Glycerin, 15 mM KCl, 2 mM MgCl2, 2 mM 2-Mercaptoethanol, 1 mM EDTA und 1 mM Ethylenglykol-bis-(β-aminoethylether)-N,N,N',N',N′-TETRAESSIGSÄURE (EGTA) mischen.
ACHTUNG: 2-Mercaptoethanol, DTT und EDTA sind giftig/gesundheitsschädlich - Bereiten Sie Tris-gepufferte Kochsalzlösung (TBS; 0,1 M Glycin HCl, pH 3,5) Puffer zur Herstellung von ANTI-FLAG M2 Affinitätsgel vor.
- Bereiten Sie Puffer A-100 vor, indem Sie 25 mM HEPES pH 7,5, 0,02 % Tween-20, 10 % Glycerin, 100 mM KCl, 1 mM DTT, 1 mM EDTA und 1 mM EGTA mischen.
- Bereiten Sie Dialysepuffer vor, indem Sie 25 mM HEPES pH 7,5, 50 mM Natriumacetat, 10 mM Magnesiumacetat, 10 % Glycerin und 1 mM DTT hinzufügen.
- Bereiten Sie TBS-Puffer vor, indem Sie 50 mM Tris-HCl, pH 7,5 und 150 mM NaCl mischen.
- Bereiten Sie den Resuspensionspuffer A vor, indem Sie 25 mM 4-(2-Hydroxyethyl)-1-piperazinethensulfonsäure (HEPES) pH 7,5, 0,02 % Tween-20, 10 % Glycerin, 15 mM KCl, 2 mM MgCl2, 2 mM 2-Mercaptoethanol, 1 mM EDTA und 1 mM Ethylenglykol-bis-(β-aminoethylether)-N,N,N',N',N′-TETRAESSIGSÄURE (EGTA) mischen.
- Resuspendieren Sie das Pellet aus einer 4-Liter-Kultur von Hi-Five-Zellen in 200 ml Kaltresuspensionspuffer A, ergänzt mit Proteasehemmer-Cocktailtabletten. Mischen Sie die Röhrchen durch Inversion, bis das gesamte Pellet vollständig resuspendiert ist.
- Übertragen Sie die resuspendierten Zellen in einen vorgekühlten 40 mL Dounce Homogenisator. Um so viele Zellen wie möglich zurückzugewinnen, waschen Sie die Röhrchen, die für das Einfrieren der Zellpellets verwendet werden, mit der gleichen Art von Puffer und geben Sie sie in den Dounce-Homogenisator. Stellen Sie sicher, dass das Gesamtvolumen im Homogenisator den empfohlenen Grenzwert nicht überschreitet. Lysieren Sie die Zellen auf Eis mit 60-70 Hüben.
HINWEIS: Stellen Sie sicher, dass der Kolben den Boden des Röhrchens erreicht, aber üben Sie nicht zu viel Druck aus, da der Homogenisator brechen kann. Achten Sie darauf, den Kolben nicht über dem Flüssigkeitsspiegel herauszunehmen, da dies zu Blasen führt. - Wiederholen Sie den vorherigen Schritt, bis die gesamte Probe homogenisiert ist, und mischen Sie sie im vorgekühlten Becherglas. Beurteilen Sie das endgültige Volumen der Probe. Tropfen für Tropfen filtriertes KCl bis zur Endkonzentration von 100 mM zugeben und vorsichtig mischen.
- Die Zentrifuge vorher auf 4 °C vorkühlen. Gießen Sie die Probe in Zentrifugationsröhrchen und stellen Sie sicher, dass das vom Hersteller empfohlene Volumen erreicht wird. Balancieren Sie die Röhren auf einer Waage aus.
HINWEIS: Röhrchen mit zu kleinem Volumen können während der Zentrifugation brechen. - Zentrifugieren Sie die lysierten Zellen bei 23.500 x g für 15-30 min bei 4 °C. Bereiten Sie in der Zwischenzeit das ANTI-FLAG M2 Affinitätsgel entsprechend dem nächsten Schritt vor.
- Mischen Sie vorsichtig die Flasche mit dem ANTI-FLAG M2 Affinitätsgel (50% Suspension). Schneiden Sie das Ende einer P-100-Pipettenspitze ab (um die Spitze zu verbreitern) und verwenden Sie sie, um sofort 4 ml Suspension (2 ml Kügelchen) in eine 20-ml-Poly-Prep-Chromatographiesäule zu überführen. Um die in der Spitze festsitzenden Perlen zu bergen, waschen Sie die Spitze mit TBS und fügen Sie sie der Säule hinzu. Lassen Sie alle Perlen in der Säule absetzen, aber achten Sie darauf, sie nicht zu trocknen.
- Waschen Sie das Harz mit 3x Kügelchen Volumen (insgesamt 6 mL) von 0,1 M Glycin HCl pH 3,5. Waschen Sie anschließend die Kügelchen mit 3-5 Kügelchen Volumen TBS (6-10 mL). Waschen Sie anschließend die Kügelchen 3 Mal (6 ml) mit Puffer A-100.
HINWEIS: Lassen Sie die Kügelchen nicht länger als 20 Minuten in der 0,1 M Glycin HCl pH 3,5 Lösung. Um den Fließdruck und die Waschgeschwindigkeit zu erhöhen, decken Sie die Säule mit einem Deckel ab. Lassen Sie das Harz an keiner Stelle trocknen. - Lassen Sie nach der Vorbereitung des Harzes 2 mL Puffer A-100 über dem Harz und schließen Sie den Auslass der Säule.
- Nach der Zentrifugation wird der Überstand vorsichtig in vorgekühlte 50-ml-Röhrchen gegossen. Achten Sie darauf, das Pellet nicht zu beschädigen. Nehmen Sie ein kleines Aliquot (~10 μL), um es später durch SDS-PAGE-Elektrophorese zu validieren.
- Geben Sie eine gleiche Menge ANTI-FLAG M2-Kügelchen (gemäß dem vorherigen Schritt vorbereitet) in jedes der 50-ml-Röhrchen. Versuchen Sie, alle Kügelchen aus der Säule zu entfernen, indem Sie sie mit ein paar zusätzlichen Millilitern Puffer A-100 resuspendieren und diese Resuspension auf die Röhrchen übertragen.
- Die Probe (Überstand) wird mit dem Harz ANTI-FLAG M2 inkubiert und 2,5 Stunden lang bei 4 °C gedreht.
- Nach der Inkubation wird die Probe bei 1.000 x g 5 min bei 4 °C geschleudert. Entfernen Sie mit einer Pipette den größten Teil des Überstands. Mit einer abgeschnittenen Spitze die Kügelchen in einigen Millilitern des restlichen Überstands resuspendieren und in zwei 15-ml-Röhrchen überführen. Um an den Wänden der 50-ml-Röhrchen haftende Kügelchen zu entfernen, fügen Sie einige Milliliter Buffer A-100 hinzu und übertragen Sie diesen ebenfalls auf die 15-ml-Röhrchen.
- Die 15-ml-Röhrchen bei 1.000 x g für 5 min bei 4 °C drehen. Entfernen Sie den Überstand.
- Waschen Sie die Kügelchen, indem Sie ~14 mL Puffer A-100 in jedes Röhrchen geben, gefolgt von einer kurzen, sanften Drehung bei 4 °C und anschließendem Schleudern bei 1000 x g für 5 min bei 4 °C. Wiederholen Sie die Wäschen zweimal. Nehmen Sie kleine Aliquots von jeder Wäsche (~10 μL), um sie später durch SDS-PAGE-Elektrophorese zu validieren.
- Dieser Schritt (2.16) ist eine Alternative zu den vorherigen Schritten (2.13-2.15).
- Nach der Inkubation mit ANTI-FLAG M2 Harz gießen Sie die Probe in zwei 20 mL Poly-Prep-Chromatographiesäulen.
- Wenn Sie sich für diese Methode entscheiden, lassen Sie die Kügelchen am Boden der Säule absetzen und die ungebundene Probe durch die Schwerkraft durch die Säule laufen. Anschließend wird der Puffer A-100 direkt in die Säule gegeben und die Probe durch Schwerkraft gewaschen.
- Übertragen Sie die Beads (resuspendiert in Puffer A-100) auf zwei 10-ml-Poly-Prep-Chromatographiesäulen.
- Lassen Sie Puffer A-100 durch das Harz passieren, schließen Sie den Auslass, wenn der Pufferspiegel die Oberseite des Harzes erreicht, und eluieren Sie das Protein aus den Kügelchen, indem Sie Puffer A-100 hinzufügen, der mit 200 μg/ml (DYKDDDDK) Peptid (FLAG-Elutionspuffer) ergänzt ist.
- Für die erste Elution fügen Sie 3 mL des FLAG-Elutionspuffers hinzu und drehen Sie die geschlossene Säule vorsichtig 15 Minuten lang bei RT. Öffnen Sie den Auslass, um die erste Elutionsfraktion aufzufangen.
- Schließen Sie den Ausgang und fügen Sie jeder Säule 2 ml des FLAG-Elutionspuffers hinzu. Drehen Sie für weitere 10 Minuten und sammeln Sie die zweite Elutionsfraktion.
- Kombinieren Sie alle eluierten Fraktionen miteinander (~10 mL) und halten Sie sie bei 4 °C. Nehmen Sie ein kleines Aliquot der eluierten Fraktion (~10 μL), um es später durch SDS-PAGE-Elektrophorese zu validieren.
- Filtrieren Sie die eluierte Probe mit 0,22 μm Spritzenfiltern.
- Bereiten Sie im Voraus die CaptoHiRes Q (5/50)-Säule (hochauflösende Ionenaustauschchromatographie-Säule) vor, die an ein Proteinreinigungssystem angeschlossen ist, gemäß dem Protokoll des Herstellers. Anschließend wird die Säule mit dem Puffer A-100 äquilibriert und die gefilterte Probe auf die äquilibrierte Säule geladen.
- Waschen Sie die Säule mit 20 Säulenvolumina (CV) Puffer A-100 (~20 mL insgesamt, da das Volumen der Säule ~1 mL beträgt).
- Um das Protein zu eluieren, bereiten Sie im Voraus zwei Puffer vor: Puffer A-100 und Puffer B mit der gleichen Zusammensetzung wie Puffer A-100, jedoch mit 550 mM KCl anstelle von 100 mM KCl. Richten Sie die Gradientenelution für 20 CV (~20 mL) mit steigender Salzkonzentration von 100 mM auf 550 mM KCl ein. Sammeln Sie 0,3-0,5 mL Elutionen in einem Fraktionssammler.
HINWEIS: Das CMG sollte bei einer Konzentration von etwa 70 % bis 75 % von Puffer B eluieren. Nehmen Sie kleine Aliquots der wasch- und eluierten Fraktionen für die SDS-PAGE-Elektrophorese. - Führen Sie eine SDS-PAGE-Elektrophorese durch, um zu bestätigen, dass CMG in den ausgewählten Fraktionen vorhanden ist. Reinigen Sie in der Zwischenzeit die Säule und das Reinigungssystem gemäß dem Protokoll des Herstellers.
- Inkubieren Sie die ausgewählten Fraktionen über Nacht mit TEV-Protease, indem Sie 50 μl TEV-Protease (1 mg/ml) pro 1 ml Probe mischen.
- Befeuchten Sie einen Dialyseschlauch und stellen Sie sicher, dass die Membran nicht beschädigt wird. Geben Sie das CMG/TEV-Gemisch in den Dialyseschlauch und geben Sie es in ein 2-Liter-Becherglas mit 1,5 l vorgekühltem Puffer A-100 und dem darin befindlichen Magnetstab. Dialysieren Sie unter leichtem Rühren über Nacht bei 4°C.
- Mischen Sie die Probe mit 50 μM LD655-markiertem Peptid und 10 μg/ml Sortase-Enzym in Gegenwart von 5 mM CaCl2. Inkubieren Sie die Reaktion bei 4 °C und drehen Sie sie 30 Minuten lang, wobei Sie sicherstellen, dass das Röhrchen lichtgeschützt ist. Nehmen Sie vor und nach der Markierung für die SDS-PAGE-Elektrophorese ein kleines Aliquot vor.
- Filtrieren Sie die markierte Probe mit 0,22-μm-Zentrifugenfiltern, bevor Sie sie in das Aufreinigungssystem laden, da das Peptid ausfallen kann.
- Bereiten Sie die hochauflösende Ionenaustauschchromatographie-Säule vor, wie zuvor beschrieben (Schritt 2.20). Decken Sie dieses Mal die Säule und das Fraktionssammlersystem mit Aluminiumfolie ab, um die Probe vor Licht zu schützen.
- Um das freie Peptid zu entfernen, laden Sie die gefilterte Probe auf die hochauflösende Ionenaustauschchromatographie-Säule. Führen Sie die Reinigung auf die gleiche Weise wie zuvor beschrieben durch (Schritte 2.21-2.22). Stellen Sie sicher, dass das markierte CMG bei einer ähnlichen Konzentration von Puffer-B wie zuvor eluiert.
- Führen Sie eine SDS-PAGE-Elektrophorese durch, um die Qualität der ausgewählten Fraktionen zu validieren. Um die Fluoreszenz abzubilden, kochen Sie die Proben vor dem Laden nicht und stellen Sie sicher, dass der Elektrophoresetank vor Licht geschützt ist. Visualisieren Sie zuerst die Proteinfluoreszenz mit einem Gel-Imaging-System, färben Sie das Gel dann mit Coomassie-Farbstoff und bilden Sie es erneut ab, um alle Proteine sichtbar zu machen.
- Dialysieren Sie ausgewählte Fraktionen über Nacht bei 4 °C gegen 1,5 l Dialysepuffer. Falls erforderlich, konzentrieren Sie die Probe.
- Frieren Sie das Protein in flüssigem Stickstoff ein und lagern Sie es bis zur weiteren Verwendung bei -80 °C.
3. Vorbereitung der Durchflusszelle (Abbildung 3)
- Biotin-PEG-Deckgläser nach einem zuvor beschriebenen Protokoll11 vorbereiten, wobei der Backschritt im Ofen weggelassen wird. Biotin-PEG-Deckgläser sind unter Vakuum bei RT mindestens 1 Monat stabil. Zur Vorbereitung der Durchflusszelle wird das Biotin-PEG-Deckglas (24 mm x 60 mm) in zwei Hälften (ca. 24 mm x 30 mm) geschnitten.
HINWEIS: Achten Sie darauf, den mittleren Bereich des Deckglases, in dem sich die Strömungskanäle befinden, nicht zu berühren. - Bereiten Sie kleine Glasstücke vor, indem Sie einen Objektträger ätzen und in ca. 2,4 cm x 1 cm große Stücke einrasten.
- Bohren Sie mit einem 0,8 mm diamantbeschichteten Bohrer zwei Löcher im Abstand von 1,4 mm, eines etwas breiter als das andere (0,043" = Einlauf, 0,048" = Auslass). Testen Sie, ob die Löcher die richtige Größe haben, indem Sie versuchen, ein Stück des Einlass-/Auslassschlauchs einzuführen und das Loch mit demselben Bohrer zu erweitern, bis der Schlauch passt. Stellen Sie sicher, dass der Schlauch fest genug sitzt, um nicht leicht herauszufallen.
HINWEIS: Es werden schmalere Einlassschläuche verwendet, um das Totvolumen zu minimieren. - Schneiden Sie doppelseitiges Klebeband in der gleichen Form wie die Glasstücke.
- Richten Sie den Schieber auf dem Klebeband aus und stechen Sie mit einer Nadel durch jedes Loch, um ihre Positionen auf dem Band zu markieren. Schneide mit einer Rasierklinge einen Kanal, der beide Löcher umfasst.
HINWEIS: Schneiden Sie den Kanal nicht zu lang, da überschüssiger Raum auf der anderen Seite jedes Lochs trocken bleiben kann, wenn Flüssigkeit eingeströmt wird, und zu Problemen mit Luftblasen während der Strömung führen kann. Versuchen Sie beim Schneiden, die langen Kanten in einem einzigen sauberen Schnitt zu schneiden, da ausgefranste Kanten durch mehrere Schnitte die Qualität des Strömungskanals beeinträchtigen können. Schneiden Sie zu einem ähnlichen Zweck nicht in die Ränder des Strömungskanals, da Schnitte in das verwendbare Stück den Flüssigkeitsfluss beeinträchtigen oder sogar in der zusammengebauten Durchflusszelle auslaufen können. - Reinigen Sie das Glasstück mit Aceton und Taschentuch, bis es trocken ist, und legen Sie es dann auf eine saubere Oberfläche. Ziehe eine Seite des Klebebandes ab und klebe es so auf das Glasstück, dass beide Löcher vollständig im Kanal liegen.
- Versiegeln Sie das Klebeband mit einer p1000-Pipettenspitze, um Blasen zu entfernen, indem Sie mit festem, aber mäßigem Druck auf die Oberfläche drücken. Gehen Sie über die gesamte Oberfläche, um sie zu versiegeln.
- Ziehe die zweite Kante des Klebebandes von beiden Glasstücken ab und lege sie mit der klebrigen Seite nach oben auf eine saubere Oberfläche. Platzieren Sie die Objektträger nahe beieinander, aber berühren Sie sich nicht in der Position, in der sie am Deckglas befestigt werden sollen (lassen Sie genügend Platz zwischen ihnen, damit das Epoxidharz jedes Glasstück vollständig abdichtet).
- Nehmen Sie das halbe Biotin-PEG-Deckglas mit einer Kunststoffpinzette auf, halten Sie es nur an der Kante fest (berühren Sie nicht den Bereich, der den Strömungskanal bildet), und senken Sie die PEG-funktionalisierte Seite auf den Klebstoff ab. Sichern Sie es, indem Sie es mit einem Finger nach unten drücken, um es an Ort und Stelle zu fixieren, und vervollständigen Sie dann die Versiegelung, indem Sie die Oberfläche mit einer Pipettenspitze mäßig fest abreiben, um Luftblasen zu entfernen (nicht im Bereich des Strömungskanals, da diese brechen können), und drehen Sie es dann um.
HINWEIS: Behandeln Sie das Deckglas mit Vorsicht. - Schneiden Sie für jede Durchflusszelle ~10 cm von jedem Typ von Polyethylenschlauch (PE20 und PE60) ab. Verwenden Sie das schmalere Rohr als Einlass in die Durchflusskammer, um das Totvolumen zu reduzieren.
- Führen Sie den Schlauch von Hand in die Löcher in der Rutsche ein. Wenn der Durchmesser der Löcher korrekt ist, stellen Sie sicher, dass der Schlauch nach dem Einführen von selbst im Loch steht. Schneiden Sie die Schlauchspitze in einem leichten Winkel <45° ab, um das Einführen des Schlauchs etwas zu erleichtern und zu verhindern, dass der Schlauch eine Dichtung gegen das Deckglas bildet, was bei flachem Ende der Fall sein kann (dies kann den Durchfluss behindern).
HINWEIS: Der genaue Einbau des Schlauchs in das Glasstück macht die Konstruktion der Durchflusszelle zuverlässiger und hilft, das Einbringen von Blasen in die Durchflusszelle zu vermeiden. - Mischen Sie die Epoxidkomponenten gut und tupfen Sie dann mit einer P200-Spitze Epoxidharz ab, um den Schlauch abzudichten und eine Abdichtung um jedes Glasstück zu schaffen. Fügen Sie so viel Epoxidharz hinzu, dass es bis zum Rand des Deckglases rund geht, da dies auch das empfindliche Deckglas verstärkt. Achten Sie jedoch darauf, dass kein Epoxidharz auf die Unterseite des Deckglases gelangt, da es verhindern kann, dass es flach auf dem Tisch liegt.
HINWEIS: Bei Bedarf können kleine Mengen überschüssiges Epoxidharz mit einer Rasierklinge von der Unterseite abgekratzt werden. - Lassen Sie es mindestens 30-60 Minuten einwirken, damit das Epoxidharz vollständig ausgehärtet ist.
HINWEIS: Wenn die Durchflusszelle nicht sofort verwendet werden soll, lagern Sie sie nach vollständiger Aushärtung unter Vakuum bei RT. - Nach der Verwendung der Durchflusszelle können Sie die Glasstücke zurückgewinnen und auf unbestimmte Zeit wiederverwenden. Ziehen Sie den Schlauch heraus und legen Sie die Durchflusszelle für mindestens 24 Stunden in ein Diagefäß mit Aceton, um das Epoxidharz und den doppelseitigen Klebstoff aufzuweichen, damit die Glasstücke leicht entfernt werden können. Reinigen Sie die Glasstücke, bevor Sie sie wiederverwenden, indem Sie sie mit einem Schwamm und Seifenwasser schrubben, trocknen Sie sie ab und schrubben Sie sie mit Aceton und Seidenpapier.

Abbildung 3: Grafische Darstellung der Vorbereitung der Durchflusszelle. (A) Schneiden Sie das doppelseitige Klebeband so zu, dass es der Größe des Glasstücks entspricht. Richten Sie die Folie oben auf dem Klebeband aus und markieren Sie die Position jedes Lochs mit einer Nadel. Schneide mit einer Rasierklinge um jeden Griff herum, um einen Kanal zu erzeugen. (B) Ziehen Sie eine Seite des Klebebandes ab und kleben Sie das Klebeband auf das Glasstück. Stellen Sie sicher, dass sich beide Löcher innerhalb des Kanals befinden. Ziehen Sie das zweite Ende des Klebebandes ab und kleben Sie das Biotin-PEG-Deckglas darauf. (C) Führen Sie den Polyethylenschlauch in jedes Loch ein und versiegeln Sie den Schlauch mit Epoxidharz, wobei Sie jedes Glasstück auch mit dem Deckglas versiegeln. (D) Nachdem Sie beide Kanäle verwendet haben, ziehen Sie den Schlauch heraus und setzen Sie die Durchflusszelle in ein mit Aceton gefülltes Färbegefäß ein. Nach ca. 24 h sind das Epoxidharz und das Klebeband weich geworden und die Schichten der Durchflusszelle können auseinander gezogen werden. Die Glasstücke können zurückgewonnen und in Aceton gelagert werden, um auf unbestimmte Zeit für die Herstellung der nächsten Durchflusszelle wiederverwendet zu werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Einzelmolekül-TIRF-Assay zur Visualisierung der CMG-vermittelten DNA-Entwindung
- Bereiten Sie die folgenden Puffer vor.
- Bereiten Sie den Blockierungspuffer vor (20 mM Tris, pH 8,0; 50 mM NaCl; 2 mM EDTA; 0,2 mg/ml BSA). Den Puffer mit einem 0,22 μm Spritzenvorsatzfilter filtrieren und bei 4 °C lagern.
- Bereiten Sie 10x Reaktionspuffer 1 (250 mM HEPES pH 7,5; 50 mM NaCl; 100 mM Magnesiumacetat) vor. Den Puffer mit einem 0,22 μm Spritzenvorsatzfilter filtrieren und bei 4 °C lagern.
- Bereiten Sie 10x Reaktionspuffer 2 (250 mM Tris, pH 7,5; 100 mM Magnesiumacetat; 1,25 M Kaliumglutamat; 1 mM EDTA; 0,025% Tween-20) vor. Den Puffer mit einem 0,22 μm Spritzenvorsatzfilter filtrieren und bei 4 °C lagern.
- Entgasen Sie ca. 2,5 mL Blockierungspuffer und 5 mL Reinstwasser, indem Sie die offenen Röhrchen in einen Exsikkator legen und 15 Minuten lang unter Vakuum belassen.
HINWEIS: Füllen Sie die Schläuche nicht bis zum Rand, da Blasen während der Entgasung dazu führen können, dass Flüssigkeit spritzt. - Stellen Sie die Durchflusszelle auf einen Mikroskoptisch und befestigen Sie sie mit einem Klebekitt an jedem Ende. Es ist wichtig, gut zu sichern, um zu verhindern, dass sich die Durchflusszelle später während der Aufnahme bewegt.
- Verbinden Sie den Auslassschlauch der Durchflusszelle mit der Spritzenpumpe, an der am anderen Ende ein Schlauch mit einer Nadel an der Spritze befestigt ist. Führen Sie diese Nadel in den Auslassschlauch ein, um die Durchflusszelle anzuschließen.
- Schalten Sie die Objektivheizung auf 30 °C ein.
- Befestigen Sie 1-2 ml entgastes Wasser in einem Röhrchen an einem separaten Stück Klebekitt in der Nähe der Durchflusszelle, so dass der Einlassschlauch eingeführt werden kann und den Boden des Röhrchens erreicht.
- Lassen Sie Wasser durch den Kanal fließen und verwenden Sie dann einen schnelleren Durchfluss, um eingeschlossene Blasen in der Nähe des Einlassschlauchs zu entfernen, und fügen Sie bei Bedarf mehr Wasser hinzu. Andernfalls können sich später in den Experimenten große Blasen lösen und den Kanal passieren. Lassen Sie es einige Minuten einwirken, bis der Fluss vollständig gestoppt ist, da der schnelle Fluss nach dem Stoppen zu einem Restziehen der Flüssigkeit führen kann, da sich der Druck in der Spritze stabilisiert.
HINWEIS: Jede DNA, die mit einer Blase in Kontakt kommt, wird unbrauchbar, selbst wenn sie rehydriert ist. - 100 μl entgaster Blockierungspuffer werden zu einem 20 μl Aliquot von 1 mg/ml Streptavidin gegeben. Befestigen Sie den offenen Schlauch an der Klebespachtel und übertragen Sie den Zulaufschlauch aus dem Wasser auf das Streptavidin. Mit einer Geschwindigkeit von 40 μl/min für 2 min (insgesamt 80 μl) fließen und 5 min inkubieren.
- Überschüssiges Streptavidin mit dem Blockierungspuffer auswaschen (50 μl/min für 100 μl).
- Fluss in biotinylierter DNA, verdünnt in Blockierungspuffer mit 25 nM SYTOX orange. Bild im Live-View mit dem 532-nm-Laser, um zu sehen, wie sich die DNA in Echtzeit an die Oberfläche bindet.
- Wenn die ungefähre Dichte der DNA auf der Oberfläche erreicht ist, wird ein Blockierungspuffer eingegossen, ebenfalls mit 25 nM SYTOX (50 μL/min für 100 μL), um die freie DNA auszuwaschen.
- Fluss in biotinyliertem Anti-Digoxigenin-Antikörper (~10 μg/ml), verdünnt in Blockpuffer mit 25 nM SYTOX orange (100 μL/min für 300 μL).
HINWEIS: Das SYTOX-Orange interkaliert zwischen den Basenpaaren der DNA und verlängert so die Länge der Kontur. Dies bedeutet, dass die End-to-End-Länge der DNA in SYTOX-Orange zunimmt, so dass sich der zweite Strang weiter von dem ersten entfernt an der Oberfläche anheften kann, was zu einer besseren Dehnung der DNA führt, wenn die SYTOX-Orange später ausgewaschen wird. Die Verwendung einer anderen Einheit an jedem DNA-Ende ermöglicht eine gleichmäßigere Bindung der DNA an die Deckglasoberfläche (Abbildung 4). Wenn beide Enden biotinyliert sind, binden sie sich im selben Schritt, was zu einer erheblichen Variation ihrer Befestigungspositionen führt. Die Verwendung von Digoxigenin an einem Ende gibt mehr Kontrolle über die DNA-Abdeckung, bevor das zweite Ende angebunden wird. Dadurch kann auch die DNA-Bedeckung durch Inkubation erhöht werden. - Waschen Sie den biotinylierten Anti-Digoxigenin-Antikörper und die SYTOX-Orange mit Blockierungspuffer (50 μL/min für 100 μL) aus.
- Bilden Sie 120 μl "ATP-g-s-Mix" (1x Reaktionspuffer 1, 0,75 mg/mL BSA, 1,25 mg/mL Kasein, 8 mM DTT, 0,33 mM ATP-g-s), geben Sie 30 μl in ein neues Röhrchen und fließen Sie dann 50 μl aus dem ursprünglichen Röhrchen in die Durchflusszelle (50 μl/min für 50 μl). Dies trägt dazu bei, die Auswirkungen der Puffermischung durch Rückfluss oder Diffusion aus dem Auslassschlauch zu minimieren.
- Gereinigtes CMG auf ~100 nM final in 30 μl ATP-g-s-Mischung geben und dann mit 20 μl/min für 20 μl einströmen. 15 min inkubieren.
- Bilden Sie 120 μl "ATP/RPA-Mix" (1x Reaktionspuffer 1, 0,75 mg/ml BSA, 8 mM DTT, 3,3 mM ATP, 20 nM EGFP-hRPA) und fließen Sie mit 40 μl/min für 80 μl in die Durchflusszelle.
- Beginnen Sie sofort mit der Aufnahme von Bildern. Erfassen Sie alle 30 s 6 x 6 Sichtfelder für jedes Bild (oder eine geeignete Bildrate für das Experiment). Niedrigere Bildraten können die Laserbelichtung und das Fluorophor-Photobleaching reduzieren. Visualisieren Sie EGFP-hRPA mit einem 488-nm-Laser bei 1 % Leistung. Wenn CMG markiert ist, z. B. LD655, visualisieren Sie CMG mit einem 640-nm-Laser (maximale Leistung = 30 mW) bei 10 % Leistung. Visualisieren Sie SYTOX orange-gefärbte DNA mit einem 532-nm-Laser bei 0,5 % Leistung. Verwenden Sie jeden Laser mit einer Belichtungszeit von 50-100 ms.
HINWEIS: Jedes Mal, wenn der Einlassschlauch der Durchflusszelle von einem Flüssigkeitsschlauch in einen anderen übertragen wird, tupfen Sie die Spitze des Schlauchs mehrmals auf den Boden des Schlauchs, bevor Sie ihn aus der Flüssigkeit entfernen. Dies hilft, das Eindringen von Blasen während der Übertragung zu verhindern. Minimieren Sie die Zeit, in der die Spitze des Einlassschlauchs aus der Flüssigkeit gehalten wird, und richten Sie die Spitze nicht nach oben oder heben Sie die Spitze nicht deutlich an, da dies dazu führt, dass die Flüssigkeit in Richtung des Kanals abfließt und Luft in die Spitze zieht. Versuchen Sie beim Übertragen des Einlassschlauchs, den Schlauch nicht unter Spannung zu setzen, da dies ihn aus dem Epoxidharz ziehen und an der Durchflusszelle befestigen kann. Für die Bildgebung der DNA-Abwicklung in Gegenwart von SYTOX Orange zur Visualisierung doppelsträngiger DNA ist 10x Reaktionspuffer 1 durch 10x Reaktionspuffer 2 für den ATP/RPA-Mix zu ersetzen. SYTOX Orange bindet unter diesen Bedingungen besser an die DNA. Entleeren Sie die Spritzenpumpe am Ende jedes Versuchs vollständig. Gelegentlich waschen, indem Sie mit Wasser befüllen und mehrmals entleeren oder demontieren und mit Seifenwasser waschen.

Abbildung 4: DNA-Bindung an die Oberfläche. (A) Beim Anbinden von DNA-Substraten mit Biotin an beiden Enden kann der Abstand zwischen den beiden Fesseln variieren, je nachdem, wie die Enden die Oberfläche berühren (i). Durch die Verwendung von Digoxigenin an einem Ende kann die Tethering an jedem Ende zeitlich getrennt werden, um konsistentere Tether-Abstände und gleichmäßiger gedehnte DNA zu erreichen (ii). (B) Beispiel für ein Sichtfeld, das DNA zeigt, die an beiden Enden angebunden ist (Digoxigenin-markiert) und mit fluoreszierender interkalierender Nukleinsäurefärbung gefärbt ist. Die DNA, die an beiden Enden gebunden ist, erscheint als Linie, während die DNA, die nur an einem Ende gebunden ist, als Punkte erscheint. Im Idealfall sollte die DNA so dicht wie möglich angebunden werden, ohne andere DNA zu überlappen. Das Bild ist 512 x 512 Pixel groß (Pixelgröße = 154,6 nm). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Repräsentative Ergebnisse
Wenn CMG die DNA abwickelt, wächst im Laufe der Zeit ein charakteristischer RPA-Trakt (Abbildung 5). Das 5'-Ende der abgewickelten DNA ist an die Oberfläche gebunden; Daher wird es als linearer Abschnitt des RPA-Signals zwischen dem Tether und der Gabel angesehen. Das 3'-Ende ist nicht angebunden und bewegt sich daher mit der Gabel und wird als kompaktes EGFP-RPA-Signal betrachtet. Die Position des verdichteten, abgewickelten Translokationsstrangs entspricht in etwa der Position der Replikationsgabel, die sich zusammen mit LD655-CMG bewegt und über einen 640-nm-Laser sichtbar gemacht wird.
Es ist wichtig, Schäden am DNA-Substrat zu minimieren, da Schäden wie einzelsträngige DNA-Kerben die Anzahl der beobachtbaren Abwicklungsereignisse verringern und die Menge der Daten, die gesammelt werden können, begrenzt (Abbildung 6).
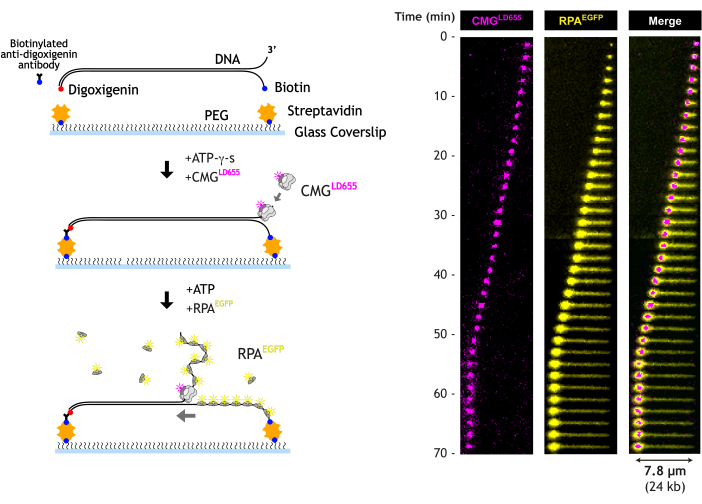
Abbildung 5: Einzelmolekül-DNA-Entwindungs-Assay. Das DNA-Substrat ist an eine Deckglasoberfläche gebunden. Gereinigtes CMG, das mit LD655 markiert ist, wird mit der DNA 15 Minuten lang in ATP-g-s inkubiert. ATP und gereinigtes EGFP-markiertes RPA werden hinzugefügt, wodurch eine umfangreiche DNA-Entwindung durch CMG eingeleitet wird. Gezeigt werden ein Cartoon-Schema (links) und ein Kymograph mit repräsentativen Daten (rechts). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
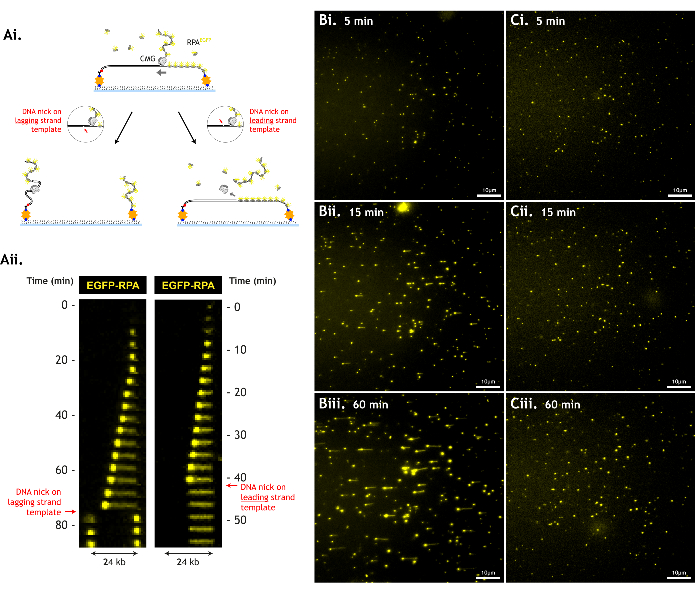
Abbildung 6: DNA-Schäden verringern den Assay-Durchsatz. (A) CMG kann DNA nicht über einen Bruch im DNA-Rückgrat hinaus abwickeln (DNA-Nick). Eine Kerbe an der führenden Strangvorlage führt dazu, dass CMG von der DNA abrutscht, und sowohl CMG als auch die führende Strangvorlage gehen verloren. Eine Kerbe an der nacheilenden Strangschablone bewirkt, dass sich die nacheilende Strangschablone vom Rest der DNA trennt und jedes DNA-Stück auf sein jeweiliges Seil zurückgezogen wird. Dies wird durch (i) Cartoon-Schaltpläne und (ii) Kymographen dieser Ereignisse (ii) veranschaulicht. Repräsentative Daten mit (B) einem minimal geschädigten DNA-Substrat versus (C) einem stärker geschädigten DNA-Substrat bei (i) 5 min, (ii) 15 min und (iii) 60 min in einem einzigen Sichtfeld. Das stärker geschädigte DNA-Substrat erzeugt keine langen Abwickelstrecken, da die CMGs trotz ähnlicher Abwickelaktivität früher auf Kerben stoßen (ähnliche Dichte an wachsenden RPA-Spots bei 5 min, was auf eine ähnliche CMG-Lade-/Abwickeleffizienz hinweist). Das Sichtfeld beträgt 512 x 512 Pixel (Pixelgröße = 154,6 nm). 1 % Laserleistung (488 nm) für die Bildgebung von EGFP-RPA. Maßstabsbalken mit 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Dieser Assay bietet eine Plattform, um die Echtzeitdynamik einzelner CMGs sowohl isoliert als auch im Kontext der gewünschten zusätzlichen Faktoren zu beobachten und zu untersuchen. Wie bei vielen Einzelmolekül-Fluoreszenztechniken gibt es jedoch einige häufige Herausforderungen, die optimiert werden müssen, um sie zu bewältigen. Diese beziehen sich in der Regel auf die Abbildung von Fluorophoren über lange Zeiträume (Photobleichung, Helligkeit), die DNA-Substratvorbereitung (DNA-Schäden), die Qualität der Oberfläche der Flusszelle (Hintergrundrauschen, unspezifische Wechselwirkungen) oder die Qualität der gereinigten Proteinpräparation (Nukleasekontamination, Markierungseffizienz).
Jedes Fluorophor variiert in seiner Photostabilität und Helligkeit, daher ist es wichtig, ein geeignetes Molekül auszuwählen. Bei der Abbildung von fluoreszenzmarkierten oligomeren Proteinen wie RPA kann eine geringere Laserleistung verwendet werden, da viele Fluorophore in unmittelbarer Nähe angeregt werden und ein sichtbares Signal erzeugen. Für die Abbildung einzelner Fluorophore, z. B. CMG, die auf einer einzelnen Untereinheit markiert sind, ist eine höhere Laserleistung erforderlich, um das Fluorophor klar zu beobachten. Die Lebensdauer von Fluorophoren kann durch Minimierung der Laserbelichtung verlängert werden, z. B. durch Verringerung der Häufigkeit, mit der Bilder aufgenommen werden. Darüber hinaus erzeugt die Anregung eines Fluorophors reaktive Sauerstoffspezies (ROS), die zur Photobleiche beitragen können. Die Aufnahme eines Sauerstofffängersystems in den Bildgebungspuffer kann die Lebensdauer von Fluorophoren verlängern, indem ROS eliminiert werden. Einige Sauerstofffängersysteme können jedoch denpH-Wert von 12 beeinflussen.
Bei der Präparation von DNA-Substraten ist es entscheidend, DNA-Schäden, wie z.B. Kerben oder einzelsträngige Lücken, zu minimieren. Übermäßige Beschädigung verhindert eine umfangreiche DNA-Entkopplung, was die Menge an Daten, die gesammelt werden kann, einschränkt. Schäden können durch mechanische Scherung, übermäßige Erwärmung als Folge von Nukleasekontamination oder ROS entstehen, die während der Bildgebung entstehen. Das Scheren kann minimiert werden, indem die DNA-Probe vorsichtig gehandhabt wird, indem Spitzen mit breiter Öffnung zum Pipettieren verwendet werden, langsam pipetiert wird und ein Schnippen der Probe vermieden wird. Der Effekt von ROS kann minimiert werden, indem entweder die Laserbelichtung reduziert oder ein Sauerstoffabscheidesystem in den Bildgebungspuffer aufgenommen wird. Nach der Vorbereitung des DNA-Substrats ist es möglich, kommerzielle DNA-Reparaturkits zu verwenden, um den Schaden zu reparieren, bevor eine Abwickelreaktion durchgeführt wird.
Die Effizienz der DNA-Entwindung hängt auch von der Reinheit und Aktivität von CMG ab. Es empfiehlt sich, die Reinheit der Probe nach jedem Aufreinigungsschritt durch SDS-PAGE-Elektrophorese zu beurteilen, um festzustellen, wo eine Optimierung erforderlich ist. Wenn nach dem letzten Schritt zu viele Verunreinigungen beobachtet werden, kann es hilfreich sein, die Salzgradientenvolumina zu modifizieren, die für die Elution aus der CaptoHiRes Q (5/50)-Säule verwendet werden. Es ist auch sehr wichtig, überschüssiges fluoreszierendes Peptid, das für die Proteinmarkierung verwendet wird, zu entfernen, da es einen unerwünschten Hintergrund auf der Deckglasoberfläche erzeugen kann. Es ist auch wichtig, eine Nukleasekontamination zu vermeiden, da diese das DNA-Substrat abbauen kann. Nach einem Experiment kann das Färben der verbleibenden DNA mit SYTOX-Orange eine gute Möglichkeit sein, um zu überprüfen, ob die DNA signifikant abgebaut wurde oder nicht. Ein gewisses Maß an DNA-Schäden ist im Laufe eines Experiments unvermeidlich, aber signifikante Schäden deuten oft auf eine problematische Nukleasekontamination hin.
Der Assay ist auch von Natur aus durch die Auflösung von beugungsbegrenzten Spots begrenzt, so dass fluoreszierende Proteine Hunderte von Basenpaaren (wenn nicht mehr) entfernt sein müssen, um sie als getrennt zu unterscheiden. Dies schränkt die Details ein, in denen CMG-Progression und Wechselwirkungen beobachtet werden können.
Die Anzahl der Abwicklungsereignisse, die wir bei jeder Analyse beobachten, variiert. Für ein erfolgreiches Experiment erwarten wir mindestens mehrere RPA-Strecken mit ausreichender Länge pro Sichtfeld von 512 x 512 Pixeln (Pixelgröße = 154,6 nm). Mehrere Sichtfelder können im selben Experiment abgebildet werden, was bei Bedarf eine weitere Datenerfassung ermöglicht. Die Trakte müssen nicht gleich lang sein oder das Ende der DNA erreichen, um nützlich zu sein. Zum Beispiel kann der durchschnittliche Tether-Abstand für jedes Experiment bestimmt werden, indem die Länge der SYTOX-gefärbten DNA vor der Zugabe von CMG gemessen wird. Dies kann verwendet werden, um abzuschätzen, wie viel DNA für einen RPA-Trakt abgewickelt wurde (solange genügend DNA abgewickelt ist, um die Gabel sichtbar zu bewegen), indem die Entfernung von "μm zurückgelegt" in "kb abgewickelt" umgerechnet wird.
CMG zeigt eine Abwickelaktivität auf einer Vielzahl von DNA-Substraten, aber es ist wichtig, ein freies 3'-DNA-Ende auf einem PolyT-Lappen von mindestens 30 nt bereitzustellen, um den Fußabdruck von CMG10 aufzunehmen. Der Einbau mehrerer Biotin-Anteile an der Gabel sorgt für eine robuste Oberflächenanbindung. Der Rest des DNA-Substrats kann auf vielfältige Weise neu gestaltet werden, z. B. um verschiedene DNA-Sequenzen, Längen und chemische Modifikationen einzubeziehen. Die Konformation der DNA kann durch die Verwendung unterschiedlicher Konzentrationen von Magnesiumacetat verändert werden. Bei höheren Konzentrationen (≥10 mM) von Magnesiumacetat wird das RPA-beschichtete ssDNA-Filament verdichtet, was dazu führt, dass die DNA beim Abwickeln durch RPA-Bindung gezogen wird. Dies kann nützlich sein, da es verhindert, dass sich die DNA übermäßig bewegt, so dass die Position des CMG und des Abwickelverlaufs genauer gemessen werden kann. Bei niedrigen Konzentrationen (~3 mM) von Magnesiumacetat bleibt die RPA-ssDNA durchgehend entspannt.
Der beschriebene Einzelmolekül-Assay stellt eine Plattform dar, auf der aufgebaut und modifiziert werden kann, um weitere Aspekte der DNA-Replikation zu untersuchen. Während der DNA-Replikation fungiert CMG als Kern, um den sich das Replisom und seine Komponenten zusammensetzen. Daher können diesem Assay zusätzliche gereinigte Proteine hinzugefügt werden, einschließlich akzessorischer Faktoren wie TIMELESS, TIPIN und CLASPIN, um deren Wirkung auf die CMG-Dynamik zu untersuchen. Es wurde gezeigt, dass diese Proteine die Rate der Replikationsgabelnbeeinflussen 13, aber es ist nicht klar, wie sie die Rate der CMG-Abwicklung beeinflussen. Daher wäre es interessant zu untersuchen, wie sich verschiedene Replisomenproteine mit diesem Assay auf CMG auswirken. Die Zugabe von DNA-Polymerasen kann einen besseren Einblick in die DNA-Replikation geben, die über die alleinige DNA-Abwicklung hinausgeht, wie zuvor mit Hefeproteinen beschrieben14. Darüber hinaus kann die Aufreinigung von modifiziertem CMG ein besseres Verständnis dafür liefern, wie sich bestimmte Mutationen oder posttranslationale Modifikationen auf die Helikaseaktivität auswirken15,16. Darüber hinaus kann das Design verschiedener DNA-Substrate die Untersuchung der DNA-Abwicklung durch CMG unter einer Vielzahl von Bedingungen ermöglichen, die den Replikationsstress nachahmen17. Zu diesen Modifikationen gehören DNA-Hindernisse9, 18, Quervernetzungen zwischen den Strängen19, 20, 21 und Diskontinuitäten in den DNA-Strängen22.
Offenlegungen
Die Autoren haben keine konkurrierenden finanziellen Interessen oder andere Interessenkonflikte.
Danksagungen
Wir danken Gheorghe Chistol für die Bereitstellung des pGC261-Plasmids und der Francis Crick Institute Chemical Biology Facility für die Peptidsynthese und -markierung. Diese Arbeit wurde vom Francis Crick Institute finanziert, das eine Grundfinanzierung von Cancer Research UK, dem UK Medical Research Council und dem Wellcome Trust (CC2133) erhält.
Materialien
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
Referenzen
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819 (2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159 (2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713 (2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331 (2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten