Method Article
Quantifizierung von Adeno-assoziierten viralen Genomen in gereinigten Vektorproben durch digitale Tröpfchenpolymerase-Kettenreaktion
In diesem Artikel
Zusammenfassung
Die genaue Quantifizierung von Vektorgenomkopien des Adeno-assoziierten Virus (AAV) ist von entscheidender Bedeutung, aber ein standardisiertes Protokoll muss noch etabliert werden. Dieses Protokoll beschreibt eine validierte Methode zur Aufbereitung gereinigter AAV-Proben und zur Durchführung einer digitalen Tröpfchenpolymerase-Kettenreaktion (dd_PCR), um den viralen Genomtiter zuverlässig zu quantifizieren.
Zusammenfassung
Das Adeno-assoziierte Virus (AAV) ist ein nicht-pathogenes Virus, das als Transportvehikel für den Transfer therapeutischer Gene in Patienten verwendet wird. Die genaue Quantifizierung der Kopienzahl des AAV-Genoms in Vektorpräparaten ist für die Optimierung des Bioprozesses und die Dosisberechnung sowohl in präklinischen als auch in klinischen Studien mit AAV-basierten Gentherapieprodukten unerlässlich. Derzeit fehlt ein Konsensprotokoll für die Titration des viralen AAV-Genoms. In dieser Arbeit stellen wir ein digitales Tröpfchen-PCR-Protokoll (dd_PCR) zur Quantifizierung viraler Genome in gereinigten Vektorproben vor. Die Proben werden mit DNase I behandelt, um unverpackte Schadstoff-DNA zu eliminieren. DNase-behandelte Proben werden dann mit einem geeigneten Primer-Sonden-Set (entsprechend dem Ziel-AAV-Genom entwickelt) und PCR-Reagenzien gemischt und dann in einen Tröpfchengenerator geladen. Die vorbereiteten Tröpfchen werden in eine PCR-Platte überführt, wo die PCR-Amplifikation durchgeführt und analysiert wird. Der Titer des viralen Genoms wird auf der Grundlage der Konzentration (Kopien/μl) unter Berücksichtigung der Probenverdünnungen berechnet. Eine erfolgreiche Messung zeigt eine klare Trennung der positiven und negativen Tröpfchenwolken, hat mindestens 10.000 akzeptierte Tröpfchen, zeigt einen Wert zwischen 10 Kopien/μL und 10.000 Kopien/μL und hat einen Variationskoeffizienten (CV) zwischen den Wiederholungen von weniger als 20%. Eine zuverlässige Titration des viralen Genoms wird die Entwicklung sicherer und wirksamer AAV-basierter Gentherapieprodukte unterstützen.
Einleitung
Die Gentherapie ist eine therapeutische Modalität, die häufig zur Behandlung genetischer Störungen eingesetzt wird. Das Design einer bestimmten Gentherapie ist spezifisch für die damit verbundene Pathologie der Zielindikation, aber alle Gentherapien beinhalten die intrazelluläre Abgabe von genetischem Material an die Zielzellen, um eine therapeutische Wirkung hervorzurufen1. Die Gentherapie kann weiter in mehrere Kategorien eingeteilt werden, darunter Genersatz bei Funktionsverlustmutationen, Gen-Silencing bei Funktionsgain-of-Function-Anomalien und Gen-Editing-Techniken. Unabhängig von der spezifischen Strategie muss das therapeutische Nukleinsäurematerial (das sogenannte Transgen) in einen Vektor verpackt werden, um eine gezielte intrazelluläre Verabreichung zu erreichen2.
Obwohl eine Vielzahl von viralen und nicht-viralen Vektorsystemen für die Entwicklung von Gentherapien zur Verfügung steht, werden aufgrund des breiten viralen Tropismus und der geringen Immunogenität, die mit dieser Gruppe von Viren verbunden sind, häufig Adeno-assoziierte Viren (AAVs) gewählt 1,2. Bisher haben sieben Gentherapien, die AAV für die therapeutische Genverabreichung nutzen, entweder von der Europäischen Arzneimittelagentur (EMA) oder der Food and Drug Administration (FDA) zugelassen und zielen auf Krankheiten ab, die von Hämophilie (z. B. Roctavian) bis hin zu spinaler Muskelatrophie (z. B. Zolgensma) reichen3.
Die Produktion von AAV-basierten Gentherapien beruht auf dem Verständnis des Wildtyp-AAV selbst. AAV ist ein kleines DNA-Virus innerhalb der Familie der Parvoviridae, das aus 13 Hauptserotypen (AAV1-13)3 besteht. Das AAV-Genom besteht aus einem ~4,7 kb großen, einzelsträngigen DNA-Molekül, das zwei offene Hauptleserahmen (ORFs) enthält, die für die Genomreplikation, die Kapsidassemblierung und die Verpackung (rep, cap) kodieren. Das virale Genom wird sowohl am 5'- als auch am 3'-Ende von palindromischen Nukleotidsequenzen flankiert, die als inverted terminal repeats (ITRs) bezeichnet werden. Diese ITRs bilden haarnadelartige Strukturen, die eine entscheidende Rolle bei der Genomreplikation und der Verpackung von de novo viralen Genomen in neu synthetisierte virale Kapside spielen. AAV ist ein helferabhängiges Virus und benötigt daher die Expression von Zusatzgenen aus anderen Viren, wie dem Herpes-simplex-Virus (HSV) oder dem Adenovirus (AdV), um replikationskompetent zu werden1.
Um AAVs herzustellen, wird ein geeignetes zellbasiertes Expressionssystem eingesetzt, um die Expression der viralen Kapsidproteine und die anschließende Assemblierung in de novo virale Partikel zu erleichtern, gefolgt von der Verkapselung eines ITR-flankierten selektierten Transgens (auch als Vektorgenom bezeichnet). Bei diesem Verfahren wird üblicherweise ein Dreifachplasmidsystem verwendet, das (1) ein Plasmid umfasst, das Helfergene enthält, die von einem Helfervirus abgeleitet sind, (2) ein Plasmid, das für die essentiellen viralen Elemente (rep/cap) kodiert, und (3) ein Plasmid, das die therapeutische Expressionskassette trägt (allgemein als Transferplasmid bezeichnet)4. Das einzigartige Vorhandensein von Inverted Terminal Repeat (ITR)-Verpackungssignalen, die die therapeutische Expressionskassette im Transferplasmid flankieren, gewährleistet eine spezifische Verpackung des Transgens, während die auf den anderen Plasmiden vorhandenen viralen Gene meist ausgeschlossen werden. Die Co-Transfektion dieses Drei-Plasmid-Systems in eine zellbasierte Expressionsplattform (typischerweise HEK293T Zellen) führt zur Produktion von transduktionskompetenten, replikationsdefizienten Viruspartikeln, die für den Einsatz in Gentherapieanwendungen geeignet sind 3,4.
Es gibt eine Reihe von kritischen Qualitätsmerkmalen (CQAs) im Zusammenhang mit der Herstellung von AAV-basierten Gentherapien, die bewertet werden müssen, um die Wirksamkeit, Reinheit und Sicherheit des beabsichtigten Arzneimittels zu gewährleisten4. Zu diesen CQAs gehören Virustiter, Kapsidinhalt und Aggregation. Der Virustiter selbst ist eine Kombination aus der Anzahl der Viruspartikel (Kapsidtiter) und der Anzahl der Vektorgenome (Vektorgenomtiter), die in einem bestimmten Präparat vorhanden sind. Im Idealfall sollte das Verhältnis zwischen diesen beiden Titern 1:1 betragen, da jedes Viruspartikel ein Vektorgenom enthalten sollte, aber Ineffizienzen bei der Verpackung des Vektorgenoms während der Biosynthese führen zur Koproduktion von leeren oder teilweise gefüllten Kapsiden (solche, die partielle Vektorgenomsequenzen und/oder Nicht-Vektorgenomsequenzen enthalten)5. Das Vorhandensein solcher Verunreinigungen kann möglicherweise ungerechtfertigte Immunreaktionen hervorrufen und um Vektorbindungsstellen konkurrieren, wodurch das Risiko einer Immuntoxizität erhöht und die Transduktionsrate von Vollkapsiden verringertwird 6. Eine genaue Quantifizierung des AAV-Genoms ist daher für die Bestimmung des Virustiters und des Kapsidgehalts unerlässlich. Dies wirkt sich sowohl auf die Grundlagenforschung als auch auf die Gentherapie-Industrie aus, die eine genaue Dosierung erfordert, um sowohl die Sicherheit als auch die Wirksamkeit von Arzneimitteln zu erhalten.
Die digitale Tröpfchenpolymerase-Kettenreaktion (dd_PCR) ist eng mit der Quantifizierung des Virustiters verbunden, da sie zur Bestimmung der Anzahl der in einem bestimmten Präparat vorhandenen Vektorgenome verwendet werden kann7. Die digitale PCR selbst wurde erstmals in den 1990er Jahren eingeführt 8,9 und dd_PCR ist eine Verbesserung dieser Technologie, die eine Probenverarbeitung mit hohem Durchsatz ermöglicht10,11. In dd_PCR wird eine 20-μl-Real-Time-PCR-Reaktion in etwa 20.000 ölumhüllte Tröpfchen aufgeteilt, was bei Unterbringung auf einer Standardplatte mit 96 Vertiefungen bis zu 96 solcher Reaktionen ergibt. Im Vergleich zur herkömmlichen quantitativen PCR (qPCR) bietet dd_PCR mehrere Vorteile, darunter eine erhöhte Sensitivität, eine höhere Präzision und eine direktere und absolutere Quantifizierung der Zielsequenzen, ohne dass Standardkurven erforderlich sind. Darüber hinaus reduziert das hohe Maß an Partitionierung in dd_PCR den Einfluss von PCR-Inhibitoren und minimiert das Potenzial für Verzerrungen durch bevorzugte Amplifikation bestimmter Templates, was es zu einer attraktiven Option für die analytische Messung der Vektorgenom-Titration macht.
Protokoll
Das hier beschriebene Protokoll ist darauf ausgelegt, den viralen Genomtiter eines gereinigten, eigens hergestellten AAV9-Vektors mit grün fluoreszierendem Protein (GFP) als Transgen12 mit hoher Präzision zu quantifizieren (Tabelle 1). Dieses Protokoll ist jedoch auf jeden AAV-Serotyp und jedes Vektorgenomdesign anwendbar, vorausgesetzt, dass die Primer-/Sondensätze so konzipiert sind, dass sie auf das spezifische Vektorgenom von Interesse abzielen. Einzelheiten zu den in dieser Studie verwendeten Reagenzien, Primern, Sonden und Geräten sind in der Materialtabelle aufgeführt.
1. Herstellung von Stammlösungen
HINWEIS: Bereiten Sie alle Stammlösungen, die für die Probenverdünnung erforderlich sind, an einer PCR-Workstation vor, um eine Kontamination durch Fremd-DNA zu vermeiden.
- Bereiten Sie einen 10x PCR-Assay-Puffer vor, der aus 500 mM KCl, 100 mM Tris-HCl, 15 mM MgCl2 und 0,01 % (w/v) BSA besteht. Messen Sie den pH-Wert der Lösung und stellen Sie ihn auf 8,3 ein.
HINWEIS: Diese Lösung kann bis zu 1 Monat bei Raumtemperatur aufbewahrt werden. - Bereiten Sie den AAV-Verdünnungspuffer vor, der aus 1x PCR-Assay-Puffer, 0,2 ng/μl gescherter Lachsspermien-DNA (sss) und 0,1 % pluronischem F-68 besteht. Stellen Sie diese Lösung bei jedem Assay frisch her und halten Sie sie bei 4 °C.
HINWEIS: Wenn pluronic F-68 nicht verfügbar ist, kann eine Alternative wie Poloxamer 188 nichtionisches Tensid verwendet werden.
2. Vorbereitung der Proben - DNase I-Behandlung und serielle Verdünnung
HINWEIS: Um DNA-Kontaminanten zu entfernen, die die Genauigkeit der Vektorgenom-Titration verringern könnten, kann die in der AAV-Probe vorhandene freie DNA (einschließlich verbleibender Plasmid-DNA oder nicht verkapselter viraler Genome) vor der PCR-Amplifikation mit DNase entfernt werden. Dies ist möglich, da die Vektorgenome in den AAV-Kapsiden eingekapselt sind und daher erst im Denaturierungsschritt der PCR-Reaktion selbst zugänglich sind (siehe Schritt 5). Da die Vektorgenomnummer in einer bestimmten Probe unbekannt ist, ist es außerdem notwendig, eine serielle Verdünnung der Proben durchzuführen, um sicherzustellen, dass die Messungen innerhalb der oberen und unteren nachweisbaren Grenzen bleiben. Führen Sie alle Probenmanipulationen an einer PCR-Workstation durch. Bewahren Sie alle Proben auf Eis auf, sofern nicht anders angegeben.
- Mischen Sie die für die Analyse ausgewählte AAV-Probe, indem Sie sie kurz vortexen und dann zentrifugieren, um sicherzustellen, dass die gesamte Flüssigkeit am Boden des Röhrchens mit der Probe verbleibt.
- Für die DNase-Behandlung geben Sie 45 μl einer Lösung, die 1x DNase I-Puffer, 0,1 % pluronic F-68 und 0,04 U/μl DNase I in DNase-freiem Wasser enthält, in ein 0,2 mL PCR 8-Röhrchen-Streifenröhrchen.
- 5 μl der Probe in das Röhrchen mit der DNAse-Lösung überführen. Wirbeln Sie die Proben vor und zentrifugieren Sie kurz, um sicherzustellen, dass die gesamte Flüssigkeit am Boden bleibt.
- In einem Thermocycler werden die Proben 1 h lang bei 37 °C inkubiert und dann auf 4 °C abgekühlt. So schnell wie möglich auf Eis legen, nachdem 4 °C erreicht sind.
- Nehmen Sie unter Verwendung eines frischen 0,2-ml-PCR-Streifens mit 8 Röhrchen geeignete Verdünnungen der DNase-behandelten Probe in AAV-Verdünnungspuffer vor. Die empfohlenen Probenverdünnungen hängen vom erwarteten vg-Titer ab (Tabelle 2), zielen jedoch auf mindestens 2 verschiedene Verdünnungen pro Probe ab und führen jede Verdünnung in doppelter Ausführung durch. Stellen Sie sicher, dass die Proben 5 s lang rigoros (2.000-3.000 U/min) vortexen, bevor Sie Volumen herausnehmen.
- Nach der seriellen Verdünnung die Proben vortexen und zentrifugieren, um sicherzustellen, dass die gesamte Flüssigkeit am Boden der Röhrchen verbleibt.
- Dazu gehören eine Positivkontrolle, von der der vg-Titer bekannt ist, und eine Negativkontrolle (nur AAV-Verdünnungspuffer), die auch als No-Template-Kontrolle (NTC) bezeichnet wird.
HINWEIS: Bei der Positivkontrolle kann es sich um ein intern hergestelltes AAV oder ein kommerzielles AAV-Referenzmaterial handeln, das die Zielsequenz der Primer/Sonde enthält.
3. Zubereitung dd_PCR Mastermixes
HINWEIS: Wählen Sie eine geeignete Zielsequenz innerhalb des Vektorgenoms aus und entwerfen Sie einen Forward-Primer, einen Reverse-Primer und Hydrolysesonden, die mit FAM- oder HEX-Reporterfluorophoren gemäß den veröffentlichten Richtlinien13 markiert sind. Primer, die auf das Transgen abzielen, werden gegenüber Primern bevorzugt, die auf die ITRs abzielen, da das Transgen spezifisch für das Design des Vektorgenoms ist und die Sekundärstruktur, die mit der Haarnadelbildung der ITRs verbunden ist, eine effiziente Primerbindung behindern kann. Darüber hinaus könnten ITR-Primer AAV-Titer überschätzen, wenn teilweise gefüllte Kapside vorhanden sind, die verkürzte Vektorgenomfragmente enthalten, die immer noch eine der ITR-Sequenzen14 enthalten. Bereiten Sie den dd_PCR Mastermix an einem eigenen, separaten Arbeitsplatz (Pre-PCR-Raum) vor. Dieser Raum sollte von dem Raum getrennt sein, in dem die Proben vorbereitet werden, um Kreuzkontaminationen zu vermeiden.
- Lassen Sie den Primer, die Sonde und die dd_PCR Supermix-Reagenzien Raumtemperatur (20-25 °C) erreichen. Anschließend alle Reagenzien vortexen und die Röhrchen kurz zentrifugieren, um die gesamte Flüssigkeit am Boden des Röhrchens zu sammeln.
- Bereiten Sie ein Volumen dd_PCR Mastermixes in einem separaten Mikrozentrifugenröhrchen entsprechend der erforderlichen Anzahl von Reaktionen vor, indem Sie die Vorwärts- und Rückwärtsprimer, die Sonde dd_PCR Supermix und das DNase-freie Wasser gemäß den in Tabelle 3 beschriebenen Volumina und Konzentrationen kombinieren.
- Den Mastermix vertexen und dann das Röhrchen kurz zentrifugieren, um sicherzustellen, dass die gesamte Flüssigkeit am Boden des Röhrchens verbleibt.
HINWEIS: Bereiten Sie immer mehr dd_PCR Mastermix-Volumen als erforderlich (n + 1) vor, um den Verlust des Pipettiervolumens auszugleichen.
- Den Mastermix vertexen und dann das Röhrchen kurz zentrifugieren, um sicherzustellen, dass die gesamte Flüssigkeit am Boden des Röhrchens verbleibt.
- Übertragen Sie 19,8 μl des Mastermixes in jedes Röhrchen auf einen frischen 0,2 mL PCR 8-Röhrchen-Streifen.
4. Tröpfchen-Erzeugung
HINWEIS: Proben und dd_PCR Mastermix werden separat gemischt, bevor sie in eine tröpfchenerzeugende Kartusche geladen werden. Führen Sie alle Manipulationen an einem PCR-Arbeitsplatz durch, vorzugsweise an einem anderen als dem, an dem die Proben vorbereitet wurden, um eine Kontamination der Amplikons zu vermeiden. Alternativ können Sie den PCR-Arbeitsplatz nach der Probenvorbereitung gründlich reinigen.
- Übertragen Sie 2,2 μl der verdünnten Probe in das 19,8 μl Volumen dd_PCR Mastermixes in jedes Röhrchen des PCR-8-Röhrchen-Streifens und mischen Sie es gut. Vortexen und kurz zentrifugieren, um sicherzustellen, dass die gesamte Flüssigkeit am Boden bleibt.
- Übertragen Sie 20 μl dieser Lösung in die Vertiefungen in der mittleren "Proben"-Reihe einer tröpfchenerzeugenden Kartusche. Vermeiden Sie Blasen in der tröpfchenerzeugenden Kartusche; Dies kann dazu führen, dass das Gerät Fehler ausgibt und Tröpfchen nicht generiert werden können.
HINWEIS: Da die Kartuschen ebenso wie die PCR-Röhrchenstreifen im 8-Well-Format vorliegen, wird empfohlen, eine 8-Kanal-Pipette zu verwenden. - Übertragen Sie 60 μl des Tröpfchenerzeugungsöls in die Vertiefungen, die sich in der unteren "Öl"-Reihe der Tröpfchenerzeugungspatrone befinden.
HINWEIS: Lassen Sie niemals eine Vertiefung der tröpfchenerzeugenden Kartusche leer. Füllen Sie die leere Probenvertiefung mit dd_PCR Puffersteuerung für Sonden (mit dem entsprechenden oben beschriebenen Volumen). - Setzen Sie eine Gummidichtung über die Tröpfchenpatrone und setzen Sie sie in den Tröpfchengenerator ein.
- Nachdem die Tröpfchen erzeugt wurden, verwenden Sie eine 8-Kanal-Pipette, um die 42,5-μl-Lösung langsam vom dd_PCR-Chip auf eine Multi-Well-PCR-Platte zu übertragen.
HINWEIS: Pipettieren Sie langsam und in einem 45-Grad-Winkel, um Tröpfchenscherungen in der Flüssigkeit beim Pipettieren zu vermeiden. Überprüfen Sie die Platte visuell, so dass alle Vertiefungen mit der gleichen Menge Flüssigkeit gefüllt sind und die Tröpfchen als undurchsichtige Schicht in der Vertiefung sichtbar sind. - Versiegeln Sie die Platte mit einer Alufolienabdeckung in einer Heißsiegelmaschine für 5 s bei 180 °C.
HINWEIS: Überprüfen Sie visuell, ob alle Vertiefungen versiegelt sind. Wenn nicht, wiederholen Sie den Vorgang weitere 5 s bei 180 °C. Die versiegelte Platte kann bis zu 4 h bei 4 °C aufbewahrt werden, bevor dd_PCR in einem Thermocycler verstärkt wird.
5. dd_PCR Verstärkung
HINWEIS: Der Thermocycler sollte in einem vom Prä-PCR-Raum getrennten Raum aufgestellt werden, um die Prä-PCR-Aktivitäten räumlich von der PCR-Amplifikation zu trennen und falsch-positive Ergebnisse aufgrund einer Amplikonkontamination zu vermeiden.
- Legen Sie die Platte für die PCR-Amplifikation in einen Thermocycler (mit einem 96-Deep-Well-Reaktionsmodul) und verschließen Sie sie fest.
- Konfigurieren Sie den Thermocycler so, dass er unter den in Tabelle 4 beschriebenen Bedingungen und bei einem Reaktionsvolumen von 40 μl läuft. Nehmen Sie einen Vorheizschritt in die Programmeinstellungen auf und stellen Sie sicher, dass der Deckel auf 99 °C erhitzt wird.
- Wenn das PCR-Programm beendet ist, halten Sie die Platte mindestens 15 Minuten lang bei 4 °C, um sicherzustellen, dass die Platte vollständig abgekühlt ist.
HINWEIS: Die Platte kann 48-72 h bei 4 °C aufbewahrt werden, bevor die Tröpfchen abgelesen werden müssen.
6. Tröpfchen-Ablesung
- Legen Sie die Platte in ein Tröpfchenlesegerät, geben Sie die folgenden Informationen in die Systemsoftware ein und setzen Sie den Messvorgang fort.
HINWEIS: Versuchstyp = Direkte Quantifizierung; Supermix = dd_PCR Supermix für Sonden (kein untUTP); Assay-Typ = einzelnes Ziel pro Kanal; Zielinfo, Signalkanal 1 = FAM-Kanal oder HEX-Kanal je nach verwendeter Sonde. - Versehen Sie die Proben mit Anmerkungen, so dass die Position jeder Probe anhand der Datendatei effektiv bestimmt und identifiziert werden kann.
7. Datenanalyse
- Führen Sie die Datenanalyse mit der kompatiblen Software durch, die vom Hersteller des Tröpfchenlesers zur Verfügung gestellt wird.
- Führen Sie einen Systemeignungstest durch, um die Gesamtleistung (und damit die Zuverlässigkeit) des Assays zu bewerten.
- Überprüfen Sie die Ereignisanzahl (Anzahl der Tröpfchen), die für jede Vertiefung auf der PCR-Platte gemessen wurde. Im Idealfall liegt die Anzahl der Ereignisse zwischen 15.000 und 20.000. Wenn die Ereignisanzahl 10.000 < beträgt, muss dieser Wert aus der Analyse ausgeschlossen werden.
- Werten Sie das 1D- oder 2D-Amplitudendiagramm aus, und prüfen Sie, ob Tröpfchenregen vorhanden ist.
- Gehen Sie zur Registerkarte Datentabelle und exportieren Sie die Daten in eine Excel-Datei.
- Berechnen Sie den vg-Titer mit den folgenden Schritten:
- Der Arbeitsbereich von dd_PCR beträgt 10-10 000 Kopien/μl. Alle Werte <10 Kopien/μl sind von der Analyse auszuschließen. Der empfohlene Bereich für die dd_PCR Ausrüstung, die hierin verwendet wird, beträgt 25-5000 Kopien/μL.
HINWEIS: Die Datenanalysesoftware liefert eine Vektorgenomnummer in Übereinstimmung mit dem PCR-Reaktionsvolumen (40 μl), dies berücksichtigt jedoch nicht die Verdünnungsfaktoren, die während der Probenvorbereitung angewendet werden. - Berechnen Sie den vg-Titer der Probe pro Volumen (typischerweise ausgedrückt als vg/ml), indem die verschiedenen Verdünnungsfaktoren während der Probenvorbereitung unter Verwendung der folgenden Beziehung berücksichtigt werden:
HINWEIS: Konzentration (aus der Datenanalyse erhaltener Wert) x 10 (DNase I-Verdünnung vor der Behandlung) x 10 (PCR-Mastermix-Verdünnung) x 1000 (μl bis ml) Verdünnung der ausgewählten Probe (in AAV-Verdünnungspuffer - z. B. Tabelle 2). - Berechnen Sie den Variationskoeffizienten (%CV) zwischen den Replikaten und zwischen den verschiedenen Verdünnungen ein und derselben Probe.
HINWEIS: %CV wird berechnet, indem die Standardabweichung durch den Mittelwert dividiert wird. Beträgt der %CV zwischen den verschiedenen Verdünnungen >20 %, sollte der Wert nicht als genau angesehen werden. - Überprüfen Sie die Werte der Negativ- und Positivkontrolle. Die Negativkontrolle sollte weniger als 5 Kopien/μl betragen.
- Der Arbeitsbereich von dd_PCR beträgt 10-10 000 Kopien/μl. Alle Werte <10 Kopien/μl sind von der Analyse auszuschließen. Der empfohlene Bereich für die dd_PCR Ausrüstung, die hierin verwendet wird, beträgt 25-5000 Kopien/μL.
Ergebnisse
Die Ergebnisse können mit entsprechender Software visualisiert werden. Für jedes Well zeigt das 1D-Amplitudendiagramm alle Tröpfchen relativ zu ihrer Amplitude an. Es wird eine klare Trennung zwischen positiven und negativen Tröpfchen erwartet. Wenn mehr als 10 % der gesamten Tröpfchen zwischen der positiven und der negativen Tröpfchenwolke gefunden werden (ein Phänomen, das als Tröpfchenregen bezeichnet wird), muss die Probe erneut gemessen werden (siehe Abbildung 1). Weitere Informationen zum Thema Tröpfchenregen finden Sie in der Diskussion.
Es kann eine Datentabelle erstellt werden, in der alle aufgezeichneten Informationen zusammengefasst werden, einschließlich des Probennamens, der Anzahl der akzeptierten Tröpfchen (Ereigniszählungen) und der Konzentration (Kopien/μl). Im Idealfall sollte die Ereignisanzahl zwischen 15.000 und 20.000 akzeptierten Droplets liegen. Wenn die Ereignisanzahl für ein Bohrloch unter 10.000 liegt, sollte der Datenpunkt aus der Analyse ausgeschlossen werden. Ein Beispiel für die Ausgabedaten ist in Tabelle 5 enthalten.
Der vg-Titer kann auf der Grundlage der Konzentration (Kopien/μL) unter Berücksichtigung der Probenverdünnungen berechnet werden. Der Arbeitsbereich von dd_PCR beträgt 10-10.000 Kopien/μl. Werte unter 10 Kopien/μl sollten von der Analyse ausgeschlossen werden. Es sollten der vg-Titer für die Proben sowie die Positiv- und Negativkontrollen berechnet werden. Der gemessene Wert der Positivkontrolle sollte einen Variationskoeffizienten (%CV) von weniger als 20 % bezogen auf den theoretischen Wert haben. Die Negativkontrolle sollte weniger als 5 Kopien/μl betragen. Zusätzlich sollte der %CV zwischen Wiederholungen und verschiedenen Verdünnungen für jede Probe berechnet werden. Wenn der %CV zwischen den verschiedenen Verdünnungen 20 % überschreitet, kann der Wert als ungenau angesehen werden, und die Probe muss möglicherweise erneut gemessen werden.
Eine erfolgreiche Messung zeichnet sich durch eine klare Trennung der positiven und negativen Tröpfchenwolke, mindestens 10.000 akzeptierte Tröpfchen, einen Wert zwischen 10 Kopien/μL und 10.000 Kopien/μL und einen %CV zwischen den Wiederholungen von weniger als 20% aus.
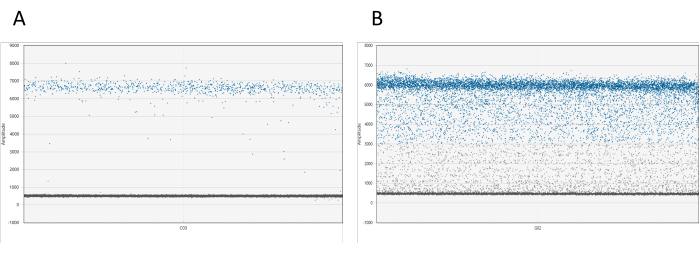
Abbildung 1: Visualisierung von dd_PCR Tröpfchen. (A) Das 1D-Amplitudendiagramm zeigt eine klare Trennung von positiven und negativen Tröpfchen, was auf eine erfolgreiche Tröpfchenpartitionierung hinweist. (B) Das Diagramm zeigt eine schlechte Trennung von positiven und negativen Tröpfchen, die als Tröpfchenregen bezeichnet wird, was auf eine suboptimale Aufteilung oder mögliche Probleme mit dem Assay hindeutet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
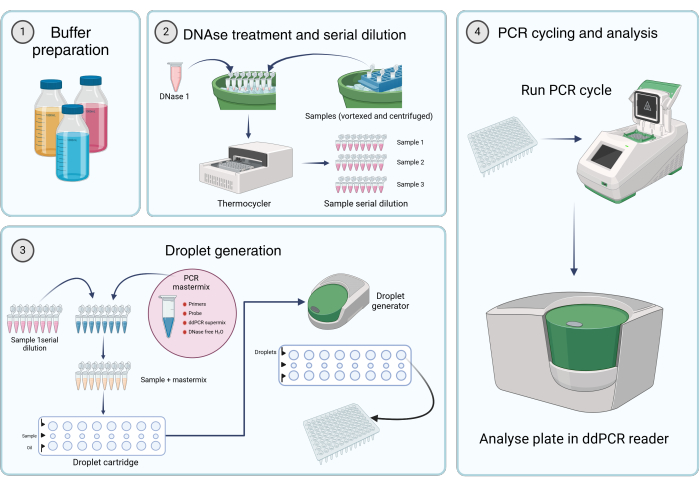
Abbildung 2: Arbeitsablauf zur Quantifizierung viraler Genome mittels digitaler Tröpfchen-PCR. (1) Puffer, Reagenzien und Lösungen werden gemäß den Anweisungen des Herstellers oder Schritt 1 des Protokolls (Herstellung von Stammlösungen) hergestellt. (2) 45 μl der DNase I-Lösung werden in jedes Röhrchen eines 8-Röhrchen-PCR-Streifens aliquotiert. Nach dem Vortexen und kurzen Zentrifugieren der Proben werden 5 μL der Probe in eines der Röhrchen gegeben. Die DNase I und der probenhaltige PCR-Streifen mit acht Röhrchen werden in einem Thermocycler für 1 h bei 37 °C inkubiert, gefolgt von seriellen Verdünnungen der Proben. (3) Der PCR-Mastermix wird wie beschrieben hergestellt und 19,8 μl werden in jedes Röhrchen eines 8-Röhrchen-PCR-Streifens aliquotiert. Proben-Reihenverdünnungen aus Schritt 2 werden dem Mastermix zugesetzt. 20 μL des Mastermix plus
Die Probenlösung wird in die mittlere Reihe der Kartusche geladen, und 60 μl Öl zur Tröpfchenerzeugung werden in die unteren Vertiefungen einer tröpfchenerzeugenden Kartusche geleitet. Die Kartusche wird dann in den Tröpfchengenerator eingelegt und läuft unter festgelegten Bedingungen. Nach der Tröpfchenerzeugung werden 42,5 μl aus der oberen Reihe der Kartusche auf eine Multi-Well-PCR-Platte übertragen. (4) Die PCR-Platte wird in einen PCR-Thermocycler eingelegt und gemäß den bereitgestellten Bedingungen betrieben. Die Analyse der Platte erfolgt mit einem dd_PCR Reader. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Genauigkeitsdaten. Diese Tabelle enthält Präzisionsdaten von vier Sätzen von Qualitätskontrollproben (QC) mit jeweils fünf Konzentrationen (QC1-QC5). Jede QC wurde fünfmal gemessen. Dargestellt ist der Variationskoeffizient (%CV) zwischen verschiedenen Durchläufen, der die Wiederholgenauigkeit (A) darstellt, und zwischen verschiedenen Probensätzen, der die Zwischengenauigkeit (B) darstellt. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Verdünnungen der Proben. Die Tabelle zeigt die empfohlenen Probenverdünnungen basierend auf dem erwarteten viralen Genomtiter (vg/mL). Es werden insgesamt drei Verdünnungen empfohlen, um sicherzustellen, dass mindestens zwei Werte innerhalb des Arbeitsbereichs des Assays liegen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: dd_PCR Mastermix-Komposition. In dieser Tabelle ist die Zusammensetzung des dd_PCR Mastermixes aufgeführt, der einen Vorwärts- und Rückwärtsprimer (909 nM), eine Sonde (227 nM) und dd_PCR Supermix für Sonden (ohne dUTP, 1x) umfasst. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 4: Bedingungen für Thermozyklen. In der Tabelle ist das empfohlene PCR-Programm aufgeführt, das Folgendes umfasst: (1) eine 10-minütige Inkubation bei 95 °C zur Kapsidaufschluss und Enzymaktivierung, (2) 40 Zyklen à 30 s bei 94 °C für die DNA-Denaturierung und 1 min bei 60 °C für das Glühen und die Verlängerung, (3) eine 10-minütige Inkubation bei 98 °C zur Deaktivierung des Enzyms, und (4) bei 4 °C halten. Die Glühtemperatur kann je nach verwendetem Primer-/Sondensatz optimiert werden müssen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 5: Beispiel für Ausgabedaten aus einem tatsächlichen AAV-Lauf. Die Tabelle enthält ein Beispiel für Ausgabedaten aus einem tatsächlichen AAV-Lauf, bei dem eine Probe in zwei verschiedenen Verdünnungen gemessen wurde, jeweils in doppelter Hinsicht. Der Titer des viralen Genoms (vg) (in vg/ml) wird nach folgender Formel berechnet: 10 (DNase I-Vorbehandlung) x 10 (Verdünnung im Mastermix) x 1.000 (μl zu ml) x Verdünnung in AAV-Verdünnungspuffer. STDEV stellt die Standardabweichung dar, und CV gibt den Variationskoeffizienten an. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Die genaue Quantifizierung von AAV-Vektorgenomkopien in Vektorpräparaten ist für die Entwicklung von AAV-basierten Gentherapieprodukten unerlässlich. Es gibt mehrere Methoden zur Bestimmung des vg-Titers, wobei die quantitative PCR (qPCR) und die dd_PCR die beiden am weitesten verbreiteten und akzeptierten Techniken sind. dd_PCR wird aufgrund ihrer Unabhängigkeit von der Amplifikationseffizienz, der höheren Präzision und der größeren Robustheit oft der qPCR vorgezogen15. In der Literatur sind zahlreiche Protokolle für die Vektorgenomtitration durch dd_PCR verfügbar, jedes mit seinen eigenen Probenvorbereitungsmethoden 15,16,17. Es fehlt jedoch an einem qualifizierten Konsensprotokoll. In diesem Artikel wird ein validiertes, zweckmäßiges dd_PCR Protokoll für die Quantifizierung von AAV-Vektorgenomen in gereinigten Vektorproben vorgestellt (Abbildung 2).
Bei der Durchführung dieses Protokolls ist die genaue Beachtung der Probenhandhabung von entscheidender Bedeutung. Kreuzkontaminationen können eine große Herausforderung bei der genauen Beurteilung von vg-Titern darstellen. Daher werden die Proben am besten unter einer PCR-Workstation verarbeitet, um eine Kontamination mit fremder DNA zu vermeiden. Darüber hinaus ist die korrekte Durchführung der DNase-Inkubation von entscheidender Bedeutung, um unverpackte Kontaminanten-DNA zu eliminieren, ohne das Kapsid zu stören und möglicherweise das verpackte Vektorgenom zu verdauen. Viele Protokolle umfassen Hitzeinaktivierung und Proteinase-K-Behandlung 16,17,18. Bei der internen Methodenentwicklung wurde jedoch festgestellt, dass eine übermäßige Erhitzung der VG-Titration abträglich war und eine Proteinase-K-Behandlung unnötig war (Daten nicht gezeigt).
Da dd_PCR für die Quantifizierung viraler Genome immer beliebter wird, haben die Hersteller detaillierte Anwendungsleitfäden mit Richtlinien zur Gestaltung und Optimierung dd_PCR Assays veröffentlicht19. Informationen zur Fehlerbehebung sind ebenfalls verfügbar. In der Praxis gehören zu den häufigsten Problemen, wenn ein Assay korrekt konzipiert ist, Tröpfchenregen und eine geringe Tröpfchenzahl. Tröpfchenregen wird oft durch eine schlechte Zugänglichkeit des Primers/der Sonden zum Amplikon verursacht, aber es gibt mehrere Strategien, die dieses Problem lösen können. So kann beispielsweise ein Temperaturgradientenexperiment helfen, die optimale Glühtemperatur zu ermitteln. Darüber hinaus kann die Verdünnung von Proben, um die DNA-Menge zu senken (empfohlen auf weniger als 66 ng) oder die Durchführung eines Restriktionsverdaus mit spezifischen Enzymen, die außerhalb der Amplikonregion schneiden, die Zugänglichkeit der Matrize verbessern. Eine Empfehlung von 10 U Restriktionsenzym pro μg DNA ist in der Regel wirksam. Das Problem der niedrigen Tröpfchenzahl (weniger als 10.000) ist häufig auf ein schlechtes Pipettieren der Probe und des Öls in den Kartuschen zurückzuführen. Es wird empfohlen, langsam mit geeigneten Spitzen zu pipettieren, um ein Scheren der Tröpfchen zu vermeiden. Obwohl der dd_PCR Assay robust ist, ist eine Einschränkung die lange Zeit bis zum Ergebnis. Der Prozess von der Probenbehandlung bis zur Tröpfchenablesung dauert mehrere Stunden, was ein Nachteil sein kann, wenn schnell Ergebnisse benötigt werden.
Eine genaue Quantifizierung von AAV-Genomkopien in Vektorpräparaten ist in allen Phasen des Lebenszyklus der AAV-Gentherapie erforderlich. Insbesondere ist es wichtig für die Optimierung von Produktions- und Reinigungsprozessen, die Durchführung präklinischer Studien und die Bestimmung klinischer Dosierungen für AAV-basierte Gentherapieprodukte. Das hier vorgestellte dd_PCR Protokoll ist breit anwendbar und kann auf gereinigte AAV-Produkte mit unterschiedlichen Serotypen und Transgenen angewendet werden.
Offenlegungen
IVH, BM und EH sind Mitbegründer von Tavira Therapeutics. Die anderen Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Diese Forschung wurde durch den Zuschuss "Flanderner Resilienz" der flämischen Regierung ermöglicht, der aus der "Europäischen Aufbau- und Resilienzfazilität" (VV021/13) stammt. Abbildung 2 wird mit Biorender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 8-channel pipette 10 µL | Eppendorf | 3,12,50,00,010 | |
| 8-channel pipette 200 µL | Eppendorf | 3,12,50,00,036 | |
| 8-channel pipette 300 µL | Eppendorf | 3,12,50,00,052 | |
| 8-well PCR strip | Sarstedt | 72.991.002 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A9418 | |
| C1000 Touch Thermal Cycler with 96–Deep Well Reaction Module | Bio-Rad | 1851197 | |
| ddPCR Buffer Control for Probes 9 mL (2 x 4.5 mL) | Bio-Rad | 1863052 | |
| ddPCR Supermix for Probes (No dUTP) (2 x 1 mL) | Bio-Rad | 1863023 | |
| ddPCR 96-Well Plates (pkg of 25) | Bio-Rad | 12001925 | |
| ddPCR Droplet Reader Oil (2 x 1L) | Bio-Rad | 1863004 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges for QX200/QX100 (pkg of 24) | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 (pkg of 24) | Bio-Rad | 1863009 | |
| DNase I (10U/µL) + buffer | Roche | 4716728001 | |
| Droplet Generation Oil for Probes (10 x 7 mL) | Bio-Rad | 1863005 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 0.1-10 μL, 34 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078500 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 20-300 μL, 55 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078560 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 2-100 μL, 53 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078543 | |
| Forward lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Forward primer: 5'-GAACGGCATCAAGGTGAACT-3' | |
| Lyopohilized probe and master stock at 100 µM | IDT | GFP as target sequence. PrimeTime Eco Probe: /56-FAM/CAAGATCCG/ZEN/CCACAACATCGAGGA/3IABkFQ/ | |
| Magnesium Chloride (MgCl2) | Chemlab Analytical | CL00.1381 | |
| Nuclease free water | IDT | 11-04-02-01 | |
| PCR Plate Heat Seal, foil, pierceable (pkg of 100) | Bio-Rad | 1814040 | |
| Pluronic F-68 non-ionic surfactant (100x) | Thermo Fisher Scientific | 24040032 | |
| Potassium Chloride (KCl) | Honeywell research chemicals | 31248 | |
| QX manager software | Bio-Rad | Software to analyse ddPCR data | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | 1864003 | |
| Reagent reservoir | VWR | 613-1181 | |
| Reverse lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Reverse primer: 5'-TGCTCAGGTAGTGGTTGTCG-3' | |
| SafeSeal reaction tube, 1.5 mL | Sarstedt | 72.706.200 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Thermo Fisher Scientific | AM9680 | |
| TE buffer | IDT | Accompanied by primers when ordering | |
| Tris hydrochloride (Tris-HCl) | Roche | 10812846001 |
Referenzen
- Balakrishnan, B., Jayandharan, G. R. Basic biology of adeno-associated virus (AAV) vectors used in gene therapy. Curr Gene Ther. 14 (2), 86-100 (2014).
- Srivastava, A. In vivo tissue-tropism of adeno-associated viral vectors. Curr Opin Virol. 21, 75-80 (2016).
- Pupo, A., et al. AAV vectors: The Rubik's cube of human gene therapy. Mol Ther. 30 (12), 3515-3541 (2022).
- Gimpel, A. L., et al. Analytical methods for process and product characterization of recombinant adeno-associated virus-based gene therapies. Mol Ther Methods Clin Dev. 20, 740-754 (2021).
- Brimble, M. A., et al. Preventing packaging of translatable P5-associated DNA contaminants in recombinant AAV vector preps. Mol Ther Methods Clin Dev. 24, 280-291 (2022).
- Wright, J. F. Product-related impurities in clinical-grade recombinant AAV vectors: Characterization and risk assessment. Biomedicines. 2 (1), 80-97 (2014).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, (2019).
- Sidransky, D., et al. Identification of ras oncogene mutations in the stool of patients with curable colorectal tumors. Science. 256 (5053), 102-105 (1992).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci USA. 96 (16), 9236-9241 (1999).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Anal Chem. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Anal Chem. 84 (2), 1003-1011 (2012).
- Sanmiguel, J., Gao, G., Vandenberghe, L. H. Quantitative and digital droplet-based AAV genome titration. Methods Mol Biol. 1950, 51-83 (2019).
- Bio-Rad Laboratories Inc. Planning droplet digital PCR experiments. , https://www.bio-rad.com/en-in/life-science/learning-center/introduction-to-digital-pcr/planning-ddpcr-experiments (2024).
- Dorange, F., Le Bec, C. Analytical approaches to characterize AAV vector production & purification: Advances and challenges. Cell Gene Ther Insights. 4 (2), 119-129 (2018).
- Lock, M., et al. Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Ther Methods. 25 (2), 115-125 (2014).
- Prantner, A., Maar, D. Genome concentration, characterization, and integrity analysis of recombinant adeno-associated viral vectors using droplet digital PCR. PLoS One. 18 (1), e0280242(2023).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, 1570(2019).
- Suoranta, T., Laham-Karam, N., Yla-Herttuala, S. Optimized protocol for accurate titration of adeno-associated virus vectors. Hum Gene Ther. 32 (19-20), 1270-1279 (2021).
- Bio-Rad. Droplet digital PCR applications guide. Bulletin 6407. , https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6407.pdf (2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten