Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Mikrobiota von Attinanameisengärten: Visualisierung einer mikrobiellen Landschaft mit Hilfe der Rasterelektronenmikroskopie
In diesem Artikel
Zusammenfassung
Wir schlagen ein optimiertes Rasterelektronenmikroskopie-Protokoll vor, um sehr heterogene und empfindliche Proben zu visualisieren, die pflanzliche und pilzliche Biomasse zusammen mit Mikrobiota und Biofilm enthalten. Dieses Protokoll ermöglicht es, die räumlichen Dimensionen der Mikrobiota-Organisation zu beschreiben.
Zusammenfassung
In makroskaligen Ökosystemen, wie Regenwäldern oder Korallenriffen, ist die räumliche Lokalisierung von Organismen die Grundlage für unser Verständnis der Ökologie von Lebensgemeinschaften. Auch in der mikrobiellen Welt sind mikroskalige Ökosysteme weit davon entfernt, eine zufällige und homogene Mischung von Organismen und Lebensräumen zu sein. Der Zugang zur räumlichen Verteilung von Mikroben ist von grundlegender Bedeutung für das Verständnis der Funktionsweise und Ökologie der Mikrobiota, da es wahrscheinlicher ist, dass zusammenlebende Arten interagieren und sich gegenseitig in ihrer Physiologie beeinflussen.
Ein mikrobielles Ökosystem zwischen den Reichen ist das Herzstück der pilzzüchtenden Ameisenkolonien, die Basidiomyceten-Pilze als Nahrungsquelle kultivieren. Attinameisen suchen nach verschiedenen Substraten (meist pflanzlich), die vom kultivierten Pilz verstoffwechselt werden, während sie eine schwammige Struktur bilden, einen "mikrobiellen Garten", der als externer Darm fungiert. Der Garten ist ein verflochtenes Geflecht von Pilzhyphen, die durch Verstoffwechselung des Substrats wachsen und Nischen öffnen, in denen sich eine charakteristische und angepasste Mikrobiota etablieren kann. Es wird angenommen, dass die Mikrobiota zum Substratabbau und zum Pilzwachstum beiträgt, obwohl ihre räumliche Organisation noch nicht bestimmt ist.
Hier beschreiben wir, wie wir die Rasterelektronenmikroskopie (REM) einsetzen, um die räumliche Organisation der Mikrobiota und des Biofilms in verschiedenen Pilzkultursystemen von pilzzüchtenden Ameisen mit bisher unerreichter Detailgenauigkeit zu untersuchen. Die REM-Bildgebung hat eine Beschreibung der räumlichen Struktur und Organisation der Mikrobiota geliefert. REM zeigte, dass sich Mikrobiota häufig in Biofilmen ansammeln, einer weit verbreiteten Struktur der mikrobiellen Landschaften in der Pilzkultur. Wir stellen die Protokolle vor, die zum Fixieren, Dehydrieren, Trocknen, Sputtern, Beschichten und Abbilden einer solch komplexen Gemeinschaft eingesetzt werden. Diese Protokolle wurden für den Umgang mit empfindlichen und heterogenen Proben optimiert, die sowohl pflanzliche und pilzliche Biomasse als auch die Mikrobiota und den Biofilm umfassen.
Einleitung
Ökosysteme bestehen aus Organismen, die durch Prozesse an einem bestimmten geografischen Ort (d. h. der Umwelt) miteinander verbunden sind. Organismen interagieren im Laufe der Zeit mit ihrer Umwelt, wodurch komplexe und heterogene räumliche Muster entstehen. Räumliche Muster bestimmen die ökologische Vielfalt und Stabilität und letztlich das Funktionieren des Ökosystems 1,2,3,4. In makroskaligen Ökosystemen wie Feuchtgebieten, Savannen, Korallenriffen und trockenen Ökosystemen korrelieren räumliche Muster mit Ressourcenfluss und -konzentration. Durch die Optimierung von Ressourcen führen räumliche Heterogenität und Musterbildung zu widerstandsfähigeren Ökosystemen als homogeneÖkosysteme 2. Die räumliche Lokalisierung von Organismen, die die Grundlage der Ökologie von Lebensgemeinschaften bildet, wird auch auf die mikrobielle Welt übertragen.
Mikrobielle Ökosysteme, weit entfernt von Organismen, die zufällig und homogen in Mikrohabitaten gemischt sind, weisen räumliche Muster auf, die einen Großteil ihrer Funktionsweise bestimmen 5,6,7. Von Winogradsky-Säulen bis hin zu umwelt- und wirtsassoziierten Mikrobiota sind diese Ökosysteme heterogen im Raum organisiert, wobei räumliche Anordnungen unterschiedliche phänotypische Reaktionen hervorrufen. Zusammenlebende Arten interagieren mit größerer Wahrscheinlichkeit und beeinflussen die Physiologie der anderen. So grenzt die räumliche Organisation der Gemeinschaft, mehr als ihre Zusammensetzung an sich, Ökosystemeigenschaften und ökologische Nischenab 5,7,8. Zur Veranschaulichung dieser Konzepte scheinen Veränderungen in der räumlichen Musterung mit dem pathologischen Fortschreiten von Zahnbelag, Karies, Zahnfleischerkrankungen 9,10, entzündlichen Darmerkrankungen11, Mukoviszidose, Lungeninfektionen, chronischen Wundinfektionen12,13, Darmkrebs und Adenomen14 zu korrelieren.
Im Rahmen der mikrobiellen Biogeographie (der Untersuchung der Verteilung und Musterung der biologischen Vielfalt über Raum und Zeit auf einer Mikroskala) ist das Wissen über mikrobielle Ökosysteme durch das Verständnis ihrer räumlichen Muster von großem Nutzen 6,13,15,16,17. Wir haben uns die räumlichen Muster eines von Insekten aufgebauten mikrobiellen Ökosystems angesehen, das sich im Kern der charismatischen pilzzüchtenden Attinameisenkolonien (Hymenoptera: Formicidae: Myrmicinae: Attini: Attina) befindet. Es gibt einen "mikrobiellen Garten", der sich um einen Basidiomyceten-Pilz aus dem Tribus Leucocoprinae (Basidiomycota: Agaricaceae) oder aus der Familie der Pterulaceae (Basidiomycota: Agaricales) dreht18,19,20,21,22. Der Garten ist eine schwammige Struktur, die aus einem ineinander verschlungenen Netz von Hyphen entsteht, das durch Verstoffwechselung des meist pflanzlichen Substrats wächst, das von den Ameisen eingebaut wird (Abbildung 1). Dazu können je nach den attinischen Gattungen gehören: trockene Pflanzenteile, Insektenfrasse und -kadaver, frisch geschnittene Blätter, Samen und Blütenteile23,24. Analog zu einem externen pflanzenfressenden Darm wandelt der Garten widerspenstige Polymere enzymatisch und chemisch in labile Nahrungsressourcen um und versorgt die Ameisen mit essentiellen Aminosäuren, Lipiden und löslichen Zuckern 21,25,26,27,28.
Ultrastrukturelle, enzymatische und transkriptomische Analysen, die für Gärten der Blattschneidergattungen Atta und Acromyrmex durchgeführt wurden, deuten darauf hin, dass diese Umgebungen ein Kontinuum von Substratabbau und Nährstoffflecken strukturieren 26,29,30,31,32. Junge Teile des Gartens sind durch das frisch eingearbeitete Substrat nach dem Fragmentieren tendenziell dunkler. Diese neu hinzugekommenen Substrate werden oft von den Rändern aus besiedelt, die von Ameisenarbeitern geschnitten und mit Myzelklumpen beimpft wurden. Pilzhyphen, die von Schnittkanten ausgehen, breiten sich über das Substrataus 29,32,33. Die Hyphenhäufigkeit nimmt mit fortschreitendem Substratabbau zu, was zu weißlichen und stoffwechselaktiven Regionen führt 30,31,32. Ältere Regionen mit stärker abgebautem Substrat und einer reichlich vorhandenen Mikrobiota29,32 neigen zu bräunlichen Tönen und höherer Luftfeuchtigkeit. Die Arbeiter entfernen Fragmente dieser Region und trennen sie in Abfallhaufen, wo sie auch Substrate entnehmen, die dem Pilzsymbionten 34,35,36 schaden. Obwohl Abfallhaufen physisch vom Garten getrennt sind, sind sie ein Ort des kontinuierlichen Substratabbaus und des Nährstoffkreislaufs durch die reichlich vorhandene bewohnte Mikrobiota 29,32,37,38,39.
Eine Mikrobiota, die hauptsächlich aus Enterobacter, Klebsiella, Pantoea, Pseudomonas und Serratia besteht, bewohnt ebenfalls den Garten, die anscheinend von verschiedenen Attine-Pilzzuchtsystemen geteilt wird. Die Mikrobiota kodiert für Stoffwechselwege, die den Pilzstoffwechsel ergänzen könnten, und ist möglicherweise an den physiologischen Reaktionen des Gartens beteiligt 40,41,42,43,44. Metagenomische Daten deuteten nicht nur darauf hin, dass die Mikrobiota vorhanden war41,42, sondern auch die Rasterelektronenmikroskopie (REM) Analyse der Pilzkultur von Blattschneiderameisen zeigte hauptsächlich stäbchenförmige Bakterien über dem Pflanzensubstrat32. Obwohl Bakterien (einschließlich zellulolytischer Stämme) aus dem gesamten Garten isoliert wurden, wurden sie nur in älteren Teilen des Gartens und in Abfallhaufen sowie in dem ersten Pellet sichtbar, das von den Gründerköniginnengetragen wurde 29,32. Es war auch unsicher, ob die Mikrobiota in vivo (d. h. im Garten und im Abfall) Biofilme bilden kann, wie ihre metabolische Kapazitätvermuten lässt 42 und in vitrobeobachtet wurde 44.
Hier haben wir SEM eingesetzt, um die räumliche Organisation der Mikrobiota in den Gartenregionen weiter zu verstehen, indem wir die physikalischen Wechselwirkungen zwischen Mikrobiota-Substrat und Mikrobiota-Hyphen detailliert beschrieben haben. Durch die Bereitstellung von Bildern mit größerer Brenntiefe ermöglicht das REM die Beobachtung dreidimensionaler mikroskopischer Strukturen in hoher Auflösung und ermöglicht so eine gründliche Analyse der räumlichen Muster der Gartenmikrobiota. Wir beschreiben die Schritte zum Fixieren, Dehydrieren, Trocknen, Sputtern, Beschichten und Abbilden solcher heterogenen und empfindlichen Proben auf Pilzbasis. Durch den Wegfall des Postfixationsschritts mit Osmiumtetroxid (OsO4) und die Verkürzung der Dehydratisierungszeit haben wir die Protokolle 32,33,45 für die Vorbereitung von Garten- und Abfallproben für die REM-Analyse vereinfacht. Dieses angepasste Protokoll bewahrt die Hyphenstrukturmuster sowie die räumliche Organisation der Mikrobiota und des Biofilms und könnte auf andere empfindliche mikrobielle Ökosysteme und Biofilme angewendet werden.
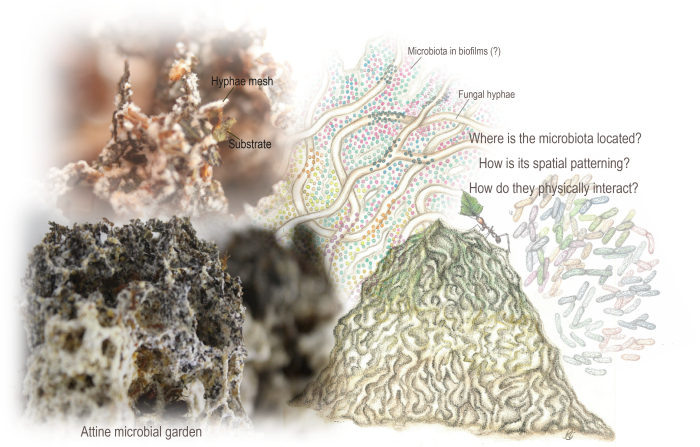
Abbildung 1: Mikrobielle Gärten von Atti. Der Garten ist eine schwammartige Struktur, die aus einem ineinander verschlungenen Geflecht von Hyphen entsteht, das durch Verstoffwechselung des meist pflanzlichen Substrats wächst, das von den Ameisen eingebaut wird. Ebenfalls im Garten wohnt die Mikrobiota, die Stoffwechselwege kodiert, die den Stoffwechsel der Pilze ergänzen könnten. Metagenomische Daten und frühere Rasterelektronenmikroskopie-Analysen deuteten auf sein Vorhandensein hin, obwohl wir nur wenige Kenntnisse über seine räumliche Organisation und seine physikalischen Wechselwirkungen mit dem Substrat und den Pilzhyphen hatten. Wir setzten SEM ein, um die räumliche Organisation und Musterung der Mikrobiota und des Biofilms zu enthüllen. Illustrationen von Mariana Barcoto (Garten und Mikrobiota adaptiert von Barcoto und Rodrigues 94) und Fotos von Mariana Barcoto und Enzo Sorrentino. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Probenahme von Feldvölkern
HINWEIS: Wenn Sie Ameisenkolonien sammeln, bestätigen Sie, dass vor dem Sammeln alle von der örtlichen Gesetzgebung geforderten Genehmigungen eingeholt wurden. In unserem Fall wurde die Sammelgenehmigung #74585 vom Instituto Chico Mendes de Conservação e Biodiversidade (ICMBio) ausgestellt. Wenn die Proben aus einer Laborkolonie stammen, fahren Sie mit Abschnitt 2 fort.
- Lokalisiert und markiert die Kolonie. Heben Sie einen Graben aus, der den Nestbereich umgibt, bis die Gartenkammer freigelegt ist (Abbildung 2A).
HINWEIS: Einige Attinasarten können ihre Kolonien unter der Laubstreu oder in morschen Baumstämmen aufbauen. Drehen Sie in solchen Fällen die Einstreu vorsichtig oder brechen Sie vorsichtig die Stämme zum Sammeln der Proben. Für detaillierte Informationen über das Auffinden, Sammeln und Halten lebender Kolonien verschiedener Attinameisenarten siehe Sosa-Calvo et al.46. - Öffnen Sie die Gartenkammer seitlich, um zu verhindern, dass die Erde auf die Gartenoberfläche fällt. Entnehmen Sie vorsichtig Gartenproben mit einer entomologischen Pinzette, einem Löffel oder einer Küchenskimmer, je nach Gartengröße.
HINWEIS: Achten Sie darauf, die Werkzeuge vor dem Sammeln zu sterilisieren. Tragen Sie beim Sammeln von Blattschneiderameisen in den Gärten dicke Stoffhandschuhe, um Arbeiterbisse zu vermeiden (oder zumindest abzuschwächen). Für andere Attine-Arten sind Stoffhandschuhe optional. - Füllen Sie die Gartenproben in einen sauberen Kunststoffbehälter mit einer Schicht Putz um, um die Feuchtigkeit im Garten auszugleichen. Nach dem Umsetzen der Garten- und Ameisenarbeiter den Behälter hermetisch verschließen, um ein Austrocknen der Probe zu vermeiden. Lagern Sie die Gartenproben bis zur Verarbeitung bei 23-25 °C.
- Verschließen Sie den Graben mit zuvor entfernter Erde.
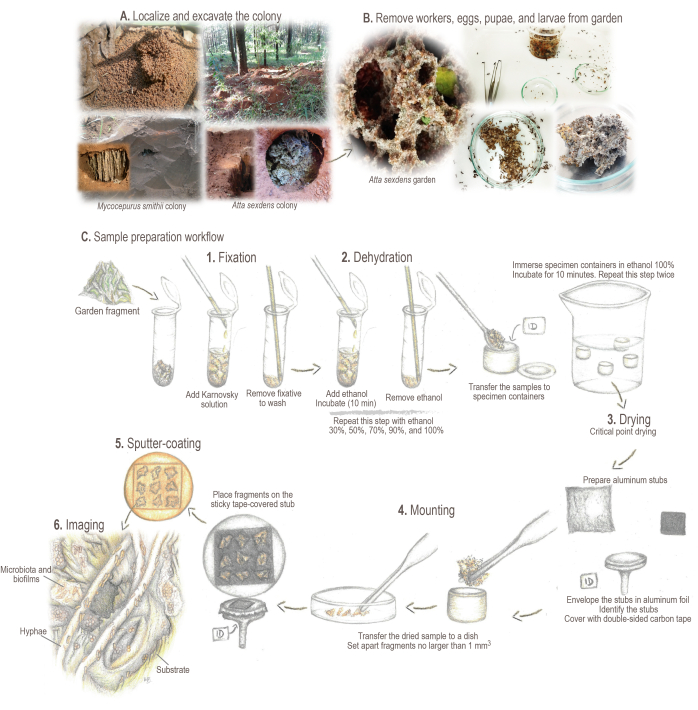
Abbildung 2: Protokoll der Probenvorbereitung. (A) Probenahme von Feldvölkern. (B) Verarbeitung von Proben. (C) Kurze Grundlagen und Arbeitsabläufe für die Probenvorbereitung: 1. Fixierung: zur Verstärkung und Erhaltung der Probenstruktur. 2. Dehydratisierung: Der Wassergehalt der Proben wird gegen Ethanol ausgetauscht. 3. Trocknung an der kritischen Stelle: Flüssiges CO2 ersetzt Ethanol und wird verdampft. 4. Montage: Probe wird zur Analyse angezeigt. 5. Sputterbeschichtung mit Gold: Verhindern Sie das Auffüllen der Probe. 6. Bildgebung. Illustrationen und Fotos von Mariana Barcoto. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Reagenzien
HINWEIS: Beachten Sie, dass die folgenden Lösungen im Voraus vorbereitet werden sollten.
- Bereiten Sie 0,2 M Natriumcacodylatpuffer vor. Lösen Sie dazu 42,8 g Natriumcacodylat in 800 ml destilliertem Wasser auf und rühren Sie bis zur Auflösung und pH-Stabilisierung bei 7,2 (falls erforderlich, stellen Sie den pH-Wert mit Salzsäure ein). Vergrößern Sie das Volumen der Lösung auf 1 l, indem Sie destilliertes Wasser hinzufügen. Lagern Sie die Lösung bei 4 °C (~1 Monat lang).
ACHTUNG: Glutaraldehyd und Paraformaldehyd sind giftig und sollten in einem Abzug gehandhabt werden. Tragen Sie beim Umgang mit solchen Reagenzien ständig Nitrilhandschuhe und eine Schutzbrille. - Bereiten Sie das Karnovsky-Fixiermittel (modifiziert von Karnovsky) vor47. Geben Sie dazu 10 ml 25%ige wässrige Lösung aus Glutaraldehyd und 10 ml wässrige Lösung aus 20 % Paraformaldehyd zu 25 ml 0,2 M Natriumcacodylatpuffer und mischen Sie. Fügen Sie 1 ml 0,1 M Calciumchlorid (CaCl2) hinzu und erhöhen Sie das Volumen durch Zugabe von destilliertem Wasser auf 100 ml. Lagern Sie die Lösung bei 4 °C (bis zu 1 Monat).
ACHTUNG: Das Karnovsky-Fixiermittel ist schädlich, wenn es eingeatmet wird, und kann Haut- und Augenreizungen verursachen. Vermeiden Sie daher das Einatmen von Dämpfen, indem Sie es nur im Freien oder in einem gut belüfteten Bereich verwenden. Tragen Sie beim Umgang mit der Lösung ständig Nitrilhandschuhe und eine Schutzbrille. - Bereiten Sie unter Verwendung von absolutem Ethanol (analytische Qualität) 30 %, 50 %, 70 % und 90 % Ethanollösungen in destilliertem Wasser vor. 100% Ethanol ist ebenfalls erforderlich.
3. Fixierung der Probe
HINWEIS: Fixiermittel härten und konservieren Proben unter Beibehaltung morphologischer Merkmale. Aldehyde (wie Paraformaldehyd und Glutaraldehyd) sind nicht gerinnungshemmende Fixiermittel vom Vernetzungstyp, die Vernetzungen innerhalb und zwischen Proteinen und Nukleinsäuren induzieren48.
- Entnehmen Sie Arbeiterinnen, Eier, Puppen und Larven mit einer entomologischen Pinzette aus den Gartenproben (Abbildung 2B). Gartenfragmente, die nicht größer als 5 mmsind, beiseite stellen 3. Geben Sie die Fragmente in ein 2-ml-Röhrchen (Abbildung 2C).
- Verwenden Sie eine Pasteur-Glaspipette, um ~1 ml Karnovsky-Fixierlösung zu den Röhrchen mit den Proben hinzuzufügen (stellen Sie sicher, dass die Probe vollständig bedeckt ist). Durch leichtes Rühren wird die Probe eingeweicht und mindestens 24 Stunden lang bei 4 °C inkubiert, bevor die Probenverarbeitung fortgesetzt wird (Abbildung 2C.1).
HINWEIS: Wenn die folgenden Dehydratisierungsschritte nicht unmittelbar nach der Fixierung durchgeführt werden, kann das Protokoll bei diesem Schritt pausiert werden und die Proben können ~1 Jahr bei 4 °C gelagert werden.
Wir empfehlen die Verwendung einer Pasteur-Glaspipette, da sie aus einem inerten Material besteht und für die spätere Wiederverwendung leichter zu reinigen ist. Gartenmaterial ist stark hydrophob und neigt dazu, über die Oberfläche der Fixierlösung zu schwimmen. In der Regel dauert es bis zu 5 Minuten, bis das Fixiermittel vollständig eingeweicht ist. Stellen Sie sicher, dass das Fixiermittel die Gartenfragmente bedeckt, da sein Volumen tendenziell reduziert wird, wenn es in die Poren der Probe gelangt. Wir haben empirisch bestätigt, dass die Bestandteile der Probe (insbesondere das empfindliche Pilzmyzel) nach dem Einweichen und Befeuchten anfällig für Zerbrechen wurden, wenn sie weiter gemischt wurden. Wir empfehlen daher, das Schütteln der Proben so weit wie möglich zu vermeiden.
4. Dehydrierung der Probe
HINWEIS: Bei der Ethanol-Waschserie wird das Wasser in den Proben nach und nach gegen Ethanol ausgetauscht. Es ist wichtig, mit einer niedrig konzentrierten Ethanollösung (siehe unten) zu beginnen, um eine übermäßige Beschädigung oder einen Kollaps solcher empfindlichen Probenzu vermeiden 49.
- Die Karnovsky-Fixierlösung mit einer Glaspipette vollständig entfernen, wobei darauf zu achten ist, dass die Probe nicht beschädigt wird (Abbildung 2C.1).
HINWEIS: Entsorgen Sie das Karnovsky-Fixiermittel in einem Behälter, der ordnungsgemäß für die Behandlung giftiger chemischer Rückstände gekennzeichnet ist. - Unmittelbar nach dem Entfernen des Fixiermittels 1 ml 30%iges Ethanol hinzufügen, darauf achten, dass die Probe nicht gestört wird, und 10 Minuten bei Raumtemperatur inkubieren (Abbildung 2C.2).
HINWEIS: Die Proben müssen immer in Lösung eingeweicht werden. Stellen Sie sicher, dass die Lösungen während der Ethanol-Waschserie schnell ausgetauscht werden. Da der Garten sein poröses Aussehen verliert und sich am Röhrchenboden ansammelt, reicht 1 ml Ethanol in der Regel aus, um die Probe zu bedecken (wenn die Probe nicht größer als 5 mmist 3). Wenn die Proben jedoch nicht vollständig bedeckt sind, fügen Sie Ethanol hinzu, bis die Probe vollständig bedeckt ist. - Entfernen Sie 30 % Ethanol vollständig mit einer Glaspipette und achten Sie darauf, die Probe nicht zu stören. 30% Ethanol ordnungsgemäß entsorgen.
HINWEIS: Entsorgen Sie das Ethanol für die gesamte Serie der schrittweisen Ethanolwäsche in einem Behälter, der ordnungsgemäß für die Behandlung giftiger chemischer Rückstände gekennzeichnet ist. - 1 ml 50%iges Ethanol zugeben und 10 Minuten bei Raumtemperatur inkubieren. Entfernen Sie 50 % Ethanol vollständig mit einer Glaspipette und achten Sie darauf, die Probe nicht zu stören. Entsorgen Sie 50% Ethanol ordnungsgemäß.
- 1 ml 70%iges Ethanol zugeben und 10 Minuten bei Raumtemperatur inkubieren. Entfernen Sie 70 % Ethanol vollständig mit einer Glaspipette und achten Sie darauf, die Probe nicht zu stören. 70% Ethanol ordnungsgemäß entsorgen.
HINWEIS: Beim Waschschritt mit 70 % Ethanol kann der Benutzer das Protokoll bei Bedarf pausieren, da Probenröhrchen über Nacht bei 4 °C gelagert werden können, wenn das Material nicht sofort verarbeitet wird. - 1 ml 90%iges Ethanol zugeben und 10 Minuten bei Raumtemperatur inkubieren. Entfernen Sie 90 % Ethanol vollständig mit einer Glaspipette und achten Sie darauf, die Probe nicht zu beschädigen. 90% Ethanol ordnungsgemäß entsorgen.
- 1 ml 100%iges Ethanol zugeben und 10 Minuten bei Raumtemperatur inkubieren. Entfernen Sie 100 % Ethanol vollständig mit einer Glaspipette und achten Sie darauf, die Probe nicht zu stören. Entsorgen Sie 100% Ethanol ordnungsgemäß.
- Übertragen Sie die Proben mit einer Pinzette und/oder einem Spatel vorsichtig in Probenbehälter für den Critical Point Dryer (CPD), die Probenidentifikationsetiketten enthalten (zuvor aus Papier und Bleistift hergestellt). Um ein Austrocknen der Proben zu vermeiden, wird dieser Transfer durchgeführt, indem der Behälter in eine Petrischale gestellt wird, die mit 100 % Ethanol bedeckt ist.
- Setzen Sie die Deckel auf die Behälter und tauchen Sie sie in ein Messglas, das 100 % Ethanol enthält, das ausreicht, um die Behälter zu untertauchen. Decken Sie das Becherglas ab und inkubieren Sie es 10 Minuten lang bei Raumtemperatur; Entsorgen Sie dann 100% Ethanol ordnungsgemäß.
- Übertragen Sie die Probenbehälter in ein anderes Messglasbecherglas, das genügend 100 % Ethanol enthält, um die Behälter zu tauchen. Decken Sie das Becherglas ab und inkubieren Sie es 10 Minuten lang bei Raumtemperatur; Übergeben Sie dann die Probenbehälter in den Trockner für kritische Punkte.
HINWEIS: Nach der Waschserie sollten Glaspipetten und Becher reichlich mit destilliertem Wasser gespült werden, und dieses Restwasser sollte in einem Behälter entsorgt werden, der für die Behandlung giftiger chemischer Rückstände ordnungsgemäß gekennzeichnet ist. Nach dem Spülen können Glasteile mit neutralem Reinigungsmittel gewaschen, mit Leitungswasser gespült und an der Luft getrocknet werden.
5. Trocknung an kritischen Punkten
HINWEIS: Ein Critical-Point-Trockner tauscht das Ethanol in den Proben gegen flüssiges Kohlendioxid (CO2) aus, das bei höherer Temperatur und höherem Druck aus der Probe verdampft. Bitte befolgen Sie die Anweisungen des Herstellers für solche Verfahren.
- Schalten Sie das Gerät ein.
- Öffnen Sie die Kammer, stellen Sie die Probenbehälter hinein und fügen Sie 100 % Ethanol hinzu, bis es die Behälter bedeckt. Schließen Sie die Kammer.
- Aktivieren Sie die Option Abkühlen und warten Sie, bis die Temperatur 10 °C erreicht.
- Öffnen Sie das CO2 -Flaschenventil und aktivieren Sie die Option Rührer .
- Aktivieren Sie die Option CO2 in und überprüfen Sie immer die Kammer, um zu überprüfen, wie viel CO2 sie bereits gefüllt hat. Wenn die Kammer fast gefüllt ist, deaktivieren Sie die Option CO2 in und aktivieren Sie die Option Austausch , wobei Sie sie so lange aktiviert lassen, bis genügend CO2 vorhanden ist, um die Behälter abzudecken. Achten Sie darauf, dass die Behälter immer mit CO2 bedeckt sind (d.h. deaktivieren Sie die Option Austausch , bevor das gesamte CO2 die Kammer verlassen hat). Wiederholen Sie Schritt 5.5 6x.
- Aktivieren Sie ein letztes Mal CO2 und füllen Sie die Kammer, bis die Behälter bedeckt sind.
- Aktivieren Sie die Option Hitze und deaktivieren Sie die Option Rührer . Das Ventil der CO2 -Flasche schließen.
- Warten Sie, bis die Temperatur auf 35 °C ansteigt; Aktivieren Sie dann die Option Gas out .
HINWEIS: Bei ca. 30 °C erreicht die Kammer einen Druck von 70-80 bar und erreicht damit den kritischen Punkt, an dem die Flüssigkeit verschwindet. - Wenn der Kammerdruck 1 bar erreicht, ist der gesamte Gasgehalt entfernt. Öffnen Sie die Kammer und entfernen Sie die Behälter.
- Schalten Sie das Gerät aus.
6. Montage
- Bereiten Sie REM-Probenhalter vor (z. B. Aluminium-Stubs; Abbildung 2C.4).
- Wickeln Sie die Stummel mit einem Stück Alufolie ein, die nur die Oberseite bedeckt, um die Reinigung der Stummel nach der Analyse zu erleichtern.
- Identifizieren Sie die Stubs, indem Sie den Probencode/die Probennummer auf die Unterseite des Halters schreiben, um die Identifizierung dessen zu gewährleisten, was oben platziert ist.
- Decken Sie den oberen Teil mit doppelseitigem Carbonband ab. Legen Sie die Stummel in einen Probenhalter.
- Öffnen Sie den Deckel des Probenbehälters und geben Sie die getrocknete Probe vorsichtig mit einer Pinzette und einem Spatel in eine Petrischale aus Glas.
HINWEIS: Kritisch punktgetrocknete Gärten neigen dazu, sich zu agglomerieren und stark gepackte Proben zu bilden, die sorgfältig als Fragmente mit einer Größe von nicht mehr als 1 mm3auseinandergesetzt werden sollten. - Legen Sie die Fragmente vorsichtig auf die klebrige Oberfläche des mit Klebeband bedeckten Stummels. Sobald das Gartenfragment das Klebeband berührt, ist es sehr schwierig, es (wieder) zu bewegen, also achten Sie darauf, es nicht an unerwünschten Stellen oder Positionen zu platzieren. Fügen Sie bis zu neun Fragmente pro Stub hinzu.
- Die Schritte 6.2 und 6.3 werden für jede Probe wiederholt.
7. Sputter-Beschichtung mit Gold
HINWEIS: Eine Beschichtung der Probe ist erforderlich, um ein Aufladen zu verhindern. Befolgen Sie die Anweisungen des Herstellers, um Einstellungen wie den Betriebsgasdruck (0,5 × 10-1 mm Hg Gasdruck in diesem Protokoll), die Sputterzeit (220 s), die Dicke der Goldschicht (~120 Å), den Strom (50 mA) und die Spannungsversorgung anzupassen. Das Sputtern folgt in der Regel einem gemeinsamen Arbeitsablauf, obwohl die Geräte verschiedener Hersteller leicht unterschiedlich funktionieren können.
- Öffnen Sie den klappbaren Zielarm und legen Sie die Stummel auf den Probentisch.
- Schließen Sie den klappbaren Zielarm und prüfen Sie, ob der Splitterschutz der Glasvakuumkammer richtig eingelassen ist.
- Öffnen Sie das Argonflaschenventil und schalten Sie den Hauptschalter ein.
- Verfolgen Sie den Anstieg des Vakuums in der Geräteanzeige, bis er die Markierung von 0,5 × 10-1 mm Hg in der Anzeige erreicht. Aktivieren Sie dann Spülen. Wiederholen Sie den Vorgang 5x.
- Schalten Sie das Wasserzirkulationssystem ein, aktivieren Sie die Option HV On und öffnen Sie den Goldfoliendeckel. Vergewissern Sie sich, dass die Plasmafarbe rosa ist. Um diesem Protokoll zu folgen, stellen Sie ein Sputtern von 220 s mit 50 mA Spannung ein, das eine Goldschicht von ~120 Å (12 nm) abscheidet.
- HV On wird automatisch abgeschaltet. Schalten Sie das Wasserzirkulationssystem aus und schließen Sie den Goldfilmdeckel.
- Schalten Sie den Hauptschalter aus, damit Luft in die Vakuumkammer eindringen kann. Wiederholen Sie für Garten- und Abfallproben die Schritte 7.1-7.7 3x.
HINWEIS: Wenn die Bildgebung nicht unmittelbar nach der Probenvorbereitung erfolgt, lagern Sie die Stummel in einem hermetischen Behälter, der mit einer Kieselsäureschicht gefüllt ist, um eine Rehydrierung der Proben zu vermeiden.
8. Bildgebung
HINWEIS: Befolgen Sie die Anweisungen des Herstellers zum Anpassen der REM-Einstellungen, wie z. B. Objektivblendendurchmesser, Betriebsspannung, Ausrichtung des Elektronenstrahlsystems, axiale Ausrichtung und Stigmatoren.
- Legen Sie die Stummel in den Probenhalter und notieren Sie sich die Position jeder Probe.
HINWEIS: Tragen Sie beim Einsetzen oder Entfernen des Probenhalters Handschuhe und halten Sie den Halter so sauber wie möglich. - Starten Sie die Betriebssoftware vom Desktop aus.
- Wählen Sie die Instrumenteneinstellungen aus. Visualisieren Sie Gartenproben mit einem objektiven Aperturdurchmesser von 30 μm (d. h. in der zweiten Stufe), die im Hochvakuum arbeiten, Signale von Sekundärelektronen (SED) detektieren, eine Beschleunigungsspannung von 20 kV, einen Arbeitsabstand von 15 - 20 mm, einen Sondenstrom von 40,0 (im Hochstrommodus) und Variieren der Vergrößerung je nach Probe.
- Befolgen Sie die Navigationsanweisungen, drücken Sie auf das Entlüftungssymbol und warten Sie, bis die Probenkammer entlüftet ist. Ein Fortschrittsbalken zeigt den Status des Vakuums an.
- Wenn Atmosphärendruck erreicht ist, öffnen Sie die Probenkammer und setzen Sie den Probenhalter vorsichtig ein.
- Schließen Sie vorsichtig die Kammertür und drücken Sie das Evac-Symbol , um das Instrument zu evakuieren, und verfolgen Sie den Vakuumstatus durch den Fortschrittsbalken. Das Navigationssystem zeigt die Position der beweglichen Bühne an und stellt ein Halterdiagramm bereit, wenn die Bewegung abgeschlossen ist. Drücken Sie auf das Kamerasymbol , um ein Foto des Halters aufzunehmen und eine Draufsicht zu erhalten, mit der Sie während der Bildgebung zwischen den Proben navigieren können.
- Drücken Sie auf das Beobachtungssymbol , um die Elektronenkanone einzuschalten, und warten Sie, bis sich das Bild gebildet hat. Verwenden Sie die manuelle Benutzeroberfläche (manuelle Steuerung), um die Z-Achse manuell auf die richtige Höhe zu bewegen (basierend auf der Probenhöhe und dem ermittelten Arbeitsabstand).
HINWEIS: Um die Lebensdauer des Wolframfilaments zu verlängern, pausieren Sie die Beobachtungsfunktion , wenn nicht aktiv abgebildet wird. - Verwenden Sie entweder die auf dem Bildschirm angezeigten Optionen oder die manuelle Steuerung, um die Rasterrate und die Bildposition zu ändern. Verschieben Sie den Tisch, um einen vollständigen Überblick über die Probe zu erhalten, und verwenden Sie die RDC-Funktion , um sich auf bestimmte Bereiche zu konzentrieren. Wenn Sie eine Zielstruktur (oder eine interessante) Struktur beobachten, passen Sie die Vergrößerung, den Fokus, die Helligkeit, den Kontrast und die Stigmatisierung entsprechend an. Korrigieren Sie die Stigmatisierung, indem Sie die manuelle Benutzeroberfläche verwenden, um die Bühne in X- und Y-Richtung zu verschieben.
- Stellen Sie die Vergrößerung zwischen 100x und 700x ein, um allgemeine Gartenaspekte (z. B. Hyphendichte, Substrat und Besiedlungsmuster) zu visualisieren.
- Stellen Sie die Vergrößerung zwischen 700x und 1.500x ein, um die räumlichen Muster der Mikrobiota zu visualisieren.
- Stellen Sie die Vergrößerung zwischen 1.500x und 3.000x ein, um die physikalischen Wechselwirkungen zwischen Mikrobiota und Biofilm zu beobachten.
- Stellen Sie die Vergrößerung zwischen 3.000x und 4.000x ein, um sich auf bestimmte mikrobielle Cluster zu konzentrieren.
- Um ein Bild zu speichern, verwenden Sie die Funktion Einfrieren , klicken Sie auf das Fotosymbol und richten Sie den Dateipfad ein. Analysieren Sie standardmäßig mindestens drei Gartenfragmente und bilden Sie jedes in allen in Schritt 8.8 genannten Vergrößerungsbereichen ab. Dies führt zu mindestens 12 Bildern pro Probe, obwohl mehr Bilder detailliertere Beschreibungen unterstützen können. Wir empfehlen, dass 15-25 Bilder pro Probe, die alle Vergrößerungen in Schritt 8.8 abdecken, dazu neigen, feine Details für die Probenbeschreibung zu liefern.
HINWEIS: Das Rastern und Speichern eines Bildes dauert einige Sekunden, während derer Vibrationen auf dem schwebenden REM-Tisch vermieden werden sollten. - Drücken Sie nach Abschluss der Bildgebung die Option Entlüftung, um die Kammer zu entlüften, und verfolgen Sie den Vakuumstatus durch den Fortschrittsbalken. Entfernen Sie vorsichtig die Halterung, schließen Sie vorsichtig die Kammertür und drücken Sie Evac, um die Kammer zu evakuieren.
- Sobald die Evakuierung abgeschlossen ist, beenden Sie die Betriebssoftware.
- Passen Sie die Helligkeit und den Kontrast an, um die Visualisierung mit einem Bildeditor zu verbessern.
Ergebnisse
Hier haben wir ein vereinfachtes Protokoll vorgestellt, um die Bestandteile von Attingarten- und Abfallproben, wie Pilzhyphen, Substrat, Mikrobiota und Biofilme, zu visualisieren. SEM hat unser Verständnis dafür verbessert, wie der Garten und der Abfall die Strukturmuster der Mikrobiota bilden (Abbildung 3). In Attingärten sind Pilzhyphen astartige Strukturen, die Teile der Substratoberfläche bedecken. Da Pilzhyphen dazu neigen, sehr empfindlich auf Dehy...
Diskussion
Beim REM wird die Probe mit einem Elektronenstrahl abgetastet und so vergrößert, dass dreidimensionale Mikrostrukturen in hoher Auflösung sichtbar gemacht werden können. Da das REM im Hochvakuum arbeitet, ist die Entfernung von bis zu /mehr als 99 % des Wassers aus den Proben erforderlich. In der REM-Vakuumkammer können teilweise hydratisierte Proben dehydrieren und kollabieren, zusätzlich zur Streuung von Elektronen. Für die hochauflösende Bildgebung im REM sollte die Probenvorb...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Autoren danken der Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP) für die finanzielle Unterstützung (Grant #2019/03746-0). MOB dankt für das Promotionsstipendium von FAPESP (Prozess 2021/08013-0) und Coordenação de Aperfeiçoamento de Pessoal de Nível Superior - Brasilien (CAPES) - Finanzcode 001. AR dankt auch dem Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) für ein Forschungsstipendium (#305269/2018). Die Autoren danken Marcia Regina de Moura Aouada und Antonio Teruyoshi Yabuki für die Hilfe bei den Pilotversuchen zur Probenvorbereitung, Renato Barbosa Salaroli für die technische Unterstützung und Enzo Sorrentino für die Hilfe beim Fotoshooting. Diese Studie wurde im Rahmen der Access Genetic Heritage Authorization # SISGen AA39A6D durchgeführt.
Materialien
| Name | Company | Catalog Number | Comments |
| 2 mL tube | Axygen | MCT-200-C-BRA | To fix and dehydrate samples |
| Calcium chloride anhydrous | Merck | C4901 | CaCl2 anhydrous to prepare Karnovsky’s fixative |
| Critical point dryer | Leica | EM CPD 300 | For critical point drying |
| Double Sided Carbon Conductive Tape, 12 mm (W) X 5 M (L) | Electron Microscopy Sciences | 77819-12 | For mounting samples |
| Entomological forceps | No specific supplier | To manipulate garden samples | |
| Ethyl alcohol (=ethanol), pure (≥99.5%) | Sigma-Aldrich | 459836 | For dehydration |
| Forceps | No specific supplier | To manipulate garden samples | |
| Glass beaker | No specific supplier | For dehydration | |
| Glass Petri dish | No specific supplier | To manipulate garden samples | |
| Glass pipette | No specific supplier | To fix and dehydrate samples | |
| Glutaraldehyde (Aqueous Glutaraldehyde EM Grade 25%) | Electron Microscopy Sciences | 16220 | To prepare Karnovsky’s fixative |
| Gold target | Ted Pella, Inc. | 8071 | To sputter coat with gold |
| Hydrochloric acid | Sigma-Aldrich | 320331 | For adjusting solutions pH |
| Image editor | Photoshop | any version | To adjust images |
| Paraformaldehyde (Paraformaldehyde 20% Aqueous Solution EM Grade) | Electron Microscopy Sciences | 15713 | To prepare Karnovsky’s fixative |
| Propilene recipient | No specific supplier | For maintaining alive ant colonies | |
| Scanning Electron Microscope | JEOL | IT300 SEM | For sample imaging |
| Sodium cacodylate trihydrate | Sigma-Aldrich | C0250 | For preparing sodium cacodylate buffer |
| Spatula | No specific supplier | To manipulate garden samples | |
| Specimen containers with 15 mm dia. x 10 mm high | Ted Pella, Inc. | 4591 | For critical point drying |
| Sputter coater | Baltec | SCD 050 | To coat with gold |
| Stub (Aluminium mount, flat end pin) 12.7 mm x 8 mm | Electron Microscopy Sciences | 75520 | For mounting samples |
Referenzen
- Turner, M. G. Landscape ecology: the effect of pattern on process. Annu Rev Ecol Evol Syst. 20 (1), 171-197 (1989).
- Rietkerk, M., Van de Koppel, J. Regular pattern formation in real ecosystems. Trends Ecol Evol. 23 (3), 169-175 (2008).
- Schmitz, O. J. Spatial dynamics and ecosystem functioning. PLOS Biol. 8 (5), e1000378 (2010).
- Pringle, R. M., Doak, D. F., Brody, A. K., Jocqué, R., Palmer, T. M. Spatial pattern enhances ecosystem functioning in an African savanna. PLOS Biol. 8 (5), e1000377 (2010).
- Wimpenny, J. W. Spatial order in microbial ecosystems. Biol Rev. 56 (3), 295-342 (1981).
- Martiny, J. B. H., et al. Microbial biogeography: putting microorganisms on the map. Nat Rev. Microbiol. 4 (2), 102-112 (2006).
- McCallum, G., Tropini, C. The gut microbiota and its biogeography. Nat Rev Microbiol. 22 (2), 105-118 (2024).
- Lamont, R. J., Hajishengallis, G., Koo, H. Social networking at the microbiome-host interface. Infec Immun. 91 (9), e00124-e00223 (2023).
- Welch, J. L. M., Rossetti, B. J., Rieken, C. W., Dewhirst, F. E., Borisy, G. G. Biogeography of a human oral microbiome at the micron scale. Proc Natl Acad Sci USA. 113 (6), E791-E800 (2016).
- Kim, D., et al. Spatial mapping of polymicrobial communities reveals a precise biogeography associated with human dental caries. Proc Natl Acad Sci USA. 117 (22), 12375-12386 (2020).
- Swidsinski, A., Weber, J., Loening-Baucke, V., Hale, L. P., Lochs, H. Spatial organization and composition of the mucosal flora in patients with inflammatory bowel disease. J Clin Microbiol. 43 (7), 3380-3389 (2005).
- Ibberson, C. B., Barraza, J. P., Holmes, A. L., Cao, P., Whiteley, M. Precise spatial structure impacts antimicrobial susceptibility of S. aureus in polymicrobial wound infections. Proc Natl Acad Sci USA. 119 (51), e2212340119 (2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Dejea, C. M., et al. Microbiota organization is a distinct feature of proximal colorectal cancers. Proc Natl Acad Sci USA. 111 (51), 18321-18326 (2014).
- Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., Martiny, J. B. Beyond biogeographic patterns: processes shaping the microbial landscape. Nat Rev Microbiol. 10 (7), 497-506 (2012).
- Adade, E. E., Al Lakhen, K., Lemus, A. A., Valm, A. M. Recent progress in analyzing the spatial structure of the human microbiome: Distinguishing biogeography and architecture in the oral and gut communities. Curr Opin Endocr. 18, 275-283 (2021).
- Mony, C., Bohannan, B. J., Leibold, M. A., Peay, K., Vandenkoornhuyse, P. Microbial landscape ecology: Highlights on the invisible corridors. Front Ecol Evol. 9, 753213 (2021).
- Hölldobler, B., Wilson, E. O. . The Ants. , (1990).
- Chapela, I. H., Rehner, S. A., Schultz, T. R., Mueller, U. G. Evolutionary history of the symbiosis between fungus-growing ants and their fungi. Science. 266 (5191), 1691-1694 (1994).
- Mueller, U. G., Rehner, S. A., Schultz, T. R. The evolution of agriculture in ants. Science. 281 (5385), 2034-2038 (1998).
- Mueller, U. G., Gerardo, N. M., Aanen, D. K., Six, D. L., Schultz, T. R. The evolution of agriculture in insects. Annu Rev Ecol Evol Syst. 36, 563-595 (2005).
- Dentinger, B. T., Lodge, D. J., Munkacsi, A. B., Desjardin, D. E., McLaughlin, D. J. Phylogenetic placement of an unusual coral mushroom challenges the classic hypothesis of strict coevolution in the Apterostigma pilosum group ant-fungus mutualism. Evolution. 63 (8), 2172-2178 (2009).
- Schultz, T. R., Brady, S. G. Major evolutionary transitions in ant agriculture. Proc Natl Acad Sci USA. 105, 5435-5440 (2008).
- de Fine Licht, H. H., Boomsma, J. J. Forage collection, substrate preparation, and diet composition in fungus-growing ants. Ecol Entomol. 35 (3), 259-269 (2010).
- Martin, M. M. The biochemical basis of the fungus-attine ant symbiosis: A complex symbiosis is based upon integration of the carbon and nitrogen metabolisms of the two organisms. Science. 169 (3940), 16-20 (1970).
- Grell, M. N., et al. The fungal symbiont of Acromyrmex leaf-cutting ants expresses the full spectrum of genes to degrade cellulose and other plant cell wall polysaccharides. BMC Genomics. 14, 928 (2013).
- Lange, L., Grell, M. N. The prominent role of fungi and fungal enzymes in the ant-fungus biomass conversion symbiosis. Appl Microbiol Biotechnol. 98, 4839-4851 (2014).
- Huang, E. L., et al. The fungus gardens of leaf-cutter ants undergo a distinct physiological transition during biomass degradation. Environ Microbiol Rep. 6 (4), 389-395 (2014).
- Craven, S. E., Dix, M. W., Michaels, G. E. Attine fungus gardens contain yeasts. Science. 169 (3941), 184-186 (1970).
- De Fine Licht, H. H., Boomsma, J. J., Tunlid, A. Symbiotic adaptations in the fungal cultivar of leaf-cutting ants. Nat Commun. 5 (1), 5675 (2014).
- Aylward, F. O., et al. Enrichment and broad representation of plant biomass-degrading enzymes in the specialized hyphal swellings of Leucoagaricus gongylophorus, the fungal symbiont of leaf-cutter ants. PLoS One. 10 (8), e0134752 (2015).
- Moreira-Soto, R. D., Sanchez, E., Currie, C. R., Pinto-Tomás, A. A. Ultrastructural and microbial analyses of cellulose degradation in leaf-cutter ant colonies. Microbiology. 163 (11), 1578-1589 (2017).
- Erthal Jr, M., Silva, C. P., Cooper, R. M., Samuels, R. I. Hydrolytic enzymes of leaf-cutting ant fungi. Comp Biochem. 152 (1), 54-59 (2009).
- North, R. D., Jackson, C. W., Howse, P. E. Communication between the fungus garden and workers of the leaf-cutting ant, Atta sexdens rubropilosa, regarding choice of substrate for the fungus. Physiol Entomol. 24 (2), 127-133 (1999).
- Herz, H., Hölldobler, B., Roces, F. Delayed rejection in a leaf-cutting ant after foraging on plants unsuitable for the symbiotic fungus. Behav Ecol. 19 (3), 575-582 (2008).
- Schiøtt, M., De Fine Licht, H. H., Lange, L., Boomsma, J. J. Towards a molecular understanding of symbiont function: identification of a fungal gene for the degradation of xylan in the fungus gardens of leaf-cutting ants. BMC Microbiol. 8, 40 (2008).
- Bot, A. N., Currie, C. R., Hart, A. G., Boomsma, J. J. Waste management in leaf-cutting ants. Ethol Ecol Evol. 13 (3), 225-237 (2001).
- Scott, J. J., et al. Microbial community structure of leaf-cutter ant fungus gardens and refuse dumps. PLOS One. 5 (3), e9922 (2010).
- Lewin, G. R., et al. Cellulose-enriched microbial communities from leaf-cutter ant (Atta colombica) refuse dumps vary in taxonomic composition and degradation ability. PLOS One. 11 (3), e0151840 (2016).
- Suen, G., et al. An insect herbivore microbiome with high plant biomass-degrading capacity. PLOS Genet. 6 (9), e1001129 (2010).
- Aylward, F. O., et al. Convergent bacterial microbiotas in the fungal agricultural systems of insects. MBio. 5 (6), e02077 (2014).
- Barcoto, M. O., et al. Fungus-growing insects host a distinctive microbiota apparently adapted to the fungiculture environment. Sci Rep. 10 (1), 12384 (2020).
- Francoeur, C. B., et al. Bacteria contribute to plant secondary compound degradation in a generalist herbivore system. mBio. 11, e02146-e02220 (2020).
- Martiarena, M. J. S., Deveau, A., Montoya, Q. V., Flórez, L. V., Rodrigues, A. The hyphosphere of leaf-cutting ant cultivars is enriched with helper bacteria. Microb Ecol. 86 (3), 1773-1788 (2023).
- Leal-Dutra, C. A., et al. Evidence that the domesticated fungus Leucoagaricus gongylophorus recycles its cytoplasmic contents as nutritional rewards to feed its leafcutter ant farmers. IMA Fungus. 14 (1), 19 (2023).
- Sosa-Calvo, J., Jesovnik, A., Okonski, E., Schultz, T. R. Locating, collecting, and maintaining colonies of fungus-farming ants (Hymenoptera: Myrmicinae: Attini). Sociobiology. 62 (2), 300-320 (2015).
- Karnovsky, M. J. A formaldehyde glutaraldehyde fixative of high osmolality for use in electron microscopy. J Cell Biol. 27, 137-138 (1965).
- Eltoum, I., Fredenburgh, J., Myers, R. B., Grizzle, W. E. Introduction to the theory and practice of fixation of tissues. J Histotechnol. 24 (3), 173-190 (2001).
- Gusnard, D., Kirschner, R. H. Cell and organelle shrinkage during preparation for scanning electron microscopy: effects of fixation, dehydration and critical point drying. J Microsc. 110 (1), 51-57 (1977).
- Surman, S. B., et al. Comparison of microscope techniques for the examination of biofilms. J Microbiol Methods. 25 (1), 57-70 (1996).
- Augustin, J. O., et al. Yet more "weeds" in the garden: fungal novelties from nests of leaf-cutting ants. PLOS One. 8 (12), e82265 (2013).
- Montoya, Q. V., Martiarena, M. J. S., Polezel, D. A., Rodrigues, A. More pieces to a huge puzzle: Two new Escovopsis species from fungus gardens of attine ants. MycoKeys. 46, 97 (2019).
- Varanda-Haifig, S. S., et al. Nature of the interactions between hypocrealean fungi and the mutualistic fungus of leaf-cutter ants. Antonie van Leeuwenhoek. 110, 593-605 (2017).
- Custodio, B. C., Rodrigues, A. Escovopsis kreiselii specialization to its native hosts in the fungiculture of the lower attine ant Mycetophylax morschi. Antonie van Leeuwenhoek. 112, 305-317 (2019).
- Schröttner, H., Schmied, M., Scherer, S. Comparison of 3D surface reconstruction data from certified depth standards obtained by SEM and an Infinite Focus Measurement Machine (IFM). Microchim Acta. 155, 279-284 (2006).
- Zhou, W., Apkarian, R., Wang, Z. L., Joy, D., Zhou, W., Wang, Z. L. Fundamentals of scanning electron microscopy (SEM). Scanning microscopy for nanotechnology: Techniques and applications. , 1-40 (2007).
- Kannan, M., Raja, K., Subramanian, K. S., Kannan, M. Scanning electron microscopy: Principle, components and applications. A Textbook on Fundamentals and Applications of Nanotechnology. , 81-92 (2018).
- Ruffolo, J. J. Critical point drying of protozoan cells and other biological specimens for scanning electron microscopy: apparatus and methods of specimen preparation. Trans Am Microsc. 93 (1), 124-131 (1974).
- Echlin, P. Recent advances in specimen coating techniques for electron microscopy. Scanning Electron Microscopy 1981/1. , 79-90 (1981).
- Ris, H. The cytoplasmic filament system in critical point-dried whole mounts and plastic-embedded sections. J Cell Biol. 100 (5), 1474-1487 (1985).
- Bray, D. F., Bagu, J., Koegler, P. Comparison of hexamethyldisilazane (HMDS), Peldri II, and critical-point drying methods for scanning electron microscopy of biological specimens. Microsc Res Tech. 26 (6), 489-495 (1993).
- Bergmans, L., Moisiadis, P., Van Meerbeek, B., Quirynen, M., Lambrechts, P. Microscopic observation of bacteria: review highlighting the use of environmental SEM. Int Endod J. 38 (11), 775-788 (2005).
- Kirk, S. E., Skepper, J. N., Donald, A. M. Application of environmental scanning electron microscopy to determine biological surface structure. J Microsc. 233 (2), 205-224 (2009).
- Kemmenoe, B. H., Bullock, G. R. Structure analysis of sputter-coated and ion-beam sputter-coated films: a comparative study. J Microsc. 132 (2), 153-163 (1983).
- Kinden, D. A., Brown, M. F. Technique for scanning electron microscopy of fungal structures within plant cells. Phytopathology. 65, 74-76 (1975).
- Masaphy, S., Levanon, D., Tchelet, R., Henis, Y. Scanning electron microscope studies of interactions between Agaricus bisporus (Lang) Sing hyphae and bacteria in casing soil. Appl Environ Microbiol. 53 (5), 1132-1137 (1987).
- Massicotte, H. B., Melville, L. H., Peterson, R. L. Scanning electron microscopy of ectomycorrhizae potential and limitations. Scanning Microsc. 1 (3), 58 (1987).
- Visen, A., Singh, P. N., Chakraborty, B., Singh, A., Bisht, T. S. Scanning electron microscopy indicates Pseudomonad strains facilitate AMF mycorrhization in litchi (Litchi chinensis Sonn.) air-layers and improving survivability, growth and leaf nutrient status. Curr Res Microb Sci. 2, 100063 (2021).
- Fleeman, R. M., Mikesh, M., Davies, B. W. Investigating Klebsiella pneumoniae biofilm preservation for scanning electron microscopy. Access Microbiol. 5 (2), 000470-000473 (2023).
- Wells, M., Mikesh, M., Gordon, V. Structure-preserving fixation allows scanning electron microscopy to reveal biofilm microstructure and interactions with immune cells. J Microsc. 293 (1), 59-68 (2024).
- Nadell, C. D., Drescher, K., Foster, K. R. Spatial structure, cooperation and competition in biofilms. Nat Rev Microbiol. 14 (9), 589-600 (2016).
- Madsen, J. S., et al. Coexistence facilitates interspecific biofilm formation in complex microbial communities. Environ Microbiol. 18 (8), 2565-2574 (2016).
- Madsen, J. S., Sørensen, S. J., Burmølle, M. Bacterial social interactions and the emergence of community-intrinsic properties. Curr Opin Microbiol. 42, 104-109 (2018).
- Flemming, H. C., Wuertz, S. Bacteria and archaea on Earth and their abundance in biofilms. Nat Rev Microbiol. 17 (4), 247-260 (2019).
- Bringhurst, B., Allert, M., Greenwold, M., Kellner, K., Seal, J. N. Environments and hosts structure the bacterial microbiomes of fungus-gardening ants and their symbiotic fungus gardens. Microb Ecol. 86 (2), 1374-1392 (2023).
- Bringhurst, B., Greenwold, M., Kellner, K., Seal, J. N. Symbiosis, dysbiosis and the impact of horizontal exchange on bacterial microbiomes in higher fungus-gardening ants. Sci Rep. 14 (1), 3231 (2024).
- Vargas, S., et al. Body-plan reorganization in a sponge correlates with microbiome change. Mol Biol Evol. 40 (6), msad138 (2023).
- Apprill, A., et al. Humpback whale populations share a core skin bacterial community: towards a health index for marine mammals. PLOS One. 9 (3), e90785 (2014).
- Fraune, S., et al. Bacteria-bacteria interactions within the microbiota of the ancestral metazoan Hydra contribute to fungal resistance. ISME J. 9 (7), 1543-1556 (2015).
- Gowen, R., Gamal, A., Di Martino, L., McCormick, T. S., Ghannoum, M. A. Modulating the microbiome for Crohn's disease treatment. Gastroenterology. 164 (5), 828-840 (2023).
- Moya, P., et al. Molecular phylogeny and ultrastructure of the lichen microalga Asterochloris mediterranea sp. nov. from Mediterranean and Canary Islands ecosystems. Int J Syst Evol Microbiol. 65 (6), 1838-1854 (2015).
- Zettler, E. R., Mincer, T. J., Amaral-Zettler, L. A. Life in the "plastisphere": microbial communities on plastic marine debris. Environ Sci Technol. 47 (13), 7137-7146 (2013).
- Porter, K. R., Kallman, F. The properties and effects of osmium tetroxide as a tissue fixative with special reference to its use for electron microscopy. Exp Cell Res. 4 (1), 127-141 (1953).
- Ligon, J. J., Abraham, J. L., Boyd, A. S. Traumatic osmium tetroxide inoculation. J Am Acad Dermatol. 45 (6), 949-952 (2001).
- Friedova, N., et al. Osmium absorption after osmium tetroxide skin and eye exposure. Basic Clin Pharmacol Toxicol. 127 (5), 429-433 (2020).
- Fratesi, S. E., Lynch, F. L., Kirkland, B. L., Brown, L. R. Effects of SEM preparation techniques on the appearance of bacteria and biofilms in the Carter Sandstone. J Sediment. 74 (6), 858-867 (2004).
- Dassanayake, R. P., et al. Identification of a reliable fixative solution to preserve the complex architecture of bacterial biofilms for scanning electron microscopy evaluation. PLOS One. 15 (5), e0233973 (2020).
- Desiro, A., et al. Detection of a novel intracellular microbiome hosted in arbuscular mycorrhizal fungi. ISME J. 8 (2), 257-270 (2014).
- Morales, D. P., et al. Advances and challenges in fluorescence in situ hybridization for visualizing fungal endobacteria. Front Microbiol. 13, 892227 (2022).
- Shi, H., et al. Highly multiplexed spatial mapping of microbial communities. Nature. 588 (7839), 676-681 (2020).
- Valm, A. M., Welch, J. L. M., Borisy, G. G. CLASI-FISH: principles of combinatorial labeling and spectral imaging. Syst Appl Microbiol. 35 (8), 496-502 (2012).
- Fernandez-Brime, S., Muggia, L., Maier, S., Grube, M., Wedin, M. Bacterial communities in an optional lichen symbiosis are determined by substrate, not algal photobionts. FEMS Microbiol Ecol. 95 (3), fiz012 (2019).
- Schluter, S., Eickhorst, T., Mueller, C. W. Correlative imaging reveals holistic view of soil microenvironments. Environ Sci Technol. 53 (2), 829-837 (2018).
- Barcoto, M. O., Rodrigues, A. Lessons from insect fungiculture: from microbial ecology to plastics degradation. Front Microbiol. 13, 812143 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten