Method Article
Isolierung von Bodenmikroorganismen mit der iChip-Technologie
In diesem Artikel
Zusammenfassung
Die iChip-Technik nutzt ein kostengünstiges und einfaches In-situ-Isolationsgerät , das die Raten der Entdeckung neuartiger Mikroorganismen aus dem Boden erhöht. Neuartige Mikroorganismen können unter anderem für weitere Studien im Zusammenhang mit dem Bodenmikrobiom oder der Entdeckung von Naturstoffen verwendet werden.
Zusammenfassung
Bei der iChip-Isolationstechnik wird eine In-situ-Isolationsvorrichtung verwendet, die die Kultivierbarkeit von zuvor nicht kultivierbaren Mikroorganismen erhöht. Mikroorganismen sind eine wichtige Quelle für neuartige Chemikalien und potenziell bioaktive Moleküle. Allerdings kann nur 1% der Mikroorganismen aus der Umwelt mit herkömmlichen Labormethoden kultiviert werden. Mit der Zunahme von Antibiotikaresistenzen ist die Suche nach neuen Medikamenten zur Bekämpfung von Infektionen und Krankheiten von größter Bedeutung, und eine entscheidende Methode zur Suche nach neuen Molekülen ist die Entdeckung neuer Mikroorganismen. Durch die Inkubation von Kolonien von Bodenmikroorganismen in den Vertiefungen einer 96-Well-Platte, die mit einer semipermeablen Membran versiegelt und auf dem Boden inkubiert wird, kommen die Mikroben in Kontakt mit Wasser und Wachstumsfaktoren aus dem Boden, was die Isolierung neuartiger Mikroben in einer Laborumgebung ermöglicht. Nach einer Zeit der Domestizierung in einem iChip können Mikroorganismen möglicherweise auf herkömmliche Medien subkultiviert und für weitere Studien verwendet werden. Dieses Gerät ist wertvoll für die Entdeckung bioaktiver Moleküle und die Erforschung des Bodenmikrobioms und wurde bereits in beiden Anwendungen eingesetzt.
Einleitung
Umweltbakterien sind eine reichhaltige Quelle für Naturstoffe (NPs)1. Diese Metaboliten sind für das Überleben der Mikroorganismen nicht lebenswichtig, sondern werden produziert, um ihre Besiedlung zu erleichtern, indem sie andere Mikroorganismen in ihrer Umgebung verdrängen2. Die Evolution hat die chemischen Strukturen und Aktivitäten von NPs fein abgestimmt und sie zu wirksamen antimikrobiellen Wirkstoffen gemacht. Ein solches Beispiel ist Daptomycin, ein Antibiotikum, das 2003 von der FDA zugelassenwurde 3. In den letzten Jahren hat die Häufigkeit und der Schweregrad der mikrobiellen Resistenz zugenommen, und neuartige Medikamente wie Daptomycin gelten als letzte Mittel der Wahl gegen Infektionen, die Resistenzen gegen ältere Antibiotika entwickelt haben4. Die Entwicklung neuer Antibiotika und anderer Medikamente ist unerlässlich, um häufige Infektionen und Krankheiten behandelbar zu halten.
Obwohl NPs oft gute Wirkstoffe sind, wandte sich die Pharmaindustrie nach dem goldenen Zeitalter der Antibiotika in den 1950er bis 1960erJahren synthetischen Methoden für die Wirkstoffforschung zu 5. In den 1970er Jahren wurden dieselben Mikroorganismen und antimikrobiellen Metaboliten immer wieder neu isoliert, wobei immer weniger neuartige Wirkstoffkandidaten entdeckt wurden 1,6. Das Bodenmikrobiom enthält eine große mikrobielle Vielfalt, aber nur eine geringe Anzahl von Mikroorganismen kann unter herkömmlichen Laborbedingungen isoliert werden. Die überwiegende Mehrheit der Bodenmikroorganismen von Mikroorganismen, die durch Genomik nachgewiesen wurden, wird bei der Kultivierung mit herkömmlichen Kultivierungsmethoden nicht beobachtet, was dazu führte, dass das Thema als "große Plattenzahlanomalie" bezeichnet wurde7. Diese nicht kultivierbaren Mikroben wurden als mikrobielle Dunkle Materie bezeichnet, da bekannt ist, dass sie existieren, aber nicht in vitro untersucht werden können, bis sie als Reinkulturen isoliert werden können. Diese unkultivierbaren Mikroorganismen können wahrscheinlich eine Fülle von Medikamentenkandidaten produzieren, was im Zeitalter der mikrobiellen Resistenz von großer Bedeutung ist.
Die iChip-Technik ist eine Methode, mit der die Rückgewinnung neuartiger Mikroorganismen aus der Umwelt erhöht werdenkann 8,9. Diese Technologie hilft dabei, die natürliche Umgebung von Bakterien während der Inkubation zu simulieren und so die Kultivierbarkeit von Mikroorganismen zu erhöhen, die sonst unter herkömmlichen Laborbedingungen nicht überleben könnten10. Modifizierte iChips wurden bereits entwickelt und verwendet, um Mikroorganismen aus vielen verschiedenen Quellen zu isolieren, wie z. B. Boden, Sedimente, Meeresumgebungen und Tierdärme 11,12,13,14,15,16. Der vielleicht einflussreichste Fall für den Einsatz dieser Technologie war NovoBiotic Pharmaceuticals, wo ein neuartiges Bakterium, Eleftheria terrae, entdeckt wurde17. Es wurde festgestellt, dass dieser Mikroorganismus Teixobactin produziert, ein Antibiotikum einer neuen Klasse, das mehrere resistente Bakterienarten hemmt, die für die menschliche Gesundheit relevant sind, ohne dass eine Resistenzentwicklung festgestellt wird 5,18. Dies war eine wirkungsvolle Entdeckung, da Teixobactin die erste neue Klasse von Antibiotika ist, die seit Jahrzehnten entdeckt wurde, und ein Zeichen dafür ist, dass diese Technik ein vielversprechender Weg für die Entdeckung neuartiger Medikamente und die Überwindung der großen Plattenzahlanomalie19 ist. Darin wird ein modifizierter iChip vorgestellt, der auf einer früheren Veröffentlichung von Berdy et al. basiert und im Hinblick auf Benutzerfreundlichkeit und Kontaminationsvermeidung optimiert wurde9.
Protokoll
1. Medienvorbereitung und Sterilisation
- Bereiten Sie Succinat mit minimalen Salzen (SMS) vor und sterilisieren Sie es, das aus 0,1 g Kartoffelstärke, 1 g Casaminosäuren, 0,125 g Kaseinverdau und 15 g bakteriologischem oder technischem Agar in 1 l Wasser besteht.
- Sterilisieren Sie das Medium und 100 ml Wasser in molekularer Qualität im Autoklaven mit einem 20-minütigen Flüssigkeitszyklus bei 121 °C.
HINWEIS: Stellen Sie sicher, dass alle anderen Gegenstände, die mit dem Agar-Zell-Gemisch in Berührung kommen, steril gekauft werden, einschließlich Pipettenspitzen und Zentrifugenröhrchen, andernfalls autoklavieren Sie sie ebenfalls.
2. Modifizierte iChip-Konstruktion
- Entfernen Sie die Böden der Vertiefungen in vier 96-Well-Platten mit einem 5-mm-Agar-Stanzwerkzeug.
- Schneiden Sie 0,05 μm Polycarbonat-Membranen in 7,6 cm x 11 cm große Rechtecke oder entsprechen den Abmessungen des Bodens der 96-Well-Platte.
- Kleben Sie mit einem Silikondichtstoff 0,05 μm Polycarbonat-Membranen auf den Boden der 96-Well-Platten, wobei Sie darauf achten, dass der Klebstoff die Wells abdichtet, aber die Öffnungen der Wells nicht vollständig bedeckt. Mindestens 24 Stunden trocknen lassen oder den Anweisungen auf dem Kleber folgen.
HINWEIS: Das Dichtmittel muss wasserdicht, ungiftig und zu 100 % aus Silikon bestehen. Die meisten Aquarienversiegelungen funktionieren gut.
3. Herstellung von Zellsuspensionen
- Beschriften Sie vier 15-ml-Zentrifugenröhrchen A-D und vier 50-ml-Zentrifugenröhrchen E-H und fügen Sie jeweils 4,5 mL steriles Wasser hinzu.
- Messen Sie 1 g Erde in ein 50-ml-Zentrifugenröhrchen, fügen Sie 10 mL steriles Wasser hinzu und wirbeln Sie es 10 Minuten lang auf.
- Lassen Sie die Bodensuspension 10 Minuten einwirken.
- 0,5 ml des Überstands, der Zellen aus dem Boden enthält, in das Röhrchen A pipettieren und gründlich mischen.
- Geben Sie 0,5 mL der Zellsuspension in Röhrchen A in Röhrchen B und mischen Sie es gründlich. 0,5 ml Lösung B in Röhrchen C überführen und gründlich mischen. Wiederholen Sie diesen Vorgang für alle verbleibenden Zentrifugenröhrchen und führen Sie eine Reihe von 10-fachen Verdünnungen über die acht Zentrifugenröhrchen durch.
- Entfernen Sie 0,5 mL aus Röhrchen H, um in allen Röhrchen das gleiche Volumen zu haben.
HINWEIS: Diese Konzentrationen sind für die lokalen Böden optimiert. Wenn Sie dieses Verfahren zum ersten Mal ausprobieren, führen Sie einen größeren Verdünnungsbereich durch, um die geeignete Probenkonzentration zu finden.
4. Modifizierte iChip-Inokulation
- Tauchen Sie die iChips für mindestens 15 min vollständig in 95%iges Ethanol.
- Nehmen Sie die Platten vom Ethanol und legen Sie sie auf ein steriles Papiertuch. Während Sie das Ethanol verdampfen lassen, schalten Sie den UV-Sterilisator in der Laminar-Flow-Haube für 15 Minuten ein, um sie weiter zu sterilisieren.
- Pipettieren Sie 360 μl sterile SMS-Medien in die erste Säule der Platte, um als Kontrollvertiefungen zu dienen.
- Geben Sie 45 ml auf 50 °C gekühltes SMS in die Zellsuspension im Röhrchen E und mischen Sie gründlich, um die Zellsuspension und den Agar zu kombinieren.
- Pipettieren Sie 360 μl des Agar-Zell-Gemisches aus Schritt 4.4 in alle anderen Vertiefungen der 96-Well-Platte.
HINWEIS: Eine Mehrkanalpipette wird empfohlen, da sich der Agar schnell verfestigt. Es wird nicht empfohlen, das Agar-Zell-Gemisch nach dem Mischen erneut zu erhitzen, da die Gefahr besteht, dass die Mikroorganismen abgetötet werden. - Sobald das Medium ausgehärtet ist, versiegeln Sie die Oberseite der Platten mit einer PCR-Plattenabdeckung.
- Wiederholen Sie die Schritte 3.3 bis 3.6 mit den Röhrchen F-H, um insgesamt vier iChips mit zehnfachen Konzentrationsunterschieden zu füllen.
HINWEIS: Das Medium kann bei Bedarf 30 s lang mit einer gelösten Kappe in der Mikrowelle erhitzt oder in einem 60 °C heißen Wasserbad zwischen den Platten aufbewahrt werden, um sicherzustellen, dass es für jede Platte geschmolzen bleibt.
5. Inkubation
- Legen Sie die Platten mit der Membranseite nach unten in einen Kasten, der etwa einen Zentimeter des in Schritt 3.2 verwendeten Bodens enthält, und setzen Sie die Abdeckung ein.
- Die Platten im abgedeckten Behälter an einem dunklen Ort bei 25 °C inkubieren.
HINWEIS: Untersuchen Sie nach einer Woche den Inhalt der modifizierten iChips, um sicherzustellen, dass es während der Einrichtung nicht zu einer Kontamination gekommen ist. Eine Kontamination wird durch das übermäßige Wachstum eines einzelnen oder weniger Mikroorganismen in allen Vertiefungen einer Platte angezeigt. - Untersuchen Sie nach 6 Wochen die iChips. Verwenden Sie die Platten, die das Wachstum in weniger als 25 % der Vertiefungen enthalten, für die Isolierung der Kolonie.
6. Isolierung der Kolonie
- Spülen Sie die modifizierten iChips 3x mit sterilem Wasser ab, um alle Schmutzpartikel zu entfernen.
- Wischen Sie die Oberseite und die Seiten der Platte mit 95 % Ethanol ab und vermeiden Sie dabei die Seite mit der halbdurchlässigen Membran.
- Schneiden Sie mit einer sterilen Klinge die Plattenabdeckung um eine Vertiefung mit einer Kolonie durch.
- Mit einem sterilen Streifenwerkzeug wird die Kolonie und das Streifen auf den SMS-Agar in 100 x 15 mm großen Platten im Vier-Quadranten-Verfahren durchstochen.
- Wiederholen Sie dies für alle anderen Völker in den iChips, die eine Kolonie pro Vertiefung wachsen.
HINWEIS: Bei winzigen Kolonien kann die Platte wieder auf den Boden gesetzt und so lange inkubiert werden, bis sie groß genug ist, um eine Subkultur durchzuführen, solange die Plattenabdeckung nicht um den Brunnen herum entfernt wurde. - Inkubieren Sie die SMS-Platten bei 25 °C und überwachen Sie das Wachstum.
- Untersuche Kolonien, um sicherzustellen, dass axenische Kulturen isoliert wurden.
7. Identifizierung von Mikroorganismen
- Sequenzieren Sie die Genome der wiedergefundenen Isolate20.
- Bewerten Sie die Genome mit JSpeciesWS oder einer anderen genombasierten Identifizierungsplattform, um den Grad der Ähnlichkeit jedes Isolats mit bekannten Mikroorganismen in GenomesDB zu bestimmen.
- Kategorisierung von Isolaten, die unterhalb der von der Gemeinschaft akzeptierten Schwellenwerte für die Identifizierung von Arten als vermeintlich neu liegen.
Ergebnisse
Eine visuelle Übersicht über das Protokoll ist in Abbildung 1 dargestellt. Ein erfolgreiches modifiziertes iChip-Experiment führt dazu, dass mindestens eine Platte in weniger als 25 % der Wells Wachstum aufweist, während in den Kontrollwells kein Wachstum stattfindet. Diese Anzahl von Brunnen mit Wachstum stellt in der Regel sicher, dass einzelne Völker in Vertiefungen isoliert werden. Zusätzlich sollte die Anzahl der Kolonien oder die Anzahl der Vertiefungen, die Kolonien enthalten, mit jeder weiteren Platte, die aus der Verdünnungsreihe hergestellt wird, um das Zehnfache abnehmen. Repräsentative Ergebnisse sind in Abbildung 2 dargestellt. wobei die Platten 7 und 8 erfolgreiche iChips sind, da sie das Wachstum in weniger als 25 % der Vertiefungen enthalten, und der Prozentsatz der Vertiefungen mit Kolonien mit jeder Platte abnimmt, wie in Tabelle 1 gezeigt. Wenn alle Platten in einem Versuch mehr als eine Kolonie pro Vertiefung oder keine Völker in einer Vertiefung enthalten, stellt dies ein negatives Ergebnis dar.
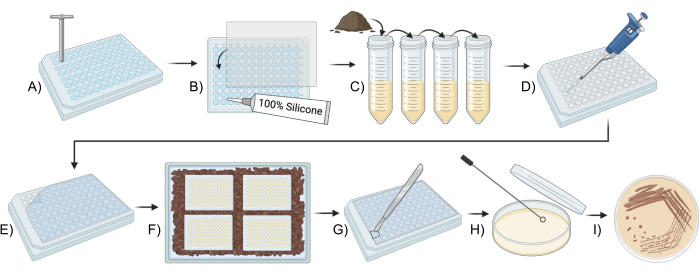
Abbildung 1: Schematische Übersicht für die Verwendung eines modifizierten iChips zur Kultivierung von Bodenmikroben. (A,B) Aufbau der Platte (Schritt 2). (C) Vorbereitung des Inokulums (Schritt 3). (D,E) iChip-Einrichtung (Schritt 4). (F) Inkubation des iChips (Schritt 5). (G-I) Überführung der Isolate in herkömmliche Medien (Schritt 6). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Ichip (Englisch) | Konzentration des Bodens in Agar-Suspension | Brunnen mit Kolonien | Koloniebildende Einheiten |
| E | 5,0 x 10-6 g/ml | Alle | Zu viele, um sie zu zählen |
| F | 5,0 x 10-7 g/ml | Alle | Zu viele, um sie zu zählen |
| G | 5,0 x 10-8 g/ml | 22 | 22 |
| H | 5,0 x 10-9 g/ml | 6 | 6 |
Tabelle 1: Repräsentative Daten aus einem erfolgreichen iChip-Experiment, bei dem die Anzahl der Vertiefungen mit Kolonien mit jeder Verdünnung in Reihe abnimmt.
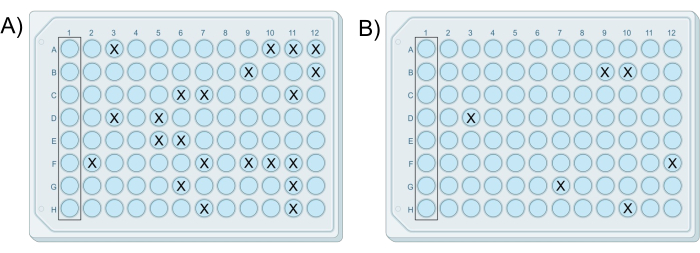
Abbildung 2: Beispielhafte Ergebnisse von zwei erfolgreich modifizierten iChips. Die iChips stammten aus demselben Experiment, in dem (A) die aus Zell-Agar-Gemisch G hergestellte Platte 22 Kolonien enthält und (B) die aus Zell-Agar-Gemisch H hergestellte Platte sechs Kolonien enthält, was mit der 10-fachen Verdünnungsdifferenz zwischen den Mischungen übereinstimmt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Man kann Kolonien mit bloßem Auge oder mikroskopisch sichtbar machen, wie in Abbildung 3A-G gezeigt. Kolonien können auf der Oberfläche des Agars wachsen, wie in Abbildung 3B zu sehen, oder in den Agar eingebettet sein, wie die Kolonie in Abbildung 3F. Es kann Fälle geben, in denen sich zwei Kolonien in derselben Vertiefung in einem erfolgreichen modifizierten iChip befinden, wie in Abbildung 3G. In einigen Fällen sind die Völker weit genug voneinander entfernt, dass sie separat mit einer Nadel durchstochen und subkultiviert werden können; Es ist jedoch unwahrscheinlich, dass die in Abbildung 3G gezeigten Mikroorganismen leicht als axenische Mikroorganismen kultiviert werden können. Beispiele für Kolonien, die vom iChip auf herkömmliche SMS-Medien übertragen wurden, sind in Abbildung 4 dargestellt.
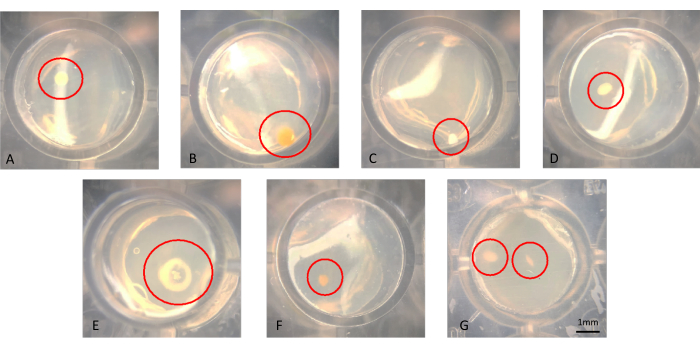
Abbildung 3: Repräsentative mikroskopische Ansicht von Völkern, die nach 6-wöchiger Inkubation in einem iChip wachsen. (A-F) Es werden einzelne Völker beobachtet, aus denen subkultiviert werden sollte, und in (G) werden zwei Völker in unmittelbarer Nähe beobachtet, die als axenische Kulturen schwer zu gewinnen sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
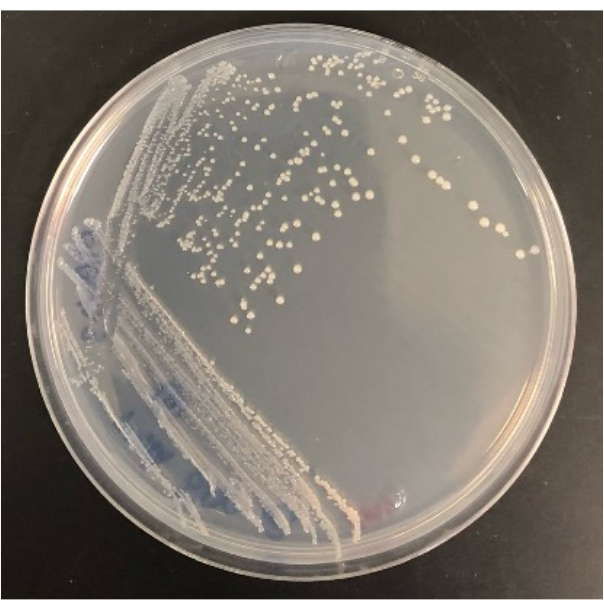
Abbildung 4: Repräsentatives Ergebnis eines Bakteriums, das aus dem modifizierten iChip isoliert wurde, nachdem es auf einer SMS-Agarplatte gewachsen war. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Auch die Möglichkeit, Kolonien aus dem modifizierten iChip auf herkömmliche Agarplatten zu isolieren, entscheidet über den Erfolg des Experiments. Nicht alle Völker, die im modifizierten iChip wachsen, überleben den Transfer auf herkömmliche Platten. In einem iChip werden die Völker Wachstumsfaktoren und Bodennährstoffen ausgesetzt, wodurch ihre Kultivierbarkeit erhöht wird. Für einige Mikroorganismen reicht eine Zeit der Domestizierung nicht aus, um sie unter herkömmlichen Bedingungen kultivierbar zu machen. Die Rückgewinnungsrate von Kolonien von einem iChip auf Platten variiert von Experiment zu Experiment, aber einige sollten in Platten wachsen, da diese Methode auch leicht zu kultivierende Mikroorganismen kultivieren kann. Zum Beispiel enthielten die modifizierten iChips in Abbildung 2 insgesamt 28 Kolonien, und die Anzahl der Kolonien, die nach der Subkultur auf Agarplatten wuchsen, betrug 16, während 12 den Transfer nicht überlebten. Isolate können identifiziert und für weitere Studien verwendet werden, sobald sie auf Agarplatten in voller Größe kultiviert wurden, wie in Abbildung 4 gezeigt.
Ein erfolgreiches modifiziertes iChip-Experiment sollte zur Isolierung von Mikroorganismen führen, die sich auf Speziesebene von bekannten Mikroorganismen unterscheiden. Der Grad der Ähnlichkeit jedes Isolats mit bekannten Mikroorganismen wird durch den Vergleich der isolierten DNA mit den Genomen bekannter Mikroorganismen ermittelt. Die Tetra-Z-Werte der am nächsten verwandten Mikroorganismen zu jedem Isolat beschreiben die prozentuale Ähnlichkeit. Abbildung 5 zeigt, dass von den zwölf isolierten Bakterien vier neue Arten oder höher, zwei wahrscheinlich neue Stämme und sechs bekannte Mikroorganismen waren. Die Wiederfindung mehrerer neuer Spezies oder Stämme stimmt mit zuvor veröffentlichten iChip-Experimenten überein, was darauf hindeutet, dass diese modifizierte iChip-Konstruktion die Steigerung der Wiederfindung neuer Spezies nicht behindert11.
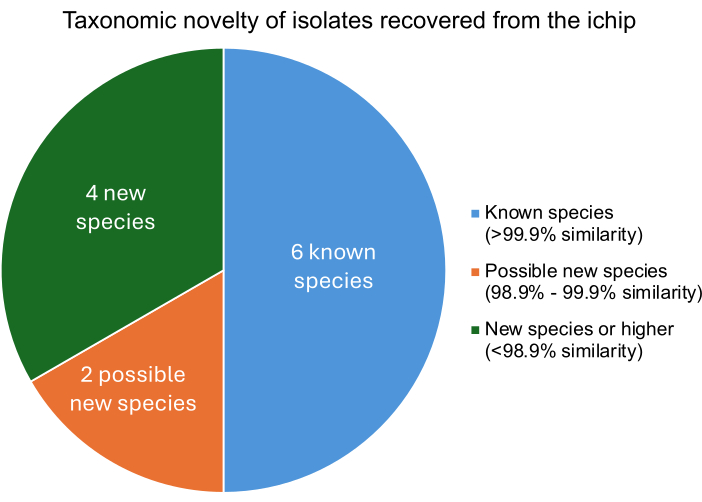
Abbildung 5: Die taxonomische Klassifizierungsstufe von Isolaten, die aus einem iChip-Experiment gewonnen wurden. Die prozentuale Ähnlichkeit wurde auf der Grundlage der Tetra-Z-Scores bestimmt, die die Ähnlichkeit der Isolate mit bekannten Bakterien beschreiben, wobei >99,9 % Ähnlichkeit auf eine Übereinstimmung der Spezies, 98,9 % bis 99,9 % Ähnlichkeit auf eine mögliche neue Spezies und <98,9 % Ähnlichkeit auf eine neue Spezies oder höher hinweist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Zahlreiche Methoden, wie z. B. Genome Mining und die Untersuchung stiller Biosynthesewege, haben in den letzten Jahren die Entdeckung neuartiger bioaktiver Chemikalien erleichtert21,22. NPs, die mit solchen Methoden entdeckt werden, weisen jedoch oft eine hohe strukturelle Ähnlichkeit mit bekannten Verbindungen auf. Der Zugang zu bisher unkultivierten Mikroorganismen wird eine größere chemische Vielfalt und NPs mit neuartigen Wirkmechanismen freisetzen, die besser bei der Bekämpfung mikrobieller Resistenzen helfen können. Es hat sich gezeigt, dass die iChip-Isolationstechnik die Kultivierbarkeit neuartiger Mikroorganismen erhöht, die zum Aufbau von Bibliotheken von Mikroorganismen für die Entdeckung neuartiger NPs verwendet werden können.
Die früheste Konzeptualisierung eines iChips wurde 2002 veröffentlicht, der aus einzelnen Metallringen bestand, die eine Agarzellmischung enthielten, die mit semipermeablen Membranen versiegelt und in situinkubiert wurde 23. Später wurde er im Jahr 2010 iteriert, um einen kleinen Chip mit vielen Wells11 zu enthalten, gefolgt von der Entwicklung eines kostengünstigen iChips, der aus gängigen Labormaterialien in einer einflussreichen Nature Protocols-Veröffentlichung im Jahr 2017hergestellt wurde 9. In dieser Veröffentlichung wurden mehrere Änderungen am Nature-Protokoll vorgenommen, um seine Praktikabilität und Benutzerfreundlichkeit zu verbessern. Beim Nature-Protokoll wird eine semipermeable Membran auf beide Seiten eines Chips geklebt, im Vergleich zu dieser Methode, bei der auf einer Seite eine Klebeplattenabdeckung verwendet wird. Bei der Verwendung von Silikonkleber zur Befestigung der Membran nach dem Laden des iChips kann es zu Problemen kommen. Unseres Wissens geben alle ungiftigen Silikonklebstoffe beim Abbinden Essigsäure ab, die sich auf die Lebensfähigkeit der Zellen auswirken kann24. Das in diesem Video beschriebene Protokoll reduziert auch den Aufwand für die Handhabung nach der Inokulation erheblich, indem die PCR-Plattenabdeckung zur Versiegelung der gefüllten Platte verwendet wird, wodurch die Sterilität weiter gewährleistet und die Einrichtungszeit verkürzt wird. Basierend auf der Identifizierung mehrerer neuartiger Isolate aus diesem Experiment hemmt die Verwendung von nur einer einzigen semipermeablen Membran die Zunahme der Entdeckungsraten neuartiger Organismen, die für iChips berichtet wurde, die mit zwei semipermeablen Membranen konstruiert wurden. Es wären jedoch weitere Experimente mit einer größeren Stichprobengröße erforderlich, um die Auswirkungen zu quantifizieren.
Eine weitere Modifikation der Methode besteht darin, nur Subkulturen aus den modifizierten iChips zu verwenden, die das Wachstum in weniger als 25 % der Wells enthalten. Wenn Kolonien in den meisten Vertiefungen einer Platte wachsen, gibt es wahrscheinlich einige Vertiefungen, die mehrere Mikroorganismen enthalten, auch wenn sie nicht leicht sichtbar sind. Bei der Methodenentwicklung wurde festgestellt, dass es weitgehend unmöglich war, axenische Kulturen zu erhalten, wenn aus Brunnen mit mehr als einer Kolonie subkultiviert wurde. Die Isolierung von nicht-axenischen Kulturen stellt nachgelagerte Kulturen in Bezug auf Bioaktivitätsassays und -identifizierung erhebliche Probleme dar. Der Einfachheit halber wird daher empfohlen, nur modifizierte iChips mit Kolonien, die in einem Teil der Wells wachsen, zu subkultivieren.
Das größte Problem, das bei iChip-Methoden auftreten kann, ist die Kontamination von Vertiefungen oder ganzen iChips mit anderen Mikroorganismen. Eine Kontamination wird durch Kolonien angezeigt, die in den Kontrollvertiefungen wachsen, oder durch denselben Mikroorganismus, der über mehrere Vertiefungen oder einen ganzen Bereich des modifizierten iChips wächst. Die Kontaminationsquelle kann auf eine unzureichende Sterilisation der Materialien während der Einrichtung oder eine unsachgemäße aseptische Technik zurückzuführen sein. Stellen Sie in solchen Fällen sicher, dass alle verwendeten Materialien wie in der Methode angegeben im Autoklaven oder Ethanol sterilisiert sind, und stellen Sie sicher, dass kein Kontakt zwischen unsterilen Gegenständen und dem modifizierten iChip mit Ausnahme des Agar-Zell-Gemischs auftritt. Wenn ein Überwachsen eines einzelnen Mikroorganismus am Boden oder an der Oberseite mehrerer modifizierter iChip-Wells beobachtet wird, ist dies höchstwahrscheinlich das Ergebnis einer unvollständigen Abdichtung zwischen den Wells und der Membran. Stellen Sie in diesem Fall sicher, dass der verwendete Klebstoff zu 100 % aus Silikon besteht, das sich nicht in Ethanol abbaut, und stellen Sie sicher, dass die semipermeable Membran und die PCR-Plattenabdeckung während des Baus vollständig um jede Vertiefung herum versiegelt sind.
Die im aktuellen Protokoll verwendete Verdünnungsreihe sollte für die meisten Bodentypen mit einer Lagerdauer von weniger als einer Woche eine angemessene Verdünnung bieten, da sie einen tausendfachen Bereich von Verdünnungen umfasst. Es kann jedoch zu erheblichen Unterschieden in der Anzahl der Mikroorganismen zwischen den Bodentypen kommen. Wenn die Kontrollwells kein Wachstum enthalten, aber in jeder Vertiefung aller vier modifizierten iChips mehrere Kolonien wachsen, waren die verwendeten Zellkonzentrationen nicht niedrig genug. Die Verdünnungsreihe sollte modifiziert werden, um niedrigere Zellkonzentrationen in den Zell-Medien-Gemischen zu erreichen, die zum Aufbau der modifizierten iChips verwendet werden. Wenn in keiner dieser Platten ein Wachstum beobachtet wird, sollten die in den Zell-Medien-Gemischen verwendeten Konzentrationen erhöht werden. Alternativ ist es möglich, dass die Temperatur des verwendeten Mediums zu hoch für das Überleben von Mikroorganismen war. In einem solchen Fall sollte das Medium so weit wie möglich abkühlen gelassen werden, ohne sich zu verfestigen, bevor es der verdünnten Zellsuspension zugesetzt wird.
Die Technologie ist ein wichtiger Fortschritt zur Überwindung der großen Plattenzahlanomalie. Sie ist jedoch immer noch durch die Untauglichkeit herkömmlicher Kultivierungstechniken begrenzt, was sich an der Anzahl der Mikroorganismen zeigt, die den Transfer vom modifizierten iChip auf herkömmliche Agarplatten nicht überleben. In früheren Veröffentlichungen wurde berichtet, dass mehrere Runden der Subkultur und Inkubation in iChips die Kultivierbarkeit der Mikroorganismen weiter erhöhen. Die verlängerte Domestizierungszeit und die Exposition gegenüber Bodenwachstumsfaktoren während der Einnahme von Agar im iChip erhöhen die Chancen, dass eine Kolonie nur auf Agar wächst. Es wurde jedoch nicht berichtet, dass dieser Ansatz eine höhere Wahrscheinlichkeit für neuartige Mikroorganismen aufweist als eine einzelne iChip-Inkubation25,26.
Viele andere Taktiken werden erforscht und modifiziert, um die Kultivierbarkeit neuartiger Mikroorganismen zu erhöhen. Zum Beispiel wurde vorgeschlagen, eine ganze iChip-Vorrichtung aus einem semipermeablen Material herzustellen, um die Co-Kultur von Mikroorganismen in benachbarten Vertiefungen27 zu erleichtern. Ein Vorteil der Konstruktion, der in dieser Veröffentlichung beschrieben wird, sind die geringen Kosten, wobei die Kosten für den Bau einer Platte etwa 12 US-Dollar entsprechen (4 US-Dollar pro 96-Well-Platte, 8 US-Dollar pro Membran, 2 US-Dollar pro PCR-Abdeckung). Darüber hinaus ist es durch seinen einfachen Aufbau ein unkompliziertes Werkzeug, wenn es wie beschrieben verwendet wird und bietet viele Möglichkeiten zur Individualisierung. Obwohl dieses Protokoll ein für Bakterien selektives Medium verwendet, kann der Versuchsaufbau theoretisch so abgestimmt werden, dass er auf eine gewünschte mikrobielle Population abzielt, indem das verwendete Medium modifiziert wird, z. B. die Verwendung von bakterienunterdrückenden Medien zur Bekämpfung von Pilzen oder nährstoffarmer Agar für sporulierende Mikroorganismen.
Offenlegungen
Die Autoren haben erklärt, keine Interessenkonflikte zu haben.
Danksagungen
Wir danken für die J-Base-Finanzierung (J-001757, J-001842), die von Agriculture and Agri-Food Canada bereitgestellt wurde und dieses Projekt ermöglicht hat. Wir danken Brett van Heyningen für das Filmen des Videoinhalts für dieses Protokoll. Wir möchten uns auch bei Ron Matters für das Sammeln von Bodenproben bedanken, die in den in dieser Veröffentlichung beschriebenen Experimenten verwendet wurden.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
Referenzen
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten