Method Article
Kompetitives Risiko-Nomogramm zur Vorhersage des krebsspezifischen Überlebens bei mehreren primären Darmkrebspatienten nach einer Operation
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Studie ergab, dass das männliche Geschlecht, der schlechte Tumorgrad und das fortgeschrittene Stadium der Tumorknotenmetastasierung bei Patienten mit mehreren primären Darmkrebserkrankungen (MPCC) nach der Operation mit einem schlechteren krebsspezifischen Überleben (CSS) verbunden waren. Wir haben ein Nomogramm entwickelt, um das CSS von MPCC-Patienten vorherzusagen und zur Entscheidungsfindung in der klinischen Behandlung beizutragen.
Zusammenfassung
Das krebsspezifische Überleben (CSS) bei Patienten mit multiplem primärem Darmkrebs (MPCC) wird kompetitiv durch den Tod aus anderen Ursachen beeinflusst. Ziel dieser Studie war es, CSS und die damit verbundenen Risikofaktoren durch konkurrierende Risikoanalysen bei MPCC-Patienten zu untersuchen. Die Daten dieser Studie stammen aus der SEER-Datenbank. Unter Verwendung einer univariablen und multivariablen Analyse des kompetitiven Risikomodells, um die Auswirkungen von Wettbewerbsereignissen abzuschwächen, untersucht es die Risikofaktoren von CSS und entwickelt ein Nomogrammmodell. Anschließend wird die Leistung des Modells durch die ROC-Kurve, die Kalibrierungskurve und den DCA überprüft. Die Studie umfasst insgesamt 8931 Patienten, davon 6255 in der Trainingskohorte und 2676 in der Validierungskohorte. Univariable und multivariable Analysen zeigten, dass das Geschlecht, das Stadium der Tumorknotenmetastasierung (TNM) und der Tumorgrad unabhängige Risikofaktoren für das krebsspezifische Überleben bei MPCC-Patienten sind. Basierend auf den Risikofaktoren haben wir ein Diagrammmodell zur Vorhersage von CSS entwickelt. ROC-Kurve, Kalibrierungskurve und DCA zeigen ebenfalls gute Ergebnisse. Zusammenfassend wird ein Nomogramm entwickelt, das als wertvolles Instrument zur Vorhersage von CSS bei MPCC-Patienten dient und Klinikern wichtige Erkenntnisse für die personalisierte Behandlungsplanung liefert.
Einleitung
Darmkrebs, als einer der weltweit häufigsten Tumoren des Verdauungssystems, hat in den letzten Jahrzehnten einen kontinuierlichen Anstieg seiner Inzidenz erlebt. Die anhaltend hohen Inzidenz- und Mortalitätsraten haben große Aufmerksamkeit erregt. Nach den neuesten Statistiken steht Darmkrebs weltweit an dritter Stelle unter den häufigsten bösartigen Tumoren, die Sterblichkeitsrateliegt weltweit an zweiter Stelle. Der Multiple Primäre Darmkrebs (MPCC) ist eine spezielle Unterform des Darmkrebses, die in den letzten Jahren zunehmend in den Fokus der Forschung gerückt ist. Es ist definiert als die Diagnose von zwei oder mehr unabhängigen Darmkrebsläsionen bei ein und demselben Patienten mit einem Abstand von mehr als 5 cm zwischen den Rändern der Läsionen. Wenn bei MPCC mehrere Tumoren gleichzeitig oder innerhalb von weniger als 6 Monaten diagnostiziert werden, wird es als synchroner Darmkrebs (SCRC) definiert, während es bei einem Intervall zwischen den Diagnosen von mehr als 6 Monaten als metachroner Darmkrebs (MCRC) definiert wird2,3,4,5.
Der Anteil von MPCC bei allen kolorektalen Karzinomen ist relativ gering, und die Berichte über die Inzidenz variieren je nach Region und Studien. Jüngste Studien haben berichtet, dass MPCC zwischen 2 % und 10 % der Darmkrebserkrankungen ausmacht 2,3,4,5. Im Vergleich zum solitären kolorektalen Karzinom hat MPCC eine schlechtere Prognose 3,6. Derzeit stützt sich die klinische Beurteilung der MPCC-Prognose hauptsächlich auf das Tumorlymphknotenmetastasierungssystem (TNM) des American Joint Committee of Cancer, das das am weitesten fortgeschrittene Stadium multipler Läsionen als tatsächliches Stadium der MPCC verwendet. Es ist jedoch nicht ausreichend, Prognosevorhersagen ausschließlich auf TNM-Staging zu stützen. Es fehlt immer noch an einem effektiven Instrument, um die Prognose der MPCC nach der Operation vorherzusagen. In der heutigen Ära der Präzisionsmedizin werden klinische Vorhersagemodelle zur Quantifizierung von Risiken häufig in der klinischen Entscheidungsfindung und bei der Beurteilung der Patientenprognose eingesetzt 7,8. In den letzten Jahren haben sich Nomogramme, die auf unabhängigen prognostischen Risikofaktoren basieren, für die Vorhersage der Tumorprognose weitgehend durchgesetzt9. Nomogramme können komplexe statistische Modelle visualisieren, wodurch sie im klinischen Umfeld leichter angewendet werden können. Bei der Beurteilung der Tumorprognose sind das Gesamtüberleben (OS) und das krebsspezifische Überleben (CSS) häufig verwendete Outcome-Indikatoren9. OS bezieht sich auf die Zeit von der Bestätigung einer Krebserkrankung bei einem Patienten bis zum Tod aus beliebiger Ursache. CSS bezieht sich auf die Zeit von der Diagnose eines Tumors bis zum Tod, der spezifisch durch die Krebserkrankung verursacht wird, und bietet eine genauere Abbildung des Risikos, an der Krebserkrankung zu sterben. Wenn CSS als Ergebnisindikator verwendet wird, können Todesfälle, die durch andere Faktoren verursacht werden, die Wahrscheinlichkeit einer krebsspezifischen Mortalität beeinflussen, was zu einer konkurrierenden Risikobeziehung zwischen den beiden Ereignissen führt10,11. Daher sollten Studien zum tumorspezifischen Überleben konkurrierende Risikomodelle verwenden, um die Auswirkungen konkurrierender Ereignisse zu eliminieren. Frühere Studien haben Modelle zur Vorhersage der Prognose von MPCC erstellt, aber diese waren auf synchronen Darmkrebs beschränkt und haben keine konkurrierenden Risikomodelle verwendet, um den Einfluss konkurrierender Ereignisse auf CSS zu berücksichtigen 12,13,14.
In dieser Studie untersuchten wir die konkurrierenden unabhängigen Risikofaktoren, die das krebsspezifische Überleben von MPCC nach einer Operation beeinflussen. Die Begründung für die Verwendung eines konkurrierenden Risikomodells ergibt sich aus seiner Fähigkeit, die Möglichkeit zu berücksichtigen, dass Patienten an anderen Ursachen als Krebs sterben können, was für die Erlangung unvoreingenommener Überlebensschätzungen entscheidend ist15,16. Herkömmliche Überlebensanalysetechniken, wie z. B. das Cox-Modell, können die Überlebenswahrscheinlichkeiten bei konkurrierenden Ereignissen überschätzen, so dass die Verwendung konkurrierender Risikomodelle in diesen Szenarien angemessener ist17.
Basierend auf den identifizierten unabhängigen Risikofaktoren haben wir ein Nomogramm erstellt, um die Überlebenswahrscheinlichkeit vorherzusagen und seine Leistung zu validieren. Nomogramme haben im klinischen Umfeld an Popularität gewonnen, da sie eine benutzerfreundliche grafische Darstellung komplexer statistischer Modelle bieten, die es Klinikern ermöglicht, individuelle Risikobewertungen einfach zu berechnen18,19. Im Gegensatz zu anderen prädiktiven Instrumenten berücksichtigen Nomogramme mehrere Risikofaktoren und liefern genauere, personalisierte Prognoseschätzungen. Dieser Ansatz wurde zunehmend bei verschiedenen Krebsarten validiert und zeigt eine überlegene Leistung gegenüber herkömmlichen Staging-Systemen 20,21,22. Unser Tool zielt darauf ab, Ärzte dabei zu unterstützen, personalisierte und genauere Prognosen während der Diagnose und Behandlung vorzunehmen und so die Entscheidungsfindung bei der Behandlung von MPCC zu verbessern.
Protokoll
Diese Studie umfasst zwei Schritte. Zunächst wurden klinische Daten und Überlebensdaten von MPCC aus der SEER-Datenbank gewonnen. Anschließend wurde die R-Software (Version 4.3.3) verwendet, um ein konkurrierendes Risikomodell zu analysieren und zu konstruieren. Das Workflow-Diagramm der Studie ist in Abbildung 1 dargestellt. Für diese Studie ist keine ethische Genehmigung und Zustimmung erforderlich, um teilnehmen zu können. Die in dieser Studie verwendeten Daten wurden aus Datenbanken gewonnen.
1. Datenerfassung
- Laden Sie die SEER. Stat 8.4.3 von der SEER-Datenbank-Website (http://seer.cancer.gov/about/overview.html). Registrieren Sie sich und melden Sie sich bei SEER an. Stat 8.4.3, um relevante Patientendaten zu erhalten.
- Nachdem Sie sich bei SEER. Stat 8.4.3, klicken Sie auf Case Listing Session > Data und wählen Sie die Datenbank Inzidenz SEER Research Data, 17 Registries, Nov 2022 Sub (2000-2020) aus.
- Klicken Sie auf Auswahl > Bearbeiten und wählen Sie {Rasse, Geschlecht, Jahr Dx. Jahr der Diagnose} = '2004', '2005', '2006', '2007', '2008', '2009', '2010', '2011', '2012', '2013', '2014', '2015' UND {Standort und Morphologie. Neukodierung der Stelle ICD-O-3/WHO 2008} = 'Dickdarm und Rektum' AND {Lokalisation und Morphologie. Diagnostische Bestätigung} = 'Positive Histologie' UND {Mehrere Primärfelder. Laufende Nummer}! = 'Nur eine Primärperson'.
- Klicken Sie auf OK und speichern Sie die Auswahl. Klicken Sie auf Tabelle und wählen Sie in der Benutzeroberfläche Verfügbare Variablen die Option Altersrekodierung mit Einzelalter und 85+, Geschlecht, Standort-Rekodierung ICD-O-3/WHO 2008, CS-Tumorgröße, Grad-Rekodierung (bis 2017), Abgeleitetes AJCC T, 6. Auflage (2004-2015), Abgeleitetes AJCC N, 6. Aufl. (2004-2015), Abgeleitetes AJCC M, 6. Aufl. (2004-2015), Strahlenrekodierung, Chemotherapie-Rekodierung (ja, nein/unk), SEER-ursachenspezifische Todesklassifikation, SEER Klassifikation der anderen Todesursache, Überlebensmonate, Patienten-ID, RX Summ-Surg Prim Site (1998+) und klicken Sie auf Spalte.
- Klicken Sie auf Ausgabe, benennen Sie die Daten und klicken Sie auf Ausführen , um die Daten auszugeben und zu speichern.
- Die SEER-Datenbank gibt nicht direkt Auskunft darüber, ob ein Patient MPCC hat. Nachdem Sie den Datendownload wie oben beschrieben abgeschlossen haben, verwenden Sie die Patienten-ID, um Patienten mit MPCC herauszufiltern, d. h. solche, bei denen zwei oder mehr Fälle diagnostiziert wurden. Berechnen Sie nach der Identifizierung von MPCC-Patienten das Intervall zwischen den Tumordiagnosen basierend auf ihren Überlebenszeiten. Klassifizieren Sie die Patienten anhand von 6 Monaten in SMPCC und MMPCC.
- Kategorisieren Sie das Alter der Patienten basierend auf ihrem Alter zum Zeitpunkt der Erstdiagnose als = 65 Jahre und> 65 Jahre. Für den Tumorgrad entspricht Grad I einer guten Differenzierung, Grad II entspricht einer moderaten Differenzierung, Grad III entspricht einer niedrigen Differenzierung und Grad IV entspricht einer Undifferenzierung. Klassifizieren Sie die Tumorlokalisation basierend auf der Verteilung mehrerer Tumoren als rechter Dickdarm, linker Dickdarm und gesamter Dickdarm.
- Es gibt offensichtliche Unterschiede in der Embryonalentwicklung und den biologischen Eigenschaften zwischen dem proximalen und dem distalen Kolorektum, die die Tumorlokalisation auf der Grundlage der Milzflexurteilen 23. Der rechte Dickdarm ist als proximal der Milzflexur definiert, einschließlich des Blinddarms, des aufsteigenden Dickdarms, der Leberflexur und des Dickdarms transversal, während das linke Kolorektum die Milzflexur, den Colon descending, das Colon sigmoideus, den Übergang rectosigmoideus und das Rektum umfasst. Wenn sich die Tumoren eines Patienten vollständig im rechten Dickdarm befinden, definieren Sie die Tumorposition als rechten Dickdarm; Wenn sich alle Tumoren im linken Dickdarm befinden, definieren Sie die Lokalisation als linkes Dickdarm; Wenn es Tumore sowohl im rechten Dickdarm als auch im linken Dickdarm gibt, definieren Sie die Lokalisation als gesamtes Dickdarm.
- Bestimmen Sie die Tumorgröße, indem Sie den größten Tumordurchmesser unter mehreren Tumoren im selben Patienten auswählen. Basierend auf dem Tumordurchmesser kategorisieren Sie die Größe als = 5 cm oder >5 cm. Insgesamt wurden 8.931 Patienten in die Studie eingeschlossen. Um eine Trainingskohorte und eine Validierungskohorte zu erstellen, dividieren Sie alle Fälle nach dem Zufallsprinzip in einem Verhältnis von 7:3.
2. Modellbau
- Laden Sie RStudio (2023.12.1+402) und die R-Software (4.3.3) herunter. Öffnen Sie RStudio, um die R-Software auszuführen. Klicken Sie auf Neue Datei und wählen Sie R-Skript aus, um eine neue R-Programmierschnittstelle zu erstellen. Geben Sie den entsprechenden Code in den Code-Editor ein und klicken Sie auf Ausführen , um den Code auszuführen.
HINWEIS: Die Sprache R und ihre Funktionen bieten eine Vielzahl von Parametern. Das Hinzufügen oder Ändern dieser Parameter kann die Datenanalyse und -visualisierung verbessern. - Verwenden Sie den folgenden Code, um eine univariate Analyse durchzuführen und die CIF-Kurve darzustellen.
Bibliothek(TidyCMPRSK)
Bibliothek(gtZusammenfassung)
Bibliothek(ggsurvfit)
Bibliothek(ggprism)
data <-read.csv('data.csv')"
data$status<-factor(data$status, levels=c(0,1,2),labels=c("0","CSS","OCS"))
CIF <- tidycmprsk::cuminc(Surv(Zeit, Status) ~ Geschlecht, Daten = Daten)
ggcuminc(CIF,outcome= c("CSS", "OCS"),size=1.5)
Dabei handelt es sich data.csv um Daten, die aus der SEER-Datenbank abgerufen werden. - Nachdem Sie den obigen Code ausgeführt haben, klicken Sie auf Exportieren, klicken Sie dann auf Als Bild speichern und schließlich auf Speichern , um das Bild zu speichern. Die Methode zum Speichern nachfolgender Bilder ist die gleiche wie in diesem Schritt. Ersetzen Sie Geschlecht im obigen Code nacheinander durch andere Faktoren, um eine univariate Analyse für alle Faktoren durchzuführen.
- Verwenden Sie den folgenden Code, um BSR- und multivariate Analysen und Visualisierungen durchzuführen.
Bibliothek(Sprünge)
library(riskRegression)
Bibliothek(prodlim)
Bibliothek(Waldgrundstück)
leaps<-regsubsets(status==0~Geschlecht+Größe+Klasse+T+N+M,Daten = Daten)
plot(leaps,scale="adjr2")
multi <- FGR(Hist(Zeit,Status)~Geschlecht + T + N + M +Klasse+ Größe,Ursache=1,Daten=Daten)
Zusammenfassung(mehrere)
Multi <- read.csv("multi.csv",Kopfzeile = T)
forestplot(labeltext=as.matrix(Multi [,1:4]), mean= Multi $HR_mean, ower= Multi $HR_1, upper= Multi $HR_2)
Dabei stammen die Daten in multi.csv aus den Ergebnissen des vorherigen Codes. - Folgen Sie Schritt 2.3. , um das Bild zu speichern.
- Verwenden Sie den folgenden Code, um das Nomogramm, die ROC-Kurve, die Kalibrierungskurve und die DCA-Kurve darzustellen.
Bibliothek(QHScrnomo)
Bibliothek(RMS)
library(riskRegression)
Bibliothek(prodlim)
Daten <- read.csv("data.csv")
d <- datadist(Daten)
Optionen(datavert = "d")
e <-cph(Surv(Zeit,Status==1)~Geschlecht + T + N + M +Note,Daten = Daten,
x=T, y=T, surv=T,time.inc=60)
nomo <- crr.fit(e,failcode=1,cencode = 0)
nomogram.crr(fit =nomo, lp = F, xfrac = 0.5, fun.at =seq(from=0, to=1, by= 0.1) , failtime =c(12,36,60),funlabel = c("Kumulative CSS-Inzidenz über 1 Jahr", "Kumulative CSS-Inzidenz über 3 Jahre","Kumulative CSS-Inzidenz über 5 Jahre"))
set.seed(123)
data$pro <- tenf.crr(m3,time = 60)
groupci(x=data$pro, ftime = data$time, fstatus = data$status, failcode = 1, cencode = 0, ci = TRUE)
f <- CSC(Hist(Zeit,Status)~Geschlecht + T + N + M +Note,Daten = Daten)
x <- Score(list(model1=f), Hist(time,status)~1, data=data, cause=1, times=c(12,36,60), se.fit=1L, plots="roc", metrics="auc")
g <- as.data.frame(x$AUC$score)
h <- g[g$times %in% c(12,36,60),]
col = c("darkcyan","tomate","lila")
plotROC(x, xlab="1-Spezifität", ylab="Sensitivität",col=col[1], cex=1.5, legend="", auc.in.legend = F, Zeiten = 12)
plotROC(x,col=col[2],legend = '', cex=1.5,times =36,auc.in.legend = F,add=T)
plotROC(x,col=col[3], times =60, add=T, cex=1.5, legend = '',auc.in.legend = F)
leg <- paste0(c("1-Jahr: ","3-Jahr: ","5-Jahr: "),substr(a$AUC,1,5)) - Folgen Sie Schritt 2.3. , um das Bild zu speichern.
- Verwenden Sie das ggscidca-Paket, um die DCA-Kurve darzustellen.
Bibliothek(ggscidca)
df_surv<-read.csv("data.csv",Kopfzeile = T)
cox_model <- coxph(Surv(Zeit,Status==1)~Sex + T + N+ M +Note, Daten = df_surv)
cox_model1<-newcrr(cox_model)
scidca(cox_model1, newdata= df_surv,threshold.line = T,threshold.text = T)
Dabei handelt es sich data.csv um Daten, die aus der SEER-Datenbank abgerufen werden. - Folgen Sie Schritt 2.3. , um das Bild zu speichern.
Ergebnisse
Eigenschaften des Patienten
Insgesamt wurden 8.931 Patienten in die Studie eingeschlossen. Bezogen auf die Geschlechterverteilung war der Anteil der männlichen Patienten (56 %) im Vergleich zu den weiblichen Patienten (44 %) höher. Hinsichtlich der Verteilung der Tumorlokalisation war die Mehrzahl der Tumoren über das gesamte Kolorektum verteilt, während die wenigsten Tumoren im rechten Dickdarm lokalisiert waren (21%). In Bezug auf den Tumorgrad war der häufigste Grad II mit 6.251 Patienten, was 70 % der Gesamtzahl ausmachte; Es folgte der Grad III mit 2.026 Patienten (23 %). Die Patienten der Grade I und IV hatten 355 bzw. 299 Patienten, was 4 % bzw. 3,3 % der Gesamtzahl ausmachte. In Bezug auf die Tumorinvasion waren die Stadien T3 und T4 am häufigsten. Mehr als die Hälfte der Patienten hatte keine Lymphknotenmetastasen, während 17% der Patienten Fernmetastasen aufwiesen. 1.446 Patienten (16 %) erhielten eine Strahlentherapie und 3.843 Patienten (43 %) eine Chemotherapie. SCRC war häufiger, mit 5.327 Patienten, die 60 % der Gesamtzahl ausmachten; Es gab 3.604 Patienten mit metachronem Darmkrebs, was 40 % entspricht. Die Patienten wurden nach dem Zufallsprinzip in Trainings- und Validierungssets im Verhältnis 7:3 eingeteilt. Es gab keine statistisch signifikanten Unterschiede in den Ausgangsdaten zwischen den Trainings- und Validierungskohorten, wie in Tabelle 1 gezeigt.
Univariable Analyse
Nach der Kontrolle des Einflusses kompetitiver Ereignisse zeigten die Ergebnisse der univariaten Analyse, dass Geschlecht, Tumorgrad und -größe, TNM-Stadium, Bestrahlung, Chemotherapie, synchroner oder metachroner Status und Tumorlokalisation die prognostischen Faktoren waren, die die CSS bei MPCC-Patienten beeinflussten. Nur das Alter ist kein prognostischer Faktor für CSS bei MPCC-Patienten. Wir stellen fest, dass es eine signifikante Überschneidung von CIF in Bezug auf Bestrahlung, Chemotherapie, Lokalisation und synchronen oder metachronen Status gibt, was darauf hindeutet, dass die kurz- und langfristigen prognostischen Auswirkungen der Bestrahlung auf MPCC-Patienten unterschiedlich waren, ebenso wie die Chemotherapie, die Lokalisation und der synchrone oder metachrone Status. Die kumulative Risikokurve der einzelnen Teilgruppen ist in Abbildung 2 dargestellt.
Multivariable Analyse
Die prognostischen Faktoren, die durch die univariable Analyse erhalten wurden, wurden in die Regression der besten Teilmengen (BSR) und die multivariable Analyse des Wettbewerbsrisikomodells integriert. Unter ihnen sind Bestrahlung, Chemotherapie, Lokalisation und synchroner oder metachroner Status aufgrund ihrer doppelten Auswirkungen auf die Prognose ausgeschlossen. Die Ergebnisse der BSR und der multivariablen Analyse zeigten beide, dass Geschlecht, TNM-Stadium und Tumorgrad unabhängige Risikofaktoren für CSS bei MPCC-Patienten waren. (Abbildung 2)
Aufbau und Verifizierung des Nomogramms
Basierend auf den unabhängigen Risikofaktoren, die durch multivariable Analyse erhalten wurden, konstruieren wir ein Liniennomogramm, um CSS vorherzusagen und die Leistung des Vorhersagemodells zu verifizieren (Abbildung 3). Anschließend verwenden wir die ROC-Kurve, die Kalibrierungskurve und den DCA, um das Modell zu bewerten. Die ROC-Kurve zeigte, dass die AUCs von 1 Jahr, 3 Jahren und 5 Jahren der Trainingskohorte 0,762, 0,742 und 0,734 betrugen, und die AUCs von 1 Jahr, 3 Jahren und 5 Jahren der Verifizierungskohorte 0,801, 0,740 und 0,743. In der Trainingskohorte und der Verifizierungskohorte zeigte die Kalibrierungskurve eine hohe Übereinstimmung zwischen der projizierten Wahrscheinlichkeit und den tatsächlichen Daten (Abbildung 4). Um die Leistung des Modells in der klinischen Anwendung zu verifizieren, haben wir DCA verwendet, um den klinischen Wert des Modells zu bewerten. Die Ergebnisse zeigen, dass das Modell einen guten Nettonutzen aufweist (Abbildung 5).
Abbildung 1: Workflow-Diagramm der Studie. Diese Studie besteht aus zwei Schritten: Zunächst wurden die Daten mit SEER gewonnen. Die Statistik und die anschließende Datenanalyse und -visualisierung wurden mit R durchgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
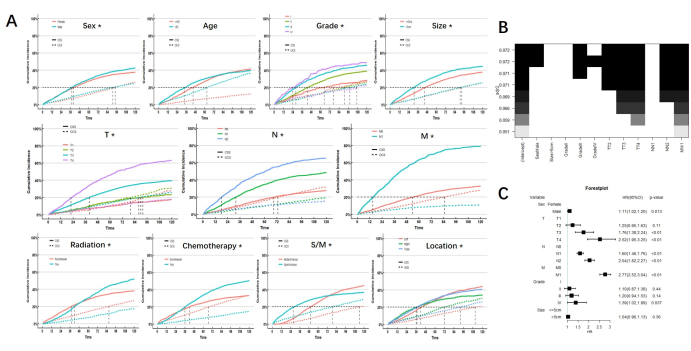
Abbildung 2: Analyseergebnisse. (A) CIF der Untergruppen. * zeigt p <0,05 an. (B) Regression der besten Teilmengen. Unter der besten Passform wurden Geschlecht, Note und TNM-Stadium für die Aufnahme in Betracht gezogen (oben als schwarze Blöcke dargestellt). (C) Die multivariate Analyse zeigte auch, dass Geschlecht, Grad und TNM-Stadium unabhängige Risikofaktoren sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
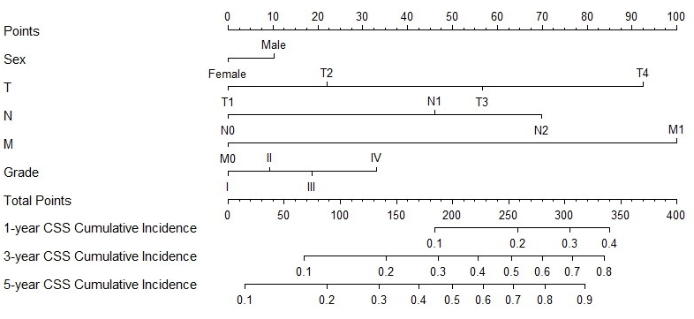
Abbildung 3: Nomogramm für CSS bei MPCC-Patienten. Die Gesamtpunktzahl des Patienten kann berechnet werden, indem die Punktzahlen addiert werden, die jedem Faktor entsprechen. Basierend auf der Gesamtpunktzahl kann die Wahrscheinlichkeit eines krebsspezifischen Todes nach 1, 3 und 5 Jahren vorhergesagt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
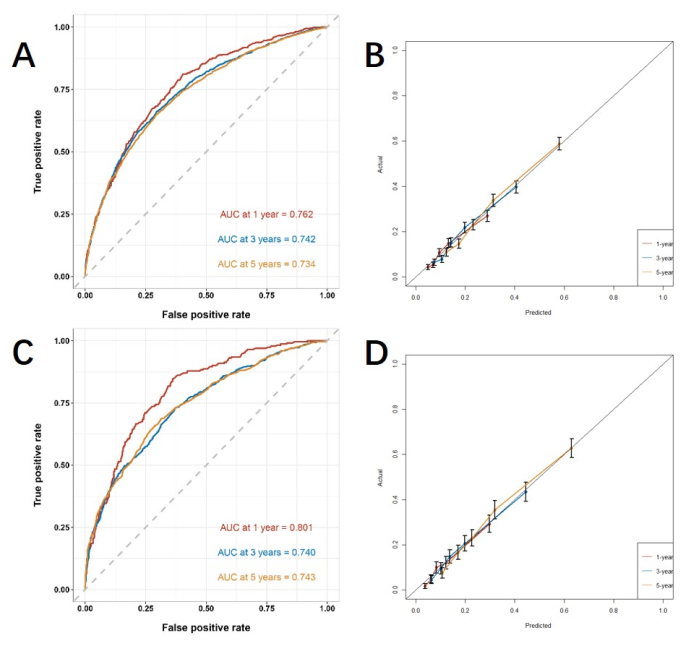
Abbildung 4: ROC-Kurve und Kalibrierungskurve. (A, B) Die Kurven in der Trainingskohorte (n=6255) und der (C, D) Validierungskohorte (n=2676). Je näher der AUC-Wert an 1 liegt, desto besser ist die Klassifizierungsleistung des Modells. Die Fehlerbalken zeigen das 95 %-Konfidenzintervall für die Wahrscheinlichkeit des Eintretens des tatsächlichen Ereignisses an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
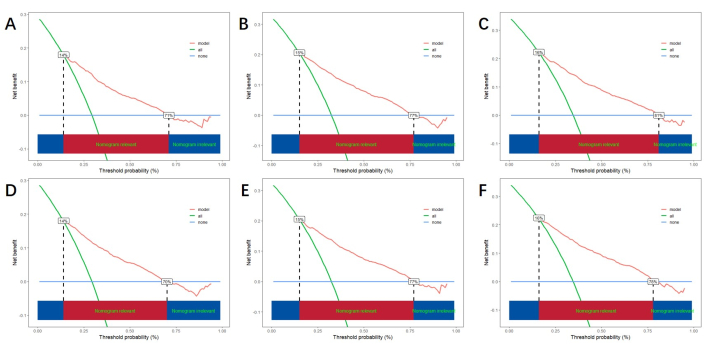
Abbildung 5: DCA für 1-Jahres-, 3-Jahres- und 5-Jahres-Kohorten. (A, B, C) DCA in der Trainingskohorte und (D, E, F) Validierungskohorte. Die grüne Linie stellt den Nettonutzen aller positiven Aspekte dar, die blaue Linie den Nettonutzen aller positiven Aspekte und die rote Linie den Nettonutzen des Modells. Der rote Bereich unten stellt den Nutzen des Modells dar, der den aller positiven und nicht positiven Aspekte übersteigt, und zeigt den tatsächlichen Nutzenbereich des Modells an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Klinisch-pathologische und Ausgangscharakteristika der Patienten. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Da es sich um einen häufigen Tumor des Verdauungssystems handelt, wurden Faktoren, die die Prognose des solitären Darmkrebses beeinflussen, in früheren Forschungen untersucht und bestätigt24. Es gibt jedoch nur begrenzte Untersuchungen zu prognostischen Faktoren für MPCC-Patienten. Diese Studie umfasste 8.931 MPCC-Patienten, die sich zwischen 2004 und 2015 aus der SEER-Datenbank einer Operation unterzogen hatten. Wir verwendeten ein konkurrierendes Risikomodell, um die Risikofaktoren zu untersuchen, die CSS beeinflussen, und konstruierten ein Vorhersagemodell. In dieser Studie starben 39,1 % der verstorbenen Patienten an anderen Ursachen als Krebs, was die Notwendigkeit unterstreicht, ein konkurrierendes Risikomodell zur Analyse von Risikofaktoren für CSS zu verwenden.
Frühere Studien haben ergeben, dass das fortgeschrittene Alter ein Risikofaktor ist, der das OS von MPCC-Patienten beeinflusst12. In dieser Studie haben wir jedoch festgestellt, dass das Alter kein Risikofaktor für CSS ist. Ältere Patienten weisen oft einen schlechteren Ausgangszustand und mehr Komorbiditäten auf, was im Vergleich zu jüngeren Patienten zu einem kürzeren OS führen kann. Ältere Menschen sterben auch häufiger an anderen Ursachen, wie z. B. kardiovaskulären Ereignissen und schweren Infektionen, was sie anfälliger für diese Faktoren macht als jüngere Patienten. In dieser Studie konzentrierten wir uns auf die konkurrierende Beziehung zwischen Todesfällen aufgrund anderer Ursachen und Todesfällen aufgrund von Krebs selbst. Durch die Verwendung eines konkurrierenden Risikomodells und unter Ausschluss der Beeinflussung von Todesfällen, die durch andere Ursachen verursacht wurden, stellten wir fest, dass unter diesen Bedingungen das fortgeschrittene Alter kein Risikofaktor mehr für CSS von MPCC war. Dies deutet darauf hin, dass MPCC-Patienten in verschiedenen Altersgruppen mit ähnlichen Tumorlasten konfrontiert sein können. Insgesamt haben junge und ältere Patienten mit dieser Krankheit eine ähnliche CSS, was eine wertvolle Orientierungshilfe für die Entwicklung personalisierter Behandlungsstrategien bietet.
Diese Studie ergab auch, dass die Inzidenz und Prognose von MPCC geschlechtsspezifische Tendenzen aufweisen, mit einem höheren Anteil und einer schlechteren CSS bei männlichen Patienten. Dies stimmt mit der Situation überein, die beim solitären Darmkrebs beobachtet wurde. Frühere Studien haben gezeigt, dass solitärer Darmkrebs bei Männern häufiger auftritt und männliche Patienten eine schlechtere Prognose haben als weibliche Patienten. Dies kann auf den Einfluss von Östrogen auf das Auftreten und Fortschreiten von Darmkrebs zurückzuführensein 25. Darüber hinaus deuten andere Untersuchungen darauf hin, dass die Darmmikrobiota und die Darmmetaboliten bei männlichen Patienten einer der Gründe für die bei Darmkrebspatienten beobachteten geschlechtsspezifischen Unterschiede sein könnten26. Eine Studie in Deutschland ergab, dass Typ-2-Diabetes einen größeren Einfluss auf Darmkrebs bei Frauenhat 27. Darüber hinaus hat Vitamin D bei Frauen eine schützende Wirkung gegen kolorektale Neoplasien, bei Männern wurden jedoch keine ähnlichen Ergebnisse gefunden28.
Konkurrierende univariate Analysen ergaben, dass MPCC-Patienten mit größeren Tumorgrößen eine schlechtere CSS aufwiesen. In früheren Studien wurde die Tumorgröße oft als Indikator für die Aggressivität des Tumors angesehen 29,30,31. Die Ergebnisse konkurrierender multivariater Analysen unterstützten jedoch nicht die Tumorgröße als unabhängigen Risikofaktor. Dies deutet darauf hin, dass die Tumorgröße in Hohlorganen wie dem Dickdarm möglicherweise nur eine begrenzte Fähigkeit hat, die Aggressivität des Tumors widerzuspiegeln. Dieses Phänomen kann auf verschiedene Faktoren zurückzuführen sein, darunter die Komplexität der Tumorbiologie und die unterschiedlichen Muster des Tumorwachstums und der Ausbreitung über verschiedene Organe.
Ein schlechter Tumorgrad deutet auf stärkere invasive und migratorische Fähigkeiten von Tumorzellen hin, was mit den Schlussfolgerungen dieser Studie übereinstimmt: Ein schlechterer Tumorgrad deutet auf einen schlechteren CSS für MPCC-Patienten hin. Das TNM-Stadium ist die am häufigsten verwendete klinische Methode zur Steuerung der Patientenbehandlung und zur Vorhersage der Prognose32,33. Diese Studie ergab, dass das krebsspezifische Überleben des Patienten umso schlechter ist, je tiefer die Tumorinfiltration ist, je größer die Anzahl der metastasierenden Lymphknoten und das Vorhandensein von Organmetastasen ist. Dies steht im Einklang mit dem klinischen Konsens.
Bei Darmkrebs variiert die Prognose je nach Lokalisation des Tumors. Viele Studien haben ergeben, dass der rechte Dickdarm im Vergleich zum linken Dickdarm eine schlechtere Prognose hat 34,35,36,37. Einige Untersuchungen deuten jedoch darauf hin, dass bei resezierbarem Darmkrebs die Seite der Tumorlokalisation keinen Einfluss auf die Langzeitprognose hat38. In dieser Studie hatte MPCC im rechten Dickdarm kurzfristig eine schlechtere CSS, aber eine bessere Langzeitprognose. In ähnlicher Weise hatten MPCC-Patienten, die eine Bestrahlung und Chemotherapie erhielten, eine bessere kurzfristige CSS, aber eine schlechtere langfristige CSS. SCRC hatte im Vergleich zu MCRC eine schlechtere kurzfristige CSS, aber eine bessere langfristige CSS. Die Gründe für diese Doppeleffekte auf die Prognose sind noch unklar.
Diese Studie hat einige Einschränkungen. Aufgrund der inhärenten Einschränkungen der SEER-Datenbank konnten wir einige bekannte prognostische Indikatoren, wie z.B. die Spiegel des karzinoembryonalen Antigens (CEA) und den Mikrosatellitenstatus, nicht ermitteln oder analysieren. Darüber hinaus konnten wir Patienten mit entzündlichen Darmerkrankungen, erblichem nichtpolyposösem Darmkrebs und familiärer adenomatöser Polyposis, die ein höheres Risiko für MPCC39 haben, nicht ausschließen. Darüber hinaus ist die Immuntherapie zu einem immer wichtigeren Aspekt der Darmkrebsbehandlung geworden, aber wir konnten keine Daten dazu erhalten. Der retrospektive Charakter unserer Analyse und die Abhängigkeit von einem einzigen Datensatz können zu inhärenten Verzerrungen führen.
Die Studie, die auf der SEER-Datenbank basiert, ergab, dass das männliche Geschlecht, der schlechte Tumorgrad und das fortgeschrittene TNM-Stadium bei MPCC-Patienten nach der Operation mit einer schlechteren CSS verbunden waren. Es ist wichtig, Patienten mit diesen Risikofaktoren bei der Diagnose und Behandlung große Aufmerksamkeit zu schenken. Darüber hinaus wurde in dieser Studie ein Nomogramm zur Vorhersage des CSS von MPCC-Patienten entwickelt, das die Prognose genau vorhersagen und zur klinischen Behandlungsentscheidung beitragen kann.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Nichts.
Materialien
| Name | Company | Catalog Number | Comments |
| forestplot package | Comprehensive R Archive Network (CRAN) | forest plot 3.1.3 | A forest plot that allows for multiple confidence intervals per row, custom fonts for each text element, custom confidence intervals, text mixed with expressions, and more. |
| ggprism package | Comprehensive R Archive Network (CRAN) | ggprism 1.0.5 | The ggprism package provides various themes, palettes, and other useful functions to customise ggplots and give them the ‘GraphPad Prism’ look. |
| ggscidca package | Comprehensive R Archive Network (CRAN) | ggscidca package | The 'ggscidca' package adds coloured bars of discriminant relevance to the traditional decision curve. Improved practicality and aesthetics. |
| ggsurvfit package | Comprehensive R Archive Network (CRAN) | ggsurvfit 1.1.0 | The ggsurvfit package eases the creation of time-to-event (aka survival) summary figures with ggplot2. The concise and modular code creates images that are ready for publication or sharing. |
| gtsummary package | Comprehensive R Archive Network (CRAN) | gtsummary 2.0.0 | The gtsummary package provides an elegant and flexible way to create publication-ready analytical and summary tables using the R programming language. The {gtsummary} package summarizes data sets, regression models, and more, using sensible defaults with highly customizable capabilities. |
| QHScrnomo package | Comprehensive R Archive Network (CRAN) | QHScrnomo 3.0.1 | The goal of QHScrnomo is to provide functionality to construct nomograms in the context of time-to-event (survival) analysis in the presence of competing risks. It also contains functions to build, validate, and summarize these models. |
| R Software | R Core Team | R 4.3.3 | Free software environment for statistical computing and graphics |
| riskRegression package | Comprehensive R Archive Network (CRAN) | riskRegression 1.3.7 | Risk Regression Models and Prediction Scores for Survival Analysis with Competing Risks |
| rms package | Comprehensive R Archive Network (CRAN) | rms 6.8-1 | rms does regression modeling, testing, estimation, validation, graphics, prediction, and typesetting by storing enhanced model design attributes in the fit. |
| RStudio | RStudio, Public Benefit Corporation(PBC) | Rstudio 2023.12.1+402 | Integrated Development Environment (IDE) used for running R scripts, data analysis, and model development. Provides a user-friendly interface for R programming with advanced features like script editing, debugging, and version control. |
| SEERstat | National Cancer Institute (NCI) | SEERstat 8.4.3 | Software for statistical analysis of SEER and other cancer-related databases |
| tidycmprsk package | Comprehensive R Archive Network (CRAN) | tidycmprsk 1.0.0 | The tidycmprsk package provides an intuitive interface for working with the competing risk endpoints |
Referenzen
- Bray, F., et al. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 74 (3), 229-263 (2024).
- Derwinger, K., Gustavsson, B. A study of aspects on gender and prognosis in synchronous colorectal cancer. Clin Med Insights Oncol. 5, 259-264 (2011).
- Chin, C. C., Kuo, Y. H., Chiang, J. M. Synchronous colorectal carcinoma: predisposing factors and characteristics. Colorectal Dis. 21 (4), 432-440 (2019).
- Nikoloudis, N., et al. Synchronous colorectal cancer. Tech Coloproctol. 8 (Suppl 1), s177-s179 (2004).
- Huang, C. S., et al. Synchronous and metachronous colorectal cancers: Distinct disease entities or different disease courses. Hepatogastroenterology. 62 (140), 838-842 (2015).
- He, W., et al. Prognosis of synchronous colorectal carcinoma compared to solitary colorectal carcinoma: a matched pair analysis. Eur J Gastroenterol Hepatol. 31 (12), 1489-1495 (2019).
- Ranstam, J., Cook, J. A., Collins, G. S. Clinical prediction models. Br J Surg. 103 (13), 1886 (2016).
- Smith, T., et al. Comparison of prognostic models to predict the occurrence of colorectal cancer in asymptomatic individuals: a systematic literature review and external validation in the EPIC and UK Biobank prospective cohort studies. Gut. 68 (4), 672-683 (2019).
- Iasonos, A., Schrag, D., Raj, G. V., Panageas, K. S. How to build and interpret a nomogram for cancer prognosis. J Clin Oncol. 26 (8), 1364-1370 (2008).
- Conroy, T., et al. Five-year outcomes of FOLFIRINOX vs Gemcitabine as adjuvant therapy for pancreatic cancer: A randomized clinical trial. JAMA Oncol. 8 (11), 1571-1578 (2022).
- Zhong, M., et al. Impact of lung metastasis versus metastasis of bone, brain, or liver on overall survival and thyroid cancer-specific survival of thyroid cancer patients: A population-based study. Cancers. 14 (13), 3133 (2022).
- Xu, Y., Wang, X., Huang, Y., Ye, D., Chi, P. A LASSO-based survival prediction model for patients with synchronous colorectal carcinomas based on SEER. Transl Cancer Res. 11 (8), 2795-2809 (2022).
- Zhang, X., Zhao, L., Hu, Y., Deng, K., Ren, W. A novel risk prediction nomogram for early death in patients with resected synchronous multiple primary colorectal cancer based on the SEER database. Int J Colorectal Dis. 38 (1), 130 (2023).
- Zhang, X., et al. Developing prognostic nomograms to predict overall survival and cancer-specific survival in synchronous multiple primary colorectal cancer based on the SEER database. J Cancer Res Clin Oncol. 149 (15), 14057-14070 (2023).
- Austin, P. C., Lee, D. S., Fine, J. P. Introduction to the analysis of survival data in the presence of competing risks. Circulation. 133 (6), 601-609 (2016).
- Lau, B., Cole, S. R., Gange, S. J. Competing risk regression models for epidemiologic data. Am J Epidemiol. 170 (2), 244-256 (2009).
- Nolan, E. K., Chen, H. Y. A comparison of the Cox model to the Fine-Gray model for survival analyses of re-fracture rates. Arch Osteoporos. 15 (1), 86 (2020).
- Park, S. Y. Nomogram: An analogue tool to deliver digital knowledge. J Thorac Cardiovasc Surg. 155 (4), 1793 (2018).
- Balachandran, V. P., Gonen, M., Smith, J. J., DeMatteo, R. P. Nomograms in oncology: more than meets the eye. Lancet Oncol. 16 (4), e173-e180 (2015).
- Liang, W., et al. Development and validation of a nomogram for predicting survival in patients with resected non-small-cell lung cancer. J Clin Oncol. 33 (8), 861-869 (2015).
- Chen, D., et al. Predicting postoperative peritoneal metastasis in gastric cancer with serosal invasion using a collagen nomogram. Nat Commun. 12 (1), 179 (2021).
- Niu, X., et al. A prognostic nomogram for patients with newly diagnosed adult thalamic glioma in a surgical cohort. Neuro Oncol. 23 (2), 337-338 (2021).
- Luo, S., et al. Comparison of left- and right-sided colorectal cancer to explore prognostic signatures related to pyroptosis. Heliyon. 10 (7), e28091 (2024).
- Keum, N., Giovannucci, E. Global burden of colorectal cancer: emerging trends, risk factors and prevention strategies. Nat Rev Gastroenterol Hepatol. 16 (12), 713-732 (2019).
- Foster, P. A. Oestrogen and colorectal cancer: mechanisms and controversies. Int J Colorectal Dis. 28 (6), 737-749 (2013).
- Wang, L., et al. Male-biased gut microbiome and metabolites aggravate colorectal cancer development. Adv Sci. 10 (25), e2206238 (2023).
- Krämer, H. U., et al. Type 2 diabetes mellitus and gender-specific risk for colorectal neoplasia. Eur J Epidemiol. 27 (5), 341-347 (2012).
- Aigner, E., et al. Gender- and site-specific differences of colorectal neoplasia relate to vitamin D. Aliment Pharmacol Ther. 40 (11-12), 1341-1348 (2014).
- Dai, W., et al. Does tumor size have its prognostic role in colorectal cancer? Re-evaluating its value in colorectal adenocarcinoma with different macroscopic growth pattern. Int J Surg. 45, 105-112 (2017).
- Rössler, O., et al. Tumor size, tumor location, and antitumor inflammatory response are associated with lymph node size in colorectal cancer patients. Mod Pathol. 30 (6), 897-904 (2017).
- Zhang, Q., et al. Prognostic impact of tumor size on patients with metastatic colorectal cancer: a large SEER-based retrospective cohort study. Updates Surg. 75 (5), 1135-1147 (2023).
- Greene, F. L. TNM: our language of cancer. CA Cancer J Clin. 54 (3), 129-130 (2004).
- Ueno, H., et al. Optimal colorectal cancer staging criteria in TNM classification. J Clin Oncol. 30 (13), 1519-1526 (2012).
- Yahagi, M., Okabayashi, K., Hasegawa, H., Tsuruta, M., Kitagawa, Y. The worse prognosis of right-sided compared with left-sided colon cancers: a systematic review and meta-analysis. J Gastrointest Surg. 20 (3), 648-655 (2016).
- Holch, J. W., Ricard, I., Stintzing, S., Modest, D. P., Heinemann, V. The relevance of primary tumour location in patients with metastatic colorectal cancer: A meta-analysis of first-line clinical trials. Eur J Cancer. 70, 87-98 (2017).
- Petrelli, F., et al. Prognostic survival associated with left-sided vs right-sided colon cancer: A systematic review and meta-analysis. JAMA Oncol. 3 (2), 211-219 (2017).
- Zheng, C., Jiang, F., Lin, H., Li, S. Clinical characteristics and prognosis of different primary tumor location in colorectal cancer: a population-based cohort study. Clin Transl Oncol. 21 (11), 1524-1531 (2019).
- Karim, S., Brennan, K., Nanji, S., Berry, S. R., Booth, C. M. Association between prognosis and tumor laterality in early-stage colon cancer. JAMA Oncol. 3 (10), 1386-1392 (2017).
- Lindberg, L. J., et al. Risk of multiple colorectal cancer development depends on age and subgroup in individuals with hereditary predisposition. Fam Cancer. 18 (2), 183-191 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten