Method Article
Hi-C: Un método para estudiar la arquitectura tridimensional de los genomas.
* Estos autores han contribuido por igual
En este artículo
Erratum Notice
Resumen
El método de Hi-C permite la identificación imparcial de todo el genoma de las interacciones entre la cromatina (1). Hi-C parejas ligadura de proximidad y de secuenciación masiva en paralelo. Los datos resultantes se pueden utilizar para estudiar arquitectura genómica a diferentes escalas, los resultados iniciales identificaron características tales como los territorios de cromosomas, la segregación de la cromatina abierta y cerrada, y la estructura de la cromatina en la escala de megabase.
Resumen
El plegamiento tridimensional de los cromosomas del genoma y compartimenta y puede traer distante elementos funcionales, tales como promotores y potenciadores, en estrecha proximidad espacial 2-6. Descifrar la relación entre la organización de los cromosomas y la actividad del genoma ayudará a comprender los procesos genómicos, como la replicación y transcripción. Sin embargo, poco se sabe sobre cómo los cromosomas veces. Microscopía es incapaz de distinguir un gran número de loci simultáneamente o en alta resolución. Hasta la fecha, la detección de interacciones con la captura de los cromosomas cromosoma conformación (3C) y sus adaptaciones posteriores requiere la elección de un conjunto de lugares de destino, por lo que todo el genoma estudios imposible 7-10.
Hemos desarrollado Hi-C, una extensión de 3C que es capaz de identificar las interacciones de largo alcance en una evaluación imparcial de todo el genoma de la moda. En Hi-C, las células se fijan con formaldehído, causando loci que interactúan para ser unidos entre sí por medio de la covalentes proteína-ADN enlaces cruzados. Cuando el ADN es posteriormente fragmentado con una enzima de restricción, estos lugares permanecen vinculados. Un residuo de biotina se ha incorporado como el 5 'domina se llenan in A continuación, de extremo romo ligadura se realiza bajo condiciones que favorecen los eventos diluir la ligadura entre los fragmentos de ADN reticulado. Esto se traduce en una biblioteca de todo el genoma de la ligadura de los productos, lo que corresponde a los pares de fragmentos que originalmente estaban muy cerca uno del otro en el núcleo. Cada producto de ligación se marca con biotina en el sitio de la unión. La biblioteca está cortado, y las uniones se tira hacia abajo con perlas de estreptavidina. Las uniones purificado posteriormente pueden ser analizados mediante un secuenciador de alto rendimiento, lo que resulta en un catálogo de los fragmentos de la interacción.
El análisis directo de la matriz de contacto resultante revela numerosas características de la organización genómica, tales como la presencia de los territorios de los cromosomas y la asociación preferencial de pequeños cromosomas ricos en genes. El análisis de correlación se puede aplicar a la matriz de contactos, lo que demuestra que el genoma humano se separa en dos compartimentos: un compartimiento menos densa que contiene cromatina abierta, accesible y eficaz, y un compartimiento cerrado que contiene más denso, las regiones inaccesibles de la cromatina, y las personas inactivas. Finalmente, el análisis conjunto de la matriz de contacto, junto con derivaciones teóricas y las simulaciones computacionales, reveló que en la escala de megabase Hi-C revela características compatibles con una conformación glóbulo fractal.
Protocolo
Este método se utilizó en la investigación publicada en Lieberman-Aiden et al., Science 326, 289-293 (2009) .
I. El entrecruzamiento, la digestión, la indicación del Fin del ADN, y la ligadura de extremo romo
- Hi-C comienza con entrecruzamiento de las células, que es un hilo común entre todos los métodos basados en 3C. Para comenzar, crecer entre 2 x 10 7 y 2,5 x 10 7 células de mamíferos, ya sea adherente o en suspensión, y el entrecruzamiento de las células. (Para más detalles sobre la reticulación de las células, por favor vea: 11
- Lisis de las células en 550 lysisbuffer l (500 l 10 mM Tris-HCl pH 8,0, 10 mM NaCl, 0,2% Igepal CA-630 y 50 l inhibidores de la proteasa) con un homogeneizador. Haga girar la cromatina a 5.000 rpm y lavar el pellet dos veces con 500 l 1x NEBuffer 2.
- Resuspender la cromatina en 1x NEBuffer 2, alícuota en 5 tubos numerados y añadir 1x NEBuffer 2 a un volumen final de 362 microlitros. Agregar 38 l SDS al 1%, mezclar cuidadosamente y se incuba a 65 ° C durante 10 minutos. Coloque los tubos de nuevo en hielo inmediatamente después de la incubación.
- Apagar la SDS mediante la adición de 44 l de Triton X-100 y mezclar cuidadosamente. Recopilación de la cromatina mediante la adición de 400 unidades de HindIII y se incuba a 37 ° C durante la noche durante la rotación.
- Los próximos pasos son Hi-C específicos e incluyen los extremos de ADN marcado con biotina y la realización de extremo romo ligadura de fragmentos entrecruzados. Este paso permitirá a la ligadura de las uniones de ser purificado posteriormente. Un tubo no debe someterse a la etapa de biotinilación y en su lugar deben mantenerse separados y servir como un control de 3C para asegurarse de que la digestión, y las condiciones de ligadura fueron óptimos.
- Para llenar los voladizos de fragmentos de restricción y marcar los extremos de ADN con la biotina en el resto de 4 tubos, añadir 1,5 l de 10 mM dATP, 1,5 l de 10 mM dGTP, 1,5 l de 10 mM dTTP, 37,5 l 0,4 mM biotina-14-dCTP, y 10 Klenow l 5U/μl a los tubos 2-5. Mezclar cuidadosamente e incubar durante 45 minutos a 37 ° C.
- Coloque los tubos en hielo. Para inactivar las enzimas, añadir 86 l 10% SDS a los tubos 1-5. Incubar los tubos a 65 ° C durante 30 minutos exactos y colocarlos en hielo inmediatamente después.
- La ligadura se realiza bajo condiciones extremadamente diluidas, para favorecer la ligadura de eventos entre los fragmentos entrecruzados. De Trabajo sobre hielo, agregar 7,61 ml mezcla de ligación [745 l 10% de Triton X-100, 745 l de amortiguación ligadura de 10x (500 mM Tris-HCl pH 7,5, 100 mM MgCl 2, 100 mM DTT), 80 l 10 mg / ml de BSA , 80 l 100 mM ATP y 5,96 ml de agua] para cada uno de los cinco números tubos de 15 ml. La transferencia de cada mezcla de la cromatina digerido a un tubo de 15 ml, que corresponde.
- Para la ligadura de 3C regular, añadir 10 l 1U/μl ligasa T4 DNA en el tubo 1. De extremo romo ligadura de Hi-C, se añaden 50 l 1U/μl ligasa T4 ADN a los tubos 2-5. Mezclar invirtiendo los tubos e incubar todos los tubos de 5 por 4 horas a 16 ° C.
- Entrecruzamientos se invierten y la proteína se degrada por la adición de 50 l 10 mg / ml de proteinasa K por tubo e incubar los tubos durante la noche a 65 ° C. Añadir un adicional de 50 l 10 mg / ml de proteinasa K por tubo al día siguiente y continuar la incubación a 65 ° C durante 2 horas.
- Se enfría la mezcla de reacción a temperatura ambiente y la transferencia de los cinco tubos de 50 ml cónicos. Purificar el ADN de estos tubos por la realización de una extracción con fenol. Agregar 10 ml de fenol pH 8,0 y agitar durante 2 minutos. Girar los tubos durante 10 minutos a 3.500 rpm y cuidadosamente transferir la mayor cantidad de la fase acuosa como sea posible a un tubo nuevo de 50 ml.
- Repetir la extracción con fenol pH 8.0: cloroformo (1:1) y precipitar el ADN con etanol. (Para más detalles sobre la purificación del ADN, por favor vea: 11
- Después de la centrifugación del ADN precipitada con etanol, se disuelven cada pellet de ADN en 450 l TE 1x (10 mM Tris-HCl pH 8,0, 1 mM EDTA). Transfiera la mezcla de ADN en un tubo de 1.7 ml de centrífuga.
- Otra ronda de la purificación se lleva a cabo haciendo dos fenol: cloroformo extracciones. Añadir 500 l de fenol pH 8.0: cloroformo (1:1) y agitar durante 1 minuto. Centrifugar los tubos durante 5 minutos a 14.000 rpm y la transferencia de la fase acuosa a un tubo nuevo. Después de la segunda extracción, precipitar el ADN mediante la adición de 0,1 x volumen de NaOAc, el volumen de 2x de etanol al 100% e incubar 30 minutos a -80 º C.
- Después de girar por el ADN precipitado, lavar cada pastilla de ADN con etanol 70% y resuspender cada pellet de ADN en 25 l TE 1x. Degradan cualquier ARN que pueden estar presentes mediante la adición de un l 1 mg / ml RNasa un tubo por e incubar los tubos durante 30 minutos a 37 ° C. La piscina del Hi-C contenido de los tubos 2-5, manteniendo un tubo por separado como un control de 3C.
- Ahora es una buena oportunidad para examinar la marca Hi-C y la eficiencia de la ligadura. Estos controles son excelentes indicadores de si una biblioteca Hi-C va a tener éxito.
- Para comprobar la calidad y cantidad de las bibliotecas, de ejecución 2 y 6 l alícuotas de diluciones 1:10 de ambos 3C y las bibliotecas de Hi-C en un 0,8% en gel de agarosa. (Ver Figura 2A)
- Hi-C marca Hi-C y la eficiencia de la ligadura se verifica mediante un ensayo de PCR digerir. El éxito de relleno y la ligadura de un sitio HindIII (AAGCTT) crea un sitio para la enzima de restricción NheI (GCTAGC). Un producto de ligación particular, formada por dos fragmentos de restricción cercana es amplificada por PCR (como en 3C 11 con 0,2 l de cada biblioteca como plantilla. Los productos de PCR son posteriormente digerido con HindIII, NheI o ambos. Después de correr las muestras en un gel al 2% , el número relativo de 3C y Hi-C ligadura de eventos se puede estimar mediante la cuantificación de la intensidad de las bandas de corte y sin cortar (ver Figura 2B).
- Algunos fragmentos no han sido ligados: para evitar tirar de ellos en otro momento, retirar la biotina de estos fines no ligado con la actividad exonucleasa de la ADN polimerasa T4.
- Biotina-14-dCTP en los extremos de ADN no ligado se elimina con la actividad exonucleasa de la ADN polimerasa T4. Mezclar 5 g de Hi-C de la biblioteca con un l 10 mg / ml de BSA, 10 NEBuffer l 10x 2, 1 l 10 mM dATP, dGTP 1 l 10 mM y 5 unidades de T4 DNA polimerasa en un volumen total de 100 l, y se incuba la mezcla a 12 ° C durante 2 horas. Si es posible, múltiples reacciones de 5 mg se llevan a cabo.
- La reacción se detiene mediante la adición de 2 l de EDTA 0,5 M pH 8,0.
- Para purificar el ADN, un pH 8,0 fenol: cloroformo (1:1) la extracción se hace seguido de precipitación con etanol.
- El sobrenadante se desecha y los pellets de ADN se resuspendió y se agruparon en un volumen total de 100 l de agua.
II. Selección del tamaño de corte y
- Para hacer que el ADN biotinilado adecuado para secuenciación de alto rendimiento, el ADN debe ser cortado a un tamaño de 300 a 500 pares de bases con un instrumento S2 Covaris (ciclo de trabajo 5, la intensidad de 5, ciclos / ráfaga 200, de 60 segundos por 4 ciclos) .
- Para reparar los extremos del ADN cortado, añadir 14 l de amortiguación ligadura de 10x, 14 l 2,5 mM dNTP mix, 5 l de ADN polimerasa T4, 5 l T4 polinucleótido quinasa, una polimerasa de ADN Klenow l de agua y 1 l. Incubar durante 30 minutos a temperatura ambiente.
- Después de la incubación, el uso de una columna MinElute Qiagen para purificar el ADN de acuerdo con las recomendaciones del fabricante. Eluir el ADN dos veces con 15 l 1x Tris-Bajo-EDTA (TLE: 10 mM Tris pH 8,0, 0,1 mM EDTA). A continuación, coloque un dATP a los extremos 3 'de la final de reparar el ADN mediante la adición de 5 l NEBuffer2 10x, 10 l 1 mM dATP, 2 l de agua y 3 Klenow l (exo-). Incubar la reacción durante 30 minutos a 37 ° C.
- Para desactivar el fragmento de Klenow, incubar las reacciones durante 20 minutos a 65 ° C y, posteriormente, enfriar la reacción en hielo. El uso de un speedvac, reducir los volúmenes de reacción de 20 microlitros.
- A continuación, cargar el ADN en un 1,5% en gel de agarosa con 1X TAE y una duración de 3,5 horas a 80-90V. Después de la tinción del gel con SYBR verde, visualizar el ADN en un DarkReader. Fragmentos de ADN de los impuestos especiales entre 300 y 500 pares de bases y purificarlos con un kit de extracción de Qiagen gel usando 2-4 columnas en función del peso del gel. Eluir el ADN con 50 TLE l 1x.
- Combine los eluidos de las columnas Qiaquick y llevar el volumen final de 300 l con 1 TLE. Por último, determinar la concentración de ADN con el ensayo Quant-iT con el fluorómetro Qubit y calcular la cantidad total de ADN.
III. Biotina Pull-down y secuenciación pares de fin de
- En esta sección del protocolo, la ligadura de las uniones se purifican a partir de la piscina de ADN, que permite la identificación eficiente de interactuar fragmentos de cromatina por secuenciación dos a dos final. Realizar todos los pasos posteriores en los tubos de LoBind ADN.
- Preparar cuentas de biotina desplegable mediante el lavado l 150 resuspendido bolas magnéticas estreptavidina dos veces con 400 l de Tween Buffer (TB: 5 mM Tris-HCl pH 8,0, 0,5 mM EDTA, 1 M NaCl, 0,05% de Tween).
Lava estos y futuros consisten en cinco pasos:- Añadir buffer a las perlas
- Transfiera la mezcla a un tubo nuevo
- Rotar la muestra durante 3 minutos a temperatura ambiente
- Recuperar las cuentas utilizando un concentrador de partículas magnéticas
- Eliminar el sobrenadante
- Volver a suspender las bolas en 300 2x l No Buffer Tween (2x NTB: 10 mM Tris-HCl pH 8,0, 1 mM EDTA, 2 M NaCl) y se combinan con 300 l Hi-C del ADN. Deje que la biotina etiquetados Hi-C de ADN para unirse a las cuentas de estreptavidina mediante la incubación de la mezcla a temperatura ambiente durante 15 minutos con la rotación.
- Recuperar el ADN vinculados cuentas estreptavidina con la Concentr partículas magnéticasAtor, y eliminar el sobrenadante. Lavado de los granos en 400 l NTB 1x (5 mM Tris-HCl pH 8,0, 0,5 mM EDTA, 1 M NaCl), seguido de 100 l de amortiguación ligadura de 1x. Volver a suspender las bolas en 50 l de amortiguación ligadura de 1x y transferir la mezcla a un tubo nuevo.
- Para preparar el ADN para la secuenciación de Illumina final emparejados, tome la cantidad total de ADN utilizado como insumo para la biotina pull-down, que se calculó en el paso 2.6, y se divide por 20 para calcular la cantidad de Hi-C de ADN que tiene sido derribado y se encuentra disponible para la ligadura. Añadir 6 picomoles de Illumina adaptadores final emparejados por g de Hi-C de ADN disponibles para la ligadura. Use 1.200 unidades ADN ligasa de T4 para ligar los adaptadores para el ADN. Incubar durante 2 horas a temperatura ambiente.
- Expulsar a los no ligados pares adaptadores de final de la recuperación de las cuentas de Hi-C ADN unido y el lavado de las cuentas dos veces con 400 l 1x TB.
- Lavar los granos con 200 l NTB 1x, seguido de 200 l, y de 50 l 1x NEBuffer 2. Después del último lavado, resuspender las cuentas en 50 l 1x NEBuffer 2 y transferir a un tubo nuevo.
- Para determinar el número de ciclos necesarios para generar suficiente producto de PCR para la secuenciación, creado cuatro pruebas de reacciones de PCR con los ciclos de 6, 9, 12 o 15. (Para más detalles de la amplificación por PCR, por favor vea: 12 Determinar el número de ciclos óptimo mediante la ejecución de las reacciones de PCR en un gel de poliacrilamida al 5% y tinción con SYBR Green, asegurando la ausencia de bandas espurias y la presencia de una mancha de 400 -. 600 pares de bases, que es la longitud de los productos cortados después de la ligadura de los adaptadores.
- Amplificar el resto de las cuentas de Hi-C-biblioteca con destino estreptavidina en una PCR a gran escala con el número óptimo de ciclos de PCR. Piscina de los productos PCR de los pozos por separado y recuperar las bolas. Mantener el 1% del producto de PCR a gran escala por separado para ejecutarse en un gel y purificar el resto del producto de PCR con 1.8x cuentas Ampure volumen de acuerdo con las recomendaciones del fabricante.
- Eluir el ADN con 50 l de amortiguación TLE 1x y comparar el 1% del producto Ampure bolas de PCR purificado de la alícuota del 1% del producto original de PCR en un gel de poliacrilamida al 5%, lo que garantiza la eliminación con éxito de la PCR.
- También se recomienda la clonación de un l de la biblioteca de Hi-C y determinar el producto de cerca de 100 clones usando secuenciación de Sanger. Esto le permitirá evaluar el número relativo de alignable Hi-C se lee en la mezcla de PCR. Para los resultados típicos, ver la Figura 3B.
- Secuencia de la biblioteca de Hi-C con la secuenciación Illumina final emparejados. Alinear los extremos de forma independiente mediante Maq (http://maq.sourceforge.net/) para identificar los fragmentos de cromatina que interactúan.
IV. Representante de Hi-C Resultados
- Los siguientes resultados se espera que el protocolo de Hi-C se ejecuta técnicamente bien y se puede considerar las normas de control de calidad.
- Medidas de control de calidad se evidencia que las dos bibliotecas 3C y Hi-C ejecutar las bandas que más bien estrechas de más de 10 kb. Una prueba de ADN indica la eficiencia de la ligadura de los pobres. Por lo general, la eficiencia de la ligadura es ligeramente inferior en una biblioteca de Hi-C, en comparación con una plantilla de 3C (ver Figura 2a).
- Marca Hi-C y la eficiencia de la ligadura se puede estimar por la digestión de un producto de PCR utilizando los cebadores generado 3C. 3C uniones son cortadas por HindIII y no por NheI. Lo contrario es cierto para Hi-C uniones. Este ensayo de PCR resumen muestra que el 70% de Hi-C amplicones son cortadas por NheI y no por HindIII, lo que confirma el marcado eficiente de los cruces de la ligadura (ver Figura 2b).
- El análisis de la secuencia se lee debe demostrar que se lee de las interacciones tanto intracromosomal y interchromosomal, indicado por las líneas azules y rojas, alinear mucho más cerca de los sitios de restricción HindIII en comparación con los generados al azar lee, que se muestra en color verde (ver Figura 3a).
- En un experimento exitoso, el 55% de las parejas alignable leer representar las interacciones interchromosomal. El quince por ciento representan las interacciones entre los fragmentos intracromosomal menos de 20 kb aparte y el 30% son parejas intracromosomal leído que son más de 20 kb aparte (ver Figura 3B). Esta distribución puede ser muestreados antes de la secuenciación de alto rendimiento, como una forma de control de calidad, la clonación y secuenciación de Sanger de cerca de 100 clones suele ser suficiente.
- Las interacciones de la cromatina puede ser visualizado como un mapa de calor, donde x e y ejes representan loci con el fin de genómica y cada píxel representa el número de interacciones observadas entre ellos. Por lo general, los fragmentos de ADN que están muy cerca el uno al otro en el genoma lineal tendrá la tendencia a interactuar con frecuencia con los demás. Esto se ve en los mapas de calor intracromosomal como un destacado diagonal (ver Figura 4).
- Los siguientes resultados muestran diferentes maneras de analizar los datos para mostrar los distintos niveles de la organización del genoma. El trazado de la probabilit contactodistancia y genómica contra (ver Figura 5) muestra que la probabilidad de contacto disminuye en función de la distancia genómica, llegando a una meseta. A cualquier distancia, las interacciones intracromosomal, que se muestra en la línea continua, se enriquecen con respecto a las interacciones interchromosomal, representada por las líneas punteadas. Esto implica directamente la presencia de los territorios de los cromosomas.
- El cálculo del número observado / esperado de contactos interchromosomal entre todos los pares de cromosomas revela asociación preferencial entre los pares de cromosomas en particular. Cromosomas pequeños ricos en genes preferentemente interactuar unos con otros, se indica por el color rojo brillante (Ver Figura 5B).
- Cromosomas individuales también puede ser examinado. El mapa de calor en bruto se puede ajustar con un mapa de calor se espera para tener en cuenta la distancia genómica entre pares de loci, lo que resulta en un mapa de calor observado / esperado. Entonces, una matriz de correlación puede ser producido por la correlación de las filas y columnas de la mapas de calor observado / esperado. Utilizando el análisis de correlación, se demuestra que la separa del genoma humano en dos compartimentos. Esto se ilustra por el escocés-patrón de la correlación de mapas de calor (ver Figura 4-D). (Para más detalles sobre el análisis de datos Hi-C, por favor ver: 1.
- Utilizando datos de Hi-C, las nuevas ideas se consiguieron que se retire de la cromatina en la escala de megabase. El modelo clásico de polímero de condensación sugiere que los paquetes de la cromatina en un glóbulo de equilibrio. Probabilidad de trazado de contacto en función de la distancia pone de manifiesto que las escalas de probabilidad de contacto con una ley de potencia con la distancia genómica cuya pendiente es de aproximadamente -1 (Ver Figura 6). Esto no es consistente con el comportamiento de un glóbulo de equilibrio, pero la altura de las expectativas de una estructura alternativa se conoce como un glóbulo fractal (véase la Figura 6B).
- Aquí, dos estructuras globulares se muestran. Coloración corresponde a la distancia de un punto final, que van desde el azul al cian, verde, amarillo, naranja y rojo (Vea las Figura 6C). A diferencia de los glóbulos de equilibrio, glóbulos fractal falta enredos. En un glóbulo fractal, los lugares que están cerca a lo largo del contorno tienden a estar cerca en 3D, lo que lleva a la presencia de bloques monocromática (Ver Figura 6C medio). Estos bloques no se encuentran en el glóbulo de equilibrio (Ver abajo la figura 6C).
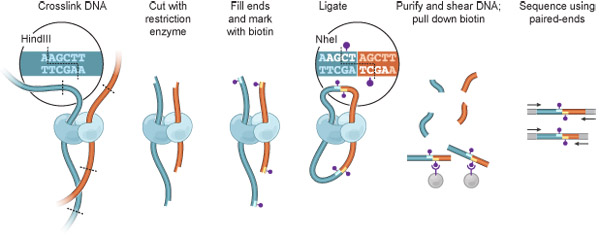
Figura 1. Hi-C visión. Las células se entrecruzan con formaldehído, dando lugar a enlaces covalentes entre los segmentos de la cromatina espacialmente adyacentes (fragmentos de ADN: azul oscuro, rojo, proteínas, que pueden mediar en estas interacciones, se muestran en azul claro y azul). Cromatina se digiere con una enzima de restricción (en este caso, HindIII, sitio de restricción: La línea de puntos, ver recuadro). Los extremos que resulta pegajosa se rellenan con los nucleótidos, de los cuales uno es con biotina (punto morado). La ligadura se realiza bajo condiciones extremadamente diluidas a favor de la ligadura de eventos intramolecular, el sitio HindIII se ha perdido y un sitio NheI se crea (recuadro). ADN se purifica y se recorta, y las uniones de biotina son aislados con bolas de estreptavidina. Fragmentos de la interacción se identificaron mediante la secuenciación dos a dos final.

Figura 2. Hi-C biblioteca de controles de calidad. (A) aumento de las cantidades de un control de 3C y una biblioteca de Hi-C se resolvieron en un 0,8% en gel de agarosa. Ambas bibliotecas se ejecute como una banda bastante estrecha de más de 10 kb. La eficiencia de la ligadura típica en una biblioteca de Hi-C es ligeramente inferior a lo que se observa en una plantilla de 3C, y es indicado por la mancha en los carriles de Hi-C. (B) PCR digerir control. Una unión ligadura formada por dos fragmentos de cerca se amplifica mediante PCR estándar 3C condiciones. Hi-C ligadura de los productos se pueden distinguir de los producidos en los convencionales 3C por la digestión de la ligadura de sitio. Hi-C uniones son cortadas por NheI no, HindIII, lo contrario es cierto para los cruces 3C. 70% de Hi-C amplificados fueron cortadas por NheI, lo que confirma eficiente marcación de salida ligadura. Dos réplicas se realizaron para garantizar una cuantificación fiable.

Figura 3. Hi-C lea los controles de calidad. (A) Lee a partir de fragmentos correspondientes a los dos intracromosomal (azul) y interchromosomal (rojo) las interacciones alinear mucho más cerca de los sitios de restricción HindIII, en comparación a las lecturas generadas al azar (verde). Tanto el intracromosomal lee y lee las curvas interchromosomal disminuir rápidamente a medida que la distancia desde el sitio HindIII aumenta hasta una meseta que se alcanza a una distancia de ~ 500 pb. Esto se corresponde con el tamaño de los fragmentos máximo utilizado para la secuenciación. (B) Por lo general, el 55% de las parejas alignable leer representar las interacciones interchromosomal. El quince por ciento representan las interacciones entre los fragmentos intracromosomal menos de 20 kbaparte y el 30% son parejas intracromosomal leído que son más de 20 kb de distancia. Esta distribución puede ser muestreados antes de la secuenciación de alto rendimiento, como una forma de control de calidad, la clonación y secuenciación de Sanger de cerca de 100 clones suele ser suficiente.

Figura 4. El análisis de correlación demuestra que el núcleo se separa en dos compartimentos. (A) Mapa de calor correspondientes a las interacciones intracromosomal en el cromosoma 14. Cada pixel representa todas las interacciones entre un lugar de 1 Mb y otro lugar de 1 Mb, la intensidad se corresponde con el número total de lecturas (rango: 0-200 lecturas). Las marcas aparecen cada 10 MB. El mapa de calor muestra subestructura en forma de una intensa diagonal y una constelación de grandes bloques. (Cromosoma 14 es acrocéntricos;. El brazo corto no se muestra) Con el conjunto de datos Hi-C para calcular la probabilidad de contacto medio de un par de lugares a una distancia dada genómica, una matriz de expectativas se produce (B) corresponde a lo que sería observar si no hay estructuras de largo alcance. El cociente de estas dos matrices es una matriz observada / esperada (C), donde el agotamiento se muestra en azul y el enriquecimiento en rojo [rango: 0,2 (azul) a 5 (rojo)]. El patrón de bloques se hace más evidente. La matriz de correlación (D) ilustra la correlación [rango: -1 (azul) y 1 (rojo)] entre los perfiles de interacción intracromosomal de cada par de loci a lo largo de cromosoma 14. El patrón a cuadros sorprendentes indica la presencia de dos compartimentos en el cromosoma.

Figura 5. La presencia y la organización de los territorios de los cromosomas. (A) Probabilidad de contacto disminuye en función de la distancia genómica en el cromosoma 1, hasta alcanzar una meseta a ~ 90MB (azul). El nivel de contacto interchromosomal (rayas negro) es diferente para los diferentes pares de cromosomas, los loci en el cromosoma 1 son más propensos a interactuar con los loci en el cromosoma 10 (rayas verdes) y con menos probabilidades de interactuar con los loci en el cromosoma 21 (rayas rojas). Interacciones interchromosomal se agotan en relación con las interacciones intracromosomal. (B) observada / esperada serie de contactos interchromosomal entre todos los pares de cromosomas. El color rojo indica el enriquecimiento y el azul indica el agotamiento [rango: 0,5 (azul) y 2 (rojo)]. Pequeños, ricos en genes cromosomas tienden a interactuar más con otros.

Figura 6. El embalaje locales de la cromatina es consistente con el comportamiento de un glóbulo fractal. (A) la probabilidad de contacto en función de la distancia genómica, un promedio de todo el genoma (en azul). La multiplicación de ley de potencia importante se observa entre 500kb y 7Mb (región sombreada) con una pendiente de -1,08 (ajuste se muestra en azul). (B) Resultados de la simulación de la probabilidad de contacto en función de la distancia de equilibrio (rojo) y fractal glóbulos (azul). La pendiente de un glóbulo fractal está muy cerca de -1 (cyan), lo que confirma nuestra predicción teórica novela 1. La pendiente de un glóbulo de equilibrio es de -3 / 2, que coincide con las expectativas previas teórico. La pendiente para el glóbulo fractal se asemeja mucho a la pendiente observada en los resultados de Hi-C, mientras que la pendiente de un glóbulo de equilibrio no se ve en los datos de Hi-C. (C) Arriba: Una cadena de polímero se desarrolló, 4000 monómeros de largo. Coloración corresponde a la distancia de un punto final, que van desde el azul al cian, verde, amarillo, naranja y rojo Medio:. Un ejemplo típico de un glóbulo fractal extraídos de nuestro conjunto. Glóbulos fractal falta enredos. Loci que están cerca a lo largo del contorno tienden a estar cerca en 3D, dando lugar a la presencia de grandes bloques monocromática que se hacen visibles en la superficie y en la sección inferior:. Un glóbulo de equilibrio. La estructura es muy enredado; lugares que están cerca a lo largo del contorno (color similar) no necesita estar cerca en 3D.
Discusión
Se presenta un método de estudio de la arquitectura en 3 dimensiones del genoma mediante la asignación de las interacciones de la cromatina en una evaluación imparcial de todo el genoma manera. El paso más crítico experimental lo que diferencia a esta tecnología, aparte de trabajos anteriores - es la incorporación de los nucleótidos de biotina en la restricción de los extremos de los fragmentos entrecruzados antes de la ligadura de extremo romo. Realizar este paso con éxito permite a profundidad la secuencia de todas las conexiones de la ligadura, y da Hi-C de su alcance y poder.
El número de lecturas en última instancia, determinar la resolución de los mapas de la interacción. Aquí, un mapa de 1 Mb de interacción para el genoma humano se presenta con aproximadamente 30 millones de lecturas alineable. Con el fin de aumentar la resolución de "todo uso" por un factor de n, el número de lecturas debe ser incrementado por un factor de n 2.
La técnica de Hi-C pueden ser fácilmente combinados con otras técnicas, como la captura híbrida después de la generación de la biblioteca (para apuntar las partes específicas del genoma) y después de la ligadura inmunoprecipitación de cromatina (para examinar el entorno de la cromatina de las regiones asociadas a proteínas específicas).
Divulgaciones
Agradecimientos
Damos las gracias a A. Kosmrlj para el debate y el código; AP Aiden, Bao XR, M. Brenner, Galas D., W. Gosper, Jaffer A., A. Melnikov, A. Miele, Giannoukos G., C. Nusbaum, AJM Walhout , L. Wood y Zeldovich K. para las discusiones, y L. Gaffney y Wong B. para ayudar con la visualización.
Apoyado por una Fannie y John Hertz Fundación Graduate Fellowship, una ciencia de la Defensa Nacional y de postgrado de Ingeniería de becas, una beca de la NSF de posgrado, el National Space Biomedical Research Institute, y la subvención no. T32 HG002295 de la National Human Genome Research Institute (NHGRI) (EL); i2b2 (Informática para la Integración de la biología y la cabecera), el NIH-apoyados por el Centro de Informática Biomédica en el Brigham and Women s Hospital (LAM); concesión no. HG003143 del NHGRI, y un premio de la Fundación Keck distinguido joven estudiante (JD). Primas y se asigna Hi-C datos de la secuencia ha sido depositada en la base de datos GEO ( www.ncbi.nlm.nih.gov/geo/ ), la adhesión no. GSE18199. Visualizaciones adicionales están disponibles en http://hic.umassmed.edu .
Materiales
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
Referencias
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados