Method Article
Pantallas competitiva Genómica de Bibliotecas de levadura con código de barras
* Estos autores han contribuido por igual
En este artículo
Resumen
Hemos desarrollado completa e imparcial en todo el genoma para comprender las pantallas de genes de drogas y las interacciones genético-ambientales. Métodos para la detección de estas colecciones mutantes se presentan.
Resumen
En virtud de los avances en las tecnologías de secuenciación de próxima generación, tenemos acceso a nuevas secuencias del genoma casi a diario. El ritmo de estos avances se está acelerando, prometiendo una mayor profundidad y amplitud. A la luz de estos avances extraordinarios, la necesidad de métodos rápidos, en paralelo a definir la función del gen se vuelve cada vez más importante. Las colecciones de mutantes de deleción en todo el genoma de las levaduras y E. coli han servido como bestias de carga para la caracterización funcional de la función del gen, pero este método no es escalable, actual deleción del gen enfoques requieren cada uno de los miles de genes que componen un genoma que se eliminan y verifican. Sólo después de este trabajo está completo podemos seguir de alto rendimiento fenotipificación. Durante la última década, nuestro laboratorio ha refinado con una cartera de la competencia, en miniatura, de alto rendimiento de todo el genoma ensayos que se pueden realizar en paralelo. Este paralelismo es posible gracias a la inclusión de "etiquetas" de ADN, o "códigos de barras," en cada mutante, con el código de barras que sirve como indicador de la mutación y se puede medir la abundancia de código de barras que evaluará su aptitud mutante. En este estudio, se busca llenar el vacío entre la secuencia de ADN y con código de barras colecciones de mutantes. Para lograr esto, se introduce una interrupción transposón combinado-código de barras del enfoque que abre los ensayos paralelos de código de barras que recientemente secuenciado, pero mal caracterizado microbios. Para ilustrar este enfoque se presenta una nueva colección de Candida albicans interrupción de código de barras y describir cómo los microarrays basado en la próxima generación basada en la secuencia de plataformas se pueden utilizar para recoger 10.000 - 1.000.000 gen-gen y las drogas las interacciones entre genes en un solo experimento.
Protocolo
1. Información general
Hay varias formas de generar mutantes que llevan etiquetas de código de barras. El actual estándar de oro es el golpe de gracia de levadura (YKO) colección creada por un consorcio de laboratorios y se terminó en 2002 1. Desde el YKO original fue introducido, otras colecciones de levadura se han generado, en los fondos de cepa diferente, utilizando la sobre-expresión construye, y en otros microbios como E. coli 2. Al mismo tiempo, el esfuerzo de crear códigos de barras bibliotecas del shRNA está avanzando rápidamente, y de hecho, muchos de los principios de diseño de las colecciones de mamíferos se han adoptado a partir de la levadura. Para demostrar cómo los transposones código de barras puede ser una estrategia rápida y ampliamente aplicable para la creación sistemática de colecciones mutantes, nos centramos en una colección que creó recientemente en el hongo patógeno humano Candida albicans. Nuestro trabajo en Candida se basó en el éxito de las pantallas de códigos de barras en el S. cerevisiae, y se utiliza aquí como ejemplo de un organismo. El protocolo de la muestra, con pequeñas modificaciones puede ser utilizado para detectar cualquier organismo que puede crecer en cultivo en suspensión. Debido a que algunos organismos tienen las tasas más altas de la necesaria transformación y recombinación mitótica eficiente que se necesita para crear mutantes perfectos eliminación, por lo tanto hemos desarrollado un protocolo que utiliza el transposón mutagénesis in vitro para mutagenizar una biblioteca de ADN genómico, y luego transformado a estos fragmentos del genoma de código de barras en Candida albicans 3 , 4. Inspirado por el éxito de la colección original de YKO y su papel en los descubrimientos fundamentales sobre la naturaleza de las redes de genes 5-8, todo el genoma haploinsuficiencia 9, blanco de la droga y el mecanismo de acción de 10,11, y la esencialidad de los genes en el genoma 12 prevemos ampliar este enfoque a otros microbios será muy fructífera.
El protocolo siguiente se supone que la colección deseada mutante se ha creado (por ejemplo, YKO o Candida albicans colección de interrupción) y está disponible como cepas individualmente archivados. Para una descripción detallada de la construcción de la cepa ver 1,13,14.
2. Combine los mutantes individuales en un solo grupo
- Permitir una semana para generar agrupados alícuotas de células (se puede almacenar indefinidamente a -80 ° C).
- Descongele los stocks de glicerol congelado para las cepas de interés por completo, pero no deje que las células permanecen descongelados durante> 2 horas.
- Esterilizar una herramienta pin de 96 pozos, sumerja la herramienta de pin en el agua para eliminar cualquier resto de las células, seguido de dos caídas en los baños de etanol al 70% (por ejemplo, tapas de cajas pipeta de punta), llama la herramienta de pin y deje enfriar por 1 minuto. Tenga cuidado de la llama de la herramienta de pines de los baños de etanol. El nivel de los baños de etanol debería superar el nivel en el baño de agua para asegurar que todos traspaso de las células son flameado y se retira. Cambie el agua cada 4-6 apuntalamientos.
- Inserte la herramienta de pin estériles de 96 pocillos en un descongelado placa de 96 pocillos, revuelva suavemente y la transferencia de las células a una bandeja que contiene Nunc Omni YPD-agar como el antibiótico apropiado. Crecen colonias hasta que alcanzan el tamaño máximo a 30 ° C (2-3d). Para conservar las placas, nos parece más útil para consolidar cuatro placas de 96 pozos en una sola bandeja con Omni-~ 384 cepas.
- Después de las colonias han crecido, tomar nota de cualquier falta o cepas de crecimiento lento y repin estos a ~ 2 veces la masa de células que el resto de las cepas.
- Trabajar en un ambiente de microbiología (con fuego y material de laboratorio estéril, la placa de las inundaciones con los medios de 5-10 ml, en remojo durante 5 minutos y volver a suspender las colonias con un difusor de la célula. Vierta el líquido más células en un tubo de centrífuga cónico de 50 ml, y añadir glicerol 15% o DMSO al 7% (vol / vol).
- Mida el diámetro exterior de 600 de la piscina y el ajuste (por dilución o centrifugación) a una concentración final de 50 600 OD / ml con los medios de comunicación que contiene 15% de glicerol o 7% de DMSO.
- Alícuota en 40 volúmenes l en tiras de tubos PCR y se congelan a -80 º C.
3. Crecimiento de la piscina experimental
Este procedimiento se describe en la figura 1.
- Alícuotas de deshielo de la piscina (en tubos de PCR) en el hielo. Si no está utilizando la robótica, con el paso 5.
- Inmediatamente diluir (gently!) la piscina en los medios de comunicación con las drogas o la condición de la elección, inoculando a un OD 600 de 0,0625 en un volumen total de 700 l en una placa de 48 pocillos. Incluir al menos un disolvente adecuado control de la placa. Para los experimentos que se extienden más allá de cinco generaciones de crecimiento (es decir, más de un bien), llenar los pozos adyacentes con los medios de comunicación o la condición de la elección, pero las células NO.
- Sellado con un sello de placa de plástico, si la condición requiere el crecimiento aeróbico (por ejemplo, no fermentable fuentes de carbono), el uso de una aguja de calibre 21 para perforar los agujeros en el sello de la membrana hacia el lado de cada pozo.
- Crecer en un espectrofotómetro, agitando a 30 º C con una determinación experimentalned sacudiendo el régimen (por ejemplo, movimiento de 14 minutos en posición más alta (o con temperatura controlada agitador), leer los pozos, reanudar agitación). Parte de la suspensión de células pueden ser cosechadas por el robot y se guarda en un plato frío en la cubierta de robótica en los puntos definidos por el usuario período de generación, por lo general 5, 10, 15 y 20 generaciones de crecimiento. (Http://med.stanford.edu/sgtc/technology/access.html, los datos de contacto Nislow C o Giaever G.).
- Para el crecimiento celular manual, inocular un cultivo de 50 ml a un punto de partida OD 600 de 0.002 en un frasco de cultivo de 250 ml. Agite a 30 ° C a 250 rpm hasta que las células llegar a una final OD 600 de 2,0 (para cerevisiae o Candida) de aproximadamente 10 generaciones de crecimiento. Generaciones adicionales de crecimiento se puede obtener por las células de la dilución en una OD 600 de 2,0 a 0,02 de nuevo en un frasco de dulce.
- Cosechar por lo menos 2 ~ OD 600 unidades de las células de cada muestra / punto de tiempo en tubos de microcentrífuga Safe-Lock.
Nota: Siempre recoger una muestra de células de partida (es decir, un "punto de tiempo T0") para evaluar la representación de la tensión inicial en cualquier piscina de nueva creación mediante la adición de 2.1 OD 600 de la piscina directamente de las alícuotas del congelador a un tubo de 1.5 ml y el tratamiento como se describe a continuación.
4. Extracción de ADN genómico, PCR e hibridación de microarrays o secuenciación
- Purificar el ADN genómico a partir de ~ 2 600 OD de las células con el Zymo Investigación YeaStar kit de acuerdo con las instrucciones del fabricante (Protocolo I si se utiliza el ADN de levadura), u otro método apropiado específico para el organismo de interés (estándar de extracción con fenol / cloroformo seguido de precipitación con alcohol funciona bien para diversos microbios). Si se utiliza el kit YeaStar, eluir el ADN con 300 L de 0,1 X TE en lugar de la l 60 de 1X TE especificado en el protocolo. ADN genómico se puede almacenar indefinidamente a -80 ° C.
- La creación de dos reacciones de PCR para cada muestra, una para el uptags y otro para el downtags, con las condiciones de reacción de la siguiente manera: 33 l ddH2O, 6 l buffer 10X PCR sin MgCl 2, 3 l 50 mM MgCl 2, 1,2 l 10 mM dNTPs, 1,2 l 50 M Up o Down mix primer, 0,6 l 5 U / l de Taq polimerasa, ~ 0,1 ug de ADN genómico en 15 microlitros. El volumen total es de 60 microlitros. Thermocycle bajo las siguientes condiciones: 94 ° C 3 min, 30 ciclos de 94 ° C 30, 55 ° C 30, 72 ° C 30 y luego 72 ° C 3 minutos, y mantener a 4 ° C. Revise los productos resultantes de PCR en un gel, un producto de 60 pb para ambas PCR se espera para amplicones utilizaron para la hibridación y 130bp para amplicones para la secuenciación de códigos de barras). Los productos de PCR se pueden almacenar a -80 ° C por tiempo indefinido.
- Precalentar el horno de hibridación de temperatura a 42 ° C y establecer un baño de agua hirviendo y un cubo de hielo que contienen una mezcla de hielo y agua.
- Humedecer previamente las matrices de llenado lentamente con 120 l de hibridación buffer 1X.
- Incubar en el tampón de hibridación a 42 ° C y 20 rpm durante 10 minutos.
- Preparar 90 l de mezcla de hibridación por muestra, y un poco más como un amortiguador uno, de la siguiente manera: 75 l buffer de hibridación 2X, 0,5 l B213 oligonucleótidos de control (0,2 fm / l), 12 l oligonucleótidos mixtos (24:05 / l), l 3 solución de Denhardt 50X) en la cerradura-top tubos de 0,5 ml.
- Añadir 30 l de PCR uptag y 30 l PCR downtag a 120 l de mezcla de hibridación para un volumen total de 150 mL. Hervir durante 2 minutos y poner en agua con hielo durante al menos 2 minutos. En pocas palabras girar los tubos antes de su uso.
- Retire el buffer de pre-hibridación de las matrices, añadiendo 90 hibridación l / mezcla de PCR. Para evitar la evaporación, cubrir las juntas matriz con una situación difícil. Hibridar durante 16 horas a 42 ° C, 20 rpm.
- Recién preparar 600 mix l etiquetado biotina por ejemplo, más uno extra, de la siguiente manera: 180 l 20X SSPE, 12 l Denhardt 50X, 6 l 1% de Tween 20 (vol / vol), 1 l de 1 mg / ml de estreptavidina-ficoeritrina, 401 l ddH 2 O. Guarde todas las muestras de estreptavidina-PE en la oscuridad. Alícuota de 600 l en tubos de 2 ml. Quitar Tough-Spots de chips.
- Retire lentamente la mezcla de hibridación de las matrices con una pipeta y llenar microarrays con 120 l Lave A. El primer de la estación de fluidos Affymetrix.
- Lave las matrices con un Affymetrix fluidos estación de acuerdo con las instrucciones del fabricante, utilizando el "Gene-Flex_Sv3_450" protocolo con las siguientes modificaciones: 1 paso adicional con lavado A (ciclo 1 º, 2 mezclas) antes de la tinción, temperatura de lavado B 42 ° C en lugar de 40 ° C, la mancha a 42 ° C en lugar de 25 ° C. También es posible llevar a cabo el lavado posterior hibridación, la tinción de la biotina, y el lavado posterior tinción manual (véase p. 396, en referencia 15). A raíz de las operaciones de fluidos, ejecute la estación de fluidos "SHUTDOWN_450" protocolo.
- Tras el lavado, asegurarse de que no hay burbujas de aire. Si es necesario añadir 90 l lavado A y la pipeta lentamente hasta que las burbujas desaparezcan. Si hay alguna marca o smudges en la superficie de la matriz, limpia la ventana de cristal con isopropanol y un pañuelo que no suelte pelusa. Aplicar nuevas Tough-Spots en las juntas / tabiques y el lugar de las matrices en el escáner.
- Exploración en un escáner Affymetrix GeneArray una longitud de onda de emisión de 560 nm.
5. Matriz de análisis (ver Figura 2 para ver un ejemplo obtenido utilizando la colección Candida albicans interrupción)
- Enmascaramiento atípico: Dado que la matriz Affymetrix tag4 contiene 5 réplicas de cada complemento de código de barras dispersos al azar cualquier sonda de código de barras que se desvía de las réplicas se pueden enmascarar y se desecha. Para ello, para cada función de matriz que parece ser un caso atípico en función de su señal en comparación con las otras cuatro réplicas de esta función, el software examina en primer lugar las 5 características que rodean el valor atípico sospechoso, produciendo una matriz de 25 funciones con el sospechoso característica en el centro. Si> 13/25 sondeos en esta región difieren de cada uno de sus su persona significa replicar recortado (la media de las medias tres repeticiones, excluido el de mayor y menor repeticiones) en más del 10%, esta sonda es entonces descartada del análisis. Debido a que los valores extremos son más a menudo el resultado de las inconsistencias de lavado post-hibridación, que se expanden o "pad" de la región que contiene sondas sospechoso. Pad como sondas mediante la inclusión de todas las sondas en un radio de 5-sonda, tal como se define por ((x 1-x 2) 2 + (y 1-y 2) 2) ½ <6, donde x 1, x 2, y 1, y y 2 son las coordenadas X e Y para las dos funciones. Por último, descartar las características para que la desviación estándar (incluido en el archivo. Cel de Affymetrix arrays) de píxeles función / media píxeles función. Después de la eliminación de valores atípicos, los valores promedio de la intensidad de todas las demás réplicas.
- La eliminación de etiquetas inservibles: Etiquetas con los valores de baja intensidad se da de baja calidad y los resultados deben ser eliminados. Un corte de exclusión de estas sondas de baja intensidad se puede calcular de la siguiente manera:
- Para cualquier par el tratamiento de control de matrices, calcular log 2 ((i c-b g) / (i t-b g)) para cada etiqueta, donde c es la intensidad del control, t i es la intensidad del tratamiento, y b g es la intensidad media de las sondas de etiqueta sin asignar.
- Par el uptag y ratios downtag por la tensión y para cada par de etiquetas, tener la intensidad mínima para las dos marcas en las dos muestras. Clasificar los pares relación de esta intensidad mínima.
- Use un tamaño de ventana deslizante de 50 en la relación de parejas clasificadas, calcular la correlación de uptag y downtag pares de relación dentro de la ventana. Asimismo, el cálculo de la media de las intensidades mínimas calculadas en el paso anterior.
- Deslice la ventana por 25 pares, y repita el paso anterior hasta que todas las parejas se han cruzado.
- Parcela de la intensidad media mínima en comparación con la correlación uptag-downtag para todas las ventanas.
- Por último, elige un umbral de intensidad, por lo general se utiliza el valor de la intensidad en la primera correlación alcanza el 80% de su nivel máximo. Marcar y quitar de los nuevos análisis de las etiquetas por debajo de este límite.
- Corrección de la saturación: Debido a que cada función en el microarray de código de barras puede llegar a saturarse, la señal de la matriz tag4 no se relaciona linealmente con la concentración de la etiqueta. Para corregir esta saturación de seguir el protocolo descrito en la referencia 16.
- Normalización matriz: Para cada matriz, normalizar la uptags y downtags por separado. Para normalizar las cuantiles, el rango de los valores obtenidos de cada matriz de uptags downtags y en orden de intensidad creciente. En el sentido de normalizar para cada conjunto de uptags y downtags, se divide por la media. La normalización significa de todas las matrices es un segundo paso después de la transformación de los datos en bruto a la media de cada matriz.
- El cálculo de las puntuaciones de sensibilidad para las comparaciones de tratamientos de control: Para el uso diario de 2 proporciones como un indicador de sensibilidad: Para cada cepa, calcular log 2 ((μ g c-b) / (μ g t-b)), donde μ es la c la intensidad media de las muestras de control, μ t es la intensidad media de las muestras de tratamiento, y b g es la intensidad media de las sondas sin asignar. Las cepas con un registro positivo 2 son sensibles a la relación entre el tratamiento, y aquellos que son resistentes a tener efectos negativos log 2 ratios.
6. Evaluar la aptitud de las cepas de levadura con código de barras mediante la secuenciación
- Aislar el ADN de las piscinas de eliminación tal como se describe para los microarrays.
- Amplificar cada código de barras 20-mer uptag con cebadores compuesto formado por las secuencias de los cebadores de códigos de barras comunes y las secuencias necesarias para la hibridación de la célula de flujo Illumina (Ver Tabla de primers Illumina y la Figura 3 para ver un diagrama de la amplificación). Estosprimers se puede utilizar desalado sin purificación adicional. PCR se realiza en 100μL, utilizando Invitrogen Platino PCR Supermix (Cat. N º 11306-016) con las siguientes condiciones: 95 ° C / 3 min, 25 ciclos de 94 ° C/30 seg, 55 ° C/30 seg, 68 ° C/30 segundos, seguido por el 68 ° C/10 min.
- Purificar el producto de PCR (~ 130bp) con Qiagen MinElute 96 UF PCR Purificación Kit (Cat. N º 28.051).
- Después de purificación de PCR, con la cuantificación de ADN Invitrogen Quant-iT dsDNA BR Kit de ensayo (Cat. No. Q32853). No confíe en 260/280 lecturas!
- Normalizar la concentración de ADN a los volúmenes de 10μg/ml y piscina iguales de ADN normalizado.
- ADN se separan agrupados en un gel de poliacrilamida TBE 12% durante 3-4 horas dependiendo del voltaje utilizado.
- Tinción de geles con bromuro de etidio (SYBR Green debería funcionar también) durante 30 minutos.
- Localice la banda de interés en una caja de luz ultravioleta de onda larga (el uso de protección para la cara adecuada), se corta y se extrae el ADN utilizando el método de aplastar y absorber 17 seguido de precipitación con etanol.
- Confirmar que el ADN de tamaño apropiado (130bp) ha sido aislado y que los cebadores se han eliminado con el Agilent Bioanalyzer alta sensibilidad ADN kit (Cat. No. 5,067 hasta 4626).
- Muestra la secuencia:
- Illumina GAIIx plataforma:
- Generar clusters en una celda de flujo de una sola lectura utilizando el Kit de Grupo Generación CBOT y solo lectura (Cat. N º DG-300-1001). Para Leer 1, arriba y abajo-tag modificado cebadores de secuenciación se agrupan en una concentración de valores 100um y se añade a una tira de tubo (0.6μL de cada cebador de secuenciación 100um en 120 l HT1). SR_Amp_Block_StripTubeHyb_v7.0 receta se utiliza para generar grupos de R1.
- La secuenciación del genoma IIx Analyzer. Después de 18 ciclos de secuenciación, el módulo de pares de gama se utiliza para pelar la hebra sintetizado por primera vez y rehybridize la célula de flujo, con el R1 Illumina (abajo). Los clusters son regenerados y secuenciado para 5 ciclos para capturar la etiqueta de índice.
- Illumina GAIIx plataforma:
- La secuencia de la etiqueta de índice se utiliza para las secuencias en los contenedores bin experimental.
- Dentro de cada contenedor experimental, las secuencias de código de barras de levadura se cuentan para dar un número total de cargos para cada código de barras.
- Que cuenta son cuantil normalizado para cada experimento tiene la distribución misma cuenta. Por analogía con los experimentos de microarrays de código de barras de fitness, gimnasio ratios defecto para cada cepa se calculan y se expresa como la relación de log 2 (control / tratamiento). Resultados positivos defecto físico significar una disminución en la abundancia de la tensión durante el tratamiento de drogas y sugieren que la versión de tipo salvaje del gen suprimido en que la tensión es necesaria para la resistencia a ese medicamento o inhibidor.
Nota: Considerando Bar-ss como una alternativa a la hibridación matriz. Con los costos de secuenciación de alto rendimiento decreciente, utilizando secuenciación de alto rendimiento como una lectura de la abundancia de etiqueta se está convirtiendo en factible y en muchos casos, es más rentable 18. De esta manera, amplificación de PCR se mide directamente como 'cuentas' y no como intensidad de la señal híbrido en una matriz. Esto elimina los falsos negativos y positivos que surgen de la etiqueta de la contaminación cruzada, la saturación, o los problemas derivados de la intensidad de señal muy alta o muy baja. Además, los experimentos se pueden combinar varios antes de la secuencia de la adición de una base de ADN 4.8 índice de 19. Debido a que los códigos de barras son la levadura de 20 pb, uno, dos pasos de lectura de 26-28 bases captura el índice de multiplex y código de barras único, lo que permite extrema de 100 multiplexación. En el momento de escribir estas líneas, Bar-ss ofrece una ventaja económica en microarrays de códigos de barras, y, además, Bar-ss es inherentemente flexible de tal manera que como aumenta el número de lecturas / ejecución, el nivel de multiplexación puede aumentar los costos de reducir aún más . Varios "a mediados de la capacidad" de todos los secuenciadores de los fabricantes de las principales plataformas aún más la democratización de Bar-ss, con la secuencia de probabilidades de convertirse en la lectura de la elección.
Este protocolo también ha sido validado en la Illumina HiSeq2000.
Una excelente demostración de la utilización de Bar-ss para hacer frente a una cuestión fundamental en el control biológico Saccharomyces cerevisiae el crecimiento se presenta en un estudio reciente de Gresham et al. 20 que destacan varias importantes del diseño experimental y pautas de interpretación.
7. La validación de los datos del cribado combinado
Los resultados de cualquier pantalla de la genómica funcional debe ser verificada con las cepas individuales en la cultura aislada. Debido a que cada experimento será diferente en función del número de cepas sensibles, seleccionar el número de cepas candidatas para confirmar es algo arbitrario. Como una guía, la clasificación de las cepas más sensibles por el log 2 ratio o z-score y las pruebas de la parte superior del 25-50% de los candidatos (que normalmente se traduce en 3.2 estándar dedesviaciones de la media de todas las cepas en la piscina) es un buen balance entre costos y beneficios. Confirmaciones individuales se puede realizar en cualquier frasco, sino que realizan estas pruebas desde hace 5 generaciones de crecimiento en placas de 96 pocillos con un inóculo de partida de 0,06 OD 600 en 100 l de los medios de comunicación en un espectrofotómetro de agitación, tomando medidas cada 15 minutos (ver Figura 4) .
8. Resultados representante
Una vez que una pantalla de todo el genoma se ha completado, y las matrices se han normalizado y el comportamiento de cada cepa en comparación con un tratamiento de control (por ejemplo, comparando la intensidad de microarrays o secuenciación cuentas / cepa) los datos son más fáciles de manipular en un archivo de Excel con los genes calificadas por la log2 ratios de control / experimento. De esta manera, cuanto mayor es la proporción de log2 negativo, el más sensible que la cepa en particular es la condición de prueba. Estos archivos de Excel se pueden trazar en una variedad de paquetes de software de gráficos. Encontramos más simple que para trazar las relaciones log2 en el eje Y y el gen o los nombres de ORF en el eje X. En el ejemplo que se muestra en la Figura 2a, como una parcela de tratamiento de clotrimazol (un agente antifúngico conocido) se muestra. Todos los cepa que son significativamente más sensibles al tratamiento con una proporción de 2 log2 se resaltan en rojo, y normalmente verificar muchas de estas cepas en los ensayos de crecimiento individual de cada mutante en presencia de la misma concentración de la droga. En este ejemplo, se destacan cuatro cepas, NCP1, ERG2 y 2 alelos independientes de ERG11, el blanco de la proteína conocida de clotrimazol. Cada uno de estos cuatro genes directamente implicados en la biosíntesis de ergosterol, la levadura equivalente de colesterol. Por ejemplo, NCP1 codifica una reductasa NADP-P450 citocromo que participa en la biosíntesis de ergosterol y que está asociado y regulado de forma coordinada con Erg11. Este ejemplo pone de relieve el hecho de que el blanco de la droga conocida (Erg11) se identifica en esta pantalla imparcial, así como varios otros componentes clave de la vía de destino. Finalmente, varios de los genes en rojo representan los genes que pueden estar implicados en la biosíntesis de ergosterol o en diferentes procesos biológicos. Como se mencionó anteriormente, cada cepa detectada como sensibles en una pantalla agrupados deben ser verificados en cuanto a su sensibilidad en el ensayo de crecimiento individual. En el ejemplo que se muestra en la Figura 2b, cuatro cepas se confirmó a ser sensibles al clotrimazol sobre la base de su crecimiento reducido en relación con la cepa de tipo salvaje, BWP17. Estas curvas de crecimiento individuales resaltar una característica importante de estos medicamentos combinados gen-pantallas, es decir el grado absoluto de una cepa en particular no refleja necesariamente su nivel exacto de la sensibilidad. Por otra parte, la Figura 2b muestra también el valor de tener múltiples alelos para cada gen, en este caso, los dos mutantes erg11 interrupción tiene ligeramente diferentes sensibilidades. La correlación de la naturaleza de estos trastornos con el grado de sensibilidad puede proporcionar información adicional sobre el mecanismo de acción de las drogas.
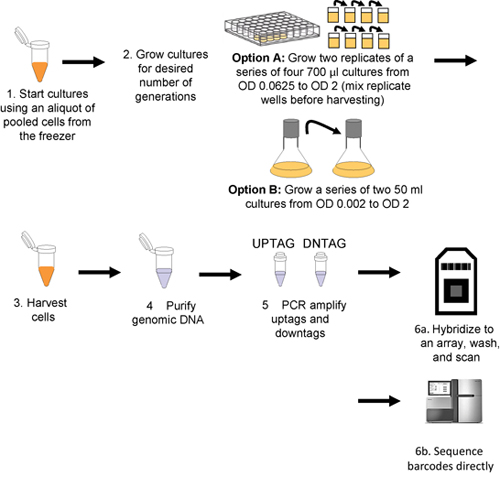
Figura 1. Flujo de trabajo para el ensayo de crecimiento común y de detección de códigos de barras. Las culturas son inoculadas con alícuotas de las células descongeladas agrupados (paso 1), y entonces ha crecido el número deseado de las generaciones (paso 2) o bien un robot (opción A) o manual (opción B). Se toman las células por centrifugación (paso 3) y el ADN genómico se aísla de las células recogidas (paso 4), y uptags downtags se amplifica de manera independiente (paso 5), y se hibridan a una matriz (el paso 6a o secuenciados directamente el paso 6b).
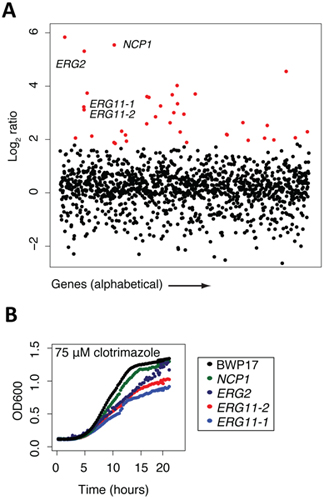
Figura 2. Datos de las muestras recogidas en ciertos puntos del protocolo. (A) Datos de ejemplo de resultados de la prueba (adaptado de 13). El grupo de mutantes marcados se cultivó durante 20 generaciones en la presencia de clotrimazol y DMSO (control). Log 2 ratio (control de intensidad / tratamiento de intensidad) se calculó y se representa como una función del gen. Cepas de alta sensibilidad (rojo) incluye el objetivo de conocer de clotrimazol, ERG11p. Tenga en cuenta que este ensayo con frecuencia descubre otros mutantes sensibles, además de objetivo real del compuesto. Por lo general, estos mutantes son los que actúan de forma sintética con el objetivo, los que forman parte de un general de la tensión / la respuesta al tratamiento, o son falsos positivos que no confirman. (B) Ejemplo de los datos de confirmación (adaptado de 13). Los resultados de los ensayos de crecimiento acumulada puede ser validado por la creciente tensión en la cultura individual y se compara con el crecimiento de tipo salvaje (negro).
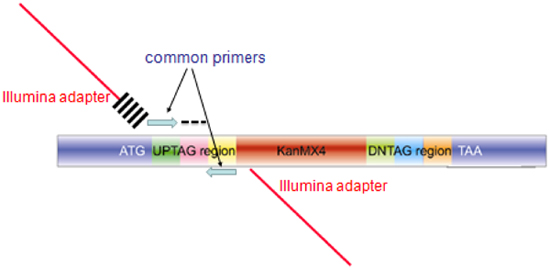
Figura 3. Estructura de la amplificación producida a partir de los ensayos de código de barras agrupadas para la hibridación de microarrays o secuencia de código de barras. El amplicón producido para cada mutante in la colección contiene homología con el genoma de la integración (zonas azules y etiquetados ATG TAA), códigos de barras únicos (con la etiqueta AG y se indica mediante un guión negro). Para la hibridación de microarrays, los primers comunes azules se utilizan para amplificar una sonda para la hibridación de microarrays 60bp. Para la secuencia de código de barras, primers extendidas se usan en la reacción de PCR, compuesto por secuencias que codifican el adaptador de Illumina (barra roja), y el índice de 6bases cruzada escotillas) y el primer azul común para el primer aguas arriba, y menos el mismo primer compuesto ( la base del índice 6) para el primer segundo.
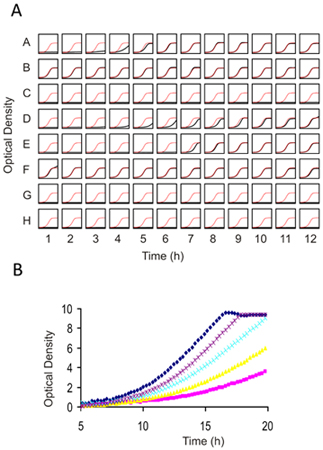
Figura 4. Ensayos individuales de crecimiento para 1) Preselección compuestos contra la de tipo salvaje de levadura para determinar la dosis adecuada de todo el genoma de detección y 2) que confirma los resultados de las pantallas de todo el genoma. (A) Una placa de fondo plano de 96 pocillos se llena con 100 l de suspensión celular en un diámetro exterior de 0.062. Cada pozo puede contener la misma cepa (por dosis-determinación), o diferentes combinaciones de tensión y de medicamentos (para los ensayos de confirmación). 2 l de compuesto (normalmente disuelto en DMSO) y se las células se cultivan con agitación constante durante 16-20 horas a 30 ° C. La concentración final de DMSO no debe superar el 2%. En este ejemplo, en cada pocillo de la placa de la curva de crecimiento se ha trazado en negro sobre una parcela de la curva de crecimiento de control en rojo. (B) Mayor resolución de imagen de varios prescreens obtenidos con un fármaco ejemplo superpuestos uno encima del otro. En esta serie de titulación, un IC 10-15 se obtiene con la dosis de púrpura y sería apropiado para la eliminación de perfiles (HIP HOP, y). Debido a la no linealidad a altas densidades ópticas, Tecan (o cualquier lector de placas similares) OD debe ser calibrado con los obtenidos con una "tradicional" de 1 mm de longitud de recorrido de la cubeta.
Discusión
En este sentido, se indica un protocolo que, con la modificación de modesto, se puede adaptar fácilmente a una amplia gama de las colecciones existentes de código de barras colecciones de mutantes de microorganismos diferentes para crear colecciones de mutantes marcados. Hacemos hincapié en que, si bien nos han informado de un protocolo sobre la mutagénesis de transposones etiquetados para el patógenos levadura C. albicans, un protocolo muy similar podría ser adaptado a una amplia variedad de hongos unicelulares. Modificado, este protocolo funciona muy bien en las bacterias 13, y en la actualidad las colecciones de un número adicional de genomas de hongos y bacterias se encuentran en construcción. En la actualidad, este ensayo proporciona la única completa de todo el genoma de pantalla imparcial de las interacciones gen-molécula pequeña. Una característica particularmente atractiva de este ensayo es que ningún conocimiento previo del gen o una molécula pequeña es necesaria. A pesar del alcance y el poder de estos ensayos, que se puedan transferir a otros laboratorios se ha visto obstaculizada tanto por la inversión de capital inicial y las herramientas informáticas para el análisis de los resultados. Con la adopción de una lectura de la generación siguiente secuencia en combinación con potentes herramientas para el análisis, se espera su aprobación para aumentar.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Damos las gracias a Ron Davis, Deutschbauer Adán, y el laboratorio de HIP HOP todo en la Universidad de Toronto para el debate y asesoramiento. CN es apoyado por becas de la National Human Genome Research Institute (número de concesión HG000205), RO1 HG003317, CIHR MOP-84305, y la Sociedad Canadiense del Cáncer (# 020380). JO fue apoyada por el Programa de Entrenamiento de Stanford Genome (número de concesión T32 HG00044 de la National Human Genome Research Institute) y los Institutos Nacionales de Salud (número de concesión P01 GH000205). GG es apoyado por el NHGRI HG003317 SR1 y la Sociedad Canadiense del Cáncer, subvención n º 020380, TD y el Centro de Secuenciación Donnelly es apoyado en parte por subvenciones de la Fundación Canadiense para la Innovación a los Dres. Brenda Andrews y Greenblatt Jack. AMS es apoyado por una beca de la Universidad de Toronto en Abrir.
Materiales
| Name | Company | Catalog Number | Comments |
| Antibióticos | Proveedor y números de catálogo | ||
| Carbenicilina | Sigma, parte # C1613 | ||
| Kanamicina | Sigma, parte # K1876 | ||
| espectinomicina | Sigma, parte # S0692 | ||
| Cloranfenicol | Sigma, parte # C0378 | ||
| ADN de limpieza y kits de concentración | |||
| QIAprep Spin Miniprep Kit | Qiagen, parte # 27106 | ||
| Kit Hi-Speed Maxi plásmido | Qiagen, parte # 12663 | ||
| QIAquick PCR kit de purificación | Qiagen, parte # 28106 | ||
| QIAquick Gel Extraction Kit | Qiagen, parte # 28704 | ||
| PCR y los reactivos de electroforesis | |||
| Taq ADN polimerasa (Mg libre) Buffer | New England Biolabs, parte # M0320L | ||
| Mezcle la solución desoxinucleótidos | New England Biolabs, parte # N0447L | ||
| 25 mM MgCl 2 | Sigma, parte # 63036 | ||
| Agarosa, colorante de carga, y el ácido nucleico mancha adecuada para electroforesis en gel | Varios | ||
| 10X tampón TAE | Sigma, parte # T8280 | ||
| Más de 1 Kb DNA Ladder | Invitrogen, parte # 10787026 | ||
| YPD caldo | |||
| 10 g de extracto de levadura | Sigma, parte # Y1625 | ||
| 20 g de Bacto peptona | BD Biosciences, parte # 211677 | ||
| 20 g de dextrosa | Sigma, parte # D9434 | ||
| Material de laboratorio | |||
| Pipetas multicanal (1000, 200, y el l 20) | Varios | ||
| Depósitos desechables pipeteo | Varios | ||
| 15 y 50 tubos de centrifugación ml | Varios | ||
| 96 - y 384-Pozo Profundo placas | Axygen Scientific, parte # P-2ML-SQ-CS & P 384240SQCS- | ||
| 96 y 384 así-PCR placas y película de sellado | Varios | ||
| Compactador para el sellado de varias placas | Sigma, parte # R1275 | ||
| 30 ° C y 37 ° C agitando incubadoras para el cultivo de bacterias y levaduras en las placas y tubos | Varios | ||
| Transposón mutagénesis in vitro | |||
| EZ-Tn5 transposasa | Epicentro biotecnologías, parte # TNP92110 | ||
| De alto rendimiento de la transformación | |||
| Seqprep 96 HT Kit de Preparación plásmido | Borde Biosystems, part # 84359 | ||
| polietilenglicol, peso molecular 3350 | Varios | ||
| acetato de litio | Sigma, parte # 517992 | ||
| Placas de 6 pocillos, estériles | Corning, parte # 3335 | ||
| 50 mg / ml uridina | Sigma, parte # U3750 | ||
| 100X Tris-EDTA solución | Sigma, parte # T9285 | ||
| 1X TE/0.1M LiOAc | Varios | ||
| salmón testículos ADN | Sigma, parte # 1626 | ||
| El crecimiento de las colecciones de código de barras | |||
| 48 placas, y si en las placas de crecimiento de cultivos | Greiner, parte # M9437 | ||
| Sellos adhesivos placa | ABgene, parte # AB-0580 | ||
| 200 ml frascos de cultivo | Varios | ||
| Espectrofotómetro capaz de absorbancia | Varios | ||
| Con control de temperatura agitador de matraces de 250 ml o espectrofotómetro temblor | Varios | ||
| Safe-Lock Microcentrífuga tubo, 2 mL | Eppendorf, parte # 0030 120.094 | ||
| Hibridación equipo | |||
| Horno de hibridación 640 | Affymetrix, parte # 800138 | ||
| GeneChip neumático Estación 450 | Affymetrix, pieza # 00-0079 | ||
| GeneArray Scanner 3000 | Affymetrix, pieza # 00-0212 | ||
| Baño de agua hirviendo con bastidor flotante | Varios | ||
| Consumibles de hibridación | |||
| Genflex etiqueta 16K matriz v2 | Affymetrix, parte # 511331 | ||
| Solución de Denhardt, concentrado 50 veces | Sigma, parte # D2532 | ||
| Estreptavidina, R-ficoeritrina conjugado (SAPE) | Invitrogen, parte # S866 | ||
| Safe-Lock Microcentrífuga tubos de 0,5 mL | Eppendorf, parte # 0030 123.301 | ||
| Teeny Tough-Spots | Diversificación de la biotecnología, parte # LTTM-1000 | ||
| EDTA 0,5 M | BioRad, part # 161-0729 | ||
| 10% de Tween | Sigma, parte # T2700 | ||
| MES monohidrato de ácido libre | Sigma, parte # M5287 | ||
| MES sal de sodio | Sigma, parte # M5057 | ||
| 5 M NaCl | Sigma, parte # 71386 | ||
| 20X PEES | Sigma, parte # S2015 | ||
| La biología molecular del agua de grado | Sigma, parte # W4502 | ||
| Primers hibridación | Varios proveedores (desalación estándar) | ||
| Uptag | 5 'GATGTCCACGAGGTCTCT 3' | ||
| Buptagkanmx4 | 5 'con biotina GTCGACCTGCAGCGTACG 3' | ||
| Dntag | 5 'CGGTGTCGGTCTCGTAG 3' | ||
| Bdntagkanmx4 | 5 'con biotina GAAAACGAGCTCGAATTCATCG 3' | ||
| B213 | 5 'con biotina CTGAACGGTAGCATCTTGAC 3' | ||
| Uptagkanmx | 5 'GTCGACCTGCAGCGTACG 3' | ||
| Dntagkanmx | 5'GAAAACGAGCTCGAATTCATCG 3 ' | ||
| Uptagcomp | 5'AGAGACCTCGTGGACATC 3 ' | ||
| Dntagcomp | 5'CTACGAGACCGACACCG 3 ' | ||
| Upkancomp | 5'CGTACGCTGCAGGTCGAC 3 ' | ||
| Dnkancomp | 5'CGATGAATTCGAGCTCGTTTTC 3 ' | ||
| Secuenciación Primers: Plataforma Illumina | Varios proveedores | ||
| UpTag Adelante (100um) | 5 'AAC GCA GAA GAC GGC ATA CGA GCT CTT CCG ATC GAT T GTC CAC GAG GTC TCT 3' | ||
| UpTag inversa (100um) | 5 'AAT GAT ACG GCG ACC ACC GAC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T NNNNN GTC CAG CAG CTG CGT ACG 3' | ||
| DownTag Adelante (100um) | 5 'AAC GCA GAA GAC GGC ATA CGA GCT CTT CCG ATC T AAC GAA GAG CTC GAA TTC ATC G 3' | ||
| DownTag inversa (100um) | 5 'AAT GAT ACG GCG ACC ACC GAC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T NNNNN CGG TGT TCT CGG CGT AG 3' | ||
| Leer 1 UP-tag siguientes cebadores (100um) | 5 'GTC CAG CAG CTG CGT ACG 3' | ||
| Leer 1 ABAJO-tag imprimación siguientes (100um) | 5 'TGT CGG CGG CGT TCT AG 3' | ||
| Lea 2 cebador de secuenciación Índice (de serie Illumina imprimación R1) (100um) | 5 'AC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T 3' | ||
| Reactivos adicionales Secuenciación / Equipo | |||
| Qiagen MinElute 96 UF Kit de purificación de PCR | Qiagen, parte # 29051 | ||
| Bomba de vacío | Cualquier proveedor | ||
| Macherey-Nagel vacío en el colector | Macherey-Nagel, parte # 740 681 | ||
| Invitrogen Quant-iT dsDNA BR Kit de ensayo | Invitrogen, parte # Q32853 | ||
| Invitrogen Qubit tubos de ensayo | Invitrogen, parte # Q32856 | ||
| 40% de acrilamida y 1% de N, N'-metilen-bis-acrilamida, 37.5:1 | Bio Rad, part # 161-0148 | ||
| Tris Base | Sigma, parte # T1503-1kg | ||
| Ácido bórico | Sigma, parte # B6768-500G | ||
| 0,5 M EDTA, pH 8,0 | Teknova, parte # E0306 | ||
| Persulfato de amonio | Sigma, parte # A3678-25G | ||
| TEMED | Bioshop, parte # TEM001.25 | ||
| Solución de bromuro de etidio | Bioshop, parte # ETB444.10 | ||
| Acetato de amonio 0,5 M | Teknova, parte # A2000 | ||
| 10 mM de acetato de magnesio tetrahidratado | Sigma, parte # M0631-100G | ||
| 1 mM EDTA, pH 8,0 | Ver 0,5 M EDTA, pH 8,0 | ||
| Etanol | Varios | ||
| Acetato de sodio, pH 5,2 | Teknova, parte # S0297 | ||
| Velocidad de vacío | Varios | ||
| Lea solo racimo kit de generación de | Illumina, parte # GD-300-1001 | ||
| 36c Secuenciación Kit v4 | Illumina, parte # FC-104-4002 | ||
Receta de TBE 10X
| Cantidades | Reactivos |
| 108 gramos | Tris Base |
| 55 gramos | Ácido bórico |
| 40 ml | 0,5 M EDTA (pH 8,0) |
| Añadir dH 2 O a 1 litro marca | |
12% de poliacrilamida gel receta
| Volúmenes | Reactivos |
| 5,8 ml | 40% de acrilamida y 1% de N, N'-metilen-bis-acrilamida, 37.5:1 |
| 12 ml | dH 2 O |
| 2 ml | TBE 10X |
| 140 l | 10% de persulfato de amonio |
| El volumen total de: 20 ml |
Uptag mezcla de cebadores:
Resuspender Uptag y Buptagkanmx4 cada uno en ddH2O a 100 M, a continuación, mezclar en una proporción de 1:1 para una concentración final de 50 M cada uno. Almacenar a -20 ° C.
Downtag mezcla de cebadores:
Resuspender Dntag y Bdntagkanmx4 cada uno en ddH2O a 100 M, a continuación, mezclar en una proporción de 1:1 para una concentración final de 50 M cada uno. Almacenar a -20 ° C.
Oligonucleótidos mixtos:
Resuspender cada uno de los siguientes ocho oligos (estándar desalada) en ddH2O a 100 M:
Uptag, Dntag, Uptagkanmx, Dntagkanmx, Uptagcomp, Dntagcomp, Upkancomp, Dnkancomp.
Mezclar un volumen igual de los ocho oligonucleótidos para una concentración final de 12,5 M cada uno.
12X MES acciones:
De 10 ml, disolver 0,7 g de monohidrato de ácido libre MES y 1,93 g de sal de sodio MES en 8 ml de agua de grado molecular biología. Después de mezclar bien, verificar el pH y ajustar si es necesario a un pH 6.5 a 6.7. Añadir agua hasta un volumen total de 10 ml. Filtro de esterilizar y almacenar a 4 ° C protegido de la luz (por ejemplo, envolver el tubo de papel de aluminio). Reemplace si la solución se vuelve visiblemente amarilla o después de 6 meses.
Tampón de hibridación 2X:
De 50 ml, mezclar 8,3 ml de 12X MES acciones (de 02/09/14), 17,7 ml de 5 M NaCl, 4,0 ml de EDTA 0,5 M, 0,1 ml de 10% de Tween 20 (vol / vol), y 19,9 ml de filtrado ddH 2 O. Filtro de esterilización.
Lavar A: Mezclar 300 ml de 20X SSPE, 1 ml de Tween 10% (vol / vol), 699 ml ddH 2 O. Filtro de esterilización.
Lave B: Mezcle 150 ml 20X SSPE, 1 ml de Tween 10% (vol / vol), 849 ml ddH 2 O. Filtro de esterilización.
Cebadores de secuenciación de códigos de barras
En primer secuencias UP-etiqueta de los 5 'de cola (en negrita) son secuencias de Illumina adaptador específico incorporado en el primer F y R. La secuencia variable (cursiva) representa la etiqueta de indexación 5-mer utilizados en la multiplexación / índice de lectura. La cola de la 3 '(subrayado) representa el primer comunes que flanquean el código de barras uptag y es necesario ampliar los códigos de barras de levadura.
En ABAJO-tag imprimación secuencias 5 'es idéntica a la cola 5' de la cola de la UP-etiqueta de cebadores (secuencia específica de iluminación), sin embargo, el 3 'de cola (subrayado) se sustituye con los primers comunes que se utilizan para amplificar la baja etiqueta de código de barras.
Referencias
- Giaever, G. Functional profiling of the Saccharomyces cerevisiae genome. Nature. 418, 387-391 (2002).
- Baba, T. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular systems biology. 2, 2006.0008-2006.0008 (2006).
- Claus, H., Frosch, M., Vogel, U. Identification of a hotspot for transformation of Neisseria meningitidis by shuttle mutagenesis using signature-tagged transposons. Mol Gen Genet. 259, 363-371 (1998).
- Hava, D. L., Camilli, A. Large-scale identification of serotype 4 Streptococcus pneumoniae virulence factors. Molecular microbiology. 45, 1389-1406 (2002).
- Costanzo, M. The Genetic Landscape of a Cell. Science. 327, 425-431 (2010).
- Tong, A. H. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Pan, X. A robust toolkit for functional profiling of the yeast genome. Molecular cell. 16, 487-496 (2004).
- Schuldiner, M. Exploration of the Function and Organization of the Yeast Early Secretory Pathway through an Epistatic Miniarray Profile. Cell. 123, 507-519 (2005).
- Deutschbauer, A. M. Mechanisms of haploinsufficiency revealed by genome-wide profiling in yeast. Genetics. 169, 1915-1925 (2005).
- Giaever, G. Chemogenomic profiling: identifying the functional interactions of small molecules in yeast. Proceedings of the National Academy of Sciences of the United States of America. 101, 793-798 (2004).
- Lum, P. Y. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116, 121-137 (2004).
- Hillenmeyer, M. E. The chemical genomic portrait of yeast: uncovering a phenotype for all genes. Science. 320, 362-365 (2008).
- Oh, J. A universal TagModule collection for parallel genetic analysis of microorganisms. Nucleic acids research. 38, e146-e146 (2010).
- Oh, J. Gene annotation and drug target discovery in Candida albicans with a tagged transposon mutant collection. PLoS pathogens. 6, (2010).
- Nislow, C., Giaever, G., Stark, I., Stansfields, M. J. R. Chapter 387. Yeast Gene Analysis. , 387-414 (2007).
- Pierce, S. E., Davis, R. W., Nislow, C., Giaever, G. Genome-wide analysis of barcoded Saccharomyces cerevisiae gene-deletion mutants in pooled cultures. Nature protocols. 2, 2958-2974 (2007).
- Sambrook, J., Russell, D. W. . Molecular cloning : a laboratory manual. , (2001).
- Smith, A. M. Quantitative phenotyping via deep barcode sequencing. Genome Res. , (2009).
- Hamady, M., Walker, J. J., Harris, J. K., Gold, N. J., Knight, R. Error-correcting barcoded primers for pyrosequencing hundreds of samples in multiplex. Nature. 5, 235-237 (2008).
- Gresham, D. System-Level Analysis of Genes and Functions Affecting Survival During Nutrient Starvation in Saccharomyces cerevisiae. Genetics. 187, 299-317 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados