Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Cribado de alto rendimiento de la actividad endoglucanasa por hongos en Escherichia coli
En este artículo
Resumen
Se describe un método de bajo costo, de alto rendimiento para la detección de hongos en la actividad endoglucanasa E. coli. El método se basa en una lectura simple y visual de la degradación del sustrato, no requiere purificación de la enzima, y es altamente escalable. Esto permite la detección rápida de las grandes bibliotecas de variantes de la enzima.
Resumen
Celulasa enzimas (endoglucanasas, celobiohidrolasas, y β-glucosidasas) hidrolizar la celulosa en azúcares componentes, que a su vez se puede convertir en una alcoholes carburantes. El potencial para la hidrólisis enzimática de biomasa celulósica para suministrar energía renovable ha intensificado sus esfuerzos para diseñar celulasas para 2 económica de producción de combustible. De particular interés son las celulasas fúngicas 3.8, que ya están siendo utilizados en la industria de los alimentos y el procesamiento de textiles.
La identificación de variantes de activos entre una biblioteca de celulasas mutante es fundamental para el proceso de ingeniería; mutantes activo puede realizar más pruebas para mejorar las propiedades y / o sometidas a mutagénesis adicionales. Ingeniería eficiente de las celulasas fúngicas se ha visto obstaculizada por la falta de herramientas genéticas para los organismos nativos y por las dificultades en la expresión de las enzimas en los ejércitos heteróloga. Recientemente, Morikawa y colaboradores desarrollaron un método para expresar en E. coli los dominios catalíticos de endoglucanasas de H. jecorina 3,9, un hongo industrial importante con la capacidad de secretar celulasas en grandes cantidades. Funcionales E. expresión coli también se ha informado de celulasas de otros hongos, como Macrophomina phaseolina 10 y Phanerochaete chrysosporium 11-12.
Se presenta un método de cribado de alto rendimiento de la actividad endoglucanasa hongos en E. coli. (Fig. 1) Este método utiliza el común microbiana colorante rojo Congo (RC) para visualizar la degradación enzimática de la celulosa carboximetil (CMC) por las células que crecen en medio sólido. El ensayo de la actividad requiere de reactivos de bajo costo, mínima manipulación, y da resultados sin ambigüedades como las zonas de degradación ("halos") en el sitio de la colonia. Aunque una medida cuantitativa de la actividad enzimática no se puede determinar por este método, hemos encontrado que el tamaño del halo de correlaciona con la actividad enzimática en el total de células. Además de la caracterización de cada uno de los clones positivos se determine, la aptitud relativa de proteínas.
Tradicionales de bacterias de células enteras CMC / CR 13 ensayos de actividad implica verter CMC agar que contiene a las colonias, que está sujeta a la contaminación cruzada, o culturas incubando en los pozos de agar CMC, que es menos susceptible a la experimentación a gran escala. Aquí se presenta un protocolo mejorado, que modifica los actuales métodos de lavado de 14 para la actividad de celulasa: las células cultivadas en placas de agar CMC se retiran antes de la tinción CR. El protocolo reduce de forma significativa la contaminación cruzada y es altamente escalable, que permite la detección rápida de miles de clones. Además de la H. jecorina enzimas, que han expresado y se seleccionan variantes endoglucanasa de la aurantiacus Thermoascus y Penicillium decumbens (como se muestra en la Figura 2), lo que sugiere que este protocolo se aplica a las enzimas a partir de una amplia gama de organismos.
Protocolo
1. Detección preparación placa
- Agregar 25 g de crecimiento medio LB, agar 15 g, y 1,5 g de carboximetilcelulosa (CMC) del sustrato de 1 litro de agua destilada y autoclave para esterilizar.
- Después del autoclave, deje a medio enfriar y añadir los antibióticos adecuados, y IPTG a una concentración final de 100μM.
- Vierta 200 ml de medio cada uno en cinco grandes platos de Petri cuadrados / bandejas de bioensayo (240 x 240 x 20 mm o más). Permita que las placas se seque por completo.
2. Endoglucanasa generación de biblioteca y de la pantalla
- Generar una biblioteca de dominio endoglucanasa catalítico (s). Esto se puede lograr de muchas maneras (por ejemplo, dirigida a un sitio o mutagénesis aleatoria, la recombinación, etc) y se determina por el investigador.
- Clon de la biblioteca en un vector de tal manera que están bajo el control del promotor T7 con el operador lac, y contienen una secuencia señal pelB para orientar a la periplasma. Un ejemplo de un vector adecuado es pET22b (+) de Novagen.
- Transformar la biblioteca en endoglucanasa E. coli cepa BL21 (DE3) y la placa de colonias aisladas.
- Incubar transformación durante la noche a 37 ° C.
- Con mondadientes estéril, inocular los clones en placas de 96 pozos profundos, así que contenían medio LB 400μL complementado con los antibióticos adecuados. Se incuba a 37 ° C durante la noche con agitación a 250 rpm.
- Añadir glicerol estéril a 96 culturas y noche a la mañana a una concentración final del 10% para el almacenamiento a largo plazo a -80 ° C. Este será el plato principal.
- El uso de un sello de 96 pines, se replican cultivos en placa durante la noche sobre las placas de detección que contiene IPTG y CMC. Hasta 5 grupos de cultivos durante la noche puede ser estampado en cada plato de detección.
- Etiqueta de las placas de selección de modo que la identidad y la orientación de cada placa de 96 pozos que se conoce.
- Incubar las bibliotecas sellada a temperatura ambiente (17-25 ° C) durante la noche o hasta que las colonias visibles se han formado.
- Enjuagar la placa con agua destilada hasta que todos los restos de las colonias se eliminan.
- Vierta Congo Roja (CR, 0,5% en agua) en la placa de lavado. Asegúrese de añadir sólo lo suficiente CR para cubrir la placa.
- Incubar 15 minutos a temperatura ambiente. No exceder los 15 minutos de incubación CR. Ya los tiempos de incubación dará lugar a halos menos clara.
- Vierta CR y añadir una cantidad abundante (más que suficiente para cubrir la placa) de NaCl 1M.
- Incubar 15 minutos a temperatura ambiente.
- Vierta NaCl para revelar halos CMC degradación. Para una mejor resolución de los halos, ya los tiempos de incubación se lava con NaCl NaCl o múltiple puede ser requerida.
- Con un palillo estéril, elegir clones enzimas activas de la placa principal para una mayor caracterización.
3. Los resultados representativos:
Un ejemplo de la lectura de esta pantalla de alto rendimiento se muestra en la Figura 3. Los clones pueden ser identificados como inactivos, poco activo, y muy activa en función del tamaño de las zonas de degradación. Esto es especialmente útil si una enzima de actividad conocida se incluye en la biblioteca de referencia. Como se muestra aquí, la identificación de enzimas activas es robusta y reproducible. Sin embargo, tenga en cuenta que el etiquetado adecuado de las placas de detección es muy importante. El investigador debe ser capaz de identificar las variantes activas después de lavar las colonias de distancia, con el fin de elegir el plato principal se almacenan a -80 ° C para una mayor caracterización. Por último, es difícil evaluar a priori la relación entre las actividades en substratos artificiales vs natural. Por lo tanto, el investigador debe poner a prueba todos los clones positivos en el material relacionado con la industria para determinar la aptitud de proteínas.
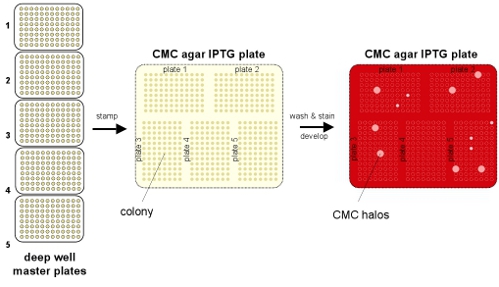
Figura 1. Esquema de los del procedimiento de selección. Las células se transforman con una biblioteca endoglucanasa y chapada selectiva de colonias aisladas. Clones recogidos y crecido durante la noche en placas de 96 pozos profundos y para (1) de almacenamiento a -80 ° C y (2) placas réplica en placas con CMC y IPTG para inducir la expresión endoglucanasa. Clones activos son recogidos de la placa principal se almacenan a -80 ° C.

Figura 2. Clonación y expresión de hongos dominios catalíticos en E. coli. (A) endoglucanasa vector de expresión. Los genes fueron clonados en pET22b (+) (Novagen) bajo el control del promotor T7. (B) del panel derecho: endoglucanasa expresan BL21 (DE3) colonias. Panel de la izquierda: CMC halos de degradación después del lavado y el desarrollo con Rojo Congo.
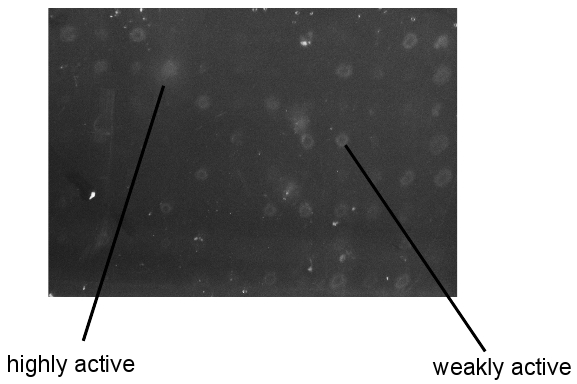
Figura 3. Resultados de las muestras de CMC / Congo pantalla roja. Las imágenes tomadas de placas de detección después del lavado y el desarrollo de Rojo Congo. Todas las colonias en screening placas eran de un tamaño similar.
Access restricted. Please log in or start a trial to view this content.
Discusión
El protocolo aquí descrito permite la identificación rápida y el rendimiento elevado de enzimas activas, con una manipulación mínima. La detección de actividad es bastante sensible, y cualitativamente refleja la cantidad de actividad dentro de la célula. Su facilidad de uso hace que este método es adecuado para una amplia gama de bibliotecas de la enzima, sólo limitado por la expresión funcional en E. coli. Además, la detección de actividad de este tipo no se limita a las celulasas fúngicas, pero p...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue financiado por la Gordon and Betty Moore Foundation, y por la Iniciativa de UNCF / Merck Ciencia.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Reactivo | Empresa | Número del producto | |
|---|---|---|---|
| IPTG | Sigma | I1284 | |
| Rojo Congo | Sigma | C6277 | |
| Carboximetil celulosa | Sigma | 360384 | |
| NaCl | Sigma | S1679 | |
| Bacto-agar | BD Biosciences | 214030 | |
| Placas de bioensayo | Thermo Scientific | 240845 |
Referencias
- Atsumi, S., Hanai, T., Liao, J. C. Non-fermentative pathways for synthesis of branched-chain higher alcohols as biofuels. Nature. 451, 86-89 (2008).
- Wilson, D. B. Cellulases and biofuels. Curr Opin Biotechnol. 20, 295-299 (2009).
- Qin, Y. Engineering endoglucanase II from Trichoderma reesei to improve the catalytic efficiency at a higher pH optimum. J Biotechnol. 135, 190-195 (2008).
- Nakazawa, H. Directed evolution of endoglucanase III (Cel12A) from Trichoderma reesei. Appl Microbiol Biotechnol. 83, 649-657 (2009).
- Heinzelman, P. A family of thermostable fungal cellulases created by structure-guided recombination. Proc Natl Acad Sci. 106, 5610-5615 (2009).
- Heinzelman, P. SCHEMA recombination of a fungal cellulase uncovers a single mutation that contributes markedly to stability. J Biol Chem. 284, 26229-26233 (2009).
- Lantz, S. E. Hypocrea jecorina CEL6A protein engineering. Biotechnol Biofuels. 8, 3-20 (2010).
- Mahadevan, S. A. Site-directed mutagenesis and CBM engineering of Cel5A (Thermotoga maritima). FEMS Microbiol Lett. 287, 205-211 (2008).
- Nakazawa, H. Characterization of the catalytic domains of Trichoderma reesei endoglucanase I, II, and III, expressed in Escherichia coli. Appl Microbiol Biotechnol. 81, 681-689 (2008).
- Wang, H., Jones, R. W. Properties of the Macrophomina phaseolina endoglucanase (EGL 1) gene product in bacterial and yeast expression systems. Appl Biochem Biotechnol. 81, 153-160 (1999).
- Howard, R. L. Enzyme activity of a Phanerochaete chrysosporium cellobiohydrolase (CBHI.1) expressed as a heterologous protein from Escherichia coli. Afr J Biotechnol. 2, 296-300 (2003).
- Howard, Characterisation of a chimeric Phanerochaete chrysosporium cellobiohydrolase expressed from Escherichia coli. Afr J Biotechnol. 3, 349-352 (2004).
- Teather, R. M., Wood, P. J. Use of Congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen. Appl Env Microbiol. 43, 777-780 (1982).
- Gilkes, N. R. Mode of action and substrate specificities of cellulases from cloned bacterial genes. J Bio Chem. 259, 10455-10459 (1984).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados