Method Article
Imágenes de células vivas de Bacillus subtilis Y Streptococcus pneumoniae Automatizada utilizando Time-lapse microscopía
En este artículo
Resumen
Este protocolo establece un procedimiento paso a paso para controlar el comportamiento de células individuales de bacterias diferentes en el tiempo el uso automatizado de fluorescencia time-lapse microscopía. Además, proporcionar directrices a analizar las imágenes de microscopía.
Resumen
Durante los últimos años los científicos se convirtió cada vez más conscientes de que los datos medios obtenidos de los experimentos basados en la población microbiana no son representativos de la conducta, el estado o el fenotipo de las células individuales. Debido a esta nueva visión de la serie de estudios célula aumenta continuamente (para una revisión reciente ver 1,2,3). Sin embargo, muchas de las técnicas aplicadas sola célula no permiten seguir la evolución y el comportamiento de una celda específica único en el tiempo (por ejemplo, la citometría de flujo o microscopía estándar).
Aquí, le ofrecemos una descripción detallada de un método de microscopía utilizado en varios estudios recientes 4, 5, 6, 7, lo que permite seguir y registrar (fluorescencia de) las células bacterianas individuales de Bacillus subtilis y Streptococcus pneumoniae a través del crecimiento y la división de muchas generaciones. Las películas resultantes pueden ser utilizados para construir los árboles filogenéticos se remontan al linaje de la historia de una sola célula dentro de una población que se originaron de un ancestro común. Este método de fluorescencia time-lapse microscopía no sólo pueden ser utilizados para investigar el crecimiento, la división y diferenciación de las células individuales, sino también para analizar el efecto de la historia de la célula y la ascendencia en el comportamiento celular específica. Además, time-lapse microscopio es ideal para examinar la dinámica de la expresión génica y localización de la proteína durante el ciclo celular de las bacterias. El método se explica cómo preparar las células bacterianas y la construcción del portaobjetos para permitir la derivación de células individuales en un microcolonias. En resumen, las células individuales son vistos en una superficie semi-sólido que consiste en el medio de cultivo complementado con la agarosa en el que crecen y se dividen en un microscopio de fluorescencia en una cámara de temperatura ambiental controlada. Las imágenes se capturan en intervalos específicos y posteriormente se analizaron mediante el software de código abierto ImageJ.
Protocolo
1. Preparación de B. subtilis culturas
- Inocular las células de -80 ° C en las poblaciones de 10 ml time-lapse microscopía (TLM) medio (62 mM K 2 HPO 4, 44 mm de KH 2 PO 4, 15 mM (NH 4) 2 SO 4, el citrato de sodio 6,5 mM, 0,8 mM MgSO4, 0,02% casaminoácidos, 27,8 mM de glucosa, 0,1 mM de L-triptófano, el pH fue ajustado a 7 con una solución de KOH), complementado con antibióticos, si es necesario.
- Crecer las células durante la noche en un frasco de agitación (30 ° C, 225 rpm).
- A la mañana siguiente, diluir las células 1:10 en pre-calentado medio químicamente definido (MDL) (62 mM K 2 HPO 4, 44 mm de KH 2 PO 4, 15 mM (NH 4) 2 SO 4, 6,5 mM de citrato sódico, 0,8 mM MgSO 4, 2,2 mm de glucosa, 2,1 ácido L-glutámico mm, 6 M de L-triptófano, 7,5 M MnCl2, mezcla de metal 0,15 x (50x preparar acciones mezcla MT (ref 8), que contiene: 0,2 M de MgCl2, 70 mM CaCl2, 5 mM MNCI 2, 0,1 mM ZnCl2 0,2 mM tiamina clorhidrato, 2 mM HCl, 0,5 mM FeCl 3 (añadir último)) sin antibióticos.
- Crecimiento de la B. subtilis células a mediados de la fase exponencial (30 ° C, 225 rpm). Por lo general, esto toma aproximadamente cuatro horas. Es importante destacar que preparar la diapositiva de agarosa una hora antes de llegar a las células a mediados de la fase exponencial (ver sección 2).
- Medir la absorbancia del cultivo a 600 nm (A 600) y diluir las células a un aproximado de un 600 de 0,035 utilizar el MDL. Esta DO se asegura de que las células individuales con una separación adecuada se vio en el portaobjetos para microscopía lapso de tiempo.
2. Preparación de la muestra de microscopio (ver también Figura 2)
Una hora antes de llegar a las células a mediados de crecimiento exponencial, preparar el portaobjetos de la siguiente manera:
- Limpie dos portaobjetos de vidrio (por ejemplo, Knittel de vidrio, 7,6 x 2,6 cm), con un 70% de etanol y agua.
- Tener un marco gen (ABgene; 1,7 x 2,8 cm) y retirar con cuidado una de las láminas de plástico de la estructura genética sin que el desmontaje de la cubierta de plástico en el otro lado de la estructura genética.
- Coloque el marco de genes en el medio de una de las láminas de vidrio por facilitar el primer contacto de un solo lado, seguido por el apego guiada por el marco de genes restantes con la uña. Evitar que las burbujas de aire mientras se coloca el marco de genes a la lámina de vidrio.
- Usar el microondas para disolver 150 mg (1,5%) de alta resolución de bajo punto de fusión de agarosa (Sigma) en 10 ml del MDL. La agarosa debe ser totalmente disuelto para obtener antecedentes mínimos requeridos para los experimentos de microscopía lapso de tiempo. Si es necesario, complementar la agarosa-MDL con el inductor o de otros compuestos en este momento
- Transferencia de 500 l de la tibia de agarosa-MDL en el medio del marco de genes. Asegúrese de que toda la zona, incluyendo (la frontera) está totalmente cubierto.
Los siguientes pasos (2,6 a 2,10) tienen que llevarse a cabo rápidamente para prevenir la sequedad excesiva de la agarosa-MDL. - Coloque la segunda lámina de vidrio en el marco de agarosa gen-CDM llena. Trate de evitar las burbujas de aire. Colocar los portaobjetos intercalado en posición horizontal durante 45 min a 4 ° C en el refrigerador para permitir a la agarosa-MDL para solidificar lo suficiente.
- Con cuidado, deslice la placa de vidrio superior. Use una hoja de afeitar para cortar las tiras de agar de ancho ~ 5 mm en el marco de genes, en el que las células se cultivan. Un máximo de tres tiras se pueden utilizar en cada diapositiva, separados por ~ espacio de 4 mm a cada lado. Estos espacios proporcionan aire que es esencial para la B. subtilis crecimiento. Si hay cuatro diferentes cepas deben ser seguidas en el tiempo, dos tiras se pueden hacer y ser cortado por la mitad para dar lugar a cuatro pequeños cuadrados. Retire cualquier medio sólido residual.
- Retire con cuidado la tapa de plástico la segunda y última de la estructura genética para exponer el lado pegajoso de la estructura genética
- Las celdas de carga única (desde el paso 1.5) en el medio sólido sin tocarlo con la punta de la pipeta. 2,5 l uso de una tira entera, o 1 l de una pequeña plaza. Comience siempre por encima de la almohadilla de agarosa y permitir que el líquido para dispersar por igual en su área de crecimiento asignados girando el deslice hacia arriba y hacia abajo. La diapositiva está listo, tan pronto como los bordes de los líquidos se corrugado y el movimiento del líquido ya no es visible al girar la tapa.
- Coloque una lámina limpia microscopio cubreobjetos (24 x 50 mm) en el marco de genes de un lado a otro (evitar las burbujas de aire). Asegurar la unión completa mediante la aplicación de presión sobre la hoja de la cubierta a lo largo de la estructura genética con la uña. Si la hoja de la cubierta se coloca en las células sin permitir que se sequen lo suficiente, las células tienden a crecer en la parte superior de uno al otro durante el experimento. También tenga cuidado de no esperar demasiado tiempo antes de aplicar la hoja de la cubierta, ya que la agarosa entonces será demasiado seco.
- Pre-caliente el portaobjetos durante 1 hora a 30 ° C. Si la tapa wOuld directamente se coloca en la cámara del medio ambiente precalentado (véase el paso 3.1) del microscopio, las fluctuaciones de temperatura podría causar problemas de enfoque automático en las primeras horas del experimento.
3. Time-lapse microscopía de fluorescencia (ver también la Figura 3 y la película 1)
- Pre-caliente de la cámara del medio ambiente en el tiempo (en nuestras manos por lo menos 2 horas antes del comienzo del experimento) con el fin de evitar problemas de enfoque automático después de comenzar el experimento. El tiempo requerido depende de la cámara ambiental utilizados, así como el sistema de calefacción y el microscopio.
- Seleccionar el objetivo adecuado, filtros y espejos dicroicos de acuerdo a su montaje experimental. Para los experimentos de larga asegurarse de que un filtro UV se coloca entre la fuente de luz y la muestra. Además, si es posible, bloquear parte de la luz de excitación el uso de filtros de densidad neutra para minimizar la exposición.
El siguiente equipo (proporcionado por DeltaVision, Reino Unido) se utilizó para los experimentos de microscopía lapso de tiempo-publicado en de Jong et al 2010 5:. IX71 microscopio (Olympus), CoolSnap HQ2 cámara (Princeton Instruments), 300W Fuente de luz de xenón, 60x brillante objetivo de campo (1,25 NA), las buenas prácticas agrarias filterset (Chroma, nm de excitación a 470/40, 525/50 nm de emisión), mCherry filterset (Chroma, nm de excitación a 572/35, 632/60 nm de emisión). El enfoque automático se ha realizado mediante la luz diascópica y el uso de la actual rutina de enfoque automático de software de DeltaVision Softworx. Cabe señalar que en la actualidad hay una serie de sistemas de enfoque automático de otros que también son adecuadas, tales como el enfoque definido Zeiss, el sistema Nikon de enfoque perfecto y el control de Leica adaptable Focus.
- El programa de su experimento de acuerdo a su puesta en marcha experimental. Es una buena idea para determinar la cantidad de luz necesaria para las construcciones específicas, así como los ajustes de enfoque automático de otro lapso de tiempo microscopios o bacterias antes de que el experimento real. Menor tiempo de exposición y menos cantidad de luz de excitación minimizar la decoloración y la fototoxicidad. Use la luz diascópica para la rutina de enfoque automático.
Los siguientes parámetros fueron utilizados para los experimentos de microscopía lapso de tiempo, publicado en de Jong et al 2010 5:. Instantáneas de películas fueron tomadas a intervalos de 8 ó 12 minutos con un 10% APLLC LED blanco exposición a la luz y la s 0,05 para las imágenes de campo claro, 10% de la luz de xenón y 0,5 s de exposición para la detección de buenas prácticas agrarias, y el 32% de luz de xenón y 0,8 s para la detección de la exposición mCherry, respectivamente. Los datos en bruto se almacenan utilizando softWoRx 3.6.0 (Applied Presicion). El enfoque automático se ha programado el 0,06 micras pasos y una gama total de 1,2 micras.
- Coloque la diapositiva preparada (sección 2) en la cámara ambiental pre-calentado del microscopio y controlar el resultado de las células individuales en una monocapa microcolonia a 30 ° C.
Consejos específicos:
- Seleccione las celdas individuales que se encuentran en el centro de la almohadilla de agar. Los bordes de la plataforma de agar se seque con mayor facilidad. Guarde la posición X, Y, Z con el software del microscopio.
- Los grandes movimientos de la etapa en la X, Y y Z dirección podría perturbar la agarosa y por lo tanto dificultan la identificación de las células por la rutina de enfoque automático. En general, para reducir al mínimo X, Y, Z movimiento, no seleccione más de 10 posiciones por experimentar, incluso si una diapositiva contiene múltiples cepas.
- Después de haber seleccionado la primera celda, sólo ajustar la Z-enfoque con el software. A partir de este momento, no alteran el enfoque manualmente en el cuerpo del microscopio con el "Z-mando" a menos que sea codificada digitalmente. Asegúrese de que después de cada rutina de enfoque automático de la nueva X, Y, Z se guarda la posición por el software.
- Compruebe si los ajustes de enfoque automático son adecuadas para el experimento, antes de comenzar la carrera. El uso de la microscopía de contraste de fase puede mejorar la rutina de enfoque automático en comparación con el uso de campo claro o microscopía DIC, debido a un mayor contraste. Sin embargo, el anillo de fase en los objetivos de contraste de fase hace menos sensible (10%) en la recogida de luz de fluorescencia. Así, por débiles muestras fluorescentes, un objetivo sin anillo de fase es más adecuado.
- Comprobar si las celdas seleccionadas están todavía en el foco cada media hora, hasta que el experimento está funcionando de manera estable. Cuando las células en este momento están fuera de foco, ajuste de forma manual. Debido a las variaciones de temperatura, así como muestras de mal seco, esto puede ser necesario durante las primeras horas. Además, debido al mayor contraste, el enfoque automático funciona mejor cuando hay más células se encuentran en el campo de visión.
- Después de que el experimento ha terminado, por separado los diferentes canales de la película y segura como archivos independientes (es decir, el contraste de fase, las buenas prácticas agrarias, mCherry) si es necesario (algunos paquetes adquisición lugar todos los canales en un solo archivo estéreo). Para su publicación, las fotos pueden ser mejoradas por deconvolución 2D, que es especial para nosotroseful para los estudios de localización de la proteína. Deconvoluir las imágenes utilizando el software de su microscopio o con un paquete comercial, como Huygens ( www.svi.nl ).
- Analizar los datos mediante ImageJ ( http://rsbweb.nih.gov/ij/ ) (utilizar los archivos en bruto, sin procesar la imagen para esto) y el diagrama de Microsoft Excel o Sigma. Pilas puede, por ejemplo, se guardará como un archivo ". Avi" archivo de película en ImageJ. Una descripción detallada de la forma de fluorescencia de las células individuales se pueden medir en el tiempo es la siguiente.
4. Análisis de los datos de la dinámica de la actividad del promotor con ImageJ
Tomamos nota de que otros paquetes de software están disponibles buenas que están especializadas en el análisis de lapso de tiempo imágenes de microscopía, como BHV software 9, 4, 10 Schnitzcell, PSICIC 11, y Microbio Tracker-12, pero aquí nos centramos en el paquete ImageJ libre disposición.
- Descargar ImageJ ( http://rsbweb.nih.gov/ij/ ) y (si es necesario) el plugin correcto para abrir (apilados) de archivos. Por ejemplo, las películas grabadas con un microscopio DeltaVision sólo se puede abrir en ImageJ con el plugin de apertura DeltaVision. Copie el DV-plugin en la carpeta plugins ImageJ e iniciar el programa. Cambiar la capacidad de memoria en editar / opción / la memoria y las discusiones de 1250. Esto permite trabajar con archivos más grandes, como los obtenidos de las películas de time-lapse.
- Para evaluar la historia de células de una sola célula, abrir la película de contraste de fase inicial de un microcolonias y desplácese hasta el último fotograma de interés en la película. Seleccione la opción "líneas segmentadas" botón de selección en el menú.
- Dibuje una línea en el fondo y presione "CTRL" + "T". Esto abrirá las regiones de interés (ROI) gerente. (El valor de fluorescencia de fondo correspondiente se puede utilizar para quitar manualmente el fondo -.. Por otra parte ver a continuación, utilizar el presente sustracción de fondo de rutina dentro de ImageJ) También trazar una línea en la célula de interés y añadir el retorno de la inversión para el administrador de retorno de la inversión. Puesto que estamos investigando las buenas prácticas agrarias fusiones promotor-en este caso de estudio y las buenas prácticas agrarias se difunde por todo el citoplasma, la célula en general debe tener similares valores de fluorescencia para cada píxel a lo largo de la célula. Desplazamiento de una estructura en el tiempo y seleccionar un retorno de la inversión nueva en la misma celda de interés. Guardar este retorno de la inversión tercero y continuar con el procedimiento hasta que el retorno de la inversión correspondiente en el primer fotograma de la película se ha guardado.
Tenga en cuenta que la fluorescencia de las células hijas pueden variar mucho después de la división celular. O bien utilizar un tinte de membrana que se puede aplicar junto con las proteínas de la fluorescencia producida por las células (por ejemplo, la combinación de la membrana tinte rojo FM 5-95 ® (Invitrogen) y GFP) para visualizar la formación del tabique. En este caso el canal de fluorescencia correspondiente y no la película de contraste de fase se debe utilizar para seguir las células en el tiempo. Alternativamente, uno puede quedarse en el lado seguro mediante la selección de retorno de la inversión en sólo la mitad de una célula. - Dentro del administrador de ROI, haga clic en "Guardar". Si la terminación del archivo es ". Roi", y luego un retorno de la inversión se ha seleccionado en la lista, y sólo éste será salvo. Si la terminación del archivo es ". Zip", y luego todo el conjunto será salvo (se requiere).
- Cerca de la película de contraste de fase y abrir la película original de fluorescencia (GFP, por ejemplo). Haga clic en "mostrar todo" y "medida" en el administrador de retorno de la inversión. Una nueva ventana se abrirá (Resultados). Copia de los resultados en una hoja de Excel y restar la media de fluorescencia de cada célula de la fluorescencia de fondo del medio. La fluorescencia neto resultante puede registrarse en el momento de revelar la actividad del promotor de la célula de interés en el tiempo.
- Por otra parte, la fluorescencia de las células de un microcolonia puede ser analizado en un determinado punto del tiempo. Con el fin de hacer esto seleccionar y guardar retorno de la inversión para el fondo y cada célula en un marco como se describe arriba, copie los valores de fluorescencia para Excel y producir histogramas usando el "histograma" en función de la "herramienta" del menú.
- Para obtener imágenes de cuadros individuales para su publicación, seleccione el marco de interés, seleccione "Imagen" - "duplicar la imagen" y cambiar el tipo de imagen a "RGB" o "de 8 bits" mediante la selección de "imagen" - "tipo" - "RGB color" o "8-bit". Guarda el cuadro duplicado como ". Tiff". Las imágenes RGB/8-bit pueden ser abiertos por programas de dibujo convencionales, tales como Corel Draw o Adobe Illustrator. Si es necesario, las imágenes se puede adaptar el uso de "imagen" - "ajuste" - "brillo / contraste" o "imagen" - "ajuste" - "de la ventana / nivel" en ImageJ.
5. La producción de películas para su publicación con ImageJ
- Abra el contraste de fase original y la película de fluorescencia correspondiente (s) en ImageJ como se describió anteriormente. Seleccione el botón de selección rectangular (1 º a la izquierda) y dibujar un rectángulo de retorno de la inversión en el primer fotograma de tal manera, que el desarrollo de microcolonias está rodeado por el retorno de la inversión throughout toda la película. Seleccione "Imagen" - "cultivo" y seguro esta versión más pequeña de la película con un nuevo nombre. Seleccione el mismo ROI a través del gestor de retorno de la inversión en la película de fluorescencia y proceder como antes.
- Para combinar las películas ya sea horizontal o verticalmente, seleccione "plugin" - "combiner pila". Si lo desea, un estampador tiempo se puede agregar a través de "plug-in" - "estampador tiempo". Guarde la pila combinado una vez como ". Dv" o ". Tiff" y otra como ". Avi" o una película QuickTime.
Adaptaciones alternativa Protocolo de Streptococcus pneumoniae (Figura 4 y la película 2):
6. Preparación de S. pneumoniae culturas
- Crecer S. pneumoniae células (encapsulado cepa D39 13, o cepa no encapsulada R6 14 en cultivos de pie en medio C + Y 15 a 37 ° C hasta una DO a 600 nm A de aproximadamente 0,4 se alcanza. Centrifugar las células durante 2 min a 14000 rpm y resuspender el pellet de células en un volumen de agua dulce C + Y medio que contiene 14,5% de glicerol (v / v), que se traduciría en un 600 nm Una de exactamente 0,4. alícuotas de las células y almacenarlos a -80 ° C para su uso futuro.
- Para microscopía lapso de tiempo, tomar una alícuota del previamente cultivadas S. células pneumoniae. Inocular 4 ml de agua dulce C + medianas Y 1:100 con las células de los -80 ° C alícuota. Crecer las células hasta mediados de la fase exponencial de un diámetro exterior de A 600nm de 0,1 a 0,2. Generalmente, esto toma alrededor de 2 horas cuando se utilizan células de -80 ° C alícuotas.
7. Preparación de la muestra microscopio
- Preparar un portaobjetos como se describe anteriormente para B. subtilis, pero asegúrese de que la agarosa contiene complejos C + Y medio. Desde el S. pneumoniae es una microaerófilo, las bolsas de aire entre las tiras de agarosa debe ser menor que la de B. subtilis (~ 1 mm de espacio en ambos lados).
- Medir la absorbancia del cultivo a 600 nm (A 600), diluir el crecimiento exponencial de S. pneumoniae a las células de un aproximado de 600 A de 0,05 con C + Y y medio de uso de esta dilución para cargar la diapositiva de agarosa.
8. Time-lapse microscopía de contraste de fase
Ajustar la configuración del microscopio en busca de S. pneumoniae: el uso de contraste de fase microscopio ya S. pneumoniae es difícil identificar con el microscopio de campo brillante. Continuar con el protocolo descrito para B. subtilis (siga los pasos 2,9 hasta 3,7). S. pneumoniae células pueden ser cultivadas en cualquiera de los 30 ° C o 37 ° C (que crecen más rápido a 37 ° C).
9. Los resultados representativos:
El experimento de fluorescencia de lapso de tiempo se ha llevado a cabo con éxito, si la bacteria se convirtió en una monocapa de microcolonias, que está completamente situado en el campo de visión al final del experimento (ver Figura 5A-C). Si las células se crecieron en la parte superior de uno al otro, no sólo es imposible rastrear su historia con precisión, sino también los niveles de fluorescencia de las células de la superposición no puede ser medido correctamente. Las células tienden a crecer en la parte superior de uno al otro, si las células no se vio suficientemente seco (paso 2.9) o si la composición del medio tiene que ser ajustada para obtener un crecimiento más lento. Si un microcolonia creció fuera de la vista, entonces la distribución de las señales de fluorescencia en una colonia no puede ser determinada. Las causas de "movimiento microcolonia" puede ser secado insuficiente de células manchado (paso 2.9), o si el software no estaba programado para realizar el seguimiento del microcolonia durante el desarrollo. Además, es importante que los parches locales mayor intensidad de fluorescencia no son detectables en el medio, ya que oculta las señales de fluorescencia procedentes de las células (véase la Figura 5D-F). Los problemas de fondo relacionados pueden surgir a partir de compuestos medios de comunicación, las burbujas de aire o disolver grupos de agarosa. Para visualizar esto, se muestra en la figura. 5F señales de fondo de esta diapositiva específica cuando la imagen fue tomada usando excitación / emisión de filtros de color rojo tintes fluorescentes. Como se ve, brillantes manchas autofluorescentes están presentes lo que perjudicaría la imagen. Para evitar tales lugares, asegúrese de que la agarosa se disuelva por completo y no hay burbujas de aire cuando se coloca el cubreobjetos sobre el portaobjetos del microscopio.
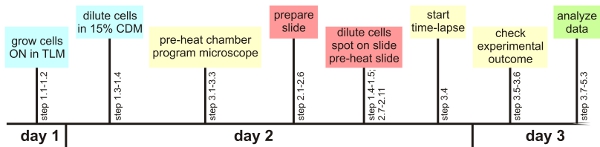
Figura 1: Experimental

Figura 2: Preparación de la muestra microscopio
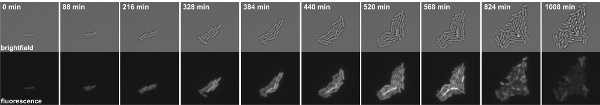
Figura 3: Time-lapse microscopía de fluorescencia de la B. subtilis células que alberga una kinB P-GFP fusión. Las instantáneas son tomadas de la película 1. Paneles superiores: campo claro, paneles de fondo: canal de las buenas prácticas agrarias.
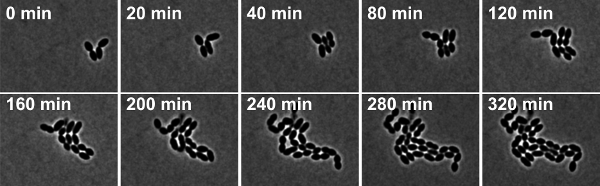
HigoUre 4: Time-lapse microscopía de contraste de fase de S. pneumoniae cepa de tipo salvaje R6. Las instantáneas son tomadas de la película 2.
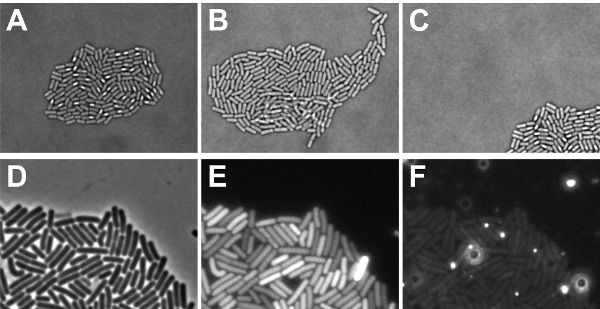
Figura 5: Ilustración de los posibles resultados de la microscopía (time-lapse). AC muestra los factores que deben tenerse en cuenta los datos obtenidos con diascópica configuración de la luz. (A) campo claro micrografía de una monocapa microcolonias (resultado positivo) de la esporulación B. células subtilis (B) Campo claro la imagen de una B. subtilis microcolonias en el que algunas células crecieron en la parte superior de cada uno (resultado negativo) (C) la imagen de un campo claro B. esporulación subtilis microcolonias que surgió del campo de concentración (resultado negativo). DF muestran que los factores deben tenerse en cuenta los datos obtenidos con episcópica configuración de la luz (D) Fase de contraste de la imagen de B. subtilis células en fase exponencial se muestra a visualizar en las señales de fluorescencia en E y F se originan a partir de (E) Las señales de las buenas prácticas agrarias de las células se muestra en la Nota D. que las señales de fondo son similares en cada pixel (resultado positivo). También tenga en cuenta que el tiempo de exposición puede ser demasiado desde un celular muestra una señal saturada (resultado negativo) (F) Las señales obtenidas por el canal rojo de las células se muestra en la Nota D. que el fondo contiene las áreas con mayores niveles de fluorescencia roja (negativos resultado).
Movie 1. Time-lapse microscopía de fluorescencia de la B. subtilis células que alberga una kinB P-GFP fusión. Las instantáneas fueron tomadas en intervalos de 8 min. Izquierda: campo claro, la derecha: GFP canal. Haga clic aquí para ver la película.
Movie 2. Time-lapse microscopía de contraste de fase de S. pneumoniae cepa de tipo salvaje R6. Las instantáneas fueron tomadas en intervalos de 10 minutos. Haga clic aquí para ver la película.
Discusión
En contraste con muchas otras técnicas de célula única, el método de time-lapse microscopía de fluorescencia se describe aquí se puede utilizar para seguir la historia de una celda específica en relación con sus antepasados, su comportamiento, y eventos de la división. En combinación con los promotores objetivo marcado con fluorescencia o las proteínas, la activación específica vía de desarrollo puede ser seguido en el tiempo y localización de la proteína, así como la dinámica de proteínas pueden ser monitoreados durante el desarrollo de bacterias.
Como se indicó anteriormente, los estudios se concentra en diferentes especies bacterianas se pueden realizar mediante la adaptación de las condiciones de crecimiento de acuerdo a los requerimientos de una bacteria específica. Las únicas limitaciones que encontramos están relacionados con las condiciones de crecimiento y tamaño de la muestra. Debido a un entorno cerrado, las condiciones del medio no se puede cambiar durante el experimento. Además, un máximo de cuatro cepas por experimento se puede controlar de manera eficiente.
Teniendo en cuenta algunas medidas críticas, el método de análisis de células individuales se describe aquí se puede aplicar fácilmente utilizando cualquier microscopio automatizado. En el siguiente, un resumen de los pasos críticos se les dará. La información detallada se puede encontrar en el texto principal de preparación general:. Es una buena idea para comprobar los ajustes de enfoque automático necesarios para una bacteria específica antes del experimento. Asimismo, se aproximan a los valores óptimos para la visualización de la fluorescencia debe ser determinada de antemano, si es posible. Además, después de un preparado de línea de tiempo ayuda a tener todo el material listo para su uso en el tiempo (pre-calentamiento de la cámara del microscopio, la programación de la configuración de microscopio, la preparación de la diapositiva una hora antes de que las células se encuentran en la fase de crecimiento deseado, ver Figura 1) . Crecimiento de la B. subtilis en TLM y el MDL: TLM y el MDL son químicamente definido los medios de comunicación en la que el hambre B. subtilis sólo crece lentamente. El período de tiempo en el que las células se cultivan en los medios de comunicación podría tener que prolongarse dependiendo de la cepa específica. El lento crecimiento impide que las células acumulan unos sobre otros Preparación de la muestra microscopio:. Burbujas de aire entre la estructura del gen, la lámina de vidrio y la hoja de la cubierta tiene que ser prevenida para evitar la sequedad amplia del medio de agarosa-based. Lo mismo vale para la interfaz de deslizamiento medio / cover. Es crucial que las células lo suficientemente seco para evitar la natación y / o el crecimiento de capas múltiples Time-lapse microscopía de fluorescencia:. Pre-calentamiento de la lámina, así como la cámara del medio ambiente es crucial para evitar mayores problemas de enfoque automático. Las células deben ser seleccionados en el medio de una almohadilla de agar, ya que estos tienen la mayor probabilidad de permanecer en el campo y centrarse en el experimento (siempre que la muestra se secó lo suficiente). Un máximo de 10 puntos por los experimentos todavía funciona correctamente. Después de haber seleccionado la primera celda de interés sólo utilizar el software para ajustar el enfoque (véase el texto para más detalles). Comprobar si las células están todavía en el foco durante las primeras horas del experimento, en intervalos de 30 minutos. Análisis: Es importante comprobar antes de los procedimientos de análisis más amplio si el fondo del medio tiene valores similares en los canales de fluorescencia. Pequeñas partículas de polvo, los componentes del medio, las lentes sucios o pequeños grupos de agarosa pueden contribuir a la fluorescencia a nivel local aumentó, lo que hace la película de difícil o imposible de analizar Resolución de problemas:. Si las células crecen en la parte superior de la otra, esta bien podría indicar que el cubreobjetos se adjunta demasiado pronto o que el medio no es adecuado para el crecimiento de las monocapas microcolonias. Si las células de interés continuamente mueren de forma prematura, mientras que otras células en el portaobjetos se dividen felizmente, es posible que desee comprobar si se pone el filtro UV en la posición. También podría ayudar a disminuir el tiempo de exposición o la intensidad de la luz durante los experimentos de largo.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
El trabajo en el grupo de JWV con el apoyo de una beca de la UE Marie-Curie Reintegración, una beca Sysmo2 (NWO-ALW/ERASysBio), un horizonte de subvención (ZonMW) y por una beca VENI (NWO-ALW). El grupo de OPK con el apoyo de varias subvenciones STW (NWO), un SYSMO1 (IGdeJ) y SYSMO2 subvención, las FSE Eurocores biología sintética, la subvención (SynMod) y por el Centro de Genómica Kluyver de Fermentaciones Industriales y el Instituto superior de Alimentación y Nutrición.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios (opcional) |
|---|---|---|---|
| Gene marco | ABgene | AB-0578 | 1,7 x 2,8 cm |
| de alta resolución de baja fusión de agarosa | Sigma | A4718 | |
| cubreobjetos grande | varios | 24 x 50 mm | |
| Si lo desea, la membrana de colorante, por ejemplo, FM 5-95 | Invitrogen | T23360 | otros tintes membrana también están disponibles: http://probes.invitrogen.com/media/pis/mp34653.pdf |
| Time-lapse microscopio con cámara ambiental | varios | ver los detalles de nuestro dispositivo en las secciones correspondientes |
Referencias
- Veening, J. W., Smits, W. K., Kuipers, O. P. Bistability, epigenetics, and bet-hedging in bacteria. Annu. Rev Microbiol. 62, 193-193 (2008).
- Dubnau, D., Losick, R. Bistability in bacteria. Mol Microbiol. 61, 564-564 (2006).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nat. Rev. Microbiol. 7 (5), 383-383 (2009).
- Veening, J. W. Bet-hedging and epigenetic inheritance in bacterial cell development. Proc. Natl. Acad. Sci. U. S. A. 105 (11), 4393-4393 (2008).
- de Jong, I. G., Veening, J. W., Kuipers, O. P. Heterochronic phosphorelay gene expression as a source of heterogeneity in Bacillus subtilis spore formation. J. Bacteriol. 192 (8), 2053-2053 (2010).
- Veening, J. W., Murray, H., Errington, J. A mechanism for cell cycle regulation of sporulation initiation in Bacillus subtilis. Genes Dev. 23 (16), 1959-1959 (2009).
- Eberhardt, A. Cellular localization of choline-utilization proteins in Streptococcus pneumoniae using novel fluorescent reporter systems. Mol Microbiol. 74 (2), 395-395 (2009).
- Vasantha, N., Freese, E. Enzyme changes during Bacillus subtilis sporulation caused by deprivation of guanine nucleotides. J Bacteriol. 144 (3), 1119-1119 (1980).
- Stewart, E. J. Aging and death in an organism that reproduces by morphologically symmetric division. PLoS. Biol. 3, 45-45 (2005).
- Rosenfeld, N. Gene regulation at the single-cell level. Science. 307 (5717), 1962-1962 (2005).
- Guberman, J. M. PSICIC: noise and asymmetry in bacterial division revealed by computational image analysis at sub-pixel resolution. PLoS. Comput. Biol. 4 (11), 1000233-1000233 (2008).
- Montero, L. lopis P. Spatial organization of the flow of genetic information in bacteria. Nature. 466, 77-77 (2010).
- Avery, O. T., Macleod, C. M., McCarty, M. Studies on the chemical nature of the substance inducing transformation of pneumococcal types: Induction of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III. J Exp. Med. 79, 137-137 (1944).
- Hoskins, Genome of the bacterium Streptococcus pneumoniae strain R6. J Bacteriol. 183, 5709-5709 (2001).
- Martin, B. The recA gene of Streptococcus pneumoniae is part of a competence-induced operon and controls lysogenic induction. Mol Microbiol. 15, 367-367 (1995).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados