Method Article
Genoma toda la pantalla de los genes miARN objetivos mediante el objetivo de la misión Carnet de identificación
* Estos autores han contribuido por igual
En este artículo
Resumen
La Biblioteca de ID de destino es un plásmido basado en el genoma colección de cDNA clonado utiliza para identificar los genes miARN objetivos. Aquí nos demuestran su uso y aplicación.
Resumen
La Biblioteca de ID de destino está diseñado para ayudar en los objetivos de descubrimiento e identificación de los microARN (miRNA). La Biblioteca de ID de destino es un plásmido basado en el genoma biblioteca de cDNA clonado en el 3'UTR aguas abajo de la proteína de fusión de dos de selección, la timidina quinasa zeocin (TKzeo). La primera ronda de selección es para transformantes estables, seguido con la introducción de un miARN de interés, y finalmente, la selección de ADNc que contienen el objetivo de miARN. ADNc seleccionados se identificaron mediante la secuenciación (véase la Figura 1-3 para flujo de trabajo de la Biblioteca de ID y los detalles).
Para asegurar una cobertura amplia del transcriptoma humano, Target ID Biblioteca cDNA se generaron a través de oligo-dT cebado con un grupo de ARN total preparado a partir de múltiples tejidos humanos y líneas celulares. Resultando gama de ADNc de 0,5 a 4 kb, con un tamaño medio de 1,2 kb, y se clonaron en el p3TKzeo doble selección plásmido (véase la Figura 4 Mapa de plásmido). Los objetivos de genes representados en la liblioteca se puede encontrar en la página web de Sigma-Aldrich. Los resultados de la secuenciación de Illumina (Tabla 3), muestran que la biblioteca cuenta con 16.922 de los 21,518 genes únicos en la UCSC RefGene (79%), o 14.000 genes con 10 o más lecturas (66%).
Protocolo
1. Transfección con la Biblioteca de ID y selección de líneas celulares estables
1. Zeocin Kill Curva
Zeocin se utiliza para seleccionar células transfectadas establemente. Sin embargo, zeocin exceso provoca respuestas indeseadas fenotípicos en la mayoría de tipos de células. Por lo tanto, un análisis de la curva de interrupción debe realizarse para establecer la dosis letal mínima.
- Placa 1,6 x 10 4 células en pocillos de una placa de 96 pocillos en 120 l de medios de comunicación.
- Al día siguiente añadir zeocin en el aumento de las concentraciones que van desde 50 mg / ml a 1 mg / ml a los pocillos apropiados.
- Examinar la viabilidad de cada 2 días.
- Vuelva a colocar los medios de comunicación que contienen zeocin cada 3 días. La concentración mínima de reactivo selección que causa la muerte celular completa después de la hora deseada debe utilizarse para ese tipo de células y el experimento. Nuestros resultados muestran que 500 mg / ml zeocin es óptima para A549, HeLa, y células MCF7.
2.Biblioteca de la transfección y selección a través de nucleofection
- Seleccione una línea de células que, o bien no se expresa o manifiesta bajos niveles de miRNA de su interés. El miRNA se presentó en la sección B para la selección de destino después de la expresión estable de la Biblioteca de ID de objetivo se logra.
- Cultura / expandir las células. Hemos obtenido excelentes resultados con 2 x 10 7 células por transfección de la biblioteca.
- Trypsinize células que están en> 80% de confluencia, y transferir 2 x 10 7 células a unos tubos de tornillo 15 ml rematados estériles.
- Pellet tripsinizaron las células a 200 xg durante 5 min.
- Quitar medio y lavar sedimento celular con HBSS o PBS 1X.
- Centrifugar a 200 xg durante 5 min y aspirar lavado.
- Repetir el paso de lavado de sedimento celular.
- Pre-caliente placas de 6 pocillos con 2 ml de medio completo a 37 º C.
- Añadir 2 g de destino Carnet de identificación (que no exceda de 10 l) por tubo de 0,5 ml para cada transfección.
- Resuspender las células en el tubo de 15 ml de arriba con 100 l de solución nucleofection Amaxa (celda específica) por 2 x 10 6 células. Por ejemplo, durante 10 Nucleofections añadir 1 ml de reactivo.
- Una reacción a la vez, añadir 100 ml de las células a la g 2 del plásmido. Mezclar con la pipeta.
- Transfiera la mezcla a una cubeta nucleofector.
- Introducir la cubeta en el instrumento nucleofector y ejecutar el programa optimizado adecuado para la línea celular (de alta eficiencia preferido sobre la viabilidad celular).
- Llene la pipeta de transferencia con pre-calentado medio. Recoger las células en misma pipeta y la transferencia a 6-así placa. Repita para cada nucleofection, uno por cada pozo.
- Volver a la cámara de crecimiento para la incubación durante la noche.
- Al día siguiente, reemplazar medio y permitir que las células de recuperar durante 3-5 días.
- Sustituir medio con medio completo que contenía el nivel apropiado de zeocin, como se determina a partir de la curva de análisis de interrupción.
- Mocélulas progenitoras de la selección para zeocin (las células que mueren).
- Vuelva a colocar medio con Zeocin cada 2-3 días.
- Una vez que confluyen en la placa de 6 pocillos, pasaje, piscina y ampliar las células en grandes frascos.
- Periodo de tiempo para la expansión de las células es el usuario y la línea celular dependiente, pero es muy recomendable para ampliar Zeocin células resistentes a la generación de crio-acciones para la detección futuro (aproximadamente 2-3 semanas, la línea celular dependiente).
2. Las células transfección de la biblioteca con la construcción-miARN expresión, seleccione la línea celular estable, y seleccionar objetivos miARN
Nota: La selección Zeocin ya no es necesario o deseado. La exposición de células a Zeocin en miARN expresión y la selección de ganciclovir (objetivo) puede resultar en la pérdida de los genes miARN objetivos.
3. Puromicina, G418 y ganciclovir Curvas Kill
- Realizar una curva de interrupción de ganciclovir con las células que expresan establemente la Biblioteca de ID de destino y con puromicina o G418 para el tipo salvajecélulas.
- Placa 1,6 x 10 4 células en pocillos de una placa de 96 pocillos con 120 l medio fresco. Al día siguiente añadir 0,1 a 10 mg / ml de puromycin/G418, o 2 a 32 mM ganciclovir a los pocillos seleccionados.
- Examinar la viabilidad de cada 2 días. Vuelva a colocar los medios de comunicación que contengan reactivo de selección de cada 3 días. La concentración mínima de reactivo selección que causa la muerte celular * completa después el tiempo deseado, se debe utilizar para ese tipo de células y el experimento. Nuestros resultados muestran que 0,25 a 1 mg / ml puromicina es óptima para A549, HeLa, y células MCF7, 0,3 microgramos / ml de G418 para células MCF7 y 8-16 mM ganciclovir son óptimas para A549, HeLa y células MCF7.
* Nota: Con la selección de ganciclovir, el crecimiento celular lento o no pueden ser observados sin muerte de la célula completa. Observar las células en el tratamiento de varios días para verificar que no se están dividiendo activamente. Si este es el caso, entonces rojo fenol en el medio permanece rojo. También puede ser necesario realizar un kianálisis de la curva II de la biblioteca de ID de destino células transfectadas si un cambio en la susceptibilidad se observa. Sin embargo, esto tendrá que ser determinado sobre una base tipo específico de célula.
4. miARN Transfección y selección a través de nucleofection Meta
- Crecer / expandir las células diana Carnet de identificación para obtener 2 x 10 7 células, y trypsinize cuando las células están en> 80% de confluencia.
- Transferencia de 2 x 10 7 células a un tornillo de 15 ml estéril rematado tubo y el pellet a 200 xg durante 5 minutos.
- Quitar medio y lavar sedimento celular con HBSS o PBS 1X.
- Centrifugar a 200 xg durante 5 min y aspirar lavado.
- Repetir el paso de lavado de sedimento celular.
- Pre-caliente placas de 6 pocillos con 2 ml de medio completo por pocillo a 37 º C.
- Añadir 2 g de miARN expresión plásmido (que no exceda de 10 l) por tubo de 0,5 ml para cada transfección. Origene plásmidos microARN de expresión (neomicina - G418 selección) o auto-miARN genes clonados en pBABE-Puro (plásmido 1764; Addgene) se han utilizado con éxito. En este último caso, la horquilla miARN con ~ 200 pb a cada lado PCR fue clonado a partir de ADN humano.
- Resuspender las células en el tubo de 15 ml de arriba con 100 l de solución nucleofection Amaxa (celda específica) por 2 x 10 6 células. Por ejemplo: Desde hace 10 Nucleofections, añadir 1 ml de reactivo.
- Una reacción a la vez, añadir 100 ml de las células a la g 2 del plásmido. Mezclar con la pipeta.
- Transfiera la mezcla a una cubeta nucleofector.
- Introducir la cubeta en el instrumento nucleofector y ejecutar el programa optimizado adecuado para la línea celular (de alta eficiencia preferido sobre la viabilidad celular).
- Llene la pipeta de transferencia con pre-calentado medio. Recoger las células en misma pipeta y la transferencia a 6-así placa. Repita para cada nucleofection, uno por cada pozo.
- Volver a la cámara de crecimiento para la incubación durante la noche.
- Reemplazar medio y permiten a las células para recuperarse durante 3-5 días.
- Sustituir medio con medio completo que contenía el nivel apropiado de puromicina o G418 como se determina a partir de la curva de interrupción prueba realizada antes de la transfección con el miARN construir.
- Monitorear las células de puromicina / G418 selección (las células que mueren).
- Vuelva a colocar medio con puromycin / G418 cada 2-3 días.
- Una vez que confluyen en la placa de 6 pocillos, pasaje, piscina y ampliar las células en grandes frascos.
- Periodo de tiempo para la expansión de las células es el usuario dependiente, pero es muy recomendable para ampliar puromycin / G418 resistentes a las células para generar crio-acciones para la detección futuro (aproximadamente 2-3 semanas, la línea celular dependiente).
- Vuelva a colocar medio con los niveles adecuados de ganciclovir (GCV) y puromycin / G418, según lo determine de la prueba de la curva de muerte a cabo antes de la transfección con la construcción de miRNA.
- Controlar las células para la selección de células (dying, Mirna focalización y caída de los conocimientos tradicionales-ZEO).
- Expandir las células en presencia de GCV y puromicina / G418.
- Preparar ADN genómico de las células GCV seleccionados.
- Amplificar por PCR se inserta con el kit de primers (ver amplificación por PCR).
- Clon en el vector TOPOTA (Invitrogen) para la secuenciación estándar o presentar producto de la PCR para la secuenciación de profundidad.
3. PCR-amplificar seleccionada Inserta la biblioteca y de secuencia
Nota: Este procedimiento se realiza con la PCR-amplificación de los objetivos de la biblioteca que han sobrevivido a la selección de ganciclovir.
5. Recoger ganciclovir células seleccionadas para preparar ADN cromosómico utilizando un GenElute mamífero genómico ADN Miniprep Kit (número de catálogo G1N10) o equivalente. Recomendamos preparar ADN de la línea celular progenitora que no contiene la biblioteca de ID para usar como un control negativo para la comparación con el ADN de las células diana seleccionados en la PCR.
- PCR Amplificar el ADN genómico con cebador de multiplicación 1 y 2 cebador de amplificación. El éxito de la amplificación se ha obtenido con la mezcla de reacción JumpStart RedTaq Readymix - para PCR (Número de catálogo P0982). Optimización de las condiciones puede ser necesario si se utiliza otra polimerasa. Consulte la Tabla 1 para una configuración de muestras de PCR y en la Tabla 2 para las condiciones del ciclo de PCR.
- Resolver 2-5 l del producto de PCR en un 1% en gel de agarosa. El producto esperado debería ser una prueba de ADN con un poco de las bandas visibles. Comparar para controlar producto de PCR del ADN genómico de las células sin la Biblioteca ID de destino.
- Proceder con la clonación y / o secuenciación profunda de producto de PCR. Si no existen productos de amplificación se observa o es idéntica para controlar el ADN, optimizar las condiciones de amplificación de PCR (es decir, la concentración de cebador, la temperatura de recocido, y ciclos).
6. La clonación y secuenciación
Nota: Recomendamos encarecidamente la clonación del producto de PCRs y luego realizando la secuenciación estándar de al menos 96 de los clones utilizando los cebadores de amplificación proporcionados en el kit. Este procedimiento se ha realizado con éxito con 96 pozos y sistemas de cultivos de una noche de purificación de plásmidos. Incluso si la secuenciación profunda se desea, los resultados preliminares de la clonación y secuenciación estándar se puede utilizar como un control de calidad para determinar si el gasto adicional de secuenciación profunda está garantizado.
- Siga el protocolo de clonación TA kit de fabricación de productos para la clonación de amplificación por PCR (supra).
- Transformar los clones en células bacterianas competentes y seleccionar durante la noche en medio que contiene antibiótico.
- Aislar y cultivar las colonias individuales en cultivo líquido que contiene antibiótico adecuado.
- Purificar el ADN plásmido. 6,5. Lleve a cabo las reacciones de secuenciación con cebadores de amplificación.
- Identificar los objetivos de genes por la alineación de secuencias BLAST con transcriptoma humano (para las transcripciones conocidas) y el genoma humano (de la novela de transcripts) *.
* Clonación de solución de problemas: Si un número elevado de inserciones de secuenciación son secuencia del plásmido, optimizar las condiciones de la clonación / secuenciación como tal:
- La amplificación del producto de PCR (insertar) la calidad y cantidad
- La ligadura de reacción
- Preparación de plásmido (calidad y cantidad)
- Secuenciación condiciones de reacción
4. Los resultados representativos
Biblioteca de la pantalla de miR-373 objetivos
Para evaluar el desempeño de la Biblioteca de la Misión Objetivo ID, miR-373 objetivos fueron seleccionados a partir de células MCF-7 de la biblioteca que expresan. MCF-7 se ha elegido porque expresa poca o ninguna detectables MIR-373 (datos no presentados). miR-373 fue elegido por su interés biológico. miR-373 promueve la expresión de la invasión tumoral y la metástasis en células MCF-7, por lo general una línea de células no metastásico [2]. Por otra parte, miR-373 ortólogos del ratón miR-290 del clúster están involucrados en la madre embrionarias de ratonesmantenimiento de las células [3], y el MIR-373 miembro de la familia, miR-372, promueve la reprogramación de fibroblastos de células madre pluripotentes inducidas [4]. Por último, hemos utilizado nucleasas de dedos de zinc para insertar un promotor de PGK-miR-373 construcción de expresión en el sitio AAVS1 en células MCF-7, y ha generado una lista de posibles objetivos de miR-373 por el análisis de microarrays de ARN (datos no mostrados).
La Biblioteca de ID de destino se transfectadas en células MCF-7, y una población estable de células se seleccionó y se amplifica en zeocin medio que contiene. Las células resultantes (MCF-7 Biblioteca) fueron transfectadas establemente con un constructo MIR-373 expresar y seleccionado en el ganciclovir que contienen medio para enriquecer las células que expresan miR-373 objetivos. Hemos observado que el control negativo células MCF-7 de biblioteca (sin MIR-373) no se haya desprendido y se redondea como se espera para las células muertas. Sin embargo, sí dejan de crecer, que se detecta fácilmente porque el rojo fenol medio que contiene no se volverá de color naranja-amarillo como se ha observado for las células que expresan el miR-373 (Fig. 5). Las células se expandió en ganciclovir medio que contiene, y secuencias diana se aislaron por PCR con cebadores que flanquean las inserciones de identificación de destino biblioteca de cDNA y DNA preparados a partir de las células supervivientes. Los productos de PCR fueron secuenciados Illumina.
Como se muestra en la Tabla 4, se obtuvieron 17.740.719 Lecturas ADNc, que asigna a 11.076 genes únicos. De estos genes únicos, 2.898 fueron detectados con más de 40 lecturas, y por lo tanto se considera jugado fiables. El vector de 13.106.469 lee se esperaba debido a que los cebadores de PCR utilizados para amplificar insertos de ADNc son 40 y 300 bases lejos del sitio de inserción. Como tal, se espera que las secuencias resultantes serían más a partir del vector diferencia más profunda secuenciación tradicional de material genómico. El catorce por ciento no se asignan a cualquier vector o ADNc, pero no se alinean con la base de datos no redundante de nucleótidos del NCBI (es decir, con éxito NR), y sólo el 1% no tenía ningún inserto de ADNc.
Una comparación inicial encontró que 10 de los genes únicos identificados con el Carnet de identificación de destino también se encuentran en la lista de los que anteriormente se habían miR-373 objetivos en TarBase (Tabla 5). Estos 10 MIR-373 objetivos fueron previamente identificados por microarreglos con ARN de las células HeLa transfectadas transitoriamente con un sintético miR-373 imitar [5]. Por otra parte, hemos detectado estos mismos 10 genes regulados a la baja en células MCF-7 que expresan el miR-373 desde el sitio AAVS1 (datos no mostrados). Por lo tanto, estos 10 son probables objetivos válidos de miR-373. Se está trabajando para caracterizar la lista de posibles objetivos y tratar de validar los accesos seleccionados experimentalmente por RT-qPCR, Western blot, y el ensayo de luciferasa.
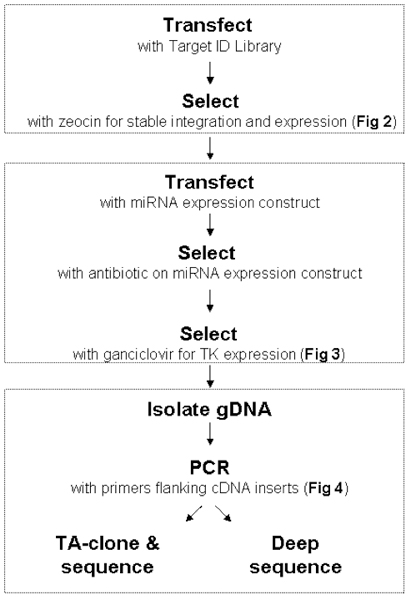
Figura 1. Flujo de trabajo para la biblioteca de ID de destino. Secciones AC: Refiérase a las secciones en el procedimiento. Cada paso se ilustra y describe en detalle en Figuras 2 y 3.

Figura 2. Transfección y selección Zeocin. La Biblioteca de ID de destino es un grupo de plásmidos (A), cada uno con un ADNc humano se inserta en la 3'-UTR después de una proteína de fusión de la timidina quinasa-zeocin (TKzeo; Fig. 4). Las células se transfectaron con Target ID de Biblioteca (B) y se dejaron recuperar durante 3-5 días. Las construcciones pueden integrarse en el genoma durante este período de recuperación (C), y expresar la transcripción codificado (D). Después de la recuperación, las células se exponen a zeocin (E). Las células que expresan la proteína de fusión TKzeo de establemente constructos integrados de ID de objetivo sobrevivir a la selección zeocin (F). Mueren las células no transfectadas (G). Además, las células que contiene una construcción que es un objetivo para un miARN endógeno o de cualquier OTHer factor de expresarse en la célula que inhibe la expresión de TKzeo de la ID de destino construcción va a morir (H).
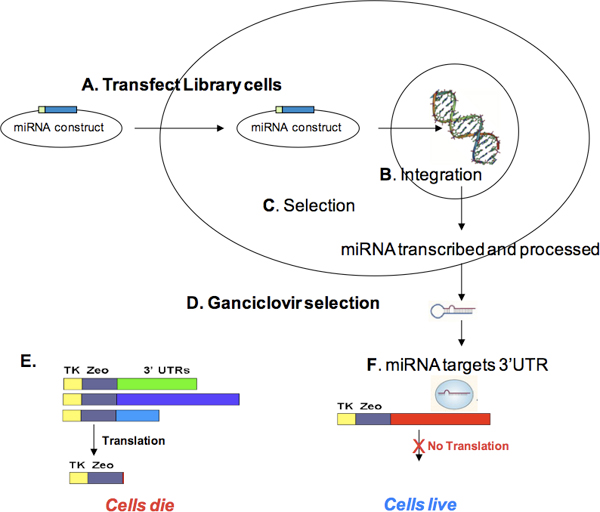
Células Figura 3. Transfección miRNA y selección de ganciclovir. Que contienen el ID de destino Biblioteca (es decir, zeocin seleccionadas las células) se transfectaron con una expresión de microARN seleccionable construir (A). Durante la recuperación, el constructo de expresión miARN puede integrar (B) y expresan el marcador seleccionable codificada en el miARN construir. Después de la selección para la integración estable (C) y la expansión de células, las células se tratan con ganciclovir (D). Las células productoras de la timidina kinasa (TK) en presencia de ganciclovir (es decir, células que expresan TKzeo no se construye dirigido por el miRNA) va a morir (E) Por otro lado, las células que contienen construcciones de la Biblioteca con el objetivo de miRNA s itas no producen TK, y por lo tanto, será sobrevivir a la selección de ganciclovir (F). Las células supervivientes se puede cultivar, ADNg aislado, y los sitios que contienen cDNA objetivo miARN amplificado por PCR utilizando los cebadores de amplificación de identificación de destino. Productos de la PCR puede ser secuenciado y alineado con el genoma humano para identificar los genes miARN objetivos.
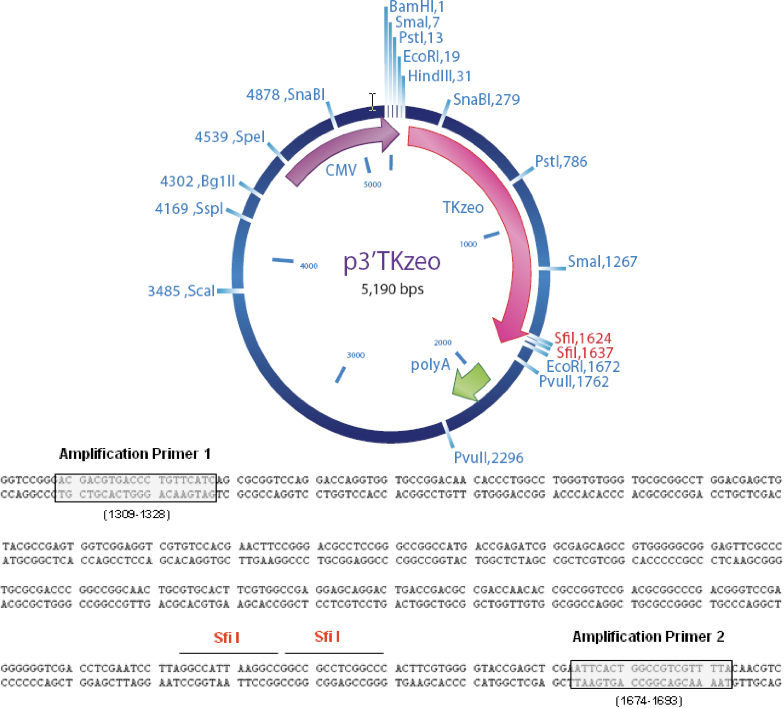
Figura 4. Mapa del plásmido y la ubicación de los cebadores de amplificación. Sfi I son los sitios de clonación de cDNA.
Primer secuencias
Objetivo de la misión ID amplificación Primer 1
5 ACGACGTGACCCTGTTCATC 3
Objetivo de la misión ID amplificación del cebador 2
5 TAAAACGACGGCCAGTGAAT 3
ig5.jpg "alt =" Figura 5 "/>
Figura 5. Efecto ganciclovir sobre el crecimiento celular. Veinticuatro placas se sembraron con dos hasta cien mil células MCF-7 o células MCF-7 contiene la biblioteca de ID de destino. Veinticuatro horas después, el medio fue reemplazado con medio que contiene 0, 8, 16 mM o ganciclovir. Después de 15 días, la placa fue fotografiado (6 pocillos superiores), a continuación, los pocillos se lavaron con HBSS y se tiñen con Azul Brillante de solución de tinción R (B6529) (6 pocillos inferiores). Nótese que el ganciclovir no tiene efecto sobre células MCF-7 sin biblioteca porque no expresan la timidina kinasa (TK). Por otro lado, células MCF-7 de Bibliotecas hacer expresa CT, pero no están completamente muertos por ganciclovir menos 8 o 16 mM, como se muestra por las células vivas que ocupan Brillinat Azul. Sin embargo, las células de Bibliotecas dejan de crecer en el ganciclovir, como se evidencia por el color del colorante rojo fenol en su soporte. Las células detenidas no acidificar su medio, y el color rojizo permanece en lugar de cambiar oange-amarillo.
| Reactivo | Volumen / reacción |
| Jumpstart RedTaq Readymix mezcla de reacción | 10 l |
| Amplificación Cebador 1 (25 mM) | 0,2 l |
| Amplificación Cebador 2 (25 mM) | 0,2 l |
| El ADN genómico | 50-200 ng |
| Agua, grado molecular | a 20 l |
Tabla 1.
| Paso | Temperatura | Tiempo | Ciclos |
| Desnaturalización inicial | 95 ° C | 5 min. | 1 |
| Desnaturalización | 95 ° C | 30 seg. | pan = "3"> 40 ciclos |
| * Recocido | 62 ° C | 30 seg. | |
| Extensión | 68 ° C | 2 min. | |
| Extensión Final | 68 ° C | 5 min. | 1 |
| Mantener | 4 ° C | Mantener |
Tabla 2.
* Las temperaturas de recocido puede variar, pero hemos observado que los mejores productos de amplificación con temperaturas de recocido que oscilan entre los 53 y los 64 ° C.
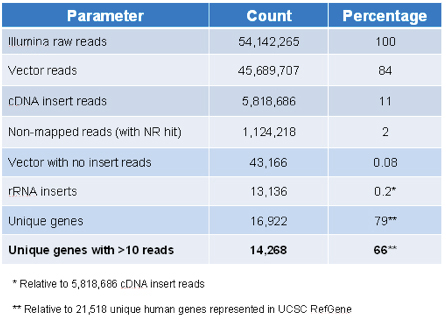
Tabla 3. ID de destino la biblioteca de contenido mediante la secuenciación de Illumina.
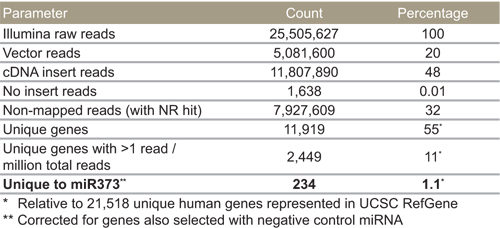
Tabla 4. MiR-373 destinos seleccionados por la secuenciación de Illumina.
les/ftp_upload/3303/3303table5.jpg "alt =" Tabla 5 "/>Tabla 5. 10 objetivos previamente identificados por el RNA microarrays.
Discusión
microRNAs son 20-24 ARN de nucleótidos que regulan la expresión de genes post-transcripcional por la inhibición de la traducción del ARNm, y con frecuencia, la desestabilización del ARNm diana (revisado en [6]). Una sola miARN puede regular varios cientos de ARNm para controlar la respuesta de una célula a las señales de desarrollo y medio ambiente. La identificación y validación de mRNAs objetivo es fundamental para determinar el papel de un miRNA y la función de estas vías. Sin embargo, la identificación de objetivos no es fácil porque, en los animales, los miRNAs y sus sitios de destino no son totalmente complementarias. La "semilla" región, basa 2 a 7 desde el extremo 5 'del miARN, es generalmente complementaria de sus objetivos. Sin embargo, hay muchas excepciones a la regla de la semilla, y aguas abajo de apareamiento de bases puede compensar un partido de la semilla imperfecta. Una serie de algoritmos computacionales se han desarrollado para predecir los genes miARN objetivos basados en la correspondencia de la semilla y la compensación aguas abajo, una estructura de destinoposición D, la secuencia de conservación, y varios otros parámetros que se hayan observado en los objetivos validados experimentalmente (revisado en [7]). Si bien las predicciones in silico son convenientes y hacer válidos los objetivos de identificar muchos genes miARN, la mayoría de los genes no predijo las pruebas experimentales de validación, y muchos objetivos reales no se predijo. Por otra parte, ya que los algoritmos informáticos se basan en las características de destino previamente determinados, que no permiten el descubrimiento de los objetivos que se apartan de lo ya conocido.
Un número de sistemas experimentales se han utilizado con éxito para identificar o descubrir los objetivos funcionales miARN en células vivas. Estos métodos de detección globales incluyen microarrays y la secuenciación del ARN, el ARN co-inmunoprecipitación (RIP), y el etiquetado de isótopos estables con aminoácidos en cultivo celular (SILAC), un método de proteómica (revisado en [7]). Cada método tiene ventajas y desventajas. Desde miARN objetivo suele desestabilizar un ARNm y conduce a su degradación, pérdida oel aumento de r en el nivel del mRNA después de la introducción de un miARN imitar o inhibidor, respectivamente, pueden identificar los genes miARN objetivos. Esta pérdida o ganancia de ARNm es fácilmente detectada por microarrays o la secuenciación de profundidad. Mientras que los métodos de detección de mRNA son más simples y más sensibles que los métodos de detección de proteínas, la detección de mRNA se falte a ninguna de los genes miARN objetivos que no se degradan. Los últimos informes del laboratorio comparando los resultados Bartell miARN objetivo de la detección de RNA con los de SILAC [8] o el perfil del ribosoma [9] indican que los miRNAs mamíferos principalmente actúa reduciendo los niveles de mRNA diana. Sin embargo, muchos otros laboratorios que trabajan con los genes miARN objetivos individuales se han detectado cambios en proteínas, pero ningún cambio en el nivel del mRNA. Los informes de lo que parece ser la regulación de traducción sin pérdida de ARNm detectable incluyen: miR-10b en la HOXD10 [10], en miR221/222 p27kip1 [11], miR-21 en Pdcd4 [12], miR-126 en p85β [13] , miR-34 en la SIRT1 [14], miR-21 en el PTEN [15], el miR-302d en Arid4b [16], el miR-200c en JAG1 [17], y miR-299, 297, 567, unº 609 de VEGF [18]. Por otra parte, Clancy et al [19] informó recientemente que la regulación de traducción por let-7 fue detectado sólo cuando las isoformas individuales de ARNm que contienen el sitio de destino vamos-7 se detectaron de forma selectiva. Lo anterior sugiere que, al menos en algunos casos, la regulación de traducción puede ser pasado por alto en un perfil compuesto - es decir, cuando todas las isoformas de mRNA se detectan como un ARNm - tal como se obtiene con microarrays muchos y los análisis de secuencias. Co-inmunoprecipitación de los complejos de mRNA de los genes miARN-a través de Argonauta (por lo general ago2) u otra proteína asociada (ARN inmunoprecipitación, o RIP) va a aislar los genes miARN objetivos con independencia del mecanismo de regulación. Además, PIR detecta endógeno, y, presumiblemente, biológicamente relevante, las interacciones. Sin embargo, no se sabe si todas las interacciones de mRNA miARN-son funcionales, y el PIR se pierda ninguna mRNA objetivos que se asocian sólo transitoriamente o se degradan rápidamente. Además, específicas de ARNm miRNA-socios debe inferirse bioinformaticamente, porque todos los pares de ARNm de miRNA-co-precipitado juntos. Finalmente, SILAC directamente identifica el producto final de la regulación miARN, la propia proteína, pero es insensible y por lo tanto se pierde proteínas raras y pequeños cambios en los niveles de proteína de plegado.
A la luz de las limitaciones con los métodos actuales de identificación de los genes miARN objetivo, nos sentimos un ensayo adicional global para identificar los objetivos funcionales miARN por un mecanismo alternativo que se necesitaba. Para cumplir con esta necesidad, la licencia de una tecnología inventada por Joop Gaken y Azim Mohamedali del King College de Londres. Su invención es una proteína de fusión de selección doble, en concreto, una timidina quinasa zeocin fusión, regulado por una biblioteca de cDNA de las posibles secuencias de miRNA en su objetivo 3'UTR. Las células transfectadas de manera estable y expresar TKzeo-cDNA construcciones se pueden seleccionar con zeocin, como se ilustra en la figura 2. Después de expresar un miARN de interés, los objetivos que Mirna se puede seleccionar wiª ganciclovir, como se ilustra en la figura 3. Ganciclovir mata las células que expresan la timidina quinasa, es decir, cualquier célula que expresa TKzeo-cDNA carece de un objetivo para el miARN de interés. Dirigida ADNc se puede aislar mediante amplificación por PCR del ADN de las células que sobreviven ganciclovir cebadores que flanquean utilizando los productos de cDNA, y la PCR puede ser secuenciado para identificar dianas.
Hemos desarrollado la tecnología del King 's College en una nueva herramienta para la identificación global y el descubrimiento de los genes miARN objetivos funcionales humanos - el objetivo de la misión Carnet de identificación. Con la Biblioteca ID de destino, los usuarios pueden aislar los genes miARN objetivos por una serie de transfección de mamífero cultivo de células y los pasos de drogas de selección. La Biblioteca es amplio, y contiene 66-79% de los genes humanos. Los resultados iniciales indican que tanto descubierto anteriormente, así como nuevos objetivos pueden ser aislados de la biblioteca MISIÓN ID de destino.
Aunque el protocolo es de cierta extensión,el uso de la Biblioteca de ID de destino requiere de técnicas estándar de laboratorio moleculares, cultivo de células mamíferas, la transfección, selección de medicamentos, PCR y secuenciación - y por lo tanto, debe estar bien al alcance de la mayoría de los biólogos. Se sugiere que se preste suficiente atención a un diseño experimental adecuado, optimizando las condiciones de cultivo, y lo más importante, antes de la prueba de cada nuevo tipo de células para determinar los niveles óptimos de drogas para los pasos de selección (kill-curvas) para asegurar el éxito con el Carnet de identificación de destino.
Al igual que con todos los métodos de identificación de genes miARN objetivo, se recomienda que los objetivos identificados por cribado de la Biblioteca de ID de destino debe ser validado con un segundo método, tales como microarrays, QRT-PCR, reportero de ensayo (es decir, la luciferasa), o el análisis occidental.
Divulgaciones
Los autores declaran las relaciones financieras con entidades comerciales que tienen un interés en los trabajos presentados.
Agradecimientos
Damos las gracias a toda la vida Sigma Misiones Científicas de la ID de destino Biblioteca equipo de desarrollo, especialmente Kevin Gutshall para encontrar la tecnología y la negociación de los términos de licencia, y Holemon Heather por su apoyo y participación en los debates fructíferos de solución de problemas. También agradecemos al Dr. Joop Gäken del King College de Londres por haber compartido los resultados no publicados y sugerencias, y Qazi Hamid de RxBiosciences para la preparación de un lote grande de cDNA y su persistencia en conseguir que clonado. Nos gustaría agradecer Nan Lin y Bahr Scott de SAFC para la realización de las matrices de cDNA y análisis de datos.
MISIÓN es una marca registrada de Sigma-Aldrich Biotecnología LP
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| JumpStart RedTaq Readymix | Sigma-Aldrich | P0982 | |
| El ganciclovir | Sigma-Aldrich | G2536 | |
| G418 | Sigma-Aldrich | G8168 | |
| Puromicina | Sigma-Aldrich | P9620 | |
| Topo TA | Invitrogen | KNM4500-01 | |
| GenElute mamíferos ADN genómico Miniprep Kit | Sigma-Aldrich | G1N10 | |
| Celular k nucleofection específicolo | Lonza | ||
| Nucleofector | Lonza | AAD-1001 | |
| Zeocin | Invitrogen | R250-01 | |
| ID de destino Biblioteca | Sigma-Aldrich | MREH01 |
Referencias
- Huang, Q., Gumireddy, K., Schrier, M. The microRNAs miR-373 and miR-520c promote tumour invasion and metastasis. Nat. Cell Biol. 10 (2), 202-210 (2008).
- Lichner, Z., Pall, E., Kerekes, A. The miR-290-295 cluster promotes pluripotency maintenance by regulating cell cycle phase distribution in mouse embryonic stem cells. Differentiation. 81 (1), 11-24 (2011).
- Subramanyam, D., Lamouille, S., Judson, R. L. Multiple targets of miR-302 and miR-372 promote reprogramming of human fibroblasts to induced pluripotent stem cells. Nat. Biotechnol. 29 (5), 443-448 (2001).
- Lim, L. P., Lau, N. C., Garrett-Engele, P. Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs. Nature. 433 (7027), 769-773 (2005).
- Huntzinger, E., Izaurralde, E. Gene silencing by microRNAs: contributions of translational repression and mRNA decay. Nat. Rev. Genet. 12 (2), 99-110 (2011).
- Thomas, M. Desperately seeking microRNA targets. Nat. Struc. Mol. Biol. 17 (10), 1169-1174 (2010).
- Baek, D. The impact of microRNAs on protein output. Nature. 455 (7209), 71-71 (2008).
- Guo, H. Mammalian microRNAs predominantly act to decrease target mRNA levels. Nature. 466 (3708), 835-840 (2010).
- Ma, L. Tumour invasion and metastasis initiated my microRNA-10b in breast cancer. Nature. 449 (7163), 682-688 (2007).
- Visone, R., et al. MicroRNAs miR-221 and miR-222, both overexpressed in human thyroid papillary carcinomas, regulate p27Kip1 protein levels and cell cycle. Endocrine-Related Cancer. 14 (3), 791-798 (2007).
- Lu, Z., et al. MicroRNA-21 promotes cell transformation by targeting the programmed cell death 4 gene. Oncogene. 27 (31), 4373-4379 (2008).
- Guo, C., et al. The noncoding RNA, miR-126, suppresses the growth of neoplastic cells by targeting phosphatidylinositol 3-kinase signaling and is frequently lost in colon cancers. Genes Chrom. Cancer. 47 (11), 939-946 (2008).
- Yamakuchi, M., et al. miR-34a repression of SIRT1 regulates apoptosis. Proc. Natl. Acad. Sci. U.S.A. 105 (36), 13421-13426 (2008).
- Wickramasinghe, N. S., et al. Estradiol downregulates miR-21 expression and increases miR-21 target gene expression in MCF-7 breast cancer cells. Nucleic Acid Res. 37 (8), 2584-2595 .
- Ciaudo, C., et al. Highly dynamic and sex-specific expression of microRNAs during early ES cell differentiation. PLoS Genetics. 5, e1000620 (2009).
- Vallejo, D. M., et al. Targeting Notch signaling by the conserved miR-8/200 microRNA family in development and cancer cells. EMBO J. 30 (4), 756-769 (2011).
- Jafarifar, F., et al. Repression of VEGFA by CA-rich element-binding microRNAs is modulated by hnRNAP. L. EMBO J. 30 (7), 1324-1334 (2011).
- Clancy, J. L., et al. mRNA isoform diversity can obscure detection of miRNA-mediated control of translation. RNA. 17 (6), 1025-1033 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados