Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis de Expresión de mamíferos enlazador histonas subtipos
En este artículo
Resumen
Se describe un conjunto de ensayos para analizar los niveles de expresión de las histonas H1 enlazador. ARNm de genes H1 individuales se mide cuantitativamente por transcripción inversa aleatoria cebador basado seguido por PCR en tiempo real, mientras que la cuantificación de proteínas de las histonas H1 se logra mediante análisis por HPLC.
Resumen
Histona H1 vinculador se une a la partícula central del nucleosoma y el ADN enlazador, lo que facilita el plegamiento de la cromatina en la estructura de orden superior. H1 es esencial para el desarrollo de mamíferos 1 y regula la expresión de genes específicos in vivo 2-4. Entre las proteínas histonas altamente conservadas, la familia de las histonas H1 linker es el grupo más heterogéneo. Hay 11 subtipos H1 en los mamíferos que son regulados diferencialmente durante el desarrollo y en diferentes tipos celulares. Estos subtipos H1 H1 somáticas son: 5 (H1a-e), el reemplazo de H1 0, 4 subtipos de células germinales específicos H1, y H1X 5. La presencia de los subtipos H1 múltiples que difieren en su afinidad de unión al ADN y la capacidad de compactación de la cromatina 6.9 proporciona un nivel adicional de modulación de la función de la cromatina. Así, el análisis cuantitativo de expresión de los subtipos H1 individuales, tanto de ARNm y proteínas, es necesario para una mejor comprensión de la regulación de la mayorPara estructura de la cromatina y la función.
Aquí se describe una serie de ensayos diseñados para el análisis de los niveles de expresión de los subtipos H1 individuales (Figura 1). la expresión de mRNA de varios genes H1 variante se mide por un conjunto de alta sensibilidad y cuantitativa de la PCR con transcripción inversa (QRT-PCR), que son más rápidos, más precisos y requieren muestras y mucho menos en comparación con el enfoque alternativo de análisis de Northern blot. A diferencia de la mayoría de los otros mensajes de ARNm celulares, ARNm para genes de la mayoría de las histonas, incluyendo la mayoría de los genes H1, carecen de una larga cola poli, pero contienen una estructura tallo-bucle en la región 3 'no traducida (UTR) 10. Por lo tanto, los ADNc se preparó a partir de ARN total por transcripción inversa usando cebadores al azar en lugar de cebadores oligo-dT. En tiempo real ensayos de PCR con primers específicos para cada uno de los subtipos H1 (Tabla 1) se llevan a cabo para obtener la medición altamente cuantitativa de los niveles de mRNA de subtipo H1 personas. La expresión de los genes de limpieza se analizan como controles para la normalización.
La abundancia relativa de las proteínas de cada subtipo H1 y histonas se obtiene mediante cromatografía líquida en fase inversa de alto rendimiento (CLAR) de análisis de las histonas total extraído de células de mamífero 11-13. El método HPLC y condiciones de elución descrito aquí dan separaciones óptimas de los subtipos H1 del ratón. Al cuantificar el perfil de HPLC, se calcula la proporción relativa de los subtipos H1 H1 individuales dentro de la familia, así como determinar la relación entre nucleosoma H1 en las células.
Protocolo
1. Preparación de la muestra y la extracción de RNA
- Antes de la extracción de RNA, todas las superficies de trabajo y pipetas deben limpiarse con 70% de etanol y se trató con solución descontaminante RNasa, tales como la RNasa Zap. Esta práctica reduce las posibilidades de contaminación RNasa y la degradación del ARN. Use guantes para todos los procedimientos.
- Para extraer el ARN de tejidos de ratón, diseccionar el órgano de interés de ratón la eutanasia, y lavar los tejidos en presencia de fosfato helada salino tamponado (PBS: 0,13 M NaCl, 5 mM de fosfato de sodio dibásico heptahidratado, 5 mM de sodio dihidrógeno heptahidratado, pH 7,4) . Proceda inmediatamente a la extracción de RNA en el paso 1.4. Si el tejido fresco no se va a procesar para extracción de RNA, las muestras de tejido deben ser congelaron rápidamente en nitrógeno líquido inmediatamente y se almacenó a -80 ° C para uso posterior.
- Si el ARN es que se extrae de cultivo de células adherentes, aspirar los medios de cultivo, enjuague con una cantidad suficiente de PBS, y añadir el reactivo Trizol (Invitrogen) sobre la placa y vaya al paso 1.4. Para las células cultivadas en suspensión, cosechar las células y las células precipitado mediante centrifugación. Desechar medios, enjuagar los gránulos brevemente con PBS, y sedimentar las células con centrifugación. Añadir el reactivo Trizol y vaya al paso 1.4.
- Reactivo Trizol suficiente es necesario para la obtención de ARN de alta calidad. Utilice 1 ml de reactivo Trizol para extraer ARN desde 50 hasta 100 mg de tejido, 5 - 10 x 10 6 células (para cultivos en suspensión) o por placa de 3,5 cm (para cultivos adherentes). Se homogeneiza el tejido en el reactivo Trizol con Polytron PT2100 homogeneizador (o equivalente). Proceda a extraer el ARN de muestras de tejido o células de acuerdo al manual del fabricante para el reactivo Trizol (Invitrogen).
- La concentración de ARN se mide utilizando Nanodrop 1000 (Thermo Scientific) y la calidad del ARN se analizaron por electroforesis en gel. Los niveles de rendimiento típicos de 1-10 mg de RNA por mg de tejido o 5-15 g de ARN por cada 1 x 10 6 células cultivadas. Para eliminar elcontaminación potencial de ARN a partir de trazas de ADN genómico, las muestras de ARN se trató con RNasa libre de DNasa (Sigma AMP-D1) de acuerdo con las instrucciones del fabricante. Repetir determinación ARN concentración y electroforesis en gel para asegurar que no degradación de ARN a partir de este tratamiento. Tienda de ARN extraído a -80 ° C.
* Nota: ARN también puede ser extraído por medio de RNAeasy Kit (Qiagen) de acuerdo con el manual de kit, o por el ADN / ARN Kit (Qiagen) si ambos ADN y el ARN se desea.
2. Cuantitativo de transcripción inversa PCR (QRT-PCR)
- El ARN total se transcriben de forma inversa en cDNA utilizando el Sistema de Síntesis III Superíndice primer capítulo (Invitrogen). Puesto que la mayoría de los ARNm de los genes H1 carecen de poli-A-colas, es crítico para utilizar hexámeros al azar en lugar de oligo-dT como cebadores para la síntesis de ADNc. Sin embargo, si el análisis de expresión de los genes con mensajes poliadenilado a niveles bajos también se desea, una mezcla de hexámeros aleatorios y shou oligo-dTLD ser utilizada en la transcripción inversa (RT) de reacción para mejorar la eficacia de la transcripción inversa de ARNm poliadenilado.
- Lleve a cabo la reacción de RT de acuerdo al manual del fabricante. Brevemente,
- En un tubo de 0,5 ml de PCR, se combinan 5 ug de RNA total, 1 l de 50 hexámeros aleatorios ng / l, 1 l de 10 mM de mezcla dNTP, y añadir tratada con DEPC H 2 O para hacer el volumen total de reacción como 10 l. Mezclar bien e incubar durante 5 minutos a 65 ° C, seguido de 1 minuto de incubación en hielo.
- Preparar 10 l de la mezcla de síntesis de ADNc: 2 l de tampón 10xRT, 4 l de 25 mM de MgCl 2, 2 l de 0,1 M DTT, 1 l de RNaseOut (40 U / l), 1 l de superíndice III RT (200 U / l) y agregarlo al ARN / imprimación mezcla.
- Incubar durante 10 minutos a 25 ° C, seguido por 50 minutos a 50 ° C y terminar la reacción a 85 ° C durante 5 minutos.
- Cada reacción típicamente produce 100-250 ng / l de producto ADNc. Guarde los productos de cDNA a -20° C o proceder de inmediato en tiempo real PCR cuantitativa (qPCR).
- qPCR puede cuantificar con precisión las copias de la secuencia de destino con una alta eficiencia y reproducibilidad 14. Nos elige qPCR medida por SYBR Green tinte, lo que da una señal fluorescente sólo cuando se intercala con el ADN de doble cadena (dsDNA). Aunque no es tan específico como Taqman ensayo de 14, este método es más económico, más fácil de adoptar en el laboratorio, y le da más versatilidad a la qPCR. Por lo tanto, es importante examinar la trama de amplificación (Figura 2A) y las curvas de fusión derivados del producto qPCR (Figura 2B) para asegurar la eficiencia de reacción y la especificidad.
| Subtipos de la histona | Ratón histona nomenclatura | Nomenclatura de las histonas Humanos | ||
| Gene el nombre de | No la adhesión. | Gen | No la adhesión. | |
| Histona H1a | Hist1h1a | NM_030609 | HIST1H1A (H1.1) | NM_005325 |
| H1b histona | Hist1h1b | NM_020034 | HIST1H1B (H1.5) | NM_005322 |
| H1c histona | Hist1h1c | NM_015786 | HIST1H1C (H1.2) | NM_005319 |
| H1d histona | Hist1h1d | NM_145713 | HIST1H1D (H1.3) | NM_005320 |
| H1e histona | Hist1h1e | NM_015787 | HIST1H1E (H1.4) | NM_005321 |
| La histona H1 ° | H1f0 | NM_008197 | H1F0 | NM_005318 |
| Histona H1oo | H1foo | NM_183811 | H1FOO | NM_153833 |
| H1t histona | Hist1h1t | NM_010377 | HIST1H1T | NM_005323 |
| H1t2 histona | H1fnt | NM_027304 | H1FNT | NM_181788 |
| H1X histona | H1fx | NM_198622 | H1FX | NM_006026 |
| Hils1 histonas | Hils1 | NM_081792 | HILS1 | AY286318 |
Tabla 1. Nomenclatura de la histona H1 en el subtipo de ratón y humano.
- Diseño de avance y retroceso cebadores de PCR específicos para cada gen H1 (Tabla 1). Debido a la similitud de secuencia elevada entre somática H1s, particularmente en la región correspondiente al dominio globular central, es crítico para asegurar que los cebadores diseñados para un subtipo H1 específica no se alinean con Otros genes H1, o cruzadas amplificar otros subtipos H1. Es también importante observar que la mayoría de los genes H1 no contienen intrones. Así intrón-que abarca los primers normalmente adoptadas para RT-PCR para evitar la contaminación genómica no están disponibles. En su lugar, las muestras de ARN debe ser pre-tratados con DNasa (véase 1,5) para eliminar cualquier cantidad traza de contaminación genómico. Además, la RT (-)-qPCR se debe realizar en paralelo para validar la ausencia de contaminación genómico en las muestras de cDNA.
- También cartillas de diseño de los genes de referencia internos, cuya expresión no cambió entre las muestras. A menudo los genes de limpieza, tales como la deshidrogenasa de gliceraldehido-3-fosfato (GAPDH) y los genes de actina beta, se eligen como genes de referencia. señales de qPCR de los genes de limpieza sirven como controles de normalización.
- Prepare cada reacción de PCR (volumen total 25 l) de la siguiente manera: 12,5 l de 2x IQ SYBR Green Supermix (Bio-Rad) (dNTPs que contienen, 50 U / ml de ADN polimerasa ITAQ, 6 mM MgCl 2, SYBR Green I y 20nM fluoresceína), 2 l de 4 ng / l de ADNc, 1,25 l de 10 nM de avance / mezcla de cebador inverso, y 9,25 l de ddH2O, y mezclar bien en Microseal placas de 96 pocillos de PCR. Utilizar juntas adhesivas 'B' Microseal (Bio-rad) para asegurar que la cubierta de la placa está sellada a la placa. Pulse o agitar brevemente la placa de PCR, y desaceleración de las mezclas de reacción de una centrifugación corta. Colocar la placa en MyiQ un solo color en tiempo real, sistema de detección por PCR (Bio-rad) para qPCR.
- Se utilizan las condiciones siguientes: qPCR 95 ° C durante 3 minutos, seguidos por 40 ciclos de 95 ° C durante 10 segundos, 60 ° C durante 20 segundos, 72 ° C durante 30 segundos. Examine las curvas de amplificación (Figura 2A) para PCR eficiencia y Ct (umbral del ciclo) los valores. La línea de umbral se puede configurar automáticamente los IQ5 2,0 ópticos versión del sistema de software.
- La eficiencia de imprimación y la concentración de ADNc óptimo necesario puede ser probada por un ensayo de curva estándar, en el que una serie de dilución de ADN genómico is utilizado para qPCR y los valores de Ct se trazan en función de registro de la cantidad de ADN molde. Un ensayo qPCR optimizado con cebadores de alta especificidad y la eficiencia dará una curva estándar lineal, con el coeficiente de determinación (R 2)> 0,98. Evite los cebadores con la longitud de amplificación más de 200 pb, que tienden a tener la eficiencia de amplificación pobres.
- Debido SYBR Green detecta cualquier dsDNA, es crítico para realizar una carrera curva de fusión después de la qPCR para asegurar que la amplificación se desea, pero no dímeros de cebadores o contaminantes, se amplifican y se detecta. Para el análisis por fusión curva, el programa del instrumento qPCR para calentar las muestras a partir de 55 ° C a 95 ° C en 0,5 ° C con incrementos de recogida de datos. La configuración predeterminada de análisis en fusión curva para MyiQ (Bio-Rad) instrumento es la siguiente: 95 ° C durante 1 minuto, 55 ° C durante 1 minuto, seguido por 81 ciclos de 10 segundos a punto de ajuste 55 ° C, se funden curva, + temp 0.5 ° C (cámara recoge los datos en cada ciclo).
- Desde temperatura de fusión (Tm) de ADN de doble cadena es dependiente de la longitud de amplificación y contenido de GC, amplicones diferentes tendrán diferentes Tm (s). Examinar las curvas de fusión de los productos derivados qPCR para confirmar la temperatura de fusión específica de amplicones deseados así como la falta de picos de ruido amplificación (Figura 2B).
- Preparar dobles o triples, las reacciones de cada ensayo para el análisis estadístico. Incluir controles negativos para QRT-PCR, tales como RT (-)-qPCR (qPCR con el ARN como molde sin transcripción inversa) y qPCR sin ARN ADNc, o fuente de ADN añadido en la mezcla de PCR. RT (-)-qPCR puede servir como control de la contaminación potencial del ADN genómico (RT (-) en la Figura 2 y 3).
- Analizar los datos qPCR con IQ5 óptico del software del sistema versión 2.0 (Bio-Rad). Normalizar los valores de expresión de los genes H1 isoforma con la expresión de limpieza gen (por ejemplo, GAPDH, beta-actina, HPRT) para obtener los niveles relativos de expresión de genes H1.
* Todos los procedimientos deben llevarse a cabo en hielo oa 4 º C.
- Diseccionar los tejidos del ratón y enjuagar con PBS enfriado con hielo. (Si no se procede a la extracción de inmediato, encaje congelar y almacenar las muestras como se describe en el paso 1.2.) Picar el tejido en trozos con hoja de afeitar. Transferir carne picada a un homogeneizador Dounce (B almirez). Añadir 10 ml de tampón de sacarosa (0,3 M de sacarosa, 15 mM de NaCl, 10 mM HEPES [pH 7,9], 2 mM EDTA, 0,5 mM PMSF, Proteasa completa Mini Tablet Inhibidor de cóctel, añadir fresco) por gramo de tejido. Homogeneizar el tejido con 10-15 golpes.
- Transferir los homogeneizados a un tubo de 15 ml, giran a 500 rpm durante 30 segundos (modelo de centrífuga: Eppendorf 5810R); transferir cuidadosamente el sobrenadante a un tubo nuevo (desechar los restos de pellets de tejidos), y se centrifuga a 2000 rpm durante 5 minutos, para precipitar las células. Continúe con el paso 3.4.
- Si las histonas y la cromatina se van a extraer a partir de células cultivadas en monocapa, enjuaguecon PBS, PBS añadir a la placa de cultivo, y cosechar las células utilizando un raspador de células, y precipitar las células por centrifugación. Para las células cultivadas en suspensión, gránulos células por centrifugación.
- Resuspender el sedimento celular en 10 ml de tampón de sacarosa suplementado con 0,5% de NP-40 (por gramo de partida cantidad de tejido o 10 (8) las células). Transferir la muestra a un homogeneizador Dounce (B almirez) y Dounce 10 golpes dentro de 20 minutos de incubación. En este punto, los núcleos se obtienen. Examinar la calidad núcleos bajo un microscopio. Pellet los núcleos por centrifugación a 2000 rpm durante 5 minutos. Eliminar el sobrenadante.
- Resuspender el sedimento en los núcleos 3 ml de tampón salino alto (0,35 M KCl, 10 mM Tris [pH 7,2], 5 mM de MgCl 2, 0,5 mM PMSF - añadir nuevo antes de cada uso) por 1 g de tejido o 10 (8) células. Transferir la muestra a una pequeña homogeneizador Dounce (B almirez) y homogeneizar con trazos 5-10.
- Alícuota de la suspensión en 3 tubos Eppendorf (1 ml cada uno), se incuban en hielo durante 20 minutos, seguido porcentrifugación a 14.000 rpm durante 10 minutos para sedimentar la cromatina. Eliminar el sobrenadante.
- Añadir 0,8 ml de 0,2 NH 2 SO 4 a cada gránulo cromatina. Use un tubo eppendorf mortero Dounce para moler la pastilla y hasta que la pastilla está completamente disociada. Se incuban las muestras en una plataforma giratoria a 4 ° C durante la noche. Histonas totales se extrajo con esta etapa de tratamiento ácido.
- Centrifugar a 14.000 rpm durante 10 minutos. Transferir el sobrenadante (extractos de las histonas) en dos tubos Eppendorf (400 l / tubo). Deseche el sedimento. Añadir 2,5 volúmenes (1 ml) de etanol enfriado con hielo a cada tubo .. Mantener las muestras a -20 ° C durante la noche.
- Centrifugar a 14.000 rpm durante 10 minutos a las histonas de pellets totales, descartar el sobrenadante. Lavar el sedimento tres veces con EtOH al 70%, dejar en el banquillo durante 20-30 minutos secar al aire. Almacenar las proteínas secas a -80 ° C o disolver en ddH 2 O y proceder a análisis por HPLC inmediatamente. Proteínas secas se pueden almacenara -80 ° C hasta por 1 año.
4. Análisis por HPLC de las histonas del vinculador
- Resuspender el sedimento en histona el volumen recomendado de ddH 2 O dependiendo de la capacidad de la columna de fase inversa y el instrumento de HPLC. Usamos columna C18 de fase inversa 250 x 4,6 mm (Vydac) y Äktapurifier UPC 900 instrumento (GE Healthcare) para el análisis por HPLC. Por lo general se disuelven 50-100 microgramos de histonas totales en 100 l de ddH 2 0 para el análisis.
- Centrifugar a 14.000 rpm durante 5 minutos para eliminar los residuos insolubles. Ensayo de Bradford proteína se utiliza para determinar la cantidad de proteína que se inyecta en la columna. Inyectar 50-100 g de proteína total en la columna de fase inversa en el sistema de HPLC. Carga de un exceso en la cantidad de proteína debe evitarse para prevenir la obstrucción de la columna.
- Fraccionar las histonas enlazador e histonas núcleo con un gradiente de acetonitrilo aumentando como se indica en la Tabla 2.
Tabla 2. Aumentar gradiente de acetonitrilo en el tiempo.
- El efluente se controló a 214 nm, y el perfil de HPLC (FigurasRe 4) se registran y analizan utilizando Äktapurifier UPC 900 (GE Healthcare), con 5.11 de software UNICORN (GE Healthcare). Las fracciones de proteína puede también recogieron con la fracción de auto-colector (Frac-920 - GE) para su posterior análisis, por ejemplo, espectrometría de SDS-PAGE y de masas.
- Los valores de A 214 de los picos de cada subtipo H1 y H2B están normalizados por el número de enlaces peptídicos de cada proteína histona correspondiente. La proporción relativa de los subtipos H1 individuales dentro de la familia de histona H1, así como la proporción de los subtipos H1 a partículas de núcleo nucleosomas puede ser calculada a partir de estos valores normalizada A 214 (Figura 5).
5. Los resultados representativos
La lista de los subtipos H1 de mamífero, el organigrama general y los resultados representativos de los análisis de expresión de los genes individuales H1 histonas se muestran en la Tabla 1, la Figura 1 y Figura 2-5, respectivamente.Figura 2A muestra curvas típicas de amplificación de H1A reacciones qPCR utilizando ADNc preparado a partir de hígado de ratón y mESCs, mientras que la figura 2B muestra las curvas de fusión derivados de los amplicones correspondientes. La curva de fusión muestra un solo pico característico a temperatura de fusión (Tm) a 86 ° C para la amplificación por PCR H1a, y carece no específicos picos de fondo, lo que sugiere una alta especificidad de H1a ensayo qPCR. Evaluación de la trama de amplificación (Figura 2A) muestra que las reacciones qPCR triplicados de cada muestra dio señales consistentes con los valores de Ct casi idénticas, lo que sugiere una alta reproducibilidad. La falta de amplicones acumulación de RT (-)-qPCR reacciones indica que la contaminación de ADN genómico no estaba presente, o mínima. Utilizando los valores de Ct de genes H1 y genes de limpieza, tales como la GAPDH, los niveles relativos de expresión de ARN de cada gen H1 se calcularon. Ejemplos de los resultados calculados para H1 ° y los genes H1A se muestran en la Figura 3. Los niveles de expresión relativos de ARNm H1a son más altos en comparación con mESCs hígado de ratón, mientras que H1 ° expresión es mucho mayor en el hígado que en mESCs.
La diferencia en la expresión de H1a o ° H1 en el hígado del ratón MESC frente a los adultos también es evidente a partir de perfiles de HPLC de las proteínas histonas (Figura 4). H1 º, el H1 diferenciación específica, se acumula a una gran cantidad de tejidos maduros, lo que representa el 27,2% del total de H1 en el hígado de un adulto (Figura 5). En contraste, H1 ° proteína es casi ausente en mESCs indiferenciados (Figura 4B). Por otro lado, H1a es altamente expresado, tanto en las transcripciones de mRNA y proteínas, en mESCs (Figura 3 y 4B). A través de la cuantificación de los picos de H1 en el perfil de HPLC, la proporción relativa de cada subtipo H1 individuo dentro de la familia H1 se determina (Figura 5). Además, los valores de subtipo H1 individual (oH1 total) por nucleosoma se puede calcular por la relación de la normalizada Un valor de pico de 214 subtipos H1 correspondiente (o suma de H1 total) para una mitad de la normalizada A 214 valores para H2B (Figura 5B).
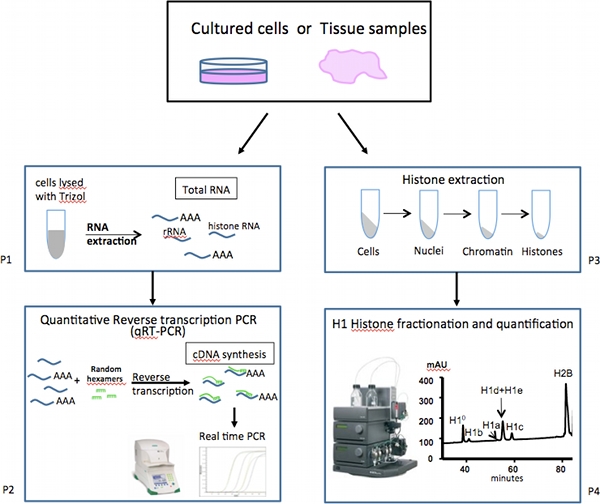
Figura 1. Esquema general de análisis de expresión de mamíferos enlazador histonas subtipos.
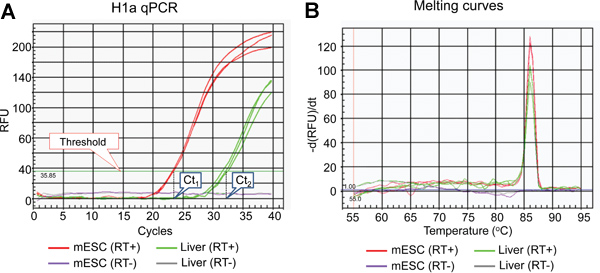
Figura 2. Los resultados representativos de H1a ensayo qPCR. (A) Amplificación parcela de H1a ensayo qPCR. La línea límite y los valores de Ct establecidas por el software del sistema óptico IQ5 se indican. (B) Derivados funden las curvas de productos qPCR se muestra en (A).
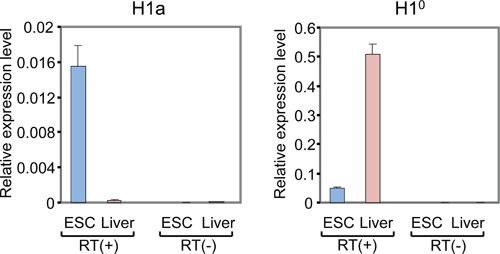
Figura 3. QRT-PCR análisis de los niveles de mRNA de H1a y H1 º en mESCs y m para adultosouse hígado. El eje Y representa los niveles relativos de expresión de genes H1 a la de la GAPDH referencia gen. qPCR con RT (-) (muestras de ARN sin transcripción inversa) muestra señales mínimas o ninguna.
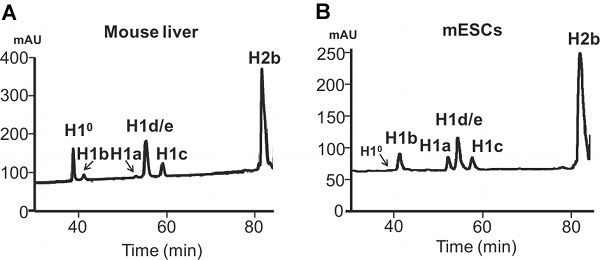
Figura 4. El análisis por HPLC de las histonas extraídos de células de mamífero. HPLC en fase inversa análisis de 100 microgramos histonas total extraído de hígado de ratón adulto (A) y CES de ratón (B). Eje X: tiempo de elución. Eje Y: MAU, mili-unidades de absorbencia.
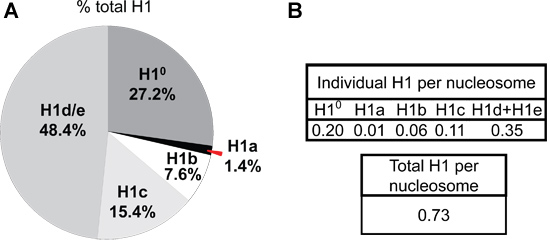
Figura 5. Composición subtipo H1 y H1 por razones de nucleosomas en el hígado de ratón adulto. Los valores de A 214 de la superficie del pico para cada isoforma H1 y H2B se calculan utilizando el software UNICORN 5,11 (GE Healthcare), y normalizado por el número de enlaces peptídicos presentes en la proteína histona correspondiente. La suma de A normalizada214 valores de todos los subtipos H1 se obtiene como el valor de H1 total. El porcentaje de H1 total para cada subtipo H1 (A), así como la relación de H1 a nucleosoma (representado por una mitad de la normalizada A 214 valores de H2B) (B) en el hígado de ratón adulto se calcula a partir del perfil de HPLC muestra en la Figura 4A.
Discusión
El conjunto de ensayos que aquí se presenta permitir el análisis exhaustivo de los niveles de expresión de mamíferos subtipos de histonas linker. Correctamente diseñados QRT-PCR proporcionar mediciones de alta sensibilidad y precisión de los mensajes de ARN de los genes histona H1 de mamífero. La parte crítica de la QRT-PCR para los genes del subtipo enlazador histonas es la preparación de cDNA utilizando cebadores aleatorios basados en la transcripción inversa. ARNm de genes más histonas, incluyendo lo...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo es apoyado por el NIH subvención GM085261 y un Premio de la Coalición de Georgia Cancer Académico Distinguido (a la fiebre amarilla).
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| RNasa Zap | Applied Biosystems | AM9780 | |
| Reactivo Trizol | Invitrogen | 15596-018 | |
| SuperScriptIII | Invitrogen | 18080-051 | |
| Etanol absoluto | Fisher Scientific | BP2818-4 | |
| IQ SYBR Green | Bio-Rad | 170-8880 | |
| RNeasy Mini Kit | Qiagen | 74104 | |
| Desoxirribonucleasa I | Sigma | AMP-D1 | |
| Microseal placas de 96 pocillos PCR | Bio-Rad | MSP-9605 | |
| Los sellos adhesivos 'B' Microseal | Bio-Rad | MSB-1001 | |
| Sacarosa | Agros Organics | AC40594 | |
| El fosfato de sodio dibásico heptahidratado (Na 2 HPO 4 · 7H 2 O) | Fisher Scientific | BP332 | |
| Cloruro sódico (NaCl) | América Bioanalítica | AB01915 | |
| Dihidrógeno de sodio heptahidratado (NaH 2 PO 4 · 7H2O) | Fisher Scientific | BP-330 | |
| HEPES | Fisher Scientific | BP310 | |
| Completa el inhibidor de la proteasa cóctel mini tablet | Roche Ciencias Aplicadas | 11836153001 | |
| EDTA | Sigma | E-5134 | |
| Fenilmetanosulfonilo fluoruro (PMSF) | América Bioanalítica | AB01620 | |
| Nonidet-40 (NP-40) | América Bioanalítica | AB01425 | |
| El cloruro de potasio (KCL) | Fisher Scientific | BP366 | |
| Tris [hidroximetil aminometano] | América Bioanalítica | AB02000 | |
| Cloruro de magnesio (MgCl2) | Fisher Scientific | BP214 | |
| El ácido sulfúrico (H2SO4) | VWR | VW3648-3 | |
| De hidróxido de amonio (NH 4 OH) | Agros Organics | AC42330 | |
| Bradford ensayo de proteínas | Bio-Rad | 500-0001 | |
| Acetonitrilo | EMD | AX0145-1 | |
| Ácido trifluoroacético (TFA) | JT Baker | 9470-01 |
Referencias
- Fan, Y. H1 linker histones are essential for mouse development and affect nucleosome spacing in vivo. Mol. Cell Biol. 23, 4559-4572 (2003).
- Woodcock, C. L., Skoultchi, A. I., Fan, Y. Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17-25 (2006).
- Shen, X., Gorovsky, M. A. Linker histone H1 regulates specific gene expression but not global transcription in vivo. Cell. 86, 475-483 (1996).
- Alami, R. Mammalian linker-histone subtypes differentially affect gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 100, 5920-5925 (2003).
- Happel, N., Doenecke, D. Histone H1 and its isoforms: contribution to chromatin structure and function. Gene. 431, 1-12 (2009).
- Clausell, J., Happel, N., Hale, T. K., Doenecke, D., Beato, M. Histone H1 subtypes differentially modulate chromatin condensation without preventing ATP-dependent remodeling by SWI/SNF or NURF. PLoS One. 4, 0007243-0007243 (2009).
- Khadake, J. R., Rao, M. R. DNA- chromatin-condensing properties of rat testes H1a and H1t compared to those of rat liver H1bdec; H1t is a poor condenser of chromatin. Biochemistry. 34, 15792-15801 (1995).
- Orrego, M. Differential affinity of mammalian histone H1 somatic subtypes for DNA and chromatin. BMC Biol. 5, 22-22 (2007).
- Th'ng, J. P., Sung, R., Ye, M., Hendzel, M. J. H1 family histones in the nucleus. Control of binding and localization by the C-terminal domain. J. Biol. Chem. 280, 27809-27814 (2005).
- Wang, Z. F., Sirotkin, A. M., Buchold, G. M., Skoultchi, A. I., Marzluff, W. F. The mouse histone H1 genes: gene organization and differential regulation. J. Mol. Biol. 271, 124-138 (1997).
- Brown, D. T., Sittman, D. B. Identification through overexpression and tagging of the variant type of the mouse H1e and H1c genes. J. Biol. Chem. 268, 713-718 (1993).
- Fan, Y., Sirotkin, A., Russell, R. G., Ayala, J., Skoultchi, A. I. Individual somatic H1 subtypes are dispensable for mouse development even in mice lacking the H1(0) replacement subtype. Mol. Cell. Biol. 21, 7933-7943 (2001).
- Fan, Y., Skoultchi, A. I. Genetic analysis of H1 linker histone subtypes and their functions in mice. Methods Enzymol. 377, 85-107 (2004).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Res. 6, 986-994 (1996).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados