Method Article
Producción y focalización de monovalente Quantum Dots
* Estos autores han contribuido por igual
En este artículo
Resumen
We provide detailed instructions for the preparation of monovalent targeted quantum dots (mQDs) from phosphorothioate DNA of defined length. DNA wrapping occurs in high yield, and therefore, products do not require purification. We demonstrate the use of the SNAP tag to target mQDs to cell-surface receptors for live-cell imaging applications.
Resumen
The multivalent nature of commercial quantum dots (QDs) and the difficulties associated with producing monovalent dots have limited their applications in biology, where clustering and the spatial organization of biomolecules is often the object of study. We describe here a protocol to produce monovalent quantum dots (mQDs) that can be accomplished in most biological research laboratories via a simple mixing of CdSe/ZnS core/shell QDs with phosphorothioate DNA (ptDNA) of defined length. After a single ptDNA strand has wrapped the QD, additional strands are excluded from the surface. Production of mQDs in this manner can be accomplished at small and large scale, with commercial reagents, and in minimal steps. These mQDs can be specifically directed to biological targets by hybridization to a complementary single stranded targeting DNA. We demonstrate the use of these mQDs as imaging probes by labeling SNAP-tagged Notch receptors on live mammalian cells, targeted by mQDs bearing a benzylguanine moiety.
Introducción
La dinámica de las moléculas individuales en células vivas contribuye a su función biológica. Formación de imágenes de fluorescencia sola molécula es un método popular para estudiar la dinámica de moléculas individuales en la superficie de la célula 1,2,3. Sin embargo, las sondas de imagen más comúnmente utilizados en estos estudios tienen varias desventajas importantes. Por ejemplo, los tintes orgánicos convencionales y proteínas fluorescentes proporcionan brillo moderado, aproximadamente 10 5 -10 6 M -1 cm -1, pero son fotoquímicamente inestable, blanqueo después de la emisión de aproximadamente 10 5 -10 6 fotones en condiciones típicas de formación de imágenes de células vivas 4,5. Por el contrario, las nanopartículas semiconductoras, llamados con frecuencia puntos cuánticos (QDs), son significativamente más brillante y más estable, con coeficientes de extinción en el rango de 10 6 -10 7 M -1 cm -1 y superior a 10 7 -10 8 fotones emitidos antes photobleaching 5. El brillo mejorado y fotoestabilidad de los puntos cuánticos más fluoróforos orgánicos permite la observación de moléculas individuales a velocidades de cuadro significativamente mayores y más mucho más tiempo trayectorias 6.
A pesar de sus ventajas y disponibilidad comercial, varios pasivos se mantienen para estos poderosos agentes de imagen. Primero, han valencia de focalización, que puede resultar en la reticulación de biomoléculas específicas 6 mal definida. En segundo lugar, por lo general tienen un gran tamaño hidrodinámico (> 20 nm) que limita la accesibilidad a ciertos ambientes celulares de hacinamiento 7. En tercer lugar, han limitado la orientación modularidad 7. Varias estrategias han tratado de abordar estos problemas 8,9,10, pero generalmente requieren unos conocimientos y reactivos especializados de implementar.
Para hacer frente a estos problemas, nos informó recientemente una estrategia de "exclusión estérica" para preparar monovalente, pequeñas ymodulares puntos cuánticos 11. Los puntos cuánticos se envuelven con un solo polímero de ADN fosforotioato largo (ptDNA). El ptDNA une a la superficie QD a través de múltiples interacciones Zn-S entre la superficie expuesta átomos de Zn y de los grupos fosforotioato del polímero ptDNA. Un único polímero unido electrostáticamente y excluye estéricamente la unión de equivalentes adicionales del polímero sin aumentar significativamente el tamaño global de la partícula (aproximadamente 2 nm). Todos los reactivos están disponibles comercialmente, se forman productos con un alto rendimiento, y el proceso requiere pocos pasos de desalinización para la purificación. Una vez marcado, puntos cuánticos envueltos con una sola ptDNA (MQDS) se unen a las hebras de ADN complementarias que llevan dominios dirigidos (por ejemplo, bencilguanina (BG), benzylcytosine, o haluros de alquilo).
Estas funcionalidades se dirigen a los MQDS específicamente para etiquetas enzimáticas tales como SNAP, CLIP Y HALO que se fusionan genéticamente para la proteína de interés. Se trata de un protocolo para la síntesis, Focalización y imágenes de células vivas de MQDS producidas por la exclusión estérica.
Protocolo
1 Producción de monovalente Quantum Dots
- De transferencia de fase de los puntos cuánticos de orgánica a la fase acuosa
- Diluir 200 l de una solución 1 M de puntos cuánticos fase orgánica con 400 l de cloroformo en un vial de vidrio de 5 ml.
- Mezclar 400 l de una solución 0,3 M de bromuro de tetrabutilamonio (TBAB) cloroformo con 36 l de puro tiol mPEG (CH 3 O (CH 2 CH 2 O) 6 C 2 H 5 SH) y agitar O / N.
- Añadir 800 l de una solución acuosa 0,2 M de NaOH y se agita durante 30 seg. Una transferencia de fase se produce en unos pocos minutos, indicados por la transferencia de las partículas coloreadas a la fase acuosa por encima de la fase orgánica densa.
- Si las partículas de agregado en una tercera fase entre las fases acuosa y orgánica, aumentar el tiempo de incubación con el tiol mPEG. Si la fase acuosa queda clara, los puntos cuánticos no lo hicieron de transferencia de fase (véase la Figura 3A). Alternativamente, aumentar la concentración de mPEG tiol en el paso 2 para aliviar transferencia de fase pobre.
- Recuperar el (a color) fase acuosa y luego concentrar los puntos cuánticos recogidos con una columna de centrifugación Centricon (30 kDa de peso molecular de corte) a 1 ml.
- Añadir la solución de QD se concentró en una columna de Sephadex NAP10 pre-equilibrada con 10 mM de tampón Tris que contenía NaCl 30 mM (pH 8,0). La elución de los puntos cuánticos con 1,5 ml de tampón de elución por flujo por gravedad.
- Medir la concentración de los puntos cuánticos con la espectroscopia de absorción a 350 nm.
- Preparación de MQDS
- Compra (o sintetizar) ptDNA. Este protocolo utiliza la secuencia 5'-A S 50 (CT) 10 (ACTG) 5 -3 '(ver Tabla 1).
| ADN | Secuencia |
| 5'-A S 50 (CT) 10 (ACTG) 5 3 ' | A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S A S CTCTCTCTCTCTCTCTCTCTACTGACTGACTGACTGACTG |
| 5'-NH 2 - (CT) 10 (CAGT) 5 -3 ' | NH 2 - / C6spacer / CTCTCTCTCTCTCTCTCTCTCAGTCAGTCAGTCAGTCAGT |
| BG-ADN | BG / C6spacer / CTCTCTCTCTCTCTCTCTCTCAGTCAGTCAGTCAGTCAGT |
Tabla 1. secuencias de ADN usadas para producir y MQDS objetivo.
- Preparar 1 ml de solución de QD 100 nM en tampón Tris 10 mM que contenía NaCl 30 mM (pH 8).
- Agregar gota a gota 500 l de la solución 100 nM ptDNA a los puntos cuánticos acuosas durante 1 minuto mientras se agita vigorosamente. Revuelva o colocar en un agitador durante una 9 horas más.
Nota: Esto debería producir en teoría una relación 1: 2 de ptDNA: QD. Sin embargo, la estequiometría real es nunca perfecto - generalmente el resultado está más cerca de 1: 1,5. Con el fin de producir una proporción de 1: 1 de ptDNA: QD, densitometría después de electroforesis en gel es necesaria para cuantificar la proporción exacta de conjugación dados los volúmenes conocidos utilizados anteriormente. - Retire ~ 10 l de la mezcla de QD para ejecutarse en un gel de agarosa analítico. Retire también una concentración similar de los puntos cuánticos no conjugados en fase acuosa (del paso 1.2.1).
- Añadir ~ 2 μ; L de tampón de carga 6x Ficoll (para aumentar la densidad de la solución) y ejecutar estas dos muestras juntas en un gel de 0,8% p / v de agarosa en tampón de borato de sodio durante 15 min a 150 V. Una sola banda en el carril de control no conjugada migrar cerca el bien, y dos bandas en el carril con los puntos cuánticos conjugados son vistos (ver geles en la Figura 2B).
- Calcular la fracción QD no conjugado usando la intensidad relativa de las dos bandas (ver la ecuación en la figura 2B). A continuación, utilice esa fracción para calcular el volumen adicional de 100 nM ptDNA solución requerida para que coincida con el número de puntos cuánticos.
- Repita los pasos 1.2.3 al 1.2.5, una vez más con este volumen calculado o hasta que el colapso de los puntos cuánticos conjugados en una sola banda en el gel que indica la conjugación completa de todos los puntos cuánticos.
- Añadir 100 l de 10 mM (CO 2 H) CH 2 O (CH 2 CH 2 O) 6 C 11 H 23 SH (carboxi PEG6 alcano tiol) en 10 mM de tampón Tris containing NaCl 30 mM (pH 8) a los MQDS conjugados producidos anteriormente, y agitar durante 10 min.
Nota: Estos ligandos PEG pasivar la superficie QD. Un PEG más largo será mejor pasivar los puntos cuánticos, sin embargo, también se incrementará su tamaño. La funcionalidad alcanotiol hidrófobo tiene una afinidad mucho mayor para los puntos cuánticos, en conjunto, que los menos hidrófoba PEG-tiol utilizado para la transferencia de fase. Por lo tanto, no permita que este paso para proceder demasiado largo o la ptDNA será reemplazado por el alcano-PEG-tiol. Después de una incubación de ~ 30 min, una fracción significativa de ADN han sido desplazados de los puntos cuánticos (Figura 3B). - Para eliminar el exceso de PEG-alcano tiol, añadir 0,5 ml de la solución de QD a una columna de Sephadex NAP5 pre-equilibrada con tampón de elución (10 mM de tampón Tris que contenía NaCl 30 mM). Recoge los MQDS con 1,0 ml de tampón de elución por flujo por gravedad.
- Se concentran los puntos cuánticos recogidos con una columna de centrifugación Centricon (30 kDa). Guarde estos MQDS a 4 ° C durante meses. No frEeze ellos.
Nota: Si se observa un sedimento de color en la parte inferior del tubo, los puntos han agregado y es probable ya no utilizable.
2. Producción de Orientación de ADN (Benzylguanine-)
- Producir o adquirir ADN terminado en amina. Este protocolo utiliza la secuencia 5'-NH 2 - (CT) 10 - (CAGT) 5 -3 '(ver Tabla 1). El enlazador poli-CT aumenta la accesibilidad a la funcionalidad enterrado profundamente en el glycocalyx pero puede no ser necesario para todas las aplicaciones. Si se tiene acceso a un sintetizador de ADN, producir ADN modificado amino-utilizando un 5 'amino-modificador (5' apropiada C6 amino-modificador).
- Disolver BG-N-hidroxisuccinimida (NHS-BG) en dimetil sulfóxido seco (DMSO) a una concentración de 10 mg / ml.
Nota: BG-NHS va a absorber agua del aire y dar lugar a la hidrólisis del éster de NHS-reactiva, en particular en disolventes higroscópicos tales como DMSO y dimetilformamida (DMF). BG-NHS es la mejor tienda ded a -80 ° C como un polvo en su propio vial dentro de un recipiente que contiene desecante secundaria. Calentar a temperatura ambiente antes de abrir el vial. Alternativamente, alícuota de inmediato para un solo uso el éster BG-NHS en un disolvente polar seco y congelar a -80 ° C, o reaccionar completamente con el ADN amina en la etapa 2.2. - Reaccionar mezclando el BG-NHS en DMSO a partir del paso 2.1 con ADN terminado en amina en HEPES (4-(2-hidroxietil) -1-piperazinetanosulfónico) tamponada (pH 8,5) de agua. En un típico reaccionar 20 l de 10 mg / ml BG-NHS en DMSO con 2 l de 2 mM NH 2 - (CT) 10 - (CAGT) 5 en 58 l de agua desionizada tamponada con 20 l de HEPES 0,5 M pH 8,5. Llevar a cabo las reacciones en un tubo de 1.5 ml. Mezclar rápidamente como la reacción procede rápidamente y deben competir con la hidrólisis NHS-éster.
- Sonicar durante 5-10 minutos para asegurar una buena mezcla. Incubar ~ 1 hora a TA. La relación estequiométrica de BG: ADN puede ser alterado para conducir la reacción hasta su finalización.
- desalar la reacción utilizando una columna NAP5 estándar en 100 mM de acetato de trietilamina (TEAA, pH 7).
- Se purifica el BG-ADN de la amina-ADN sin reaccionar por HPLC en fase inversa funcionamiento de un TEAA 100 mM: gradiente de acetonitrilo (ACN) entre 8% y 95% de acetonitrilo durante 30 min. La amina que no ha reaccionado-ADN se eluye antes de la ADN-BG. Recoger la fracción BG-ADN en un tubo cónico.
- Utilice una columna C18 Zorbax para la purificación de oligonucleótidos estándar y el gradiente siguiente:
0 → 15 min: 8 → 30% de ACN;
15 → 21 min: 30-90% de ACN;
21 → 25 min: 90 → 8% ACN;
25 → 30 min: 8% ACN;
- Utilice una columna C18 Zorbax para la purificación de oligonucleótidos estándar y el gradiente siguiente:
- Congelar en nitrógeno líquido y liofilizar la fracción recogida. Resuspender el ADN en agua desionizada. Para ser muy cuidadoso, liofilizar dos veces más para eliminar TEAA residual. TEAA residual puede ser tóxico para las células a concentraciones más altas.
- Resuspender el BG-ADN liofilizado en agua, por lo que asegúrese de lavarse las caras de jutubo e, y se diluye hasta ~ 25 mM de stock. Alícuota y almacenar a 4 ° C. Las muestras se almacenaron durante ~ 9 meses sin degradación apreciable.
3. Las células Etiquetado en vivo con monovalente Quantum Dots
- Opcionalmente, pasivar los MQDS justo antes de un experimento de formación de imágenes usando reactivos tales como caseína (0,5%) o albúmina de suero bovino (BSA, 1-3%).
- Las células de placas que expresan la proteína marcada con SNAP sobre el total de fluorescencia de reflexión interna (TIRF) de vidrio-calidad. En este experimento, se utiliza una línea celular U2OS expresa un receptor Notch1 etiquetados-SNAP y superficies de cristal de alta calidad.
- Después de que las células se han unido al vidrio (~ 24 h), eliminar el medio de crecimiento, lavar con PBS, y se incuban las células durante 10-30 min a RT en ~ 100-150 l de PBS o medio celular que contiene ~ 1 M BG ADN desde el paso 2.6 anterior.
- Después de la incubación con BG, lave cuidadosamente las células con PBS o medio celular. Se incuban las células de ~ 5-10 min con la famie MQDS producidos en la etapa 1.2.9 (y opcionalmente pasivados en el paso 3.1).
- Opcionalmente, lavar los MQDS no consolidados y devolver las células a un búfer / medios adecuados tanto para la imagen y la cultura. Para las células que iban a ser fotografiado en el modo de TIRF, una etapa de lavado final fue a menudo innecesario como MQDS no unidos en solución difundido tan rápidamente en comparación con MQDS obligado como para ser indetectable durante el análisis.
4. Microscopia y Análisis de
- Imagen de las células utilizando un microscopio TIRF.
Nota: El alcance debe tener la excitación separable y filtros de emisión, o un cubo de filtro QD personalizado. Puntos cuánticos son mejor entusiasmados con un láser de baja longitud de onda (405 o 488 nm). Puntos cuánticos de diferente diámetro tienen longitudes de onda de emisión característicos, normalmente asociado con el cambio de un gran Stoke. - Debido al brillo de MQDS, recoger imágenes a altas velocidades de fotogramas en TIRF mientras que imágenes de la parte basal de una célula viva.
Nota: Como alternativa, la dinámica de una sola molécula puede ser followed en otros planos focales utilizando un microscopio confocal de disco giratorio. Software automatizado de seguimiento de una sola partícula es generalmente exitosa en un seguimiento preciso de los MQDS brillantes y fotoestables.
Resultados
La transferencia de fase de los puntos cuánticos de una orgánica a una fase acuosa es crítico para la producción de MQDS, pero puede ser tanto acondicionamiento y QD-específico. Transferencia de fase en la sección 1.1 debería aparecer tan limpio como los dos primeros viales en la Figura 3. Si la transferencia se parece más a vial 3, entonces uno debe tratar de nuevo con distintas condiciones.
Una vez que los puntos cuánticos están recubiertas con el ADN cargado negativamente que deberían migrar en un gel por separado de los puntos cuánticos no envuelto. Usando una alícuota de puntos cuánticos abierto como un control, una segunda, banda más rápido-la migración debería aparecer tras la adición del ptDNA, como se ve en la primera gel en la Figura 2B. Formación completa de MQDS se demostró con la pérdida de la banda inmóvil, y su colapso en la banda móvil como visto en los últimos gel en la Figura 2B. Si una parte alícuota de su producto MQD migra como una sola banda separable de los puntos cuánticos que quiero, entonces sus MQDS son monovalentes y r Eady para ser utilizado en etapas adicionales.
Etiquetado de la célula debe ser específico para el destino de su elección y debe depender de los niveles de expresión de proteínas y la concentración MQD. Optimización empírica tanto de concentración MQD y MQD pasivación a menudo es necesario para cualquier aplicación dada. Figura 4B demuestra el etiquetado de células U2OS que expresan un receptor Notch-etiquetados SNAP con MQDS que varían en concentración de 0,5 nM a 10 nM.
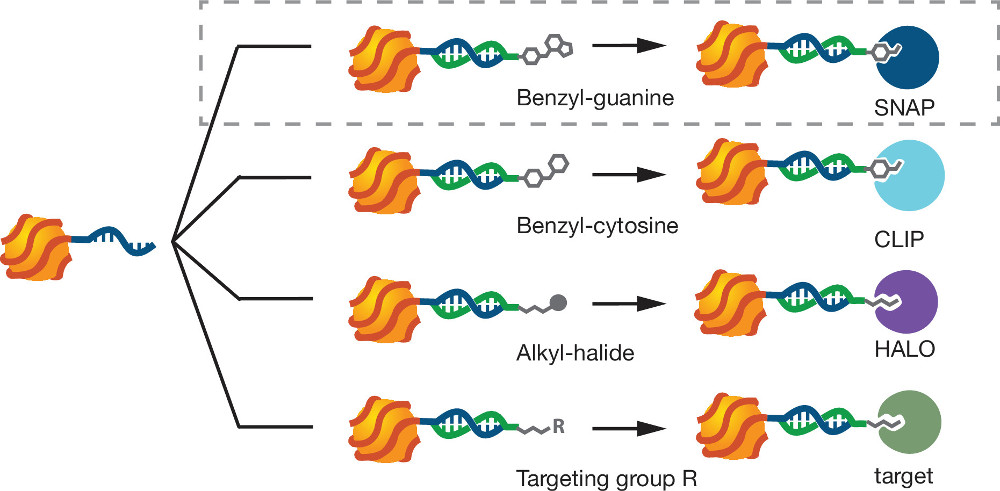
Figura 1. modular la focalización de MQDS. MQDS hibridan a ssDNA funcionalizado por varias moléculas de direccionamiento. Bencilguanina vincula-dianas de ADN MQDS a SNAP-etiquetados proteínas de fusión. Modificaciones Otros 5 'y secuencias de ADN permiten la focalización de MQDS a una variedad de otras biomoléculas./files/ftp_upload/52198/52198fig1highres.jpg "target =" _blank "> Haga clic aquí para ver una versión más grande de esta figura.
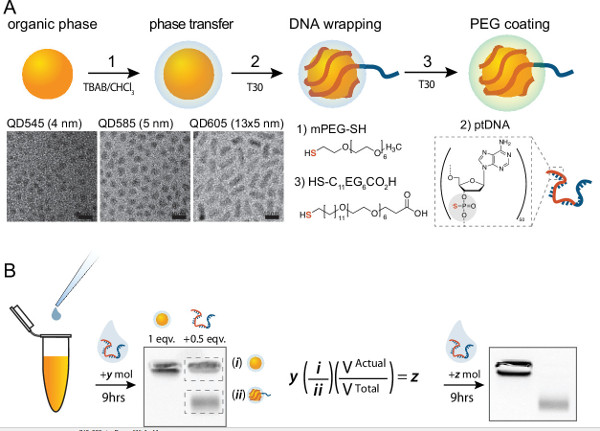
Figura 2 Esquema para la producción de MQDS. (A) los puntos cuánticos de fase orgánica se transfieren a la fase acuosa por tratamiento con mPEG tiol y TBAB, envuelto con ptDNA, y pasivado con carboxi peg6 alcano tiol. Representante imágenes de TEM son QD545 orgánica, QD585, y QD605, respectivamente. (B) Un método para determinar empíricamente la estequiometría de la interacción entre los puntos cuánticos y ptDNA en el paso dos anteriormente. QD y ptDNA estequiometrías son empíricamente verificados por densitometría de manera que la estequiometría de la reacción final es 1: 1 (QD: ptDNA). El número inicial de moles de ptDNA se multiplica por la relación de QD: MQD, y ajustado por el volumen real af Conjugación ter para dar el número de moles necesarios para alcanzar 1:. 1 Conjugación Por favor haga clic aquí para ver una versión más grande de esta figura.
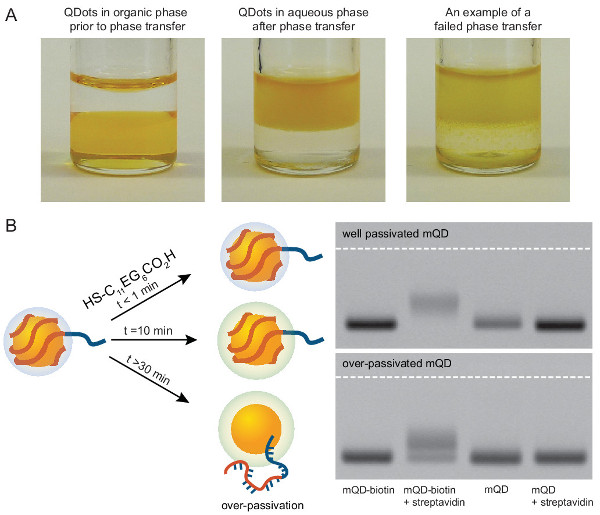
Figura 3. detalles experimentales para la producción de MQDS. (A) Fotos representativas de exitosos y fallidos de transferencia de fase. Puntos cuánticos debe transferir visualmente entre la fase orgánica densa y la fase acuosa menos densa. La separación de fases incompleta indica transferencia pobres. (B) ptDNA puede ser sustituido por el alcano-PEG-tiol. Datos de gel de Derecho es representativa de una reacción en la que una fracción significativa de ptDNA han sido desplazados de los puntos cuánticos debido a la sobre-pasivación.es.jpg "target =" _blank "> Haga clic aquí para ver una versión más grande de esta figura.
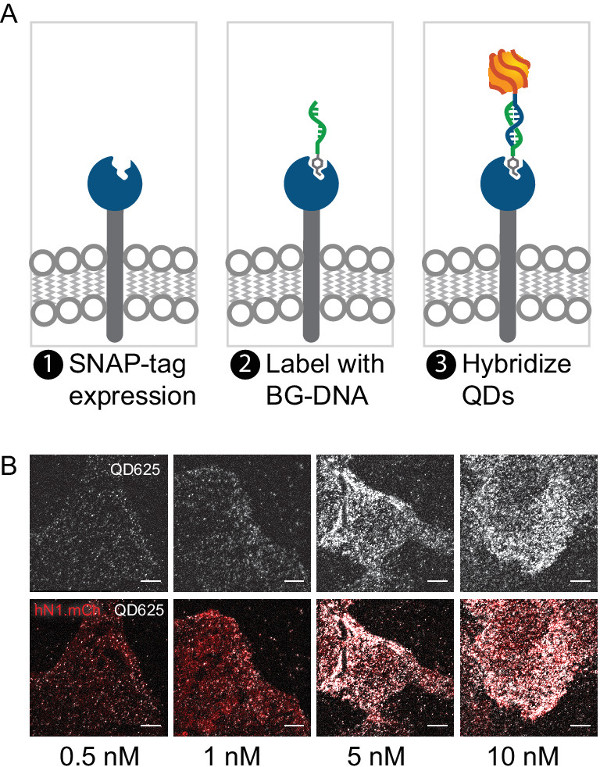
Figura 4. Etiquetado proteínas SNAP-etiquetado en células vivas. (A) Esquema que demuestra la expresión, el apego y el etiquetado de un receptor de SNAP-etiquetado con un MQD orientada BG. (B) el etiquetado Representante de células que expresan un SNAP-Notch-mCherry construir en varias concentraciones MQD. MQDS pasivadas con PEG12 colocalize con mCherry indicando etiquetado específico. Densidades de etiquetado más bajos (<0,5 nM) en general es preferible para el seguimiento de una sola partícula. La barra de escala es de 10 micras. Por favor haga clic aquí para ver una versión más grande de esta figura.
Discusión
La modularidad del diseño MQD permite un mayor grado de flexibilidad experimental. Por ejemplo, una variedad de MQDS se puede preparar rápidamente en colores únicos que permiten la formación de imágenes simultánea de múltiples objetivos. La secuencia dirigida ssDNA puede dirigir MQDS a las proteínas, azúcares, lípidos 12 y las superficies 13. Una serie de etiquetas enzimáticas están disponibles con reactividades ortogonales, lo que permite múltiples objetivos para obtener imágenes de forma simultánea con MQDS diferencialmente dirigidas. Además de la orientación con la etiqueta SNAP, el etiquetado de proteínas diana con MQDS usando la etiqueta CLIP, la etiqueta HALO, y proteínas con biotina también tuvo éxito. Este protocolo demuestra el etiquetado específico de un receptor de superficie en células vivas con estos MQDS, pero el protocolo podría ser fácilmente adaptado a una serie de contextos diferentes.
La idea metodológica significativa en la producción de MQDS con valencia definido es que ~ vinculado-fosforotioato 50se requieren bases para envolver los puntos cuánticos (y por lo tanto evitar que dos moléculas de unión de forma simultánea). A-Un poli secuencia S 50 reproducible y estable vinculado 605 puntos cuánticos nm de Life Technologies. Aunque este producto ha sido descontinuado, la estrategia de exclusión estérica es generalizable a los productos similares de otros fabricantes que tengan diferentes tamaños, formas, propiedades espectrales.
La eficiencia y la estabilidad de los puntos cuánticos envueltos-ptDNA depende críticamente de la química de la superficie y la estructura de los puntos cuánticos. Por lo tanto, el éxito de un protocolo dependerá de la estructura química y fuente comercial de los puntos cuánticos. A los fines de este protocolo, existen tres principales puntos de diferencia entre varias fuentes comerciales de los puntos cuánticos: a diferencia de las condiciones necesarias para la transferencia de fase; una diferencia en la fuerza de, posiblemente obstaculizar el desplazamiento PEG-tiol-ligando de unión inicial por el ptDNA; y la diferencia en la cantidad de núcleo CdSe expuesta, que puede leanuncio a la extinción de la QD por las condiciones de transferencia de fase tiol MPEG.
A partir de la publicación, los puntos cuánticos con la mejor estructura para la producción de MQDS son los puntos cuánticos 4-10 nm CdSe / ZnS núcleo / corteza adquiridos de Life Technologies con espectros de emisión a 545, 585, 605 y 625 nm (Figura 2A). Puntos cuánticos basados en la formulación 'Vivid' (545, 605, etc) apagan tras la adición de mPEG tiol y no son adecuados para esta aplicación. Puntos cuánticos de Aldrich y Océano Nanotech funcionan bien, pero requieren pasos de transferencia de fase más larga y el tratamiento previo con óxido trioctilfosfina. Este protocolo ha sido optimizado para los puntos cuánticos de Life Technologies.
La secuencia poli-A fosforotioato utilizado para envolver los puntos cuánticos se termina con una cola de ADN 20-mer nativo que contiene la secuencia de (ACTG) 5 al que una hebra diana puede hibridar. Esta secuencia es conveniente, ya que tiene poca o ninguna estructura secundaria, y seguirá siendo hybridized a 37 ° C en PBS. Si se tiene acceso a un sintetizador de ADN, la ptDNA por se sintetiza y después se purificó por cromatografía líquida de alta resolución en fase inversa (HPLC) usando una columna C 8. El ptDNA se eluye más tarde en la HPLC de oligonucleótidos equivalentes con una columna vertebral nativa. Normalmente nos dejamos 5 'DMT grupo protector en nuestros oligonucleótidos fosforotioato después de la purificación.
La pasivación de los puntos cuánticos envueltos-ptDNA por lo general se requiere con el fin de mejorar la estabilidad coloidal de los puntos cuánticos y reducir la unión de fondo para las aplicaciones más experimentales. El protocolo utiliza un PEG-capa para pasivar los puntos cuánticos. Carboxi PEG alcano tiol con unidades de PEG adicional ((CO 2 H) CH 2 O (CH 2 CH 2 O) C 11 H 12 23 SH, carboxi-PEG12 alcano tiol) proporciona redujo significativamente fondo, aunque los PEG más largas son tanto más grande, y generalmente más caros. MQDS recubiertas con PEG carboxi Alkaligandos tiol ne son muy estables en tampones fisiológicos tales como soluciones salinas tamponadas con fosfato y medios de cultivo. El almacenamiento a largo plazo (> 8 meses) de MQDS a 4 ° C no mostró agregación significativa o ptDNA desprendimiento 11. Dependiendo del experimento, PEG pasivación de los puntos cuánticos por sí sola no siempre suficientemente reducir la unión no específica de los MQDS. La incubación de ambas células y MQDS en tampón fosfato salino (PBS) que contenía 3% de BSA durante 20 min antes de su uso reduce sustancialmente la unión no específica a las células, aunque sí aumenta el radio hidrodinámico aparente de los MQDS por ~ 50%. La pasivación con 0,5% de caseína reduce la unión no específica aún más pero aumenta el tamaño aparente a un mayor grado que BSA.
El extremo 5 'de una hebra de ADN complementaria a las MQDS puede ser modificado para permitir la orientación de un número de diferentes biomoléculas. Hay una serie de técnicas establecidos disponibles para modificar covalentemente las proteínas, lipids y azúcares con ADN de cadena sencilla (ssDNA). En tanto que el ssDNA se presenta extracelularmente, es accesible a MQDS solubles. MQDS con las secuencias anteriores se hibridarán rápidamente con su cadena de ADN complementaria en condiciones de cultivo celular. Un 10-20x poli (CT) espaciador entre el ptDNA y la secuencia diana puede ser necesaria para una eficiente orientación a fin de elevar la secuencia de unión por encima de la glycocalyx de espesor y con carga negativa de la célula. Para este protocolo elegimos para producir un ADN-BG con una secuencia complementaria de (CAGT) 5 que a la vez hibridado a los MQDS y ligarse covalentemente a una proteína en sí SNAP-tag para el etiquetado rápida y específica. A las funciones de protocolo similares bien para acoplar otros ésteres de NHS a oligonucleótidos modificados con amino.
Para imágenes de una sola molécula, una baja densidad de etiquetado se requiere típicamente para resolver moléculas individuales. Una concentración final del MQD ~ 0,5 nM en PBS con el agente de pasivaciónes un buen objetivo. Sin embargo, estas altas diluciones veces resultaron en menos de etiquetado de las células. Si esto ocurre, MQD adicional se puede añadir hasta que se observa una densidad óptima de etiquetado. En el caso de etiquetado-SNAP Notch1 humano, las concentraciones de> 10 nM MQD producen células de etiquetado densos, mientras que 0,5 nM MQD resultó en la unión de ~ 20 MQDS en la superficie basal de la célula diana (ver Figura 4B). Etiquetado celular con MQDS fue altamente dependiente de la confluencia de las células sembradas. Demasiado células confluentes no etiquetan en sus superficies basales.
En resumen, un método simple para generar monovalente y puntos cuánticos modulares se describió. Estos MQDS encuentran utilidad en una amplia gama de aplicaciones de imágenes de células vivas, como se demuestra por formación de imágenes del receptor Notch en células U2OS en vivo. Aplicabilidad de MQDS no se limita a este caso específico, pero puede ser potencialmente extenderse por otras dianas celulares tales como otras proteínas, ácidos nucleicos y enzimas.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
Financiamiento proporcionado por el Departamento de Defensa W81XWH-10/01/1023 (ZJG), P50 GM081879 subvención del Centro de UCSF para Sistemas y Biología Sintética (ZJG), NIH 5R21EB015088-02 (YJ) y NIH 1R21EB018044 (ZJG y YJ). DS fue apoyado por Human Frontier Ciencia Programa cruzada postdoc disciplinaria beca de investigación.
Materiales
| Name | Company | Catalog Number | Comments |
| mQD Production | |||
| Phosphorothioate DNA: | |||
| (A*)x50-(ACTG)x5 | IDT | Most DNA Synthesis companies | |
| Quantum Dots: | |||
| QDots 525, 585, 605 & 625 | Invitrogen | Q21791MP (545), Q21711MP (585), Q21701MP (605) | Custom QD synthesis for QD625. |
| QD610 | Ocean Nanotech | QSP-610-10 | |
| QD 610 lamda | Aldrich | 731854 | |
| Chloroform, 99.8% | ACROS | 67-66-3 | |
| Tetrabutylammonium bromide, 98.0% | Sigma-Aldrich | 426288 | |
| mPEG thiol [2,5,8,11,14,17,20-Heptaoxadocosane-22-thiol], MW 354.5, 95% | Polypure | 11156-0695 | |
| HSC11EG6CO2H [HS-(CH2)11-(OCH2CH2)6-OCH2CO2H] | ProChimia | TH 003-m11.n6-0.1 | |
| Boric Acid, 99.5% | Sigma-Aldrich | B0394 | |
| Sodium Hydroxide, 99.0% | ACROS | S/4845 | |
| Sodium Chloride, 98% | Sigma-Aldrich | 310166 | |
| Agarose LE | U.S. Biotech Sources | G02PD-125 | |
| Ethanol | Sigma-Aldrich | 459828 | |
| BG-DNA Production | |||
| Amine DNA: | |||
| (CAGT)5-NH2 | IDT | N/A | Most DNA Synthesis companies |
| (CAGT)5(T)40-NH2 | IDT | N/A | Most DNA Synthesis companies |
| NHS-GLA-Benzylguanine | New England Biosciences | S9151 | |
| DMSO | Sigma-Aldrich | D8428 | |
| HEPES Buffer | Sigma-Aldrich | 83264 | |
| NAP5 Column | GE Healthcare | 17-0853-01 | |
| C18 Column | |||
| Acetonitrile | |||
| Mammalian Cell Culture & Imaging | |||
| Cell line expressing SNAP-tagged protein | New England Biosciences | E9100S | |
| McCoys 5A | UCSF Cell Culture Facility | Specific to U2OS culture | |
| Fetal Bovine Serum | UCSF Cell Culture Facility | Specific to U2OS culture | |
| PBS | UCSF Cell Culture Facility | ||
| Nunc Lab-tek II Chambered Coverglass | Thermo-Fischer Scientific | 155409 | |
| Matriplate 96-well plate | Brooks Life Science Systems | MGB096-1-2-LG-L | |
| BSA | |||
| 5% Alkali-soluble Casein | EMD Millipore | 70955 | Not all caseins are the same |
| (Optional) DNA Synthesis Reagents | |||
| 5’ Amine modifier C6 | Glen Research | Oct-06 | |
| dA-Thiophosphoramidite | Glen Research | 10-700 | |
| Analysis Software | |||
| FIJI | http://valelab.ucsf.edu/~schindelin/ | ||
| RyTrack.pro | http://sun.iwu.edu/~gspaldin/rytrack.html | ||
| Tracker | http://www.gartnerlab.ucsf.edu/more/software | ||
Referencias
- Sako, Y., Minoghchi, S., Yanagida, T. Single-molecule imaging of EGFR signaling on the surface of living cells. Nature Cell Biology. 2 (3), 168-172 (2000).
- Luo, W., He, K., Xia, T., Fang, X. Single-molecule monitoring in living cells by use of fluorescence microscopy. Analytical and Bioanalytical Chemistry. 405 (1), 43-49 (2013).
- Chung, I., et al. Spatial control of EGF receptor activation by reversible dimerization on living cells. Nature. 464 (7289), 783-787 (2010).
- Fernández-Suárez, M., Ting, A. Fluorescent probes for super-resolution imaging in living cells. Nature Reviews Molecular Cell Biology. 9 (12), 929-943 (2008).
- Sark, W. G. J. H. M., Frederix, P. L. T. M., Vanden Heuvel, D. J., Gerritsen, H. C. Photooxidation and photobleaching of single CdSe/ZnS quantum dots probed by room-temperature time-resolved spectroscopy. The Journal of Physical Chemistry B. 105 (35), 8281-8284 (2001).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Michalet, X., et al. Quantum dots for live cells, in vivo imaging, and diagnostics. Science. 307 (5709), 538-544 (2005).
- Howarth, M., et al. Monovalent, reduced-size quantum dots for imaging receptors on living cells. Nature Methods. 5 (5), 397-399 (2008).
- Iyer, G., et al. Aromatic aldehyde and hydrazine activated peptide coated quantum dots for easy bioconjugation and live cell imaging. Bioconjugate Chemistry. 22 (6), 1006-1011 (2011).
- Tikhomirov, G., et al. DNA-based programming of quantum dot valency, self-assembly and luminescence. Nature Nanotechnology. 6 (8), 485-490 (2011).
- Farlow, J., Seo, D., Broaders, K. E., Taylor, M. J., Gartner, Z. J., Jun, Y. W. Formation of targeted monovalent quantum dots by steric exclusion. Nature Methods. 10 (12), 1203-1205 (2013).
- Gartner, Z. J., Bertozzi, C. R. Programmed assembly of 3-dimensional microtissues with defined cellular connectivity. Proceeding of the National Academy of Sciences USA. 106 (12), 4606-4610 (2009).
- Selden, N. S., et al. Chemically programmed cell adhesion with membrane-anchored oligonucleotides. Journal of the American Chemical Society. 134 (2), 765-768 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados