Method Article
Instantánea de todo el genoma de los Reguladores de la cromatina y los Estados en
En este artículo
Resumen
La cuestión de cómo los reguladores y los estados de la cromatina cromatina afecta al genoma in vivo es fundamental para nuestra comprensión de cómo los primeros se toman las decisiones del destino celular en el embrión en desarrollo. Chip-Sec-el método más popular para investigar las características de la cromatina en un nivel se describe aquí mundial para los embriones de Xenopus.
Resumen
The recruitment of chromatin regulators and the assignment of chromatin states to specific genomic loci are pivotal to cell fate decisions and tissue and organ formation during development. Determining the locations and levels of such chromatin features in vivo will provide valuable information about the spatio-temporal regulation of genomic elements, and will support aspirations to mimic embryonic tissue development in vitro. The most commonly used method for genome-wide and high-resolution profiling is chromatin immunoprecipitation followed by next-generation sequencing (ChIP-Seq). This protocol outlines how yolk-rich embryos such as those of the frog Xenopus can be processed for ChIP-Seq experiments, and it offers simple command lines for post-sequencing analysis. Because of the high efficiency with which the protocol extracts nuclei from formaldehyde-fixed tissue, the method allows easy upscaling to obtain enough ChIP material for genome-wide profiling. Our protocol has been used successfully to map various DNA-binding proteins such as transcription factors, signaling mediators, components of the transcription machinery, chromatin modifiers and post-translational histone modifications, and for this to be done at various stages of embryogenesis. Lastly, this protocol should be widely applicable to other model and non-model organisms as more and more genome assemblies become available.
Introducción
The first attempts to characterize protein-DNA interactions in vivo were reported about 30 years ago in an effort to understand RNA polymerase-mediated gene transcription in bacteria and in the fruit fly1,2. Since then, the use of immunoprecipitation to enrich distinct chromatin features (ChIP) has been widely adopted to capture binding events and chromatin states with high efficiency3. Subsequently, with the emergence of powerful microarray technologies, this method led to the characterization of genome-wide chromatin landscapes4. More recently, chromatin profiling has become even more comprehensive and high-resolution, because millions of co-immunoprecipitated DNA templates can now be sequenced in parallel and mapped to the genome (ChIP-Seq)5. As increasing numbers of genome assemblies are available, ChIP-Seq is an attractive approach to learn more about the genome regulation that underlies biological processes.
Here we provide a protocol to perform ChIP-Seq on yolk-rich embryos such as those of the frog Xenopus. Drafts of the genomes of both widely used Xenopus species—X. tropicalis and X. laevis—have now been released by the International Xenopus Genome Consortium6. The embryos of Xenopus species share many desirable features that facilitate and allow the interpretation of genome-wide chromatin studies, including the production of large numbers of high-quality embryos, the large size of the embryos themselves, and their external development. In addition, the embryos are amenable to classic and novel manipulations like cell lineage tracing, whole-mount in situ hybridisation, RNA overexpression, and TALEN/CRISPR-mediated knockout technology.
The following protocol builds on the work of Lee et al., Blythe et al. and Gentsch et al.7-9. Briefly, Xenopus embryos are formaldehyde-fixed at the developmental stage of interest to covalently bind (cross-link) proteins to their associated genomic DNA. After nuclear extraction, cross-linked chromatin is fragmented to focus subsequent sequencing on specific genomic binding or modification sites, and to minimize the contributions of flanking DNA sequences. Subsequently, the chromatin fragments are immunoprecipitated with a ChIP-grade antibody to enrich those containing the protein of interest. The co-immunoprecipitated DNA is stripped from the protein and purified before creating an indexed (paired-end) library for next-generation sequencing (NGS). At the end, simple command lines are offered for the post-sequencing analysis of ChIP-Seq data.
Protocolo
NOTA: Todos los trabajos de Xenopus se ajusta plenamente a la Ley de 1986 como ejecutado por el Instituto Nacional para la Investigación Médica MRC Animales del Reino Unido (Procedimientos Científicos).
1. Preparativos
- Estimar el número de embriones necesarios para el experimento de chip (véase el debate).
- Preparar las siguientes soluciones que se almacenan a temperatura ambiente: 500 ml de 10x de Marc Ringers Modificado (MMR) sin EDTA, pH ajustado a 7,5 y se esterilizó por tratamiento en autoclave (1 M NaCl, 20 mM KCl, 20 mM CaCl2, 10 mM MgSO 4, 50 mM HEPES pH 7,5) 10, 1 ml de tampón de elución SDS (50 mM Tris-HCl pH 8,0, EDTA 1 mM, 1% SDS) y 1 ml de tampón de carga de ADN 5x (0,2% de Orange G, 30% de glicerol, 60 EDTA mM pH 8,0).
- Preparar las siguientes soluciones que se almacenan a 4 ° C: 50 ml de tampón de HEG (50 mM HEPES-KOH pH 7,5, 1 mM EDTA pH 8,0, 20% de glicerol), 500 ml de tampones de extracción E1 (50 mM HEPES-KOH pH 7,5, 150 mM NaCl, 1 mM EDTA. 10% de glicerol, 0,5% de Igepal CA-630, 0,25% de Triton X-100), E2 (10 mM Tris-HCl pH 8,0, NaCl 150 mM, EDTA 1 mM, 0,5 mM EGTA) y E3 (mM Tris-HCl 10 pH 8,0, NaCl 150 mM, EDTA 1 mM, 1% de Igepal CA-630, 0,25% de Na-desoxicolato, 0,1% SDS), 500 ml de tampón RIPA (50 mM HEPES-KOH pH 7,5, LiCl 500 mM, EDTA 1 mM , 1% de Igepal CA-630, 0,7% de Na-desoxicolato) y 50 ml de tampón TEN (10 mM Tris-HCl pH 8,0, EDTA 1 mM, NaCl 150 mM).
- Partitura y recortar unos 15 ml tubo de poliestireno cónico en la marca de 7 ml. Utilice este tubo para contener extractos nucleares sometidos a sonicación.
- Para el análisis posterior a la secuenciación, utilice un ordenador multinúcleo operativo estilo Unix con al menos 8 GB de RAM y 500 GB de espacio libre en disco. Instale el siguiente software localmente de los cuales la mayoría se utilizan en la línea de comandos: FastQC, Illumina Casava-1.8 filtro de calidad, Bowtie 11, SAMtools 12, HOMER 13, MACS2 14, IGV 15,16, Cluster3 17, Java TreeView, BLAST + 18, y b2g4pipe 19. Revise las instrucciones de instalación y los requisitos para los compiladores y software de terceros.
- Construir un índice de Bowtie para alinear corto NGS lee al genoma Xenopus. Un ejemplo se muestra aquí por la X. tropicalis v7.1 genoma (noviembre de 2011), que puede ser descargado como un archivo FASTA (genome.fa) desde el servidor ftp Xenbase (/ pub / Genómica / JGI). Mueva el archivo FASTA al subdirectorio índice de Bowtie.
- Utilice la siguiente línea de comandos (en este caso después de que el personaje prompt>) para generar archivos de índice xenTro7:
> Pajarita-build /path/to/bowtie/index/genome.fa xenTro7
> Exportación BOWTIE_INDEXES = / ruta / a / pajarita / index /
- Utilice la siguiente línea de comandos (en este caso después de que el personaje prompt>) para generar archivos de índice xenTro7:
- Descargue el archivo de anotación de genes (GTF) a partir de la UCSC Genome Browser o el sitio espejo en el servidor RMIN de las versiones más recientes del genoma (genomes.nimr.mrc.ac.uk) a través de Herramientas / Explorador de tablas. Utilice el archivo FASTA genoma y el archivo GTF de personalizar HOMER de Xenopus (por ejemplo, X. tropicalis genoma v7.1, - nombre xenTro7).
- Como alternativa, utilice los paquetes HOMER pre-construidos para algunas versiones antiguas del genoma Xenopus.
> LoadGenome.pl -name xenTro7 -org nula -fasta /path/to/genome.fa -gtf ruta / a / genes.gtf
- Como alternativa, utilice los paquetes HOMER pre-construidos para algunas versiones antiguas del genoma Xenopus.
- Crear una pista genoma (.genome archivo) para el navegador genoma IGV por subir un archivo FASTA indexada (genome.fa con genome.fa.fai archivo en la misma carpeta) y el archivo de anotación (genes.gtf). Crear un índice de andamio genoma (genome.fa.fai) para el genoma Xenopus como sigue:
> Samtools faidx /path/to/genome.fa - Utilice BLAST + para pasar Gene Ontología (GO) términos de varias especies modelo (humano, ratón, pez cebra, mosca de la fruta y de la levadura) sobre genes Xenopus de la siguiente manera:
- Descarga todas las secuencias codificadoras (CDS) como un solo archivo FASTA (cds.fa) de la UCSC Genome Browser a través de Herramientas/ Explorador de tablas y actualización BLAST + con la base de datos BLAST preformateado de proteínas no redundante (nr):
> Update_blastdb.pl nr - Buscar humana (txid9606), ratón proteínas de la NCBI sitio a través de su función de búsqueda avanzada (txid10090), el pez cebra (txid7955), mosca de la fruta (txid7227) y levadura (txid4932) (http: //www.ncbi.nlm.nih. gov / proteína / avanzado) y enviar la lista GI resultante (identificador de secuencia) (sequence.gi.txt) al ordenador.
- Asignar genes Xenopus a las proteínas más similares de la lista GI ejecutando BLASTx con cierta esperar (E) valor de corte (aquí 10 -20). Asegúrese de que el formato de salida es xml (-outfmt 5 salida privado blastx_results.xml). Hacer uso de hilos de ahorro de tiempo (-num_threads) que se corresponden con el número de núcleos de computación disponibles.
> Blastx -db / path / to / nr -gilist /path/to/sequence.gi.txt -query / path / to / cds.fa -evalue 1e-20
-outfmt 5 salida privado /path/to/blastx_results.xml -num_threads [# hilos] - Abra el archivo b2gPipe.properties de la carpeta b2g4pipe con un editor de texto y actualizar las propiedades de base de datos a Dbacces.dbname = b2go_sep13 y Dbacces.dbhost = publicdb.blast2go.com. B2g4pipe Ejecutar desde la carpeta de instalación.
> Java -cp Xmx1000m *: ext / *: es.blast2go.prog.B2GAnnotPipe -en /path/to/blastx_results.xml
Resultados salida privado / xenTro7 -prop b2gPipe.properties -v -annot
NOTA: extractos Este programa GO términos para cada BLAST golpeado y les asigna a los genes de Xenopus correspondientes (xenTro7.annot). La configuración de bases de datos más actualizados se pueden encontrar en Herramientas / Ajustes generales / Ajustes DataAccess de la aplicación Blast2GO Java Web Start (ver 9.11.1).
- Descarga todas las secuencias codificadoras (CDS) como un solo archivo FASTA (cds.fa) de la UCSC Genome Browser a través de Herramientas/ Explorador de tablas y actualización BLAST + con la base de datos BLAST preformateado de proteínas no redundante (nr):
2. La cromatina reticulación
- Fertilizar los huevos de Xenopus, de-la jalea y la cultura embryos acuerdo con protocolos estándar 20.
- La transferencia de los embriones dejellied (máximo 2500 X. laevis o 10.000 X. tropicalis) en la etapa de desarrollo de interés para un niño de 8 ml de muestra de vidrio frasco con tapa y lávelos brevemente una vez con MMR 0,01x.
- Fijar los embriones con 1% de formaldehído en 0,01x MMR (por ejemplo, añadir 225 l de 36,5 a 38% de formaldehído a 8 ml MMR 0,01x) para 15 a 40 min a RT (ver Discusión para el tiempo de fijación y el número de embriones requerido por experimento CHIP).
NOTA: El formaldehído es corrosivo y altamente tóxico. Es peligroso en caso de contacto con ojos y piel, la indigestión, y la inhalación. Utilice la campana de humos cuando se añade formaldehído al vial. - Detener la fijación lavando brevemente los embriones tres veces con MMR 0,01x frío. No deje que os embry hacen contacto con la superficie del líquido, porque la tensión superficial hace que se rompan.
- Alícuota de los embriones en 2 ml tubos de microcentrífuga en hielocon un máximo de 250 embriones por tubo, que ocupan un volumen de aproximadamente 250 l (X. tropicalis) o 600 l (X. laevis) antes de la eclosión.
- Pipeta lejos tanto MMR 0,01x posible. Omita el siguiente paso si continúa inmediatamente con la sección 3.
- Equilibrar embriones en 250 l de tampón HEG frío. Una vez que los embriones han depositado en el fondo del tubo de eliminar la mayor cantidad de líquido posible y snap-congelado en nitrógeno líquido. Almacenar a -80 ° C.
3. La cromatina Extracción
NOTA: La siguiente extracción de cromatina reticulada a partir de embriones de Xenopus funciona más eficientemente con los tiempos de fijación indicadas en el paso 2.3 y 50 a 80 X. tropicalis o 25 a 40 X. laevis embriones por ml de tampón de extracción E1, E2 y E3. Cada etapa de extracción se repite, de manera que se requerirá el doble del volumen calculado de tampón. Para escalado, use múltiples 2 ml microcentritubos FUGE o 50 ml tubos de centrífuga. Mantener las muestras y tampones en hielo durante la extracción de la cromatina.
- Suplemento volúmenes adecuados de tampones E1, E2 y E3 con DTT 1 mM y comprimidos de inhibidor de proteasa. Si la realización de chip con un fosfato de anticuerpos específicos, más buffers de suplementos con 5 mM NaF y 2 mM Na 3 VO 4.
- Homogeneizar embriones fijos con E1 pipeta hacia arriba y hacia abajo. Centrifugar homogeneizados en una centrífuga refrigerada (4 ° C) a 1000 xg durante 2 min (o 5 min en el caso de utilizar tubos de 50 ml). Aspirar el sobrenadante y cualquier lípidos unidos a la pared.
- Resuspender pellet en E1. Mantener las muestras en hielo durante 10 min. Centrifugar y descarte sobrenadantes como en el paso 3.2.
- Resuspender pellet en E2. Centrifugar y descarte sobrenadantes como en el paso 3.2.
- Repita el paso 3.4, pero mantener las muestras en hielo durante 10 minutos antes de la centrifugación.
- Resuspender pellet en el E3. Mantener las muestras en hielo durante al menos 10 minutos. Centrífuga y deseche supernatants como en el paso 3.2.
NOTA: En esta etapa, los resuspensiones deberían ser bastante transparente. Los detergentes aniónicos en E3 extraer núcleos reticulados por la prestación de la mayoría de las plaquetas yema restantes solubles. - Volver a suspender y piscina pellets de núcleos reticulados (normalmente de color marrón de los gránulos de pigmento insolubilizados) en un volumen total de 1 ml de E3. Diluir la muestra con E3 a 2 o 3 ml si aparece muy viscosa y es difícil de pipeta. Mantener en hielo oa 4 ° C para continuar con el paso 4 en el mismo o al día siguiente. Snap-congelación en nitrógeno líquido y se almacena a -80 ° C para su uso posterior.
4. La cromatina Fragmentación
NOTA: La sonicación se utiliza tanto para solubilizar y para cizallar la cromatina reticulada. Éstos son los parámetros para ejecutar el Misonix Sonicator 3000 equipado con un microtip cónico 1/16 pulgadas y sonido envolvente. Si se utilizan otras sonicadores, siga las recomendaciones del fabricante para esquilanreticulado cromatina o utilizar de 6 a 12 W durante 4 a 8 min en total.
- Transferir la muestra nuclear desde el paso 3.7 en un tubo hecho a la medida para el tratamiento con ultrasonidos (paso 1.4). Mantenga la muestra refrigerada durante la sonicación por tener el tubo conectado a un recipiente de plástico de 800 ml llena de agua de hielo a través de una abrazadera termómetro corto.
- Colocar el vaso de precipitados sobre un gato de laboratorio. Ajuste el conector de manera que la micropunta sonicador se sumerge en la muestra a aproximadamente dos tercios de la profundidad de volumen y centrada sin tocar la pared del tubo.
- Sonicar la muestra durante 7 min en total, interrumpido cada 30 segundos con pausas de 1 min. Ajuste de potencia a 1,0. Iniciar el tratamiento con ultrasonidos y aumentar inmediatamente el ajuste de potencia (normalmente de 2 a 4) para llegar a una lectura de 9 a 12 W. Pausa de inmediato si la muestra comienza a espumar. Vuelva a colocar el tubo y se reiniciará cuando la espuma ha desaparecido por completo.
- Transferir la cromatina esquilada en tubos de 1,5 ml preenfriados microcentrífuga y vuelta a toda velocidad (> 15,000 xg) durante 5 min a 4 ° C.
- Transferir el sobrenadante a pre-refrigerada 1,5 ml tubos de microcentrífuga. Recoge 50 l del sobrenadante (idealmente contiene la cromatina de alrededor de 400.000 o más núcleos) para visualizar el grado de fragmentación de la cromatina (sección 5). Utilice el resto del sobrenadante para Chip (artículo 6).
- Almacene las muestras a 4 ° C para un máximo de un día. Muestras Snap-congelantes como alícuotas (uno por cada chip experimento) en nitrógeno líquido para almacenamiento a largo plazo a -80 ° C.
5. Imaging cromatina Fragmentación
- Añadir 50 l de tampón de elución SDS, 4 l de NaCl 5 M y 1 l de proteinasa K (20 mg / l) a 50 l del sobrenadante de la etapa 4.6.
- Incubar durante 6 a 15 hr (O / N) en una hibridación horno a 65 ° C.
- Se purifica el ADN utilizando un kit comercial de purificación de PCR. Si es necesario, utilizar 3 M de acetato de sodio (pH 5,2) para ajustar el pH como se recomienda por el fabricante. Eluir laADN dos veces con 11 l de tampón de elución (Tris-HCl 10 mM pH 8,5).
- Añadir 0,4 l de RNasa A (20 g / l) y 5 l de tampón de carga de ADN 5x antes de ejecutar toda la muestra junto a un 100 pb y una escalera de ADN de 1 kb en un gel de agarosa al 1,4% mediante electroforesis. Para obtener resultados óptimos, gel mancha con una solución de tinción ácido nucleico seguro después de la electroforesis.
6. La cromatina Immunoprecipitation
NOTA: En esta sección, use baja retención de 1,5 ml tubos de microcentrífuga y al menos 1 ml de tampón indicada por tubo para lavar perlas magnéticas durante 5 min a 4 ° C. Antes de retirar el tampón de las perlas, dejar los tubos en la gradilla magnética durante 20 a 30 seg cada vez o hasta que la solución es clara.
- Transferencia de 10 y 30 l del sobrenadante (cromatina esquilada) de la etapa 4.6 a un nuevo tubo que se utilizará más adelante como muestra de entrada, que corresponde a aproximadamente el 1% de la cromatina total utilizado para la viruta. Almacenar a 4° C hasta que las muestras de chip están listos para revertir los enlaces cruzados.
- Transferir la cromatina restante a un nuevo tubo. Para los experimentos chip-qPCR que requieren un control de anticuerpos, distribuir volúmenes iguales de la cromatina a dos tubos.
- Añadir el anticuerpo chip-grado (o el control correspondiente anticuerpo) a la cromatina. Como una guía general, utilizar aproximadamente 1 mg de anticuerpo por un millón de células que expresan el epítopo de interés.
- Para estimar con mayor precisión la cantidad de anticuerpo requerida por experimento ChIP, ejecute el mismo chip con diversas cantidades de anticuerpo (por ejemplo, 0,25 g, 1 g y 2,5 g) y comparar el rendimiento en negativo y positivo loci de control por chip-qPCR (ver sección 10). Como un control de anticuerpos, utilizar suero normal de las mismas especies de isotipo y al animal huésped como el anticuerpo.
- Incubar en un rotador (10 rpm) O / N a 4 ° C.
- Lavar una cantidad adecuada de perlas magnéticas compatibles con anticuerpos una vez con E3 durante 5 min unat 4 ° C. Comprobar la especificación del fabricante para la capacidad de unión del anticuerpo de las perlas (por lo general de 5 a 20 l de perlas se une 1 mg de anticuerpo IgG).
- Añadir los granos lavados al anticuerpo cromatina pre-incubadas. Además incubar en un rotador (10 rpm) durante 4 hr.
- Lavar los granos de cuatro veces (chip-qPCR) o diez veces (chip-Seq) con tampón RIPA enfriado previamente, y luego una vez con tampón enfriado previamente TEN.
- Sólo llevar a cabo este paso si la realización de un experimento de chip Sec.
- Resuspender lava cuentas en 50 l de tampón TEN por tubo. Piscina todas las bolas de un solo chip experimento transfiriéndolos a un nuevo tubo. Utilice la gradilla magnética y refrigerado (4 ° C) centrifugación a 1000 xg para recoger los granos en la parte inferior del tubo. Deseche todo el líquido posible sin perturbar el sedimento de perlas.
- Material de Gaza chip las perlas resuspendiendo las perlas en 50 a 100 l de tampón de elución SDS y vortexing continuamente con un termomezclador (1000 rpm) durante 15 min a 65 ° C. Después de que centrifugar a toda velocidad (> 15.000 xg) durante 30 segundos. Transferir el sobrenadante (eluido CHIP) a un nuevo tubo.
- Repita el último paso y combinar los eluidos chip.
7. La cromatina Reverse Cross-linking y purificación de ADN
- Añadir suficiente tampón de elución SDS a la muestra de entrada (paso 6.1) para alcanzar el volumen de la muestra de chip, que es de 100 a 200 l (paso 6,10). Suplemento ambas muestras de chip y de entrada con 1/20 volumen de NaCl 5 M. Incubar las muestras de 6 a 15 hr (O / N) a 65 ° C en un horno de hibridación.
- Añadir 1 volumen de tampón TE y RNasa A en 200 mg / ml. Incubar durante 1 hora a 37 ° C.
- Añadir proteinasa K a 200 mg / ml. Incubar durante 2 a 4 horas a 55 ° C.
- Se purifica el ADN con fenol: cloroformo: alcohol isoamílico, seguido de extracción por precipitación con etanol como se describe previamente 9. Para chip-Sec, añadir 32l de tampón de elución (Tris-HCl 10, pH 8,5) para disolver el sedimento de ADN. Deja muestras en hielo durante 30 min para asegurar que el ADN se disuelve completamente.
NOTA: Commercial kits de purificación de PCR tienen la recuperación de ADN inferior, pero son más convenientes, y pueden ser utilizados para las muestras de chip-qPCR. - Para chip-Sec, determinar la concentración de 1 l de ADN chip y de entrada utilizando métodos basados en fluorometría. Siga las instrucciones del fabricante y asegúrese de que la concentración de ADN se encuentra dentro del rango de detección fiable del fluorómetro.
8. chip-Sec Biblioteca Construcción y Validación
NOTA: Los métodos actuales para la preparación de biblioteca de ADN permiten la construcción de las bibliotecas de alta complejidad para NGS 1 a 2 ng. A expensas de cierta complejidad, las bibliotecas se pueden hacer desde tan poco como 50 pg de ADN (véase la Tabla de específico Materiales / Equipo). Utilice la misma cantidad de ADN para los dos chip y biblioteca de entrada. En pocas palabras, a make indexados (final emparejado) bibliotecas-chip Sec, chip y ADN de entrada tienen que ser reparado en sus extremos, se ligó a adaptadores especiales (véase la Tabla de Materiales Específicos / Equipo), seleccionado por tamaño y amplificado por PCR.
- Siga las instrucciones del fabricante para hacer bibliotecas-chip Sec. Véase la discusión para más recomendaciones.
- Eluir cada biblioteca en 12 l de tampón de elución y determinar la concentración de 1 l de cada biblioteca de chip y de entrada utilizando un fluorómetro. Esperar concentraciones de 5 a 25 ng / l. Considere reducir el número de ciclos de PCR (menos de 18 ciclos) si las concentraciones son superiores a 25 ng / l.
NOTA: cuantificación precisa es clave para lograr resultados óptimos NGS. Las bibliotecas con concentraciones tan bajas como 1 ng / l después de 18 ciclos de PCR pueden ser secuenciados, pero con frecuencia son de menor complejidad. - Utilice 1 l de biblioteca para determinar la distribución del tamaño del fragmento y para comprobar si hay contaminación dímero adaptador (banda alrededor de 120 pb) por cbasado en la cadera electroforesis capilar. Repita la fase sólida de purificación de inmovilización reversible con una relación de perlas a la muestra de 1: 1 (en lugar de 1,6: 1) si la biblioteca contiene dímeros de adaptador.
- Realizar qPCR en loci control positivo y negativo validado (ver sección 10) para comprobar si las tendencias de enriquecimiento de ADN similares se observaron antes y después de la preparación de la biblioteca. Presentar control de calidad bibliotecas aprobados para la secuenciación.
9. Análisis Post-secuenciación y Visualización de Datos
NOTA: En la actualidad, NGS menudo se lleva a cabo por en la empresa o las instalaciones de secuenciación comerciales (véase el análisis de algunas pautas de NGS). La salida estándar son uno o varios archivos comprimidos con gzip FASTQ (*) .fastq.gz almacenar millones de secuenciación lee. Normalmente, multiplexado lee ya están separados de acuerdo con su índice y cada lectura contiene un identificador de secuencia y una puntuación de control de calidad (Phred + 33 para Illumina 1.8+) por cada base llamada. Este enfoque aquí es sólo una de tantas maneras de cómo analizar los datos NGS. Se alienta al lector a comprobar si alguna de las siguientes líneas de comando requieren cambios como este campo avanza rápidamente y las actualizaciones se están produciendo con regularidad.
- Concatenar archivos FASTQ comprimidos con gzip y comprobar la calidad de los datos de secuenciación mediante el script FastQC. Ejecutar esta y la mayoría de los comandos siguientes para ambos datos chip y la secuencia de entrada (Ejemplos muestran para CHIP) desde el terminal:
> Gato /path/to/*.fastq.gz> ChIP.fastq.gz
> Fastqc ChIP.fastq.gz
NOTA: Los datos en bruto de la secuenciación con éxito de una biblioteca-chip Sec alta complejidad deben pasar la mayor parte de las pruebas. Las fallas se originan principalmente de pobres carreras de secuenciación y artefactos experimentales, como la amplificación por PCR sesgada o contaminación adaptador. Se espera que un cierto grado de duplicación (redundancia) como redundantes lee puede representar de buena fe enriquecimiento de ADN 21. Sin embargo, una tarde puede restringir las etiquetas de lectura - el extremo 5 'o lee - a uno por par de bases para eliminar cualquier redundante lee sin afectar la sensibilidad de detección de picos (paso 9.4) 21. - Datos de secuenciación de Pre-proceso para eliminar la contaminación adaptador (homerTools recortar -3 ) que permite un desajuste (-mis 1). Utilice las primeras 20 bases de la (indexado) adaptador (5 'a 3') proximal al fragmento de ADN de interés tras la unión (que se muestra para el adaptador que aparece en la Tabla de Materiales Específicos / Equipo).
> Gzip -CD ChIP.fastq.gz | fastq_illumina_filter -vn> ChIP.fastq
> homerTools recortar -3 GATCGGAAGAGCACACGTCT -mis 1 -min 36 ChIP.fastq
NOTA: La eliminación de filtrado lee (-N) sólo es necesaria por defecto en archivos FASTQ generados por Illumina 1.8. Omita el comando fastq_illumina_filter (es decir,. '| Fastq_illumina_filter -vn')si una versión anterior a la 1.8 genera el identificador de secuencia. - Alinear pre-procesado lee al genoma de referencia (xenTro7) utilizando Bowtie. Lee sólo mantener asignada única (-m 1) con la configuración predeterminada, es decir, máximos dos desajustes en los primeros 28 bases y una Phred + 33 puntuación total de calidad de todas las desigualdades por lectura de máxima 70. Informe alineación en formato SAM (-S) . Aumentar el número de megabytes por hilo (--chunkmbs) si la memoria trozo se ha agotado:
> Pajarita -m 1 -S -p [# hilos] --chunkmbs [por ejemplo, 200] xenTro7 ChIP.fastq.trimmed> ChIP.sam
NOTA: Bowtie espera Phred + 33 puntuaciones de calidad de forma predeterminada. Incluya la opción - phred64-Quals si el archivo FASTQ se generó con Phred + 64 puntuaciones de calidad de Illumina mayores de 1,8. - Utilice dos comandos HOMER para transformar la alineación (SAM) de archivo en un archivo pez gordo (.bw):
> MakeTagDirectory chip / -single -tbp 1 ChIP.sam
> MakeUCSCfile chip /-bigWig / path / to / genome.fa.fai -fsize 1e20-norma 1E7 -o ChIP.bw
NOTA: La transformación requiere el índice de andamio (genome.fa.fai) del genoma de referencia (paso 1.8). Aquí el perfil se limita a una etiqueta por par de bases (-tbp 1) y normalizado a 10 millones de lee (1E7-norma). bigwig es uno de los formato preferido para visualizar dinámicamente perfiles de cromatina con un genoma navegador tales como IGV (paso 9,12). - Determinar la distribución de etiquetas (CHIP -d /) en puntos de referencia del genoma (por ejemplo, +/- 10 kb con 25 contenedores pb, -si ze 20.000 -hist 25), como el inicio de la transcripción (TSS, ejemplo que se muestra aquí) y terminación ( TTS) sitios. Ejecute el annotatePeaks.pl HOMER script de perl con anotaciones de Xenopus xenTro7 (paso 1.7):
> AnnotatePeaks.pl tss xenTro7 -size 20.000 -hist 25 -d chip /> ChIP_tagDensity.tss - Encuentra picos significativos de enriquecimiento de ADN entre el chip(-t ChIP.sam) y entrada (Input.sam -c) en la X. tropicalis genoma utilizando MACS2 con un FDR de corte 1% (-q 0,01) y fragmentos de ADN (después de tratamiento con ultrasonidos) de 200 pb (--bw = 200) para la construcción de modelos. Añadir la --broad bandera a esta línea de comandos si esperara una amplia distribución de la función de la cromatina de interés, como las marcas de las histonas o ARN polimerasa.
> Macs2 callpeak -t ChIP.sam -c -f Input.sam chip SAM -n -g 1.4376e9 -q 0,01 --bw = 200
NOTA: El tamaño efectivo de la X. tropicalis v7.1 genoma de montaje es de aproximadamente 1,437,600,000 pb (1.4376e9 -g). MACS2 genera un archivo CAMA (ChIP_peaks.bed) alistar picos con sus lugares del genoma. - Comparar varios perfiles de la cromatina en forma de un mapa de calor en clúster:
- Crear una matriz de distribución de etiqueta de directorios densidad etiqueta de interés (-d chip / other_ChIP /) en los picos MACS2 (por ejemplo, +/- 1 kb con 25 contenedores pb, -size 2000 -hist 25 -ghist):
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -size 2000 -hist 25 -ghist -d chip / other_ChIP /> ChIP.matrix - Utilice la interfaz gráfica de usuario de Cluster3 subir archivos ChIP.matrix y jerárquicamente agrupar estas densidades de etiquetas basado en la distancia mínima euclidiana al centroide más cercano. Abra el archivo CTD generada en Java TreeView para visualizar el agrupamiento.
- Crear una matriz de distribución de etiqueta de directorios densidad etiqueta de interés (-d chip / other_ChIP /) en los picos MACS2 (por ejemplo, +/- 1 kb con 25 contenedores pb, -size 2000 -hist 25 -ghist):
- Encuentra novela y motivos de unión previamente conocidos, que se enriquecen en las cumbres de pico +/- 100 pb (-size 200). Utilice annotatePeaks.pl mapear motivo de los sucesos y para trazar densidades motivos:
> FindMotifsGenome.pl ChIP_peaks.bed xenTro7 ChIP_motifs / -size 200 -p [# hilos]
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -m motif1.motif> ChIP_peaks.motif1
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -m motif1.motif -size 800 -hist 25> motif1.density
NOTA: El infe guión findMotifsGenome.plrs el enriquecimiento de la comparación de las secuencias seleccionadas al azar fondo genocéntrica. El motivo novela más enriquecido se guarda con motif1.motif en el formato de una posición de peso matriz. Se alienta al lector a corroborar estos resultados con otros métodos de novo motivo descubrimiento como cisFinder 22 y MEME 23. - Anotar picos mediante el cálculo de su distancia al gen más cercano y por la determinación de su recuento normalizado de lectura dentro de 400 bp ventanas (-size 400):
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -Tamaño 400 -d chip / input /> ChIP_peaks.genes - Resumir la salida usando el comando awk siguiente para enumerar el número (N), la ubicación y el recuento de lectura normalizada del individuo (R) y todos (LR) picos por gen (objetivo) más cercano.
> Awk 'begin {FS =' t '} $ 7> = -5,000 && $ 7 <= 1000
{N [$ 8] + = 1; R [$ 8] + = $ 9; LR [$ 8] = LR [$ 8] ', y# 39, $ 7 '(' $ 9 ')'} END {for (i en N)
{Print i ' t' N [i] ' t' R [i] ' t' substr (LR [i], 2)}} 'ChIP_peaks.genes> ChIP_peaks.summary
NOTA: El número después de $ se refiere al número de columna, que pueden necesitar modificación para el archivo ChIP_peaks.genes creado en el paso anterior. Este ejemplar guión filtra los picos más allá de 5 kb aguas arriba y 1 kb aguas abajo de la SAT. $ 7, $ 8 y $ 9 se refieren a la distancia a la SAT, la identificación de genes y el recuento normalizado lectura por pico, respectivamente. - Realizar el análisis de los términos de GO enriquecido entre genes diana de la siguiente manera:
- Inicie la interfaz gráfica de usuario de Blast2GO desde la línea de comandos a través de Java Web Start (javaws) 19.
> Javaws http://blast2go.com/webstart/blast2go1000.jnlp - Siga las instrucciones de los desarrolladores para cargar la anotaciónpresentar para los genes de Xenopus (xenTro7.annot) como generada en 1.9.4 y un archivo plano de genes diana identificados. Asegúrese de que los mismos identificadores de genes se utilizan en ambos archivos.
- Inicie la interfaz gráfica de usuario de Blast2GO desde la línea de comandos a través de Java Web Start (javaws) 19.
- Visualizar perfiles cromatina añadiendo pez gordo (ChIP.bw, Input.bw) y archivos CAMA (ChIP_peaks.bed) en IGV como pistas. Datos del complemento con RNA-Seq pistas si está disponible para el mismo nivel de desarrollo. Guardar los resultados como una sesión.
- Utilice la programación de plataformas R (www.r-project.org) o MATLAB para manipular más y visualizar datos como generada anteriormente. Alternativamente, trazar pequeños conjuntos de datos con Excel.
10. chip-qPCR para Pruebas chip y Confirmando chip-Sec
- Utilice la plataforma en línea Primer3 para diseñar cebadores que rodean aproximadamente 100 bp de ADN a 60 ° C (T m) para ambos loci de control (pico específico) y negativos positivos. Confirmar la especificidad del cebador usando el in silico Búsqueda PCR implementada en la UCSC Genome Browser.
- Crear una curva estándar de 8 puntos de diluciones de tres veces a partir de aproximadamente el 1% de entrada o utilizar el 2 - método ΔΔC (T) 8,24 para la cuantificación de enriquecimiento de ADN.
- Ejecutar PCR en tiempo real por triplicado técnicos para todas las muestras, es decir, chip, control y, en caso necesario, muestras de la curva estándar.
- Parcela ADN enriquecimiento como el porcentaje de ADN de entrada o como una proporción de ChIP frente muestra de control en ambos loci de control positivas y negativas.
Resultados
Se espera que los resultados equivalentes a los que aquí se presenta si el protocolo es bien ejecutado y el anticuerpo en uso es de calidad chip-grado (véase la discusión). Este protocolo permite la extracción de los núcleos de embriones de Xenopus de formaldehído-fijo y la esquila eficiente de la cromatina por sonicación (Figura 1A-C). Cromatina esquilada muestra una distribución asimétrica de los fragmentos de ADN que van principalmente de 100 a 1000 pb y alcanzando un máximo entre 300 y 500 pb (Figura 1C). Se requiere un mínimo de 50 pg de ADN immunoprecipitated de realizar con éxito una gama emparejado biblioteca-chip Sec indexados con insertos de ADN de tamaño similar (Figura 2A). La biblioteca debe ser en gran medida carente de dímeros de adaptador, que se pueden ver en el electroferograma aproximadamente a 120 pb.
Tras la secuenciación por síntesis, pre-procesado lee se asignan al genoma (Figura 2 B, C). En un experimento exitoso con X. embriones tropicalis, normalmente 50 a 70% de un solo extremo lee de 40 pb puede ser asignada única para el conjunto de genoma de v7.1 con como máximo dos desajustes. Mientras lee de entrada alinear bastante uniformemente en todo el genoma, la alineación de Chip lee los resultados en enriquecimientos específicos de cadena que flanquean la característica de la cromatina de interés. Esto es porque todos los fragmentos son secuenciados desde el extremo 5 '(Figura 2C) 25. La extensión de la alineación en la dirección de lectura a un tamaño medio fragmento produce perfiles exactos para las características individuales de la cromatina, tales como eventos de unión a factor de transcripción. Estas ocupaciones de ADN aparecen como picos cuando se visualizan en el IGV o cualquier otro genoma navegador compatible. Las personas que llaman pico como MACS se utilizan para determinar la localización de estos picos (Figura 3A). De esta manera decenas de miles de sitios de unión han sido determinados en el X. tropicalis genoma de factores de transcripción T-box como Vegt 26. Chip-qPCR expementos deben confirmar el enriquecimiento local de encontrado por chip-Sec (Figura 3B).
Experimentos-chip Sec permiten explorar las características de todo el genoma de las características de la cromatina. Por ejemplo, el cálculo de la distribución de lectura sobre los elementos genómicos, como inicio de la transcripción y sitios de terminación puede resaltar cualquier preferencia vinculantes espaciales alrededor de genes (Figura 3C). Del mismo modo, un mapa de calor de las distribuciones de lectura en lugares pico se utiliza para comparar las diferentes características de la cromatina en un genoma de gran escala (Figura 3D). Ciertos factores de transcripción se unen secuencia de ADN específica. De novo motivo de análisis de picos subyacentes de ADN genómico puede recuperar este tipo de información, incluyendo enriquecida con Co motivos de co-factores potenciales (Figura 3E). La gran mayoría de los genes diana mostrar ocupación de ADN a un menor en vez de mayor nivel (Figura 3F). Esta característica libre de escala parece ser bastante común entre los trfactores anscription y sugiere que sólo una pequeña fracción de los genes diana están regulados directamente con relevancia biológica 27,28. El análisis de los términos de GO enriquecido u otros atributos como la expresión diferencial de genes diana puede revelar más conocimientos sobre la función biológica de la función de la cromatina en el embrión de Xenopus (Figura 3G).
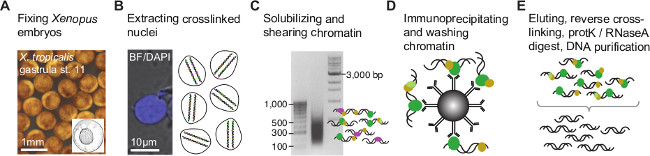
Figura 1. La cromatina procedimiento de inmunoprecipitación para los embriones de Xenopus. Embriones (A) son formaldehído-fijo en la etapa de desarrollo de interés para unen covalentemente (entrecruzamiento) cualquiera de las proteínas asociadas con el ADN genómico. Tras la extracción nuclear (B), la cromatina reticulada se fragmenta para reducir de unión al ADN genómico o sitios de modificación de la cromatina minimizando el DN flanqueandoUna secuencia (C). Posteriormente, los fragmentos de cromatina se inmunoprecipitaron con un anticuerpo chip-grado para enriquecer los que contienen el epítopo de interés (D). El ADN co-immunoprecipitated se despojó de la proteína y se purifica (E) antes de crear la biblioteca de fragmentos chip para NGS (Figura 2). Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Preparación de la biblioteca-chip Sec, la secuenciación por síntesis, la cartografía y el llamado pico. (A) El electroferograma muestra una buena biblioteca-chip Sec con moldes de ADN de 250 a 450 pb. Estas plantillas implican el inserto de ADN de interés flanqueado por lo universal (58 pb) y el (63 pb) adaptador de indexado. (B) Millones de grupos, con cada grupo que contiene plantillas idénticas, son de base secuenciados por base en presencia de los cuatro nucleótidos que poseen reversible, fluoróforo distinto y propiedades de terminación idénticos. Fluorescente imágenes se procesan en tiempo real para llamar correspondientes bases, que en última instancia se ensamblan en las lecturas. (C) Sólo lee ese mapa única para el genoma Xenopus se mantienen. Como todos los fragmentos se secuencian a partir del extremo 5 ', el mapeo de Chip lee los resultados en picos específicos de cadena que flanquean la función de la cromatina de interés. De este modo, las personas que llaman pico detectar el enriquecimiento que se origina de inmunoprecipitación y extender la lee a una longitud media fragmento de localizar con precisión las características de la cromatina. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Un ejemplo de análisis post-secuencia y la visualización de datos por medio del factor cigóticos T-box transcripción Vegt (zVegT). Todos los leídos recuentos mostrados aquí están normalizados a 10 millones asignan de forma única y lee no redundante. (A) Extracto del perfil de todo el genoma de zVegT unión en X. embriones tropicalis gástrula (etapa 11 a 12,5 después de Nieuwkoop y Faber 29). Cada pico, un choque en cadena de lecturas extendida, representa un sitio de unión. Estos picos son llamados por MACS2 con una tasa de falso descubrimiento (FDR) de menos de 1%. Cada gen MESP muestra zVegT muy proximal y aguas arriba de unión, pero sólo ENTHERMICS MEDICAL SYSTEMS y mespb se expresan en esa etapa (datos de RNA-Seq 30). (B) los niveles de ocupación de ADN de zVegT según lo determinado por chip-qPCR en varios loci (incluyendo un no región -bound 0,5 kb aguas arriba de β-actina) confirm el enriquecimiento específico encontrado por chip-Sec. Comparar los resultados de ENTHERMICS MEDICAL SYSTEMS con pico llamado (barra roja) en (A). El nivel de ocupación de ADN se visualiza como un porcentaje de entrada para ambos, el chip con el anticuerpo Vegt (policlonal de conejo de isotipo IgG) y el chip con el control de anticuerpo (IgG de conejo normal). Las barras de error representan la desviación típica de dos réplicas biológicas. (C) MetaGene análisis muestra zVegT unión preferencial (etiquetas binned más de 25 pb) al promotor con respecto a cualquier otra región genómica alrededor y dentro de los cuerpos de genes. (D) Heatmap muestra k-media agrupados niveles (k = 5) de ocupación de ADN (etiquetas binned más de 25 pb) de zVegT y Smad2 / Smad3 (datos-chip Sec 31) relativa a todas las regiones con destino a zVegT en la etapa de gástrula. El mapa de calor es log 2 basado y centrado a 5 etiquetas por pb. (E) De novo motivo de análisis descubre la canónica factor de transcripción T-box motivo de unión en el 38% de zVegT-regiones con destino si la puntuación subyacente motivo se normaliza a una tasa de descubrimiento del 5% en las secuencias de fondo. El mapa de densidad de muestra más alto enriquecimiento para el motivo de la caja en T en el centro de sitios de unión zVegT, mientras que el motivo de Smad2 / Smad3 unión canónica apenas se enriquece. (F) histograma muestra los niveles de ocupación de ADN de zVegT, que se calculan para cada gen diana de todos los picos (+/- 200 pb) entre 5 kb aguas arriba [-]. y 1 kb abajo [+] de los sitios de inicio de transcripción correspondiente (G) Top 300 genes con niveles más altos de ocupación de ADN dentro de -5 kb y 1 kb son enriquecida para los procesos biológicos del desarrollo embrionario temprano. Estos GO términos están en línea con la supuesta función de zVegT. El FDR se basa en una prueba exacta de Fisher de dos colas y corregido para múltiples pruebas. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Discusión
Nuestro protocolo describe cómo realizar y analizar los perfiles de la cromatina en todo el genoma a partir de embriones de Xenopus. Cubre cada paso de las proteínas de reticulación a loci endógenos in vivo para procesar millones de lecturas que representa sitios genómicas enriquecidas en silico. Dado el creciente número de borradores del genoma están disponibles, este protocolo debe ser aplicable a otros organismos modelo y no modelo. La sección experimental más importante, lo que diferencia a este protocolo aparte del trabajo previo 8,31,33,34, es el procedimiento posterior a la fijación para extraer núcleos reticulados. Se facilita la solubilización de la cromatina eficiente y de cizallamiento y fácil ampliación de la escala. Junto con la mejora de la eficiencia de la preparación de la biblioteca este protocolo permite la construcción de bibliotecas-chip Sec de alta complejidad de la mitad a dos millones de células que expresan el epítopo asociada a la cromatina de interés. Para los experimentos chip-qPCR, algunos diez mil de estas células son normalmente suficientepara comprobar para el enriquecimiento de ADN en unos seis loci genómicos diferentes. Estos números son estimaciones conservadoras, pero puede variar dependiendo del nivel de expresión de la proteína, la calidad del anticuerpo, la eficiencia, y la accesibilidad epítopo de reticulación. Como una guía, un solo embrión de Xenopus contiene alrededor de 4.000 células en la etapa de mediados de blástula (8,5 después de Nieuwkoop y Faber 29), 40.000 células en la última etapa de gástrula (12) y 100.000 células en la fase tailbud temprana (20).
La hora exacta de fijación para la inmunoprecipitación eficaz necesita ser determinado empíricamente por chip-qPCR (sección 10). En general, se requieren tiempos de fijación más largos si el experimento implica X. laevis embriones, primeras etapas de desarrollo, y propiedades de unión de ADN débiles (o indirectos). Sin embargo, no se recomienda la fijación de Xenopus embriones de más de 40 minutos, o el procesamiento de más embriones que los indicados (sección 3), cuya esquila cromatina se vuelve menos eficiente. Es importante noutilizar cualquier glicina después de la fijación ya que este paso común para saciar el formaldehído puede hacer extracción nuclear a partir de embriones de yema rica muy difíciles. Actualmente, no se conoce la razón de esto. Es concebible que el aducto de formaldehído glicina reacciona adicionalmente con grupos amino o residuos de arginina 35 N-terminal.
El anticuerpo es clave para cualquier experimento de chip y controles suficientes necesita ser llevado a cabo para mostrar su especificidad para el epítopo de interés (ver directrices por Landt et al. 36). Si ningún anticuerpo chip-grado se encuentra disponible, la introducción de proteínas de fusión etiquetadas con epítopo correspondiente puede ser una alternativa legítima como estas proteínas pueden ocupar sitios de unión 37 endógeno. En este caso, los embriones no inyectados son los mejores para usar como un control negativo en lugar de un chip con suero no específico. Esta estrategia también se puede aplicar si la proteína de interés se expresa en niveles bajos resultantes en el pobre recuperación de enriADN ched.
Como para la fabricación de bibliotecas-Chip SEQ, debido a la baja cantidad de ADN en uso, se recomienda optar por los procedimientos que reducen el número de pasos de limpieza y para reacciones combinan para mantener cualquier pérdida de ADN en un mínimo. Los adaptadores y cebadores tienen que ser compatibles con la secuenciación multiplex y la plataforma de NGS (véase la Tabla de Materiales Específicos / Equipo). Si se utiliza adaptadores Y (que contiene los brazos de una sola hebra de largo), es crítico para pre-amplificar la biblioteca con tres a cinco rondas de PCR antes de insertos de ADN de tamaño-seleccionar (por ejemplo., De 100 a 300 pb) mediante electroforesis en gel. Extremos de cadena simple causan fragmentos de ADN que migran de forma heterogénea. Trial se ejecuta con diferentes cantidades de ADN de entrada (por ejemplo, 0,1, 0,5, 1, 2, 5, 10 y 20 ng) se recomienda para determinar el número total de ciclos de PCR (menos de o igual a 18 ciclos) requerida para hacer un tamaño -seleccionado biblioteca de 100 a 200 ng. La reducción del número de ciclos de PCR hace que la secuenciación de redulee menos probable ndant. En fase sólida perlas de inmovilización reversibles son buena limpieza de reactivos para recuperar de manera eficiente el ADN de interés y fiable extraer cualquier adaptador y dímeros libres de la ligadura y reacciones de PCR.
En términos de número, tipo y duración de la lee, alrededor de 20 y 30 millones de fin de lecturas simples de 36 pb es suficiente para la mayoría de los experimentos-chip Sec para cubrir todo el genoma Xenopus con la profundidad suficiente. Las máquinas NGS más prevalentes son rutinariamente capaz de satisfacer estos criterios. Sin embargo, puede ser beneficioso para aumentar el número de lecturas, si se espera que un amplias distribuciones de lee, como se observa con las modificaciones de histonas, en lugar de picos agudos. Para muchos experimentos-chip Sec, 4-5 bibliotecas diferente indexados se unen y se secuenciaron en el carril celular uno de flujo usando una máquina de NGS alto rendimiento. A veces también es aconsejable extender la longitud de lectura y la secuencia de los dos extremos de la plantilla de ADN (de extremo emparejado) para aumentar mapeabilidad wanalizar gallina cromatina dentro de las regiones genómicas repetitivas.
Este protocolo se ha aplicado con éxito a una amplia variedad de características de la cromatina, tales como factores de transcripción, mediadores de señalización y las modificaciones de histonas post-traduccionales. Sin embargo, los embriones adquieren un creciente grado de heterogeneidad celular a medida que desarrollan y los perfiles de la cromatina se vuelven más difíciles de interpretar. Pasos prometedores se han hecho en Arabidopsis y Drosophila a los paisajes de la cromatina perfil específicamente mediante la extracción de tejido de tipo específico núcleos celulares 38,39. Nuestro protocolo incluye una etapa de extracción nuclear, que podría allanar el camino para que los tejidos específicos de chip-Sec en otros embriones.
Divulgaciones
The authors declare that they have no competing financial interests.
Agradecimientos
We thank Chris Benner for implementing the X. tropicalis genome (xenTro2, xenTro2r) into HOMER and the Gilchrist lab for discussions on post-sequencing analysis. I.P. assisted the GO term analysis. G.E.G and J.C.S. were supported by the Wellcome Trust and are now supported by the Medical Research Council (program number U117597140).
Materiales
| Name | Company | Catalog Number | Comments |
| 1/16 inch tapered microtip | Qsonica | 4417 | This microtip is compatible with Sonicator 3000 from Misonix and Q500/700 from Qsonica. |
| 8 ml glass sample vial with cap | Wheaton | 224884 | 8 ml clear glass sample vials for aqueous samples with 15-425 size phenolic rubber-lined screw caps. |
| Adaptor | e.g., IDT or Sigma | NA | TruSeq universal adaptor,
AATGATACGGCGACCACCGAG ATCTACACTCTTTCCCTACAC GACGCTCTTCCGATC*T. TruSeq indexed adaptor, P-GATCGGAAGAGCACACGTC TGAACTCCAGTCAC ‐NNNNNN‐ ATCTCGTATGCCGTCT TCTGCTT*G. *, phosphorothioate bondphosphate group at 5' end. NNNNNN, index (see TruSeq ChIP Sample Preparation Guide for DNA sequence). Order adaptors HPLC purified. Adaptors can be prepared by combining equimolar amounts (each 100 µM) of the universal and the indexed adaptor and cooling them down slowly from 95 °C to room temperature. Use 1.5 pmol per ng of input DNA. Store at -20 °C. |
| b2g4pipe (software) | Blast2GO | non-commercial | http://www.blast2go.com/data/blast2go/b2g4pipe_v2.5.zip |
| BLAST+ (software) | Camacho et al. | non-commercial | http://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE=BlastDocs& DOC_TYPE=Download |
| Bowtie (software) | Langmead et al. | non-commercial | http://bowtie-bio.sourceforge.net/index.shtml |
| cisFinder (software) | Sharov et al. | non-commercial | http://lgsun.grc.nia.nih.gov/CisFinder/ |
| Chip for capillary electrophoresis | Agilent Technologies | 5067-1504 | Load this chip with 1 µl DNA for library quality control. Store at 4 °C. |
| Chip-based capillary electrophoresis system | Agilent Technologies | G2940CA | The Agilent 2100 BioAnalyzer is used to check the quality of ChIP-Seq libraries. Keep reagents at 4 °C. |
| ChIP-Seq library preparation kit (KAPA Hyper Prep Kit) | Kapa Biosystems | KK8504 | Kit contains KAPA end repair and A-tailing enzyme mix, end Repair and A-tailing buffer, DNA ligase, ligation buffer, KAPA HiFi HotStart ReadyMix (2X), and KAPA library amplification primer mix (10X) (see also PCR primers). Adaptors are not included. Store at -20 °C. |
| ChIP-Seq library preparation kit (alternative, ThruPLEX-FD Prep Kit) | Rubicon Genomics | R40048 | Kit uses their own stem-loop adaptors and primers. This kit eliminates intermediate purification steps and is as sensitive as the KAPA Hyper Prep Kit. Store at -20 °C. |
| Cluster3 (software) | de Hoon et al. | non-commercial | http://bonsai.hgc.jp/~mdehoon/software/cluster |
| FastQC (software) | Simon Andrews | non-commercial | http://www.bioinformatics.babraham.ac.uk/projects/fastqc |
| Fluorometer | life technologies | Q32866 | Qubit 2.0 Fluorometer |
| Fluorometer reagents | life technologies | Q32851 | The kit provides concentrated assay reagent, dilution buffer, and pre-diluted DNA standards for the Qubit fluorometer. Store DNA standards at 4 °C, buffer and dye at room temperature. |
| Formaldehyde | Sigma | F8775-4X25ML | Formaldehyde solution, for molecular biology, 36.5-38% in H2O, stabilised with 10-15% methanol. Store at room temperature. CAUTION: Formaldehyde is corrosive and highly toxic. |
| Gel (E-Gel EX agarose , 2%) | life technologies | G4010 | Pre-cast gel with 11 wells, openable format. Leave one lane between ladder and library empty to avoid cross-contamination. Store gels at room temperature. |
| Gel electrophoresis system | life technologies | G6465 | E-Gel iBase and E-Gel Safe Imager combo kit for size-selecting ChIP-Seq libraries. |
| Gel extraction kit | Qiagen | 28706 | Store all reagents at room temperature. Use 500 µl of QG buffer per 100 mg of 2% agarose gel slice to extract DNA. Use MinElute columns (from MinElute PCR purification kit) to elute DNA twice. |
| HOMER (software) | Chris Benner | non-commercial | http://homer.salk.edu/homer/index.html |
| Hybridization oven | Techne | FHB1D | Hybridizer HB-1D |
| IGV (software) | Robinson et al. | non-commercial | http://www.broadinstitute.org/igv/home |
| Illumina CASAVA-1.8 quality filter (software) | Assaf Gordon | non-commercial | http://cancan.cshl.edu/labmembers/gordon/fastq_illumina_filter |
| Java TreeView (software) | Alok Saldanha | non-commercial | http://jtreeview.sourceforge.net |
| Laboratory jack | Edu-Lab | CH0642 | This jack is used to elevate sample in sound enclosure for sonication. |
| Ladder, 100 bp | New England BioLabs | N3231 | Keep 1x solution at room temperature. Store stock at -20 °C. |
| Ladder, 1 kb | New England BioLabs | N3232 | Keep 1x solution at room temperature. Store stock at -20 °C. |
| Low-retention 1.5-ml microcentrifuge tubes | life technologies | AM12450 | nonstick, RNase-free microfuge tubes, 1.5 ml |
| MACS2 (software) | Tao Liu | non-commercial | https://github.com/taoliu/MACS |
| Magnetic beads | life technologies | 11201D | These Dynabeads are superparamagnetic beads with affinity purified polyclonal sheep anti-mouse IgG covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 11203D | These Dynabeads are superparamagnetic beads with affinity purified polyclonal sheep anti-rabbit IgG covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 10001D | These Dynabeads are superparamagnetic beads with recombinant protein A covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 10003D | These Dynabeads are superparamagnetic beads with recombinant protein G covalently bound to the bead surface. Store at 4 °C. |
| Magnetic rack | life technologies | 12321D | DynaMag-2 magnet |
| MEME | Bailey et al. | non-commercial | http://meme.nbcr.net/meme/ |
| Na3VO4 | New England BioLabs | P0758 | Sodium orthovanadate (100 mM) is a commonly used general inhibitor for protein phosphotyrosyl phosphatases. Store at -20 °C. |
| NaF | New England BioLabs | P0759 | Sodium fluoride (500 mM) is commonly used as general inhibitor of phosphoseryl and phosphothreonyl phosphatases. Store at -20 °C. |
| NGS machine | Illumina | SY-301-1301 | Genome Analyzer IIx |
| NGS machine (high performance) | Illumina | SY-401-2501 | HiSeq |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2028 | Use as control for goat polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2025 | Use as control for mouse polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2027 | Use as control for rabbit polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Nucleic acid staining solution | iNtRON | 21141 | Use RedSafe nucleic acid staining solution at 1:50,000. Store at room temperature. |
| Orange G | Sigma | O3756-25G | 1-Phenylazo-2-naphthol-6,8-disulfonic acid disodium salt. Store at 4 °C. |
| PCR primers | e.g., IDT or Sigma | Primers to enrich adaptor-ligated DNA fragments by PCR: AATGATACGGCGACCACCGA*G and CAAGCAGAAGACGGCATACGA*G, phosphorothioate bond. Primers designed by Ethan Ford. Combine primers at 5 µM each. Use 5 µl in a 50 µl PCR reaction. Store at -20 °C. | |
| MinElute PCR purification kit | Qiagen | 28006 | for purification of ChIP-qPCR and shearing test samples. Store MinElute spin columns at 4 °C, all other buffers and collection tubes at room temperature. |
| Phenol:chloroform:isoamyl alcohol (25:24:1, pH 7.9) | life technologies | AM9730 | Phenol:Chloroform:IAA (25:24:1) is premixed and supplied at pH 6.6. Use provided Tris alkaline buffer to raise pH to 7.9. Store at 4 °C. CAUTION: phenol:chloroform:isoamyl alcohol is corrosive, highly toxic and combustible. |
| Primer3 (software) | Steve Rozen & Helen Skaletsky | non-commercial | http://biotools.umassmed.edu/bioapps/primer3_www.cgi |
| Protease inhibitor tablets | Roche | 11836170001 | cOmplete, Mini, EDTA-free. Use 1 tablet per 10 ml. Store at 4 °C. |
| Protease inhibitor tablets | Roche | 11873580001 | cOmplete, EDTA-free. Use 1 tablet per 50 ml. Store at 4 °C. |
| Proteinase K | life technologies | AM2548 | proteinase K solution (20 µg/µl). Store at -20 °C. |
| RNase A | life technologies | 12091-039 | RNase A (20 µg/µl). Store at room temperature. |
| Rotator | Stuart | SB3 | Rotator SB3 |
| SAMtools (software) | Li et al. | non-commercial | http://samtools.sourceforge.neta |
| Solid phase reversible immobilisation beads | Beckman Coulter | A63882 | The Agencourt AMPure XP beads are used to minimise adaptor dimer contamination in ChIP-Seq libraries. Store at 4 °C. |
| Sonicator 3000 | Misonix/Qsonica | Newer models are now available. Q125, Q500 or Q700 are all suitable for shearing crosslinked chromatin. | |
| Sound enclosure | Misonix/Qsonica | optional: follow the manufacturer's recommendation to obtain the correct sound enclosure. | |
| Thermomixer | eppendorf | 22670000 | Thermomixer for 24 x 1.5 mL tubes. Precise temperature control from 4 °C above room temperature to 99 °C. |
Referencias
- Gilmour, D. S., Lis, J. T. Detecting protein-DNA interactions in vivo: distribution of RNA polymerase on specific bacterial genes. Proc Natl Acad Sci U S A. 81 (14), 4275-4279 (1984).
- Gilmour, D. S., Lis, J. T. In vivo interactions of RNA polymerase II with genes of Drosophila melanogaster. Mol Cell Biol. 5 (8), 2009-2018 (1985).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Ren, B., et al. Genome-wide location and function of DNA binding proteins. Science. 290 (5500), 2306-2309 (2000).
- Johnson, D., Mortazavi, A., Myers, R., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Hellsten, U., et al. The genome of the Western clawed frog Xenopus tropicalis. Science. 328 (5978), 633-636 (2010).
- Lee, T. I., Johnstone, S. E., Young, R. A. Chromatin immunoprecipitation and microarray-based analysis of protein location. Nature Protocols. 1 (2), 729-748 (2006).
- Blythe, S. A., Reid, C. D., Kessler, D. S., Klein, P. S. Chromatin immunoprecipitation in early Xenopus laevis embryos. Dev Dyn. 238 (6), 1422-1432 (2009).
- Gentsch, G. E., Smith, J. C. Investigating physical chromatin associations across the Xenopus genome by chromatin immunoprecipitation. Cold Spring Harb Protoc. 2014 (5), (2014).
- Ubbels, G. A., Hara, K., Koster, C. H., Kirschner, M. W. Evidence for a functional role of the cytoskeleton in determination of the dorsoventral axis in Xenopus laevis eggs. J Embryol Exp Morphol. 77, 15-37 (1983).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Heinz, S., et al. Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities. Mol Cell. 38 (4), 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9 (9), R137 (2008).
- Robinson, J. T., et al. Integrative genomics viewer. Nat Biotechnol. 29 (1), 24-26 (2011).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Brief Bioinform. 14 (2), 178-192 (2013).
- Imoto, S., Nolan, J., Bioinformatics Miyano, S. . 20 (9), 1453-1454 (2004).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421 (2009).
- Conesa, A., et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Sive, H., Grainger, R., Harland, R. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Chen, Y., et al. Systematic evaluation of factors influencing ChIP-seq fidelity. Nat Methods. 9 (6), 609-614 (2012).
- Sharov, A. A., Ko, M. S. H. Exhaustive search for over-represented DNA sequence motifs with CisFinder. DNA Res. 16 (5), 261-273 (2009).
- Bailey, T. L., et al. MEME SUITE: tools for motif discovery and searching. Nucl Acids Res. 37 (2), W202-W208 (2009).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10 (10), 669-680 (2009).
- Gentsch, G. E., et al. In vivo T-box transcription factor profiling reveals joint regulation of embryonic neuromesodermal bipotency. Cell Rep. 4 (6), 1185-1196 (2013).
- Barabasi, A. L., Oltvai, Z. N. Network biology: understanding the cell's functional organization. Nat Rev Genet. 5 (2), 101-113 (2004).
- Biggin, M. D. Animal transcription networks as highly connected, quantitative continua. Dev Cell. 21 (4), 611-626 (2011).
- Nieuwkoop, P. D., Faber, J. . Normal table of Xenopus laevis (Daudin): a systematical and chronological survey of the development from the fertilized egg till the end of metamorphosis. , (1994).
- Akkers, R. C., et al. A hierarchy of H3K4me3 and H3K27me3 acquisition in spatial gene regulation in Xenopus embryos. Dev Cell. 17 (3), 425-434 (2009).
- Yoon, S. J., Wills, A. E., Chuong, E., Gupta, R., Baker, J. C. . HEB and E2A function as SMAD/FOXH1 cofactors. Genes Dev. 25 (15), 1654-1661 (2011).
- Jallow, Z., Jacobi, U. G., Weeks, D. L., Dawid, I. B., Veenstra, G. J. Specialized and redundant roles of TBP and a vertebrate-specific TBP paralog in embryonic gene regulation in Xenopus. Proc Natl Acad Sci U S A. 101 (37), 13525 (2004).
- Buchholz, D. R., Paul, B. D., Shi, Y. -. B. Gene-specific changes in promoter occupancy by thyroid hormone receptor during frog metamorphosis. Implications for developmental gene regulation. J Biol Chem. 280 (50), 41222-41228 (2005).
- Wills, A. E., Guptaa, R., Chuonga, E., Baker, J. C. Chromatin immunoprecipitation and deep sequencing in Xenopus tropicalis and Xenopus laevis. Methods. 66 (3), 410-421 (2014).
- Metz, B., et al. Identification of formaldehyde-induced modifications in proteins: reactions with model peptides. J Biol Chem. 279 (8), 6235-6243 (2004).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Res. 22 (9), 1813-1831 (2012).
- Mazzoni, E. O., et al. Embryonic stem cell-based mapping of developmental transcriptional programs. Nat Methods. 8 (12), 1056-1058 (2011).
- Deal, R. B., Henikoff, S. A simple method for gene expression and chromatin profiling of individual cell types within a tissue. Dev Cell. 18 (6), 1030-1040 (2010).
- Bonn, S., et al. Tissue-specific analysis of chromatin state identifies temporal signatures of enhancer activity during embryonic development. Nat Genet. 44 (2), 148-156 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados