Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Predicción de la silenciación génica a través del control espaciotemporal de la liberación de siRNA de Nanocarriers poliméricos fotosensibles
En este artículo
Resumen
Presentamos un nuevo método que utiliza copolímeros de bloques fotosensibles para un control espaciotemporal más eficiente del silenciamiento génico sin efectos detectables fuera del objetivo. Además, los cambios en la expresión génica se puede predecir utilizando sencillo siRNA liberación ensayos y modelado cinético simple.
Resumen
Se necesitan nuevos materiales y métodos para controlar mejor la unión frente a la liberación de ácidos nucleicos para una amplia gama de aplicaciones que requieren la regulación precisa de la actividad génica. En particular, nuevos materiales sensibles a los estímulos con un control espaciotemporal mejorado sobre la expresión génica desbloquearían plataformas traducibles en tecnologías de descubrimiento de fármacos y medicina regenerativa. Además, una mayor capacidad para controlar la liberación de ácido nucleico a partir de materiales permitiría el desarrollo de métodos aerodinámicos para predecir la eficacia del nanocarrier a priori , dando lugar a un rastreo acelerado de los vehículos de administración. Aquí, presentamos un protocolo para predecir el gen silenciamiento eficiencias y lograr spatiotemporal control sobre la expresión de genes a través de un modular fotorresistente sistema nanocarrier. El pequeño ARN interferente (ARNsi) se compleja con los polımeros de mPEG- b -poli (metacrilato de 5- (3- (amino) propoxi) -2-nitrobencilo) (mPEG- b- P (APNBMA)) foRm nanocarriers estables que pueden ser controlados con luz para facilitar tunable, on / off siRNA liberación. Describimos dos ensayos complementarios que emplean espectroscopia de correlación de fluorescencia y electroforesis en gel para la cuantificación precisa de la liberación de siRNA a partir de soluciones que imitan los ambientes intracelulares. La información obtenida de estos ensayos se incorporó en un simple modelo cinético de interferencia de ARN (RNAi) para predecir las respuestas de silenciamiento dinámico a diversas condiciones de foto-estímulo. A su vez, estas condiciones de irradiación optimizadas permitieron perfeccionar un nuevo protocolo para controlar espacialmente el silenciamiento de genes. Este método puede generar patrones celulares en la expresión génica con la resolución célula a célula y sin efectos detectables fuera de la meta. En conjunto, nuestro enfoque ofrece un método fácil de usar para predecir los cambios dinámicos en la expresión génica y controlar con precisión la actividad del siRNA en el espacio y el tiempo. Este conjunto de ensayos puede adaptarse fácilmente para probar una amplia variedad de otSus sistemas de respuesta a los estímulos para abordar desafíos clave pertinentes a una multitud de aplicaciones en la investigación biomédica y la medicina.
Introducción
Los pequeños ARNs de interferencia (siRNAs) median el silenciamiento génico post-transcripcional a través de una vía catalítica de ARNi que es altamente específica, potente y adaptable a prácticamente cualquier gen diana 1 . Estas características prometedoras han permitido que la terapéutica del siRNA avance en ensayos clínicos humanos para el tratamiento de numerosas enfermedades, incluyendo el melanoma metastásico y la hemofilia 2 , 3 . Sin embargo, persisten problemas de entrega importantes que han impedido la traducción 4 . En particular, los vehículos de entrega deben permanecer estables y proteger siRNAs de la degradación extracelular, sin embargo, también liberar la carga útil en el citoplasma [ 5] . Además, muchas aplicaciones de RNAi requieren métodos mejorados para regular el silenciamiento de genes en el espacio y el tiempo [ 6] , lo que reducirá los efectos secundarios en siARN terapéutica [ 7] y permitir transformador aDvances en aplicaciones que van desde microarrays de células para el descubrimiento de fármacos 8 a la modulación de las respuestas celulares en andamios regenerativos [ 9] . Estos desafíos destacan la necesidad de nuevos materiales y métodos para controlar mejor la unión vs liberación en los nanocarriers siRNA.
Una de las estrategias más prometedoras para controlar la liberación de siRNA y mejorar la regulación espaciotemporal es el uso de materiales sensibles a los estímulos 10 . Por ejemplo, se ha diseñado una amplia variedad de biomateriales con afinidad de unión a ácidos nucleicos variable en respuesta al potencial o potencial redox alterado, oa campos magnéticos aplicados, ultrasonidos o luz 11 . Aunque muchos de estos sistemas demuestran un control mejorado sobre la actividad del ácido nucleico, el uso de la luz como gatillo es particularmente ventajoso debido a su respuesta temporal instantánea, resolución espacial precisa y facilidad de afinación 12. Además, el potencial de las fotoprotectoras para la regulación de la expresión génica ha sido demostrado por el estado de la técnica promotor inducible y sistemas reguladores optogenéticos; Sin embargo, estos sistemas sufren de numerosos retos incluyendo capacidades limitadas para regular genes endógenos, preocupaciones de seguridad tales como inmunogenicidad, y dificultades en la entrega de ensamblajes de múltiples componentes 13 , 14 , 15 . Foto-sensores siRNA nanocarriers son ideales para superar estos inconvenientes y proporcionar un enfoque más simple y más robusto spatiotemporalmente modular la expresión génica [ 16 , 17 , 18] . Desafortunadamente, los métodos para predecir con exactitud la respuesta resultante de desmontaje de la proteína siguen siendo esquivos.
Un desafío clave es que las evaluaciones cuantitativas de la liberación de siRNA sonRara 19 , 20 , e incluso cuando se realizan estas evaluaciones, no se han acoplado a los análisis de siRNA / proteína dinámica de rotación. Tanto la cantidad de siRNA liberado y su persistencia / vida son determinantes importantes de la dinámica de silenciamiento de genes resultante; Por lo tanto, la falta de dicha información es una importante desconexión que impide una predicción precisa de la dosis-respuesta en RNAi [ 21] . Abordar este reto aceleraría la formulación de las relaciones estructura-función apropiadas en nanocarriers y mejoraría el conocimiento de los diseños de biomateriales 22 . Además, tales enfoques permitirían el desarrollo de protocolos de dosificación de siRNA más eficaces. En un intento de entender la respuesta silenciosa dinámica, varios grupos han investigado modelos matemáticos de RNAi 23 , 24 , 25 . Estos marcos fueronÉxito en la prestación de información sobre siRNA mediada por los cambios en la expresión génica y la identificación de la tasa de limitación de los pasos [ 26] . Sin embargo, estos modelos se han aplicado sólo a los sistemas comerciales de entrega de genes ( por ejemplo , Lipofectamine y polietilenimina (PEI)) que no son capaces de liberación controlada siRNA, y la complejidad de los modelos ha limitado severamente su utilidad [ 27] . Estas deficiencias resaltan una necesidad insatisfecha de nuevos materiales capaces de sincronizar con precisión la liberación de siRNA combinada con modelos dinámicos predictivos simplificados y fáciles de usar.
Nuestro método aborda todos estos retos a través de la integración de una plataforma fotovoltaica sensible a la luz con métodos acoplados para cuantificar la ARNi libre y la dinámica de RNAi modelo. En particular, la plataforma 28 controlada con precisión siRNA liberación 28 se supervisa por dos métodos complementarios para cuantificar con precisión encapsulado vs unLigado siRNA. Los datos experimentales de estos ensayos se introducen en un modelo cinético simple para predecir el gen silenciamiento eficiencias a priori [ 29] . Por último, la naturaleza on / off de los nanocarriers se explota fácilmente para generar patrones celulares en la expresión génica con control espacial en la escala de longitud celular. Por lo tanto, este método proporciona un método fácilmente adaptable para controlar y predecir el silenciamiento de genes en una variedad de aplicaciones que se beneficiarían de la regulación spatiotemporal del comportamiento celular.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Formulación de siRNA Nanocarriers
- Preparar soluciones separadas de siRNA y mPEG- b -AP (APNBMA) con volúmenes iguales diluidos en tampón de ácido 4- (2-hidroxietil) piperazina-1-etanosulfónico (HEPES) 20 mM a pH 6,0.
- Añadir siRNA en una concentración de 32 μ g / mL a 20 mM HEPES solución.
NOTA: El ARNsi era una secuencia de control negativo universal no dirigida; Sin embargo, el siRNA puede ser diseñado para dirigirse a cualquier gen de interés. - Disolver polímeros de mPEG- b- P (APNBMA) en una solución de HEPES 20 mM. Añadir una cantidad apropiada de mPEG- b- P (APNBMA) para preparar una solución de 220 μg / ml de modo que la relación N / P (N, grupos amina en mPEG- b- P (APNBMA), P, grupos fosfato en siRNA) Es 4.
NOTA: El protocolo sintético para los polímeros mPEG- b- P (APNBMA) se informa en otro lugar 30 .
- Añadir siRNA en una concentración de 32 μ g / mL a 20 mM HEPES solución.
- Añadir la solución de mPEG- b- P (APNBMA) gota a gota a un volumen igualE de la solución de siRNA mientras se mezcla suavemente en una máquina de vórtice. Continuar a agitar durante 30 s después de la adición de polímero. Incubar las muestras en la oscuridad a temperatura ambiente durante 30 min.
2. Medición de la liberación de siRNA mediante electroforesis en gel
- Formular los nanocarriers de acuerdo con los pasos 1.1-1.2, y escalar los volúmenes para acomodar el número de muestras deseadas.
- Mezclar el nanocarrier con dodecil sulfato de sodio (SDS).
- Preparar una solución de 1 mg / ml de SDS en agua. Alícuota de la cantidad de solución SDS necesaria para producir soluciones con una relación S / P (S, grupos sulfato en SDS, P, grupos fosfato en siRNA) de 15.
NOTA: Si la solución polyplex contiene 1 μg de siRNA, se deben agregar 13 μg de SDS para lograr una relación S / P de 15. - Añadir la solución de SDS a cada solución nanocarrier gota a gota mientras se mezcla suavemente en una máquina de vórtice. Continuar a vortex durante 30 s después de la adición SDS.
- Centrifugar las muestras a 3.000 xg durante 5 s. Incubar las muestras en la oscuridad a temperatura ambiente durante 30 min.
- Preparar una solución de 1 mg / ml de SDS en agua. Alícuota de la cantidad de solución SDS necesaria para producir soluciones con una relación S / P (S, grupos sulfato en SDS, P, grupos fosfato en siRNA) de 15.
- Calibre y ajuste un láser UV con un filtro de 365 nm a una intensidad de 200 W / m. Asegúrese de que la intensidad de la luz se mide desde la ubicación en la que se asentará la parte inferior de la solución de muestra.
- Cargar la solución nanocarrier / SDS en una cámara de vidrio compuesta de diapositivas de vidrio separadas por una junta de goma.
- El vidrio de prelavado se desliza en una solución de etanol / agua 7: 3 (v / v) en agua y se seca completamente. Corte un agujero (~ 2 x 3 cm rectángulo) en una junta de goma. Coloque la junta de goma en un portaobjetos de vidrio.
- Pipetee la solución de nanocarrier / SDS sobre el portaobjetos de vidrio dentro del orificio de la junta de goma. Cargar un exceso de solución (20 μl en exceso) sobre el portaobjetos de vidrio evitando el contacto con la junta de goma.
NOTA: Algún líquido se perderá durante los siguientes pasos. - Coloque el segundo vasoDeslice en la parte superior de la junta deslizante. Para evitar la generación de burbujas de aire, coloque primero un extremo de la corredera y luego baje lentamente el otro extremo.
- Sujete los clips de la encuadernación a cada lado de la cámara de vidrio para mantenerla cerrada.
- Irradiar las muestras durante el tiempo deseado ( por ejemplo , 0-60 min) usando el láser UV con un filtro de 365 nm a una intensidad de 200 W / m. Retire los clips de la encuadernación y abra la cámara.
- Pipetear 25 μL de las muestras de nanocarrier / SDS irradiadas en un tubo de microcentrífuga. Incubar las soluciones en la oscuridad a temperatura ambiente durante 30 min.
- Preparar un gel de agarosa al 2% en peso previamente teñido con 0,5 μg / mL de bromuro de etidio en solución tamponada Tris / Borato / EDTA (TBE) de acuerdo con los protocolos estándar 31 . Preparar un tampón de carga compuesto de glicerol / agua 3: 7 (v / v).
- Añadir 5 μl de la solución tampón de carga a 25 μl de cada muestra nanocarrier / SDS. Incubar las muestras en la oscuridad aTemperatura ambiente durante 10 min.
- Cargar 30 μL de cada muestra de nanocarrier / SDS en el gel de agarosa al 2%. Ejecutar el gel en la oscuridad a 100 V durante 30 min. Image el gel utilizando un sistema de imágenes de gel con filtros de bromuro de etidio. Guarde los archivos de imagen de gel y continúe con el paso 2.10 para la cuantificación de intensidad de banda. Asegúrese de que las intensidades de banda son lo suficientemente brillantes como para visualizar claramente pero no demasiado brillante que las señales están saturadas.
- Cuantificar las intensidades de banda utilizando el software ImageJ disponible públicamente 32 .
- Usando la herramienta ROI de ImageJ, determine la intensidad de fluorescencia de las bandas de siRNA libres en cada carril dibujando un rectángulo alrededor de cada banda. Trace las curvas de intensidad de cada carril e integre el área bajo las curvas dibujando una línea horizontal a través de las curvas de intensidad y haciendo clic en la varilla de trazado dentro de las áreas cerradas.
- Calcular la intensidad relativa de cada carril dividiendo el área bajo el cuRve de cada muestra por el área bajo la curva del control positivo de siRNA (no se a~nadió mPEG- b- P (APNBMA) y no se a~nadió SDS). Informe el porcentaje de siRNA liberado como la intensidad de banda normalizada de cada muestra.
3. Medición de la liberación de siRNA usando fluorescencia Correlation Spectroscopy (FCS)
- Obtener siRNA marcado con un único fluoróforo en el extremo 5 'de la cadena sentido.
NOTA: El siRNA se puede comprar previamente recocido con las etiquetas conjugadas en la ubicación deseada. El fluoróforo debe ser foto-estable y absorber / emitir entre 450 y 750 nm para evitar la extinción de luz UV y la transferencia de energía con mPEG- b- P (APNBMA). - Formular los nanocarriers de acuerdo con los pasos 1.1-1.2 usando el siRNA marcado. Escale los volúmenes para acomodar el número de muestras deseadas.
- Incubar las soluciones en SDS e irradiar durante el tiempo deseado de acuerdo con los pasos 2.2-2.6.
- PrepararDe la cámara de muestra FCS.
- Lavar un portaobjetos de vidrio con una solución de etanol / agua 7: 3 (v / v) y secar completamente el vidrio con un trapo y una corriente de aire.
- Retire los pedazos de papel de un espaciador adhesivo de doble cara para exponer el espaciador adhesivo de doble cara. Conecte el espaciador a un cubreobjetos de vidrio.
- Pipetear la solución de nanocarrier / SDS sobre el resbalón de la cubierta en el medio del agujero del espaciador adhesivo.
- Coloque la diapositiva de cristal en la parte superior del cubreobjetos. Empuje el portaobjetos de vidrio para asegurarse de que el portaobjetos de vidrio y la cubreobjetos estén bien unidos y formen un sello.
- Utilizar un microscopio confocal para las mediciones FCS 33 . Utilice un objetivo de 40x de inmersión en agua apochromat con una apertura numérica de 1,2. Utilice el canal láser de excitación apropiado (488 nm) para recoger al menos 30 mediciones de 10 s cada una por muestra 34 . Asegúrese de que la intensidad del láser y la alineación del detectorEl mismo para cada muestra.
- Además de las muestras experimentales, se miden los controles incluyendo: una muestra en blanco sin ARNsi marcado; Y una muestra de siRNA libre con siRNA marcado pero sin mPEG- b- P (APNBMA).
- Analizar los datos utilizando software específico de FCS. Identificar la tasa de recuento de referencia de cada muestra mediante la determinación de la tasa de recuento estable durante un tiempo cuando no nanocarriers están pasando a través del volumen confocal [ 29] .
- Restar la tasa de recuento de la muestra en blanco de cada valor de la cuenta de referencia de la muestra. Normalizar los valores resultantes al control de siRNA libre para calcular el porcentaje de siRNA libre 35 .
4. Modelado cinético para predecir la silenciación de genes
- Crear scripts en un lenguaje de programación matemático usando el conjunto simple de ecuaciones diferenciales ordinarias para predecir el silenciamiento de genes 29.
NOTA: Los scripts pueden estar disponibles bajo petición.- Escriba el conjunto de ecuaciones diferenciales ordinarias como:
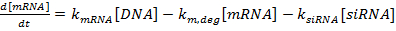 (1)
(1)  (2)
(2) 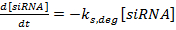 (3)
(3)
NOTA: Para las ecuaciones 1-3, los términos k mRNA , k siRNA y k prot son las constantes de velocidad para la producción de mRNA, siARN y proteína, respectivamente. Los términos k m, deg , k s, deg y kp , deg son las constantes de velocidad para la degradación del mRNA, siRNA y proteína, respectivamente. Las constantes de velocidad de degradación se calculan sobre la base de las semividas de componente y las constantes de velocidad de producción son adecuadas para asegurar que los valores de ARNm y de estado estacionario de proteína se alcanzan en ausencia deF siRNA.- Determine las semividas del ARNm y la proteína para el gen o los genes de interés, ya sea experimentalmente como se describe en la referencia 36 o de la literatura (véase la Discusión ). Además, determine el tiempo de duplicación para la línea celular. Introduzca estos valores en las expresiones de velocidad de degradación apropiadas.
- Ajuste las constantes de velocidad de producción de manera que los niveles de expresión génica permanezcan estables a un valor normalizado de 100 si no se introduce ningún ARNic. Específicamente, establezca [siRNA] a cero y varíe los valores de las constantes de k ARNm , k siRNA y k prot hasta que [ARNm] y [proteína] permanezcan dentro del 1% del valor normalizado inicial del 100% durante la duración de La simulación.
- Usando las cantidades relativas de siRNA liberado de la electroforesis en gel previamente descrita y los ensayos de FCS como estimaciones, se ajusta la concentración relativa inicial de siRNA en el guión. Específicamente, variar [siRNA] sea proporcional a la cantidad relativa de siRNA liberado, con un valor de 100 correspondiente a la cantidad máxima 29 .
- Escriba el conjunto de ecuaciones diferenciales ordinarias como:
5. Cultivo de células y suministro in vitro de siRNA
- Cultura NIH / 3T3 fibroblastos embrionarios murinos de acuerdo con los protocolos del proveedor.
- Cultivar las células en medio de crecimiento (medio de Eagle modificado por Dulbecco (DMEM) suplementado con 10% de suero fetal bovino inactivado por calor y 1% de penicilina-estreptomicina). Mantener las células a 37 ° C en un ambiente humidificado con 5% de CO 2 .
- Sembrar las células en placas de 6 pocillos tratadas con cultivo de tejidos.
- Siga el procedimiento de subcultivo recomendado del proveedor. Cuente las células usando un hemocitómetro. Diluir las células en medio de crecimiento suplementado hasta una concentración de 75.000 células / ml.
- Añadir 2 ml de suspensión celular (75.000 célulasS / ml) a cada pocillo de la placa de 6 pocillos. Deje que las células se adhieran y se recuperen durante 24 h en la incubadora.
- Preparar las células para la transfección mediante lavado con solución salina tamponada con fosfato (PBS) y añadir 1,5 ml de medio de transfección sin suero y antibiótico (véase la Tabla de Materiales ) a cada pocillo.
- Formule los nanocarriers de siRNA de acuerdo con los pasos 1.1-1.2. Añadir 25 μL de solución nanocarrier conteniendo 30 pmol de siRNA a cada pocillo. Con cuidado, pipetee los medios hacia arriba y hacia abajo para mezclarlos. Colocar las células en la incubadora durante 3 h.
- Eliminar el medio de transfección y lavar cada pocillo con PBS. Añadir 1 mL de medio de crecimiento suplementado y colocar las células en la incubadora para recuperar durante 30 min.
- Para preparar las células para el tratamiento con foto-estímulo, eliminar el medio de crecimiento suplementado. Añadir 1 ml de medio de transfección (sin rojo de fenol) a cada pocillo.
NOTA: Asegúrese de que el medio de transfección no contenga rojo fenol. - Calibre y ajuste un láser UV con un filtro de 365 nm a una intensidad de 200 W / m. Asegúrese de que la intensidad de la luz se mide desde la ubicación en la que se asentará la parte inferior de la placa de la celda.
- Coloque las células sobre una placa caliente colocada a 37 ° C. Retire la cubierta de la placa de las celdas. Irradiar las células desde arriba de la placa durante el tiempo deseado (hasta 20 min) usando el láser UV con un filtro de 365 nm a una intensidad de 200 W m -2 .
- Eliminar el medio de transfección y añadir 2 ml de medio de crecimiento suplementado. Colocar en incubadora hasta un análisis posterior ( por ejemplo , 24 h para qPCR y 48 h para Western Blot).
- Mida los cambios en la expresión génica usando una variedad de técnicas tales como Western blotting 37 y qPCR. 38 Para los genes con señales visibles, como las buenas prácticas agrarias, el uso de microscopía de fluorescencia [ 29] .
NOTA: Estas técnicas se sugieren debido a su facilidad de uso y precisiónEn la cuantificación de la expresión génica
- Mida los cambios en la expresión génica usando una variedad de técnicas tales como Western blotting 37 y qPCR. 38 Para los genes con señales visibles, como las buenas prácticas agrarias, el uso de microscopía de fluorescencia [ 29] .
6. Controlar el Silenciamiento de Genes de una manera Spatiotemporal
- Células de cultivo, semillas y transfectas de acuerdo con los pasos 5.1-5.7.
- Prepare una fotomáscara que bloquee por completo la luz de 365 nm y minimice las reflexiones.
NOTA: En este caso, se utilizaron trozos de papel de aluminio de 10 x 10 cm y papel de construcción negro para bloquear la luz y reducir los reflejos, respectivamente. La hoja de aluminio y el papel de construcción se pegaron juntos para formar una sola unidad.- Cortar / perforar / mecanizar la forma deseada en la fotomáscara. Por ejemplo, utilice una cuchilla afilada y un perforador para formar un dibujo en línea recta (~ 5 cm de largo) y un patrón circular (~ 7 mm de diámetro) en la fotomáscara, respectivamente.
- Pegue la fotomáscara al fondo de la placa de 6 pocillos con el patrón centrado debajo del pocillo que contiene las células con el lado antirreflectante ( por ejemplo , negro cPapel) frente a la placa. Asegúrese de que el pegamento no esté colocado cerca del borde (dentro de ~ 3 mm) del patrón.
- Coloque dos soportes de anillo aproximadamente 25 cm de distancia y coloque una plataforma en cada soporte de anillo para que las plataformas sean de igual altura. Suspenda la placa de la celda entre los dos soportes apoyando la placa en la parte superior de las plataformas. Asegúrese de que la placa esté nivelada.
- Irradiar las células de debajo de la muestra durante el tiempo deseado (hasta 20 min) utilizando el láser UV con un filtro de 365 nm a una intensidad de 200 W / m 2.
- Eliminar el medio de transfección y añadir 2 ml de medio de crecimiento suplementado. Colocar en la incubadora para recuperar por lo menos 24 h. Imagen de las células utilizando microscopía de fluorescencia como se describe [ 29] .
Access restricted. Please log in or start a trial to view this content.
Resultados
Después de la formulación de los nanocarriers, los ensayos de liberación de siRNA se llevaron a cabo para informar las condiciones de irradiación que se utilizarán en las transfecciones in vitro . Se aplicaron varias dosis de luz para determinar el porcentaje de siRNA que se liberó. El primer ensayo utilizó electroforesis en gel para separar las moléculas de siRNA libres de las moléculas de siRNA todavía complejadas / asociadas con el polímero. Los Nanocarriers que no...
Access restricted. Please log in or start a trial to view this content.
Discusión
Hay algunos pasos en el método que son particularmente críticos. Al formular los nanocarriers, el orden de la adición de componentes y velocidad de mezcla son dos parámetros importantes que influyen en la eficacia [ 39] . Este protocolo requiere que el componente catiónico, mPEG- b- P (APNBMA), se añada al componente aniónico, siRNA, de una forma gota a gota mientras se agita en vórtice. Dependiendo del volumen total de la formulación, este proceso de mezcla toma 3-6 s. Para pro...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Los autores agradecen al Instituto Nacional de Ciencias Médicas Generales de los Institutos Nacionales de Salud (NIH) por el apoyo financiero a través de un Premio de Desarrollo Institucional (IDeA) bajo el número de concesión P20GM103541, así como el número de la concesión P20GM10344615. Las declaraciones aquí contenidas no reflejan las opiniones de los NIH. También reconocemos al Instituto de Biotecnología de Delaware (DBI) y la Oficina de Desarrollo Económico de Delaware (DEDO) por su apoyo financiero a través del premio Bioscience CAT para el Bioscience (12A00448).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| siRNA | Sigma-Aldrich | SIC001 | non-targeted, universal negative control |
| mPEG-b-P(APNBMA) | synthesized in our lab | N/A | photo-responsive polymer |
| HEPES | Fisher Scientific | BP310-100 | |
| sodium dodecyl sulfate | Sigma-Aldrich | 436143 | |
| rubber gasket | McMaster-Carr | 3788T21 | 0.5 mL thick |
| UV laser | Excelitas Technologies | Omnicure S2000 | collimating lens and 365 nm filter used |
| agarose | Fisher Scientific | BP160-100 | |
| ethidium bromide | Fisher Scientific | BP1302-10 | |
| siRNA labelled with Dy547 | GE Healthcare Dharmacon, Inc. | custom order | fluorophore conjugated to 5’ end of sense strand |
| microscope slide | Fisher Scientific | 12-550-A3 | pre-cleaned glass |
| Secure-Seal Spacer | Life Technologies | S24735 | double-sided adhesive |
| LSM 780 | Carl Zeiss | N/A | confocal microscope |
| ZEN 2010 | Carl Zeiss | N/A | FCS analysis software |
| MATLAB | MathWorks | N/A | programming language |
| NIH/3T3 cells | ATCC | ATCC CRL-1658 | |
| DMEM | Mediatech | 10-013-CV | growth media |
| fetal bovine serum | Mediatech | 35-011-CV | heat-inactivated |
| penicillin-streptomycin | Mediatech | 30-002-CI | |
| 6-well plates | Fisher Scientific | 08-772-1B | |
| Opti-MEM | Life Technologies | 11058021 | transfection media |
Referencias
- Forbes, D. C., Peppas, N. A. Oral delivery of small RNA and DNA. J Control Release. 162 (2), 438-445 (2012).
- Davis, M. E., et al. Evidence of RNAi in humans from systemically administered siRNA via targeted nanoparticles. Nature. 464 (7291), 1067-1070 (2010).
- Bouchie, A. Companies in footrace to deliver RNAi. Nat Biotechnol. 30 (12), 1154-1157 (2012).
- Burke, P. A., Pun, S. H., Reineke, T. M. Advancing Polymeric Delivery Systems Amidst a Nucleic Acid Therapy Renaissance. ACS Macro Lett. 2 (10), 928-934 (2013).
- Gooding, M., Browne, L. P., Quinteiro, F. M., Selwood, D. L. siRNA Delivery: From Lipids to Cell-penetrating Peptides and Their Mimics. Chem Biol Drug Des. 80 (6), 787-809 (2012).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nat Chem Biol. 10 (3), 196-202 (2014).
- Wang, X., Chen, X., Yang, Y. Spatiotemporal control of gene expression by a light-switchable transgene system. Nat Methods. 9 (3), 266-269 (2012).
- Ziauddin, J., Sabatini, D. M. Microarrays of cells expressing defined cDNAs. Nature. 411 (6833), 107-110 (2001).
- Saltzman, W. M., Olbricht, W. L. Building drug delivery into tissue engineering. Nat Rev Drug Discov. 1 (3), 177-186 (2002).
- Mura, S., Nicolas, J., Couvreur, P. Stimuli-responsive nanocarriers for drug delivery. Nat Mater. 12 (11), 991-1003 (2013).
- Shim, M. S., Kwon, Y. J. Stimuli-responsive polymers and nanomaterials for gene delivery and imaging applications. Adv Drug Delivery Rev. 64 (11), 1046-1058 (2012).
- Kelley, E. G., Albert, J. N. L., Sullivan, M. O., Epps, T. H. Stimuli-responsive copolymer solution and surface assemblies for biomedical applications. Chem Soc Rev. 42 (17), 7057-7071 (2013).
- Weber, W., Fussenegger, M. Emerging biomedical applications of synthetic biology. Nat Rev Genet. 13 (1), 21-35 (2012).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Shimizu-Sato, S., Huq, E., Tepperman, J. M., Quail, P. H. A light-switchable gene promoter system. Nat Biotechnol. 20 (10), 1041-1044 (2002).
- Huschka, R., et al. Gene Silencing by Gold Nanoshell-Mediated Delivery and Laser-Triggered Release of Antisense Oligonucleotide and siRNA. ACS Nano. 6 (9), 7681-7691 (2012).
- Li, H. -J., Wang, H. -X., Sun, C. -Y., Du, J. -Z., Wang, J. Shell-detachable nanoparticles based on a light-responsive amphiphile for enhanced siRNA delivery. R Soc Chem Adv. 4 (4), 1961-1964 (2014).
- Braun, G. B., et al. Laser-Activated Gene Silencing via Gold Nanoshell-siRNA Conjugates. ACS Nano. 3 (7), 2007-2015 (2009).
- Gilleron, J., et al. Image-based analysis of lipid nanoparticle-mediated siRNA delivery, intracellular trafficking and endosomal escape. Nat Biotechnol. 31 (7), 638-646 (2013).
- Wittrup, A., et al. Visualizing lipid-formulated siRNA release from endosomes and target gene knockdown. Nat Biotechnol. 33 (8), 870-876 (2015).
- Roth, C. M. Quantitative measurements and rational materials design for intracellular delivery of oligonucleotides. Biotechnol Prog. 24 (1), 23-28 (2008).
- Mao, S., et al. Influence of polyethylene glycol chain length on the physicochemical and biological properties of poly(ethylene imine)-graft-poly(ethylene glycol) block copolymer/SiRNA polyplexes. Bioconjugate Chem. 17 (5), 1209-1218 (2006).
- Raab, R. M., Stephanopoulos, G. Dynamics of gene silencing by RNA interference. Biotechnol Bioeng. 88 (1), 121-132 (2004).
- Cuccato, G., et al. Modeling RNA interference in mammalian cells. BMC Systems Biology. 5, 1(2011).
- Varga, C. M., Hong, K., Lauffenburger, D. A. Quantitative analysis of synthetic gene delivery vector design properties. Mol Ther. 4 (5), 438-446 (2001).
- Chen, H. H., et al. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum Dot-FRET. Mol Ther. 16 (2), 324-332 (2008).
- Bartlett, D. W., Davis, M. E. Insights into the kinetics of siRNA-mediated gene silencing from live-cell and live-animal bioluminescent imaging. Nucleic Acids Res. 34 (1), 322-333 (2006).
- Foster, A. A., Greco, C. T., Green, M. D., Epps, T. H., Sullivan, M. O. Light-Mediated Activation of siRNA Release in Diblock Copolymer Assemblies for Controlled Gene Silencing. Adv Healthc Mater. 4 (5), 760-770 (2015).
- Greco, C. T., Epps, T. H., Sullivan, M. O. Mechanistic Design of Polymer Nanocarriers to Spatiotemporally Control Gene Silencing. ACS Biomater Sci Eng. 2 (9), 1582-1594 (2016).
- Green, M. D., et al. Catch and release: photocleavable cationic diblock copolymers as a potential platform for nucleic acid delivery. Polym Chem. 5 (19), 5535-5541 (2014).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Marquer, C., Leveque-Fort, S., Potier, M. C. Determination of Lipid Raft Partitioning of Fluorescently-tagged Probes in Living Cells by Fluorescence Correlation Spectroscopy (FCS). J Vis Exp. (62), (2012).
- Staaf, E., Bagawath-Singh, S., Johansson, S. Molecular Diffusion in Plasma Membranes of Primary Lymphocytes Measured by Fluorescence Correlation Spectroscopy. J Vis Exp. (120), e54756(2017).
- Buyens, K., et al. Monitoring the disassembly of siRNA polyplexes in serum is crucial for predicting their biological efficacy. J Control Release. 141 (1), 38-41 (2010).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764-768 (2011).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proc Natl Acad. Sci U S A. 76 (9), 4350-4354 (1979).
- Wittwer, C. T., Herrmann, M. G., Moss, A. A., Rasmussen, R. P. Continuous Fluorescence Monitoring of Rapid Cycle DNA Amplification. Biotechniques. 54 (6), 314-320 (2013).
- Cho, S. K., Dang, C., Wang, X., Ragan, R., Kwon, Y. J. Mixing-sequence-dependent nucleic acid complexation and gene transfer efficiency by polyethylenimine. Biomater Sci. 3 (7), 1124-1133 (2015).
- Liu, Y. M., Reineke, T. M. Poly(glycoamidoamine)s for gene delivery: Stability of polyplexes and efficacy with cardiomyoblast cells. Bioconjugate Chem. 17 (1), 101-108 (2006).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Greco, C. T., Muir, V. G., Epps, T. H., Sullivan, M. O. Efficient tuning of siRNA dose response by combining mixed polymer nanocarriers with simple kinetic modeling. Acta Biomater. 50, 407-416 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados