Method Article
Análisis automatizado de la diapositiva y segmentación en tejidos marcada con fluorescencia utilizando un sistema de análisis de la Widefield High-contenido
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí describimos un protocolo para la segmentación automática de tejidos fluorescencia etiquetadas en diapositivas, utilizando un sistema de análisis de alto contenido de campo amplio (WHCAS). Este protocolo tiene amplias aplicaciones en cualquier campo que consiste en la cuantificación de marcadores fluorescentes en tejidos biológicos, como las ciencias biológicas, ingeniería médica y Ciencias de la salud.
Resumen
Automated slide análisis y segmentación de marcada con fluorescencia de los tejidos es la manera más eficiente para analizar todo diapositivas o grandes cortes de tejido. Lamentablemente, muchos investigadores dedican grandes cantidades de tiempo y recursos desarrollando y optimizando los flujos de trabajo que sólo son relevantes para sus propios experimentos. En este artículo se describe un protocolo que puede utilizarse por aquellos que tienen acceso a un sistema de análisis de alto contenido de campo amplio (WHCAS) para cualquier tejido montado en corredera, con opciones de personalización dentro de módulos pre-construidos en el software asociado. No originalmente concebidos para el escaneo de diapositivas, los pasos detallados en este artículo permiten adquirir diapositiva imágenes en el WHCAS que se pueden importar en el software asociado. En este ejemplo, se demuestra la segmentación automática de diapositivas del tumor de cerebro, pero la segmentación automatizada de cualquier marcador marcada con fluorescencia nuclear o citoplásmico es posible. Además, hay una variedad de otros módulos de software cuantitativos incluyendo ensayos de localización/translocación de proteínas, proliferación celular/viabilidad/apoptosis y angiogénesis que se pueden ejecutar. Esta técnica se ahorra esfuerzo y tiempo que los investigadores y crear un protocolo automatizado para el análisis de diapositivas.
Introducción
La cuantificación exacta y precisa de tejidos marcada con fluorescencia en las diapositivas es una técnica altamente solicitada en muchos campos científicos. Sin embargo, los investigadores a menudo manualmente cuentan muestras o gastan cantidades considerables de tiempo desarrollando técnicas automatizadas esotéricas para lograr esto. Aquí, ofrecemos un protocolo para la diapositiva automatizado análisis y cuantificación de células usando un WHCAS y su software asociado, con las células inmunes innatas en secciones del tumor de cerebro humano congelado como ejemplo. El software asociado ofrece una amplia gama de módulos personalizables incorporados del neurite consecuencia contando a la diferenciación de célula tipo1,2,3,4,5, 6. el objetivo de este método es proporcionar a los investigadores con un protocolo de principio a fin, fácilmente reproducible para adquirir imágenes de y cuantificar entidades marcada con fluorescencia en cualquier tejidos montados en portaobjetos.
En el presente Protocolo, el WHCAS se utiliza principalmente para placas para el posterior análisis en el software asociado para la proyección de imagen aunque dispusiera de un adaptador de diapositivas y las bases para análisis7 . Era prohibitivo a las diapositivas de imagen porque la calibración espacial cuidadosa de la zona de adquisición, la selección de revistas apropiadas, la creación de equipamiento a medida y un enlace con los representantes de producto se requiere. En el cuerpo más amplio de la literatura, en lugar de comprar una proyección de imagen de diapositiva dedicada y análisis aparato8, un anterior informe tecnológico con acceso a este software había eludido la adquisición de imágenes de diapositivas en el WHCAS en conjunto9. Realizar análisis de imagen o adquisición de imágenes en diferentes plataformas, requiere un trabajo extra para asegurar que cada uno sea compatible con la otra.
La habilidad de usar el WHCAS y el software para captura de imagen evitaría las complicaciones innecesarias de búsqueda o desarrollo de un alien de flujo de trabajo a estas herramientas. En este artículo, los pasos necesitan para crear una ampliación baja Resumen la exploración y las correspondientes imágenes de gran aumento por el tratamiento de la diapositiva como un plato, y el análisis posterior mediante el módulo de segmentación de multi-longitud de onda de la célula que permiten la replanificación de la WHCAS. Este protocolo fácilmente usable proporciona una ventaja sobre técnicas alternativas porque no es necesario desarrollar algoritmos o multi-step cuenta protocolos10,11 , una vez que las imágenes se adquieren en el WHCAS. Este protocolo reduce el tiempo necesario para optimizar una técnica de cuantificación, es más preciso12 y eficiente que el conteo manual y maximiza el uso de la WHCAS. Este protocolo puede ser fácil y ampliamente utilizado ya permite la proyección de imagen y análisis de cualquier tejido marcada con fluorescencia en las diapositivas.
Protocolo
Se obtuvieron muestras de tumor según el protocolo aprobado por el Comité de ética y Junta de revisión institucional local y llevó a cabo conforme a normas nacionales. El WHCAS y su software asociado en este artículo se enumeran en la Tabla de materiales.
1. importación de las revistas
- Abra el software asociado.
- Descargar la suite diario indicada en la Tabla de materiales.
- Importar el paquete de diario a un directorio adecuado haciendo clic en el menú principal de partida revista, selección de Importación diario Suite...y haga clic en importar.
2. crear opciones para escuchar analizar adquisición
- En el cuadro de diálogo Configuración de placa de adquisición , ir a la ficha de cámara y objetivo seleccionar 4 X como la ampliación, 1 como la cámara binning y 2 como la ganancia.
- La configuración en la figura 1A en la pestaña de la placa: Slidescanningde entrada.
Nota: Estos ajustes se han creado para permitir al usuario ver y navegar un máximo de 3 diapositivas. Cada diapositiva se ha dividido en 3 pozos adyacentes para la fácil navegación y visualización de los cortes de tejido o células en ' vivo'. - En la ficha de sitios para visitar , se llenan los pozos uniformemente los sitios.
- Haga clic en Editar la placa inferior configuración... e introduzca los valores mostrados en la figura 1B.
- La ficha de Adquisición lazo , introduzca la longitud de onda deseada (cualquier tinción nuclear en este ejemplo) para el análisis de vista previa. Para el mejor contraste y enfoque basado en la imagen, utilice la señal fluorescente más brillante (por ejemplo, a menudo una tinción nuclear).
Nota: Tinción de tejidos con una mancha brillante como una tinción nuclear se recomienda ya que esto proporciona un alto contraste en comparación con el fondo. No sólo será esta ayuda en la localización de la muestra, pero cuando más de un fluoróforo en la siguiente adquisición de alta magnificación de imagen, se utilizará el color más brillante a base del proyección de imagen offset de los otros colores en. - Habilitar Corrección de sombreado en modo de adquisición de la placa para unir imágenes.
- Guardar esta configuración a configuración.
3. creando ambientes para la adquisición de diapositivas en alta magnificación
- En el cuadro de diálogo Configuración de adquisición de la placa , vaya a la ficha cámara y objetivo seleccionar 40 X como la ampliación, 1 como la cámara binning (para la más alta resolución digital) y 2 como la ganancia (para aumentar la señal y el brillo de las imágenes).
Nota: Puede usarse un valor más bajo de aumento del objetivo, pero los autores encontraron 40 X ser el ajuste óptimo para el análisis de humano microglia y macrófagos en el tejido de tumor cerebral con el módulo de Onda celular puntuación . - Asegurar todos los ajustes para las lengüetas de la placa – Slidescanning y Editar la placa inferior configuración... son los mismos que para ajuste A (ver paso 2).
- Si las imágenes obtenidas no son nítidas, cambiar la profundidad, altura de placay Placa inferior configuración para ajustar el enfoque. Si todavía hay un problema con el foco, elija Laser con la recuperación de la imagen en la sección de Opciones de enfoque automático .
- En la ficha de Adquisición lazo , introduzca el número de longitudes de onda presentes en la muestra. Compruebe las opciones de Activar láser centrado y permitir basado en imágenes centrándose (recuperación adquisición o láser) .
- En la pestaña de enfoque automático , seleccionar el foco en la placa y bien inferior.
- En la ficha publicaciones , seleccione las opciones tal como se indica en la figura 2, asegurando que evitar hardware asincrónico se mueve es también seleccionado.
- Guardar estos ajustes como configuración B.
4. colocar el portaobjetos en el sistema de análisis de campo amplio de alto contenido
- Presione F4 para obtener el Menú principal. Seleccione diapositiva exploración. Haga clic en abrir la puerta – diapositiva expulsión e insertar diapositivas y deslice el adaptador (que puede contener hasta tres diapositivas) en el WHCAS (Figura 3A y 3B). Oriente la diapositiva con el cubreobjetos hacia abajo y la etiqueta en el lado de la muesca en el adaptador de diapositivas (Figura 3A).
Nota: Las diapositivas utilizadas en este experimento fueron estándar 25 x 75 x 1 diapositivas mm. - Haga clic en cerrar la puerta – carga deslizante.
5. adquisición de una exploración de la vista previa
- Bajo el título de proyección , seleccionar la placa de adquisición y Control de... y ... Adquisición del Setup de la placa y A. de ajuste de la carga
- En la pestaña de pozos de visita , hacia el bien que contiene la muestra, utilizando el botón de clic del ratón. En la ficha de sitios para visitar , moverse a diferentes sitios con la captura de imagen en vivo hasta que las células se encuentran. Manualmente se centran en la muestra o utilizar el enfoque automático. Usar Snap para capturar la imagen.
Nota: La muestra es más fácil de localizar con más sitios y ajustando los sitios para el pozo (véase el paso 2.3). - En el menú principal, seleccione Las diapositivas vista previa.
- En el cuadro de diálogo que aparece automáticamente, seleccione la opción que refleja cuántas diapositivas se van a escanear. Escanear una diapositiva a la vez. Oprima OK. Seleccione 4 X como la ampliación. Oprima OK. Seleccione cubreobjetos abajo (0,17 mm) como la orientación de la diapositiva (uso de una corrección de cuello si se utiliza un grueso cubreobjetos diferentes). Oprima OK. Haga clic en continuar.
Nota: Si el usuario desea adquirir muestras de las exploraciones para 2 o más diapositivas, cada análisis de vista previa tiene que abrirse individualmente antes de la adquisición de la imagen correspondiente de alta magnificación. - Ajuste Deslice análisis de carga, asegúrese de que las casillas de parámetros de adquisición y calibración se comprueban. Presione Cancelar. Cuando vuelve a aparecer el cuadro de Configuración de diapositiva de exploración , seleccione continuar.
- Cuando aparezca el cuadro de diálogo Escanear diapositiva , ajuste la configuración como se indica en la Figura 4A-4 C. Seleccione cerrar cuando haya terminado.
Nota: Aumentar la cámara binning y disminuyendo el tiempo de exposición de la cámara resultará en escaneo más rápido, aunque en una menor resolución digital y rango dinámico, respectivamente, por lo tanto disminuyendo la calidad de imagen. Corrección de sombreado se asegurar una intensidad uniforme a través de un solo campo de visión. Encender Hardware y enfoque de imagen automático se asegurará de la exploración está en foco pero ralentizará la exploración. En aras de ahorrar tiempo, es aconsejable tener sombreado corrección y Hardware y enfoque automático de imágenes de mientras adquiere la exploración Ampliación baja, pero para tener estas opciones encendido paso 6 durante la proyección de imagen de alta magnificación. La resolución de la exploración de ampliación baja no afectará la resolución de la exploración de alta magnificación. - Seleccione un directorio para la diapositiva en el análisis de datos. Haga clic en Aceptar. Introduzca un nombre para el archivo. Haga clic en Aceptar.
- Automáticamente aparece el cuadro de diálogo Llamar la región . Utilice la herramienta región Rectangular (figura 5A) para seleccionar la región de exploración completa vista previa como se muestra en la figura 5B. Haga clic en continuar.
- Aparecerá el cuadro de diálogo Instalación completa . Seleccione continuar.
Nota: El escaneo de diapositivas vista previa ahora comenzará. - Al finalizar la vista previa, aparecerá el cuadro de diálogo Escaneo completo . Seleccione continuar. Cierre la ventana de exploración de vista previa (en este ejemplo la ventana de Exploración de DAPI ; ver figura 6).
Nota: La configuración y el proceso de adquisición de ampliación baja exploración tomará aproximadamente 20 minutos. El tiempo de adquisición de imagen posterior alta ampliación dependerá de la cantidad de sitios elegidos así como los tiempos de exposición para cada canal.
6. gran aumento de la exploración
- Cargar configuración B.
- Determinar que los pozos son a ser reflejada. En la pestaña de pozos de visita , hacia el bien que contiene la muestra, utilizando el botón de clic del ratón.
Nota: La técnica se demuestra aquí por proyección de imagen bien 1. - En la ficha de sitios para visitar , moverse a diferentes sitios con la característica de Live hasta las células o tejido se encuentra. Manualmente se centran en la muestra o utilizar el enfoque automático. Usar Snap para capturar la imagen.
Nota: Para ayudar en la localización de la muestra en una alta ampliación, utilice el cuadro de diálogo de placa de adquisición y Control para ajustar el tamaño de paso de 5 a 100 μm para encontrar la muestra si no Autofocus. - En la ficha de W1 DAPI , haga clic en Auto descubierto. Asegúrese de que la imagen de la Placa de adquisición Snap - DAPI es crujiente.
- En el menú principal, haga clic en diario para revelar el desplegable. Seleccione Ejecutar diario....
- Haga doble clic en la revista Deslice región adquisición instalación para ejecutarlo. Cuando aparezca el cuadro de diálogo de Instalación Deslice región adquisición , seleccione continuar.
- Cuando se le indique, seleccione correctamente el DAPI análisis para seleccionar la imagen de diapositiva de baja magnificación. Oprima OK. Del mismo modo, destacan la adquisición de placa Snap - DAPI cuando pidió el cierre de la adquisición de la placa. Haga clic en Aceptar.
- Cuando aparece el cuadro crear o regiones de carga , utilice la herramienta región Rectangular para seleccionar (a) región (es) de interés en la exploración de DAPI (figura 6). Presione continuar.
Nota: El área máxima que se puede seleccionar a 40 X es 45 columnas por filas de 35. Si se seleccionan varias regiones de interés, las regiones se redimensionará a la caja con mayor área. Las cajas pueden ajustarse después de cambiar el tamaño. - Seleccione No en el cuadro de diálogo de Carga de las regiones .
Nota: Seleccionando sí cargas guardan previamente las regiones para lotes de diapositivas con la misma región de lugares de interés. - Con la herramienta de localizador , destacan la región más grande de interés y pulse continuar en el cuadro de diálogo Seleccionar región . Cuando aparezca el cuadro de las regiones de confirmar , seleccione continuar. Decidir si la región debe guardarse cuando aparezca el cuadro de diálogo Guardar regiones .
- El siguiente cuadro de diálogo Sitio espacio, permite al usuario elegir el suelo de baldosa, que se traduce en una imagen sin superposición o traslapo de 10%. Seleccione una opción y haga clic en Aceptar. Los autores eligieron azulejos.
- Tres cuadros de diálogo aparecerá ahora. Puede ser despedida la caja Completa de configuración pulsando continuar. Mantener el cuadro de Configuración del sitio de adquisición para 40 X (Figura 7A) abierta por ahora ya que tiene claro cuántas columnas y filas que deba ser introducido en el cuadro de Configuración de adquisición de la placa (figura 7B). Mantener el cuadro de WellPositions (figura 7) que aparece en la esquina izquierda inferior abierto para la duración de la proyección de imagen.
- En el cuadro Configuración de placa de adquisición , en pozos de visita, debe ser el mismo número de pozos que las regiones de interés previamente seleccionados destacaron (figura 7).
Nota: Los autores desarrollaron este protocolo para un máximo de nueve regiones de interés por diapositiva. Si hay más, simplemente aumentar el número de columnas o filas en la pestaña de la placa - SlideScanning para reflejar el número de regiones de interés. Estos pozos no representan espacialmente la ubicación de las regiones de interés de cualquier manera, pero la cantidad debe ser resaltada (botón izquierdo del ratón). - En el cuadro Configuración de adquisición de la placa , vaya a la ficha de sitios para visitar entrar el sugerido columnas y filas (figura 7B). Recuerde hacer clic en sitios de azulejo.
- Para eliminar las conjeturas de encontrar los sitios que contiene la muestra, pulsa el Adquirir la placa bajo la pestaña de Resumen a la posición de la etapa en la región previamente seleccionada de interés. Una vez que la cámara ollas sobre un sitio que contiene células, presione Cancelar en la esquina derecha inferior para detener la adquisición de la imagen. Asegurar la etapa es ahora situado sobre la muestra.
- Optimizar la configuración de longitud de onda para asegurar que se obtiene un concentrado y una imagen de alto rango dinámico. Cuando esté satisfecho, haga clic en la ficha Resumen y Adquirir la placa.
7. Análisis de la imagen
- Seleccione el módulo personalizado deseado.
Nota: Los autores eligieron el módulo de Onda celular anotando para demostrar una segmentación automática de los portaobjetos de tumor de cerebro. - Ejecutar el análisis en todos los sitios. Si los parámetros de análisis son las mismas para muchas atracciones, el análisis puede incluirse en el cuadro Configuración de placa de adquisición en la ficha de Post adquisición .
- Una vez finalizado el análisis, excluir los sitios pobres de calidad de imagen para no sesgar los resultados. Puesto que el objetivo de este análisis es cuantificar la microglia y macrófagos en la parenquimia del tumor, seleccione la región de interés dentro de los bordes de la muestra de tumor. Además, excluir los sitios que están fuera de foco o contienen tejido pliegues o burbujas y desgarros en el tejido. Para una demostración, ver los Resultados de representante. Como alternativa, utilice la puntuación del enfoque láser generado para cada sitio dependiendo de la amplitud de la reflexión del láser como un umbral abajo sitios que deben ser excluidos (el umbral es personalizable y depende de parámetros como el tiempo de exposición y la tipo de medio utilizado).
Nota: Las imágenes de gran aumento permiten excluir específicamente estructuras como vasos sanguíneos y áreas de necrosis, si es necesario. También es posible incluir sólo las áreas de interés, como los que son hipercelular. Esta manera, la especificidad y la exactitud de los análisis pueden aumentar.
Resultados
Las imágenes pueden verse dentro del software WHCAS. La figura 8 muestra las miniaturas de alta magnificación de todos los sitios dentro de la región definida de interés. Revisión de cada sitio para identificar las que deben ser excluidas del análisis (ejemplos muestran aumento baja y alta en la figura 8 y figura 9, respectivamente). Por ejemplo, 149 sitio está fuera de foco (Figura 9A), página 219 tiene burbujas (figura 9B), y 54 del sitio contiene un doblez (figura 9) y deben ser excluidos. Es típico para excluir a 10-15% de todos los sitios de imagen. En el ejemplo proporcionado, 15,3% de los sitios fueron excluidos (25/300 tenía burbujas, 17/300 estaban fuera de foco, se plegaron 3/300 y 1/300 estaba en el borde). Figura 10A es una imagen representativa para análisis (su miniatura de ampliación baja correspondiente se muestra en la figura 8). Aquí, las plantillas correspondientes generadas por el módulo de onda celular que demuestran los resultados de la segmentación automatizada en el sitio 59, modificado para requisitos particulares a las especificaciones de los autores (anchura mínima de núcleos = 2.5 μm, ancho máximo = 7,5 μm, y la intensidad por encima del fondo local = 35 graylevels; Anchura mínima de las células CD11b-positivas = 4 μm, ancho máximo = 18 μm, mínimo manchado área = 15 μm2y la intensidad por encima del fondo local = 310 graylevels; y anchura mínima de las células CD45 positivo = 4 μm, ancho máximo = 18 μm, mínimo manchado área = 15 μm2y la intensidad por encima del fondo local = 50 graylevels). Tras la segmentación, los datos cuantitativos de la proporción de células de tinción positiva para cada marcador (6,2 ± 5,1% y 3,8 ± 2.1% de CD11b+ y CD45+ de las células, respectivamente), ambos marcadores (3,5 ± 2.1% CD11b+CD45+ las células), no hay marcadores (86,6 ± 9.0% CD11b–CD45 células– ; Figura 10B), medio manchado área (40,0 ± 9.2 μm y μm ± 36.7 7.6 de CD11b+ y CD45+ de las células, respectivamente; Figura 10) y significa la intensidad de la fluorescencia (408,9 unidades de fluorescencia relativa de ± 40.3 y 373,9 unidades de fluorescencia relativa de ± 38.1 de CD11b+ y CD45+ de las células, respectivamente; Figura 10) se puede obtener.
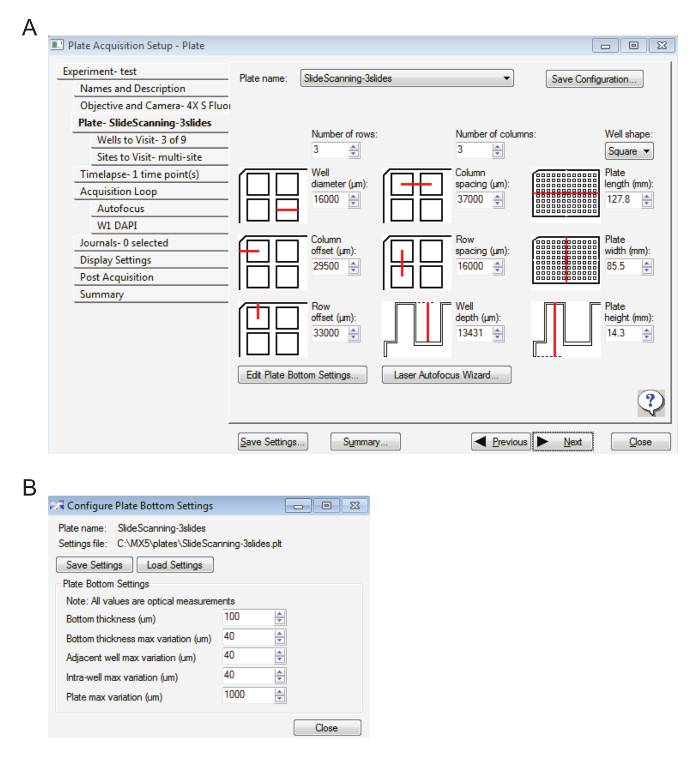
Figura 1: configuración para la adquisición de la diapositiva en la ampliación baja. A. esta imagen muestra la configuración de la placa. B., se muestran los valores de fondo de placa. Haga clic aquí para ver una versión más grande de esta figura.
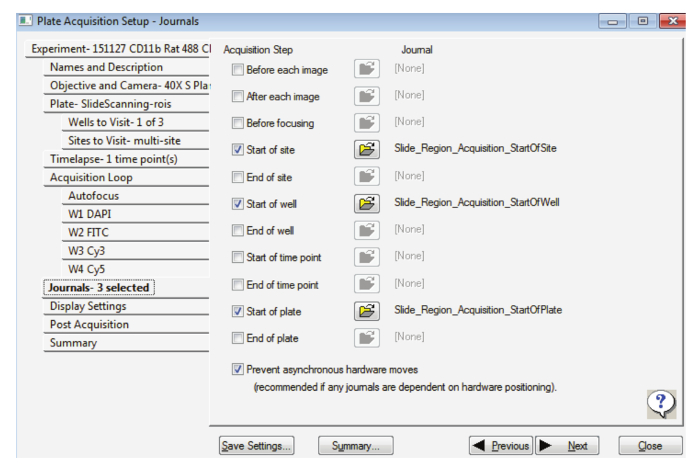
Figura 2: configuración para la adquisición de la diapositiva en gran aumento. Esta figura muestra la selección de las revistas correspondientes. Haga clic aquí para ver una versión más grande de esta figura.
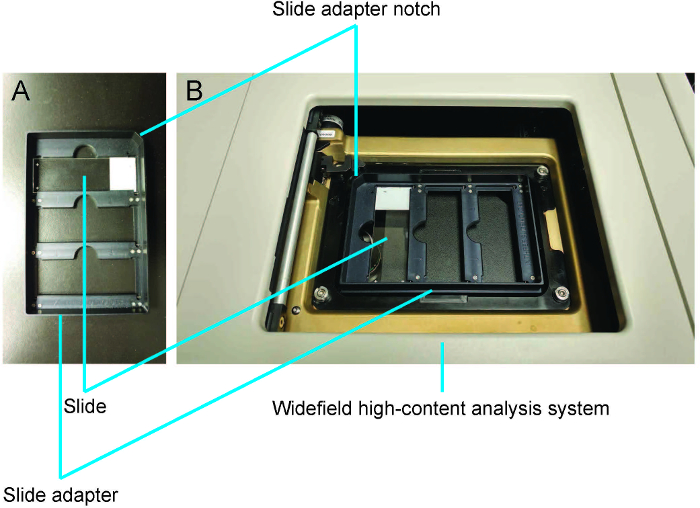
Figura 3: demostración de cómo cargar usando el adaptador de diapositivas diapositivas. A. la diapositiva se coloca el cubreobjetos hacia abajo con la etiqueta en el lado de la ranura del adaptador de diapositivas. B. Deslice el adaptador se carga en el sistema de análisis de alto contenido de campo amplio. Haga clic aquí para ver una versión más grande de esta figura.
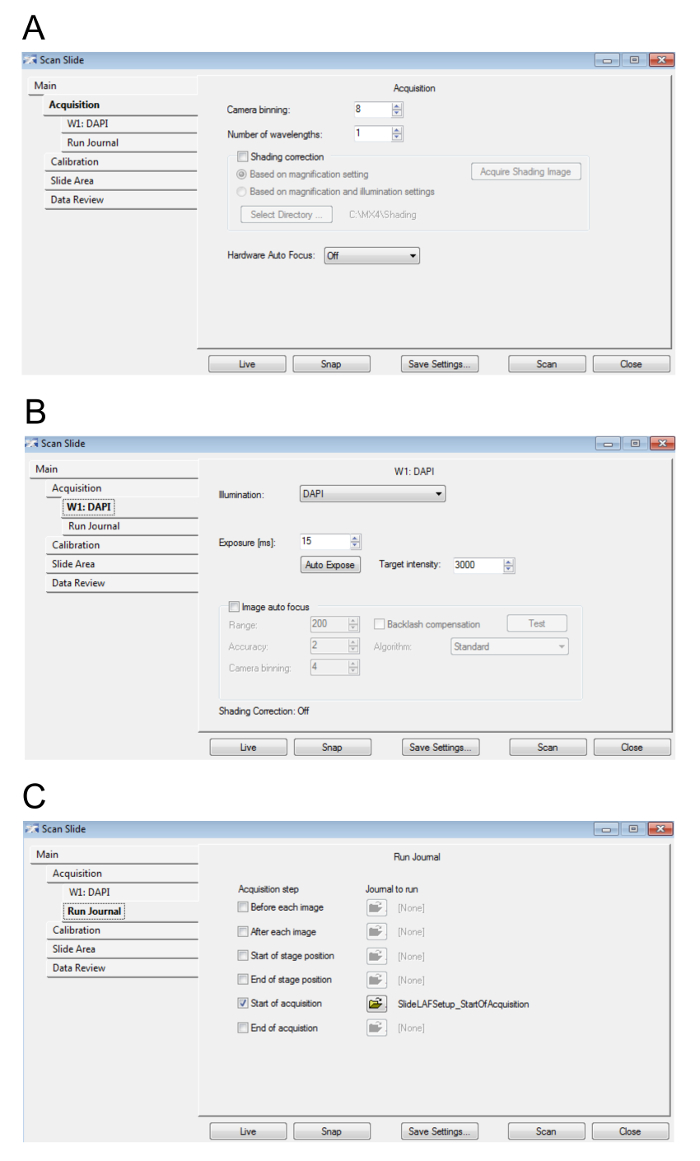
Figura 4: configuración de diapositivas vista previa. A. esta figura muestra los parámetros para la adquisición. B., se muestran los valores para los parámetros de longitud de onda de Hoescht/DAPI. C. esta imagen muestra los ajustes de diario. Haga clic aquí para ver una versión más grande de esta figura.
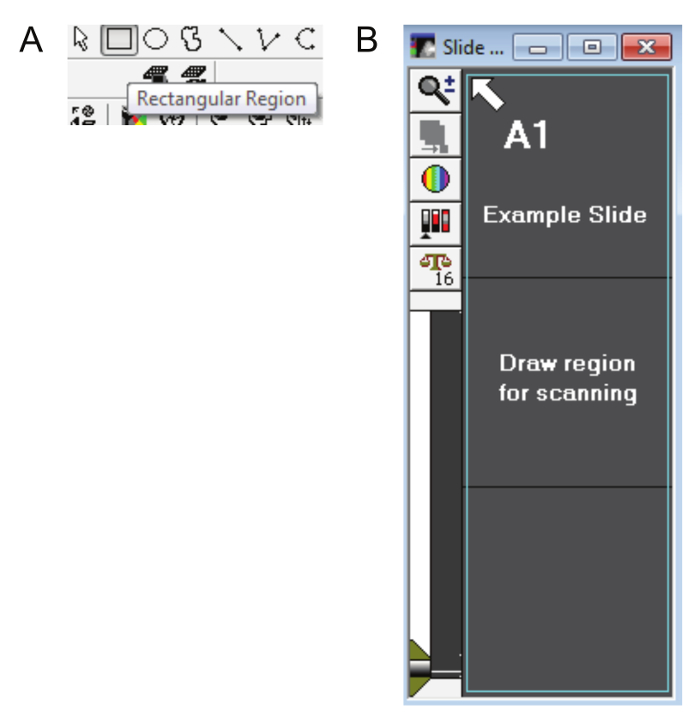
Figura 5: dibujo una región diapositiva. A. la herramienta región rectangular (no pueden utilizarse otras herramientas de dibujo) se utiliza para seleccionar el área de exploración de vista previa y crear regiones de interés en la exploración del DAPI. B. el contorno del teal en esta figura muestra cómo se debe seleccionar el área de la diapositiva (incluyendo la etiqueta). Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: creación de regiones de interés en la exploración de la vista previa. La vista previa o escanear DAPI representa el área de la diapositiva entera. En este ejemplo, hay tres secciones de tejido de cerebro serial (los contornos rectangulares blanco sólidos). El área por encima de estas secciones representa las etiquetas circulares en la diapositiva. Arreglos de sección diferente no limitará la selección posterior de las regiones de interés. La región de interés en este ejemplo concreto se muestra con un contorno rectangular blanco discontinuo. El scalebar = 1 mm. haga clic aquí para ver una versión más grande de esta figura.
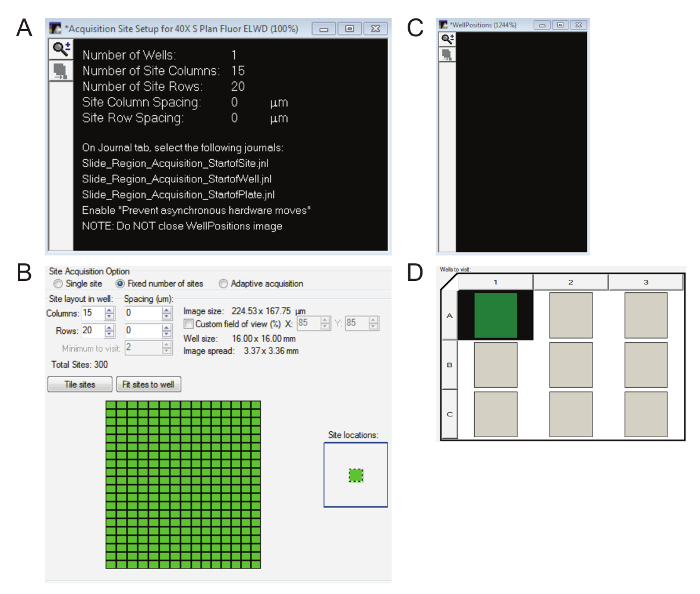
Figura 7: adquisición de la imagen de windows esenciales para alta magnificación. A. ventana de configuración de sitio de la adquisición muestra cuántas columnas y filas dentro de la región o regiones de interés. B. garantizar el número correcto de columnas y filas indicadas en la Figura 7A se introducen en el cuadro configuración de la adquisición de placa y los sitios son de cerámica. C. es necesario mantener la ventana WellPositions abierta para la duración de la adquisición de la imagen aunque sea en blanco. D. debe destacarse el número apropiado de las regiones de interés. Haga clic aquí para ver una versión más grande de esta figura.

Figura 8: imagen representativa de la región de interés. Cada sitio puede mostrarse en una miniatura compuesta de la región de interés. Los sitios representativos que han sido excluidos (marcados con rojo cajas) y un ejemplo de un sitio incluido (marcado con un cuadro verde) se muestra en un aumento mayor. Se recomienda revisar cada sitio para asegurarse de que es de calidad suficiente para el análisis. El scalebar = 1 mm. haga clic aquí para ver una versión más grande de esta figura.
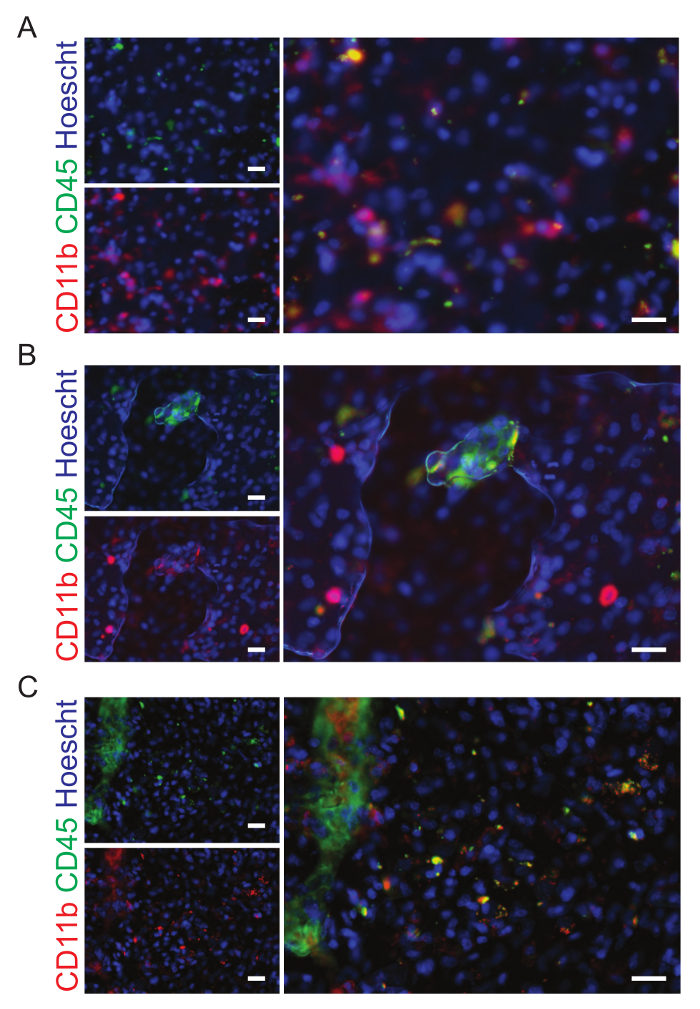
Figura 9: ejemplos de sitios que deben excluirse del análisis. A. las secciones del tumor de cerebro humano han sido manchadas con CD11b (verde) y CD45 (rojo), marcadores de microglia y macrófagos. Esta imagen está fuera de foco y se ha excluido del análisis. B. burbujas están presentes en esta imagen que ha sido excluida del análisis final. C. Este sitio fue excluido del análisis por el pliegue en el tejido. Las barras de escala son 20 μm. haga clic aquí para ver una versión más grande de esta figura.
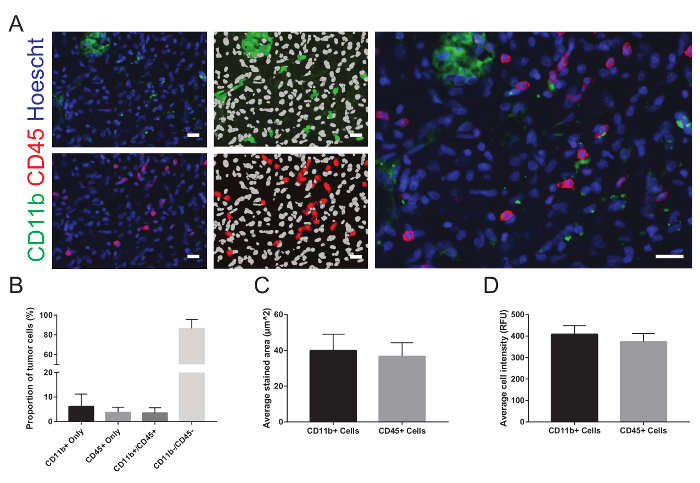
Figura 10: imágenes representativas y análisis. A. cada imagen aparece junto a las plantillas que representan los resultados de la segmentación automatizada. En el recubrimiento, los núcleos aparecen en blanco y positivamente células aparecen en verde para CD11b y rojo para CD45. Después de excluir los sitios de calidad inadecuada del análisis y combinación de los resultados de todos los sitios restantes, B. la proporción media del total de células en cada sitio que mancharon el positivo para cada marcador y conjuntamente con la etiqueta para ambos marcadores, C. la media había manchada de área y D. la intensidad media de la célula son representados gráficamente. RFU = unidades de fluorescencia relativa. Los datos se expresan como media ± desviación estándar. Las barras de escala son 20 μm. haga clic aquí para ver una versión más grande de esta figura.
Discusión
Un alambique de problema comunes que obstaculizan la eficacia de la investigación en ciencias biológicas es el desarrollo de protocolos para la cuantificación objetiva, exacta y precisa de los tejidos marcada con fluorescencia y sus estructuras. Cantidades significativas de tiempo y esfuerzo se dirigen hacia encontrar maneras de analizar diapositivas de tejidos una vez que ha sido reflejadas. Muchos métodos existentes proporcionan algoritmos para que los usuarios recrear dentro de programas12,13,14. Estos métodos son aceptables, pero la importancia de este informe es que permite al usuario establecer fácil y rápidamente una imagen holística adquisición y Protocolo de análisis si el usuario tiene acceso a un WHCAS. Ensayos que diferencian tipos de la célula y cuantifican varias estructuras y procesos, análisis de ciclo celular y translocación nuclear, por ejemplo, ya están disponibles en el software asociado.
La configuración implica algunos pasos críticos. En primer lugar, se definen los parámetros espaciales de la diapositiva como si fuera un plato. En segundo lugar, se crea un análisis Resumen de ampliación baja de que las regiones de interés se seleccionan para la proyección de imagen de alta magnificación. Por último, se excluyen los sitios que afectan la exactitud y precisión de los análisis posteriores. La mayor limitación de esta técnica es que su aplicabilidad depende de si el usuario tiene acceso a un WHCAS. Sin embargo, con la creciente necesidad de sistemas de análisis de alto contenido, muchas instituciones están ofreciendo a sus investigadores a permanecer competitivo15. Solución de problemas es necesario más comúnmente cuando las secciones de tejido no tienen el mismo grosor. Si se seleccionan varias regiones de interés, algunos estarán enfocados mientras que otras no. Idealmente, durante el seccionamiento, el usuario cuidaría para crear muestras homogéneas. Sin embargo, si las muestras son inconsistentes, el foco en la longitud de onda de tinción nuclear (o fluoróforo más brillante del usuario), que se utiliza para enfocar, puede reajustarse para cada región fuera de foco de interés e incluso para cada campo de visión. Simplemente se compensan las otras longitudes de onda de la longitud de onda de tinción nuclear, solamente esta longitud de onda necesita reajuste.
En este informe, detallamos cómo escanear y analizar diapositivas utilizando un WHCAS y software asociado. El módulo de onda de la célula que le permite contar automáticamente cualquier marcadores nucleares o citoplasmáticos fluorescencia etiquetadas. Después de ajustar los ajustes de enfoque y definir las características celulares como el ancho y área para personalizar el módulo al imagen del tejido, no hay necesidad de intervención del usuario obtener las diapositivas de imagen y datos cuantitativos. Hasta tres diapositivas puede ser reflejada en un momento y se pueden definir múltiples regiones de interés. Permite protocolo WHCAS los usuarios que necesitan analizar diapositivas aprovechar personalizable, multiusos, automatiza flujos de trabajo que no requieren poca o ninguna optimización y pueden ser aplicados en cualquier proyecto en el futuro que involucran el análisis histológico del tejido.
Divulgaciones
Los autores tienen intereses financieros en los productos descritos en este manuscrito y tienen nada más que revelar.
Agradecimientos
Este proyecto fue financiado por una subvención de los institutos canadienses de investigación en salud y Alberta Innovates - salud soluciones/Alberta Cancer Foundation. Los autores desean reconocer a la unidad de regeneración en instalaciones centrales de Neurobiología para el uso de su equipo, el trabajo de Paula Gedraitis en la construcción de la Fundación sobre la que Deslice la exploración en el WHCAS fue posible y el creador de los productos mencionados en este artículo, dispositivos moleculares.
Materiales
| Name | Company | Catalog Number | Comments |
| ImageXpress MicroXLS | Molecular Devices | NA | Apparatus for image acquisition |
| MetaXpress 5.1 | Molecular Devices | NA | Associated software for ImageXpress MicroXL (runs on a PC with the Windows operating system). |
| Slide adapter | Molecular Devices | NA | Metal slide holder that fits into ImageXpress MicroXL |
| Slide_Region_Acquisition_revA.jzp | Molecular Devices | NA | The journal can be obtained from metamorph.moleculardevices.com/forum/showthread.php?tid=218&highlight=slide or from contacting a Molecular Devices representative |
| Slide_Region_Acquisition _Setup.JNL | Molecular Devices | NA | Select this journal in Step 6.6. |
Referencias
- Hua, Y., Shun, T. Y., Strock, C. J., Johnston, P. A. High-content positional biosensor screening assay for compounds to prevent or disrupt androgen receptor and transcriptional intermediary factor 2 protein-protein interactions. Assay and Drug Development Technologies. 12 (7), 395-418 (2014).
- Rishal, I., et al. WIS-NeuroMath enables versatile high throughput analyses of neuronal processes. Developmental Neurobiology. 73 (3), 247-256 (2013).
- Kanungo, J., Lantz, S., Paule, M. G. In vivo imaging and quantitative analysis of changes in axon length using transgenic zebrafish embryos. Neurotoxicology and Teratology. 33 (6), 618-623 (2011).
- Schurmann, C., et al. Analyzing illumina gene expression microarray data from different tissues: methodological aspects of data analysis in the MetaXpress Consortium. PLoS ONE. 7 (12), e50938 (2012).
- Vogt, A., Codore, H., Day, B. W., Hukriede, N. A., Tsang, M. Development of automated imaging and analysis for zebrafish chemical screens. Journal of Visualized Experiments. (40), e1900 (2010).
- Bravo-San Pedro, J. M., et al. High-throughput quantification of GFP-LC3+ dots by automated fluorescence microscopy. Methods in Enzymology. , 71-86 (2017).
- Varga, V. S., et al. Automated multichannel fluorescent whole slide imaging and its application for cytometry. Cytometry Part A. 75 (12), 1020-1030 (2009).
- Narayan, P. J., et al. Assessing fibrinogen extravasation into Alzheimer's disease brain using high-content screening of brain tissue microarrays. Journal of Neuroscience Methods. 247, 41-49 (2015).
- Du, Y., Budman, H. M., Duever, T. A. Segmentation and quantitative analysis of apoptosis of Chinese hamster ovary cells from fluorescence microscopy images. Microscopy and Microanalysis. 23 (3), 569-583 (2017).
- Mueller, J. L., et al. Quantitative segmentation of fluorescence microscopy images of heterogeneous tissue: application to the detection of residual disease in tumor margins. PLoS One. 8 (6), e66198 (2013).
- Donnelly, D. J., Gensel, J. C., Ankeny, D. P., van Rooijen, N., Popovich, P. G. An efficient and reproducible method for quantifying macrophages in different experimental models of central nervous system pathology. Journal of Neuroscience Methods. 181 (1), 36-44 (2009).
- Kozlowski, C., Weimer, R. M. An automated method to quantify microglia morphology and application to monitor activation state longitudinally in vivo. PLoS One. 7 (2), e31814 (2012).
- Fish, K. N., Sweet, R. A., Deo, A. J., Lewis, D. A. An automated segmentation methodology for quantifying immunoreactive puncta number and fluorescence intensity in tissue sections. Brain Research. 1240, 62-72 (2008).
- Zock, J. M. Applications of high content screening in life science research. Combinatorial Chemistry & High Throughput Screening. 12 (9), 870-876 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados