Method Article
Disección de la función del reforzador mediante Multiplex basados en CRISPR potenciador de interferencia en líneas celulares
En este artículo
Resumen
Este protocolo describe los pasos necesarios para diseñar y realizar targeting multiplexados de potenciadores con la desactivación proteína de fusión SID4X-dCas9-Cascarudo, también conocido como interferencia de enhancer (potenciador-i). Este protocolo permite la identificación de los reforzadores que regulan la expresión génica y facilita la disección de las relaciones entre los potenciadores de regulación de un gen de destino común.
Resumen
Múltiples potenciadores a menudo regulan un determinado gen, sin embargo, para la mayoría de los genes, no queda claro qué Reforzadores para expresión génica, y cómo estos potenciadores se combinan para producir una respuesta transcripcional. Como millones de potenciadores han sido identificados, se necesitan herramientas de alto rendimiento para determinar la función de mejorador en una escala genoma-ancha. Los métodos actuales para estudiar la función del reforzador incluyen hacer supresiones genéticas utilizando el dominio nucleasa Cas9, pero es difícil estudiar los efectos combinatorios de múltiples potenciadores usando esta técnica, como múltiples líneas de células clonales sucesivas deben estar generados. Aquí, presentamos Enhancer-i, un método basado en la interferencia de CRISPR que permite la interrogación funcional de múltiples potenciadores simultáneamente en sus locus endógenos. Enhancer-i hace uso de dos dominios represivos fusionados a nucleasa-deficiente Cas9, SID y KRAB, para lograr la desactivación de reforzador mediante desacetilación de las histonas en loci específicos. Este protocolo utiliza transitorios de la transfección de guía RNAs para permitir que la inactivación transitoria de regiones específicas y es particularmente eficaz en bloquear las respuestas transcripcionales inducibles a estímulos en lugares de cultivo de tejidos. Reforzador es altamente específico tanto en su orientación genómica y sus efectos sobre la expresión génica global. Resultados obtenidos en este protocolo ayudan a comprender si un reforzador está contribuyendo a la expresión génica, la magnitud de la contribución, y cómo el aporte se ve afectado por otros potenciadores cercano.
Introducción
Proyectos como codificar1, Roadmap Epigenomics2y FANTOM de secuenciación a gran escala3 han identificado millones de supuestos potenciadores dentro del genoma humano a través de cientos de tipos celulares. Se estima que cada promotor se asocia con un promedio de 4,9 potenciadores y cada reforzador entra en contacto con un promedio de 2,4 genes3, sugiriendo que la expresión génica es a menudo el resultado de la integración de múltiples interacciones reguladoras distribuidas. Un reto importante restante es definir no sólo como individuales potenciadores contribuyen a la expresión génica, pero cómo combinan para afectar la expresión. Enfoques genéticos se utilizan para identificar las relaciones entre reforzadores en organismos modelo de Drosophila4 ratones5. Sin embargo, estos experimentos son desperdiciadoras de tiempo y de bajo rendimiento para el estudio de múltiples potenciadores en múltiples genes.
Un enfoque para estudiar la función del reforzador a gran escala implica ensayos reportero masivamente paralelo. Estos ensayos permiten la detección simultánea de miles de secuencias de ADN por su capacidad para la expresión de un gen reportero del6. Mientras que estos ensayos han demostrado que secuencia de ADN solo puede ser suficiente para transmitir el gen Reglamento información7, vienen con las salvedades de ser realizada fuera del contexto de la cromatina nativa y con un promotor heterólogo. Además, el tamaño de la secuencia de la DNA están analizando en ensayos reportero masivamente paralelo suele basepairs menos de 200, que puede excluir la pertinente secuencia circundante. Lo importante, como ensayos reportero sólo medir la actividad de una secuencia a la vez, no toman en cuenta las complejas relaciones que pueden existir entre reforzadores. Así, mientras que ensayos reportero masivamente paralelo pueden ser informativos sobre la actividad intrínseca de una secuencia de ADN, ellos no necesariamente nos informan de la función de esa secuencia de ADN en el contexto del genoma.
Recientemente desarrollados CRISPR/Cas9 herramientas8 han facilitado el estudio de la regulación génica, ya que permiten la supresión de los reforzadores en el locus endógeno. Sin embargo, eliminar reforzadores múltiples al mismo tiempo puede conducir a inestabilidad genómica y es lento para generar deleciones sucesivas potenciador en una línea celular única. Además, nueva secuencia genómica se crea en el sitio de la eliminación después de la reparación, y esta secuencia puede tener función reguladora. Una versión alternativa de Cas9 ha sido desarrollada específicamente para la modulación de la expresión génica, confiando en fusiones de9,10 de activación o represión de11,12 dominios a nucleasa-deficiente forma de Cas9 (dCas9). Estas proteínas de fusión son ideales para el estudio de múltiples loci simultáneamente como físicamente no alterar la secuencia del ADN y en su lugar modular Epigenética para interrogar a una región reguladora. La fusión represiva más ampliamente utilizada es KRAB, que recluta el KAP1 represor Co complejo, promover la deposición de la represión asociado histona H3 lisina 9 trimetilación (H3K9me3)13. dCas9-Cascarudo, también conocido como CRISPR interferencia14ha sido utilizado para blanco y pantalla individuales potenciadores para sus contribuciones a la expresión de gene de15,16; sin embargo, no ha sido optimizado para apuntar varias regiones al mismo tiempo. Una versión de multiplex interferencia CRISPR para potenciadores, seq mosaico17, usa sola célula RNA-seq como una lectura, pero esta tecnología es costosa y sólo es adecuado para el estudio de genes altamente expresados debido a la baja sensibilidad de la célula RNA-seq.
Se intentó desarrollar un método basado en la interferencia de CRISPR para disección función combinatoria potenciador en el contexto de una respuesta transcripcional a los estrógenos. Aproximadamente la mitad de genes estrógeno-responsivos contienen potenciadores 2 o más obligados por la alfa del receptor de estrógeno (ER) cerca de18, sugiriendo que múltiples potenciadores se pueden participar en la respuesta al estrógeno y entender que la lógica reguladora requeriría dirige a reforzadores múltiples simultáneamente. Como estudios iniciales mediante interferencia CRISPR en promotores sugieren que promotores no todos responden igual a represión mediada por KRAB19, razonamos que la adición de un dominio represivo distinto a dCas9 puede facilitar la desactivación de diversos potenciadores. Elegimos el Sin3a interacción dominio de Mad1 (SID)20 ya que conduce al reclutamiento de las histona deacetilasas21, que quitan grupos acetil de las histonas que se asocian con actividad transcripcional. Importante, el dominio SID fue eficaz para reducir la expresión del gen cuando fusionan dCas922 y23de la relatos y Sin3a ha demostrado ser un potente factor de cooperación represivo en una variedad de contextos de secuencia enhancer24. Se utilizó SID4x-dCas9-KRAB (Enhancer-i) a 10 diferentes potenciadores por el ER e identifican los sitios de unión de ER (ERBS) que son necesarios para la respuesta transcripcional de estrógeno a los 4 genes18. Hemos dirigido también las combinaciones de potenciadores para identificar los sitios que cooperan en la producción de la respuesta transcripcional de estrógeno. Hemos encontrado que hasta 50 sitios pueden potencialmente ser objeto simultáneamente con los cambios de expresión genética detectable. Uso de ChIP-seq y RNA-seq, demostramos que el reforzador es una técnica altamente específica para estudiar simultáneamente múltiples potenciadores.
En este protocolo se describen los pasos involucrados en la realización de reforzador-i, una técnica flexible que permite el estudio funcional de reforzadores múltiples simultáneamente en un entorno de cultivo de tejidos. Enhancer-i está correlacionada altamente con la canceladura genética pero proporciona desactivación transitoria que depende de las histonas deacetilasas (HDACs). Entregar guía RNAs mediante transfección transitoria frente a integración estable a través de vectores virales, este protocolo evita la deposición y difusión potencial de H3K9me3. Este diseño de guía RNA detalles de protocolo y clonación vía Asamblea de Gibson, la transfección de guían RNAs usando lipofection, y el análisis de la expresión génica resultante cambia por qPCR. También se incluyen los métodos para evaluar la especificidad del reforzador-i dirigido a nivel de genoma y transcriptoma. Mientras que esta técnica se desarrolló para estudiar la regulación génica por ER límite potenciadores en líneas celulares de cáncer humano, es aplicable a la disección de cualquier mamífero enhancer.
Protocolo
1. generación de la célula líneas expresando estable SID4X-dCas9-KRAB
Nota: Las condiciones de transfección y concentraciones de la droga aquí presentadas han sido optimizadas para las células de Ishikawa, una línea celular de cáncer de endometrio, cultivadas en Medio RPMI 1640 suplementado con 10% FBS y 1% penicilina/estreptomicina (RPMI completo). Otras líneas celulares pueden requerir condiciones diferentes de la transfección y concentraciones de la droga. Los usuarios también pueden realizar experimentos de transfección transitoria en células de tipo salvaje, en lugar de generar una línea celular estable, con un plásmido que expresa SID4X-dCas9-KRAB junto con RNA guía expresando plásmidos; sin embargo, los resultados de transfecciones transitorias pueden ser difíciles de reproducir como SID4X-dCas9-KRAB niveles pueden variar por transfección.
- Placa de células Ishikawa en por lo menos 2 pocillos de una placa de 6 pozos en confluencia de 30-50% (aproximadamente 300.000 células de Ishikawa) en 3 mL de RPMI completo.
- Aspire los medios de comunicación de las células. Lavar las células una vez con PBS 1 x (pH 7.4). Aspire el PBS y añadir tripsina (4 mL para un plato de 10 cm o 5 mL para un frasco de T-75).
- Incube las células ~ 5 minutos a 37 º C, comprobando cada 2 min para que las células separadas y sacudiendo suavemente el vaso.
- Una vez que las células se han separado, pipeta tripsinizaron células arriba y abajo unas cuantas veces y pipeta suavemente hacia abajo el lado de la embarcación para liberar las células adjuntas.
- Transferir las células a un tubo cónico de 15 mL y centrifugar las células abajo durante 5 min a 250 x g.
- Aspirar la tripsina y resuspender las células en 5-10 mL de medio. Utilice una pipeta P1000 para disociar grupos de células si es necesario.
- Contar las células y determinar el volumen necesario para ~ 300.000 células por pocillo en un volumen total de 3 mL de la placa. Añadir las células a 2 pocillos distintos de una placa de 6 pozos. Llene cada uno bien a 3 mL de RPMI completo.
- Agitar suavemente la placa cada 5 min en los primeros 15 minutos después de la galjanoplastia para asegurarse de que las células se distribuyen uniformemente en la placa. Utilizar un microscopio para asegurarse de que las células se han dispersado desde la mitad del pozo.
- Dentro de las 24 h de la galjanoplastia, realice los siguientes transfecciones utilizando un reactivo de transfección adecuada para la línea celular de interés. Para Ishikawa las celdas, utilice el procedimiento como se describe a continuación.
Nota: Este protocolo asume que el uso de reactivos de transfección basada en liposomas catiónicos. Electroporación proporciona un método alternativo para tipos de células que son muy sensibles a los reactivos o que exhiben eficacia de transfección baja con lipofection. Condiciones de transfección deben optimizarse para la línea celular de interés antes de intentar experimentos Enhancer-i.- En un tubo Eppendorf de 1,7 mL, diluir 2,5 μg de plásmido SID4X-dCas9-KRAB y 800 ng de plásmido expresan una proteína fluorescente en medio libre de suero que el volumen final del tubo es de 155 μl y la concentración final de plásmido es 0,020 μg/μl.
- En otro tubo, diluir 3,3 μg de un plásmido que contiene una cinta de resistencia a neomicina, como pCMV-GFP, en medios sin suero tal que el volumen final del tubo es de 155 μl y la concentración final de plásmido es 0,020 μg/μl.
- Vortex cada tubo brevemente y girar hacia abajo usando una microcentrífuga.
- Añadir 9,9 μl del reactivo de transfección (Tabla de materiales) a cada tubo. Mezclar con un vórtex brevemente a baja velocidad. Girar los tubos hacia abajo con una microcentrífuga.
- Incubar los tubos a temperatura ambiente durante al menos 5 minutos pero no más de 20 minutos.
- En el gabinete de bioseguridad, añadir 150 μL de la mezcla de ADN preparado: reactivo gota a gota a un pozo en la placa de la pozo 6. Repita para el otro tubo de mezcla de ADN preparado: reactivo. Las placas se mezclan agitando suavemente y vuelva la placa a la incubadora.
- En la transfección de post del día 2, cambiar los medios de comunicación y complementar con G418 a una concentración final de 600 ng/μL. Esta concentración puede necesitar ser optimizado para el tipo de célula.
- Cambiar medios RPMI completo y complementar con G418 cada tercer día durante 2-4 semanas hasta que las células transfectada de control están muertas y los pozos que contienen SID4X-dCas9-KRAB convertirse en confluentes. La cantidad exacta de tiempo necesario para que recuperarse de las células dependerá el tiempo de duplicación de las células.
- Cuando las células se convierten en confluente, paso a un buque T-25 o T-75 en completan RPMI con una dosis menor de G418 (300 ng/μL de células de Ishikawa). Durante este paso, hacer 2 alícuotas de células ~ 100.000 cada (aproximadamente 1/10th de una placa de 6 pozos) en tubos de Eppendorf de 2 mL 1,7 separado para el aislamiento de ARN y ADN, respectivamente. Girar los tubos hacia abajo (min 5, 250 g de x), eliminar tripsina mediante pipeteo y congelar los tubos a-20 ° C para su uso futuro.
- Aislar ADN genómico utilizando kits comerciales disponibles y realizar PCR utilizando el "pAC95_PCR" o "SID4X_PCR" iniciadores (tabla 1) para verificar la presencia de la proteína de fusión dentro de la línea celular. Utilizar la DNA genomic extraída de la línea parental como un control negativo y SID4x-dCas9-KRAB plásmido ADN como control positivo. Utilice una mezcla maestra de polimerasa de alta fidelidad con 50-100 ng de ADN genómico y las siguientes condiciones de ciclismo: 98 ° C por 30 s, 25 ciclos de (98 ° C por 10 s, 58 ° C por 30 s, 72 ° C por 2 min), 72 ° C por 5 min. , mantener a 4 ° C.
- Para comprobar la expresión de la proteína de fusión a nivel de RNA, realizar qPCR con ARN extraído de la línea celular usando kits disponibles en el mercado. Uso de los iniciadores de la "dCas9_qPCR" (tabla 1), y el protocolo de qPCR de paso en paso 6.3 del presente Protocolo.
- Para comprobar la expresión del nivel de la proteína de la fusión, realizar un Western blot en lisados de la línea celular. Uso de anti-FLAG o anti-HA para detectar la proteína de fusión.
2. Guía ARN diseño
Nota: Este protocolo está diseñado para uso con el RNA de la guía de U6 clonación vector creado por el laboratorio de la iglesia y está disponible en Addgene (Addgene 41824). Para crear una versión de este vector que contienen resistencia puromicina que permitió la clonación misma estrategia como 41824, nos mudamos el sitio de clonación múltiple de este vector en el vector pGL3-U6-sgRNA-PGK-puromicina (Addgene 51133). 41824 Addgene o nuestra versión con puromicina (Addgene 106404) son compatibles con la estrategia de clonación se detallan a continuación.
- Obtener 600-900 basepairs de secuencia de la DNA para cada región reguladora de interés. Use sitios de unión del factor de transcripción o accesibilidad de la cromatina para orientación en cuanto a cómo definir la región de interés (figura 2A).
Nota: Mientras que en el ejemplo en la figura 2A cuenta con potenciadores de aguas arriba y aguas abajo, también es posible orientar elementos reguladores en intrones. - Colocar todas las secuencias obtenidas en un archivo de solo texto con el formato FASTA.
- Identificar por lo menos una región de control negativo que no se espera cambiar en condiciones experimentales, como promotor de un gen que se expresa no en la línea celular de interés. Obtener la secuencia de la DNA para esta región y agregar en el archivo de texto en formato FASTA.
Nota: Utilizamos guía RNAs a IL1RN promotor25 como un control negativo para todas las regiones que apuntamos. Los usuarios también pueden seleccionar secuencia intergénica cerca de la región de interés que no contienen sitios de unión del factor de transcripción como control negativo. Sin embargo, si se apunta simultáneamente múltiples loci, una región de control negativo solo simplifica el diseño experimental e interpretación de resultados. Si el objetivo el reforzador es intronic, puede ser útil a una región intronic en el mismo lugar geométrico que contiene un elemento regulador putativo como control negativo adicional, como la fusión de dCas9 puede interferir con la transcripción de destino. - Identificar regiones de control positivo, como los promotores que son los supuestos blancos de las regiones reguladoras de interés, o promotores de los genes que se transcriben muy en la línea celular de interés. Obtener la secuencia de la DNA para estas regiones y añadir en el archivo de texto en formato FASTA.
- Utilizar un programa como fresco e26 (http://www.e-crisp.org/E-CRISP/) en las secuencias de ADN generadas para encontrar a guía RNAs con baja de-objetivos (idealmente 0-3). Guía RNAs constan de 20 nucleótidos corriente arriba de un protospacer motivo adyacente (PAM), que toma la forma "NGG" para la dCas9 de S. pyogenes.
- En el sitio web de e-patata a la inglesa, seleccionar el organismo de interés utilizando el menú desplegable. La Asamblea del genoma aparece a la derecha del nombre de la especie.
- Seleccione el botón de entrada es secuencia FASTA . Copiar las secuencias FASTA desde arriba y pegarlos en el cuadro de diálogo. Asegurar que un encabezado FASTA se incluye para cada secuencia.
Nota: Hasta 50 secuencias pueden consultar simultáneamente. - Seleccione el botón de radio medio y único diseño en el menú desplegable.
- Haga clic en el botón Iniciar búsqueda sgRNA. Se abrirá una nueva pestaña del navegador y se mostrará los resultados. Descargar las secuencias de candidatos haciendo clic en el botón descargar un Excel con formato tabular para todas las secuencias de consulta juntas.
- Abra el informe tabular usando Excel o un programa de edición de texto.
- Utilizar el navegador del genoma UCSC a BLAT candidato gRNA integral secuencias (23 basepairs) del genoma.
- En un navegador, vaya a la Web de browser del genoma UCSC (http://genome.ucsc.edu). En la sección nuestras herramientas, localizar la palabra BLAT y haga clic en él. Se abrirá la herramienta de búsqueda BLAT.
- Utilice los menús de lista desplegable situados bajo el texto BLAT búsqueda genoma para seleccionar el conjunto de organismo y genoma de interés.
- Copiar las secuencias de RNA de la guía del informe tabular generado por e-crisp y pégalos en el cuadro de diálogo. Asegúrese de que cada secuencia tiene un único encabezado FASTA, a continuación, haga clic en Enviar en la parte inferior del cuadro de diálogo.
Nota: Hasta 25 secuencias pueden ser examinadas a la vez. En la página de Resultados de la búsqueda BLAT , aparecerá alineaciones de cada guía secuencia de ARN, con cada línea que representa una alineación. Idealmente, debe existir una alineación para cada guía de RNA, indicando la singularidad de que RNA de la guía. - Evitar a las guías que alinearán a varios lugares en el genoma, si es posible.
- Para examinar la guía RNA localización y distribución dentro de la región de interés, haga clic en el enlace del navegador en la sección de acciones de uno de los RNAs guía consultado. El navegador de genoma aparecerá y estará centrado en la guía de las RNA. Utilice los botones de Zoom out en la parte superior de la página para visualizar la distribución de otra guía RNAs identificados por e-quebradizo dentro de la región de interés.
- Seleccione 4 preferiblemente sin traslapo guía secuencias de ARN que se distribuyen en toda la región de interés (figura 2B). Si la región de interés excede 600 bp, considerar la adición de guías adicionales de 1-2. Evite guía RNAs con estiramientos homopolymeric y extrema contenido de GC, ya que estas características pueden impedir el ARN guía proceso de clonación y reducir RNA guía a eficiencia.
- Una vez que las guías han sido seleccionadas, crear un archivo que contiene a la guía completa secuencia de ARN (23 nucleótidos) para cada guía deseada y luego elimina el nucleótido 5', así como la PAM (NGG) desde el extremo 3'. Este paso facilita ordenar oligo.
- Agregue la siguiente secuencia en el extremo 5' de la secuencia del oligonucleótido: GTGGAAAGGACGAAACACCG.
- Agregue la siguiente secuencia en el extremo 3' de la secuencia del oligonucleótido: GTTTTAGAGCTAGAAATAGC.
Nota: La secuencia final debe 59 nucleótidos largo y mira como esta: GTGGAAAGGACGAAACACCG-blanco (19 nt)-GTTTTAGAGCTAGAAATAGC. - Asegúrese de que cada elemento regulador para orientarse con potenciador-i tiene al menos 4 oligonucleótidos únicos diseñados para él. Ordenar estas secuencias junto con los iniciadores de la "U6_internal" aparece en la tabla 1.
3. Guía de clonación de ARN
Nota: RNA guía clonación vía Asamblea de Gibson ha demostrado para ser altamente eficiente en nuestras manos, produciendo cientos de colonias por placa, con escasas colonias presentes en el único control del vector. Tal eficiencia es fundamental para mantener la complejidad durante la clonación combinada. Otra ventaja de Asamblea de Gibson la clonación es que los usuarios no tienen que preocuparse por la presencia de una enzima de restricción corta el sitio en la guía de RNA están tratando de insertar en el vector de clonación de U6. Sin embargo, este protocolo puede ser adaptado para la enzima de restricción tradicional basado en la clonación si se desea.
- Reconstituir la guía RNA oligos a una concentración final de 100 μM en agua ultrapura (libre de Rnasa, DNasa-libre). Debe haber al menos 4 guía separada RNA oligos para cada región de interés.
- Para cada región reguladora de interés, crear una agrupación de todos los oligos correspondientes a la región de interés. En un tubo Eppendorf, se combinan 5 μL de cada oligo de RNA guía reconstituido individuales para cada región. La piscina se mezclan bien con un vórtex, entonces retire 1 μL y diluir esta alícuota 1: 200 en agua ultrapura.
Nota: Si lo desea, estas piscinas dirigidas a las regiones reguladoras individuales más combinables para generar una complejo piscina dirigidas a varias regiones. Hasta 50 regiones reguladoras pueden orientarse simultáneamente en una única agrupación (figura 3). - Realizar una PCR corto con los iniciadores del U6 para unir regiones de homología a los oligos antes del montaje de Gibson. Cerca de 40 bases se añadirá a cada oligo, que rinde un producto de bp de ~ 100 que contiene suficiente homología al vector U6 en ambos extremos.
- Para cada guía piscina de RNA, configurar un 20 μL de PCR con una mezcla maestra de polimerasa de alta fidelidad y los siguientes componentes: 1 μL de piscina oligo diluido del paso 3.2, 1 μL del primer avance de U6 (10 μM), 1 μL de U6 inversa primer (10 μM) y hasta 20 μL de agua.
- Incubar en un termociclador con las siguientes condiciones: 98 ° C por 30 s, 10 ciclos de (98 ° C por 10 s, 55 ° C por 30 s, 72 ° C por 2 min), 72 ° C por 5 min y mantener a 4 ° C.
- Ejecutar 5 μL de la reacción en un gel de agarosa al 1-2% con una escala de bajo peso molecular. El producto final debe ser ~ 100 basepairs (figura 2).
- Limpie la reacción de extensión con un kit de purificación de ADN basado en la columna y eluir en 20 μL de tampón de elución proporcionada en el kit.
Nota: Como el producto es corto, evitar el uso de base de grano de limpiezas, diseñados para excluir fragmentos pequeños de menos de 100 bp. - Cuantificar el ADN purificado con un fluorómetro o espectrofotómetro (espera que el rendimiento es de 10-20 ng/μL). Guía RNA puede ser almacenado a-20 ° C, o se puede utilizar inmediatamente en la Asamblea de Gibson con un vector linearizado de U6.
- Para preparar al receptor vector de clonación U6 para montaje de Gibson, creó una síntesis de la enzima de la restricción. Si muchas reacciones de ensamblaje de Gibson deben realizarse, configurar múltiples resúmenes para asegurar una producción suficiente de vectores de corte.
- Utilice 20 unidades de enzima AflII y 1 μg de plásmido en una reacción de 20 μL con el tampón de enzima de restricción apropiada. Incubar a 37 ° C durante 1-2 h.
- Limpiar el resumen con granos o un kit de base de columna para la purificación de ADN y eluir en 20 μL de tampón de elución. Cuantificar el ADN purificado usando un fluorómetro o un espectrofotómetro. Las muestras pueden congelarse a-20 ° C para su uso posterior.
- Realizará el montaje de Gibson en el vector preparado e inserte.
- Establecer las reacciones de ensamblaje de Gibson en el hielo. Uso 50 ng de vector y 7 ng del inserto en una reacción de 20 μL. Diluir los insertos de 1:10 en agua ultrapura para facilitar el pipeteo. Crear una vector único Gibson Asamblea la reacción, usando 50 ng de vector y reemplazar el inserto con agua.
- Incubar las reacciones de ensamblaje de Gibson durante 15 min a 50 ° C, seguido por un asimiento a 4 ° C.
- Transferencia de los productos ensamblados de hielo. Diluir los productos ensamblados 1:4 en agua ultrapura en hielo. Por ejemplo, añadir 5 μL del producto de Gibson Asamblea a 15 μL de agua ultrapura.
- Transformar los productos de montaje diluidos de Gibson.
- Descongelar células competentes de alta eficiencia en el hielo y hacer 25 alícuotas μL para cada transformación. Si se desea una piscina complejo dirigido a múltiples sitios, descongelar suficientes células en diferentes tubos para realizar múltiples transformaciones independientes de la misma agrupación de gRNA complejo.
- Para cada producto diluido, añadir 1 μL de esta dilución a un 1,7 mL tubo Eppendorf conteniendo 25 μL de células competentes. Mezclar brevemente agitando el tubo. Incubar los tubos en hielo durante 30 minutos.
- Calor del choque las células para 30 s a 42 ° C, luego se transfieren inmediatamente hielo por 2 minutos.
- Añadir 300 μL SOC los medios de comunicación (2% triptona, extracto de levadura 0.5%, 10 mM NaCl, 2,5 mM KCl, 10 mM MgCl2, 10 mM MgSO4y 20 mM de glucosa) y dejar las células recuperar por 1 h a 37 ° C con agitación (300 rpm). Durante este tiempo, caliente las placas de agar con ampicilina/carbenicilina a 37 ° C en una incubadora. Utilizar una placa para cada transformación.
- 50 μL de las células de la placa y colocar las placas en una incubadora de 37 ° C durante la noche. Para las piscinas dirigidas a sitios individuales, colocar directamente en 3-5 mL de caldo LB (Tabla de materiales) que contienen ampicilina/carbenicilina (1 mg/mL) e incubar durante la noche con agitación a 250 rpm a 37 ° C para minipreps.
- Cosecha de las células y aislar ADN.
- Para bibliotecas de guía grandes RNA dirigidas a múltiples sitios, use un raspador de placa para recoger todas las colonias de cada placa individual en un maxiprep (cultivo líquido 150 mL). Esto puede facilitarse mediante colada ~ 5 mL de LB con el antibiótico apropiado en un tubo falcon de 50 mL y raspado de las colonias en el tubo. Para las bibliotecas dirigidas a sitios individuales, quitar las placas en un miniprep (cultivo de líquido de 3 a 5 mL).
- Incubar estas culturas con el antibiótico apropiado para el 3-5 h a 37 ° C con agitación a 250 rpm.
- Realizar la extracción de ADN usando un kit que se traduce en preparaciones libres de endotoxinas.
- Cuantificar el ADN utilizando un espectrofotómetro o un Fluorímetro. Plásmidos pueden utilizarse inmediatamente en la transfección o almacenados a-20 ° C para su uso futuro.
- Para confirmar la presencia de la secuencia de ARN guía dentro del vector de U6 para pequeñas piscinas dirigidas a sitios individuales, Sanger secuenciación en la miniprep preparado con la cartilla de "U6_PCR_R" aparece en la tabla 1. Debido a la puesta en común de guía RNAs, la secuencia de destino 19 basepair gRNA cederá bases mixtas, pero el promotor U6 y andamio de RNA guía que rodea esta secuencia debe estar intactos.
4. transfección de reforzador-i
Nota: Para el bloqueo exitoso de una respuesta de estrógeno con potenciador-i en células de Ishikawa, es necesario privar a las células de estrógeno por 5-7 días antes de la transfección por mantener en rojo de fenol libre RPMI con 10% carbón despojado de FBS y 1% penicilina/estreptomicina. Las células se deben cultivar en este medio durante y después de transfección si tratando de bloquear una respuesta de estrógeno. Recomendamos el uso de la tripsina libre de rojo de fenol para paso de células en rojo fenol completa gratis RPMI.
- El día antes de la transfección, la placa de las células (tipo salvaje o expresando estable SID4X-dCas9-KRAB) en un plato 24-well en confluencia de 30-50% (~ 60.000 células por pozo para células de Ishikawa). Placa de suficientes células que transfecciones pueden realizarse por duplicado e incluyen pocillos para ser transfectadas con la guía de control de RNAs. Asegúrese de que las células se distribuyen uniformemente en el pozo agitando suavemente la placa después de galjanoplastia de la célula como en el paso 1.1.7.
Nota: Este protocolo asume que el uso de reactivos de transfección basada en liposomas catiónicos. Electroporación proporciona un método alternativo para tipos celulares que son muy sensibles a los reactivos. Condiciones de transfección deben optimizarse para la línea celular de interés antes de intentar experimentos Enhancer-i. - Al día siguiente, preparar transfecciones según las instrucciones del reactivo de transfección de elección. Para Ishikawa las células, uso 550 ng de plásmido total para cada pocillo de una placa de 24 pocillos. Diluir la plásmidos a una concentración final de 0,020 μg/μL en medios sin suero (1.1 μg de DNA en 52 μL de volumen total para transfectar 2 pozos). Utilizar 3 μL de reactivo de transfección para cada 1 μg de DNA, vortex e incubar como se describe en el paso 1.2. Añadir 25 μL de la mezcla final a cada pocillo.
Nota: A las combinaciones de objetivo de sitios, utilice el mismo peso del plásmido para cada sitio individual y después llenar el peso sobrante con un plásmido de control (RNA guía vacío clonación vector o guía RNAs dirigidos a una región de control negativo como el IL1RN promotor). De transfecciones transitorias, utilizan una relación de plásmido de 3:2 Cas9 fusión: Guía RNA. Plásmidos que contienen reporteros fluorescentes pueden agregarse para controlar eficacia de transfección. - En la transfección de 36 h post, cambiar los medios de comunicación usando rojo de fenol libre RPMI con 10% carbón despojado FBS y 1% penicilina/estreptomicina (para células de Ishikawa) y fuente de puromicina (concentración final: 1 μg/mL) y neomicina (concentración final: 300 ng/mL). Si las células son sensibles a reactivos de transfección, los medios de comunicación se pueden cambiar antes, pero deben añadirse antibióticos no antes de transfección de correos 24 h.
Nota: Espere al menos 24 h después de agregar el antibiótico antes de la cosecha de las células. Cambios de expresión debido a i el reforzador pueden ser detectados como temprano como 48 h post transfección y hasta 5 días post transfección. Si trabaja con células de Ishikawa que han sido privadas de estrógeno, realizamos un 8 h 10 nM 17β-estradiol (E2) de inducción el día después del tratamiento antibiótico y luego cosechar células inmediatamente.
5. cosecha y extracción de ARN de la célula
- Preparar el tampón de lisis con 1% β-mercaptoetanol (BME). Asegúrese de que hay bastante mezcla de lisis-BME (300 μL para cada bien se cosecha).
- Aspire los medios de comunicación utilizando un aspirador de vacío.
- Lavar las células una vez con un volumen igual de PBS 1 x (500 μL) y aspirada para eliminar tanto PBS como sea posible.
- Añadir 300 μL de solución de lisis-BME a cada pocillo con una pipeta multicanal. Pipetear la solución de lisis para arriba y abajo 8 - 10 veces y la transferencia a una placa de la pozo profundo o 1,7 mL Eppendorf tubos en hielo. RNA puede extraerse inmediatamente o lisados pueden ser congelados a-80 ° C para el procesamiento futuro.
- Para extraer RNA de lisados, utilice un kit disponible comercialmente que incluye un tratamiento de DNasa. Responsables del menor volumen recomendado de agua ultrapura (libre de Rnasa, DNasa libre) o elución de búfer y cuantificar RNA. Para una pequeña cantidad de muestras, utilice un fluorómetro o un espectrofotómetro. Para un gran número de muestras, utilice una sonda fluorescente que se detecta ARN y medida en un lector de placas. Las muestras se pueden congelar a-80 ° C antes de o después de la cuantificación.
6. cuantificación de cambios de expresión génica utilizando qPCR de un solo paso y RNA-seq
- Obtener qPCR iniciadores para los genes de interés y para gen de limpieza al menos una que se expresa cerca del nivel de los genes específicos y no cambia a través de condiciones experimentales. Idealmente, estos iniciadores desarrollará en un empalme exón-exón para evitar la amplificación de la DNA genomic.
- Prueba estos cebadores de RNA Obtenido de la línea celular de interés. Uso del derretimiento análisis de la curva para verificar la producción de un solo producto. Si no se produce un solo producto, prueba de pares de imprimación adicional.
- Para cada guía RNA tratado muestra Enhancer-i y control, identificar cuántos genes deben ensayarse en esa muestra. Este conjunto de genes debe incluir genes housekeeping, como CTCF o GAPDH.
- Establecer las reacciones de la qPCR.
- Diluir todas las muestras a la misma concentración en el agua, tal que 50 ng de RNA total se pipetea fácilmente y hay bastante diluido RNA para cada reacción. Por ejemplo, diluir RNA a ~16.6 ng/μL y 3 μL del ARN en cada reacción. Mantenga el RNA en el hielo mientras mezclas principales.
- Preparar mezclas Masters independientes para cada gen con kits de qPCR comercialmente disponibles de un solo paso. Utilice un volumen de reacción de 20 μL con 1 μL de cada primer (solución stock de 10 μM). Establecer estas reacciones en el hielo.
- En una placa de reacción que es apropiada para el termociclador, agregar las muestras de RNA seguidas de mezcla principal. Sellar con un sellador de placas y mezclar suavemente por Vortex o pipeteando. Centrifugar brevemente la placa (140 x g durante 60 s) para ese líquido está en el fondo de los pozos.
- Incube la placa en un termociclador como sigue (o como kit manda): 48 ° C por 30 min, 95 ° C durante 10 min, 40 ciclos de (95 ° C por 15 s, 60 ° C por 1 min).
- Obtener los valores de Ct de cada gen medidos en cada muestra. Utilice el método de Ct comparativo para identificar los cambios en la expresión génica.
- Reste del Ct del gen housekeeping del TC de cada gen de interés para cada muestra para generar valores normalizados de la Ct.
- Para las muestras de control de tratamiento, tomar un promedio de los valores normalizados de la Ct para cada gen. Log base 2-escala doble represión entonces se puede calcular restando la muestra normalizada de Enhancer-i tratado Ct para cada gen del mismo valor para la muestra control tratado para el gen correspondiente.
- Para determinar los cambios globales en la expresión de gene Enhancer-i tratamiento, preparar las muestras para la secuencia de RNA utilizando un kit comercial compatible con la tecnología de secuenciación del usuario. Uso ~ 500 ng de ARN para el material de partida y se preparan bibliotecas para al menos 2 réplicas biológicas.
7. verificación de genómica específica dirigida a por SID4X-dCas9-KRAB usando ChIP-seq
Nota: La proteína de fusión SID4X-dCas9-KRAB contiene una etiqueta de bandera del epítopo y una etiqueta de epítopo HA, pero se obtuvieron mejores resultados para el ChIP-seq con los anticuerpos de anti-FLAG. Si lo desea, el usuario puede realizar experimentos adicionales de ChIP-seq para factores de transcripción potencialmente afectados por reforzador-i o H3K27ac, una marca de actividad de reforzador que es disminuida por el reforzador-i. Sin embargo, cada experimento de ChIP-seq requiere 10 x 106 células, así que plan por consiguiente.
- Transfectar las células con piscinas Enhancer-i.
- Placa de 10 x 106 células en un plato de cultivo de tejidos de 15 cm. Cada plato representa 1 experimento de ChIP-seq para 1 factor de interés.
- Al día siguiente, transfectar las células con 20 μg de DNA total por plato. Para transfecciones en líneas celulares de expresión estable de SID4X-dCas9-KRAB, el ADN debe ser una piscina de plásmido de guía RNAs dirigidos a todos los sitios de interés y, opcionalmente, un plásmido que expresan la proteína fluorescente. De transfecciones transitorias, utilizan una relación de piscina de RNA de la proteína: Guía de 3:2 dCas9 fusión. Para ChIP-seq de otros TFs o modificaciones de las histonas, realizar al menos un control adicional guía RNA la transfección en otro plato.
- Tratar los platos con puromicina (1 μg/mL) y neomicina (300 ng/mL) en la transfección de 24-48 h post. Espere al menos 24 h antes de la cosecha de la cromatina.
- Cromatina de cosecha de los platos.
Nota: Para estudiar los efectos del reforzador-i en la Unión de genomic de ER en las células de Ishikawa, realizar un tratamiento de 1 h 10 nM E2 en platos transfected con la guía de control de RNAs y potenciador-i antes a la cosecha. Para estudiar los efectos del reforzador-i en H3K27ac, realizar un nM 8 h 10 E2 inducción en platos transfected con la guía de control de RNAs y potenciador-i antes a la cosecha.- 500 μL de formaldehído al 37% se aplican a cada plato (concentración final de 1%). Agitar brevemente las placas. Deje las placas a temperatura ambiente durante 10 minutos.
- Añadir 1 mL de glicina de 2,5 M (concentración final de 125 mM). Agitar brevemente las placas.
- Retirar de los medios de comunicación con formaldehído y glicina. Añadir un volumen igual (20 mL) de PBS 1 x fría.
- Retirar el PBS. Aspirado con un aspirador de vacío para quitar tanta PBS como sea posible. Coloque los platos en el hielo.
- Añadir 3-5 mL de frío 1 x PBS o Farnham tampón de lisis (pH de 5mM tubos 8.0, 85 mM KCl, 0.5% NP-40) con 1 x inhibidor de la proteasa (Añadida justo antes de usar) para cada placa. Raspar el plato con un raspador con una placa y transferir la solución a un tubo cónico de 15 mL en hielo.
- Cromatina de pellets por giro por tubos en la centrífuga por 5 min a 4 ° C a 1000 x g. descartar el sobrenadante y almacenar los pellets a-80 ° C para su uso futuro o continuar con el protocolo ChIP-seq de elección mediante un anticuerpo de anti-FLAG o anticuerpos dirigidos a otros factores de transcripción o modificaciones de las histonas de interés (H3K27ac, H3K9me3).
Resultados
La figura 1 muestra un esquema del flujo de trabajo descrito en el protocolo. Para determinar las contribuciones de los potenciadores de ER-limite cerca del gen regulado por estrógenos MMP17, que cuenta con 3 sitios de Unión cerca como se define en ChIP-seq (figura 2A), guían RNAs fueron diseñadas para cada región. Diseñar guía RNAs, una ventana de 600-900 bp de secuencia que rodea cada ER sitio de unión de intereses fue seleccionado y puesto en un programa de diseño de RNA guía. Dando como resultado la guía de secuencias de RNA con 0-2 predicha de destino sitios fueron alineados con el genoma humano con BLAT. Cuatro no superpuestos guía RNAs que abarcó la región definida por ChIP-seq y DNaseI hipersensibilidad fueron escogidos para dirigir (figura 2B). Secuencia adicional (tabla 1) se añadió a cada extremo para facilitar la clonación aguas abajo y el resultado 59 fragmentos de nucleótidos se les ordenó. A su llegada, guía RNAs se diluye y agrupados por sitio y un PCR corto fue realizada para añadir regiones de homología antes del montaje de Gibson. Figura 2 se muestra el producto de RNA guía esperado después de un corto PCR utilizando los iniciadores de la "U6_internal" (tabla 1), que se sumarán 20 basepairs de secuencia a cada extremo del basepair 59 guía fragmento de RNA, lo que resulta en una secuencia del basepair ~ 100. Tras Asamblea de Gibson, estas piscinas guía RNA se transformaron en bacterias y plásmidos minipreps fueron preparados al día siguiente. Figura 2D muestra resultados de un experimento de disección de reforzador, donde múltiples potenciadores cerca MMP17 se dirigen solos y en combinación con potenciador-i. Sitios blanco de reforzador-i se indican con un hexágono negro. Plásmidos de RNA guía dirigida a los sitios indicados fueron transfectadas en una línea de celular de Ishikawa privación de estrógeno expresando estable SID4X-dCas9-KRAB. Dos días más tarde, los medios de comunicación fue cambiado y puromicina fue agregado para enriquecer las células transfected. Al día siguiente, las células se cosecharon siguiendo un tratamiento de estradiol 8 h 10 nM. RNA fue aislado, y se realizó una qPCR de un solo paso. En este ejemplo, los sitios 1 y 2 son necesarios para una completa respuesta estrogénica de MMP17, mientras que el sitio 3 no contribuye en estas condiciones (Figura 2D, carriles ii-iv). Cuando están activos sólo los sitios 2 ó 3 (vi y vii), la respuesta del estrógeno es similar a cuando no hay sitios están activos (viii), lo que sugiere que estos sitios no pueden contribuir de forma independiente. Sitio 1 puede contribuir a una expresión por sí misma (v), pero la mayor actividad se observa cuando los sitios 1 y 2 están activos (iv).
Para manipular 10 potenciadores cerca de 4 genes diferentes simultáneamente (Figura 3A), complejo piscinas de guían RNAs se generaron que contiene 42 guías de potenciador y 16 guías de promotor. Guía RNA oligos se agruparon antes la guía inicial extensión de RNA PCR (paso 3.3), y productos PCR obtenidos fueron purificados y combinados con el vector de clonación de U6 de puromicina vacíos usa Gibson. Tras la Asamblea de Gibson, múltiples transformaciones independientes realizaron y plateadas. Las placas fueron raspadas en LB y permitir crecer durante 2-4 h antes maxiprep. Figura 3B muestra representativa reducción en la expresión génica por qPCR cuando estas piscinas de RNA de la guía fueron transfectadas en una línea de celular de Ishikawa privación de estrógeno expresando estable SID4X-dCas9-KRAB y tratadas como se describe anteriormente (Figura 2D). Reducciones de reforzador-i son similares a los obtenidos al atacar el promotor del gen putativo objetivo. Figura 3 muestra los efectos de dilución de guía RNAs en la reducción de la respuesta de estrógeno con potenciador-i. Un 1:50 dilución de una piscina de guía RNA objetivo el reforzador cerca de G0S2 todavía produce una reducción significativa en la expresión génica, sugiriendo que Enhancer-i puede utilizarse para dirigirse hasta 50 sitios a la vez. Sin embargo, la desactivación puede ser diluida, lo que indica que cientos de sitios no pueden dirigirse simultáneamente a menos que se emplean métodos de detección más sensibles.

Figura 1. Protocolo de manual de reparacion para disección potenciador multiplex con potenciador-i. Guía RNAs (rojos y azul) se diseñan utilizando e-patata a la inglesa y seleccionados usando el browser del genoma UCSC. Se eligen cuatro guía RNAs que abarcan las regiones de interés (transcripción factor sitios de Unión según lo definido por ChIP-seq). Oligonucleótidos de RNA de la guía que han sido agrupados por la región de interés (rojo y azul) se someten a una PCR para agregar regiones de homología (naranja) antes de la Asamblea de Gibson y la transformación. Piscinas de plásmido resultante son transfectadas por lipofection en líneas celulares de expresión estable de SID4X-dCas9-Cascarudo o en células de tipo salvaje junto con el plásmido SID4X-dCas9-KRAB. Guía RNA plásmido piscinas pueden transfected individualmente para llegar a un sitio, o en combinación para atacar varios sitios simultáneamente. Transfected las células se tratan con antibióticos para enriquecer de las células que contienen guía RNAs. En la transfección de post ~ 72 h, las células se cosechan. Los ácidos nucleicos se pueden extraer para qPCR, RNA-seq o ChIP-SEQ haga clic aquí para ver una versión más grande de esta figura.
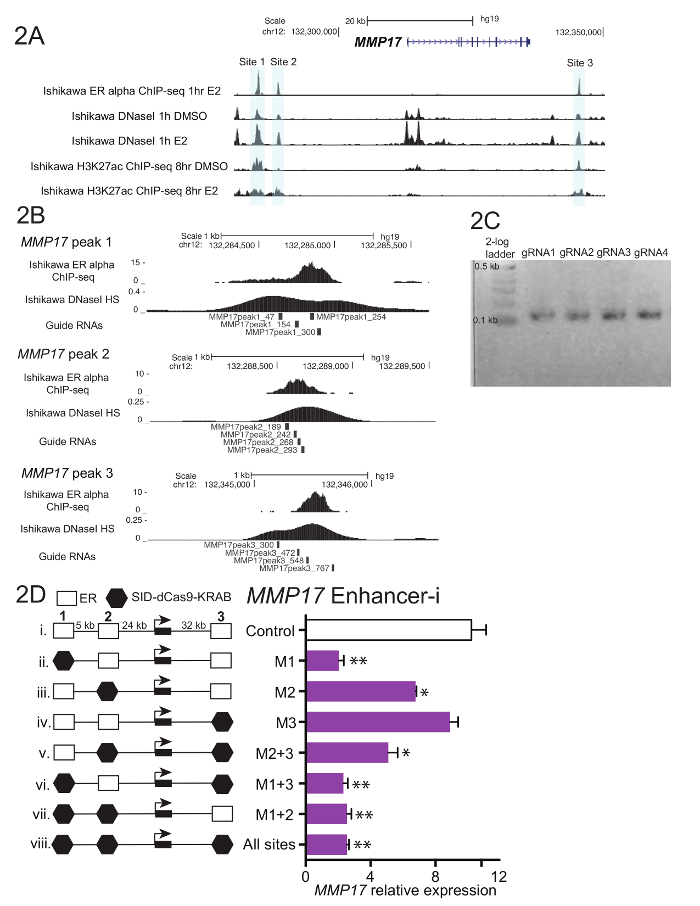
Figura 2. Guía de diseño y reforzador de la disección de RNA para MMP17. (A) captura de pantalla de navegador de genoma de los potenciadores de la alfa-limite de ER (gris) a cerca de MMP17. Esta figura ha sido modificada de Carleton, et al. 18. (B) guía de ARN se diseña para el 3 enlace sitios18. El sitio de Unión para ER como se define en ChIP-seq es el destino y el 4 guía RNAs azulejo en toda esta región. La señal de sensibilidad DNaseI, que abarca el sitio de Unión, también puede ser utilizada para definir la secuencia de destino para guía de diseño de RNA. ChIP-seq y datos DNaseI capítulo se obtuvieron de Ishikawa las células tratadas con estradiol 10 de nM para secuencias de ARN de guía representativa de (C) de 1 h. que están listos para el montaje de Gibson, haber sufrido una PCR corto para agregar regiones de homología. (D) expresión relativa de MMP17 medido mediante qPCR raíz dirigidas a regiones específicas con potenciador-i y un tratamiento de estradiol nM 8-h10. Expresión corresponde al nivel CTCF y la expresión de MMP17 en células no tratadas con estradiol. Guía de control de RNAs de destino el promotor de IL1RN. Todas las barras de error representan SEM, doble asterisco indica p < 0.01 y solo los asteriscos indican p < 0.05 en una prueba de t pareada. Esta figura ha sido modificada de Carleton, et al. 18. haga clic aquí para ver una versión más grande de esta figura.
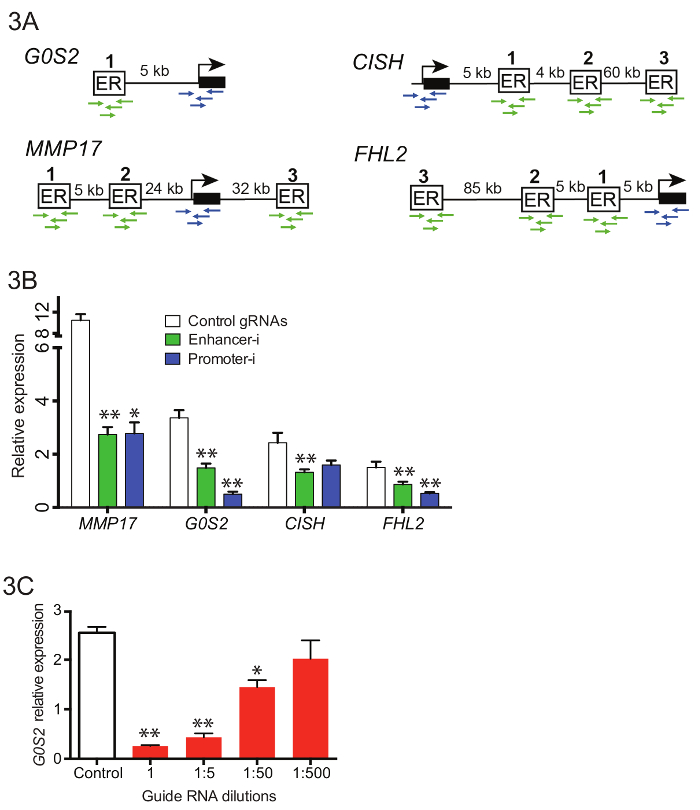
Figura 3. Dirigido a múltiples potenciadores cerca genes diferentes simultáneamente con combinaron Enhancer-i. (A) esquema de los sitios de Unión y los promotores para orientarse en combinado potenciador-i. (B) los efectos sobre la expresión según lo medido por qPCR después tratamiento E2 en Ishikawa las células transfected con piscina de plásmido de reforzador-i (verde), piscina de plásmido de promotor-i (azul) o control gRNAs (blanco)18. Se observa una reducción significativa en todos los genes con potenciador-i. Esta figura fue modificada de Carleton, et al. 18. (C) los efectos en los niveles de expresión de G0S2 después del tratamiento de E2 en células de Ishikawa transfected con diferentes cantidades de guía RNAs dirigidas a G0S2. Se aprecia una reducción significativa incluso con pequeñas cantidades de guía RNA (1:50 dilución), lo que sugiere que hasta 50 sitios puede dirigido simultáneamente. Todas las barras de error representan SEM, doble asterisco indica p < 0.01 y solo los asteriscos indican p < 0.05 en una prueba de t pareada. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre | Secuencia de |
| U6_internal_F | TTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCG |
| U6_internal_R | GACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC |
| U6_PCR_F | CCAATTCAGTCGACTGGATCCGGTA |
| U6_PCR_R | AAAAAAAGCACCGACTCGGTGCCA |
| gRNA_qPCR_F | GCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG |
| gRNA_qPCR_R | AAAAAGCACCGACTCGGTGCC |
| dCas9_qPCR_F | GTGACCGAGGGAATGAGAAA |
| dCas9_qPCR_R | AGCTGCTTCACGGTCACTTT |
| pAC95_PCR_F | AGAAGAGAAAGGTGGAGGCC |
| pAC95_PCR_R | CGTCACCGCATGTTAGAAGG |
| SID4X_PCR_F | CAATAGAAACTGGGCTTGTCG |
| SID4X_PCR_R | TCGTGCTTCTTATCCTCTTCC |
Tabla 1. Cebadores utilizados para extensión de RNA guía y secuencia, qPCR y detección de la proteína de fusión.
Discusión
Este protocolo describe un método simple y flexible para disección función del reforzador en el locus genómico endógena sin alterar físicamente la secuencia del ADN. Mientras que es similar en concepto a los previamente publicados protocolos de interferencia CRISPR usando dCas9 KRAB27, potenciador-i diferencia de estos protocolos de 3 maneras principales. En primer lugar, potenciador-i utiliza el dominio interacción SIN3A de MAD120 para lograr la desactivación de potenciador. Desactivación de Enhancer puede rescatar mediante inhibidores de las HDACS, sugiriendo que el principal mecanismo de desactivación es dependiente de las HDACS. A diferencia de la interferencia CRISPR dCas9 KRAB, potenciador-i no conduce a la deposición de H3K9me3. Esto es probable debido a que i potenciador depende introducción transitoria de guía RNAs, con las células se cosechan a los 3 días post transfección. En interferencia CRISPR, se observa un aumento de H3K9me3 en 7 días post transducción12. Finalmente, el protocolo de reforzador-i ofrece una estrategia para atacar simultáneamente varios sitios y monitorear la eficiencia de la focalización. En seq mosaico17, dCas9 KRAB es objetivo reforzadores múltiples simultáneamente, pero esta técnica se basa en la secuencia de RNA unicelular para identificar cambios de expresión, y muchos genes (por ejemplo de genes estrógeno-responsivos) pasan desapercibidos debido a la baja sensibilidad de unicelular RNA-SEQ Enhancer-i proporciona un método confiable para estudiar potenciadores individualmente y en combinación para cualquier gen.
El paso más crítico del reforzador-i es la transfección, que debe ser optimizada para la línea celular de interés. Este protocolo se basa en tratamiento de puromicina para enriquecer las células transfected, pero es posible que co transfecting guía RNAs con una proteína fluorescente y clasificación de células fluorescentes mediante citometría de flujo puede llegar a ser un mejor método de enriquecimiento para algunos celulares tipos. Se recomienda control de la expresión a nivel de guía RNAs y SID4x-dCas9-KRAB por qPCR para solucionar problemas y confirmar la transfección. Si los niveles guía RNA son bajos (ciclo umbral > 30), los usuarios también pueden considerar guía alternativa estrategias de producción de RNA como en vitro transcripción28. También es posible que a pesar de niveles altos de gRNA, RNA guía dirigida a de la proteína SID4x-dCas9-KRAB es ineficiente, en cuyo caso seleccionando diferentes guía de secuencias de ARN puede ser necesario. Al realizar el ChIP-seq de la proteína de fusión con la cromatina de células Enhancer-i Tratado, la eficacia de la focalización puede controlarse. Si hay señal de alto de SID4x-dCas9-KRAB en la región de interés, y cambios de expresión en su gen blanco supuesta no se detectan, la región probablemente no contribuyen a la expresión de este gen bajo las condiciones estudiadas.
Una limitación potencial del reforzador-i es que se pueden acumular efectos off-target si muchos sitios están dirigidos al mismo tiempo. Sin embargo, estrategias de interferencia CRISPR para caída tienen menos efectos objetivo de ARNi29, particularmente cuando se utiliza una línea celular policlonales de expresando dCas9 KRAB. Si bien hemos visto objetivo vinculante genómica de SID4X-dCas9-KRAB cuando 10 sitios al mismo tiempo, no hemos identificado cambios de expresión génica como consecuencia de los eventos de enlace. Como podrán en contacto con algunos reforzadores múltiples promotores y potenciadores de otros, es posible que muchos genes pueden cambiar la expresión en contra de un solo reforzador, aunque no está claro si esta forma de regulación génica es común. Para confirmar que los cambios de expresión observaron son debido a la orientación un potenciador específico y no objetivo efectos, los usuarios pueden realizar Enhancer-i con dos conjuntos distintos de no superposición guía RNAs a la misma región. Además, la supresión genética de la región utilizando Cas9 nucleasa competente puede confirmar aún más sus efectos sobre la expresión génica.
Como funciones de reforzador-i a través de desacetilación de histonas, es posible que sus capacidades de desactivación se limitan a reforzadores que tienen apreciables niveles de acetilación de histonas. Hay una gran variedad de fusiones represivas alternativa que puede ser más eficaz en el objetivo específicos potenciadores. Fusiones de metiltransferasa de ADN a dCas9 pueden utilizarse para reducir la expresión del gen cuando a distal potenciadores30, pero esta represión no es a menudo transitoria. Otra fusión represiva utiliza el dominio amigo de GATA1 (FOG1), que conduce a la histona H3 lisina 27 trimetilación y reprime la expresión del gen en niveles similares a dCas9 KRAB a través de una variedad de célula líneas y promotores31. Curiosamente, añadiendo más copias de FOG1 a dCas9 reduce el potencial represivo en promotores, sugiriendo que un solo ejemplar del dominio SID puede proporcionar más desactivación de potenciador de los 4 ejemplares utilizados actualmente en Enhancer-i. Es posible que algunos loci pueden beneficiarse de orientación dual por diferentes combinaciones de las anteriores fusiones dCas9. Por ejemplo, represión a largo plazo estable puede lograrse mediante transducción simultánea de dCas9 DNMT3a y KRAB dCas932. La mayoría de estas fusiones represivas sólo ha sido atacada a un locus en un momento, y no queda claro que es más eficaz en la manipulación de múltiples potenciadores simultáneamente.
Enhancer-i, mientras que un método adecuado para el estudio de combinaciones de potenciadores para un puñado de genes, es todavía algo limitado en rendimiento si el usuario desea estudiar supuestos potenciadores para cientos de genes. Futuras aplicaciones de esta técnica incorporará tecnologías basadas en la proyección de imagen para cuantificar simultáneamente múltiples genes en muestras múltiples. Lo importante, estas tecnologías son compatibles con la detección directa de moléculas de ARN de lisado, eliminando la necesidad de aislamiento de RNA desperdiciador de tiempo. Estas adaptaciones facilitarán el interrogatorio de los conjuntos más grandes de potenciadores.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por NIH/NHGRI R00 HG006922 NIH/NHGRI R01 HG008974 a J.G. y el Instituto de cáncer Huntsman. J.B.C. fue apoyado por el programa de capacitación de los NIH en T32GM007464 genética.
Materiales
| Name | Company | Catalog Number | Comments |
| ZR 96-well Quick-RNA Kit | Zymo Research | R1053 | |
| Power SYBR Green RNA-to-CT 1-Step | Applied Biosystems | 4389986 | |
| AflII restriction enzyme | NEB | R0520S | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531L | |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | |
| FuGENE HD | Promega | E2312 | |
| DNA Clean & Concentrator Kit | Zymo Research | D4013 | |
| Buffer RLT Plus | Qiagen | 1053393 | |
| b-estradiol | Sigma-Aldrich | E2758 | |
| Human: Ishikawa cells | ECACC | 99040201 | |
| H3K27ac rabbit polyclonal | Active Motif | 39133 | |
| H3K9me3 rabbit polyclonal | Abcam | ab8898 | |
| FLAG mouse monoclonal | Sigma-Aldrich | F1804 | |
| ER alpha rabbit polyclonal | Santa Cruz | sc-544 | |
| pGL3-U6-PGK-Puro plasmid | Addgene | 51133 | Shen et al., 2014 |
| gRNA_cloningVector plasmid | Addgene | 41824 | Mali et al., 2013 |
| AflII U6 puromycin plasmid | Addgene | 106404 | Carleton et al., 2017 |
| SID4X-dCas9-KRAB plasmid | Addgene | 106399 | Carleton et al., 2017 |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-10ML | |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977-023 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985070 | |
| KAPA Stranded mRNA-Seq Kit, with KAPA mRNA Capture Beads | Kapa Biosytems | KK8420 | |
| Pierce Protease and Phosphatase Inhibitor Mini Tablets | ThermoFisher Scientific | A32959 | |
| Formaldehyde solution | Sigma-Aldrich | 252549-25ML | |
| Geneticin Selective Antibiotic (G418 Sulfate) (50 mg/mL) | ThermoFisher Scientific | 10131035 | |
| LB Broth | ThermoFisher Scientific | 10855001 | |
| Quick-DNA Miniprep Kit | Zymo Research | D3020 | |
| Quick-Load Purple 2-Log DNA Ladder | NEB | N0050S |
Referencias
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Roadmap Epigenomics, C., et al. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Perry, M. W., Boettiger, A. N., Bothma, J. P., Levine, M. Shadow enhancers foster robustness of Drosophila gastrulation. Curr Biol. 20 (17), 1562-1567 (2010).
- Lam, D. D., et al. Partially redundant enhancers cooperatively maintain Mammalian pomc expression above a critical functional threshold. PLoS Genet. 11 (2), e1004935 (2015).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Savic, D., et al. Promoter-distal RNA polymerase II binding discriminates active from inactive CCAAT/ enhancer-binding protein beta binding sites. Genome Res. 25 (12), 1791-1800 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23 (10), 1163-1171 (2013).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol. , (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods. 12 (5), 401-403 (2015).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods. 12 (12), 1143-1149 (2015).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6 (3), e1000869 (2010).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Fulco, C. P., et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science. 354 (6313), 769-773 (2016).
- Joo, J. Y., Schaukowitch, K., Farbiak, L., Kilaru, G., Kim, T. K. Stimulus-specific combinatorial functionality of neuronal c-fos enhancers. Nat Neurosci. 19 (1), 75-83 (2016).
- Xie, S., Duan, J., Li, B., Zhou, P., Hon, G. C. Multiplexed Engineering and Analysis of Combinatorial Enhancer Activity in Single Cells. Mol Cell. 66 (2), 285-299 (2017).
- Carleton, J. B., Berrett, K. C., Gertz, J. Multiplex Enhancer Interference Reveals Collaborative Control of Gene Regulation by Estrogen Receptor alpha-Bound Enhancers. Cell Syst. 5 (4), 333-344 (2017).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Ayer, D. E., Laherty, C. D., Lawrence, Q. A., Armstrong, A. P., Eisenman, R. N. Mad proteins contain a dominant transcription repression domain. Mol Cell Biol. 16 (10), 5772-5781 (1996).
- Alland, L., et al. Role for N-CoR and histone deacetylase in Sin3-mediated transcriptional repression. Nature. 387 (6628), 49-55 (1997).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Rennoll, S. A., Scott, S. A., Yochum, G. S. Targeted repression of AXIN2 and MYC gene expression using designer TALEs. Biochem Biophys Res Commun. 446 (4), 1120-1125 (2014).
- Stampfel, G., et al. Transcriptional regulators form diverse groups with context-dependent regulatory functions. Nature. 528 (7580), 147-151 (2015).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods. 10 (10), 973-976 (2013).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nat Methods. 11 (2), 122-123 (2014).
- Parsi, K. M., Hennessy, E., Kearns, N., Maehr, R. Using an Inducible CRISPR-dCas9-KRAB Effector System to Dissect Transcriptional Regulation in Human Embryonic Stem Cells. Methods Mol Biol. , 221-233 (2017).
- Romanienko, P. J., et al. A Vector with a Single Promoter for In Vitro Transcription and Mammalian Cell Expression of CRISPR gRNAs. PLoS One. 11 (2), e0148362 (2016).
- Smith, I., et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map. PLoS Biol. 15 (11), e2003213 (2017).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Res. 45 (17), 9901-9916 (2017).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados