Method Article
Absorbente Microbiopsy muestreo y extracción de RNA de sangre como mínimo invasor, simultánea y análisis de la piel
En este artículo
Resumen
En este artículo, se demuestra cómo se realiza la técnica de microbiopsy absorbente y cómo se puede utilizar la muestra para la extracción de RNA para muestreo simple y simultánea de la piel y la sangre de una manera mínimamente invasiva.
Resumen
Biopsia de piel convencionales limita la investigación clínica que involucra áreas cosméticamente sensibles o aplicaciones pediátricas debido a su invasividad. Aquí, describimos el protocolo para el uso de un dispositivo absorbente de microneedle, microbiopsy absorbente, para muestreo mínimamente invasiva de la mezcla de piel y sangre. Nuestro objetivo es ayudar a facilitar un rápido progreso en la investigación clínica, el establecimiento de biomarcadores para la enfermedad de la piel y reducir el riesgo para los participantes de la investigación clínica. En contraste con técnicas de biopsia de piel convencionales, microbiopsy absorbente puede realizarse en segundos y no requiere entrenamiento intensivo debido a su diseño simple. En este informe, describimos el uso de microbiopsy absorbente, incluyendo carga y aplicación, en un voluntario. A continuación, mostramos cómo aislar RNA de la muestra absorbida. Finalmente, se demuestra el uso de cuantitativo transcripción reversa PCR (RT-qPCR) para cuantificar los niveles de expresión de mRNA de sangre (CD3E y CD19) y piel (KRT14 y TYR). Los métodos que describimos a utilizan kits de estante y reactivos. Este protocolo ofrece un acercamiento como mínimo invasor para el muestreo simultáneo de la piel y la sangre dentro de la misma matriz absorbente microbiopsy. Hemos encontrado los comités de ética humana, los médicos y voluntarios que apoyan este enfoque a la investigación dermatológica.
Introducción
Biopsia de la piel es una de las técnicas más esenciales en dermatología para la toma de muestras de piel y posterior diagnóstico de enfermedades de la piel a través de la evaluación histopatológica. La técnica de biopsia implica a un profesional médico mediante una biopsia de la lámina o punzón para eliminar la lesión sospechosa en la piel del paciente para examen1. Aunque la técnica es eficaz, es altamente invasivo y limita la investigación clínica como el punto final generalmente involucra técnicas de biología molecular2,3. Análisis molecular de enfermedades de la piel tiene el potencial para proporcionar información biológica altamente específico que no puede el análisis histopatológico, facilitando así la droga descubrimiento y enfermedad diagnóstico4,5. Además, la demanda de la muestra en técnicas moleculares más es comparativamente pequeña y puede conducir a una reducción en el uso de animales y permitir un mayor número de repeticiones. Por lo tanto, existe una clara necesidad de una técnica alternativa que permite el análisis molecular en la investigación clínica y disminuye el riesgo para los participantes.
Para hacer frente a tanta necesidad en el campo, nuestro grupo ha desarrollado una novedosa plataforma de diagnóstico base de microneedle, microbiopsy absorbente, que permite la recopilación de una pequeña cantidad de piel mezclada con la sangre en una manera simple y mínimamente invasivo6. El propósito de esta publicación es describir la microbiopsy absorbente como una herramienta de toma de muestras para facilitar el análisis molecular mediante la extracción de RNA en la investigación clínica.
Anteriormente, hemos descrito la primera versión de microbiopsy, microbiopsy de la piel, que consiste en un microneedle de un diseño de placa de acero de tres capas para extraer pedazos diminutos de piel tejidos7. La novedad de este aparato proviene de los múltiples puntos de contacto de la microneedle que permite la extracción de tejido eficiente3. En cambio, una biopsia del sacador de piel circular proporciona un único punto de contacto y simplemente se rasga la piel sin capturar cualquier muestra en algunos casos. Basado en la microbiopsy de la piel, hemos desarrollado recientemente el microbiopsy absorbente que tiene sangre y piel capacidades de muestreo. El dispositivo ha demostrado ser factible para el uso en áreas de escasos recursos en un reciente estudio epidemiológico6.
Debido a su diseño simple, la microbiopsy absorbente puede realizarse en unos pocos segundos y no requiere de un entrenamiento extensivo. Además, no se necesita anestesia local y el sitio de aplicación no causa cicatrización. El presente Protocolo permite a investigadores o profesionales de la medicina sin el correspondiente muestreo para obtener datos específicos de la piel para el análisis molecular. Esperamos microsampling dispositivos para convertirse en rutina en la investigación de la piel en el futuro.
Aunque el microbiopsy se ha reportado en otros estudios de enfermedades de la piel que las técnicas de diagnóstico molecular6,8,9,10, como el virus del papiloma humano detección de ADN, este protocolo es el primero en demostrar los detalles de las técnicas de extracción y procesamiento de muestra para la microbiopsy absorbente. Además, este es el primer informe que describe los perfiles de expresión génica relativa de la piel y las células de la sangre en las muestras de microbiopsy.
Protocolo
El estudio fue aprobado por el Comité de ética en investigación humana Metro sur y la Universidad de Queensland humano investigación Comité de ética (HREC-13-QPAH-551 y UQ2013001551) y la Universidad de South Australia humana Comité de ética (200607).
1. absorbente Microbiopsy fabricación
- Corte láser piezas de microneedle de 50 μm hoja de acero inoxidable de grado médico y capa absorbente (papel de filtro), respectivamente (figura 1).
Nota: Las partes, incluyendo el émbolo y el caso, el dispositivo se fabrican con técnicas de prototipado rápido, como la impresión 3D. - Remoje todas las piezas, excepto la capa absorbente, en 70% de etanol (EtOH) por 5 min y aire seque durante 30 minutos después.
- Montar el microneedle de la tres-capa, con la capa absorbente en el medio (Figura 1a).
- Inserte el microneedle montado en la ranura del émbolo.
- Poner en la primavera e Inserte el émbolo en el caso (figura 1C). Evitar el microneedle tocar nada en el procedimiento de montaje para asegurar la calidad de la aguja.
- Sello de la microbiopsy absorbente montado en un paquete para la esterilización de la γ-radiación antes de usar.
2. muestreo con Microbiopsy absorbente
- Use guantes desechables y spray manos y herramientas, como pinzas, con el 70% EtOH.
- Desinfectar el sitio de aplicación con un paño con alcohol.
- Retire el microbiopsy del paquete esterilizado de γ-radiación sin tocar el microneedle en el proceso.
- Cargar el dispositivo tirando el émbolo para comprimir el resorte hasta que hay un 'clic' sonido y las cerraduras del émbolo en su lugar.
- El objetivo del dispositivo, la piel que se quiera muestrear, asegurando que el área de muestreo está en una posición fija.
- Asegurar que el dispositivo sea aproximadamente perpendicular a la piel y luego aplique ≥1 kg de fuerza para el dispositivo sobre la piel.
Nota: Aplicando fuerza hacia abajo en la piel es necesaria para asegurar la penetración suficiente de la microneedle. - Presione el gatillo y mantenga el dispositivo en su lugar por un mínimo de 10 s para la muestra de sangre ser absorbido.
Nota: Si el dispositivo se libera antes de 10 s, hay menos muestra capturada.
3. extracción de RNA
Nota: Los procedimientos de extracción de RNA se modificaron del protocolo del fabricante para el óptimo aislamiento de ARN de la muestra microbiopsied. Todos los reactivos y la columna utilizada en la extracción de RNA se incluyen en el kit a menos que se indique lo contrario.
- Saque el émbolo y absorbente microneedle de la caja de las manos suavemente. Asegúrese de que la punta de la microneedle no entra en contacto con cualquier cosa durante la extracción.
- Retire el microneedle absorbente el émbolo. Retener los dos orificios en el microneedle con un par de pinzas estériles y tire de ella.
- Poner el microneedle intacto en un tubo de microcentrífuga de 2 mL (no desde el kit de aislamiento de RNA; véase Tabla de materiales) Prellenado con 50 μl de tampón de extracción, con la aguja hacia hacia la parte inferior. Para minimizar la pérdida de la muestra, dejar el tubo en hielo seco durante 5 minutos antes de procesar la muestra.
- Vortex brevemente (3 a 5 s) para eliminar algunos de los materiales muestreados de la punta.
- Incubar el microneedle del tubo en un eje de balancín durante 30 min a 42 ° C.
Nota: La solución tampón será absorbida en la absorbente capa inmediatamente. - Coloque la parte superior del nivel de microneedle (el lado opuesto de la aguja) con el borde superior del tubo de microcentrífuga de 2 mL con pinzas estériles.
- Cierre la tapa del tubo de tal forma que la parte superior de la microneedle se sostiene entre el casquillo y el tubo.
- Girar el tubo en 16.000 x g o a la máxima velocidad de 30 s para eliminar los materiales muestreados de la capa absorbente del dispositivo.
- Retire con cuidado el microneedle con fórceps sin sumergir en la solución tampón. Mantenga el tubo y deseche el microneedle.
- Centrifugar las muestras a 3.000 x g durante 2 min pipeta el sobrenadante que contiene el RNA extraído cuidadosamente en un tubo nuevo de microcentrífuga.
- Añadir 250 μl de tampón de acondicionamiento en la membrana de filtro de columna de purificación e incubar 5 min a temperatura ambiente.
- Centrifugar la columna en el tubo de la colección en 16.000 x g o a la máxima velocidad durante 1 minuto.
- Añadir 50 μl de 70% EtOH en la muestra recogida de paso 3.9. Mezclar por pipeteo suavemente hacia arriba y hacia abajo.
- Transferir la mezcla (~ 100 μL) en la columna de purificación previo.
- Centrifugar durante 2 min a 100 x g de inmediato, seguido de una centrifugación a 16.000 x g por 30 s para quitar el flujo a través.
- Añadir 100 μl de tampón de lavado 1 (W1) en la columna de purificación y centrifugue durante 1 min a 8.000 x g.
- Preparar solución de DNasa por añadir 5 μl de DNasa I solución Stock a 35 μl de tampón de RDD en una microcentrífuga separada del tubo para cada muestra. Mezclar invirtiendo suavemente.
Nota: El tratamiento de DNasa es a realizar en la columna de purificación, en vez de en la placa PCR luego, debido a la cantidad de la muestra. - Agregar 40 μl de la mezcla de incubación DNasa directamente en la membrana de la columna de purificación. Incubar a temperatura ambiente durante 15 minutos.
- Agregar 40 μl de W1 en la membrana de la columna de purificación. Centrifugue a 8.000 x g durante 15 s.
- Pipetear 100 μl de tampón de lavado 2 (W2) en la columna de purificación después del tratamiento de DNasa y centrifugue durante 1 min a 8.000 x g.
- Añadir otro 100 μl de W2 en la columna de purificación y centrifugar durante 2 min a 16.000 x g. Revise la columna de purificación para cualquier tampón de lavado residual. Volver a centrifugar a 16.000 x g durante 1 min si queda cualquier tampón de lavado.
- Transferencia a un nuevo tubo de microcentrífuga de 0,5 mL en el juego de la columna de purificación.
- Pipeta 11 μl de ARNasa-agua libre (no incluido en el kit de aislamiento de RNA), en vez de tampón de elución (EB), directamente sobre la membrana de la columna de purificación. Toque suavemente la punta de la pipeta a la superficie de la membrana mientras se distribuye el agua libre de ARNasa para asegurar la máxima absorción de agua libre de ARNasa en la membrana. Incubar a temperatura ambiente durante 2 minutos.
- Centrifugar 1 min a 1.000 x g para distribuir el agua libre de ARNasa en la columna, luego centrifugar a 16.000 x g en 1 min para eluir el RNA en la columna.
Nota: No se recomienda la cuantificación de RNA debido a la cantidad pequeña de la muestra. - Dejar de punto #1: guardar la muestra de RNA total a-80 ° C si no se realizaron inmediatamente la síntesis de cDNA.
Nota: Evite congelar si no es necesario dada la baja concentración de la muestra de RNA.
4. síntesis de cDNA
- Descongelar la muestra congelada en hielo (de paso 3.25).
- Sintetizar el cDNA del ARN total con kit de síntesis de cDNA según el siguiente procedimiento.
- Preparar la mezcla de reacción: 11 μl de RNA total, 4 μL de 5 x TransAmp Buffer, 1 μl de transcriptasa reversa, 4 μL de agua libre de DNasa/Rnasa. Mezclar suavemente por pipeteo.
- Ejecutar la reversa de la transcripción en un aparato termociclador: 25 ° C durante 10 min, 42 ° C durante 15 min, seguida por un paso de inactivación definitiva de la transcriptasa inversa a 85 ° C durante 5 minutos.
- Deje el punto #2: almacenar la muestra transcripción inversa a-80 ° C hasta su uso.
5. reacción de qPCR y análisis de datos
Nota: Los iniciadores para reacciones de qPCR fueron diseñados para abarcar los límites del intrón para evitar la amplificación de la DNA genomic uso de cartilla de NCBI BLAST (www.ncbi.nlm.nih.gov/tools/primer-blast/). El gen de referencia utilizado en este estudio es RPLP0 (véase la sección de discusión para más información).
- Descongelar la muestra congelada en hielo (paso 4.5).
- Realizar qPCR con el qPCR master mix kit según el siguiente procedimiento.
- Preparar la mezcla principal que contiene los siguientes por reacción: 5 μl de la mezcla de reacción de 2 x qPCR, 0.4 μL de primer avance de 10 μm, 0.4 μL de cebador inverso de 10 μm, 2,2 μl de agua libre de DNAsas. Mezclar suavemente por pipeteo. Establecer las muestras en triplicado.
- Pipeta 8 μl de la mezcla principal en cada pocillo de una 384-bien PCR placa primero y luego 2 μl de cDNA a partir de cada muestra (un total de 10 μl por pocillo).
Nota: No hay control de plantilla (NTC) y sin control de la transcriptasa inversa (-RT) están incluidos como controles negativos. - Selle la placa con la película adhesiva y centrifugar brevemente.
- Ejecutar la reacción de amplificación en un termociclador en tiempo real: 95 ° C por 2 min (activación de la polimerasa), seguido por 40 ciclos de 95 ° C durante 5 s (desnaturalización), 60 ° C durante 10 s (recocido) y 72 ° C durante 10 s (extensión).
- Calcular la concentración de RNA de las muestras por interpolación de una curva estándar, preparada a partir de plantillas con concentraciones conocidas.
- Crear un Nuevo experimento en el software.
- Haga clic en definir y ajustar a las normas para establecer la dilución seriada de las placas.
- Seleccionar y organizar los pocillos para muestras y estándares. Haga clic en aplicar.
- Haga clic en analizar en la pestaña de resultado para evaluar la curva estándar. Confirmar la pendiente, el valor de R2 , error y eficiencia de amplificación cumplen con los criterios experimentales.
- Verificar las cantidades de las muestras desconocidas visualmente en la curva estándar basado en los valores de Ct.
Nota: La construcción de la curva estándar, incluyendo la elección del factor de dilución, se realiza según protocolos publicados11,12,13.
- Realizar análisis de expresión génica de ARNm mediante el método ΔCt (Ct es normalizada a un gen refence).
- Restar el valor de Ct de gene de la blanco del valor de Ct del gen de referencia para obtener el valor de ΔCt.
- Promedio de los valores de ΔCt de repeticiones técnicas.
- Trazar y analizar la expresión génica con estadísticas software11,14 (véase Tabla de materiales).
Nota: Alternativamente, el análisis de expresión génica se realiza según protocolos publicados13.
Resultados
Hemos divulgado previamente que la microneedle de la microbiopsy de la piel penetra en la piel aproximadamente 500 μm profunda7. El diseño de microneedle de la piel microbiopsy es muy similar a la de microbiopsy absorbente (Figura 2a). Mientras el absorbente microbiopsy consistió en una capa absorbente en el medio para la absorción de sangre, la microbiopsy de la piel contiene un canal de captación mecánica de los tejidos de la piel. El uso de la capa absorbente también condujo a una leve diferencia en la dimensión de microneedle (absorbente: 1.50 x 0.50 x 0,21 mm vs piel: 1.50 x 0.50 x 0.15 mm).
Figura 2b muestra los sitios de aplicación 5 min después de absorbente y piel microbiopsies se aplicaron en el brazo izquierdo de volar del hombre voluntario. Debido a la similitud entre los dos diseños de microneedle, los sitios de aplicación de ambos aparatos fueron comparables, con eritema menor. Ambos sitios de aplicación no eran sensibles después de 48 h con ninguna cicatriz izquierda detrás. Esto apoya la hipótesis de que este dispositivo mínimamente invasivo tiene el potencial para ayudar a múltiples sitios del uso de la pantalla o que se realizará en una base regular.
C de la figura 2 muestra un cuadro representativo de la capa absorbente de microbiopsy absorbente después de ser aplicado en el brazo de volar de un hombre voluntario. Como se muestra en la figura, unos pedazos pequeños de piel fueron capturados cerca de la punta de la microneedle, y algo de sangre fue absorbido por el papel de filtro. Esto indica que el aparato penetró la piel y había capturado la piel y la sangre simultáneamente dentro de la misma matriz absorbente microbiopsy. Figura 2d muestra un muestreo después de la piel microbiopsy, la anterior generación de microbiopsy, para la comparación. El canal de la microbiopsy de piel capturó un pequeño trozo de piel, pero la cantidad de sangre en el microneedle fue pequeña en relación con la microbiopsy absorbente. Ambos sitios de aplicación no eran sensibles dentro de 48 h. En el experimento, se aplicó el microbiopsy absorbente con un tiempo de retención 10-s posteriores a la aplicación, mientras que la piel microbiopsy fue lanzado inmediatamente después de aplicar debido a la diferencia en el diseño.
Como se muestra en la figura 3a, dos grupos de microbiopsy absorbente participaron en este experimento basado en el protocolo de aplicación: 'Liberación inmediata' y tenencia 10 s'. Para el grupo de la "Liberación inmediata", el dispositivo se aplicó utilizando el mismo protocolo original de piel microbiopsy, con el dispositivo de ser retirado de la zona de aplicación inmediatamente después de la aplicación. Para la celebración de 10-s' grupo, el dispositivo se llevó a cabo en el lugar después de la aplicación de 10 s para mejorar la recogida de la muestra. Los dos grupos de microbiopsy absorbente se establecieron para demostrar cómo el enfoque de aplicación podría afectar la cantidad de muestra. El tiempo de mantenimiento de 10-s fue elegido como un tiempo clínicamente razonable para demostrar que el tiempo de aplicación afecta a la cantidad de muestra recuperable.
La cantidad de RNA se recuperó de los dos grupos de microbiopsy absorbente fueron 0.33 ± 0,39 ng 'Liberación inmediata' y 1.43 ± 0,88 ng para la celebración de 10-s' (figura 3a, n = 20), lo que sugiere un aumento de 4 veces con el tiempo extra. Esto indica que, aplicando el dispositivo absorbente con el tiempo de mantenimiento de 10-s dio lugar a más ARN extraído. La diferencia puede ser debido a la presencia de muestra de sangre creciente en la capa de absorbente (Figura 3C). De hecho, el grupo de 'Liberación inmediata' (figura 3b) no recoge una cantidad similar de sangre con la capa absorbente en comparación con la explotación de 10-s' grupo (Figura 3C). Debe señalarse también que microbiopsies más inmediatamente liberados estaban cerca del eje x, lo que sugiere que fueron acontecimientos negativos o muestran una muy baja cantidad de ARN. Por lo tanto, el resultado valida la hipótesis de que el tiempo tendría un impacto en el rendimiento del dispositivo ya que puede tomar tiempo para que la sangre sea absorbida por la capa absorbente.
Puesto que el dispositivo fue diseñado para la sangre y toma de muestras de piel, qPCR fue utilizado para detectar la expresión de biomarcadores de la piel y la sangre para ambos dispositivos de comparación. Utilizamos tirosinasa, TYR, como un biomarcador de melanocitos y KRT14 como un biomarcador de queratinocitos en la epidermis viable. Piel microbiopsy las muestras se incluyeron en el experimento para la comparación. Como se muestra en la figura 4, aunque la piel y microbiopsies absorbentes se aplicaron diferentemente debido a la diferencia en el diseño, la expresión los niveles de biomarcadores TYR de la piel y KRT14 fueron comparables para ambos dispositivos como se indica en la datos. Biomarcadores de glóbulos blancos (CD3E, células T y CD19, las células de B) fueron encontrados para ser más frecuentes en muestras de microbiopsy absorbente que en muestras de piel microbiopsy. Este resultado sugiere que la microbiopsy absorbente se desempeñaron mejor en la recogida de sangre pero todavía mantiene la capacidad de captura de piel en comparación con la microbiopsy de piel (n = 5).
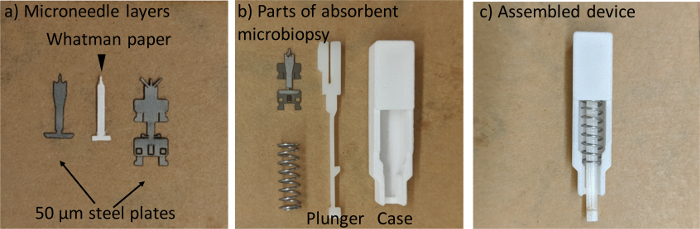
Figura 1. La fabricación de absorbente microbiopsy. El microneedle de tres capas. (b) todas las piezas del dispositivo. (c) el ensamblado microbiopsy absorbente. Haga clic aquí para ver una versión más grande de esta figura.
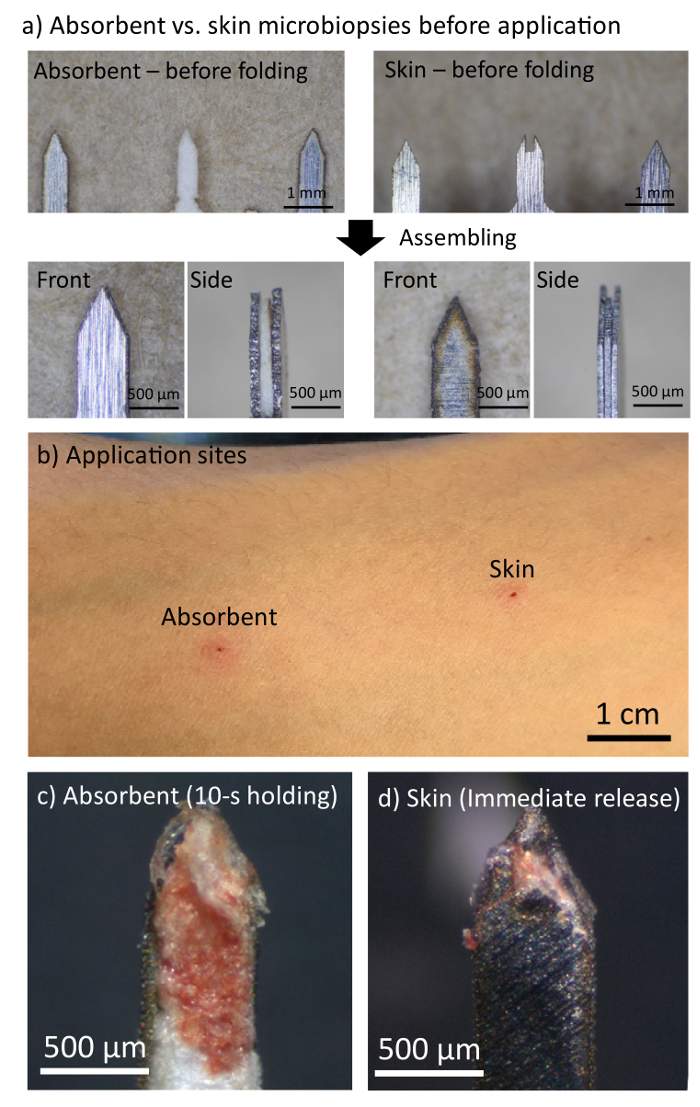
Figura 2. Microbiopsy absorbente fue capaz de capturar simultáneamente muestras de sangre y la piel sin dejar una cicatriz en el brazo izquierdo de volar del hombre voluntario. una comparación entre los microneedles de microbiopsies absorbente y de la piel. (b) aplicación sitios izquierda por absorbente y de la piel microbiopsies 5 min después de la aplicación. (c, d) Microneedles de microbiopsies absorbente y de la piel después de la aplicación. Haga clic aquí para ver una versión más grande de esta figura.
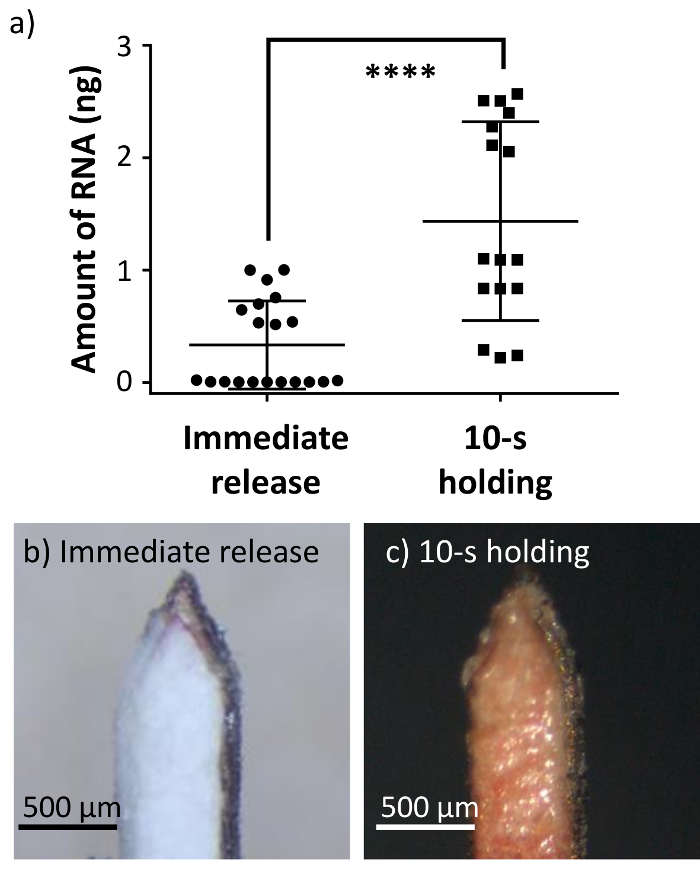
Figura 3. Microbiopsy absorbente capturado más sangre y muestra de RNA cuando se aplica por 10-s. (a) el tiempo de mantenimiento posteriores a la aplicación de 10-s dio lugar a una mayor cantidad de RNA que liberar inmediatamente el dispositivo (n = 20). Barras de error representan S.D. de la media (***p< 0.0001). (b, c) Cuadros representativos de la microbiopsies absorbente después de la aplicación con 'Liberación inmediata' y 10 s' enfoques. Haga clic aquí para ver una versión más grande de esta figura.
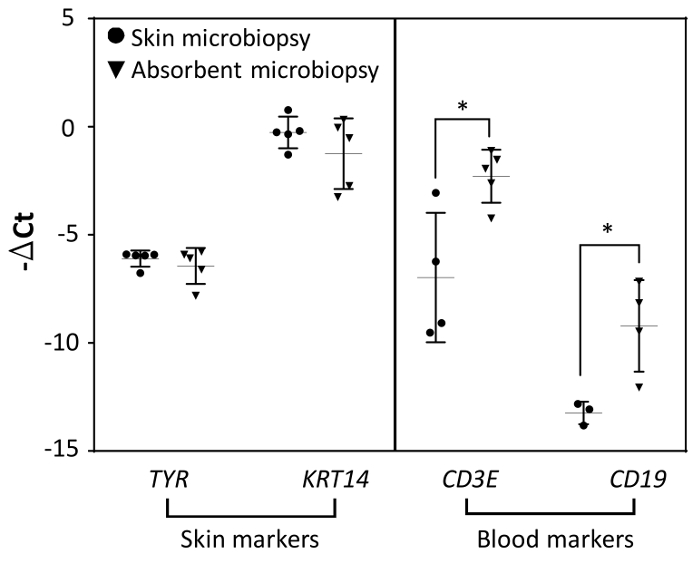
Figura 4. La comparación de los niveles de expresión de mRNA de la piel absorbente microbiopsies (n = 5). La expresión del gene había sido normalizada con gen de referencia RPLP0. Barras de error representan S.D. de la media (*p< 0.05). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Estos resultados demuestran que el microbiopsy absorbente puede utilizarse como una herramienta para muestreo simple y mínimamente invasivo de la piel y la sangre mezcla para caracterización molecular. Aplicación del dispositivo según nuestro protocolo es esencial para obtener resultados fiables como se indica por la diferencia en la cantidad de RNA con protocolos de aplicación diferentes (figura 3). Una vez extraída la muestra, la muestra posterior procesamiento paso para extracción de RNA es muy similar a protocolos establecidos15,16. Además de los pasos iniciales en la extracción de RNA que son modificados para lo microbiopsy absorbente, otro cambio clave es el uso de agua libre de ARNasa para mejorar los resultados para aplicaciones posteriores. Por otra parte, cabe mencionar que el gen de referencia utilizado en este estudio es RPLP0. RPLP0, cuya función es conocida por diferentes células y tejidos tipos17, se ha divulgado como un gen de referencia adecuados para el uso en cánceres de piel no melanoma y lesiones precancerosas18.
Una de las principales limitaciones del dispositivo es la eliminación de la microbiopsy desde el dispositivo, que es desperdiciador de tiempo y aumenta potencialmente la posibilidad de pérdida de muestra, especialmente para muestras sensibles como RNA. Sin embargo, el problema puede superarse la prerefrigeración todas las herramientas de procesamiento estéril, como los tubos de microcentrífuga de 2 mL en hielo seco.
El uso de la microbiopsy absorbente es simple y no requiere entrenamiento intensivo. Biopsia convencional no es necesaria microbiopsy permite la caracterización molecular con técnicas de diagnóstico molecular establecido, como RT-qPCR. Para cuantificar y demostrar la capacidad de muestreo de microbiopsy absorbente, se investigó la literatura anterior que involucró la extracción del RNA de tejidos de la piel humana. Biopsia en sacabocados de una típico 3.0 o 4.0 mm la piel, tres estudios han reportado la cantidad de RNA extraída osciló entre 50 y 200 ng por mg de tejido de piel19,20,21. Comparando con el 1,43 ng de RNA que se recuperó de la microbiopsy absorbente en promedio (figura 3), el peso de los tejidos de la piel muestra se espera al rango de 0.29-115 μg basada en la misma proporción de RNA en tejido de los estudios de biopsia de piel punch.
Este protocolo no está sin peligros potenciales, aunque algunos de los problemas pueden superarse fácilmente. Por ejemplo, los datos de extracción de RNA sugirieron una variación considerable con el tiempo de mantenimiento de 10-s (figura 3). Para abordar el problema, una posible solución consiste en optimizar el protocolo de aplicación. Parámetros como la fuerza aplicación a la piel y tiempo de retención debe ser probado y optimizado para reducir las variaciones en la extracción de la muestra. El otro problema potencial es la eliminación de microbiopsy desde el dispositivo, que podría afectar la integridad de la muestra y la recuperación. Aunque la eliminación de la microbiopsy para la extracción de RNA es un enfoque eficaz, todo el procedimiento es tedioso, y la muestra puede estar expuesta a la contaminación en el proceso. Por lo tanto, la modificación del Protocolo de procesamiento de muestra es altamente deseada con el fin de garantizar la integridad de la muestra y evitar la pérdida de muestra.
Una vez que se abordan las dos cuestiones anteriores, se espera que el dispositivo se convertirá en una herramienta estándar para la investigación clínica. Es importante tener en cuenta que el dispositivo captura muestra de piel y sangre al mismo tiempo y esto debería tenerse en cuenta cuando se analizan datos de expresión genética. En conclusión, este protocolo describe los detalles de la realización de microbiopsy absorbente como una herramienta fácil y mínimamente invasiva para piel combinada y tomar muestras de sangre y la posterior muestra de procesamiento para el perfil de expresión génica relativa.
Divulgaciones
No hay conflicto de interés declarado.
Agradecimientos
Nos gustaría reconocer NHMRC becas APP1109749 y APP1111216, Universidad de Queensland centenario beca y beca de investigación Postgrado Internacional.
Materiales
| Name | Company | Catalog Number | Comments |
| Absorbent microbiopsy fabrication and sampling | |||
| Absorbent Microbiopsy | Trajan Scientific and Medical | N/A | https://www.trajanscimed.com/ |
| Whatman filter paper, Grade 1 | Sigma Aldrich | WHA1001325 | N/A |
| RNA extraction | |||
| PicoPure RNA Isolation Kit | ThermoFisher | KIT0214 | Including all buffer soltuions described in protocol |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher | 10977015 | Improving RNA quality in RNA elution step |
| 2.0 mL Microcentrifuge Tube, Sterile | Thomas Scientific | 1226S74 | N/A |
| Microcentrifuge 5415R | Eppendorf | Z605212 | N/A |
| cDNA synthesis | |||
| SensiFAST cDNA Synthesis Kit | Bioline | BIO-65053 | N/A |
| CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855196 | N/A |
| qPCR reaction and data analysis | |||
| SensiFAST SYBR Lo-ROX Kit | Bioline | BIO-94005 | Including reagents described in qPCR reaction steps |
| MicroAmp Optical Adhesive Film | ThermoFisher Scinentific | 4311971 | N/A |
| MicroAmp Optical 384-Well Reaction Plate | ThermoFisher Scinentific | 4343370 | N/A |
| QuantStudio 6 Flex Real-Time PCR System | ThermoFisher Scinentific | 4485694 | N/A |
| GraphPad Prism (v6.04) | GraphPad | N/A; Windows version | Plotting and statistical analysis in qPCR data analysis steps |
| PCR primers | |||
| RPLP0 F | Sigma Aldrich | N/A | ATC AAC GGG TAC AAA CGA GTC |
| RPLP0 R | Sigma Aldrich | N/A | CAG ATG GAT CAG CCA AGA AGG |
| TYR F | Sigma Aldrich | N/A | TCA GCA CCC CAC AAA TCC TAA |
| TYR R | Sigma Aldrich | N/A | AAT CGG CTA CAG ACA ATC TGC |
| KRT14 F | Sigma Aldrich | N/A | CCT CCT CCC AGT TCT CCT |
| KRT14 R | Sigma Aldrich | N/A | ACA CCA CCT TGC CAT CG |
| CD3E F | Sigma Aldrich | N/A | CAA AGG GGA CAA AAC AAG GAG |
| CD3E R | Sigma Aldrich | N/A | GTT CTC CAG AGG GTC AGA TG |
| CD19 F | Sigma Aldrich | N/A | TTC TGC CTG TGT TCC CTT G |
| CD19 R | Sigma Aldrich | N/A | GCG TCA CTT TGA AGA ATC TCC T |
Referencias
- Wang, C. Y., Maibach, H. I. Why minimally invasive skin sampling techniques? A bright scientific future. Cutaneous and Ocular Toxicology. 30, 1-6 (2011).
- Kirstein, O. D., et al. Minimally invasive microbiopsies: a novel sampling method for identifying asymptomatic, potentially infectious carriers of Leishmania donovani. International journal for parasitology. 47, 609-616 (2017).
- Lin, L. L. . The skin microbiopsy. , (2015).
- Dhingra, N., et al. Molecular profiling of contact dermatitis skin identifies allergen-dependent differences in immune response. Journal of Allergy and Clinical Immunology. 134, 362-372 (2014).
- Chapman, P. B., et al. Improved Survival with Vemurafenib in Melanoma with BRAF V600E Mutation. New England Journal of Medicine. 364, 2507-2516 (2011).
- Kirstein, O. D., et al. Minimally invasive microbiopsies: a novel sampling method for identifying asymptomatic, potentially infectious carriers of Leishmania donovani. International journal for parasitology. 47, 609-616 (2017).
- Lin, L. L., et al. Microbiopsy engineered for minimally invasive and suture-free sub-millimetre skin sampling. F1000Research. 2, (2013).
- Tom, L. N., et al. Skin microbiopsy for HPV DNA detection in cutaneous warts. Journal of the European Academy of Dermatology and Venereology. 30, e216-e217 (2016).
- Sobarun, P., et al. Microbiopsy Biomarker Profiling in a Superficial Melanoma Resembling a Pigmented Basal Cell Carcinoma. JAMA Dermatology. 153, 334 (2017).
- Banan, P., Lin, L. L., Lambie, D., Prow, T., Soyer, H. P. Effects of Ex Vivo Skin Microbiopsy on Histopathologic Diagnosis in Melanocytic Skin Lesions. JAMA Dermatology. 149, 1107 (2013).
- . GraphPad - FAQ 1753 - Prism 3 -- Calculating "Unknown" Concentrations using a Standard Curve Available from: https://www.graphpad.com/support/faq/prism-3-calculating-unknown-concentrations-using-a-standard-curve/ (2018)
- Kubista, M., et al. The real-time polymerase chain reaction. Molecular Aspects of Medicine. 27, 95-125 (2006).
- Goni, R., García, P., Foissac, S. . The qPCR data statistical analysis. , 1-9 (2009).
- . . GraphPad Statistics Guide. , (1995).
- Goldstein, N. B., et al. Isolating RNA from precursor and mature melanocytes from human vitiligo and normal skin using laser capture microdissection. Experimental dermatology. 25, 805-811 (2016).
- Shearstone, J. R., Allaire, N. E., Campos-Rivera, J., Rao, S., Perrin, S. Accurate and precise transcriptional profiles from 50 pg of total RNA or 100 flow-sorted primary lymphocytes. Genomics. 88, 111-121 (2006).
- Bär, M., Bär, D., Lehmann, B. Selection and Validation of Candidate Housekeeping Genes for Studies of Human Keratinocytes-Review and Recommendations. Journal of Investigative Dermatology. 129, 535-537 (2009).
- Hoang, V. L. T., et al. RNA-seq reveals more consistent reference genes for gene expression studies in human non-melanoma skin cancers. PeerJ. 5, e3631 (2017).
- Döbbeling, U. Simultaneous RNA and DNA Extraction from Biopsy Material, Culture Cells, Plants, and Bacteria. Nucleic Acid Protocols Handbook, The. , 53-56 (2000).
- Bruning, O., et al. RNA isolation for transcriptomics of human and mouse small skin biopsies. BMC Research Notes. 4, 438 (2011).
- Berglund, S. R., et al. Optimized Methodology for Sequential Extraction of RNA and Protein from Small Human Skin Biopsies. Journal of Investigative Dermatology. 127, 349-353 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados