Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Tri cellulaire activé par fluorescence des protoplastes végétaux
Dans cet article
Résumé
Une méthode pour isoler des types cellulaires spécifiques du matériel végétal est démontrée. Cette technique utilise des lignes de marqueur transgéniques exprimant des protéines fluorescentes dans des types cellulaires particuliers, cellulaire dissociation et de tri cellulaire activé par fluorescence. De plus, une installation de croissance est établi ici, qui facilite le traitement des Arabidopsis thaliana Semis avant le tri cellulaire.
Résumé
Haute résolution, le type spécifique de cellules d'analyse de l'expression génique améliore considérablement la compréhension de la régulation du développement et des réponses aux stimuli de l'environnement dans tout organisme multicellulaire. Hybridation in situ et la visualisation gène rapporteur peut dans une certaine mesure être utilisés à cette fin, mais pour une grande résolution quantitative RT-PCR ou haut-débit à l'échelle d'analyse du transcriptome à l'isolement de l'ARN à partir des types cellulaires particuliers sont requis. Cellulaire dissociation des tissus exprimant un marqueur fluorescent protein dans un type cellulaire spécifique et ultérieures de tri cellulaire par fluorescence (FACS), il est possible de collecter des quantités suffisantes de matériel pour l'extraction d'ARN, d'ADNc analyse de synthèse / amplification et de biopuces.
Un vaste ensemble de cellules spécifiques au type de lignes rapporteur fluorescent est disponible à la communauté scientifique des plantes. Dans ce cas, deux lignes de marqueur de la racine d'Arabidopsis thaliana sont utilisées: P RCS:: GFP (endoderme et le centre de repos) et P WOX5:: GFP (centre de repos). Un grand nombre (des milliers) de jeunes plants sont cultivés en hydroponie ou sur plaques de gélose et d'obtenir du matériel récolté assez de racines pour une analyse ultérieure. Cellulaire de dissociation de la matière végétale est obtenue par digestion enzymatique de la paroi cellulaire. Cette procédure permet l'utilisation de haute osmolalité induite par plasmolyse et cellulases disponibles dans le commerce, pectinases et hémicellulases pour libérer protoplastes dans la solution.
FACS de GFP-positives cellules permet l'utilisation de la visualisation des verts contre les spectres d'émission rouge de protoplastes excité par un laser à 488 nm. GFP-positives protoplastes peuvent être distingués par leur ratio est passé du vert à l'émission de rouge. Les protoplastes sont généralement triés directement dans un tampon d'extraction de l'ARN et stockés pour un traitement ultérieur à une date ultérieure.
Cette technique se révèle être simple et réalisable. Par ailleurs, il est démontré qu'il peut être utilisé sans difficulté d'isoler un nombre suffisant de cellules pour l'analyse du transcriptome, même pour des types de cellules très rares (par exemple les cellules du centre de repos). Enfin, une installation de croissance des semis d'Arabidopsis est démontré que le traitement permet simple des plantes avant de tri cellulaire (par exemple pour le type spécifique de cellules d'analyse des réponses au stress biotiques ou abiotiques). Supplémentaires éventuelles utilisations des FACS des protoplastes végétaux sont discutées.
Protocole
1) Préparation du matériel végétal
- Les protoplastes peuvent être dérivées de nombreuses espèces de plantes différentes et des tissus sous réserve que la bonne combinaison de la paroi cellulaire des enzymes digérant est utilisé 1. Avant une expérimentation à grande échelle est entreprise, une digestion à petite échelle de la matière est conseillé afin d'évaluer l'efficacité protoplastes du tissu, des enzymes, etc, et d'estimer les cellules pour cent positif pour le tri cellulaire. Ici, les protoplastes dérivés à partir des racines des plants d'Arabidopsis thaliana cette cellule de type expriment spécifiquement la protéine fluorescente verte (GFP) sont utilisés. L'endoderme et le centre de repos sont marquées par P RCS:: GFP et le centre de repos par P WOX5:: GFP 2,3 (figure 1).
- Les plants sont cultivés en hydroponie dans phytatrays (Sigma; figure 2a) sur un filtre en nylon (250 mailles um; NITEX) qui permet la croissance des racines dans le milieu grâce à la croissance (0,22% p / v de Murashige et Skoog basale [sigma], 1% p / v de saccharose, 0,05% p / v MES [2 - (N-morpholino) acide éthanesulfonique], pH 5,7 avec KOH). Alternativement, les plantes peuvent être cultivées sur un filtre en nylon (maille 100 um) en position verticale des plaques d'agar à 1% (Figure 2b).
- L'utilisation des filtres ci-dessus contribue non seulement à la récolte des racines, il facilite également facile de traitement supplémentaire des plants, si désiré. Les filtres permettent les semis à être transférés en masse pour phytatrays nouvelles ou des plaques d'agar complété par un catalyseur d'intérêt. Par exemple, l'ensemble phytatray a été utilisé pour des cellules spécifiques au type d'analyse de la réponse transcriptionnelle au traitement de l'azote dans des plants d'Arabidopsis 4.
- Assurez-vous que votre microscope marqueur fluorescent est correctement exprimé (en particulier lorsque vous utilisez l'option de traitement, comme la cellule de type marqueurs spécifiques eux-mêmes pourraient être influencés par le traitement). Dans ce cas, les plants sont inspectés sous un microscope à fluorescence (Nikon, figure 1). Notez que la cellule spécifique du type lignes marqueur fluorescent devrait être caractérisé d'abord sous un microscope confocal à déterminer exactement quels types de cellules sont marquées et de déterminer la variabilité dans l'expression.
2) Préparation de la solution de protoplastes
- Dissoudre 1,25% p / v cellulase (Yakult), 0,3% p / v Macerozyme (Yakult), 0,4 M de D-mannitol, 20 mM de MES et 20 mM de KCl (à partir d'un stock de 1 M) dans de l'eau déminéralisée et ajuster le pH à 5,7 avec 1 M Tris / HCl pH 7,5. Cette solution sera légèrement trouble.
- Chauffer la solution à 55 ° C pendant 10 minutes (la solution va devenir clair) et laisser refroidir à température ambiante avant d'ajouter 0,1% p / v de BSA (sérum albumine bovine), CaCl2 10 mM et 5 mM β-mercaptoéthanol .
3) Récolte et protoplastes de la matière végétale
- Les racines sont récoltées par les grattant le filet de nylon avec un scalpel et déposés dans un flacon contenant la solution protoplastes. Généralement, 10 ml de solution protoplastes est utilisée par 1500 racines des plantules.
- Agiter les flacons en douceur (75 min) à température ambiante pendant une heure. Un temps d'incubation peut augmenter le rendement de protoplastes, mais va également ajouter à l'effet de lui-même protoplastes sur l'expression génique.
- Filtrer la solution de protoplastes avec une passoire cellule 40 um (BD Falcon) et diviser la solution sur des tubes coniques de 15 ml (BD Falcon).
- Faites tourner les tubes dans une centrifugeuse swing-seau pendant 10 minutes à 500 g. Remarquez que cette vitesse de centrifugation va dépendre du type de protoplastes utilisés, leur fragilité et de la quantité de débris de cellules produites pendant le traitement enzymatique.
- Retirez la plupart du surnageant, remettre en suspension les protoplastes dans la solution restante et les inspecter au microscope (figure 3).
- Faire usage d'un hématimètre pour estimer le nombre et la densité de protoplastes. La densité cellulaire permettra de déterminer la vitesse d'injection de l'échantillon, les événements par seconde et donc le temps total nécessaire pour trier les cellules au FACS (voir aussi rubrique 4.3).
- 3.7) Soit procéder directement à FACS ou se laver et remettre en suspension les protoplastes dans une solution d'incubation, comme W5 (154 mM NaCl, 125 mM de CaCl2, 5 mM de KCl, 5 mM MES, ajuster le pH à 5,7 avec KOH) ou de solution sans le protoplastes ajoute des enzymes. Surtout quand on regarde les changements de la transcription, il est d'importance pour minimiser l'exposition des échantillons à des conditions qui pourraient influer sur l'expression des gènes, comme le changement de la mémoire tampon, les protoplastes sont maintenus po Il est donc recommandé de garder les protoplastes dans une solution de protoplastes et de procéder au FACS dès que possible.
4) Le tri cellulaire activé par fluorescence des protoplastes
- Allumez-le et préparer le trieur de cellules. Ici, un FACSAria (BD) est utilisé.
- Mettre en place un flux de débit avec un 100 m &u; buse m et une pression de 20 psi gaine.
- La densité cellulaire et la vitesse d'injection de l'échantillon peut être ajusté à l'expérience particulière basée sur le savoir si un meilleur rendement possible, ou plus grande vitesse possible est souhaitée. Nous avons trié avec succès avec des densités atteignant 10.000.000 cellules / ml.
- Utilisez l'option d'agitation de l'échantillon sur le FACS pour empêcher la sédimentation des protoplastes. Si le colmatage du FACS est un problème, il ya trois étapes de dépannage possibles: 1. Effectuer un échantillon de rétrobalayage ligne. 2. Diluer la suspension de votre protoplastes pour réduire la densité. 3. Nettoyer la solution de protoplastes en répétant l'étape de filtration (3,3) après centrifugation et resuspension.
- Préparez l'appareil à mesurer diffusion vers l'avant (FSC), la diffusion latérale (SSC) et émission à 530/30 nm pour la GFP et 610/20 nm pour le spectre rouge autofluorescence (RSA) après excitation par un laser à 488 nm. Ce sont essentiellement les seuls paramètres utilisés pour isoler la GFP-positives protoplastes. Ici, les réglages de tension ont été comme suit: FSC - 60V, 250V SSC, la GFP 350V 335V et RSA. Notez que les réglages de tension optimale sera différente pour chaque FACS et sera même pas besoin d'être ajustée pendant toute la durée de la trieuse de cellules.
- Commencez par mettre en place un dotplot pour diffusion vers l'avant par rapport diffusion latérale. Appliquer les réglages de tension de sorte que les événements mesurés sont centrés dans le complot.
- Ensuite, créez un tracé de points de vert par rapport rouges signaux de fluorescence. Appliquer les réglages de tension de sorte que les événements mesurés donnent une population centrée en diagonale dans la parcelle en regardant une suspension de type sauvage protoplastes (non-GFP). Une suspension de protoplastes provenant d'une ligne de marqueur GFP va produire une population claires de vert fluo des événements jamais vus dans des échantillons de type sauvage.
- Régler les contraintes d'ajuster la rémunération de chevauchement spectral entre la GFP et RSA. Une bonne paramètres contrainte de compensation permettra une meilleure séparation des protoplastes GFP-positives à partir des protoplastes non-GFP et de débris. Les contraintes utilisées ici sont comme suit: RSA, moins GFP 17,91%.
- Mettre en place un portail d'identifier GFP-positives événements, un contrôle négatif de la non-GFP protoplastes devrait être utilisé pour aider à définir les limites de grille (figure 4).
- Mettre en œuvre une coupure diffusion vers l'avant afin de laisser sortir les petits débris de l'analyse. Visualisez les événements GFP-positives dans le complot FSC vs SSC pour aider à déterminer le placement de la coupure. Ici, le seuil était fixé à 5000. Notez que le FACS comptera les débris que les événements de tri et d'un échantillon avec un niveau élevé de débris peuvent avoir un pour cent événements différents GFP positives que prévu. Ce n'est pas nécessairement un problème. Cependant, le plus de débris dans l'échantillon, plus le tri va prendre.
- Selon l'expérience et l'abondance du type de cellule à analyser, de définir le mode de précision FACS, soit pour un rendement optimal ou la pureté optimale des cellules triées.
- Pour l'extraction d'ARN, de préparer des tubes de prélèvement (1,5 ml microtubes de) avec la quantité appropriée de tampon d'extraction d'ARN. Avec cette configuration, 20000 évènements de tri donnera un volume total d'environ 100 ul qui ont été triés pour 350 pi de tampon d'extraction (RNeasy Micro Kit, QIAGEN). Mélanger les échantillons après l'achèvement de la sorte que la suspension de cellules peuvent mettre en commun au sommet.
- Conserver les échantillons ou de procéder directement à extraction de l'ARN. Analyses microarray réussies ont été préformé avec l'ARN extrait d'aussi peu que 500 événements triés. Ici, nous avons utilisé un kit d'extraction RNeasy Micro (QIAGEN), le système WT-Ovation Pico amplification de l'ARN et FL-Ovation ADNc Biotine Module V2 (NuGEN).
Les résultats représentatifs
Un phytatray d'environ 1500 RCS d'une semaine, âgé P: semis GFP a donné environ 60 000 protoplastes (tel que mesuré par hématimètre). 2,6% des 65 000 évènements FACS-transformés ont été définis comme étant GFP-positives et ont été triés (figure 4b).
Huit plaques d'environ 1500 de quatre jours, âgé de P WOX5:: GFP plants chacun (12 000 au total) a donné environ 30.000.000 protoplastes (tel que mesuré par hématimètre). 0,063% des 16.000.000 d'événements FACS-traitées ont été définis comme étant GFP-positives et ont été triés (figure 4C).
10.000 manifestations triées sont généralement utilisés pour l'extraction de l'ARN et peut produire de 20 à 140 ng d'ARN total (figure 5).
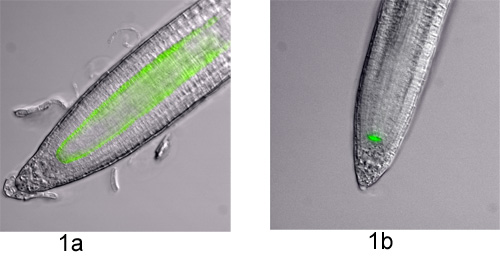
Figure 1. Cellulaire spécifique du type lignes de marqueur GFP dans la racine d'Arabidopsis. Microscopie à fluorescence images ont été prises avec un contraste d'interférence différentiel (DIC) et une GFP sur un microscope à Eclipse 90i (Nikon) fonctionnant sur le logiciel Metamorph (Molecular Devices) de filtrage. Les images DIC et GFP ont été superposées à des fins de visualisation. Les deux lignes de marqueur utilisé dans cette expérience visuelle sont présentés;a) P RCS:: GFP et b) P WOX5:: GFP.

Figure 2. Plants conditions de croissance. Ont été cultivées dans un contrôleur de l'environnement hydroponique dans phytatrays (a) ou sur gélose positionnée verticalement (b).
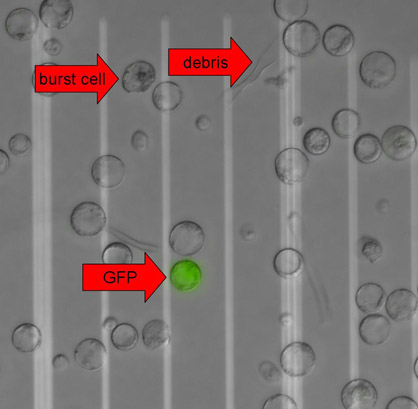
Figure 3. Protoplastes de plantes exprimant la GFP. Microscopie à fluorescence images ont été prises avec un contraste d'interférence différentiel (DIC) et un filtre GFP sur un microscope à Eclipse 90i (Nikon) fonctionnant sur le logiciel Metamorph (Molecular Devices). Les images DIC et GFP ont été superposées à des fins de visualisation. Les flèches indiquent une cellule éclatement, les débris cellulaires et d'un protoplaste GFP-positives. La distance entre deux lignes blanches est de 50 um.
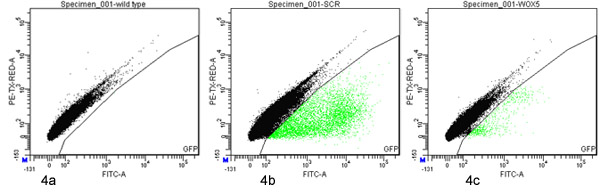
Figure 4. Fluorescence tri cellulaire activé de la GFP-positives protoplastes protoplastes issus de type sauvage (a) P RCS.:: GFP (B) ou P WOX5:: GFP (c) des lignes de marqueur ont été analysés et triés avec un FACSAria (BD) en utilisant les portes définies sur un dotplot de vert (530/30 nm; axe des x) par rapport à rouge (610/20 nm; axe Y) de fluorescence. 100 000 événements sont présentés dans chaque parcelle. Les événements relevant de la porte de tri GFP sont surlignés en vert.
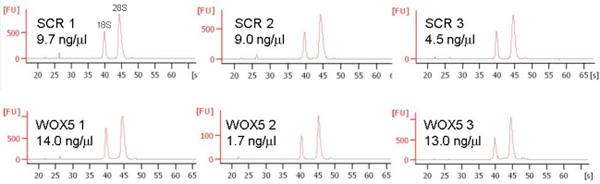
Figure 5. Représentant des extractions d'ARN à partir 10.000 cellules triées. Les cellules ont été triées directement dans l'ARN d'extraction du tampon (QIAGEN), l'ARN a été purifié et vérifiées pour la concentration, la pureté et l'intégrité sur un bioanalyseur 2100 (Agilent). Trois répliques pour les deux lignes de marqueur sont affichées.
Discussion
Les protoplastes peuvent, en principe, être dérivé d'une variété de tissus végétaux, l'optimisation des conditions favorables permettra d'améliorer grandement la qualité et la quantité d'ARN. Tant la solution protoplastes et le tampon d'incubation élective utilisés auront une influence sur cet aspect.
Beaucoup de différentes protéines fluorescentes peuvent être utilisés, selon les capacités de la FACS utilisé, par exemple, la GFP, RFP, YFP, CFP ...
Remerciements
Ce travail a été soutenu par la National Science Foundation (subvention aucune. DBI 0.519.984) et le National Institutes of Health (subvention aucune. 5R01GM078279) ..
matériels
| Name | Company | Catalog Number | Comments |
| 250 μm nylon mesh | Sefar Filtration | NITEX 03-250/50 | |
| 100 μm nylon mesh | Sefar Filtration | NITEX 03-100/47 | |
| Square petri dishes | Fisher Scientific | 08-757-10k | |
| Phytatrays | Sigma-Aldrich | P1552 | |
| Murashige and Skoog Basal Medium (MS) | Sigma-Aldrich | M5519 | |
| sucrose | Fisher Scientific | S5-3 | |
| MES | Sigma-Aldrich | M2933 | |
| KOH | Sigma-Aldrich | P1767 | 10 M stock |
| Eclipse 90i microscope | Nikon Instruments | ||
| Cellulase R-10 | Yakult Pharmaceutical | ||
| Macerozyme R-10 | Yakult Pharmaceutical | ||
| D-mannitol | Sigma-Aldrich | M9546 | |
| KCl | Sigma-Aldrich | P8041 | 1 M stock |
| BSA | Sigma-Aldrich | A3912 | |
| β-mercapt–thanol | Calbiochem | 444203 | |
| CaCl2 | Sigma-Aldrich | C2536 | 1 M stock |
| orbital shaker | Labline Instruments | ||
| 40 μm cell strainer | BD Biosciences | 352340 | |
| conical 15 ml tubes | BD Biosciences | 352196 | |
| table centrifuge | Sorvall, Thermo Scientific | Legend RT | |
| NaCl | Sigma-Aldrich | S3014 | |
| FACSAria | BD Biosciences | ||
| 1.5 ml microfuge tubes | VWR international | 20170-38 | |
| RNeasy micro kit | Qiagen | 74004 | |
| WT-Ovation Pico RNA Amplification System | NuGEN | 3300_12 | |
| FL-Ovation cDNA Biotin Module V2 | NuGEN | 4200_12 |
Références
- Sheen, J. Signal transduction in maize and Arabidopsis mesophyll protoplasts. Plant Physiol. 127, 1466-1475 (2001).
- Wysocka-Diller, J. W., Helariutta, Y., Fukaki, H., Malamy, J. E., Benfey, P. N. Molecular analysis of SCARECROW function reveals a radial patterning mechanism common to root and shoot. Development. 127, 595-603 (2000).
- Blilou, I., Xu, J., Wildwater, M., Willemsen, V., Paponov, I., Friml, J., Heidstra, R., Aida, M., Palme, K., Scheres, B. The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots. Nature. 433, 39-44 (2005).
- Gifford, M. L., Dean, A., Gutierrez, R. A., Coruzzi, G. M., Birnbaum, K. D. Cell-specific nitrogen responses mediate developmental plasticity. Proc Natl Acad Sci U S A. 105, 803-808 (2008).
- Bargmann, B. O. R., Birnbaum, K. D. Positive fluorescent selection permits precise, rapid, and in-depth overexpression analysis in plant protoplasts. Plant Physiol. 149, 1231-1239 (2009).
- Petersson, S. V., Johansson, A. I., Kowalczyk, M., Makoveychuk, A., Wang, J. Y., Moritz, T., Grebe, M., Benfey, P. N., Sandberg, G., Ljung, K. An Auxin Gradient and Maximum in the Arabidopsis Root Apex Shown by High-Resolution Cell-Specific Analysis of IAA Distribution and Synthesis. Plant Cell. 21, 1659-1668 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon