Method Article
Salut-C: une méthode d'étude de l'architecture tridimensionnelle des génomes.
* Ces auteurs ont contribué à parts égales
Dans cet article
Erratum Notice
Résumé
La méthode Salut-C permet l'identification sans parti pris, l'ensemble du génome des interactions chromatine (1). Salut-C couples ligature de proximité et le séquençage massivement parallèle. Les données obtenues peuvent être utilisées pour étudier l'architecture génomique à plusieurs échelles: premiers résultats identifié des caractéristiques telles que les territoires chromosomiques, la ségrégation de la chromatine ouverte et fermée, et la structure de la chromatine à l'échelle mégabase.
Résumé
Le pliage en trois dimensions des chromosomes du génome et compartimente et peut apporter éloignés des éléments fonctionnels, tels que les promoteurs et activateurs, dans la proximité spatiale proches 2-6. Décrypter la relation entre l'organisation des chromosomes du génome et de l'activité contribuera à la compréhension des processus génomique, comme la transcription et la réplication. Cependant, on sait peu sur la façon dont les chromosomes se coucher. La microscopie est incapable de distinguer un grand nombre de loci simultanément ou à haute résolution. À ce jour, la détection des interactions chromosomiques en utilisant la capture de conformation des chromosomes (3C) et ses adaptations ultérieures nécessaires au choix d'un ensemble de loci cibles, ce qui rend l'ensemble du génome d'études 7-10 impossible.
Nous avons développé Salut-C, une extension de 3C qui est capable d'identifier les interactions à longue portée dans un parti pris, l'échelle du génome de la mode. Dans Salut-C, les cellules sont fixées avec du formaldéhyde, causant des loci interagissent à être liés les uns aux autres par le biais de liaisons covalentes ADN-protéines des liens croisés. Lorsque l'ADN est ensuite fragmenté avec une enzyme de restriction, ces loci restent liés. Un résidu biotinylé est incorporé comme la 5 'surplombs sont remplis po Ensuite, ligature des extrémités franches est effectuée dans des conditions qui favorisent des événements diluer la ligature entre les fragments d'ADN réticulé. Il en résulte une bibliothèque de l'échelle du génome des produits de ligation, correspondant à des paires de fragments qui étaient à l'origine à proximité les uns aux autres dans le noyau. Chaque produit de ligation est marqué avec la biotine sur le site de la jonction. La bibliothèque est cisaillé, et les jonctions sont tirés vers le bas avec des billes de streptavidine. Les jonctions purifiés peuvent ensuite être analysées à l'aide d'un séquenceur à haut débit, résultant en un catalogue de fragments d'interagir.
Analyse directe de la matrice de contact qui en résulte révèle de nombreuses caractéristiques de l'organisation génomique, tels que la présence de territoires chromosomiques et l'association préférentielle des petits riches en gènes des chromosomes. L'analyse de corrélation peut être appliquée à la matrice de contact, ce qui démontre que le génome humain est séparé en deux compartiments: un compartiment moins dense contenant chromatine ouverte, accessible et active et un compartiment plus dense contenant fermé, inaccessible, et inactive les régions de chromatine. Enfin, l'analyse d'ensemble de la matrice de contact, couplé avec des calculs théoriques et des simulations informatiques, a révélé que, à l'échelle mégabase Salut-C révèle des caractéristiques compatibles avec une conformation globule fractale.
Protocole
Cette méthode a été utilisée dans la recherche présentée dans Lieberman-Aiden et al., Science 326, 289-293 (2009) .
Réticulation I., Digestion, Marquage des extrémités d'ADN, et ligature des extrémités franches
- Salut-C commence par réticulation de cellules, qui est un fil conducteur entre toutes les méthodes de 3C-base. Pour commencer, croître entre 2 x 10 7 et 2,5 x 10 7 cellules de mammifères, soit adhérentes ou en suspension, et réticulent les cellules. (Pour plus de détails sur la réticulation des cellules, s'il vous plaît voir: 11
- Lyse des cellules dans 550 ul lysisbuffer (500 pl 10 mM Tris-HCl pH 8,0, NaCl 10 mM, 0,2% Igepal CA-630 et 50 des inhibiteurs de protéase ul) en utilisant un homogénéisateur. Faites tourner la chromatine à 5000 rpm et laver le culot deux fois avec 500 pl 1x NEBuffer 2.
- Resuspendre la chromatine dans NEBuffer 1x 2, partie aliquote en 5 tubes numérotés et ajouter 1x NEBuffer 2 à un volume final de 362 pl. Ajouter 38 ul SDS 1%, mélanger soigneusement et laisser incuber à 65 ° C pendant 10 minutes. Placer les tubes de retour sur la glace immédiatement après l'incubation.
- Quench la SDD en ajoutant 44 ul de Triton X-100 et mélanger soigneusement. Digest de la chromatine en ajoutant 400 unités de HindIII et incuber à 37 ° C pendant la nuit tout en tournant.
- Les prochaines étapes sont Salut-C spécifiques et comprennent le marquage de l'ADN se termine avec la biotine et l'exécution ligature des extrémités franches des fragments réticulé. Cette étape permettra jonctions ligature à purifier plus tard. Tube 1 ne devraient pas subir l'étape de biotinylation et devrait plutôt être séparés et de servir de contrôle pour s'assurer que 3C digestion, et des conditions de ligature étaient optimales.
- Pour remplir les surplombs de fragments de restriction et de marquer l'ADN se termine avec la biotine dans les 4 autres tubes, ajouter 1,5 ul 10 mM, dATP 1,5 pl 10 dGTP mM, 1,5 mM dTTP 10 ul, 37,5 ul de 0,4 mM de biotine-14-dCTP et 10 Klenow ul 5U/μl aux tubes 2-5. Mélanger soigneusement et laisser incuber pendant 45 minutes à 37 ° C.
- Placer les tubes sur la glace. Pour inactiver les enzymes, ajouter 86 ul de 10% de SDS à des tubes 1-5. Incuber les tubes à 65 ° C pendant exactement 30 minutes et les placer sur la glace immédiatement après.
- La ligature est réalisée sous des conditions extrêmement diluées de manière à favoriser des événements ligature entre les fragments réticulé. Travail sur la glace, ajouter 7,61 ml mélange de ligature [745 ul de 10% Triton X-100, 745 pi de tampon 10x ligature (500 mM Tris-HCl pH 7,5, 100 mM de MgCl2, 100 mM DTT), 80 ul de 10 mg / ml de BSA , 80 pl 100 mM d'ATP et de 5,96 ml d'eau] à chacun des cinq numérotés tubes de 15 ml. Transfert chaque mélange chromatine digérée à un tube de 15 ml correspondante.
- Pour la ligature 3C régulières, ajouter 10 ul de ligase d'ADN T4 1U/μl au tube 1. Pour à bout émoussé Salut-C ligature, ajouter 50 ul de ligase d'ADN T4 1U/μl aux tubes 2-5. Mélanger en inversant les tubes et incuber tous les tubes de 5 pendant 4 heures à 16 ° C.
- Réticule sont inversés et les protéines sont dégradées en ajoutant 50 pl de 10 mg / ml de protéinase K par tube et incuber les tubes nuit à 65 ° C. Ajouter un supplément de 50 ul de 10 mg / ml de protéinase K par tube le jour suivant et continuer l'incubation à 65 ° C pendant 2 heures.
- Refroidir le mélange réactionnel à température ambiante et de les transférer à cinq tubes coniques de 50 ml. Purifier l'ADN dans ces tubes en effectuant une extraction phénol. Ajouter 10 ml de phénol pH 8,0 et le vortex pendant 2 minutes. Faites tourner les tubes pendant 10 minutes à 3500 rpm et transférer soigneusement autant de la phase aqueuse que possible à un nouveau tube de 50 ml.
- Répéter l'extraction en utilisant du phénol pH 8,0: chloroforme (1:1) et précipiter l'ADN en utilisant l'éthanol. (Pour plus de détails sur la purification de l'ADN, s'il vous plaît voir: 11
- Après centrifugation du précipité à l'éthanol de l'ADN, la dissolution de chaque culot d'ADN dans 450 ul de TE 1x (10 mM Tris-HCl pH 8,0, EDTA 1 mM). Transférer le mélange d'ADN à un tube de 1,7 ml par centrifugation.
- Un autre tour de purification est effectuée en faisant 2 phénol: chloroforme extractions. Ajouter 500 ul de phénol pH 8,0: chloroforme (1:1) et le vortex pendant 1 minute. Centrifuger les tubes pendant 5 minutes à 14000 rpm et le transfert de la phase aqueuse dans un nouveau tube. Après la deuxième extraction, précipiter l'ADN en ajoutant du volume de NaOAc 0,1 x, 2x volume d'éthanol à 100% et incuber 30 minutes à -80 ° C.
- Après filer le bas de l'ADN précipité, lavez chaque culot d'ADN à l'éthanol 70% et resuspendre chaque culot d'ADN dans 25 ul de TE 1x. Dégrader tout ARN qui pourraient être présents en ajoutant 1 pl 1 mg / ml par tube RNAse A et l'incubation des tubes pendant 30 minutes à 37 ° C. Mélangez le contenu Salut-C de tubes 2-5, en gardant le tube 1 séparée en tant que contrôle 3C.
- C'est maintenant une bonne occasion d'examiner le marquage Salut-C et l'efficacité ligature. Ces contrôles sont d'excellents indicateurs de savoir si une bibliothèque Salut-C va être couronnée de succès.
- Pour vérifier la qualité et la quantité des bibliothèques, exécutez 2 pl et 6 aliquotes de dilutions 1:10 à la fois de 3C et Salut-C des bibliothèques sur un gel agarose à 0,8%. (Voir figure 2a)
- Salut-C marquage et l'efficacité ligature Salut-C est vérifiée par un test PCR digérer. Succès fill-in et la ligature d'un site HindIII (AAGCTT) crée un site pour l'enzyme de restriction Nhel (GCTAGC). Un produit de ligature notamment formé de deux fragments de restriction à proximité est amplifié par la PCR (comme dans 3C 11 en utilisant 0,2 ul de chaque bibliothèque en tant que modèle. Les produits de PCR sont ensuite digéré par HindIII, Nhel, ou les deux. Après avoir exécuté les échantillons sur un gel à 2% , le nombre relatif de 3C et événements ligature Salut-C peut être estimée par la quantification de l'intensité des bandes coupées et non coupées (voir figure 2B).
- Certains fragments n'auront pas été ligaturé: pour éviter de les tirant vers le bas plus tard, retirez la biotine de ces extrémités non ligaturés en utilisant l'activité exonucléase de l'ADN polymérase T4.
- Biotine-14-dCTP aux extrémités d'ADN non ligaturé est enlevé avec l'activité exonucléase de l'ADN polymérase T4. Mélanger 5 pg de Salut-bibliothèque C avec 1 pi à 10 mg / ml de BSA, 10 NEBuffer ul 10x 2, 1 pl 10 mM de dATP, dGTP 1 pl 10 mM et 5 unités de T4 ADN polymérase dans un volume total de 100 pi et incuber la mélange à 12 ° C pendant 2 heures. Si possible, plusieurs réactions 5 mg sont effectuées.
- La réaction est stoppée par addition de 2 ul d'EDTA 0,5 M pH 8,0.
- Pour purifier l'ADN, un pH de phénol 8.0: chloroforme (1:1) d'extraction est réalisée suivie par précipitation à l'éthanol.
- Le surnageant est éliminé et les culots d'ADN sont remis en suspension et regroupées dans un volume total de 100 ul d'eau.
II. Shearing et Sélection de la taille
- Pour rendre l'ADN biotinylé approprié pour séquençage à haut débit, l'ADN doit être cisaillée à une taille de 300-500 paires de bases avec un instrument Covaris S2 (5 cyclique, l'intensité 5, cycles / éclatement 200, temps de 60 secondes pour les 4 cycles) .
- Pour réparer les extrémités d'ADN cisaillé, ajouter 14 ul de tampon 10x ligature, 14 ul 2,5 mM de dNTP mix, 5 ul polymérase ADN de T4, 5 ul kinase T4 polynucléotide, une polymérase de Klenow et l'ADN ul d'eau 1 pl. Incuber pendant 30 minutes à température ambiante.
- Après l'incubation, l'utilisation d'une colonne Qiagen MinElute pour purifier l'ADN selon les recommandations du fabricant. Éluer l'ADN à deux reprises avec 15 ul de Tris-Bas 1x-EDTA (TLE: Tris 10 mM pH 8,0, EDTA 0,1 mM). Ensuite, joignez une dATP aux extrémités 3 'de la fin réparé ADN en ajoutant 5 ul NEBuffer2 10x, 10 pi 1 mM de dATP, 2 et 3 de l'eau ul ul Klenow (exo-). Incuber la réaction pendant 30 minutes à 37 ° C.
- Pour inactiver le fragment de Klenow, incuber les réactions pendant 20 minutes à 65 ° C puis refroidir les réactions sur la glace. L'utilisation d'un speedvac, réduire les volumes de réaction de 20 ul.
- Ensuite, la charge de l'ADN dans un gel à 1,5% d'agarose TAE 1X et courir pendant 3,5 heures à 80-90V. Après coloration du gel avec SYBR green, de visualiser l'ADN sur un DarkReader. Fragments d'ADN d'accise entre 300 et 500 paires de bases et les purifier avec un kit Qiagen extraction de gel en utilisant 2-4 colonnes en fonction du poids du gel. Éluer l'ADN avec 50 TLE 1x ul.
- Combinez les éluats des colonnes QIAquick et porter le volume final à 300 ul avec 1x DFIT. Enfin, déterminer la concentration d'ADN avec le dosage Quant-à l'aide du fluorimètre qubit et calculer la quantité totale d'ADN.
III. Biotine déroulant et appariés fin Séquencement
- Dans cette section du protocole, les jonctions de ligation sont purifiés à partir du pool d'ADN, permettant une identification efficace de l'interaction des fragments de chromatine par jumelé bout de séquençage. Effectuez toutes les étapes ultérieures de tubes LoBind ADN.
- Préparer des billes pour la biotine déroulant en lavant 150 ul resuspendues billes de streptavidine magnétique deux fois avec 400 pi de tampon Tween (TB: 5 mM Tris-HCl pH 8,0, EDTA 0,5 mM, 1 M de NaCl, 0,05% Tween).
Ces lave et l'avenir se compose de cinq étapes:- Ajouter tampon pour les perles
- Transférer le mélange dans un nouveau tube
- Rotation de l'échantillon pendant 3 minutes à température ambiante
- Reclaim the perles en utilisant un concentrateur de particules magnétiques
- Enlever le surnageant
- Resuspendre les billes en 2x 300 ul aucun tampon Tween (2x NTB: 10 mM Tris-HCl pH 8,0, EDTA 1 mM, 2 M de NaCl) et de combiner avec 300 pl Salut-C 'ADN. Autoriser l'biotine Salut-C 'ADN de se lier aux billes de streptavidine en incubant le mélange à la température ambiante pendant 15 minutes avec une rotation.
- Reclaim the ADN lié billes de streptavidine avec la concentr particules magnétiquesAtor, et retirer le surnageant. Laver les billes dans 400 pi NTB 1x (5 mM Tris-HCl pH 8,0, EDTA 0,5 mM, 1 M de NaCl), suivie par 100 ul de tampon de ligature 1x. Resuspendre les perles dans 50 ul de tampon de ligature 1x et transférer le mélange dans un nouveau tube.
- Pour préparer l'ADN pour le séquençage Illumina Fin jumelés, prendre la quantité totale d'ADN utilisé comme entrée pour la biotine pull-down, qui a été calculé précédemment à l'étape 2.6, et le diviser par 20 pour estimer la quantité de Salut-C d'ADN qui a été tiré vers le bas et est disponible pour la ligature. Ajouter 6 picomoles de Illumina adaptateurs Fin jumelés par ug de Salut-C 'ADN disponibles pour la ligature. Utilisez 1200 unités ADN ligase T4 pour ligaturer les adaptateurs à l'ADN. Incuber pendant 2 heures à température ambiante.
- Retirez non ligaturé adaptateurs Fin jumelés en récupérant les perles Salut-C 'ADN lié et lavage des billes à deux reprises avec 400 To 1x ul.
- Laver les billes avec 200 ul 1x NTB, suivie par 200 pi et 50 pi 1x NEBuffer 2. Après le dernier lavage, remettre en suspension les perles dans 50 ul 1x NEBuffer 2 et le transfert dans un nouveau tube.
- Pour déterminer le nombre de cycles nécessaires pour générer suffisamment de produit de PCR pour le séquençage, mis en place quatre tester les réactions de PCR avec des cycles de 6, 9, 12 ou 15. (Pour plus de détails sur l'amplification par PCR, s'il vous plaît voir: 12 Déterminer le nombre de cycle optimal en exécutant les réactions PCR sur un gel de polyacrylamide à 5% et coloration au SYBR Green, garantissant l'absence de bandes parasites et la présence d'un frottis entre 400 -. 600 paires de base, qui est la longueur des produits cisaillé après ligature des adaptateurs.
- Amplifier le reste des billes de streptavidine Salut-C-bibliothèque lié dans une PCR à grande échelle avec le nombre optimal de cycles de PCR. Piscine des produits de PCR à partir des puits séparés et récupérer les perles. Gardez à 1% du produit de PCR à grande échelle séparée pour fonctionner sur un gel et de purifier le reste du produit de PCR avec des perles de 1.8x Ampure volume selon les recommandations du fabricant.
- Éluer l'ADN avec un tampon de 50 TLE ul 1x et de comparer à 1% du produit Ampure perles PCR purifié à l'aliquote de 1% du produit original PCR sur un gel de polyacrylamide à 5%, assurant l'évacuation réussie des amorces PCR.
- Nous recommandons également le clonage 1 pl de la bibliothèque Salut-C et déterminer le produit d'environ 100 clones en utilisant le séquençage Sanger. Cela vous permettra d'évaluer le nombre relatif de alignable Salut-C lit dans le mélange PCR. Pour des résultats typiques, voir la figure 3B.
- Séquence de la bibliothèque Salut-C avec le séquençage Illumina fin appariés. Alignez chaque extrémité de façon indépendante en utilisant Maq (http://maq.sourceforge.net/) afin d'identifier des fragments de chromatine en interaction.
IV. Représentant Salut-C Résultats
- Les résultats suivants sont attendus lorsque le protocole Salut-C est techniquement bien exécutés et peuvent être considérées comme des normes de contrôle qualité.
- Étapes de contrôle qualité devrait révéler que les deux bibliothèques 3C et Salut-exécution C bandes comme plutôt serré de plus de 10 ko. Un frottis ADN indique l'efficacité ligature pauvres. Typiquement, l'efficacité de ligature est légèrement inférieur dans une bibliothèque de Salut-C par rapport à un modèle 3C (voir figure 2a).
- Salut-C marquage et l'efficacité ligature peut être estimée par la digestion d'un produit de PCR générés en utilisant des amorces 3C. Jonctions 3C sont coupés par HindIII et non pas par Nhel. L'inverse est vrai pour Salut-C jonctions. Cette analyse PCR Digest montre que 70% de Salut-C amplicons sont coupées par Nhel et non pas par HindIII, confirmant le marquage efficace de jonctions ligature (voir figure 2B).
- L'analyse de la lecture séquencé devrait montrer que les interactions se lit de deux intrachromosomique et interchromosomique, indiqué par les lignes bleues et rouges, d'aligner beaucoup plus proche de sites de restriction HindIII par rapport à générées aléatoirement lit, représenté en vert (voir figure 3A).
- Dans une expérience réussie, 55% des paires alignable lire représenter les interactions interchromosomique. Quinze pour cent représentent les interactions entre les fragments intrachromosomique moins de 20 ko à part et 30% sont intrachromosomique paires lu que plus de 20 ko de l'autre (voir figure 3B). Cette distribution peut être échantillonné avant séquençage à haut débit, comme une forme de contrôle de qualité; clonage et de séquençage Sanger d'environ 100 clones est généralement suffisant.
- Les interactions chromatine peut être visualisé comme une heatmap, où x et y représentent les axes des loci génomiques et pour chaque pixel représente le nombre d'interactions observées entre les deux. Typiquement, des fragments d'ADN qui sont très proches les unes aux autres dans le génome linéaire aura tendance à interagir fréquemment avec eux. Cela se voit dans l'heatmaps intrachromosomique comme une diagonale de premier plan (voir figure 4A).
- Les résultats suivants montrent différentes façons d'analyser les données pour révéler les différents niveaux de l'organisation du génome. Tracé de la probabilit de contactdistance y génomique rapport (voir figure 5A) montre que la probabilité de contact diminue en fonction de la distance génomique, pour finalement atteindre un plateau. A chaque distance, les interactions intrachromosomique, indiqué dans la ligne solide, sont enrichies par rapport aux interactions interchromosomique, représentée par les lignes pointillées. Ceci implique directement la présence de territoires chromosomiques.
- Calcul du nombre observé / attendu de contacts interchromosomique entre toutes les paires de chromosomes révèle association préférentielle entre les paires de chromosomes particuliers. Chromosomes riches en gènes préférentiellement petites interagissent les uns avec les autres, indiqué par la couleur rouge vif (voir figure 5b).
- Chromosomes individuels peuvent également être examinés. Le heatmap premières peuvent être ajustées en utilisant une heatmap devrait tenir compte de la distance génomique entre les paires de loci, résultant en une heatmap observé / attendu. Ensuite, une matrice de corrélation peut être produite en corrélant les lignes et les colonnes de la heatmaps observé / attendu. En utilisant l'analyse de corrélation, il est démontré que le génome humain sépare en deux compartiments. Ceci est illustré par le plaid-modèle dans le heatmaps corrélation (voir la figure 4A-D). (Pour plus de détails sur l'analyse des données Salut-C, s'il vous plaît voir: 1.
- Utiliser Salut-C données, de nouvelles idées ont été acquises dans le pliage de chromatine à l'échelle mégabase. Le modèle classique de polymère de condensation suggère que la chromatine packs en un globule équilibre. La probabilité de contact Tracé en fonction de la distance montre que la probabilité de contact échelles comme une loi de puissance avec la distance génomique dont la pente est d'environ -1 (voir figure 6A). Ce n'est pas cohérent avec le comportement d'un globule d'équilibre, mais ne attentes match pour une autre structure connue comme un globule fractale (voir figure 6B).
- Ici, deux structures globulaires sont affichés. Coloration correspond à la distance d'une extrémité, allant du bleu au cyan, vert, jaune, orange et rouge (voir en haut figure 6C). Contrairement globules équilibre, globules fractale manque enchevêtrements. Dans un globule fractale, loci qui sont à proximité le long du contour ont tendance à être proches en 3D, ce qui conduit à la présence de blocs monochromes (voir la figure 6C milieu). Ces blocs ne sont pas trouvés dans le globule d'équilibre (voir en bas figure 6C).
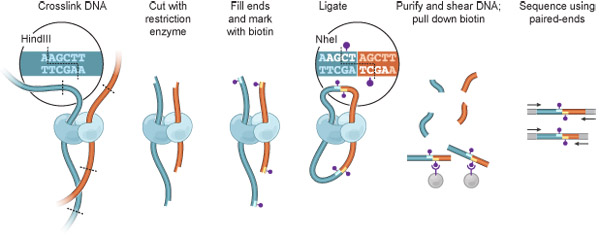
Figure 1. Salut-C aperçu. Les cellules sont réticulés avec du formaldéhyde, ce qui entraîne des liaisons covalentes entre les segments de la chromatine spatialement adjacents (fragments d'ADN: bleu foncé, rouge, les protéines, ce qui peut servir de médiateur de telles interactions, sont représentées en bleu clair et cyan). La chromatine est digéré par une enzyme de restriction (ici, HindIII; site de restriction: ligne pointillée, voir encadré). Les extrémités résultant collants sont remplis avec des nucléotides, dont l'un est biotinylé (point violet). La ligature est effectuée dans des conditions extrêmement diluées conditions favorisant des événements ligature intramoléculaire; le site HindIII est perdu et un site Nhel est créé (en médaillon). L'ADN est purifié et cisaillé, et les jonctions biotinylé sont isolées en utilisant des billes de streptavidine. Interagir fragments sont identifiés par séquençage jumelé-end.

Figure 2. Salut-C contrôle la qualité de la bibliothèque. (A) Des quantités croissantes d'un contrôle et une bibliothèque 3C Salut-C ont été résolus sur un gel agarose à 0,8%. Les deux bibliothèques d'exécution en tant que groupe assez serré de plus de 10 ko. L'efficacité ligature typique dans une bibliothèque de Salut-C est légèrement inférieure à ce qui est observé dans un modèle 3C, et est indiqué par le frottis dans les ruelles Salut-C. (B) PCR digérer contrôle. Une jonction ligature formée par deux fragments voisins est amplifié en utilisant la norme 3C conditions de PCR. Produits de ligation Salut-C peuvent être distingués de ceux produits dans conventionnelle 3C de la digestion des site de ligature. Salut-C jonctions sont coupés par Nhel, pas HindIII; l'inverse est vrai pour les jonctions 3C. 70% de Salut-C amplicons ont été coupées par Nhel, confirmant l'efficacité marquant de jonction ligature. Deux répétitions ont été effectuées afin d'assurer une quantification fiable.

Figure 3. Salut-C lire des contrôles de qualité. (A) se lit à partir de fragments correspondant à la fois intrachromosomique (bleu) et interchromosomique (rouge) d'aligner les interactions beaucoup plus proche de sites de restriction HindIII par rapport à lit générés aléatoirement (en vert). Tant le intrachromosomique lit et lit interchromosomique courbes décroissent rapidement la distance entre le site HindIII augmente jusqu'à un plateau est atteint à une distance de ~ 500 pb. Cela correspond à la taille du fragment maximum utilisé pour le séquençage. (B) En règle générale, 55% des paires alignable lire représenter les interactions interchromosomique. Quinze pour cent représentent les interactions entre les fragments intrachromosomique moins de 20 kopart et 30% sont intrachromosomique paires lu que plus de 20 ko à part. Cette distribution peut être échantillonné avant séquençage à haut débit, comme une forme de contrôle de qualité; clonage et de séquençage Sanger d'environ 100 clones est généralement suffisant.

Figure 4. L'analyse de corrélation montre que le noyau est séparé en deux compartiments. (A) correspondant à des interactions Heatmap intrachromosomique sur le chromosome 14. Chaque pixel représente toutes les interactions entre un lieu de 1 Mo et d'un autre locus 1-Mo; intensité correspond au nombre total de lectures (gamme: 0-200 lectures). Les graduations apparaissent tous les 10 Mb. La sous-structure heatmap expositions sous la forme d'une diagonale intense et une constellation de grands blocs. (Chromosome 14 est acrocentriques;. Le bras court n'est pas représenté) Utilisation de l'ensemble de données Salut-C pour calculer la probabilité de contact moyenne pour une paire de loci génomiques à une distance donnée, une matrice d'attente est produite (B) correspondant à ce qui serait observée s'il n'y avait pas de structures à longue portée. Le quotient de ces deux matrices est une matrice observé / attendu (C) où l'épuisement est représentée en bleu et l'enrichissement en rouge [plage: 0,2 (bleu) à 5 (rouge)]. Le schéma bloc devient plus évidente. La corrélation (D) illustre la corrélation matricielle [plage: -1 (bleu) à 1 (rouge)] entre les profils d'interaction intrachromosomique de chaque paire de loci long du chromosome 14. Le modèle de plaid frappante indique la présence de deux compartiments dans le chromosome.

Figure 5. La présence et l'organisation des territoires chromosomiques. (A) Probabilité de contact diminue en fonction de la distance génomique sur le chromosome 1, pour finalement atteindre un plateau à ~ 90MB (bleu). Le niveau de contact interchromosomique (tirets noirs) diffère pour les différentes paires de chromosomes; loci sur le chromosome 1 sont les plus susceptibles d'interagir avec des loci sur le chromosome 10 (tirets verts) et les moins susceptibles d'interagir avec des loci sur le chromosome 21 (tirets rouges). Interactions interchromosomique sont épuisés par rapport aux interactions intrachromosomique. (B) observé / attendu nombre de contacts interchromosomique entre toutes les paires de chromosomes. Le rouge indique l'enrichissement et bleue indique l'épuisement [plage: 0,5 (bleu) à 2 (rouge)]. Petit, riches en gènes des chromosomes ont tendance à interagir davantage avec l'autre.

Figure 6. L'emballage locale de la chromatine est cohérente avec le comportement d'un globule fractale. (A) la probabilité de contact en fonction de la distance génomique, en moyenne sur tout le génome (en bleu). Une mise à l'échelle de puissance de premier plan loi est observée entre 500kb et 7Mo (ombrée) avec une pente de -1,08 (ajustement montré en cyan). (B) Les résultats de simulation de la probabilité de contact en fonction de la distance d'équilibre (rouge) et fractale (en bleu) des globules. La pente d'un globule fractale est très près -1 (cyan), confirmant ainsi notre prévision roman théorique 1. La pente d'un globule équilibre est -3 / 2, ce qui correspond aux attentes théorique préalable. La pente pour le globule fractale ressemble étroitement à la pente observée dans les résultats Salut-C, alors que la pente pour un globule équilibre n'est pas vu dans les données de Salut-C. (C) En haut: Une chaîne de polymère déplié, 4000 monomères de long. Coloration correspond à la distance d'une extrémité, allant du bleu au cyan, vert, jaune, orange et rouge-Orient:. Exemple typique d'un globule fractale tirées de notre ensemble. Globules Fractal manque enchevêtrements. Loci qui sont à proximité le long du contour ont tendance à être proches en 3D, ce qui conduit à la présence de grands blocs monochromes qui sont apparentes à la surface et en coupe du bas:. Un globule de l'équilibre. La structure est très enchevêtré; loci qui sont à proximité le long du contour (couleur similaire) ne doivent pas être à proximité en 3D.
Discussion
Nous présentons une méthode d'étude de l'architecture 3-dimensions du génome par la cartographie des interactions chromatine dans un parti pris, l'échelle du génome manière. L'étape la plus critique expérimentale ce qui distingue cette technologie en dehors du travail précédent - est l'incorporation de nucléotides biotinylés à la restriction extrémités des fragments réticulé avant ligature des extrémités franches. L'exécution de cette étape permet succès profonde séquençage de tous les carrefours ligature, et donne Salut-C sa portée et sa puissance.
Le nombre de lectures détermineront finalement la résolution des cartes d'interaction. Ici, une carte 1 Mo d'interaction pour le génome humain est présentée en utilisant ~ 30 millions de lectures alignable. Afin d'augmenter la résolution «tout usage» par un facteur de n, le nombre de lectures doit être augmenté par un facteur de N 2.
La technique de Salut-C peuvent être facilement combinés avec d'autres techniques, telles que la capture hybride après la génération de la bibliothèque (pour cibler des parties spécifiques du génome) et immunoprécipitation de la chromatine après ligature (pour étudier l'environnement des régions de la chromatine associée à des protéines spécifiques).
Déclarations de divulgation
Remerciements
Nous remercions A. Kosmrlj pour les discussions et le code; AP Aiden, XR Bao, M. Brenner, D. Galas, W. Gosper, A. Jaffer, A. Melnikov, A. Miele, G. Giannoukos, C. Nusbaum, AJM Walhout , L. Wood, et K. Zeldovich pour les discussions, et L. Gaffney et B. Wong pour aider à la visualisation.
Soutenu par une Fannie et John Hertz Fondation bourse d'études supérieures, une défense nationale des sciences et de génie bourse d'études supérieures, une bourse d'études supérieures NSF, la National Space Biomedical Research Institute, et d'accorder aucune. T32 HG002295 du National Human Genome Research Institute (NHGRI) (EL); i2b2 (Informatique pour l'intégration de la biologie et la recherche clinique), le NIH soutenus Centre de Calcul Biomédical au Brigham and Women s Hospital (LAM); accorder aucune. HG003143 du NHGRI, et un Prix d'excellence de la Fondation Keck jeune érudit (JD). Raw et cartographié les données de séquence Salut-C a été déposé à la base de données GEO ( www.ncbi.nlm.nih.gov/geo/ ), accession no. GSE18199. Visualisations supplémentaires sont disponibles à http://hic.umassmed.edu .
matériels
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
Références
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon