Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Microscopie dispersion optique basé sur des filtres de Gabor bidimensionnelle
Dans cet article
Résumé
Nous démontrons une méthode de microscopie en champ sombre repose sur Gabor-like de filtrage pour mesurer la dynamique subcellulaire dans les cellules vivantes unique. La technique est sensible aux modifications dans la structure des organelles, comme la fragmentation mitochondriale.
Résumé
Nous démontrons un instrument microscopiques qui peuvent mesurer la texture subcellulaire découlant de la morphologie et l'organisation des organites dans les cellules vivantes non colorées. L'instrument proposé s'étend de la sensibilité de l'étiquette sans la microscopie optique à des changements dans la taille nanométrique des organelles et la forme et peut être utilisé pour accélérer l'étude de la relation structure-fonction se rapportant à la dynamique sous-jacente organelle processus biologiques fondamentaux, tels que la mort cellulaire programmée ou cellulaire différenciation. Le microscope peut être facilement mis en œuvre sur les plates-formes de microscopie existant, et peut donc être diffusé aux laboratoires individuels, où les scientifiques peuvent mettre en œuvre et utiliser les méthodes proposées avec un accès illimité.
La technique proposée est en mesure de caractériser la structure subcellulaire par l'observation des cellules à travers deux dimensions filtres de Gabor optique. Ces filtres peuvent être réglés à l'échelle nanométrique sens avec (10 nm de) la sensibilité, des attributs spécifiques morphologiques relatives à la taille et l'orientation de la non-sphériques organites subcellulaires. Bien que basé sur le contraste généré par la diffusion élastique, la technique ne repose pas sur un modèle de diffusion inverse détaillée ou sur la théorie de Mie pour extraire des mesures morphométriques. Cette technique est donc applicable aux non-sphériques organites pour lesquels une description précise de dispersion théorique n'est pas facile étant donné, et fournit des paramètres morphométriques distinctifs qui peuvent être obtenues dans les cellules vivantes non colorées pour évaluer leur fonction. La technique est avantageuse par rapport à traitement d'images numériques en ce qu'elle opère directement sur le champ de l'objet plutôt que de transformer l'intensité de l'objet discrétisé est. Il ne repose pas sur les taux d'échantillonnage d'image et peut donc être utilisé pour dépister rapidement l'activité morphologique au sein de centaines de cellules à un moment, ce qui facilite grandement l'étude de la structure des organelles delà organite la segmentation individuelle et de la reconstruction par microscopie confocale de fluorescence fortement agrandie des images numériques des champs de vision limité.
Dans cette démonstration, nous montrons les données d'une diatomée marine pour illustrer la méthodologie. Nous montrons aussi les données préliminaires recueillies auprès des cellules vivantes pour donner une idée de comment la méthode peut être appliquée dans un contexte biologique pertinent.
Protocole
1. Obtenir des cellules prêtes
- Les cellules qui ont été étalées la veille doivent être étiquetés avec le vert Mitotracker pour l'imagerie de fluorescence de la mitochondrie.
- Retirer 100 uM solution stock de vert dans le DMSO Mitotracker faites précédemment dans le congélateur de 4 ° C, et à température ambiante avec la main. Veuillez également prendre des cellules endothéliales aortiques bovines (CEAB) milieu de culture cellulaire aussi préalablement préparée et chaude à 37 ° C au bain-marie paillasse.
- Une fois que le milieu de culture et de Mitotracker sont réchauffés, les situer dans le capot en prenant soin de stériliser vos mains gantées et toutes les surfaces extérieures des conteneurs avec une solution d'éthanol à 70%. Ne pas allumer la lumière capot, comme le marqueur fluorescent est sensible à la lumière et va rapidement photobleach à la lumière ambiante.
- Rendre la bonne concentration pour l'étiquetage des mitochondries est très important. Trop peu de ne pas étiqueter les mitochondries de manière efficace, tout en Mitotracker trop peut avoir des effets toxiques. Une concentration de 100 nM de Mitotracker incubées pendant 45 minutes avec les cellules fonctionne bien. Préparer cette concentration en ajoutant 100 uL de stock Mitotracker à 10 ml milieu de culture dans un tube de 15 ml. Cela fera beaucoup pour au moins une expérience.
- Remplacer le support existant avec le milieu étiquetés en aspirant l'ancien milieu avec une pipette Pasteur connecté à la ligne de vide. Puis, aussitôt, ajouter 2 mL du milieu étiquetés à chaque culture occupait bien dans la plaque de 6 puits.
- Parce que le marqueur fluorescent est sensible à la lumière, de remplacer les cellules dans l'incubateur rapidement sans exposer à la lumière ambiante directe. Couvrant la plaque 6 puits avec les mains qui fonctionne bien pour cela. Les cellules resteront dans l'incubateur pendant 45 minutes.
2. Obtenir le montage optique prête
- Bien que les cellules d'attendre dans l'incubateur, nous devons nous tourner sur le banc optique. Dans la salle de l'optique, d'abord allumer la lampe à arc au mercure, suivie par l'ordinateur, le microscope, les caméras, et le laser. Ensuite, branchez l'appareil micromiroirs numériques (DMD) et le diffuseur de tourner.
- Assurez-vous que le lancement optique est aligné en regardant à travers l'oculaire du microscope afin de s'assurer que le champ de vision est brillamment éclairée par la lumière laser.
- Nettoyez l'objectif en pliant un morceau de papier pour objectif dans un carré serré et bien saisir avec une pince hémostatique. Trempez le papier pour lentilles en ammoniac solution sans nettoyage du verre pour absorber une quantité minuscule dans le papier. Rap du hémostatique sur votre main libre à plusieurs reprises pour enlever l'excédent. Essuyez l'objectif en appliquant fermement un propre, balayage continu à travers l'objectif d'un bout à l'autre, allant sur la lentille dans le milieu. Ne pas ré-glisser ou frotter. Jeter le papier utilisé.
- Pour charger l'échantillon, placez le réticule sur l'objectif à immersion 63x en laissant tomber 1-2 gouttes d'huile d'immersion petits sur l'objectif alors que l'objectif est tout en bas. Ensuite, placez le réticule dans le stade. Puis levez l'objectif pour que l'huile «attrape» de l'échantillon. Concentrer l'échantillon dans l'oculaire.
- Pour aligner le condenseur, ajuster la hauteur du condenseur afin qu'il soit aligné au centre éclairage de Köhler en concentrant le bord hexagonale de l'arrêt de terrain condenseur. Centre sur le terrain du condenseur arrêter sur le champ de vue, si nécessaire en tournant les boutons du condenseur de centrage. Le diaphragme du condenseur doit être fermé.
- Démarrez le programme IPLab et les paramètres d'entrée pour faire fonctionner le Cascade RoperScientific 512 caméras. Vérifiez que l'appareil est réglé pour encadrer le mode de transfert. Démarrer l'aperçu en direct en exécutant la "Acquérir focus" de commande. Régler le préfixe d'index et l'emplacement du fichier à laquelle les images seront sauvegardées.
- Démarrez le programme RSImage et les paramètres d'entrée pour faire fonctionner le programme de Coolsnap. Mode Clocking devrait être fixé à la normale.
- Démarrez le logiciel DMD et le lieu de l'iris en champ sombre sur le menu script, suivi par le "Load and Reset" commande et exécutez le script.
- Envoyer la lumière de la DMD et Cascade 512 caméras en réglant le microscope et optovar viewport à LSM. Ce sera d'envoyer l'image à travers la DMD et les optiques alignées, projetant l'image sur le CCD DF. Le champ sombre (DF) l'image doit apparaître sur l'aperçu en direct déjà en cours dans IPLab. Réglez la mise au point fine du microscope si nécessaire pour focaliser l'image sur l'aperçu en direct.
- Prendre un instantané du champ de vision avec le «acquérir seule commande". Réglez le temps d'exposition suffisamment élevé pour assurer au moins 10 000 chefs d'accusation de signal dans l'image. Après l'acquisition, l'utilisation du "enregistrer sous indexé" commande pour enregistrer l'image sur le disque. Cette image du réticule mesures de la taille du champ de vision (FOV).
- Maintenant, déplacez le réticule de l'échantillon de sorte que le réticule est au-delà des FOV afin de fond ne soit visible. Acquérir une image de fond du champ à unlongs temps d'exposition suffisant pour s'assurer qu'au moins 5000 points de signal est acquis. Cette image sera une aide dans le fond la soustraction des images non filtrées.
3. Chargement de la batterie de filtres et d'utiliser l'installation d'acquérir des images filtrées-fond
- Maintenant, nous avons besoin d'acquérir Gabor filtré des images de l'arrière-plan. Chargez le filtre de Gabor bancaire script pour le logiciel de contrôle de DMD. Exécuter le script entier pour amortir les filtres à la mémoire embarquée de la DMD, ce qui pourrait prendre quelques minutes.
- Une fois le script entier est tamponné, nous pouvons maintenant acquérir des images filtrées sur le fond. Utilisez le démarrage et l'arrêt des marqueurs dans le logiciel DMD de charger le DMD de charger un seul ensemble de filtres correspondant à une Gabor-comme le filtre à la fois, et lancez le script. La prévisualisation des images en direct devrait changer de champ sombre à l'image filtrée pour ce filtre.
- Ouvrez le script d'acquisition dans IPLab partir du disque. Réglez le temps d'exposition afin d'assurer qu'au moins 2000 points de signal sont en cours d'acquisition. Comme le script DMD est en marche, d'annuler l'aperçu en direct de IPLab et exécuter le script d'acquisition. Cela va automatiquement acquérir, enregistrer et de conserver l'image filtrée sur le disque.
- Une fois la première image est acquise, arrêtez le script qui s'exécute dans le logiciel DMD et supprimer les commandes utilisées par le script. Remplacez les marqueurs de début et d'arrêt au début et à la fin de l'ensemble de filtre suivant. Répétez l'acquisition en IPLab.
- Répétez l'étape 3.4 jusqu'à ce que la totalité de filtres ont été utilisés et toutes les images filtrées ont été acquises et enregistrées.
4. Placage des cellules
- A présent, les cellules seront bientôt prêts à l'assiette pour l'expérience. Branchez le fer à souder sur la paillasse du laboratoire. Retirez le support L15 visualisation et de la chaleur à 37 ° C. Faire un poste de travail avec une serviette en papier et un Kimwipe. Faire plusieurs mèches en déchirant et virevoltant Kimwipes. Les mèches aidera à transfert de fluide vers et à partir de la plaque cellulaire.
- Après cela, nous avons besoin de plaque de l'échantillon. Nous utilisons les détenteurs usinées échantillon métallique à l'assiette de nos cellules, faisant un "sandwich lamelle" avec la plaque de métal entre les deux. Appliquez un mince cordon de graisse à vide à partir d'une seringue à la périphérie supérieure du trou plaque métallique s'étendant sur environ la moitié de l'extrémités des rainures de chaque côté. Appuyez doucement sur une aucune propre. Une lamelle sur la graisse. Retournez la plaque sur et appliquer de la graisse autour du trou. Éteignez les lumières chambre.
- Maintenant nous obtenons les cellules de l'incubateur, la manipulation du contenu incubateur avec des gants en nitrile examen stérilisés avec de l'éthanol à 70%. Retirer la plaque de cellules issues de l'incubateur, en retenant votre souffle tandis que la porte de l'incubateur est ouvert. Soyez prudent de minimiser l'exposition à la lumière ambiante.
- Enlever les lamelles qui seront utilisées pour l'expérience de la plaque de six puits, notant que le côté qui était face vers le haut dans le puits est le côté avec les cellules ci-joint. Séchez soigneusement la lamelle sur les deux côtés jusqu'à ce qu'il soit presque totalement sec, tout en gardant une trace de quel côté de la lamelle a les cellules. Ensuite, appuyez sur le côté pile lamelle, en bas, dans la plaque de métal graissé sur le trou d'observation, en s'assurant qu'aucun entrefers restent dans la couche de graisse. La graisse doit former un joint étanche à l'eau pour permettre de nous charger les cellules avec le milieu L15. Lorsque vous êtes certain de cela, retournez la plaque arrière plus.
- Pipeter le support dans les cellules L15 plaqués en forçant le liquide à travers la gorge entre la lamelle supérieure et la plaque métallique. Pipetage 200 ul à un moment fonctionne bien. La pipette premier volume devrait remplir l'espace en sandwich entre les lamelles avec le liquide qui s'étend presque à la rainure de l'autre côté.
- Pipeter une autre uL 200 de moyenne dans les cellules plaqués, mais cette fois, tenir une mèche à la palmeraie de s'opposer pour que le fluide circule d'un côté à l'autre. Ce lave les cellules et supprime toute trace de l'ancien milieu. Soyez prudent pour éviter toute formation de bulles dans le liquide pendant cette étape. Répétez ce processus 2-3 fois en utilisant une mèche neuve pour chaque rinçage.
- Rappelez-vous le fer à souder, nous branché? C'est maintenant le temps qu'il s'habitue. Retourner la plaque à l'envers une fois de plus, en soutenant la plaque sur les bords de sorte que le liquide est piégé dans le réservoir de cellules et ne peut pas goutte à goutte vers le bas. Tremper le fer à souder dans le bécher valap. Ce sera rapidement fondre certains des valap qui seront ensuite s'accrocher à la pointe du fer à souder. Soigneusement appliquer les valap fondue sur les bords de la lamelle du bas (qui est maintenant tournée vers le haut) en utilisant la pointe du fer à souder comme un applicateur. Continuez à descendre et l'application jusqu'à ce qu'un, vous aller tout le chemin autour du périmètre lamelle, la lamelle d'étanchéité à la plaque de métal.
- La lamelle du bas a des cellules en croissance sur elle, et peut avoir des résidus dans le milieu asséché sur le côté exposé. Nettoyer tout résidu de la surface de la lamelle par Balling un Kimwipe et Cleaning la lamelle dans un seul mouvement coulissant tout comme le nettoyage de l'objectif. Cela garantit que la lamelle est plus propre dans le centre où il sera consulté.
- Débranchez le fer à souder et de retourner la plaque de 6 puits d'observation de l'incubateur le confinement et les mêmes procédures de stérilité. Prenez les cellules plaquées au laboratoire optique et monter sur l'objectif tel que décrit dans les étapes 2.4 et 2.5.
5. Déroulement de l'expérience
- Trouver un beau champ d'apparence saine des cellules.
- Acquérir une image en champ sombre du champ de vue. Aligner le microscope en contraste d'interférence différentiel (DIC) et acquérir une image DIC. Assurez-vous que le temps d'exposition sont suffisamment longs pour s'assurer que le signal est suffisante.
- Maintenant, nous devons acquérir les images fluorescentes sur l'autre caméra. Pour obtenir des images sur le DIC Coolsnap, nous utilisons une LED bleue attachée au condenseur, la remplacer et de le retirer si nécessaire. Alors que le microscope est toujours alignée dans la DIC, envoyer la lumière à l'Coolsnap en définissant la optovar microscope pour 1.0x et fenêtre pour jumelles de 100%. Détourner l'image de l'oculaire de la caméra. Placer la LED sur le condenseur pour éclairer le champ et de prévisualiser le FOV dans RSimage et ajuster au point fine, si nécessaire. Acquérir une image DIC et enregistrer sur le disque. Notez comment le champ de vision est différente de celle obtenue par la caméra de Cascade. Ces images devront être enregistrées au cours de la phase d'analyse après l'expérience.
- Obtenir une image de fluorescence en ajustant le cube de filtre à la FILTERCUBE fluorescéine. Acquérir une image par une brève tourner sur l'excitation de fluorescence à l'aide du microscope, puis l'éteindre dès que l'acquisition est terminée. Depuis nous nous sommes concentrés dans l'échantillon DIC, l'image de fluorescence est axé aussi bien. Cela permet d'économiser sur les temps d'exposition en fluorescence, ce qui ralentit photoblanchiment. Enregistrer l'image de fluorescence sur le disque.
- Maintenant, nous devons acquérir les images filtrées. Réinitialiser le microscope à fond noir et renvoyer la lumière à travers le port LSM comme dans 2.9.
- Exécutez le script de filtres de Gabor ensemble comme dans 3.3 à 3.5. Nous avons maintenant complété l'acquisition de données pour un seul point du temps.
6. Commutation de la moyenne pour exposer les cellules à la staurosporine (STS), et maintenant le milieu à travers l'expérience
- Alors que les cellules sont encore sur la scène et sans perturber le champ de vision, passer régulièrement le milieu L-15 pour la même contenant une solution 1 M de STS fait à partir d'une solution 4 mM de STS dans le DMSO. Utilisez la méthode décrite dans les étapes mèche de 4,6 à passer dans les médias.
- Maintenant, nous répétez les étapes 5.2 à 5.6 pour les points de temps ultérieurs. Nous répétons ce processus jusqu'à ce que l'expérience est terminée.
- Au cours de l'expérience, plus de support devra être ajoutée afin de l'échantillon ne pas dessécher. Ceci est accompli par pipetage moyennes dans le bosquet de la plaque cellulaire sans avoir à retirer de la scène et sans perturber le FOV.
7. Les résultats représentatifs
A l'issue de l'expérience, les données collectées comprennent un grand nombre d'images filtrées qui doivent être traitées pour en extraire les données structurelles subcellulaire. Deux exemples sont présentés pour une banque filtre optique composé de 9 Gabor-like avec des filtres = période de filtre 0.95μm S, écart gaussien enveloppe standard s = S / 2 = 0.45μm, et les orientations Φ = 0 ° à Φ = 160 ° en 20 ° sur. (Voir aussi [1] pour plus de détails).
Exemple 1: diatomée marine
Nous avons d'abord appliqué notre orientation banque sensibles filtre à un échantillon de diatomées marines (Carolina Biological Supply Company) avec des fonctionnalités orientées qui étaient clairement visibles dans les champ sombre (DF) d'imagerie (fig. 1). Les images filtrées sont optiquement montré aux côtés de l'image non filtrée de l'échantillon de comparaison.
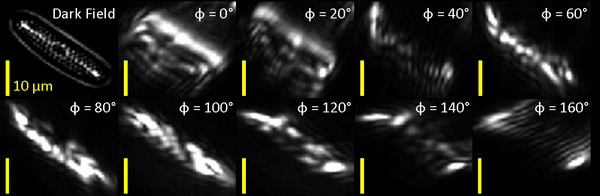
Figure 1: Fond noir (DF) et l'image filtrée optiquement des diatomées marines. Nous allons analyser les diatomées dans le coin inférieur droit de l'image (flèche blanche dans le panneau le plus à gauche).
L'ensemble de neuf Gabor filtré des images de la diatomée ont été traitées pixel par pixel pour l'orientation objet et de rondeur. Traitement consisté (1) en additionnant les réponses mesurées de l'ensemble des neuf Gabor filtré des images à chaque pixel afin de déterminer l'ampleur globale de la réponse du signal de ce fait l'importance d'encodage de réponse, et (2) de trouver l'orientation filtre de Gabor, Φ, à laquelle la réponse est maximisée et prenant le rapport de cette réponse maximale à la moyenne des réponses pour tous les angles ainsi codant pour la mesure dans laquelle les objets à chaque pixel ont une orientation préférentielle. Le degré d'orientation est étroitement liée à l'aspect ratio géométrique de la particule. Dans la Fig. 2B, ee de réponse global du pixel à la banque de filtre (paramètre 1) et le degré d'orientation ou de ratio d'aspect (paramètre 2) sont encodées dans la saturation des couleurs et la teinte, respectivement. Un aspect ratio proche de 1 (bleu) est présent dans les zones où il n'ya pas de réponse d'angle préféré, tandis que des valeurs plus grandes (rouge) indiquent les domaines dans lesquels une réponse angle supérieur préféré est présent. L'orientation des particules sous-structure est encodée dans un complot carquois (figure 2C), où chaque ligne de près d'accord avec l'orientation objet local qui sous-tendent visible dans filtré en champ sombre (Fig. 2A).

Figure 2: A: image de champ obscur de diatomées. B: orientation de l'image de l'objet. Échelle de couleur indique le degré d'orientation (aspect ratio), tandis que la luminosité encode signification de la réponse totale filtre de Gabor. C: l'orientation des objets avec une intensité de réponse ≥ 10% du maximum. Segment de ligne indique axe long de la structure correspondant.
Exemple 2: Les cellules apoptotiques
Ici, nous montrons des images filtrées des cellules endothéliales bovines traitées avec la staurosporine (STS) qui ont été traitées de la même manière que les diatomées. Fig. 3 montre une non filtrée en champ sombre (DF) l'image des cellules avec les neuf images filtrées à l'instant T =- 180 min. avant le traitement STS.
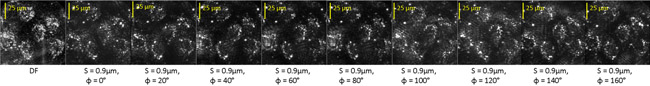
Figure 3: Fond noir (DF) et des images optiquement filtrées d'un champ contenant plusieurs cellules vivantes endothéliales.
Les images filtrées ont ensuite été acquises toutes les 20 minutes pendant une période de trois heures après le traitement STS. Fig. 4a montre une carte de rapport d'aspect des cellules en fonction du temps. Dans ce cas, la teinte couleur représente le degré d'orientation (orientation vers étiqueté) comme pour la nuance de couleur dans la Fig. 2b ci-dessus. Toutefois, la luminosité ratio d'aspect n'a pas été pondérés par le moyen de réponse du filtre. En vous inscrivant nos cartes ratio d'aspect avec des images de fluorescence de la mitochondrie étiquetés dans ces cellules (fig. 4b), nous avons déterminé que la baisse mesurée aspect ratio était confinée aux régions cellulaire contenant des mitochondries et a été concomitante avec la fragmentation mitochondriale qui pourraient être observés directement en les images de fluorescence de ces mêmes cellules. Fig. 5 montre parcelles de temps illustrant le changement de ratio d'aspect en fonction du temps dans des cellules en apoptose. Dans chaque cellule, il ya une baisse de ratio d'aspect à T = 60 à 100 min dans les régions qui enregistrent fluorescentes mitochondries, mais pas dans les régions qui enregistrent les zones de fond de fluorescence DIM.
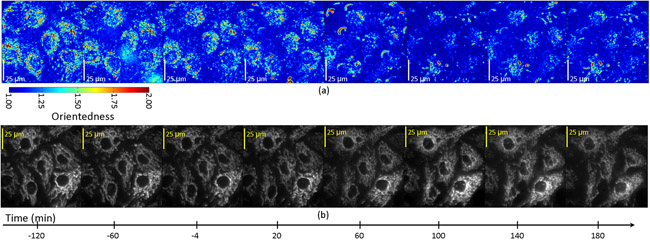
Figure 4: Aspect ratio (a) et fluorescence (b) les images des cellules endothéliales traitées avec l'inducteur d'apoptose, la staurosporine.
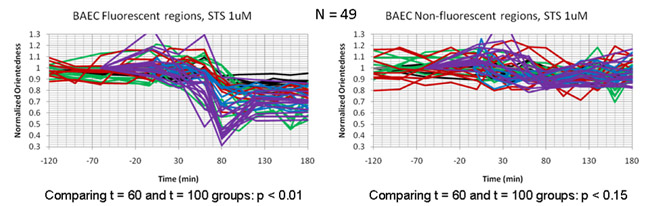
Figure 5: parcelles de temps à comparer la diminution de ratio d'aspect de particules (orientation vers) dans les cellules endothéliales traitées avec la staurosporine. Les traces individuelles représentent parcelles temps dans des cellules individuelles. La baisse est limitée à orientation vers les régions des cellules qui s'inscrivent auprès fluorescentes mitochondries (à gauche du panneau) et est absente des régions de fond reste de fluorescence (à droite du panneau).
Maintenant que nous avons déterminé que la baisse de ratio d'aspect correspond à la fragmentation mitochondriale, on peut induire l'apoptose dans ces cellules, mesure de la fragmentation en utilisant notre méthode de dispersion optique sans avoir à marquer les cellules, et d'étudier l'effet de différentes conditions génétiques et expérimentales sur ce dynamique.
Discussion
La méthode décrite ci-dessus cartes rendements morphométrique de l'objet qui peut coder la taille des particules ou l'orientation par exemple. Cette information structurale peut être utilisé de plusieurs façons:
- Il peut être utilisé comme un écran initial pour identifier les régions de tissus ou de cellules qui sont modifiées au cours d'un traitement spécifique et ensuite d'analyser plus avant ces régions avec des dosages moléculaires et biochimiques.
- Il peut être ut...
Déclarations de divulgation
Remerciements
Le dispositif de micro-miroirs dans cette recherche a été financée par subvention de la Fondation Whitaker RG-02-0682 à N. Boustany. Les travaux en cours est financé par la NSF accorde-DBI-0852857 à N. Boustany. Pasternack RM a été partiellement soutenue par une bourse de Rutgers présidentielle supérieures. Nous tenons également à remercier le Dr E. White pour les cellules iBMK utilisé dans nos études et le Dr Metaxas DN pour la discussion utile sur les stratégies de filtrage optique.
matériels
| Name | Company | Catalog Number | Comments |
| DMEM | Invitrogen | Low glucose DMEM | |
| Liebowitz L15 medium | Invitrogen | Without phenol red | |
| L-glutamine | Invitrogen | ||
| Mitotracker Green | Invitrogen | ||
| Bovine Brain Extract | Clonetics | ||
| Fetal Bovine Serum | Gemini Bio Products | ||
| Heparin | Sigma-Aldrich | ||
| Staurosporine | Sigma-Aldrich | ||
| Dymethylsulfoxide | Sigma-Aldrich | ||
| Inverted microscope | Carl Zeiss, Inc. | Axiovert 200M | |
| DMD | Texas Instruments | TI 0.7 XGA DMD 1100 | |
| CCD | Roper Scientific | Cascase 512B | High (16 bit) dynamic range CCD |
| CCD | Roper Scientific | Coolsnap cf |
Références
- Pasternack, R. M., Qian, Z., Zheng, J. -. Y., Metaxas, D. N., White, E., Boustany, N. N. Measurement of Subcellular Texture by Optical Gabor-Like Filtering with a Digitial Micromirror Device. Optics Letters. 33 (19), 2209-2211 (2008).
- Pasternack, R. M., Qian, Z., Zheng, J. -. Y., Metaxas, D. N., Boustany, N. N. Highly sensitive size discrimination of submicron objects using optical Fourier filtering based on two-dimensional Gabor filters. Optics Express. 17 (14), 12001-12012 (2009).
- Zheng, J. -. Y., Pasternack, R. M., Boustany, N. N. Optical scatter imaging with a digital micromirror device. Optics Express. 17 (22), 20401-20414 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon