Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
L'ADN des isotopes stables de palpage (ADN-SIP)
Dans cet article
Résumé
L'ADN des isotopes stables de sondage est une méthode de culture-indépendante pour identifier et caractériser des communautés actives de microorganismes qui sont capables d'utiliser des substrats spécifiques. L'assimilation de substrat enrichi en isotope lourd conduit à l'incorporation des atomes marqués dans la biomasse microbienne. Ultracentrifugation en gradient de densité récupère l'ADN marqué en aval des analyses moléculaires.
Résumé
L'ADN des isotopes stables de sondage (DNA-SIP) est une technique puissante pour identifier les microorganismes actifs qui assimilent substrats carbonés particulier et des nutriments dans la biomasse cellulaire. Comme telle, cette technique de culture-indépendante a été une méthode importante pour assigner la fonction métabolique avec les diverses communautés vivant dans un large éventail de milieux terrestres et aquatiques. Suite à l'incubation d'un échantillon de l'environnement avec des isotopes stables des composés marqués, extraits d'acides nucléiques est soumis à une ultracentrifugation en gradient de densité et de fractionnement du gradient subséquente de séparer les acides nucléiques de densités différentes. Purification de l'ADN à partir de chlorure de césium récupère étiquetés et non étiquetés d'ADN pour la caractérisation moléculaire ultérieure (par exemple les empreintes digitales, les puces, les bibliothèques clone, métagénomique). Ce protocole fournit la vidéo JoVE visuelle étape par étape, des explications sur le protocole d'ultracentrifugation en gradient de densité de fractionnement du gradient, et la récupération d'ADN marqué. Le protocole comprend également des échantillons de données SIP et met en évidence d'importants conseils et avertissements qui doivent être considérées pour assurer un succès DNA-SIP analyse.
Protocole
1. Préparation des réactifs
DNA-SIP nécessite l'utilisation de réactifs qui doivent être préparés à l'avance de la procédure actuelle. Les orientations pour la préparation de chaque réactif sont répertoriés dans cette section et sont modifiées à partir d'un précédent protocole SIP 1.
- Chlorure de césium (CsCl), solution pour la préparation des gradients SIP - Préparer une solution de 7,163 M CsCl par dissoudre progressivement 603,0 g de CsCl dans l'eau distillée et désionisée (FD 2 O) pour un volume final de 500 ml. Attention à ne pas dépasser 500 ml! Le réchauffement de la solution légèrement en remuant aidera à dissoudre toutes les CsCl. Aliquoter la solution finale en aliquots scellés. Dans notre laboratoire, une pratique commune de stockage est de préparer 100 ml aliquotes de 125 ml fioles de sérum, qui sont ensuite sertir scellés avec des bouchons en caoutchouc butyle. Les aliquotes scellés peuvent être conservés indéfiniment à température ambiante (20 ° C). Les joints aider à prévenir l'évaporation et CsCl "croûte" de formation. Déterminer la densité de la solution en pesant triple de 100 uL aliquotes, ou en utilisant un réfractomètre numérique (par exemple Reichert AR200) qui a été soigneusement calibrée pour des solutions de CsCl. Une fois calibré avec succès, l'AR200 Reichert est cohérente et fournit des lectures précises pour plusieurs années. A température ambiante (20 ° C), la densité finale de cette solution varie généralement de 1.88 à 1.89 g -1 ml. La densité varie légèrement à chaque fois un nouveau stock est préparé.
- Solution de chlorure de césium pour la préparation de gradients de bromure d'éthidium (BET) - Combiner 250 g de CsCl avec 250 ml de solution stérile ddH 2 O eau. Aliquoter cette solution dans séparés fioles de sérum qui ont été scellés avec sertissage des joints en caoutchouc butyle comme décrit en 1.1.
- Tampon Dégradé - Mélanger 50 ml de 1 M Tris-HCl, 3,75 g de KCl et 1 ml d'EDTA 0,5 M à 400 ml d'eau. Dissoudre le KCl, puis ajouter ddH 2 O à 500 ml. Filtre-stériliser et autoclave. La solution finale est de 0,1 M de Tris, 0,1 M de KCl et 1 mM d'EDTA.
- Polyéthylène glycol (PEG) solution - Préparer la solution de PEG en dissolvant 150 g de polyéthylène glycol 6000 et 46,8 g de NaCl dans l'eau stérile ddH 2 O pour un volume total de 500 mL (PEG 30%, 1,6 M de NaCl). Autoclave.
Note: Cette solution se sépare en deux phases avec autoclave. Inclure un barreau dans la bouteille autoclave afin que la solution ne peut être correctement mélangés lorsque cela se produit. - Tampon TE - Préparer une solution de 10 mM Tris-HCl (pH 8,0) et EDTA 1 mM (pH 8,0) dans les stériles ddH 2 O eau, en utilisant des solutions mères autoclave de 1 EDTA M Tris-HCl (pH 8,0) et 0,5 M ( pH 8,0). Stériliser par filtration et autoclave.
- L'éthanol à 70% - Combinez 350 ml d'éthanol de haute pureté avec 150 ml d'eau stérile ddH 2 O.
2. L'incubation des échantillons et d'extraction d'ADN
Pour l'ADN-SIP incubations, les échantillons sont généralement incubées avec lourd des isotopes du carbone (13 C) du substrat. La période d'incubation et les conditions (par exemple, la supplémentation en nutriments, l'humidité, la lumière) varie selon le type d'échantillon qui est incubé et la nature du substrat. DNA-SIP expériences ont été réalisées avec succès en utilisant une variété de composés de carbone monoparois 2,3, multi-composés de carbone 4,5,6, et en utilisant l'azote ou l'oxygène étiquetés 7,8 9. Cependant, un inconvénient à l'utilisation de 15 N-ou O-18 composés marqués est la séparation physique de diminution d'acide nucléique marquée, principalement en raison de la présence de moins d'azote et des atomes d'oxygène dans l'ADN et par rapport aux atomes de carbone ARN.
Un contrôle critiques pour l'ADN-SIP expériences est une incubation identiques établis avec natif (par exemple 12 C) du substrat. Cette incubation permet une comparaison ultérieure afin de s'assurer que tout étiquetage apparente de l'acide nucléique ne fut pas un artefact de l'ultracentrifugation ou G + C Densité différences de contenu dans l'ADN contribue à la séparation 10. Il est également important de garder le matériel de l'échantillon congelé pour les comparer aux «légères» et «lourd» de l'ADN, et la valeur, y compris un contrôle sans substrat pour évaluer les changements de population de fond tout au long de l'incubation SIP.
- Incuber les échantillons environnementaux dans des microcosmes contenant substrat marqué (figure 1). Dans notre expérience, nous avons trouvé que l'incorporation minimale comprise entre 500 à 500 pmol de carbone 13 C par gramme d'échantillon sera adapté pour les échantillons contenant une biomasse élevée comme une des échantillons de sol. Pour les échantillons contenant moins de biomasse aquatique que les sols, 1-100 pmol de carbone C 13 incorporé par litre peut donner un détectables lourds-isotopiques signature 1. Le montant de l'amendement de carbone, la proportion de carbone incorporé dans la biomasse et l'exigence d'ajout d'éléments nutritifs supplémentaires pour l'assimilation tout dépendra des caractéristiques des échantillons analysés et la cible organisms d'intérêt. Un seul ensemble de directives d'incubation de l'échantillon ne sera pas applicable pour tous les échantillons. Fait important, la concentration du substrat utilisé pour l'incubation SIP devraient idéalement être aussi proche que possible de la concentration habituellement rencontrées in situ; biais expérimentaux peuvent être une conséquence des conditions de culture d'enrichissement 10.
- Après l'incubation de l'échantillon avec le substrat isotopes stables étiquetés, extraire l'ADN de microcosmes utilisant un protocole d'extraction rigoureux (pour la PCR ou clonage petit insert) ou une lyse enzymatique de confiance pour le clonage de haut poids moléculaire (par exemple les grands-insert métagénomique). ARN co-extraction n'affecte généralement pas l'analyse, de sorte que le rendement en ARN protocoles ainsi que l'ADN peut être utilisé. L'ultracentrifugation de l'ADN extrait ne sera pas plus courte que les fragments de cisaillement ~ paires ko 50 1.
- Quantifier l'ADN extrait avant l'installation des tubes de CsCl ultracentrifugation en gradient. Quantifier l'ADN en utilisant un spectrophotomètre (par exemple Nanodrop 2000) si le protocole d'extraction des rendements que l'ADN (par exemple colonne de base de kits). Alternativement, de quantifier en utilisant électrophorèse sur gel.
3. Préparation des solutions de gradient pour Ultracentrifugation
Cette procédure implique l'ajout d'ADN pour ultracentrifugeuse tubes. Il ya plus d'un type de tube et le rotor de sorte que le protocole exact variera et dépendra des instructions du fabricant. Cela dit, nous recommandons l'utilisation d'un rotor vertical et pour assurer le maximum possible séparation de l'ADN légers et lourds. Nous utilisons un Beckman-Coulter Vti 65.2 rotor avec 16 puits pour la tenue de 5,1 ml tubes polyallomère Quickseal et le protocole fournira les étapes et les considérations relatives à ces conditions.
- En utilisant les concentrations d'ADN déterminée dans l'étape 2.3, calculer le volume nécessaire de l'ADN extrait qui est nécessaire pour fournir 0,5 g - 5 ug d'ADN dans les tubes d'ultracentrifugation.
- Combinez l'ADN extrait (0,5 - 5 mg) avec le tampon de dégradé (voir étape 1.3) et 4,8 ml de 7,163 M CsCl à un volume total de ~ 6 ml dans un stérile jetable de 15 ml tube. Notez que la densité de la solution de CsCl peut varier, même à la molarité mêmes (voir étape 1.1). L'équation suivante peut être utilisée pour déterminer le volume de gradient de tampon / ADN mélange qui est requis pour générer un rapport de mélange approprié:
Volume de solution tampon et l'ADN dégradé (ml) = (densité de CsCl solution stock - densité finale souhaitée) x volume de la solution stock de CsCl ajouté x 1,52
Indiquez le volume de la solution stock de CsCl à 4,80 ml. La densité finale désirée devrait être 1,725 g ml -1. La densité de la solution d'actions a été déterminé à l'étape 1.1.
Notez également que les volumes relatifs de CsCl et gradient de tampon / ADN se traduira par un volume combiné de plus de 5,1 ml. Préparation des volumes supérieurs à la capacité du volume maximal des tubes d'ultracentrifugation (supérieur à 5,1 ml) fera en sorte qu'il y ait assez de solution pour remplir complètement le tube. - Mélanger en inversant 10 fois. L'ADN est stable à température ambiante dans le CsCl.
4. Créant un gradient de contrôle EtBr (facultatif)
Parce EtBr est un colorant intercalant que les complexes avec l'ADN de le rendre visible sous lumière UV, les gradients de contrôle contenant EtBr sont utiles car ils fournissent une confirmation visuelle immédiate de la formation de gradient avant le fractionnement des tubes de prélèvement (par exemple figure 1). L'inclusion d'un tube témoin contenant EtBr et un mélange des deux C-12 de l'ADN et l'ADN-C 13 (ou 14 N-DNA et 15 N-ADN) permet une visualisation immédiate de la formation de bandes dans les tubes à la fin de l'ultracentrifugation. Ceci est important car un tube perforé lors ultracentrifugation ou conditions de fonctionnement mal programmées peuvent entraîner la formation de gradient échoué. Liés à l'ADN, EtBr diminue la densité de l'ADN et, par conséquent, un protocole différent est suivie pour préparer les gradients. Notez que d'autres taches d'acides nucléiques peuvent être utilisés au lieu des 11 EtBr mais le protocole exigera l'optimisation avec des fluorophores autres.
- Le gradient de contrôle nécessite deux volumes de l'ADN génomique: un étiquetage complet avec des isotopes stables et un sans étiquette. Nous utilisons généralement soit Sinorhizobium meliloti cultivées dans un milieu contenant 13 C ou 12 C-glucose comme seule source de carbone, ou Bain Methylococcus souche capsulatus cultivée en présence de 13 C ou 12 C-méthane comme nos contrôles.
- Combinez une quantité de 5 à 10 mg à la fois du 12 C-DNA et 13 C-ADN avec tampon de gradient pour un volume final de 1,00 ml dans un jetables de 15 ml à bouchon à vis tube.
- Ajouter 1,00 g de CsCl solide pour le même tube. Mélanger par inversion.
- Ajouter 110 uld'une solution de 10 mg ml -1 EtBr et 4,3 ml d'une solution de 1 g ml -1 stock de CsCl à la même bouchon à vis tube utilisé dans l'étape 4.2. La densité finale de la solution sera approximativement celle de la solution stock de CsCl originale.
- Une solution supplémentaire "vide" de contrôle contenant EtBr sera également nécessaire pour contrebalancer la solution créée à l'étape 4.4. Combinez 1,00 ml de tampon de Dégradé, 1,00 g de CsCl, 110 ul d'une solution de 10 mg ml -1 EtBr et 4,3 ml d'une solution de 1 g ml -1 stock de CsCl dans une séparée de 15 ml à bouchon à vis tube et mélanger par retournement.
5. Ultracentrifugation
- En utilisant une ampoule et une pipette Pasteur, remplir soigneusement les tubes d'ultracentrifugation avec des solutions de gradient préparé à l'étape 3.2 (ou 4.4 si des mesures préparant un gradient de contrôle EtBr). Ajouter avec précaution les solutions pour les tubes à l'aide d'une pipette Pasteur. Etiqueter les tubes sur l'épaule du tube avec un marqueur permanent fines. ATTENTION: S'assurer que les tubes sont remplis exactement à la base du col du tube. Tubes insuffisamment rempli sont susceptibles d'éclater lors de l'ultracentrifugation.
- Quand tous les tubes nécessaires sont remplis avec des solutions de l'échantillon, enregistrer la masse précise de chaque tube. Paire tubes et les équilibre à l'intérieur 0-10 mg. Pour l'équilibrage, trouver des paires presque égalé et ajouter ou supprimer des quantités infimes de solution jusqu'à ce qu'elles soient équilibrées, en gardant le niveau de la solution au plus près de la base du cou du tube que possible. Notez que pour le pesage des tubes, nous utilisons une inversion de 15 ml à bouchon à vis du tube qui a été coupé en deux comme un porte-tube pour le solde.
- Sceau des tubes en utilisant un «tube topper" conformément aux instructions du fabricant.
- Vérifiez que les tubes sont correctement scellés en les inversant et en appliquant une pression modérée. Peser les tubes à nouveau pour vérifier qu'ils sont toujours équilibrés après scellement au sein de 0-10 mg.
- Vérifiez chaque rotor ainsi soigneusement pour s'assurer que les puits sont propres et exemptes de débris ou de poussières qui pourraient perforer les tubes lors de l'ultracentrifugation.
- Insérer les tubes dans le rotor avec les paires équilibrées face de l'autre. Notez l'emplacement du rotor de chaque échantillon, car le processus d'ultracentrifugation peut causer des étiquettes de marqueur d'être endommagées ou effacées. Bien sceller les puits de rotor comme indiqué par le fabricant.
- Chargez le rotor dans le ultracentrifugeuse. Fermez la porte d'ultracentrifugation et appliquer le vide. Si vous utilisez un Vti 65.2 rotor, régler la vitesse de rotation à 44 100 rpm (~ 177 000 xg AV), la température à 20 ° C le temps, et ultracentrifugation pendant 36 à 40 heures. Sélectionnez le vide, une accélération maximale, et éteignez le frein (gradient assure pas perturbée par la décélération). Notez que la désactivation du frein va ajouter un 1-2 heures supplémentaires pour le temps d'exécution. Notez également que temps d'exécution plus courts ne peuvent pas obtenir une résolution de bande suffisante. Fonctionne ultracentrifugation longs sont recommandés, car ils conduisent à une plus grande résolution des bandes distinctes d'acides nucléiques.
- Immédiatement après l'achèvement de la procédure d'ultracentrifugation, retirer le rotor attentivement. Éviter toute inclinaison ou de heurter du rotor, retirer délicatement les tubes du rotor pour éviter de perturber les gradients dans les tubes. Dans de rares circonstances, un tube va éclater pendant la course. Si oui, il ya une chance que les gradients dans les autres tubes ne se forme pas correctement. Si un gradient de contrôle a été inclus, vérifiez attentivement ce tube sous lumière UV afin de confirmer la formation de gradient. Si la pente n'est pas formé correctement dans le tube de contrôle, il est préférable de répéter tous l'étape 5. Notez que le tube de contrôle EtBr et son contrôle vierge peuvent être stockées dans l'obscurité et réutilisés pour un maximum de six mois. Prenez soin de nettoyer le rotor soigneusement selon les instructions du fabricant une fois le tube rafale a été supprimé. Ne pas utiliser de brosses métalliques ou de nettoyants abrasifs pour nettoyer les puits de rotor afin d'éviter les rayures sur les puits du rotor! Rotor spécifique brosses et de solution de nettoyage peuvent être achetés auprès de Beckman.
6. Fractionnement Dégradé
Il existe deux méthodes qui sont actuellement utilisés pour récupérer l'ADN à partir des tubes d'ultracentrifugation: fractionnement et d'extraction de l'aiguille. Ce protocole sera seulement décrire le processus d'extraction de l'ADN en utilisant la technique de fractionnement. C'est parce que pour des expériences de la plupart des SIP, l'ADN marqué ne peut être visualisée avec EtBr et doit plutôt être détectée en comparant la lumière équivalente et les fractions lourdes de tubes d'échantillons multiples. Une pompe à seringue est fortement recommandé de récupérer les fractions égales gradient de densité à partir des tubes d'ultracentrifugation. Nous utilisons une pompe BSP perfusion modèle (Braintree Scientific Inc). Une pompe à faible débit péristaltique ou une pompe HPLC peuvent également être utilisés.
- Remplir une solution stérile de 60 ml stérile seringue avec ddH 2 O contenant suffisamment de colorant bleu de bromophénol à fournir une couleur bleu foncé. Placez la seringue dans le bras de chargement de la pompe à seringue.Fixez tube de pompe équipée d'un calibre 23 1 "aiguille et tourner sur la pompe jusqu'à ce que certains ddH 2 O est venu jusqu'à la fin de la Note d'aiguille. Que toutes les bulles d'air dans cette offre ddH 2 O affectera négativement le processus de fractionnement.
- Fixer l'un des tubes d'ultracentrifugation d'un stand de serrage. Assurez-vous que la pince est suffisamment serré pour empêcher le tube de se déplacer, mais pas telle que la pression sur le tube causerait un communiqué de la solution de CsCl lorsque le tube est percé. Percez le fond même du tube le long du joint du tube en utilisant un nouveau calibre 23 1 "aiguille. Pour les meilleurs résultats, percer le tube de manière contrôlée, rapide et confiant. Cela est très difficile de bien faire, la pratique plusieurs fois avant cela est d'abord tenté avec des tubes d'échantillons.
- Pour chaque échantillon, préparer 12 stérile de 1,5 ml microtubes avec des étiquettes indiquant le numéro de l'échantillon et la fraction (1-12; lourdes à la lumière). Utilisation de l'aiguille fixée à la tubulure de la pompe (étape 6.1), percer le haut du tube sur l'épaule du tube supérieur, le long de la couture. Recueillir la solution de gradient utilisant les microtubes. Comme réalisée pour le fond du tube, percer le tube de manière rapide et contrôlé. Pratique à l'avance et être très prudent d'utiliser un mouvement contrôlé de traction pour éviter que l'aiguille contraints de passer par le tube et dans le petit doigt! Utiliser un taux de pompe préalablement étalonné qui donnera 12 x 425 ul fractions en 12 minutes (425 minutes ul -1).
- Utilisez un réfractomètre numérique (par exemple Reichert AR200; recommandé) ou une balance analytique pour vérifier la densité des fractions d'un gradient de confirmer la formation de gradient adéquat. Vous aurez besoin d'utiliser ~ 50 ul d'échantillon pour cet essai. Nous incluons souvent l'ADN culture pure dans un tube (comme décrit pour la préparation des gradients de contrôle EtBr) pour servir de contrôle pour le fractionnement et l'utiliser pour déterminer la densité. Attendez-vous à la densité varie de 1,690 à 1,760 ~ -1 g ml, avec une densité médiane de ~ 1,725 g -1 ml.
7. L'ADN des précipitations
- Précipiter l'ADN de toutes les fractions en ajoutant d'abord 20 pg de polyacrylamide linéaire comme support pour les précipitations. Mélanger par inversion. Ajouter 2 volumes de solution de PEG (voir étape 1) et mélanger par retournement. Notez que d'un transporteur pour les précipitations (p.ex. glycogène ou de polyacrylamide linéaire) est critique pour la récupération quantitative de l'ADN de factions dégradé, mais la prudence doit être utilisé si le glycogène est utilisé comme support pour la précipitation de ce protocole. Les préparatifs du glycogène a été démontré d'être contaminés par des bactéries d'acides nucléiques et la contamination peut facilement confondre l'interprétation des fractions du gradient SIP 12.
- Laisser les tubes à température ambiante pendant 2 heures pour permettre à l'ADN de précipité. Si désiré, les tubes peuvent être laissé une nuit à température ambiante.
- Centrifuger à 13000 g pendant 30 minutes avec le dos des tubes vers l'extérieur pour une orientation cohérente du tube dans le rotor. Soigneusement aspirer et jeter le surnageant. Une pastille doit être visible, mais peut être très difficile de voir à ce stade. Travailler sous une source lumineuse (lampe de bureau par exemple) pour aider à la visualisation de la pastille.
- Laver le culot avec 500 ul d'éthanol à 70%. Centrifuger à 13000 g pendant 10 minutes. Soigneusement aspirer et jeter le surnageant. Le culot sera généralement plus visible de cette étape, mais va se dissocier de la paroi du tube plus facilement.
- Autoriser le culot sécher à température ambiante pendant 15 minutes.
- Suspendre chaque pastille dans 50 ul de tampon TE (voir étape 1.5). Exécuter 5 ul de chaque fraction sur un gel d'agarose selon les protocoles de laboratoire standard.
8. Caractérisation de fractions
La méthode utilisée pour caractériser les fractions de gradient pour évaluer le succès d'une incubation de SIP varie selon le laboratoire et la disponibilité des équipements. En utilisant une méthode d'empreintes ciblant le gène ARNr 16S est une approche commune et des méthodes telles que la restriction de longueur de fragments terminaux polymorphisme (T-RFLP) ou électrophorèse sur gel dénaturant de gradient (DGGE) sont appropriés (figure 1). En suivant le protocole décrit ci-dessus, attendez l'ADN de lumière pour être associés à des fractions 9-11 (~ 1,705 à 1,720 g ml -1) et les empreintes ADN lourd pour être associés au sein des fractions 5-8 (~ 1.720 à 1,735 g ml -1 ). Empreintes digitales uniques associés à des fractions 5-8 du isotopes stables échantillons incubés, mais pas avec les contrôles natifs substrat incubé fournit des preuves solides liant les organismes spécifiques avec le métabolisme du substrat marqué en particulier. Si insuffisante d'ADN marqués reste pour certaines applications (hybridation, métagénomique), l'amplification par déplacement multiple peut être utilisé pour produire de plus grandes quantités de 13 à 15, mais cela peut introduire des chimères dans l'ADN amplifié 14,16.
9. Résultats
ADN typique SIP résultats montrent une séparation de l'ADN marqué et non marqué dans le gradient formé par ultracentrifugation. Idéalement, une résolution complète de matériel de haut poids moléculaire génétique (par exemple 13 C, 15 N) à partir de matériaux non étiquetés seront atteints. La résolution peut être témoin visuel en observant la formation de bandes dans des tubes de contrôle EtBr. Les concentrations d'ADN génomique récupéré contenues dans les fractions de gradient individuels peuvent également être utilisés pour confirmer la formation de gradient adéquat.
Pour ce protocole, nous incluons des résultats représentatifs de l'ultracentrifugation en gradient réalisée à l'aide d'acides nucléiques à partir de deux cultures pures (figure 2). Le gradient fractionné inclus ici a été préparé en utilisant l'ADN génomique extrait de S. meliloti (ATCC 1021), et marqués au 13 C M. str capsulatus. Bath. Suite à la récupération d'ultracentrifugation, le fractionnement et l'ADN, étiquetés et non étiquetés ADN génomique séparées en fractions de gradient respectives avec des densités différentes (figure 2A). Heavy-isotopes d'ADN marqués peuvent être observés dans les fractions 4-5, alors que l'ADN non étiquetés se trouve à des concentrations élevées dans les fractions 9-10. L'ADN de chaque fraction a été caractérisé par électrophorèse sur gel en gradient dénaturant 17 et les produits amplifiés par PCR générés discrète profils de bandes correspondant aux deux organismes inclus dans le gradient (figure 2B). La densité des fractions allant de ~ 1,580 à 1,759 g ml -1, et ils sont présentés dans un ordre décroissant de la densité de gauche à droite.
Bien que la séparation des 13 pur C et 12 C-ADN peut être prononcée (figure 2), les incubations des échantillons environnementaux peuvent être plus difficiles à interpréter. Par exemple, nous avons incubé la toundra sols de Resolute Bay (Nunavut, Canada) avec soit 12 C ou 13 C-étiquetés de glucose pour une période de 14 jours à 15 ° C. Les gels d'agarose de l'ADN gradient fraction purifiée a démontré que l'ADN génomique a été «barbouillé» à travers les fractions 7-10 pour deux C-12 et C 13-incubations (figure 3A et 3C, respectivement). Dans ce cas, 13 C-enrichissement de la biomasse microbienne du taxons particuliers ne peut être déterminée avec une approche comme celle-DGGE des gènes ARNr 16S. Le 12 C-glucose ADN de sol incubé génère des tendances similaires dans toutes les fractions du gradient (figure 3B), mais le 13 C-glucose de l'échantillon incubé généré empreintes DGGE qui sont typiquement associés à des fractions 5-8 (figure 3D). D'intérêt particulier sont les bandes conservées indiquées par les flèches. Cette dominante »phylotype est constante dans toutes les fractions du gradient, mais se déplace vers fractions plus lourdes pour l'ADN obtenu à partir de 13 C-glucose sols incubés. Séquençage génétique subséquente de cette bande et / ou d'un clone d'analyse bibliothèque serait de confirmer l'identité de ce gène particulier ARNr 16S et guide métagénomique ultérieures ou la culture des approches.
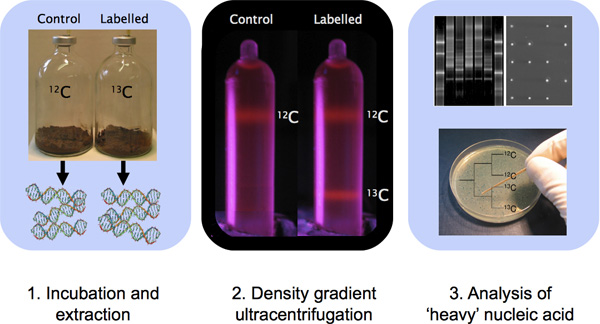
Figure 1. Esquisse d'une expérience de l'ADN-SIP comportant l'incubation de l'échantillon, l'extraction d'ADN, CsCl ultracentrifugation en gradient de densité et de caractérisation de l'ADN avec des techniques moléculaires.
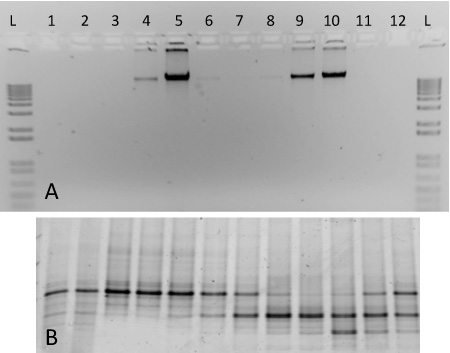
Figure 2. Résultats attendus pour un fractionnement du gradient SIP y compris l'ADN provenant de deux cultures pures. (A) Des aliquotes de l'ADN de 1 à 12 fractions du gradient ont été exécutés sur un gel d'agarose 1% à partir d'un gradient contenant marqués au 13 C M. Bain souche capsulatus (fractions 4-6) et 12 C-étiquetés S. meliloti (fractions 8-10). Une échelle de 1 kb est inclus pour comparaison (B) amplifiée par PCR ADN à partir des mêmes fractions ont été effectués sur un gel DGGE 10%. Modèles d'empreintes digitales révèlent des différences marquées entre les fractions 5 et 9, par exemple.
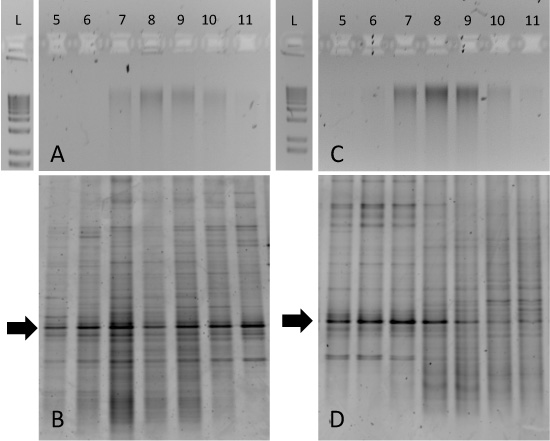
Figure 3. Résultats attendus pour les fractionnements gradient SIP de incubations d'échantillons de sol. Aliquotes des fractions de gradient des deux 12 C-glucose sol amendé (A) et 13 C-glucose sol amendé (C) ont été effectuées sur des gels d'agarose 1% et une échelle de 1 kb est inclus pour comparaison. DGGE empreintes correspondantes pour chacun de ces échantillons sont présentés dans (B) et (D). Empreintes des fractions révèle l'enrichissement des taxons bactériens particulier dans le 13 C-glucose de l'échantillon modifié en fractions 5-8 (D).
Discussion
Une bonne conception des isotopes stables expériences de sondage est d'une importance critique pour l'obtention de l'ADN marqué au-dessus de la communauté de fond non étiquetés. Considérations relatives à l'échantillon des durées d'incubation, les concentrations de substrat, les conditions d'incubation (par exemple, les nutriments, l'humidité du sol), cross-alimentation et de reproduction ont été discutées ailleurs 10,18 et nous recommandons au lecteur de consulter ces...
Déclarations de divulgation
Remerciements
Ce travail a été soutenu par le projet stratégique et subventions à la découverte de JDN en sciences naturelles et en génie du Canada (CRSNG).
matériels
| Name | Company | Catalog Number | Comments | |
| Bromophenol Blue | Reagent | Fisher Scientific | BP115-25 | |
| Cesium chloride | Reagent | Fisher Scientific | BP210-500 | |
| Ethanol, reagent grade | Reagent | Sigma-Aldrich | 652261 | |
| Ethidium bromide | Reagent | Sigma-Aldrich | E1510 | |
| Hydrochloric acid | Reagent | Fisher Scientific | 351285212 | |
| Linear polyacrylamide | Reagent | Applichem | A6587 | |
| Polyethylene Glycol 6000 | Reagent | VWR international | CAPX1286L-4 | |
| Potassium Chloride | Reagent | Fisher Scientific | AC42409-0010 | |
| Sodium Chloride | Reagent | Fisher Scientific | S2711 | |
| Sodium Hydroxide pellets | Reagent | Fisher Scientific | S3181 | |
| Tris base | Reagent | Fisher Scientific | BP1521 | |
| Dark Reader | Equipment | Clare Chemical | DR46B | |
| Microcentrifuge | Equipment | Eppendorf | 5424 000.410 | |
| Nanodrop 2000 | Equipment | Fisher Scientific | 361013650 | |
| Infusion pump | Equipment | Braintree Scientific, Inc. | N/A | Model Number: BSP See www.braintreesci.com for ordering details. |
| Tube sealer | Equipment | Beckman Coulter Inc. | 358312 | |
| Ultracentrifuge | Equipment | Beckman Coulter Inc. | ||
| Ultracentrifuge rotor | Equipment | Beckman Coulter Inc. | 362754 | |
| Ultraviolet light source | Equipment | UVP Inc. | 95-0017-09 | Any UV source will suffice |
| Ultraviolet light face shield | Equipment | Fisher Scientific | 114051C | |
| Butyl rubber stoppers, gray | Material | Sigma-Aldrich | 27232 | |
| Centrifuge tubes | Material | Beckman Coulter Inc. | 342412 | |
| Hypodermic needle, 23 gauge, 2” length | Material | BD Biosciences | 305145 | |
| Microfuge tubes, 1.5 mL | Material | DiaMed | AD151-N500 | |
| Open center seals, 20 mm diameter | Material | Sigma-Aldrich | 27230-U | |
| Pasteur pipettes, glass | Material | Fisher Scientific | 13-678-6C | |
| Pipet tips | Material | DiaMed | BPS340-1000 | Catalogue number is for 200 μl tips. 10 or 20 μl tips may be purchased from the same source |
| Pump tubing 1.5 mm bore x 1.5 mm wall | Material | Appleton Woods | ||
| Screw-cap tubes, 15 mL | Material | DiaMed | AD15MLP-S | |
| Serum vials, 125 mL volume | Material | Sigma-Aldrich | Z114014 | |
| Syringe, 60 mL | Material | BD Biosciences | 309653 |
Références
- Neufeld, J. D. DNA stable-isotope probing. Nat. Protocols. 2, 860-866 (2007).
- Neufeld, J. D., Boden, R., Moussard, H., Schäfer, H., Murrell, J. C. Substrate-specific clades of active marine methylotrophs associated with a phytoplankton bloom in a temperate coastal environment. Appl. Environ. Microbiol. 74, 7321-7328 (2009).
- Nercessian, O., Noyes, E., Kalyuzhnaya, M. G., Lidstrom, M. E., Chistoserdova, L. Bacterial populations active in metabolism of C1 compounds in the sediment of Lake Washington, a freshwater lake. Appl. Environ. Microbiol. 71, 6885-6899 (2005).
- Padmanabhan, P. Respiration of 13C-labelled substrates added to soil in the field and subsequent 16S rRNA gene analysis of 13C-labelled soil DNA. Appl. Environ. Microbiol. 69, 1614-1622 (2003).
- Bernard, L. Dynamics and identification of soil microbial populations actively assimilating carbon from 13C-labelled wheat residue as estimated by DNA- and RNA-SIP techniques. Environ. Microbiol. 9, 752-764 (2007).
- Haichar, e. l. Z. a. h. a. r., F, . Identification of cellulolytic bacteria in soil by stable isotope probing. Environ. Microbiol. 9, 625-634 (2007).
- Addison, S., McDonald, I., Lloyd-Jones, G. Stable isotope probing: Technical considerations when resolving 15N-labelled RNA in gradients. J. Microbiol. Meth. 80, 70-75 (2009).
- Buckley, D. H., Huangyutitham, V., Hsu, S. -. F., Nelson, T. A. Stable isotope probing with 15N achieved by disentangling the effects of genome G + C content and isotope enrichment on DNA density. Appl. Environ. Microbiol. 73, 3189-3195 (2007).
- Schwartz, E. Characterization of growing microorganisms in soil by stable isotope probing with H218O. Appl. Environ. Microbiol. 73, 2541-2546 (2007).
- Neufeld, J. D., Dumont, M. G., Vohra, J., Murrell, J. C. Methodological considerations for the use of stable isotope probing in microbial ecology. Microb. Ecol. 53, 435-442 (2007).
- Martineau, C., Whyte, L., Greer, C. Development of a SYBR safe technique for the sensitive detection of DNA in cesium chloride density gradients for stable isotope probing assays. J. Microbiol. Meth. 73, 199-202 (2008).
- Bartram, A. K., Poon, C., Neufeld, J. D. Nucleic acid contamination of glycogen used in nucleic acid precipitation and assessment of linear polyacrylamide as an alternative co-precipitant. Biotechniques. 47, 1019-1022 (2009).
- Chen, Y. Revealing the uncultivated majority: combining DNA stable-isotope probing, multiple displacement amplification and metagenomic analyses of uncultivated Methylocystis in acidic peatlands. Environ. Microbiol. 10, 2609-2622 (2008).
- Neufeld, J. D., Chen, Y., Dumont, M. G., Murrell, J. C. Marine methylotrophs revealed by stable-isotope probing, multiple displacement amplification and metagenomics. Environ. Microbiol. 10, 1526-1535 (2008).
- Kalyuzhnaya, M. High-resolution metagenomics targets specific functional types in complex microbial communities. Nat. Biotechnol. 26, 1029-1034 (2008).
- Binga, E. K., Lasken, R. S., Neufeld, J. D. Something from (almost) nothing: the impact of multiple displacement amplification on microbial ecology. ISME J. 2, 233-241 (2008).
- Green, S. J., Leigh, M. B., Neufeld, J. D., Timmis, K. N. . Microbiology of Hydrocarbon and Lipid Microbiology. , 4137-4158 (2010).
- Neufeld, J. D., Wagner, M., Murrell, J. C. Who eats what, where and when? Isotope-labelling experiments are coming of age. ISME J. 1, 103-110 (2007).
- Gallagher, E., McGuinness, L., Phelps, C., Young, L. Y., Kerkhof, L. J. DNA shortens the incubation time needed to detect benzoate-utilizing denitrifying bacteria by stable-isotope probing. Appl. Environ. Microbiol. 71, 5192-5196 .
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon