Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse de la fonction des petites GTPases par micro-injection de plasmides dans les cellules épithéliales polarisées
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Cet article détaille les procédures impliquées dans la surexpression et l'analyse des petites GTPases dans les cellules épithéliales polarisées en utilisant la technique de microinjection.
Résumé
Les cellules épithéliales polarisent leur membrane plasmatique dans biochimiquement et fonctionnellement distinctes domaines apical et basolatéral où le domaine apical face à la «libre» des surfaces et la membrane basolatérale est en contact avec le substrat et les cellules voisines. Les deux domaines membranaires sont séparés par des jonctions serrées, qui forment une barrière de diffusion. Apicale-basolatérale polarisation peut être récapitulé avec succès dans la culture lorsque les cellules épithéliales rénales canines telles que Madin-Darby (MDCK) sont ensemencées à haute densité sur des filtres de polycarbonate et cultivées pendant plusieurs jours 1 2. Création et maintien de la polarité cellulaire est régulée par un ensemble de petites GTPases de la superfamille Ras, comme RalA, Cdc42, Rab8, Rab10 et Rab13 3 4 5 6 7. Comme tous ces protéines GTPases cycle entre une inactifs PIB lié état et un actif lié au GTP Etat. Des mutations spécifiques dans les régions de fixation du nucléotide interférer avec ce vélo 8. Par exemple, Rab13T22N est définitivement verrouillée dans le PIB-forme et donc surnommé «dominant négatif», alors que Rab13Q67L ne peut plus hydrolyser le GTP et est donc bloqué dans un état «dominante active» 7. Afin d'analyser leur fonction dans les cellules à la fois dominante et dominante négative allèles active de GTPases sont habituellement exprimés à des niveaux élevés d'interférer avec la fonction des protéines endogènes 9. Une manière élégante d'atteindre des niveaux élevés de la surexpression dans un court laps de temps est d'introduire les plasmides codant pour les protéines pertinentes directement dans les noyaux des cellules polarisées cultivées sur filtre prend en charge en utilisant la technique de microinjection. Ceci est souvent combiné avec la co-injection de plasmides qui codent les récepteurs journaliste de la membrane plasmique qui sont spécifiquement triés sur le domaine apical ou basolatéral. Une cargaison fréquemment utilisés pour analyser les cargaisons de tri pour le domaine basolatéral est un allèle thermosensible de la glycoprotéine du virus de stomatite vésiculaire (VSVGts045) 10. Cette protéine ne peut pas se replier correctement à 39 ° C et sera donc retenue dans le réticulum endoplasmique (RE), alors que la protéine de régulation d'intérêt est assemblé dans le cytosol. Le passage à 31 ° C permettra alors de plier VSVGts045 correctement, laisser la salle d'urgence et de voyage à la membrane plasmique 11. Cette chasse est généralement effectuée en présence de cycloheximide pour empêcher la synthèse des protéines supplémentaires menant à l'assainissement de résultats. Ici nous décrivons en détail la procédure de micro-injection des plasmides dans les cellules polarisées et incubations ultérieures, y compris les changements de température qui permettent une analyse complète des protéines de régulation impliqués dans le tri basolatérale.
Protocole
1. L'isolement de l'ADN plasmidique
- Utilisez une endotoxine de Sigma-Aldrich kit maxiprep pour préparer l'ADN endotoxines libres selon le protocole du fabricant. Ce kit fonctionne pour nous, car elle élimine de manière fiable toute endotoxines des préparations d'ADN. Endotoxines qui sont injectés avec de l'ADN dans les noyaux des cellules entraîne la mort cellulaire.
- Ajouter 100 ul de phénol / chlorofom / alcool isoamylique (25:24:1) à l'ADN isolé, vortex et spin pendant 1 min à 13000 rpm dans un microcentrifugeuse Eppendorf. Transférer la phase aqueuse supérieure dans un nouveau tube et ajouter 100 ul de chloroforme / alcool isoamylique (24:1), vortex et spin comme ci-dessus. Transférer la phase aqueuse supérieure contenant l'ADN à un nouveau tube. Cette étape est nécessaire pour éliminer toute protéine de l'ADN, ce qui empêche le colmatage de l'aiguille de microinjection.
- Précipiter l'ADN par addition d'acétate de Na (pH 6,0) à une concentration finale de 300 mm et 2 X vol d'éthanol 100%. Incuber à -20 ° C la nuit. Spin ADN pendant 20 min à 13000 rpm dans un microcentrifugeuse Eppendorf, se laver une fois avec 70% d'éthanol, et remettre en suspension dans 300 ul sans endotoxine de l'eau (Sigma-Aldrich).
- Déterminer la concentration d'ADN. Une concentration de l'ADN typique varie de 1 à 5 ug / ul.
2. Culture de cellules MDCK
- Fractionner les cellules MDCK. Compter les cellules et les graines de 4 X 10 5 cellules sur des filtres clairs Transwell de 12 mm (0,4 um taille des pores, Corning Costar, 3460). Pour une expérience de contrôle contenant une injection maquette et deux différentes protéines Rab mutant dont vous avez besoin pour ensemencer trois filtres.
- Cellules MDCK culture dans un milieu MEM avec 2 mM de L-glutamine, 0,1 mg / ml de pénicilline / streptomycine et 7% (vol / vol) de sérum bovin foetal (= milieux de croissance MEM) à 37 ° C et CO 2 5%.
- Changement des médias dans la chambre basolatérale chaque jour. Ceci facilite le processus de polarisation, parce qu'elle imite la situation des cellules épithéliales chez l'animal.
- 2 jours après l'ensemencement vérifier dans un microscope si les cellules se multiplient en monocouche fermée. Si vous ne pouvez pas détecter tous les trous dans la monocouche, effectuez votre expérience au jour 3. S'il ya encore des trous, effectuer votre expérience sur 4 jours.
3. Procédure microinjection et post-injection incubations
- Le jour de l'expérience de préparer 5 ml de milieu MEM de croissance et 50 mM d'HEPES dans un plateau 60 x 15 mm pour chaque filtre et le placer dans un incubateur à 39 ° C. En outre, mis en place un puits d'une plaque 12 puits avec les médias 1 ml de croissance MEM et 50 mM d'HEPES et 0,1 mg / ml de cycloheximide par filtre, et le placer dans un incubateur 31 ° C.
- Allumez votre microscope microinjection et mis son stade chauffé à 39 ° C. Nous utilisons un microscope inversé (Axiovert 200, Carl Zeiss, Inc) avec une scène chauffée, 10X et 32X objectifs, et une FemtoJet Eppendorf (InjectMan NI2). Enfin, ouvrez le réservoir d'azote qui alimente la table de l'air à l'azote.
- Diluer l'ADN avec de l'eau filtrée (utiliser filtre de 0,2 um) à une concentration finale de 0,2 mg / ml dans un volume total de 10 - 100 pi. Par la suite, le spin de l'ADN dans microcentrifugeuse Eppendorf à 13000 rpm pendant 30 min. Retirer la partie supérieure et le placer dans un nouveau tube. Cette étape assure que lorsque vous chargez votre ADN dilué dans l'aiguille de microinjection vous pouvez exactement la charge "propre" ADN. Votre ADN est maintenant prêt pour la microinjection.
- Préparez vos cellules en prenant la sortie de son premier filtre boîte de culture. Avec une lame chirurgicale (Lame Plume chirurgical, acier inoxydable, n ° 11), découper le filtre du porte-filtre et le placer dans 5 ml, 39 ° C au chaud milieux de croissance MEM et 50 mM HEPES (60 x 15 mm Plaque). Courbez le filtre dans la plaque de culture avec la lame chirurgicale que vous avez utilisé pour le découper. Placez-le sur le filtre tel que le trou de la lame chirurgicale entoure le milieu du filtre. Placez votre plaque de culture sur la scène chauffée du microscope.
- Charge 2-3 ul de votre ADN dilué (dans le cas des plasmides cet encodage VSVGts045-GFP = contrôle d'injection maquette) dans une aiguille de microinjection (Femtotip II, Eppendorf, 930 000 043), tournez le couvercle de protection de l'aiguille et le laisser tomber sur le plancher . Maintenant que l'aiguille est prête à être vissée dans son support. Pour ce faire, appuyez sur la touche menu de votre InjectMan et s'assurer que le robinet est fermé. Vissez l'aiguille dans son support, méfiez-vous de ne pas visser l'aiguille trop serré car cela pourrait conduire à la rupture. Maintenant, appuyez sur la touche menu, l'application P c sera empêcher les médias d'être aspirée dans l'aiguille lors de la procédure microinjeciton. Enfin, appuyez sur le joystick pour effacer homing stockées.
- Pour abaisser l'aiguille sur les cellules, utilisez l'objectif 10X et amener l'aiguille dans le faisceau de lumière au-dessus du liquide. Maintenant se concentrer sur les cellules, mettre l'accent à nouveau en tournant la molette de mise en place de 180 °, et de trouver l'aiguille. Par la suite, se déplacer lentement l'aiguille dans le foyer, puis sortir de concentrer à nouveau (travailler à amener les cellules en se concentrer à nouveau), ramener l'aiguille dans desUC à nouveau. Répétez jusqu'à ce que l'aiguille touche la surface du support, à quel point tout ce que vous allez voir est un halo. Lorsque vous atteignez un point où les cellules sont en discussion, mais l'aiguille est encore floue (c'est à dire hors du plan focal) modification de l'objectif 32X et fine des paramètres grossiers.
- Régler la limite de z-en touchant la membrane apicale avec la pointe de l'aiguille et en soustrayant environ 10 um que les noyaux sont la pose d'environ 10 um sous la membrane apicale.
- Après avoir défini le z-limite, trouver la pression d'injection de droite. Départ à 95 PSI. Si la pression est trop élevé, vos cellules vont exploser. Si votre tension est trop basse, vous verrez un point blanc qui reste, mais rien ne se passe. Avec une injection réussie, vous verrez un changement de phase sans changer la taille des cellules. Pour l'injection, amener l'aiguille de quelques microns de la plan focal aussi vite que possible. Visez avec l'aiguille au-dessus du noyau (ie au milieu d'une cellule individuelle) et appuyez sur le bouton d'injection de votre joystick, mais ne pas maintenir le bouton enfoncé.
- Injecter 100 à 500 cellules dans le trou de votre lame chirurgicale qui se trouve sur vos cellules. Lorsque vous avez terminé, placez vos cellules avec la boîte de culture et d'une lame chirurgicale dans un incubateur à 39 ° C, et incuber pendant 2 h. Pendant ce temps, VSVGts045-GFP est exprimée et sécrétée dans le RE. Cependant, à 39 ° C, VSVGts045-GFP ne peut pas se replier correctement et donc ne peuvent pas quitter la salle d'urgence. En attendant, la petite GTPase d'intérêt va s'accumuler dans le cytosol des expériences de co-injection.
- Après 2 h, placez vos cellules dans 1 ml de croissance des médias MEM et 50 mM d'HEPES et 0,1 mg / ml de cycloheximide dans une plaque de 12 puits et incuber pendant 2 h à 31 ° C. Durant cette période, chasse, VSVGts045-GFP se replier correctement, laisser la salle d'urgence et est livré à la surface cellulaire.
- Répétez les étapes 03.01 à 03.10 pour les filtres à deux (injecter des plasmides codant pour la GFP et VSVGts045-par exemple-V5 taggés Rab13T22N) et trois (injecter des plasmides codant VSVGts045-GFP et par exemple V5-taggés Rab13Q67L). Chaque injection durera environ 20 - 60 min. Cependant, il est absolument crucial pour traiter tous les filtres de la même façon tout au long du incubations changement de température et une coloration de surface jusqu'à ce que vous fixer les cellules. Après fixation, vous pouvez effectuer le reste de votre protocole de coloration pour tous les filtres dans le même temps.
4. Coloration de surface pour analyse par immunofluorescence
Remarque, afin d'éviter le blanchiment du signal GFP de VSVGts045-GFP, protéger les spécimens de la lumière en couvrant avec du papier d'aluminium pendant toutes les procédures ultérieures.
- Si vous souhaitez effectuer une coloration de surface, placez vos cellules dans une boîte de culture sur une plaque de métal sur la glace et lave-toi les cellules 1X avec PBS glacé 2 + (PBS [0,2 g / litre de KCl, 0,2 g / litre de KH 2 PO 4, 8 g / litre de NaCl et 2,17 g / litre de Na 2 HPO 4 x 7 H 2 O] et 0,1 g / litre de CaCl 2 et 0,1 g / litre de MgCl 2 x 6 H 2 O). Par la suite, placer 30 pl d'un anticorps qui reconnaît l'ectodomaine de vos protéines, dans ce cas, l'anticorps monoclonal de souris TK1 (IgG 1, obtenu à partir de la fin des années Kreis Thomas), sur du parafilm nettoyage placé sur la plaque de métal sur la glace. Placez le filtre avec vos cellules à l'envers sur la goutte et ajouter quelques gouttes d'anticorps sur la face arrière du filtre. Incuber pendant 1 h sur la glace.
- Cellules de lieu dans une plaque de 12 puits, laver 3x avec PBS glacé 2 + (ou RT chaude PBS 2 + sans coloration de surface avant), et fixer avec du paraformaldéhyde 3% pendant 15 min à température ambiante.
- Laver les cellules une fois avec du PBS 2 + et de laisser en PBS 2 + pendant 5 min.
- Incuber les cellules dans un tampon bloquant perméabilisation / (BPB) (2% [poids / vol] BSA, 0,4% [p / v] saponine dans le PBS 2 +) avec 10% [vol / vol] du sérum de chèvre. Incuber 1 h à température ambiante.
- Diluer anticorps primaires pour détecter la GTPase Rab exprimé, dans cet exemple anti-V5 (monoclonal de souris d'anticorps IgG 2a, Invitrogen), 1:200 dans BPB. Spin anticorps dilué dans une microcentrifugeuse Eppendorf pendant 10 min à 13000 rpm. Placer 30 ul de la solution d'anticorps sur du parafilm propres placés dans une chambre humide. Placez les cellules sur le filtre à l'envers sur la baisse des anticorps et incuber pendant 1 h à température ambiante.
- Remettre les cellules (côté droit) dans une plaque de 12 puits et laver 5 fois en 30 min avec BPB à la température ambiante.
- Diluer appropriée anticorps secondaire, dans ce cas, de chèvre anti-souris IgG 1 Alexa 594-étiquetées (pour VSVG détection sur la surface, Invitrogen), et Cy5-anticorps secondaires marqués à reconnaître vos GTPase Rab, dans cette chèvre par exemple IgG anti-souris 2a Cy5-étiquetées (Jackson ImmunoResearch), 1:200 dans BPB et de spin comme ci-dessus. Placer 30 ul de solution d'anticorps sur des parafilm propre dans une chambre humide et les cellules de lieu à l'envers du filtre vers le bas sur la baisse des anticorps. Incuber 1 h à température ambiante.
- Répétez l'étape 4.6.
- Tremper les cellules sur le filtre en 3X eau déminéralisée et le lieu rSide Up ight sur microslides. Ajouter 10-15 ul montage (10% [poids / vol] DABCO, 50% [poids / volume] du glycérol dans l'eau distillée) sur le dessus des cellules. Placez 18X18 mm couvercle en verre micro sur le dessus et en utilisant des tissus du visage appuyez doucement sur le couvercle en verre sur le micro-cellules. Sceller avec du vernis à ongles.
- Analyser les échantillons avec un microscope confocal. Nous avons utilisé un Microsystem LSM 510, Carl Zeiss, Inc qui a été équipé d'une lentille à immersion d'eau 63X.
- Pour la préparation des figures, ajuster et de combiner des images en utilisant des programmes comme Adobe Photoshop et Adobe Illustrator.
5. Les résultats représentatifs
Pour des exemples sur la façon dont la co-expression des petites GTPases interfère avec le tri VSVG veuillez vous reporter aux articles publiés soit pour missorting apicale 3, 4 ou l'inhibition de 7 de livraison de surface.
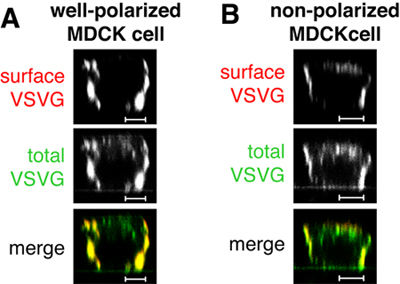
Figure 1. Dans l'injection maquette, c'est à dire une injection de seulement le plasmide codant pour la GFP-VSVGts045, la protéine est livré à la surface basolatérale de bien-polarisée des cellules MDCK en juger par la coloration de surface (en rouge, la figure 1 A). Notez que tous les VSVGts045-GFP est livré à la membrane basolatérale pendant la poursuite à 31 ° C comme en témoigne le signal intracellulaire vaste pour les protéines totales (en vert, les figures 1 A et B). Dans les cellules qui ne sont pas bien polarisée VSVGts045-GFP sera livré aussi à la membrane apicale (figure 1 B). Si vos échantillons de contrôle ressemble à la cellule de la figure 1 B, vous ne pouvez pas faire confiance à vos données et à répéter l'expérience avec une meilleure polarisation des cellules. Barres d'échelle sont de 5 um.
Discussion
Les étapes les plus critiques pour une expérience réussie de microinjection sont la qualité et la pureté de l'ADN et la polarité de vos cellules. Sans cellules polarisées, votre commande d'injection aura déjà mistargeted VSVG et l'expérience ne peut pas être utilisé. Si l'ADN est de mauvaise qualité, l'ADN peut obstruer l'aiguille d'injection entraînant peu ou pas d'expression de la protéine désirée du tout. Aussi, il est conseillé d'utiliser des plasmides d'expr...
Déclarations de divulgation
Remerciements
Ce travail a été financé par une subvention des National Institutes of Health (GM070736) à H. Fölsch. SF Ang a été soutenue par un prix A * STAR bourses d'études supérieures, et RS Kang a été soutenue par la base cellulaire et moléculaire du programme de formation des maladies (GM8061)
matériels
| Name | Company | Catalog Number | Comments |
| Nom de réactif | Société | Numéro de catalogue | |
| Axiovert 200 Microscope à platine chauffante | Carl Zeiss Inc | Afin personnalisé | |
| InjectMan NI2 FemtoJet micromanipulateur | Eppendorf | Afin personnalisé | |
| Femtotips II (aiguilles microinjection) | Eppendorf | 930000043 | |
| Microloader astuces | Eppendorf | 930001007 | |
| Clair 12 mm transwell filtre prend en charge | Corning Costar | 3460 | |
| Endotoxines-libres Bluetooth plasmide maxiprep | Sigma-Aldrich | NA0400 |
Références
- Mellman, I., Nelson, W. J. . Nature reviews. 9 (11), 833-833 (2008).
- Rodriguez-Boulan, E., Kreitzer, G., Musch, A. . Nature reviews. 6 (3), 233-233 (2005).
- Ang, A. L., Fölsch, H., Koivisto, U. M. . The Journal of cell biology. 163 (2), 339-339 (2003).
- Kroschewski, R., Hall, A., Mellman, I. . Nature cell biology. 1 (1), 8-8 (1999).
- Moskalenko, S., Henry, D. O., Rosse, C. . Nature cell biology. 4 (1), 66-66 (2002).
- Schuck, S., Gerl, M. J., Ang, A. . Traffic (Copenhagen, Denmark). 8 (1), 47-47 (2007).
- Nokes, R. L., Fields, I. C., Collins, R. N. . The Journal of cell biology. 182 (5), 845-845 (2008).
- Collins, R. N. Molecular cell. 12, 1064-1064 (2003).
- Hall, A. . Science (New York, N.Y. 279 (5350), 509-509 (1998).
- Scales, S. J., Pepperkok, R., Kreis, T. E. . Cell. 90 (6), 1137-1137 (1997).
- Keller, P., Toomre, D., Diaz, E. . Nature cell biology. 3 (2), 140-140 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon