Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Photobleaching essais (FRAP et FLIP) pour mesurer la dynamique des protéines de la chromatine dans Vivre cellules souches embryonnaires
Dans cet article
Résumé
Nous décrivons les méthodes de récupération, y compris photoblanchiment de fluorescence après photoblanchiment (FRAP) et la perte de fluorescence En Photobleaching (FLIP) pour suivre la dynamique des protéines dans la chromatine souches embryonnaires (ES) cellules. La dynamique des protéines de la chromatine, ce qui est considéré comme l'un des moyens d'étudier la plasticité de la chromatine, est augmentée dans les cellules pluripotentes.
Résumé
Récupération de fluorescence après photoblanchiment (FRAP) et la perte de fluorescence En Photobleaching (FLIP) permettra l'étude de la dynamique des protéines dans les cellules vivantes avec une résolution spatiale et temporelle bien. Nous décrivons ici comment effectuer FRAP et FLIP dosages des protéines de la chromatine, notamment H1 et HP1, chez la souris souches embryonnaires (ES) cellules. Dans une expérience de FRAP, les cellules sont transfectées, soit transitoire ou stable, avec une protéine d'intérêt fusionnée à la protéine fluorescente verte (GFP) ou leurs dérivés (YFP, CFP, cerise, etc.) Dans le transfectées, les cellules fluorescentes, un faisceau laser intense concentré blanchit une région relativement petite d'intérêt (ROI). La longueur d'onde laser est choisie en fonction de la protéine fluorescente utilisée pour la fusion. La lumière du laser blanchit de manière irréversible le signal de fluorescence des molécules dans le retour sur investissement et, immédiatement après le blanchiment, la récupération du signal fluorescent dans la zone blanchie - médiatisé par le remplacement des molécules blanchie avec les molécules non blanchie - est contrôlée au moyen d'imagerie laps de temps. Les courbes générées récupération de fluorescence fournir des informations sur la mobilité de la protéine. Si les molécules fluorescentes sont immobiles, aucune récupération de fluorescence est observée. Dans une approche complémentaire, la perte de fluorescence dans le Photobleaching (FLIP), le faisceau laser blanchit au même endroit à plusieurs reprises et l'intensité du signal est mesuré ailleurs dans la cellule fluorescente. Expériences FLIP donc mesurer la carie signal plutôt que la récupération de fluorescence et sont utiles pour déterminer la mobilité des protéines ainsi que la protéine navette entre compartiments cellulaires. Contraignante transitoire est une propriété commune de la chromatine protéines associées. Bien que la fraction majeure de chaque protéine chromatine est lié à la chromatine à tout moment à l'état stationnaire, la liaison est transitoire et la plupart des protéines de chromatine ont un chiffre d'affaires élevé sur la chromatine, avec un temps de séjour dans l'ordre des secondes. Ces propriétés sont cruciales pour générer une grande plasticité dans l'expression du génome 1. Expériences de photoblanchiment sont donc particulièrement utiles pour déterminer la plasticité chromatinienne utilisant la GFP-fusion des versions de la chromatine des protéines structurelles, en particulier dans les cellules ES, où l'échange dynamique de protéines de chromatine (y compris les protéines hétérochromatine 1 (HP1), un linker histone H1 et histones) est plus élevé que dans les cellules différenciées 2,3.
Protocole
1. Placage des cellules ES
T = 0 h
Plaquage MEF
- Manteau de l'imagerie direct 8-même μ-Slides (ibidi; Munich, Allemagne) avec la gélatine ou dans des verres à couvercle chambrés (Lab-Tek; Rochester, NY) ou en boîtes de culture à fond de verre (MatTek; Ashland, MA). Départ pour 5-30 min et aspirer l'écart de la gélatine sans.
- Graine 22000 FAE / puits dans 250 ul volume total de DMEM [complété avec 10% sérum de veau fœtal (FBS)]. Permettent aux cellules de se développer dans un incubateur de culture tissulaire (37 ° C, 5% de CO2).
T = 6 heures
Placage de cellules ES
- Aspirer DMEM.
- Graine de chaque MEF revêtement bien avec 15 000 cellules R1 / puits dans 250 ul du volume total des médias de cellules ES [complété avec 10% ESC-grade du sérum de veau fœtal (FBS), pyruvate de sodium 1 mM, 0,1 mM acides aminés non essentiels, 0,1 mM β- mercaptoéthanol, et 1000 la leucémie facteur U / ml inhibitrice (FRV)], pour obtenir 30% à 50% de confluence le jour suivant.
2. Transfection des cellules ES
T = 24 h.
Transfection transitoire
- Remplacez le support de cellules ES avec 250 ul / puits de Fresh Media cellules ES.
- Dans un tube de 1,5 ml de test stérile, ajouter 100 ul de sérum médias libres [Opti-MEM (Gibco)], puis ajouter 10-Transit LT1 ul réactif de transfection (Mirus) directement dans les médias sans sérum. Mélanger doucement par pipetage et incuber à température ambiante pendant 5-20 min.
- Ajouter 1,5 mg ADN de fusion GFP plasmide (H1E, H1o ou HP1) à l'diluée transit LT1 réactif. Mélanger à la pipette doux et incuber à température ambiante pendant 15-30 min.
- Ajouter 13,5 l / puits du mélange de transfection. Agiter le 8-même μ-Slides pour assurer une dispersion. Après 24 heures de remplacer les anciens médias de cellules ES avec 250 pi de Fresh Media cellules ES.
3. Exécution FRAP et FLIP
T = 48-72 heures
- L'expérience peut être effectuée sur un microscope confocal à balayage laser (CLSM) mais depuis une normale FRAP / FLIP expérience, de nombreuses images consécutives sont acquises, il est recommandé d'utiliser un microscope confocal à disque rotatif, qui permet une vitesse d'acquisition et assure que personne ne l'échantillon indésirable blanchiment aura lieu après l'événement initial de blanchiment intentionnel. Ici, nous vous recommandons d'utiliser la Révolution filer disque système confocal (www.Andor.com), avec le Yokogawa CSU-X tête du disque en rotation. Ce système a la capacité dual photobleach utilisant un module spécialisé Frappa avec un système de points de numérisation et de rapidement passer la lumière en arrière pour recueillir des images en utilisant le disque en rotation. Les trois protéines fluorescentes les plus couramment utilisés pour les expériences de photoblanchiment sont GFP, YFP et Cherry. Si la GFP ou YFP sont utilisés, un laser ~ 488 nm est nécessaire. Pour Cherry, l'utilisation d'un laser ~ 560 nm. Dans tous les cas, nous recommandons d'utiliser des lasers à semi-conducteurs. Ayant une étape automatisée est utile mais pas nécessaire. Depuis les cellules vivantes sont imagés, il est indispensable d'utiliser une chambre environnementale (nous utilisons un de LIS, Suisse), l'oxygène de contrôle, l'humidité, le CO 2 et la température. FRAP est réalisée en utilisant au maximum l'intensité du laser tout en imagerie se fait avec la puissance du laser minimale requise (généralement dans le domaine de 10%, lorsque le niveau de fluorescence est suffisant).
Observez les cellules avec une lumière fluorescente de longueur d'onde appropriée et sélectionnez une cellule exprimant la GFP à l'aide d'une lentille d'huile d'immersion 60X. Assurer une bonne distribution subcellulaire. Parfois, lorsque les niveaux d'expression sont trop élevés, la localisation de la protéine peut «déversement» pour les autres compartiments tels que le nucléole. Ces cellules ne doivent pas être sélectionné. - Maintenant établir un protocole d'imagerie: collecter 3-5 cadres avant le photoblanchiment, puis photobleach au euchromatine ou de l'hétérochromatine (considérés comme des foyers condensée GFP) et recueillir 90 à 120 images après l'photobleach, avec des intervalles ms 250-1000: H1E-GFP, 1000 ms, H1o et HP1-GFP, 250 ms (intervalle de temps des changements en fonction de la dynamique des protéines, où les protéines hautement dynamiques nécessitent du temps plus court intervalle). Nous utilisons normalement l'intensité du laser 80-100% pour les photoblanchiment avec une impulsion laser de 20 à 40 μseconds (1-2 itérations), mais ces chiffres peuvent changer en fonction de la protéine analysée et les niveaux d'expression. Lorsque photoblanchiment est approprié, vous devriez observer un «trou noir» dans votre fluorescence de la GFP. Le trou noir est progressivement re-rempli avec récupération après fluorescence. Bien que le disque tourne peut obtenir jusqu'à près de 60 images par seconde (avec une bonne intensité de la fluorescence et quand en zoomant sur une seule cellule), nous ne recommandons pas d'utiliser le système à de telles vitesses élevées en raison de la qualité d'image à faible et le potentiel de phototoxicité augmenté.
- Pour une expérience FLIP, mis en place un protocole d'imagerie différentes: collecter 3-5 cadres avant le blanchiment, puis lancez répétées de blanchiment, au même endroit tout en collectant des images. Pour H1E-GFP, eau de javel toutes les 5 sec, pour H1o-GFP, eau de javel toutes les 2 sec, et pour HP1-GFP javel toutes les 1 sec. Bleach uned recueillir des images à plusieurs reprises tout au long de l'expérience entière.
- Pour ces deux techniques, répétez le processus sur 20-30 cellules. À des fins statistiques, répéter l'expérience 3 fois ou plus, de préférence à des jours différents. Dans les populations homogènes et paramètres corrects, la déviation standard est généralement faible (<5%).
Pour les deux FRAP et FLIP, la taille et la forme de la région blanchi influe sur la dynamique de récupération et doit rester constante au sein d'une expérience. Aussi, lorsque deux cellules sont comparés, les protocoles identiques doivent être utilisés et les cellules doivent être analysés séquentiellement sur la même journée que la puissance laser et d'autres conditions peuvent fluctuer et peuvent affecter le résultat de l'expérience.
4. FRAP et FLIP Data Analysis
- Dans tous les cadres du PAF recueillies, de mesurer l'intensité de fluorescence dans le ROI (retour sur investissement b = surface blanchie), la zone de fond (ROI BG), et la zone non-blanchie nucléaire (ROI NB) en fonction du temps avant et après le blanchiment. Quand la région est négligeable blanchi le noyau tout entier peut être sélectionné à des fins de normalisation.
- Pour chaque point du temps, de normaliser les données selon la formule: (ROI b - ROI bg) / (nb ROI - retour sur investissement bg) / (b pbROI - pbROI bg) / (nb pbROI - pbROI bg), PB indique pré-blanchi. Pour pré-blanchiment des images, vous devriez obtenir une valeur d'environ 1. La première image après la javel va indiquer la profondeur Bleach. Soustraire la valeur de 1 pour la valeur réelle profondeur Bleach. Répétez l'opération pour chaque cellule et en moyenne 20 à 30 cellules de chaque expérience.
- Dans tous les cadres FLIP recueillies, de mesurer l'intensité de fluorescence dans le domaine non-blanchie nucléaire, et la zone de fond (ROI nb = non blanchi région, ROI bg = fond). Calcul des données FLIP est semblable à une courbe de FRAP, seul le ROI analysé (ROI nb) doit être différent de la région réelle blanchis, ce qui n'est pas utilisé pour le calcul: (ROI NB - ROI bg) / (nb pbROI - pbROI bg) . Il est également possible d'utiliser une cellule voisine (cellule n ROI voisin =) à des fins de normalisation: (ROI NB - ROI bg) / (n ROI - retour sur investissement bg) / (nb pbROI - pbROI bg) / (n pbROI - pbROI bg ).
Après la collecte des données, il est possible d'ajuster les données expérimentales à la simulation informatique. Ceci permet le calcul, avec une proximité bien, la fraction mobile, la fraction immobile et la demi-maximale. Nous n'allons pas discuter des aspects mathématiques et statistiques d'analyses FRAP ici et renvoyons le lecteur à d'autres excellentes publications 4-9. La profondeur de l'eau de Javel se réfère à la distance (sur l'axe des y) entre le pré-blanchiment (100%) du signal et la première image, après l'eau de Javel, la fraction mobile se réfère à la distance (sur l'axe des y) entre la profondeur et l'eau de Javel le signal récupéré lors de la cinétique atteint un plateau, et la fraction immobile se réfère à la distance (sur l'axe des y) entre le signal récupéré et le pré-blanchiment (100%) du signal (voir les figures 1B et 2B). Au-delà de cette analyse, il ya de bons modèles mathématiques pour ajuster les données. Pour un exposant unique, l'équation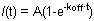
où t est le temps, A est la fraction mobile, 1-A est la fraction immobile et k est hors la constante de dissociation, peut être utilisé pour ajuster les données, et une estimation directe du taux hors de liaison (k off) peut être obtenus, ainsi que pour le paramètre A, qui peut être utilisé pour calculer le taux de l'association.
5. Les résultats représentatifs:
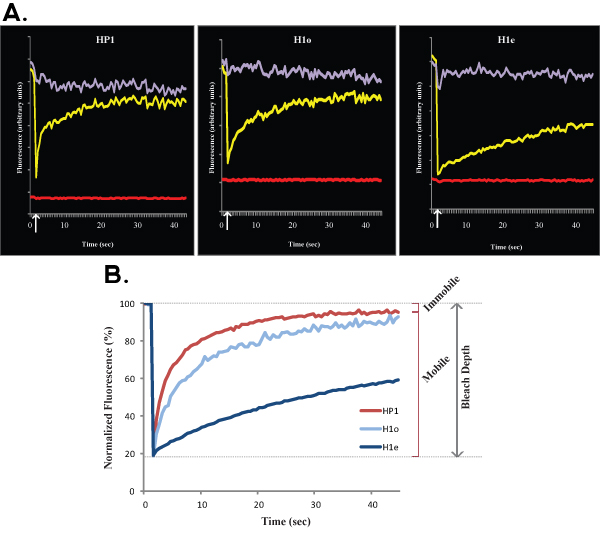
Figure 1. A et B montrent des courbes représentatives du FRAP HP1 (à gauche), H1o (milieu) et H1E (à droite) dans des cellules ES R1. Pour la figure simplicité et la clarté 1A montre les données brutes d'une seule cellule, avant toute normalisation et de calcul. La courbe jaune correspond à la région blanchis, la courbe mauve correspond à la zone non-blanchie nucléaire (lorsque la région est négligeable blanchi le noyau tout entier peut être sélectionné à des fins de normalisation), et la ligne rouge correspond à la fluorescence de fond, qui est minime dans ce cas. Flèche verticale représente le temps l'eau de Javel. Données normalisées et une moyenne est montré dans la figure 1B. Notez la reprise plus lente de H1 (bleu) par rapport à HP1 (rouge). Aussi la variante H1E (bleu foncé) est plus lente que la variante H1o (bleu clair). Fractions mobiles et immobiles et profondeur Bleach sont indiqués pour HP1.
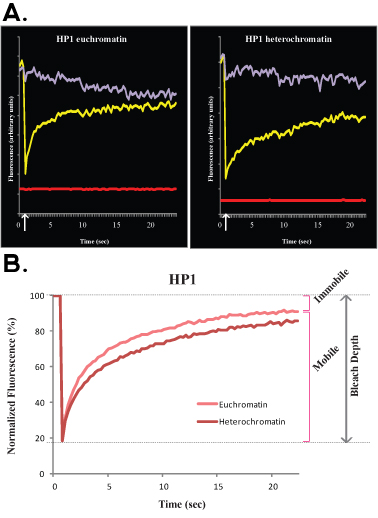
Figure 2. A et B montrent des courbes représentant FRAP comparant euchromatine (à gauche) avec l'hétérochromatine (à droite) de HP1 dans des cellules ES R1. De même à la figure 1, la figure 2A montre les données brutes d'une cellule unique, courbe jaune correspond à la région blanchis, courbe mauve correspond à la zone non-blanchie nucléaire, et la ligne rouge correspond à la fluorescence de fond. Flèche verticale représente le temps l'eau de Javel. Données normalisées et une moyenne est montré dans la figure 2B. Notez la reprise plus lente de l'hétérochromatine (rouge foncé) par rapport à l'euchromatine (lumière rouge). Fractions mobiles et immobiles et profondeur Bleach sont indiqués pour l'euchromatine.
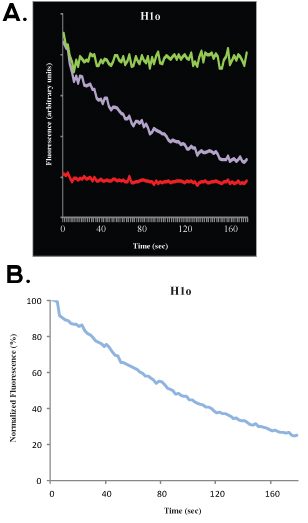
Figure 3.. Une expérience typique FLIP de cellules ES H1o R1 est montré dans la figure 3A (crus, non normalisées de données) et B (données normalisées et moyennées). Dans cette expérience, la courbe mauve correspond à la zone non-blanchie nucléaire, la ligne verte correspond à un noyau de la cellule voisine et la ligne rouge correspond à la fluorescence de fond.
Discussion
Contrairement à la plupart des techniques disponibles, qui impliquent la chromatine purifiée à partir de populations de cellules ou de cellules fixes, des expériences FRAP suivre l'évolution de la dynamique des protéines de la chromatine dans les cellules vivantes. Nous avons trouvé la dynamique des protéines de chromatine d'être un bon indicateur de la plasticité chromatinienne. Cependant, parce qu'il exige la fusion des gènes d'intérêt avec la GFP, l'ajout du marqueur fluorescent peut ...
Déclarations de divulgation
Remerciements
Nous remercions les membres du laboratoire Meshorer, surtout Shai Melcer, Adi Alajem, Raghu Ram Edupuganti, Badi Sri Sailaja, Anna Mattout et Alva Biran, pour les commentaires critiques et pour le dépannage des expériences de photoblanchiment sur une base quotidienne. EM est un H. Joseph et Belle R. Braun Senior Lecturer en Sciences de la Vie et est soutenu par la Fondation des sciences d'Israël (ISF 943/09), le ministère israélien de la Santé (6007) de l'Union européenne (IRG-206872 et 238176), l'Israel Cancer Research Foundation, l'interne Applicative Subventions de médecine de l'Université hébraïque et de la psychobiologie Institut israélien.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | |
|---|---|---|---|
| DMEM | Sigma | D5671 | |
| Gélatine | Merck | 1,04078 | |
| Opti-MEM | Gibco | 31985 | |
| Transit-LT1 | Mirus | MIR2300 | |
| 8-même μ-Slides | ibidi | 80826 |
Références
- Phair, R. D. Global nature of dynamic protein-chromatin interactions in vivo: three-dimensional genome scanning and dynamic interaction networks of chromatin proteins. Mol Cell Biol. 24, 6393-6402 (2004).
- Meshorer, E., Girard, L. . Imaging chromatin in embyonic stem cells in StemBook. , (2008).
- Meshorer, E. Hyperdynamic plasticity of chromatin proteins in pluripotent embryonic stem cells. Dev Cell. 10, 105-116 (2006).
- Bancaud, A., Huet, S., Rabut, G., Ellenberg, J. . Fluorescence perturbation techniques to study mobility and molecular dynamics of proteins in live cells: FRAP, photoactivation, photo conversion, and FLIP. , (2009).
- Dundr, M., Misteli, T. Measuring dynamics of nuclear proteins by photobleaching. Curr Protoc Cell Biol. Chapter 13, Unit 13-Unit 13 (2003).
- Ellenberg, J. Nuclear membrane dynamics and reassembly in living cells: targeting of an inner nuclear membrane protein in interphase and mitosis. J Cell Biol. 138, 1193-1206 (1997).
- Lenser, T., Weisshart, K., Ulbricht, T., Klement, K., Hemmerich, P. Fluorescence fluctuation microscopy to reveal 3D architecture and function in the cell nucleus. Methods Cell Biol. 98, 2-33 (2010).
- Mueller, F., Mazza, D., Stasevich, T. J., McNally, J. G. FRAP and kinetic modeling in the analysis of nuclear protein dynamics: what do we really know. Curr Opin Cell Biol. 22, 403-411 (2010).
- Phair, R. D., Misteli, T. Kinetic modelling approaches to in vivo imaging. Nat Rev Mol Cell Biol. 2, 898-907 (2001).
- Poser, I. BAC TransgeneOmics: a high-throughput method for exploration of protein function in mammals. Nat Methods. 5, 409-415 (2008).
- Sigal, A. Generation of a fluorescently labeled endogenous protein library in living human cells. Nat Protoc. 2, 1515-1527 (2007).
- Cohen, A. A. Dynamic proteomics of individual cancer cells in response to a drug. Science. 322, 1511-1516 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon