Method Article
Imagerie de cellules vivantes de la Et Streptococcus pneumoniae À l'aide automatisée de microscopie time-lapse
Dans cet article
Résumé
Ce protocole prévoit une procédure étape par étape pour surveiller le comportement seule cellule de bactéries différentes dans le temps en utilisant la fluorescence automatisée de microscopie time-lapse. En outre, nous fournir des lignes directrices comment analyser les images de microscopie.
Résumé
Durant les dernières années les scientifiques de plus en plus conscients que la moyenne des données obtenues par des expériences de la population microbienne base ne sont pas représentatifs du comportement, le statut ou le phénotype des cellules individuelles. Grâce à cette nouvelle perspective le nombre d'études seule cellule augmente continuellement (pour des revues récentes, voir 1,2,3). Cependant, la plupart des techniques cellule unique appliquée ne permettent pas de suivre l'évolution et le comportement d'une seule cellule spécifique dans le temps (par exemple, la cytométrie en flux ou la microscopie standard).
Ici, nous fournir une description détaillée d'une méthode de microscopie utilisée dans plusieurs études récentes 4, 5, 6, 7, ce qui permet de suivre et d'enregistrer (de fluorescence des) personne cellules bactériennes de Bacillus subtilis et Streptococcus pneumoniae par la croissance et la division pour de nombreuses générations. Les films qui en résulte peut être utilisé pour construire des arbres phylogénétiques lignée en remontant l'histoire d'une cellule unique dans une population qui provenaient d'un ancêtre commun. Cette méthode time-lapse de fluorescence microscopie peut être utilisé non seulement pour enquêter sur la croissance, la division et la différenciation des cellules individuelles, mais aussi d'analyser l'effet de l'histoire de la cellule et l'ascendance sur le comportement cellulaire spécifique. Par ailleurs, la microscopie time-lapse est idéalement adapté à examiner la dynamique de l'expression génique et la localisation des protéines au cours du cycle cellulaire de la bactérie. La méthode explique comment préparer les cellules bactériennes et de construire la lame du microscope pour permettre à l'excroissance de cellules individuelles dans une microcolonie. En bref, des cellules individuelles sont repérés sur une surface semi-solide composé d'un milieu de croissance complétées par d'agarose sur lequel ils croissent et se divisent sous un microscope à fluorescence dans un environnement contrôlé température de la chambre. Les images sont capturées à des intervalles spécifiques et sont ensuite analysés en utilisant le logiciel open source ImageJ.
Protocole
1. Préparation de la B. cultures subtilis
- Inoculer les cellules de -80 ° C stocks dans 10 ml de microscopie time-lapse (TLM) moyen (62 mM K 2 HPO 4, 44mm KH 2 PO 4, 15 mM (NH 4) 2 SO 4, 6,5 mM citrate de sodium, 0,8 mM MgSO 4, 0,02% casaminoacides, 27,8 mM de glucose, 0,1 mM de L-tryptophane, le pH a été fixé à 7 en utilisant une solution de KOH) complété avec des antibiotiques, si nécessaire.
- Cultiver les cellules la nuit dans un flacon d'agitation (30 ° C, 225 rpm).
- Le lendemain matin, diluer les cellules à 1:10 dans préchauffé milieu chimiquement défini (CDM) (62 mM K 2 HPO 4, 44mm KH 2 PO 4, 15 mM (NH 4) 2 SO 4, 6,5 mM citrate de sodium, 0,8 mM MgSO 4, 2.2mm de glucose, 2,1 d'acide L-glutamique mm, 6 uM de L-tryptophane, 7,5 uM MnCl2, 0,15 x combinaison de métal (préparer 50x stock de mélange de MT (ref 8) qui contient: 0,2 M de MgCl 2, 70 mM CaCl 2, 5 mM MnCI 2, 0,1 mM ZnCl 2, 0,2 mM thiamine chlorhydrate, 2 mM HCl, 0,5 mM FeCl 3 (ajouter en dernier lieu)), sans antibiotiques.
- Cultiver la B. cellules B. subtilis à phase mi-exponentielle (30 ° C, 225 rpm). Habituellement, cela prend environ quatre heures. Surtout, préparer la diapositive agarose une heure avant les cellules atteignent phase mi-exponentielle (voir section 2).
- Mesurer l'absorbance de la culture à 600nm (600 A) et diluer les cellules à une approximative de 0,035 A 600 à l'aide du MDP. Cette OD assure que des cellules isolées, avec un espacement appropriés sont repérés sur la lame du microscope pour la microscopie time-lapse.
2. Préparation de l'échantillon au microscope (voir aussi la figure 2)
Une heure avant la mi-cellules atteignent une croissance exponentielle, de préparer la lame du microscope comme suit:
- Nettoyer deux lames de verre de microscope (par exemple Knittel verre, 7,6 x 2,6 cm), avec 70% d'éthanol et d'eau.
- Prenez un châssis gène (ABgene; 1,7 x 2,8 cm) et retirez soigneusement l'un des films plastiques à partir du cadre de gènes sans causer le démontage de la couverture en plastique de l'autre côté du cadre de gènes.
- Fixer le cadre de gènes dans le milieu de l'une des lames de verre par contact facilitant d'abord sur un seul côté, suivie par l'attachement guidée de la trame de gènes restants avec un ongle. Empêcher les bulles d'air tout en attachant le cadre de gènes à la lame de verre.
- Utiliser un micro-ondes pour dissoudre 150 mg (1,5%) à haute résolution à faible point de fusion d'agarose (Sigma) dans 10 ml du MDP. L'agarose doit être entièrement dissous pour obtenir de fond minimal requis pour les expériences de microscopie time-lapse. Si nécessaire, compléter l'agarose-MDP avec un inducteur ou d'autres composés à cette époque
- Transférer 500 ul de l'échauffement d'agarose-MDP dans le milieu du cadre de gènes. Assurez-vous que toute la région, y compris (les frontières) est entièrement couvert.
Les étapes suivantes (02/06 à 02/10) doivent être effectués rapidement pour empêcher une déshydratation excessive de l'agarose-MDP. - Placez la lame de verre secondes sur le châssis d'agarose-MDP gène remplis. Essayez d'éviter les bulles d'air. Placer les lames en sandwich dans une position horizontale pendant 45 min à 4 ° C dans le réfrigérateur pour permettre à l'agarose-MDP de solidifier suffisamment.
- Soigneusement glisser la lame de verre supérieure. Utilisez une lame de rasoir pour découper des bandes de largeur agar ~ 5 mm dans le cadre de gènes, sur laquelle les cellules seront cultivées. Un maximum de trois bandes peut être utilisé par diapositive, séparés par un espace ~ 4 mm de chaque côté. Ces places offriront de l'air qui est essentiel pour B. la croissance subtilis. Si quatre souches différentes ont besoin d'être suivis dans le temps, deux bandes peuvent être faites et être réduite de moitié à raison de quatre petits carrés. Retirez tout support solide résiduel.
- Retirer délicatement le couvercle du second et dernier en plastique à partir du cadre de gènes pour exposer le côté collant du cadre de gènes
- Charger des cellules individuelles (de l'étape 1.5) sur le milieu solide, sans le toucher avec la pointe de la pipette. Utiliser 2,5 pi pour une bande de totalité, ou 1 pl pour une petite place. Il faut toujours commencer sur le dessus du pavé agarose et laisser le liquide à disperser également sur sa zone de croissance assignés en tournant le glisser vers le haut et le bas. La lame est prête, dès que les bords du liquide devient ondulé et le mouvement du liquide n'est plus visible lorsque vous tournez la diapositive.
- Placer un bordereau de microscope propre glisser le couvercle (24 x 50 mm) sur le cadre de gènes d'un côté à l'autre (éviter les bulles d'air). Assurer l'attachement complète en appliquant une pression sur la lamelle le long du cadre de gènes avec votre ongle. Si la lamelle est placée sur les cellules sans leur permettre de sécher suffisamment longtemps, les cellules ont tendance à croître sur le dessus de l'autre pendant l'expérience. Aussi soyez prudent de ne pas attendre trop longtemps avant d'appliquer la lamelle, depuis l'agarose sera alors trop sec.
- Pré-chauffer le glisser pendant 1 h à 30 ° C. Si la lame wOuld être directement placé dans la chambre de pré-chauffée de l'environnement (voir étape 3.1) du microscope, les fluctuations de température pourrait causer des problèmes autofocus dans les premières heures de l'expérience.
3. Time-lapse de microscopie par fluorescence (voir aussi la figure 3 et Movie 1)
- Préchauffer la chambre de l'environnement sur le temps (dans nos mains au moins 2h avant le début de l'expérience) afin de prévenir les problèmes de mise au point automatique après le démarrage de l'expérience. Le temps nécessaire dépend de la chambre de l'environnement utilisées ainsi que le système de chauffage et le microscope.
- Sélectionnez l'objectif approprié, les filtres et le miroir dichroïque en fonction de votre configuration expérimentale. Pour les expériences à long s'assurer qu'un filtre UV est placé entre la source lumineuse et l'échantillon. Aussi, si possible, de bloquer certains de la lumière d'excitation en utilisant des filtres de densité neutre pour minimiser l'exposition.
Les équipements suivants (fournis par DeltaVision, UK) a été utilisée pour les expériences de microscopie time-lapse publié par de Jong et al 2010 5:. IX71 Microscope (Olympus), Coolsnap HQ2 caméra (Princeton Instruments), Source 300W Xénon, 60x lumineux objectives sur le terrain (1,25 NA), la GFP filterset (Chroma, excitation à 470/40 nm, émission 525/50 nm), mCherry filterset (Chroma, excitation à 572/35 nm, émission 632/60 nm). Autofocus a été réalisée en utilisant la lumière diascopique et en utilisant le présent de routine autofocus dans le logiciel de DeltaVision Softworx. Il faut noter qu'il ya maintenant un certain nombre de systèmes autofocus d'autres qui sont également appropriés comme la Focus Zeiss définitive, le système de mise au point parfaite de Nikon et le contrôle de Leica Adaptive Focus.
- Programmez votre expérience en fonction de votre configuration expérimentale. Il est sage de déterminer la quantité de lumière nécessaire pour des constructions spécifiques, ainsi que les réglages autofocus pour d'autres time-lapse microscopes ou les bactéries avant l'expérience réelle. Temps d'exposition plus courts et moins de quantité de lumière d'excitation sera de minimiser le blanchiment et la phototoxicité. Utilisez la lumière diascopique pour la routine autofocus.
Les paramètres suivants ont été utilisés pour les expériences de microscopie time-lapse publié par de Jong et al 2010 5:. Instantanés pour les films ont été prélevés à intervalles de 8 ou 12 minutes en utilisant 10% APLLC LED blanche exposition à la lumière et 0,05 s pour des images en champ clair, 10% de lumière au xénon et 0,5 s pour la détection de l'exposition GFP, et 32% de lumière au xénon et 0,8 s pour la détection de l'exposition mCherry, respectivement. Les données brutes sont stockées à l'aide softWoRx 3.6.0 (Applied Presicion). L'autofocus a été programmé pour 0,06 um étapes et une gamme totale de 1,2 um.
- Placez la lame préparée (section 2) dans la chambre de pré-chauffé de l'environnement du microscope et de surveiller l'excroissance de cellules individuelles dans une monocouche microcolonie à 30 ° C.
Conseils spécifiques:
- Sélectionnez les cellules simples qui se trouvent au milieu du pad agar. Les bords de l'agar pad sèchent plus facilement. Magasin de la position X, Y, Z en utilisant le logiciel du microscope.
- Des mouvements importants de la scène dans le X, Y et Z pourrait perturber le agarose et par conséquent d'entraver l'identification de cellules par la routine autofocus. En général, afin de minimiser X, Y, Z le mouvement, nous ne sélectionnons pas plus de 10 postes par expérience, même si une diapositive contient des souches multiples.
- Après avoir sélectionné la première cellule, seulement ajuster le Z-focus avec le logiciel. De ce point, ne modifient pas la mise au point manuellement sur le corps du microscope à l'aide de la "Z-knob" sauf si cela est codée numériquement. Assurez-vous que, après chaque routine autofocus du nouveau X, Y, Z la position est stocké par le logiciel.
- Vérifiez si les réglages autofocus sont appropriés pour l'expérience, avant de commencer la course. L'utilisation de la microscopie à contraste de phase pourrait améliorer la routine autofocus comparé à l'utilisation ou la microscopie DIC fond clair, en raison de contraste amélioré. Cependant, l'anneau de phase dans les objectifs à contraste de phase rend moins sensibles (environ 10%) dans la collecte de lumière de fluorescence. Donc, pour la faiblesse des échantillons fluorescents, un objectif sans bague de phase est mieux adapté.
- Vérifiez si les cellules sélectionnées sont encore en discussion chaque demi-heure, jusqu'à ce que l'expérience fonctionne stablement. Lorsque les cellules sont à ce point hors de discussion, d'ajuster manuellement. En raison de variations de température, ainsi que des échantillons mal séchés, cela peut être nécessaire pendant les premières heures. Par ailleurs, en raison d'un meilleur contraste, l'autofocus fonctionne mieux lorsque plusieurs cellules sont dans le champ de vision.
- Après l'expérience est terminée, séparer les différents canaux du film et de sécurité entre eux comme des fichiers séparés (ie contraste de phase, la GFP, mCherry) si nécessaire (les paquets d'acquisition de certains va placer tous les canaux dans un seul fichier empilés). Pour la publication, les photos peuvent être améliorées par déconvolution 2D, qui est surtout nouseful pour les études de localisation de la protéine. Déconvoluer les images en utilisant le logiciel de votre microscope ou avec un logiciel commercial comme Huygens ( www.svi.nl ).
- Analyser les données en utilisant ImageJ ( http://rsbweb.nih.gov/ij/ ) (utiliser les premières, les fichiers image brute pour cela) et Microsoft Excel ou Sigma Plot. Stacks peut par exemple être enregistré en tant que ". Avi" fichier de film dans ImageJ. Une description détaillée de la façon dont la fluorescence des cellules individuelles peut être mesurée dans le temps est donnée ci-dessous.
4. L'analyse des données de la dynamique de l'activité du promoteur en utilisant ImageJ
Nous notons que d'autres logiciels sont disponibles bonnes qui sont spécialisés dans l'analyse des images de microscopie time-lapse tels que BHV logiciels 9, 4, 10 Schnitzcell, PSICIC 11, et Microbe-Tracker 12, mais ici nous nous concentrons sur le paquet disponible gratuitement ImageJ.
- Télécharger ImageJ ( http://rsbweb.nih.gov/ij/ ) et (si nécessaire) le plugin correcte pour ouvrir votre (empilés) fichier. Par exemple, les films enregistrés en utilisant un microscope DeltaVision ne peut être ouvert dans ImageJ avec le plugin ouvre DeltaVision. Copiez le DV-plugin dans le dossier plugin ImageJ et démarrer le programme. Changer la capacité de mémoire de modifier / option / mémoire et des threads pour 1250. Cela permet de travailler avec des fichiers plus gros tels que ceux obtenus à partir time-lapse films.
- Afin d'évaluer l'histoire de cellules d'une seule cellule, ouvrez le film phase initiale de contraste d'un microcolonie et faites défiler jusqu'à la dernière image de l'intérêt dans le film. Sélectionnez le bouton "segmenté lignes" dans le menu de sélection.
- Tracez une ligne en arrière-plan et appuyez sur "Ctrl" + "T". Cela va ouvrir les régions d'intérêt (ROI) gestionnaire. (La valeur correspondante de fond de fluorescence peut être utilisée manuellement soustraire le fond -.. Voir ci-dessous Sinon, utilisez le présent de routine soustraction de fond au sein de ImageJ) Aussi tracer une ligne dans la cellule d'intérêt et d'ajouter le retour sur investissement pour le gestionnaire de ROI. Puisque nous étudions promoteur-GFP fusions dans cette étude de cas et de la GFP est diffusée partout dans le cytoplasme, la cellule entière devrait avoir les mêmes valeurs de fluorescence pour chaque pixel sur la longueur de la cellule. Faites défiler une image dans le temps et sélectionnez une nouvelle ROI dans la même cellule d'intérêt. Sauvegarder cette ROI troisième et continuer avec la procédure jusqu'à ce que le retour sur investissement correspondant à la première image du film a été enregistré.
Soyez conscient que la fluorescence des cellules filles peuvent varier considérablement après la division cellulaire. Soit utiliser un colorant membrane qui peuvent être appliquées conjointement avec des protéines de fluorescence produite par les cellules (telles que la combinaison du rouge à membrane colorants FM ® 5-95 (Invitrogen) et GFP) pour visualiser la formation du septum. Dans ce cas, le canal correspondant à fluorescence et non le film à contraste de phase doit être utilisée pour suivre les cellules dans le temps. Alternativement, on peut rester sur le côté sécuritaire en choisissant le ROI en une seule moitié d'une cellule. - Dans le gestionnaire de ROI, cliquez sur "enregistrer". Si la fin du fichier est ". ROI", puis un retour sur investissement est sélectionné dans la liste, et seulement celui-ci sera sauvé. Si la fin du fichier est ". Zip", puis l'ensemble sera sauvé (obligatoire).
- Fermer le film à contraste de phase et d'ouvrir le film original de fluorescence (GFP, par exemple). Cliquez sur "Afficher tous" et "mesure" dans le gestionnaire de ROI. Une nouvelle fenêtre va s'ouvrir (Résultats). Copier les résultats dans une feuille Excel et soustraire la moyenne de fluorescence de chaque cellule unique à partir de la fluorescence de fond du milieu. La fluorescence résultante nette peut être comploté contre le temps de révéler l'activité du promoteur de la cellule d'intérêt dans le temps.
- Alternativement, la fluorescence de toutes les cellules d'une micro peut être analysée à un moment précis du temps-point. Pour ce faire, sélectionner et enregistrer un retour sur investissement pour le fond et chaque cellule unique dans un cadre tel que décrit ci-dessus, copiez les valeurs de fluorescence pour Excel et produire des histogrammes en utilisant le "histogramme" de fonction dans le menu "outil".
- Pour obtenir des photos à partir d'images unique pour la publication, sélectionnez le cadre de l'intérêt, sélectionnez "image" - "double image" et changer le type d'image sur "RGB" ou "8-bit" en sélectionnant "image" - "type" - "RVB" ou "8-bit". Sauvegardez l'image dupliquée comme «. Tiff». Les photos RGB/8-bit peut être ouvert par des programmes de dessin classiques, comme CorelDraw ou Adobe Illustrator. Si nécessaire, les images peuvent être adaptés en utilisant "l'image" - "ajuster" - "Luminosité / Contraste" ou "image" - "ajuster" - "window / level" dans ImageJ.
5. Produire des films pour publication avec ImageJ
- Ouvrez le contraste de phase originale et le film de fluorescence correspondant (s) dans ImageJ comme décrit ci-dessus. Sélectionnez le bouton rectangulaire sélections (1 er à gauche) et dessinez un rectangle de ROI dans la première image de telle façon, que le développement de microcolonies est entouré par le Throu ROIghout tout le film. Sélectionnez "Image" - "culture" et sûre de cette version plus petite du film sous un nouveau nom. Sélectionnez le ROI mêmes via le gestionnaire ROI dans le film de fluorescence et de procéder comme avant.
- Pour combiner les films soit horizontalement ou verticalement, sélectionnez "plugin" - "combinateur pile". Si désiré, une matrice de temps peuvent être ajoutées via "plugin" - "Stamper temps». Enregistrer la pile combinée, dès que ". Dv» ou «. Tiff" et une fois comme ". Avi" ou un film QuickTime.
Adaptations protocole alternatif pour Streptococcus pneumoniae (figure 4 et Film 2):
6. Préparation de S. cultures pneumoniae
- Cultivez S. cellules pneumoniae (souche capsulée D39 13, ou d'une souche non encapsulé R6 14 en tant que cultures debout milieu C + Y 15 à 37 ° C jusqu'à une DO à 600nm A d'environ 0,4 est atteint. Centrifuger les cellules pendant 2 min à 14000 rpm et remettre les culot cellulaire dans un volume de frais C + Y milieu contenant du glycérol 14,5% (v / v) qui se traduirait par une 600nm Une exactement 0,4. Aliquoter les cellules et de les stocker à -80 ° C pour une utilisation future.
- Pour la microscopie time-lapse, prélever une aliquote de préalablement cultivées S. cellules pneumoniae. Inoculer 4 ml de frais C + Y moyennes 1:100 avec des cellules de la partie aliquote de -80 ° C. Cultiver les cellules jusqu'à la mi-exponentielle phase à une DO de A 600nm de 0,1 - 0,2. Habituellement, cela prend environ 2 heures en utilisant des cellules de -80 ° C aliquotes.
7. Préparation de l'échantillon de microscope
- Préparer une lame de microscope comme décrit ci-dessus pour B. subtilis, mais assurez-vous que l'agarose contient complexe C + Y moyen. Depuis S. pneumoniae est une microaerophile, les poches d'air entre les bandes d'agarose doit être plus petite que pour B. subtilis (~ 1 mm d'espace sur les deux côtés).
- Mesurer l'absorbance de la culture à 600 nm (600 A), diluer le croissance exponentielle S. pneumoniae aux cellules une approximative A 600 de 0,05 en utilisant C + Y moyennes et de l'utilisation de cette dilution pour charger les diapositives d'agarose.
8. Time-lapse en contraste de phase de microscopie
Ajustez les paramètres de microscope pour S. pneumoniae: utiliser la microscopie à contraste de phase puisque S. pneumoniae est difficile à identifier à l'aide lumineuse microscopie à champ. Continuer le protocole décrit pour B. subtilis (suivre les étapes 2.9 à 3.7). S. pneumoniae cellules peuvent être cultivées soit à 30 ° C ou 37 ° C (ils poussent plus vite à 37 ° C).
9. Les résultats représentatifs:
L'expérience de fluorescence time-lapse a été effectuée avec succès, si les bactéries grandi dans une monocouche microcolonie, qui est complètement situé dans le champ de vue à la fin de l'expérience (voir figure 5A-C). Si les cellules a grandi sur le dessus de l'autre, il est non seulement impossible de retracer leur histoire avec précision, mais aussi les niveaux de fluorescence de cellules qui se chevauchent ne peut pas être mesurée correctement. Les cellules ont tendance à croître sur le dessus de l'autre, si les cellules n'ont pas été repérés séché suffisamment (étape 2.9) ou si la composition du milieu doit être ajustée pour obtenir une croissance plus lente. Si une micro grandi hors de la vue, puis la distribution des signaux de fluorescence au sein d'une colonie ne peut être déterminée. Les causes de «mouvement microcolonie" peut être un séchage insuffisant des cellules repéré (étape 2.9), ou si le logiciel n'a pas été programmé pour suivre la microcolonie cours du développement. Par ailleurs, il est important que des correctifs locaux de la fluorescence accrue ne sont pas détectable dans le milieu car cela obscurcit les signaux de fluorescence provenant des cellules (voir figure 5D-F). Problèmes de fond liés peuvent survenir à partir de composés médias, bulles d'air ou non dissous touffes d'agarose. Pour visualiser cela, nous montrons dans la Fig. Signaux de fond 5F de cette diapositive spécifique lorsque l'image a été prise à l'aide d'excitation / émission des filtres pour les colorants fluorescents rouges. Comme on le voit, taches lumineuses autofluorescente sont présents, qui pourraient entraver l'imagerie. Pour éviter ces taches, assurez-vous que l'agarose est complètement dissous et il n'ya pas de bulles d'air en plaçant la lamelle sur la lame de microscope.
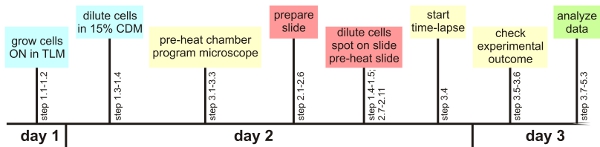
Figure 1: Vue d'ensemble expérimental

Figure 2: Préparation de l'échantillon de microscope
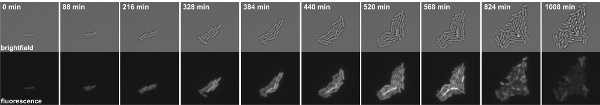
Figure 3: Time-lapse de microscopie par fluorescence de B. cellules subtilis abritant un P-GFP kinB fusion. Les instantanés sont prises à partir Movie 1. Panneaux Haut: champ lumineux, panneaux du bas: canal GFP.
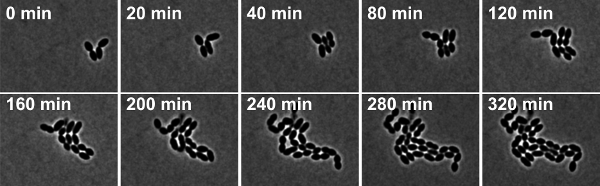
FigUre 4: Time-lapse phase de microscopie en contraste de S. pneumoniae souche de type sauvage R6. Les instantanés sont prises à partir Movie 2.
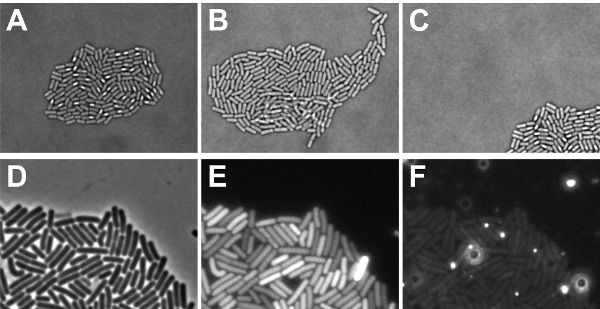
Figure 5: Illustration des résultats possibles de microscopie (time-lapse). AC indique les facteurs qui doivent être considérées pour les données obtenues avec les paramètres de la lumière diascopique. (A) Micrographie Brightfield d'une monocouche microcolonie (résultat positif) de la sporulation B. cellules subtilis (B) l'image en fond clair d'un B. subtilis microcolonie dans lequel certaines cellules se sont développées sur le dessus de l'autre (résultat négatif) (C) l'image en fond clair d'un B. sporulantes microcolonie subtilis qui a grandi hors du champ de la discussion (résultat négatif). Facteurs montrent DF qui doivent être considérées pour les données obtenues avec épiscopique réglages de lumière (D) d'image en contraste de phase de B. cellules en phase exponentielle subtilis représenté à visualiser où les signaux de fluorescence dans E et F proviennent de signaux GFP (e) des cellules montré dans la note D. que les signaux de fond sont similaires dans chaque pixel (résultat positif). Notez également que le temps d'exposition pourrait être trop car une cellule montre un signal saturé (résultat négatif) (F) Signaux obtenus par le canal rouge des cellules montré dans la note D. que le fond contient des zones avec des niveaux de fluorescence rouge augmenté (négatif résultat).
Film 1. Time-lapse de microscopie par fluorescence de B. cellules subtilis abritant un P-GFP kinB fusion. Instantanés ont été prises dans des intervalles de 8 min. A gauche: champ clair, à droite: GFP canal. Cliquez ici pour regarder le film.
Movie 2. Time-lapse en contraste de phase de microscopie de S. pneumoniae souche de type sauvage R6. Instantanés ont été prises dans des intervalles de 10 min. Cliquez ici pour regarder le film.
Discussion
Contrairement à beaucoup d'autres techniques de cellule unique, la méthode time-lapse de microscopie par fluorescence décrites ici peuvent être utilisés pour suivre l'histoire d'une cellule spécifique à l'égard de ses ancêtres, son comportement, et les événements de division. En combinaison avec des promoteurs cibles marquées par fluorescence ou de protéines, spécifiques activation de la voie de développement peuvent être suivies dans le temps et la localisation des protéines ainsi que la dynamique des protéines peuvent être surveillés pendant le développement bactérien.
Comme indiqué plus haut, les études se concentrant sur différentes espèces bactériennes peuvent être réalisées en adaptant les conditions de croissance selon les exigences pour une bactérie spécifique. Les seules limites que nous avons rencontrées sont liées aux conditions de croissance et taille de l'échantillon. En raison d'un environnement hermétique, des conditions de milieu ne peut pas être changé pendant l'expérience. Par ailleurs, un maximum de quatre souches par expérience peut être contrôlé efficacement.
Considérant quelques critiques, la méthode seule cellule d'analyse décrites ici peuvent facilement être appliqués en utilisant un microscope automatisé. Dans la suite, un aperçu de ces étapes critiques sera donnée. Des informations détaillées peuvent être trouvées dans le texte principal la préparation générale:. Il est sage de vérifier les réglages autofocus requis pour une bactérie spécifique avant l'expérience. De même, environ paramètres optimaux pour la visualisation de la fluorescence devrait être déterminé à l'avance, si possible. Par ailleurs, suite à un prêt de temps en ligne permet d'avoir tout le matériel prêt à l'emploi dans le temps (pré-réchauffement de la chambre du microscope, la programmation des réglages du microscope, la préparation de la lame, une heure avant les cellules sont en phase de croissance désiré, voir figure 1) . Croissance de B. subtilis dans le TLM et MDP: TLM et le MDP sont chimiquement défini les médias dans lesquels la famine B. subtilis ne pousse que lentement. La période de temps dans lequel les cellules sont cultivées dans les médias pourrait être prolongée en fonction de la souche spécifique. La croissance lente empêche les cellules de s'accumuler sur l'autre Préparation de l'échantillon de microscope:. Bulles d'air entre le cadre de gènes, la lame de verre et de la lamelle doit être empêché pour éviter le dessèchement vaste du milieu à base d'agarose. La même chose vaut pour l'interface de glissement moyen / couverture. Il est crucial de laisser les cellules assez sec, pour éviter la baignade et / ou de croissance de couches multiples Time-lapse de microscopie par fluorescence:. Pré-réchauffement de la lame ainsi que la chambre de l'environnement est essentielle pour prévenir les problèmes majeurs autofocus. Les cellules doivent être choisis au milieu d'un tampon d'agar, puisque ceux-ci ont le plus de chance de rester dans le champ et de se concentrer pendant l'expérience (fourni l'échantillon a été séché assez bien). Un maximum de 10 emplacements par des expériences fonctionne toujours correctement. Après avoir sélectionné la première cellule d'intérêt à utiliser le logiciel pour ajuster la mise au point (voir texte pour plus de détails). Vérifiez si les cellules sont encore en mise au point pendant les premières heures de l'expérience dans des intervalles de 30 min. Analyse: Il est important de vérifier avant de procédures d'analyse étendue si le contexte du milieu a les mêmes valeurs dans les canaux de fluorescence. Petites particules de poussière, les composantes moyennes, les verres sales ou minuscules touffes d'agarose peuvent contribuer à la fluorescence localement accrue, ce qui rend le film difficile, voire impossible d'analyser Dépannage:. Si les cellules se développent au-dessus de l'autre, cela pourrait indiquer soit la lamelle a été attachée trop tôt ou que le support n'est pas adapté à la croissance de monocouches microcolonie. Si les cellules d'intérêt sans cesse mourir prématurément, tandis que d'autres cellules sur la lame divisent heureusement, vous pouvez vérifier si vous mettez le filtre UV en position. Il pourrait également aider à diminuer le temps d'exposition ou l'intensité lumineuse au cours des expériences de long.
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Le travail dans le groupe de JWV est soutenu par une bourse Marie Curie de l'UE de réintégration, une subvention Sysmo2 (NWO-ALW/ERASysBio), un horizon (ZonMw) de subvention et par une bourse VENI (NWO-ALW). Le groupe d'OPK est soutenu par des subventions de plusieurs STW (NWO), un SYSMO1 (IGdeJ) et SYSMO2 subvention, une ESF Eurocores biologie synthétique subvention (SynMod) et par le Centre de génomique Kluyver de la fermentation industrielle et l'Institut supérieur pour l'alimentation et la nutrition.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | Commentaires (optionnel) |
|---|---|---|---|
| Gene Cadre | ABgene | AB-0578 | 1,7 x 2,8 cm |
| haute résolution basse température de fusion d'agarose | Sigma | A4718 | |
| lamelle grands | plusieurs | 24 x 50 mm | |
| si désiré, une membrane de teinture, par exemple FM 5-95 | Invitrogen | T23360 | colorants autre membrane sont également disponibles: http://probes.invitrogen.com/media/pis/mp34653.pdf |
| Time-lapse microscope avec chambre environnementale | plusieurs | voir les détails de notre dispositif dans les sections correspondantes |
Références
- Veening, J. W., Smits, W. K., Kuipers, O. P. Bistability, epigenetics, and bet-hedging in bacteria. Annu. Rev Microbiol. 62, 193-193 (2008).
- Dubnau, D., Losick, R. Bistability in bacteria. Mol Microbiol. 61, 564-564 (2006).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nat. Rev. Microbiol. 7 (5), 383-383 (2009).
- Veening, J. W. Bet-hedging and epigenetic inheritance in bacterial cell development. Proc. Natl. Acad. Sci. U. S. A. 105 (11), 4393-4393 (2008).
- de Jong, I. G., Veening, J. W., Kuipers, O. P. Heterochronic phosphorelay gene expression as a source of heterogeneity in Bacillus subtilis spore formation. J. Bacteriol. 192 (8), 2053-2053 (2010).
- Veening, J. W., Murray, H., Errington, J. A mechanism for cell cycle regulation of sporulation initiation in Bacillus subtilis. Genes Dev. 23 (16), 1959-1959 (2009).
- Eberhardt, A. Cellular localization of choline-utilization proteins in Streptococcus pneumoniae using novel fluorescent reporter systems. Mol Microbiol. 74 (2), 395-395 (2009).
- Vasantha, N., Freese, E. Enzyme changes during Bacillus subtilis sporulation caused by deprivation of guanine nucleotides. J Bacteriol. 144 (3), 1119-1119 (1980).
- Stewart, E. J. Aging and death in an organism that reproduces by morphologically symmetric division. PLoS. Biol. 3, 45-45 (2005).
- Rosenfeld, N. Gene regulation at the single-cell level. Science. 307 (5717), 1962-1962 (2005).
- Guberman, J. M. PSICIC: noise and asymmetry in bacterial division revealed by computational image analysis at sub-pixel resolution. PLoS. Comput. Biol. 4 (11), 1000233-1000233 (2008).
- Montero, L. l. o. p. i. s. . P. Spatial organization of the flow of genetic information in bacteria. Nature. 466, 77-77 (2010).
- Avery, O. T., Macleod, C. M., McCarty, M. Studies on the chemical nature of the substance inducing transformation of pneumococcal types: Induction of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III. J Exp. Med. 79, 137-137 (1944).
- Hoskins, . Genome of the bacterium Streptococcus pneumoniae strain R6. J Bacteriol. 183, 5709-5709 (2001).
- Martin, B. The recA gene of Streptococcus pneumoniae is part of a competence-induced operon and controls lysogenic induction. Mol Microbiol. 15, 367-367 (1995).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon