Method Article
Schéma de flux de travail d'une journée pour la détection de pathogènes et des bactéries tests de résistance antimicrobienne à partir d'hémocultures
Dans cet article
Résumé
La conception d'un système de flux de travail d'une journée simple pour le diagnostique des pathogènes bactériens permet la reconnaissance rapide des infections du sang. L'inclusion de huit cliniquement pertinentes cibles bactériennes et leurs profils de résistance aux antibiotiques offre au clinicien un premier aperçu sur le même jour, ce qui peut conduire à un traitement plus adéquat.
Résumé
Les bactériémies sont associées à des taux élevés de mortalité en raison de la manifestation probable de sepsis, sepsis sévère et du choc septique 1. Par conséquent, l'administration rapide d'une antibiothérapie adéquate est de première importance dans le traitement des infections sanguines. L'élément essentiel dans ce processus est le timing, qui dépend fortement sur les résultats de l'identification bactérienne et d'antibiogramme. Ces deux paramètres sont couramment obtenus par la culture basée sur des tests, ce qui prend beaucoup de temps et prend en moyenne 24-48 heures 2, 4. L'objectif de l'étude était de développer basées sur l'ADN des essais pour l'identification rapide des infections sanguines, ainsi que les tests rapides de sensibilité aux antimicrobiens. Le premier essai est un 16S ADNr eubactériennes-temps réel basé sur la PCR complétées par espèce ou du genre des sondes spécifiques 5. L'utilisation de ces sondes, bactéries à Gram négatif, y compris Pseudomonas spp., Pseudomonas aeruginosaet Escherichia coli, ainsi que les bactéries Gram-positives, y compris Staphylococcus spp., Staphylococcus aureus, Enterococcus spp., Streptococcus spp., et Streptococcus pneumoniae peuvent être distinguées. L'utilisation de ce test multisonde, une première identification de l'agent causal micro-organisme a été donné après 2 h.
Deuxièmement, nous avons développé un test moléculaire pour semi-tests de sensibilité aux antibiotiques de S. aureus, Enterococcus spp. et (facultatif) aérobies Gram-négatifs 6. Cet essai a été basée sur une étude dans laquelle la PCR a été utilisée pour mesurer la croissance des bactéries 7. Les bactéries récoltées directement à partir d'hémocultures sont incubées pendant 6 h avec une sélection d'antibiotiques, et suite à une SYBR Green-temps réel basé sur la PCR détermine l'inhibition de la croissance. La combinaison de ces deux méthodes pourraient orienter le choix d'une thérapie appropriée aux antibiotiques, le même jour (figure 1). En conclusion, L'analyse moléculaire de l 'identification et la sensibilité aux antibiotiques offre une alternative plus rapide pour la détection des agents pathogènes et pourrait améliorer le diagnostic des septicémies.
Protocole
PARTIE I: identification des agents pathogènes
1. Préparation de l'échantillon
Note: Le flux de production moléculaire est décrite dans le protocole suivant doit être effectuée conformément aux recommandations pour l'assurance qualité dans le diagnostic moléculaire 3.
- Ajouter une fraction aliquote de 0,1 ml de culture de sang à 0,9 ml de NaCl 0,9% dans un tube réactionnel de 1,5 ml à devenir un échantillon dilué 1:10. (Diluer afin d'empêcher l'inhibition qPCR).
- Centrifuger l'échantillon à 13 400 xg pendant 5 min pour obtenir un culot de l'ADN bactérien.
- Remettre en suspension le culot bactérien dans 100 ul stérile déminéralisée H 2 O.
- Conserver l'échantillon d'ADN à 4 ° C jusqu'à l'utilisation.
2. Essai d'identification: en temps réel de PCR ADNr 16S
- Préparer les mélanges de réaction comme suit. Le dosage se compose de quatre réactions séparées par échantillon. Chaque mélange comprend 12,50 maître ulmélange, 0,9 uM Amorce (5-TCCTACGGGAGGCAGCAGT-3) 7, amorce 0,6 uM inverse (5-GGACTACCAGGGTATCTAATCCTGTT-3) 8 et un panel de sondes. La quantité de sondes est donnée pour chacune des quatre réactions séparées dans ci-dessous.
- La première réaction comprend:
- 0,2 um sonde universelle (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1) 8
- 0,2 p uM sonde aeruginosa (5-JOE-CCAAAACTACTGAGCTAGAGTACG-3-BHQ1)
- La seconde réaction comprend:
- 0,2 E. uM sonde coli (5-JOE-GGAGTAAAGTTAATACCTTTGCTCATT-3-BHQ1)
- 0,2 um Pseudomonas spp. la sonde (5-NED-CCTTCCTCCCAACTTAAAGTGCTT-3-MGBNFQ)
- La troisième réaction comprend:
- 0,2 um Staphylococcus spp. la sonde (5-NED-AATCTTCCGCAATGGGCGAAAGC-3-MGBNFQ)
- 0,2 uM S. sonde aureus (5-FAM-AGATGTGCACAGTTACTTACACATAT-3-BHQ1)
- 0,2 um Enterococcus spp. (5-JOE-TCCTTGTTCTTCTCTAACAACAGAG-3-BHQ1)
- La quatrième réaction comprend:
- 0,2 um sonde universelle (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1)
- 0,3 pM Streptococcus spp. la sonde (5-NED-CCAGAAAGGGACSGCTAACT-3-MGBNFQ)
- 0,2 uM S. pneumoniae sonde (5-JOE-CCAAAGCCTACTATGGTTAAGCCA-3-BHQ1)
- Ajouter stérile déminéralisée H 2 O pour atteindre un volume total de 20 pi. Ajouter 20 ul de chaque mélange réactionnel pour les puits d'une plaque de 96 puits PCR.
- Ajouter 5 ul d'échantillon dans chaque puits.
- Utilisez un film adhésif pour sceller la plaque de 96 puits PCR.
- Exécutez la plaque sur le ABI PRISM 7900HT Système temps réel PCR en utilisant les suivantes conditions optimales de cyclage thermique:
- Préchauffage à 50 ° C pendant 10 min
- Dénaturation initiale à 95 ° C pendant 15 min
- 42 cycles de
- Dénaturation à 95 ° C pendant 15 s
- Recuit à 60 ° C pendant 1 min
3. Analyse des résultats
Régler le seuil de l'analyse Ct à 0,1 dans les paramètres d'analyse de tabulation. Cibler les configurations de base pour démarrer (Cycle): 6 et Fin (cycle): 15.
- Enregistrez le cycle seuil (Ct) la valeur pour tous les échantillons. La valeur de coupure d'envisager un résultat PCR positif que peut être réglé à une valeur Ct de 35 ans. La quantité de bactéries présentes dans les cultures de sang variait de 10 7 à 10 11 UFC / ml, générant les valeurs Ct-dessous de 35.
PARTIE II: antibiogramme
4. Isolement de bactéries à partir d'hémocultures positives 9
- Aspirer 5 ml de bouillon à partir d'un flacon d'hémoculture positive et le transférer dans un tube séparateur de sérum.
- Centrifuger le tube séparateur de sérum à 2000 xg pendant 10 min.
- Éliminer le surnageant du tube de séparation du sérum.
- Transfert bactères de la couche de gel du tube avec un écouvillon stérile dans une solution saline à 0,9% jusqu'à 0,5 McFarland une norme de suspension est obtenue.
5. L'inoculation des plaques Objectives Micro
- Diluer la suspension à 0,5 McFarland en double concentré de bouillon de Mueller-Hinton II pour former une suspension de 5 x 10 5 UFC / ml.
- Ajouter cette suspension dans les puits d'une plaque de micro titre contenant une sélection des antibiotiques (tableau 1).
- Incuber la plaque de titrage micro à 37 ° C pendant 6 h.
- Conserver une partie aliquote de la suspension à 4 ° C (comme régulation de croissance négative).
- Après 6 h d'incubation, transférer le contenu de chaque puits dans un tube stérile, ainsi que l'échantillon de croissance négatif de contrôle qui a été stocké à 4 ° C.
- Centrifuger les tubes à 16000 xg pendant 5 min.
- Retirez délicatement le surnageant, sans perturber le culot bactérien.
- Reprendre le culot dans H déminéralisée stérile 2 O.
- Diluer les échantillons de 10 fois stérile déminéralisée H 2 O.
6. En temps réel de PCR ADNr 16S 10
- Préparer le mélange de PCR comme suit:
- 12,50 pi iQ SYBR Green Supermix
- 0,5 pM avant amorce 16S-1 (5-TGGAGAGTTTGATCCTGGCTCAG-3) 11
- 0,25 um 16S-2 amorce inverse (5-TACCGCGGCTGCTGGCAC-3) 11
- stérile déminéralisée H 2 O à un volume total de 20 pi
- Ajouter 20 ul du mélange PCR pour les puits d'une plaque de 96 puits PCR.
- Ajouter 5 ul d'échantillon dans chaque puits.
- Utilisez un film adhésif pour sceller la plaque de 96 puits PCR.
- Exécutez la plaque sur le MyiQ unique couleur en temps réel système de détection par PCR, en utilisant les suivantes conditions optimales de cyclage thermique:
- Dénaturation initiale à 95 ° C pendant 4 min
- Initiale de recuit à 65 ° C pendant 30 s
- 35 cycles de
- Dénaturation à 95 ° C pendant 15 s
- Recuit à 60 ° C pendant 1 min
- Faire fondre analyse de la courbe (à partir de 60-95 ° C en 20 minutes avec des incréments de 0,57 ° C)
7. Analyse des résultats
- Calculer la valeur de coupure Ct en utilisant l'une des formules suivantes (selon le type d'antibiotique).
- En général:
Cut-off valeur Ct = valeur de Ct la croissance de contrôle positif + 0,5 x (valeur de Ct à contrôler la croissance négative - la valeur Ct de croissance de contrôle positif) - La pipéracilline, pipéracilline / tazobactam et la ceftazidime dans bacilles Gram-négatifs; Amoxicilline, oxacilline et le triméthoprime / sulfaméthoxazole dans S.aureus; Amoxicilline chez Enterococcus spp:. Cut-off valeur Ct = valeur de Ct la croissance de contrôle positif + 0,25 x (croissance Ct valeur négative de contrôle - la valeur Ct de croissance de contrôle positif)
- En général:
- Utilisez l'échantillon incubé avec stérile déminéralisée H 2 O comme régulation de croissance positive.
- Selon le micro-organisme, utilisez le contrôle de la croissance négative appropriée:
- Gram-négatif échantillon tiges incubées avec un mélange d'antibiotiques
- Enterococcus spp. Échantillon stocké à 4 ° C
- Exemple de S.aureus conservés à 4 ° C
- Déterminer la sensibilité (S) ou de la résistance (R) de la souche de l'antibiotique effectué comme suit:
- Une valeur Ct supérieur à la valeur de coupure Ct indique la sensibilité
- Une valeur Ct inférieure à la valeur de coupure Ct indique la résistance
8. Les résultats représentatifs
Deux organismes modèles, à savoir un E. Gram négatif coli et un S. Gram-positif Staphylococcus, sont choisis de visualiser la procédure combiné pour la détection et l'identification de bactéries pathogènes et la détermination de son profil antimicrobien. La première partie du protocole comprend l'identification des agents pathogènes. Spesondes spécifiques sont conçus pour la détection de micro-organismes huit cliniquement pertinentes. En présence d'une cible inclus dans le panneau bactérienne, les courbes d'amplification sont générés et les valeurs de Ct sont calculés (Figure 2). La valeur de coupure d'envisager un résultat positif de PCR comme est fixé à une valeur Ct de 35 ans. Figure 2A, le profil d'identification d'une culture de sang E. coli-infectée est représenté. La sonde 16S universel est inclus dans deux mélanges réactionnels distincts et génère par conséquent deux courbes d'amplification (Ct de 25,20 et 25,95). Le troisième signal est dérivé de la sonde spécifique pour E. coli (Ct de 27,04). L'identification d'un S. Staphylococcus infecté par hémoculture est montré dans la figure 2B. La sonde 16S universel comporte des signaux d'amplification de 33,35 et 33,71. Les deux signaux restants sont dérivées à partir des sondes spécifiques pour Staphylococcus spp. et S. aureus (Ct de 32,48 et 30,59).
Après la première partie du protocole, le micro-organisme pathogène est connu et le profil antimicrobien peut être déterminée. La figure 3 est un exemple d'un complot d'amplification tests de sensibilité aux antibiotiques, ce qui représente la souche E. coli qui a été également montré la figure 2A. Chaque ligne représente un antibiotique que l'échantillon bactérien a été incubé avec. Un échantillon avec une faible valeur Ct est un échantillon dans lequel la croissance a lieu en présence d'un antibiotique, une résistance à l'antibiotique indiquant testée. Au contraire, une valeur élevée Ct représente un échantillon dans lequel aucune croissance s'est produite en raison du fonctionnement efficace de l'antibiotique, la sensibilité à l'antibiotique indiquant testée. Le tableau 1 illustre la détermination du profil de l'antimicrobien E. coli et S. aureus. Toutes les valeurs Ct sont signalés et, en utilisant les formules mentionnées dans le texte du protocole (7.1), deux seuils sont calculés les valeurs de Ctréglementée à la distinction entre résistance et la sensibilité. La souche est résistante à l'antibiotique si la valeur déclarée Ct est inférieure à la calcule la valeur seuil Ct (et vice versa).
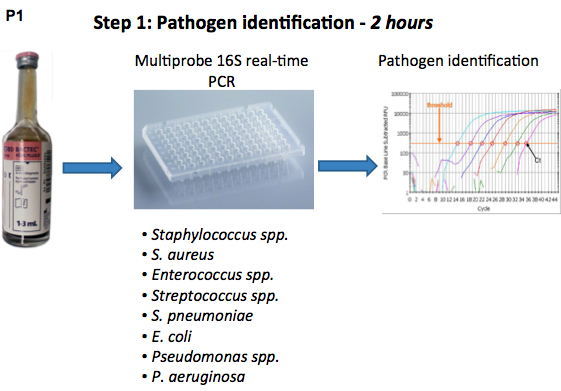
Diagramme Figure 1. De l'identification des agents pathogènes et d'antibiotiques procédure des tests de sensibilité à l'aide en temps réel ADNr 16S par PCR.
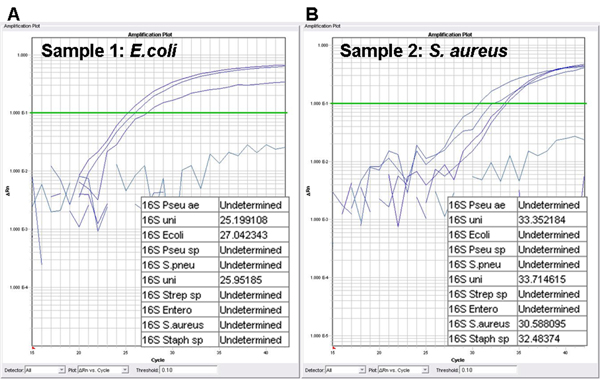
Figure 2 test d'identification:. Parcelles d'amplification et des valeurs seuils de cycle (les valeurs de Ct) Une hémoculture positive est détectée par le sonde universelle ADNr 16S, tandis que les sondes spécifiques sont utilisés pour l'identification de l'agent pathogène.. Parcelle d'amplification A. de la culture du sang contenant E. coli; parcelles d'amplification de la culture B. sang contenant S. aureus; Pseudomonas ae, Pseudomonaeruginosa; uni, 16S sonde universelle; Ecoli, Escherichia coli sonde; Pseudomonas sp, Pseudomonas spp. sonde; S. pneumoniae, Streptococcus pneumoniae sonde; Strep sp, Streptococcus spp. sonde; Entero, Enterococcus spp. sonde; S. aureus, Staphylococcus aureus sonde; Staph sp, Staphylococcus spp. Sonde.
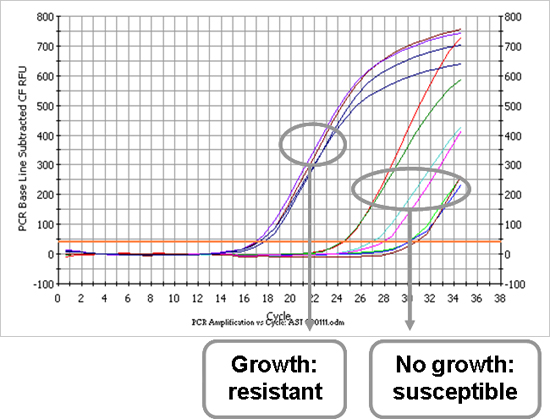
Amplification parcelle Figure 3. Des tests de sensibilité aux antibiotiques d'un E. coli isoler (échantillon 1). Chaque courbe représente un antibiotique que la souche a été incubé avec. Un signal de début est provoquée par une charge bactérienne élevée, ce qui signifie que la souche a cultivé en présence de l'antibiotique testé et n'est donc résistant à l'antibiotique. Signaux en retard indiquent que la souche a pas cultivées en présence de l'antibiotique, en d'autres termes, il est sensible.
| Exemple 1: E. coli | Exemple 2: S. Staphylococcus | ||||
| AST | Ct | R / S | AST | Ct | R / S |
| L'amoxicilline 8 mg / L | 16,83 | R | Amoxicilline 0,25 mg / L | 21,03 | R |
| L'amoxicilline-acide clavulanique 8/4 mg / L | 17,36 | R | Oxacilline 2 mg / L | 25,80 | S |
| Pipéracilline 16 mg / L | 16,67 | R | La vancomycine 2 mg / L | 25,20 | S |
| Pipéracilline-tazobactam 16 /4 mg / l | 24,15 | S | Gentamicine 4 mg / L | 25,86 | S |
| La ciprofloxacine 1 mg / L | 29,72 | S | Le triméthoprime-sulfaméthoxazole 2/38 mg / L | 24,62 | S |
| Ceftazidime 1 mg / L | 24,03 | S | |||
| Ceftazidime 8 mg / L | 26,58 | S | |||
| Gentamicine 4 mg / L | 29,83 | S | |||
| Le triméthoprime-sulfaméthoxazole 2/38 mg / L | 27,60 | S | |||
| Contrôle de la croissance négative (mélange d'antibiotiques) | 30,41 | Contrôle de la croissance négative (échantillon stocké à 4 ° C) | 27,42 | ||
| Contrôle de la croissance positive | 16,90 | Contrôle de la croissance positive | 20,22 | ||
| Cut-off valeur Ct 1 * | 21,76 | Cut-off valeur Ct 1 *** | 23,82 | ||
| Cut-off valeur Ct 2 ** | 18,75 | Cut-off valeur Ct 2 **** | 22,02 | ||
| * Pour l'amoxicilline, l'amoxicilline-acide clavulanique, la ciprofloxacine, gentamicdans, le triméthoprime-sulfaméthoxazole ** Pour pipéracilline, pipéracilline-tazobactam, la ceftazidime | *** Pour la vancomycine et la gentamicine Pour l'amoxicilline ****, oxacilline et le triméthoprime-sulfaméthoxazole |
Tableau 1. Détermination des tests de sensibilité aux antibiotiques des deux échantillons (E. coli et S. aureus). Les valeurs de Ct de la PCR-test ont été copiés dans ce fichier Excel, que ce qui peut calcule automatiquement les deux cut-off aleurs Ct de la régulation de croissance positive et négative, en utilisant les formules indiquées dans le texte du protocole. Si un antibiotique montre une valeur Ct inférieure à la valeur de coupure Ct, la souche est résistante à l'antibiotique, si la valeur Ct est plus élevé que le seuil, la souche est sensible.
Discussion
Le protocole décrit ici permet l'identification rapide des agents pathogènes et fournit un profil fonctionnel antimicrobien qui pourrait conduire à l'administration précoce d'antibiotiques adéquats, améliorant ainsi le pronostic des patients atteints d'infections sanguines. En fonction des conditions demandées d'un test, à savoir faible coût, un débit élevé, de demi-tour minimale de temps, les conditions de test peut être ajustée. Toute la procédure peut être effectuée dans un jour ouvrable. En outre, les deux parties du protocole peut être effectuée simultanément, ce qui réduit le temps de demi-tour sensiblement. Tel que présenté ici, le panneau d'identification est une sélection des bactéries les plus cliniquement pertinente dans notre hôpital. Depuis le principe est le ciblage de la région du gène 16S, des sondes spécifiques pour d'autres microorganismes peuvent être conçus et ajoutés à l'essai. Le test complet a été à l'origine destiné à l'analyse rapide des cultures de sang, mais peut aussi être utilisé pour le traitement des odes échantillons de matériaux Ther. C'est aussi le cas pour les antibiotiques qui ont été utilisées pour les tests de sensibilité aux antibiotiques: les antibiotiques plus ou autre peut être ajoutée, basée sur les profils de résistance locaux et des lignes directrices.
Déclarations de divulgation
Nous n'avons rien à communiquer.
Remerciements
Ce travail a été soutenu par l'AZM Profileringsfonds (PF245).
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| Chlorure de sodium (NaCl) | Merck Chemicals | 106404 | 0,9% dans l'eau |
| Vacutainer SST Tube séparateur de sérum 5 ml | BD Diagnostic Systems | 367986 | |
| Mueller-Hinton II bouillon | BD Diagnostic Dystems | 212322 | 44 g / L dans l'eau |
| Centrifuger Rotixa 50 RS | Andreas Hettich GmbH & Co. KG | 4910 | |
| Centrifuger 5415 D | Eppendorf | Supprimé | |
| Primaires | Sigma-Aldrich | na | |
| Sondes | Sigma-Aldrich/Applied Biosystems | na | |
| TaqManEnvironmental master mix 2.0 | Applied Biosystems | 4396838 | |
| iQ SybrGreen Supermix | Bio-Rad Laboratories BV | 170-8880 | |
| MicroAmp optique Plaque de réaction de 96 puits | Applied Biosystems | N8010560 | |
| MicroAmp Film adhésif optique | Applied Biosystems | 4311971 | |
| iQ plaques à 96 puits PCR | Bio-Rad Laboratories BV | 223-9441 | |
| Microseal B joints adhésifs | Bio-Rad travailatories BV | MSB-1001 | |
| En temps réel système de détection par PCR | Applied Biosystems | ABI PRISM 7900HT | |
| Real-Time PCR Detection System | Bio-Rad Laboratories BV | MyiQ unique couleur |
Références
- Wallet, F. Preliminary clinical study using a multiplex real-time PCR test for the detection of bacterial and fungal DNA directly in blood. Clin. Microbiol. Infect. 16, 774 (2010).
- Beekmann, S. E., Diekema, D. J., Chapin, K. C., Doern, G. V. Effects of rapid detection of bloodstream infections on length of hospitalization and hospital charges. J. Clin. Microbiol. 41, 3119 (2003).
- Raymaekers, M., Bakkus, M., Boone, E., de Rijke, B., Housni, H. E. l., Descheemaeker, P., De Schouwer, P., Franke, S., Hillen, F., Nollet, F., Soetens, O., Vankeerberghen, A. Molecular Diagnostics working group. Reflections and proposals to assure quality in molecular diagnostics. Acta. Clin. Belg. 66, 33 (2011).
- Peters, R. P. New developments in the diagnosis of bloodstream infections. Lancet Infect. Dis. 4, 751 (2004).
- Hansen, W. L., Beuving, J., Bruggeman, C. A., Wolffs, P. F. Molecular probes for the diagnosis of clinically relevant bacterial infections in blood cultures. J. Clin. Microbiol. 48, 4432-4432 (2010).
- Beuving, J. Antibiotic susceptibility testing of grown blood cultures by combining culture and real-time polymerase chain reaction is rapid and effective. PLoS ONE. 6, (2011).
- Rolain, J. M., Mallet, M. N., Fournier, P. E., Raoult, D. Real-time PCR for universal antibiotic susceptibility testing. J. Antimicrob. Chemother. 54, 538 (2004).
- Nadkarni, M. A., Martin, F. E., Jacques, N. A., Hunter, N. Determination of bacterial load by real-time PCR using a broad-range (universal) probe and primers set. Microbiol. 148, 257 (2002).
- Waites, K. B., Brookings, E. S., Moser, S. A., Zimmer, B. L. Direct susceptibility testing with positive BacT/Alert blood cultures by using MicroScan overnight and rapid panels. J. Clin. Microbiol. 36, 2052 ( ).
- Vliegen, I. Rapid identification of bacteria by real-time amplification and sequencing of the 16S rRNA gene. J. Microbiol. Meth. 66, 156 (2006).
- Hall, L., Doerr, K. A., Wohlfiel, S. L., Roberts, G. D. Evaluation of the MicroSeq system for identification of mycobacteria by 16S ribosomal DNA sequencing and its integration into a routine clinical mycobacteriology laboratory. J. Clin. Microbiol. 41, 1447 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon